このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
蛍光寿命イメージング顕微鏡を用いたがん細胞の代謝と粘度の探索
要約
ここでは、蛍光寿命イメージング顕微鏡(FLIM)を使用して、生きたがん細胞培養における細胞代謝と原形質膜粘度を順次イメージングする方法を示します。代謝評価は、内因性蛍光を検出することによって行われます。粘度は、蛍光分子ローターを使用して測定されます。
要約
粘度は、生細胞の形態学的および生理学的状態を調節するための重要なパラメータの1つであるため、生体膜の重要な物理的特性です。腫瘍細胞の原形質膜は、その組成、構造、および機能特性に有意な変化があることが知られています。グルコースと脂質の代謝の調節不全に加えて、これらの特定の膜特性は、腫瘍細胞が敵対的な微小環境に適応し、薬物療法に対する耐性を発達させるのを助けます。ここでは、蛍光寿命イメージング顕微鏡(FLIM)を使用して、生きたがん細胞培養における細胞代謝と原形質膜粘度を順次イメージングする方法を示します。代謝評価は、還元型ニコチンアミドアデニンジヌクレオチドNAD(P)Hや酸化フラビンなどの内因性代謝補因子の蛍光を検出することによって行われます。粘度は、合成粘度感受性色素である蛍光分子ローターを使用して測定され、蛍光寿命は身近な環境の粘度に強く依存します。これらの技術を組み合わせることで、がん細胞の膜状態と代謝プロファイルとの関連をより深く理解し、化学療法によって誘発される変化を視覚化することができます。
概要
細胞の悪性形質転換は、それらの形態学的および生理学的状態における複数の変化を伴う。がん細胞の急速かつ制御不能な増殖には、エネルギー生産と生合成に関与する生化学的経路の根本的な再編成が必要です。がん代謝の特徴的な特徴は、正常な酸素濃度の下でも解糖速度の向上(ワールブルグ効果)、代替燃料としてのアミノ酸、脂肪酸、乳酸の使用、高い抗酸化レベルの存在下での高ROS産生、および脂肪酸の生合成の増加です1,2.がん細胞の代謝は非常に柔軟性があり、それによりがん細胞が不利で不均一な環境に適応することができ、生存率がさらに向上することが現在では認められています3。
代謝の変化は、腫瘍細胞の膜の特定の組織と組成をサポートします。がん細胞の原形質膜の脂質プロファイルは、非がん細胞とは定量的に異なります。リピドームの主な変化は、ホスファチジルイノシトール、ホスファチジルセリン、ホスファチジルエタノールアミン、ホスファチジルコリンなどのリン脂質のレベルの増加、スフィンゴミエリンのレベルの低下、コレステロールの量の増加、脂肪酸の不飽和度の低下です4,5,6.したがって、膜の粘度、流動性の逆など、膜の物理的特性は必然的に変化します。粘度は生体膜の透過性を決定し、膜結合タンパク質(酵素、トランスポーター、受容体)の活性を制御するため、その恒常性調節は細胞機能に不可欠です。同時に、膜脂質プロファイルの調整による粘度の修飾は、細胞の遊走/浸潤や条件付き変化による生存にとって重要です。
蛍光寿命イメージング顕微鏡(FLIM)は、内因性蛍光プローブまたは外因性プローブ7を使用して、生細胞内の複数のパラメーターを非侵襲的に評価するための強力なアプローチとして登場しました。FLIMは、一般的に多光子レーザー走査型顕微鏡で実現され、(サブ)セル分解能を提供します。TCSPC(Time-Correlated Single-Photon Counting)モジュールを搭載しているため、蛍光の時間分解測定を高精度に行うことができます8。
FLIMによる細胞代謝のプローブは、デヒドロゲナーゼの内因性補因子、還元型ニコチンアミドアデニンジヌクレオチド(リン酸)NAD(P)H、および酸化フラビン-フラビンアデニンジヌクレオチドFADおよびフラビンモノヌクレオチドFMNの蛍光測定に基づいており、これらは多くの生化学反応で電子キャリアとして機能します7,9,10.NAD(P)Hの検出された蛍光は、NADHとそのリン酸化型であるNADPHからのものであり、スペクトル的にはほぼ同一です。通常、NAD(P)Hとフラビンの蛍光減衰は双指数関数に適合します。NAD(P)Hの場合、第1の成分(~0.3-0.5 ns、~70%-80%)は解糖系に関連する遊離状態に起因し、第2の成分(~1.2-2.5 ns、~20%-30%)はミトコンドリア呼吸に関連するタンパク質結合状態に起因します。フラビンの場合、短い成分(~0.3-0.4 ns、~75%-85%)をFADの消光状態に、長い成分(~2.5-2.8 ns、~15%-25%)を消光されていないFAD、FMN、およびリボフラビンに割り当てることができます。解糖、グルタミン分解、酸化的リン酸化、および脂肪酸合成の相対レベルの変化は、補因子の短寿命および長期寿命画分の変化をもたらします。また、これらの蛍光色素の蛍光強度比(酸化還元比)は、細胞の酸化還元状態を反映しており、代謝指標としても利用されています。酸化還元比は、蛍光寿命と比較して単純な指標を示しますが、データ取得の点では、蛍光寿命は蛍光色素の本質的な特性であり、特に組織における励起電力、光退色、集束、光散乱および吸収などの要因にほとんど影響されないため、FLIMはNAD(P)HおよびFADを推定するのに有利です。 放出強度とは異なります。
微生物レベルで生細胞および組織中の粘度をマッピングする便利な方法の1つは、蛍光パラメータが局所粘度11,12に強く依存する小さな合成粘度感受性色素である蛍光分子ローターの使用に基づくものである。粘性媒体では、分子内のねじれまたは回転が遅くなるため、ローターの蛍光寿命が長くなります。分子ローターの中で、ホウ素ジピロメテン(BODIPY)の誘導体は、生理学的粘度範囲での蛍光寿命の良好なダイナミックレンジ、温度依存性、単指数関数的な蛍光減衰により、簡単なデータ解釈、十分な水溶性、および低い細胞毒性13,14で蛍光寿命の良好なダイナミックレンジを備えているため、生体システムの粘度を検出するのに適しています。.BODIPYベースのローターとFLIMを使用した微小粘度の定量的評価は、以前にin vitroの癌細胞、多細胞腫瘍スフェロイド、およびin vivoのマウス腫瘍で実証されています15,16。
ここでは、FLIMによるin vitro でのがん細胞の細胞代謝と原形質膜粘度の研究のためのシーケンシャルプロービング方法論について詳しく説明します。比較的弱い内因性蛍光がBODIPYベースのローターの蛍光による汚染を避けるために、NAD(P)HとFADの蛍光を最初にイメージングして、同じ細胞層のイメージングを順次実行します。補因子の蛍光寿命は細胞質で測定され、ローターの蛍光寿命は、関心領域として対応するゾーンを手動で選択することにより、細胞の原形質膜で測定されます。このプロトコルは、さまざまながん細胞株の代謝状態と粘度を相関させ、化学療法後の変化を評価するために適用されました。
FLIMサンプル調製のプロトコルは、共焦点蛍光顕微鏡のプロトコルと変わりません。データを取得したら、主なタスクは生データから蛍光寿命を抽出することです。プロトコールの性能は、HCT116(ヒト結腸直腸癌)、CT26(マウス結腸癌)、HeLa(ヒト子宮頸癌)、およびhuFB(ヒト皮膚線維芽細胞)細胞を使用して実証されています。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. FLIMを実行するための最小限のセットアップの説明
- この実験を行うには、必要なセットアップが利用可能であることを確認してください:倒立共焦点顕微鏡、パルスレーザー、通常はpsまたはfs、同期信号、高速フォトンカウンティング検出器(時間応答150 ps)およびフォトンカウンティングエレクトロニクス、検出器とレーザーにそれぞれ使用可能な出力ポートと入力ポート、顕微鏡スキャンコントローラーからのスキャンクロックパルス、 レーザービームコンバイナーとFLIMに使用されるレーザーの波長に適したメインダイクロイックビームスプリッターを備えた顕微鏡のスキャンヘッド。
- FLIMに2光子励起を使用する場合は、顕微鏡にNDDポートが含まれていることを確認してください。
- 哺乳類細胞研究、特に長期実験では、CO2 インキュベーターを目的の温度に維持してください。
注:この実験で使用したシステムについては、 材料の表を参照してください。
2. 顕微鏡観察用細胞の調製
- 細胞をインキュベーターで37°C、5%CO2 、湿度の高い雰囲気で定期的に培養します。
- 顕微鏡検査では、細胞懸濁液を1 x 106 細胞/mLの濃度で完全な培養培地で調製します。
注:細胞濃度と培地条件は細胞に依存します。播種に使用する細胞の数とインキュベーション時間は、顕微鏡皿で70%〜80%のコンフルエンスが得られるように適合させる必要があります。 - 200 μLの自動ピペットを使用して、ガラス底の35 mm細胞培養皿(皿あたり10 μLに5 細胞×10 x)に細胞を播種します。
注:細胞の播種には格子状のガラス底皿を使用して、ダイナミクスの同じ微視的な視野で細胞を監視します。- 手動播種中は、ピペットの先端が皿の底や側面を傷つけないようにして、底の損傷を防ぎます。
- 皿をCO2 インキュベーター(37°C、5%CO2、湿潤雰囲気)に入れ、細胞を24時間インキュベートします。
- 24時間後、インキュベーターからディッシュを取り出し、光学顕微鏡で細胞の形態と合流を確認します。細胞が約80%のコンフルエント度に達しなかった場合は、さらに24時間インキュベートします。
- 1,000 μLの自動ピペットを使用して古い培地を皿から静かに取り出し、フェノールレッドを含まないDMEM培地(DMEM LifeまたはFluoroBriteなど)を2 mL加えます。
注:イメージングには、さまざまな培地を使用することができます。顕微鏡検査に細胞を使用するときは、培地中のフェノールレッドを避けてください。 - 細胞の適応を可能にするために、皿をインキュベーターに60〜120分間置きます。
3. 代謝補因子のFLIM
- 細胞(ステップ2.7から)を入れたガラス底皿を顕微鏡ステージに置きます。
- レーザー走査型顕微鏡ソフトウェア(例:ZEN - ZEISS Efficient Navigation)の [検索 ]タブをクリックし、 次に[透過灯(TL)] をクリックしてライトをオンにします。
- 接眼レンズを通して、正方形が細胞によって最大限に占められているセルの中央スライスレベル(倍率40倍)を観察することにより、サンプルの焦点面を見つけます。
- OFFボタンをクリックして、ライトをオフに切り替えます。
- [Acquisition] タブを開きます。内因性NAD(P)Hの透過率および自家蛍光強度画像を取得するには、次の設定を入力します:励起波長:2光子モード750 nm、登録範囲:450-490 nm、レーザー出力:5%(~6 mW)、画像サイズ:1024 x 1024ピクセル。
注:励起波長とレジストレーション範囲の選択は、NAD(P)H17のスペクトル特性(最大励起と最大放出)に基づいています。- 画像取得には、油浸対物レンズСプランアポクロマート40x/1.3 NAを使用します。
- スナップボタンを押して、画像をZEN形式で保存します。
- FADの透過率と自家蛍光強度の画像を取得するには、 励起波長 を900nmに変更します。 レジストレーション範囲を500-550 nm、 レーザー出力を9%(~6 mW)、 画像サイズを1024 x 1024ピクセルに設定します。
注:励起波長とレジストレーション範囲の選択は、FAD18のスペクトル特性(最大励起と最大放出)に基づいています。- 画像取得には、油浸対物レンズСプランアポクロマート40x/1.3 NAを使用します。
- スナップボタンを押して、画像をZEN形式で保存します。
- NAD(P)Hの場合、レーザー走査型顕微鏡ソフトウェアのステップ3.5で説明されているようにパラメータを設定します。 [画像サイズ ] を 256 x 256 ピクセルに変更します。
- SPCM(Single Photon Counting Modules)FLIMモジュールのオペレーティングソフトウェアのメニューに次のパラメータを入力します: 収集時間:60秒; TAC範囲:5.00E-8; CFDリミットロー:-29.41; ADC解像度:256、 画像サイズ:256 x256ピクセル。
- サンプルを60秒間スキャンし、スキャンを停止して、取得したNAD(P)HのFLIM画像を保存します。
- 取得したFLIMデータを確認します。このためには、画像ソフトウェアで生データ を開き 、細胞の細胞質のピクセルにカーソルを置いて選択し、このピクセルの蛍光減衰を分析します。ピクセル強度は、ビニング 1 での減衰曲線あたり ≥3,000 フォトンである必要があります。
注:光子の数が3,000未満の場合は、細胞の形態と光子計数率を制御しながら、レーザー出力または画像収集時間を増やします。通常、カウント率の低下が初期値の10%を超えると、光退色が起こります。 - FADの場合は、レーザー走査型顕微鏡のソフトウェアのステップ3.7で説明されているようにパラメータを設定します。画像サイズを 256 x 256 ピクセルに変更します。
- SPCM(Single Photon Counting Modules)のメニューに次のパラメータを入力しますFLIMモジュールのオペレーティングソフトウェア: 収集時間:60秒; TAC範囲:5.00E-8; CFDリミットロー:-29.41; ADC解像度:256、 画像サイズ:256 x256ピクセル。
- サンプルを60秒間スキャンします。スキャンを停止し、取得したFADのFLIM画像を保存します。
注意: 手順3.10および3.15で示されたパラメータは、使用する電子機器と検出器に固有のものです。 - 手順3.12の説明に従って、取得したデータを確認します。
- 手順3.5〜3.16を繰り返して、さまざまな視野からFLIM画像を記録します。
4. 蛍光分子ローターによる細胞の染色
注:細胞は、室温(~20°C)で洗浄せずに蛍光分子ローター溶液中でイメージングされ、ローターの内在化を遅らせます。膜の粘度は、以前の研究19,20で示されたように、温度に依存します。顕微鏡の温度制御ステージは、事前に、つまりローターを細胞に追加する前にオフにする必要があります。私たちのセットアップでは、ステージの冷却に約10分かかります。
- 蛍光分子ローターBODIPY 2(ストック1、25.7 mM)の一般的な原液を調製します。
- BODIPY 2を無菌環境で開き、正確な天びんを使用して約2mgの重量を量ります。マイクロ遠心チューブに慎重に入れます。
- 自動20 μLピペットを使用して、3 μLの適切な溶媒(DMSOなど)を追加します。
- サンプルがDMSOに完全に溶解したら、297 μLの滅菌PBSを加え、自動200 μLピペットを使用して完全に混合します。
注意: 原液は、暗い色のパッケージに入れて+ 4°Cの冷蔵庫に保管してください。再懸濁したら、冷蔵庫で数ヶ月間保存できます。
- ストック2(8.9 mM)を調製するには、25 μLの一般的なストック溶液(ストック1)を微量遠心チューブに加え、続いて48 μLの滅菌PBSを加えます。自動200μLピペットを使用して穏やかに混合します。
注:ストック2を使用して最終染色ストックを調製し、マイクロモル濃度が必要なため細胞染色に適用します。 - ディッシュ内の培地(ステップ3.1)をCa2+/Mg2+ を含まない氷冷ハンク溶液に静かに置き換え、細胞を+4°Cで3分間インキュベートします。
注:氷冷溶液を使用し、+4°Cでインキュベートすると、分子ローターの内部化が遅くなり、メンブレンの局所染色が20〜30分間持続します。 - 4.5 μMのBODIPY 2を含む最終染色溶液を調製するには、ストック2 1 μLを999 μLの氷冷ハンク溶液またはPBSに加えます。
注:最終染色溶液中のBODIPY 2の濃度は、細胞に毒性影響を与えることなく~10 μMまで増やすことができるため、より効率的な染色と収集される光子の数を増やすことができます。高濃度では、FLIM検出器の過負荷が発生する可能性があります。 - 細胞培養皿からハンク溶液を吸引し、BODIPY 2の氷冷した4.5μM溶液と交換します。細胞は、洗浄せずにBODIPY 2溶液でイメージングされます。
5. 細胞内蛍光分子ローターのFLIM
注:BODIPY 2の蛍光スペクトルは内因性補因子NAD(Р)HおよびFAD 12,17,18の発光と重なるため、代謝補因子のFLIMの後に常に蛍光分子ローターのFLIMを実行してください。
- 染色した細胞を入れた皿を顕微鏡ステージ(~20°C)に移し、イメージングします。
- レーザー走査型顕微鏡のソフトウェアで1光子モードの次のパラメータを設定します:アルゴンイオンレーザーによる488nmの波長での 励起 、 レーザー出力 1%-2%、 登録範囲500-550nm波長。
- 画像取得には、油浸対物レンズСプランアポクロマート40x/1.3 NAを使用します。
- ライブボタンを押します。スキャンを開始し、統合された電動ステージでXYとZの位置決めを行い、フォーカスを調整して、プレビューウィンドウで細胞の透過率と蛍光強度の画像を取得します。必要に応じて、取得した画像を保存します。
- オーバーラップした透過画像と蛍光画像をチェックして、ローターの蛍光が予想される場所(細胞の原形質膜)から来ているかどうかを確認します。
- FLIMモジュールのSPCMソフトウェアのメニューに次のパラメータを入力します: 収集時間:60秒; TAC範囲:5.00E-8; CFDリミットロー:-29.41; ADC解像度:256、 画像サイズ:256 x256ピクセル。
注意: FLIMに使用するシステム構成と検出器によっては、画像取得のパラメータが異なる場合があります。 - 顕微鏡のTi:Sapphireレーザーを850nmの 波長 に調整し、レーザー 出力 を1%-2%に調整します。
- レーザー走査型顕微鏡ソフトウェアの [連続 ]タブを選択し、SPCMソフトウェアの [開始 ]を押します。サンプルを60秒間スキャンし、スキャンを停止して、取得したFLIM画像を保存します。
- 取得したFLIMデータを確認します。このためには、FLIMデータ解析SPCImageソフトウェアに生データをロードし、カーソルを細胞の膜に置いてピクセルを選択し、このピクセルの蛍光減衰を分析します。ピクセル強度は、妥当な収集時間 (60 から 120 秒) で減衰 (ビニングを含む) あたり ≥5,000 である必要があります。
- 手順5.4〜5.8を繰り返して、さまざまな視野からのセルのFLIM画像を記録します。
注:BODIPY 2で染色した生細胞のFLIM測定は、BODIPY 2を添加した後~30分に制限する必要があります。
6. データ分析
- 蛍光強度分析:酸化還元比
- ImageJを使用してNAD(P)HとFADの蛍光強度の画像を開きます。
- NAD(P)H 画像内のセルフリー領域を、円または正方形のオプションを使用してハイライト表示します。 [Measure] をクリックし、[ Subtract] をクリックします (メイン パネルで [Process ] を選択し、[ Math ] と [Subtract] を選択します) して、バックグラウンド信号の取得した値を減算します。
- FADイメージに対して手順6.1.2を繰り返します。
- FAD蛍光強度をNAD(P)H蛍光強度で割ることにより、酸化還元比のイメージを取得します。これを行うには、メインパネルで [プロセス ]を選択し、[ 画像計算機 ]と [除算]を選択します。[ 新しいウィンドウを作成する]チェックボックスをオンにして、[ OK]を押します。
- 画像をTIFF形式で保存します。
- 酸化還元比を計算するには、TIFF画像上の特定の細胞の細胞質の領域を選択し、 M キーを押します。目的のすべてのセルに対して繰り返します。
- 測定値をスプレッドシートドキュメントにインポートします。
注:あるいは、細胞内のNAD(P)HおよびFADの蛍光強度を顕微鏡の標準ソフトウェアを使用して測定し、酸化還元比はスプレッドシートソフトウェアでこれらの値を除算することによって取得できます。
- FLIMデータ解析:代謝
- NAD(P)HのFLIM画像をSPCImageソフトウェアにインポートします。
- [コンポーネント] セクションに 2 を入力して、画像に 2 指数関数的減衰フィットを適用します。
- SPCImageソフトウェアの対応するボックスをチェックして、 オフセット パラメータを修正します。
- [オプション]に移動し、[モデル]を選択します。不完全多指数近似モデルと近似法MLEを使用します。
- ビニングを調整して、減衰曲線あたり ≥5000 フォトンのピクセル強度を実現します。
- χ2 の値を確認します。χ2≤ 1.20 は、使用されたモデルが妥当な適合値を提供することを示します。
- 各画像の蛍光寿命のヒストグラムを計算するには、トップメニューの [計算]をクリックし、[ Decay Matrix]をクリックします。
- 特定の細胞の細胞質内の領域を関心領域として選択します。
- 「色」オプションを使用して、寿命の短い成分と長い寿命成分(それぞれ τ1 と τ2)と寿命成分の相対振幅(a1 とa 2、a1 + a2 = 100%)を解析します。
- 測定値をスプレッドシートソフトウェアにエクスポートします。
- 目的のセルごとに手順6.2.8〜6.2.10を繰り返します。
- FADイメージに対して手順6.2.1〜6.2.11を繰り返します。
- FLIMデータ解析:粘度
- FLIM画像をFLIMデータ分析SPCImageソフトウェアにインポートします。
- スキャッターボックスのマークを削除します。
- ローターの蛍光減衰は単指数モデルに適合するはずなので、 コンポーネント セクションに1を入れてください。
- ビニングを調整して、減衰曲線あたり ≥5000 フォトンのピクセル強度を実現します。
- 原形質膜のχ2値を確認します。χ2≤1.20 の値は、使用されたモデルが妥当な適合値を提供することを示します。χ2≥ 1.20 の場合、単指数近似は適用されず、そのようなデータは色素凝集を示している可能性があるため、破棄する必要があります。凝集により、検量線やリード線を誤った粘度推定値に使用できなくなります。
注:二指数関数的減衰は、凝集を示している可能性があります。可変フィルターを備えたFLIMモジュールを搭載した顕微鏡では、参考文献21に記載されているように、モノマーおよび凝集体固有の発光波長範囲(500-550 nmおよび580-650 nm)を試験することで検出できます。 - 各画像の蛍光寿命 τ のヒストグラムを生成するには、上部のメニュー [Calculate] をクリックし、[ Decay Matrix] をクリックします。
- ROIオプションを使用して、単指数関数的崩壊を伴う個々の細胞の原形質膜の領域χ2≤1.20を選択します。
- 蛍光寿命の値をスプレッドシートソフトウェアにエクスポートします。
- 目的のセルごとに手順6.3.7〜6.3.8を繰り返します。
- 次の式を使用して、実験的に測定されたBODIPY 2の寿命(ns)を粘度値(cP)に変換します(以前はメタノール/グリセロール混合物中のBODIPYのキャリブレーションプロットに基づいて取得されました)。
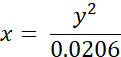
ここで、x - 粘度(cP)、y - 蛍光寿命τ(ns)です。
注意: IRF(Instrument Response Function)は、FLIMフィッティングの重要な部分です。SPCImageでは、蛍光減衰曲線の立ち上がりエッジからIRFが自動的に計算されます。一方、IRFは、セラミックなどの非蛍光サンプル、またはコラーゲン、尿素結晶、糖などのSHG(Second Harmonic Generation)シグナルを生成するサンプルを使用して記録できます。ソフトウェアで計算するオプションがある場合、記録されたIRFの使用はお勧めしません。
Access restricted. Please log in or start a trial to view this content.
結果
ここで説明するプロトコルを使用して、FLIMを使用して生きた培養細胞の代謝補因子と顕微鏡的膜粘度を視覚化しました。測定は、ヒト結腸直腸癌HCT116、マウス結腸癌CT26、ヒト子宮頸癌HeLa Kyoto、およびヒト皮膚線維芽細胞huFBなど、さまざまな癌細胞株で行われています。
蛍光強度に基づく酸化還...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
このプロトコルは、がん細胞のマルチパラメトリック、機能、および生物物理学的分析のためのFLIMの可能性を示しています。内因性蛍光に基づく光学代謝イメージングと、蛍光分子ローターによる外因性標識を用いた原形質膜粘度の測定を組み合わせることで、細胞培養中の生きたがん細胞におけるこれら2つのパラメータ間の相互接続を特徴付け、化学療法に対す?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は何も開示していません。
謝辞
代謝イメージングのプロトコルの開発は、ロシア連邦保健省(政府割り当て、登録番号АААА-А-А20-120022590098-0)によって支援されました。粘度の研究は、ロシア科学財団(プロジェクト番号20-14-00111)の支援を受けました。著者は、ビデオ制作に協力してくれたAnton Plesshanov(PRMU)に感謝しています。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Item/Device | |||
| Cell culture incubator | Sanyo | 37°C, 5% CO2, humidified atmosphere | |
| Centrifuge 5702 R | Eppendorf | 5703000010 | |
| imageJ 1.53c | Wayne Rasband (NIH) | ||
| FLIM module Simple Tau 152 TCSPC (in LSM 880) | Becker & Hickl GmbH | ||
| Laminar flow hood | ThermoFisher Scientific | ||
| Leica microscope DFC290 | Leica Microsystems | ||
| LSM 880 confocal microscope | Carl Zeiss | ||
| Ti:Sapphire femtosecond laser Mai Tai | Spectra Physics | ||
| Microscope incubator XLmulti S DARK LS | PeCon GmbH | 273-800 050 | |
| Mechanical pipettor | Sartorius mLINE | volume 0.5-10 μL; 20-200 μL; 100-1000 μL | |
| Oil-immersion objective C-Apochromat 40×/1.2 NA W Korr (in LSM 880) | Carl Zeiss | 421767-9971-790 | |
| Power-Tau 152 module with the detector HPM-100-40 | Becker&Hickl GmbH | ||
| SPCImage software | Becker & Hickl GmbH | SPC 9.8; SPCImage 8.3 | |
| ZEN software | Carl Zeiss | ZEN 2.1 SP3 (black), Version 14.0.0.201 | |
| Reagent/Material | |||
| 5-fluorouracil | Medac GmbH | 3728044 | |
| DMEM | Gibco, Life Technologies | 31885023 | |
| DMSO | PanEco | F135 | |
| FBS | Hyclone | A3160801 | |
| FluoroBright DMEM | Gibco, Life Technologies | A1896701 | |
| Hank’s solution without Ca2+/Mg2+ | Gibco, Life Technologies | 14175 | |
| l-Glutamine | PanEco | F032 | |
| Mammalian cells | HCT116, CT26, HeLa Kyoto, huFB | ||
| Molecular rotor BODIPY 2 | Synthesized and Supplied by Marina Kuimova Group, Imperial College London | ||
| Penicillin/streptomycin | PanEco | A065 | |
| Tissue culture dish with cover glass-bottom FluoroDishes | World Precision Instruments, Inc | ||
| Trypsin- EDTA 0.25% | PanEco | P034 | |
| Versen buffer | PanEco | R080p |
参考文献
- Vazquez, A., et al. Cancer metabolism at a glance. Journal of Cell Science. 129 (18), 3367-3373 (2016).
- Li, Z., Zhang, H. Reprogramming of glucose, fatty acid, and amino acid metabolism for cancer progression. Cellular and Molecular Life Sciences. 73 (2), 377-392 (2015).
- Strickaert, A., et al. Cancer heterogeneity is not compatible with one unique cancer cell metabolic map. Oncogene. 36 (19), 2637-2642 (2016).
- Szlasa, W., Zendran, I., Zalesińska, A., Tarek, M., Kulbacka, J. Lipid composition of the cancer cell membrane. Journal of Bioenergetics and Biomembranes. 52, 321-342 (2020).
- Preta, G. New insights into targeting membrane lipids for cancer therapy. Frontiers in Cell and Developmental Biology. 8, 571237(2020).
- Kojima, K. Molecular aspects of the plasma membrane in tumor cells. Nagoya Journal of Medical Science. 56, 1-18 (1993).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 071203(2020).
- Becker, W. Advanced time-correlated single photon counting applications. Springer Series in Chemical Physics. , Springer. (2015).
- Shirmanova, M. V., Shcheslavskiy, V. I., Lukina, M. M., Becker, W., Zagaynova, E. V. Exploring tumor metabolism with time-resolved fluorescence methods: From single cells to a whole tumor. Multimodal Optical Diagnostics of Cancer. 3, 133-155 (2020).
- Kalinina, S., Rück, A. FLIM and PLIM in biomedical research. An innovative way to combine autofluorescence and oxygen measurements. Photonics & Lasers in Medicine. 5 (4), 257-266 (2016).
- Kuimova, M. K. Molecular rotors image intracellular viscosity. Chimia. 66 (4), 159-165 (2012).
- Kuimova, M. K. Mapping viscosity in cells using molecular rotors. Physical Chemistry Chemical Physics. 14 (37), 12671(2012).
- Haidekker, M. A., Theodorakis, E. A. Molecular rotors-fluorescent biosensors for viscosity and flow. Organic & Biomolecular Chemistry. 5, 1669-1678 (2007).
- Liu, X., et al. Molecular mechanism of viscosity sensitivity in BODIPY rotors and application to motion-based fluorescent sensors. ACS Sensors. 5 (3), 731-739 (2020).
- Shirmanova, M. V., Shimolina, L. E., Lukina, M. M., Zagaynova, E. V., Kuimova, M. K. Live cell imaging of viscosity in 3D tumour cell models. Multi-Parametric Live Cell Microscopy of 3D Tissue Models. Advances in Experimental Medicine and Biology. Dmitriev, R. 1035, 143-153 (2017).
- Shimolina, L. E., et al. Imaging tumor microscopic viscosity in vivo using molecular rotors. Scientific Reports. 7, 41097(2017).
- Scott, T. G., Spencer, R. D., Leonard, N. J., Weber, G. Synthetic spectroscopic models related to coenzymes and base pairs. V. Emission properties of NADH. Studies of fluorescence lifetimes and quantum efficiencies of NADH, AcPyADH, [reduced acetylpyridineadenine dinucleotide] and simplified synthetic models. Journal of the American Chemical Society. 92 (3), 687-695 (1970).
- Harvey, R. A., Damle, S. A fluorescent modification of flavin adenine dinucleotide. FEBS Letters. 26 (1-2), 341-343 (1972).
- Kubánková, M., Summers, P., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Microscopic viscosity of neuronal plasma membranes measured using fluorescent molecular rotors: Effects of oxidative stress and neuroprotection. ACS Applied Materials and Interfaces. 11, 36307-36315 (2019).
- Kubánková, M., López-Duarte, I., Kiryushko, D., Kuimova, M. K. Molecular rotors report on changes in live cell plasma membrane microviscosity upon interaction with beta-amyloid aggregates. Soft Matter. 14, 9466-9474 (2018).
- Wu, Y., et al. Molecular rheometry: Direct determination of viscosity in Lo and Ld lipid phases via fluorescence lifetime imaging. Physical Chemistry Chemical Physics. 15 (36), 14986(2013).
- Shimolina, L. E., et al. Mapping cisplatin-induced viscosity alterations in cancer cells using molecular rotor and fluorescence lifetime imaging microscopy. Journal of Biomedical Optics. 25 (12), 126004(2020).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014(2007).
- Rück, A., Hauser, C., Mosch, S., Kalinina, S. Spectrally resolved fluorescence lifetime imaging to investigate cell metabolism in malignant and nonmalignant oral mucosa cells. Journal of Biomedical Optics. 19 (9), 096005(2014).
- Lee, D. -H., Li, X., Ma, N., Digman, M. A., Lee, A. P. Rapid and label-free identification of single leukemia cells from blood in a high-density microfluidic trapping array by fluorescence lifetime imaging microscopy. Lab on a Chip. 18 (9), 1349-1358 (2018).
- Lukina, M. M., et al. Interrogation of tumor metabolism in tissue samples ex vivo using fluorescence lifetime imaging of NAD(P)H. Methods and Applications in Fluorescence. 8 (1), 014002(2019).
- Alam, S. R., et al. Investigation of mitochondrial metabolic response to doxorubicin in prostate cancer cells: An NADH, FAD and Tryptophan FLIM assay. Scientific Reports. 7 (1), 10451(2017).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Lukina, M. M., et al. In vivo metabolic and SHG imaging for monitoring of tumor response to chemotherapy. Cytometry Part A. 95 (1), 47-55 (2019).
- Shah, A. T., et al. Optical metabolic imaging of treatment response in human head and neck squamous cell carcinoma. PLoS One. 9 (3), 90746(2014).
- Sergeeva, T. F., et al. Relationship between intracellular pH, metabolic co-factors, and caspase-3 activation in cancer cells during apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1864 (3), 604-611 (2017).
- Shirmanova, M. V., et al. Chemotherapy with cisplatin: insights into intracellular pH and metabolic landscape of cancer cells in vitro and in vivo. Scientific Reports. 7, 8911(2017).
- Lukina, M. M., et al. Metabolic cofactors NAD(P)H and FAD as potential indicators of cancer cell response to chemotherapy with paclitaxel. Biochimica et Biophysica Acta (BBA) - General Subjects. 1862 (8), 1693-1700 (2018).
- Druzhkova, I. N., et al. The metabolic interaction of cancer cells and fibroblasts - coupling between NAD(P)H and FAD, intracellular pH and hydrogen peroxide. Cell Cycle. 15 (9), 1257-1266 (2016).
- Lakowicz, J., Szmacinski, H., Nowaczyk, K., Johnson, M. Fluorescence lifetime imaging of free and protein bound NADH. Proceedings of the National Academy of Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Vanden Berg, P. A. W., Feenstra, K. A., Mark, A. E., Berendsen, H. J. C., Visser, A. J. W. G. Dynamic conformations of flavin adenine dinucleotide: Simulated molecular dynamics of the flavin cofactor related to the time-resolved fluorescence characteristics. The Journal of Physical Chemistry B. 106 (34), 8858-8869 (2002).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Blacker, T. S., et al. Separating NADH and NADPH fluorescence in live cells and tissues using FLIM. Nature Communications. 5, 3936(2014).
- Lu, W., Wang, L., Chen, L., Hui, S., Rabinowitz, J. D. Extraction and quantitation of nicotinamide adenine dinucleotide redox cofactors. Antioxidants and Redox Signaling. 28 (3), 167-179 (2018).
- Ranjit, S., Malacrida, L., Jameson, D. M., Gratton, E. Fit-free analysis of fluorescence lifetime imaging data using the phasor approach. Nature Protocols. 13 (9), 1979-2004 (2018).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13582-13587 (2011).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116 (48), 24019-24030 (2019).
- Wang, S., Chacko, J. V., Sagar, A. K., Eliceiri, K. W., Yuan, M. Nonparametric empirical Bayesian framework for fluorescence-lifetime imaging microscopy. Biomedical Optics Express. 10 (11), 5497-5517 (2019).
- Quinn, K. P., et al. Quantitative metabolic imaging using endogenous fluorescence to detect stem cell differentiation. Scientific Reports. 3 (1), 3432(2013).
- Hou, J., et al. Correlating two-photon excited fluorescence imaging of breast cancer cellular redox state with seahorse flux analysis of normalized cellular oxygen consumption. Journal of Biomedical Optics. 21 (6), 060503(2016).
- Wang, H., et al. Red-emitting fluorescence probe for sensing viscosity in living cells. Chemical Papers. 74, 1071-1078 (2020).
- Chen, B., et al. Sensing and imaging of mitochondrial viscosity in living cells by a red photoluminescent probe with long lifetime. Chemical Communications. 55, 7410(2019).
- Shen, B., Wang, L. F., Zhi, X., Qian, Y. Construction of a red emission BODIPY-based probe for tracing lysosomal viscosity changes in culture cells. Sensors and Actuators B: Chemical. 304, 127271(2019).
- Chen, T., Chen, Z., Liu, R., Zheng, S. NIR fluorescent probe for detection of viscosity and lysosome imaging in live cells. Organic and Biomolecular Chemistry. 17, 6398(2019).
- Angelucci, C., et al. Epithelial-stromal interactions in human breast cancer: effects on adhesion, plasma membrane fluidity and migration speed and directness. PLoS One. 7 (12), 50804(2012).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved