このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
BIFC-FRET-FLIMアッセイによるMADS型転写因子の2つのモノマーとカルシウムセンサタンパク質の三者間相互作用の測定
* これらの著者は同等に貢献しました
要約
ここでは、BiFCベースのFRET-FLIMアッセイによる蛍光タグ付きタンパク質を用いた3つのタンパク質パートナー間の三元複合体形成を可視化する方法を提示する。この方法は、 生体内のタンパク質とタンパク質相互作用複合体を研究する上で有用である。
要約
タンパク質とタンパク質の相互作用は、細胞機能の調節、維持、および修正において重要な役割を果たす細胞内のすべての生物学的プロセスの不可欠な部分です。これらの相互作用は、シグナル伝達、病原体応答、細胞と細胞相互作用、代謝および発達過程などの幅広い現象に関与しています。転写因子の場合、これらの相互作用は、サブユニットのオリゴマー化、核、細胞質などの特定の細胞下の文脈での隔離を引き起こし、下流遺伝子の発現により深い影響を及ぼす可能性がある。ここでは、蛍光生涯イメージング(FLIM)を含む二分子蛍光相補(BiFC)ベースのフェルスター共鳴エネルギー伝達(FRET)を用いて 生体内 三者間相互作用を可視化する方法論を示す。このデモンストレーションのために選択された2つのタンパク質はBiFCパートナーとして相互作用し、その再構成された蛍光活性は第3のパートナーとのFRET-FLIMのアッセイに使用される。4~5週齢の成長室で栽培された ニコティアナ・ベンタミアナ 工場が、このデモンストレーションのモデルプラントシステムとして使用されています。
概要
タンパク質相互作用(PPI)は、様々な代謝および発達過程を調節することによって真核細胞の適切な機能の基礎を形成する。PPI の中には安定しているものもありますが、一時的な PPI もあります。相互作用は、異なる間、三量体、四量体同族体、ヘテロマー1などの相互作用におけるメンバーの数と種類に基づいて分類され得る。タンパク質相互作用の同定と特性評価は、タンパク質機能と調節ネットワークの理解を深める可能性があります。
転写因子は、調節機能に関与するタンパク質です。彼らはDNAに結合することによって下流遺伝子の転写速度を調節する。時にはタンパク質によるオリゴマー化または高次複合体の形成は、それらの機能を実行するための前提条件である2.植物MADSボックス転写因子は、花の転移、花の臓器の発達、受精、種子の発達、老化、栄養発達などの様々なプロセスを調節するホメオ遺伝子です。それらはDNA3,4に結合する高次複合体を形成することが知られている。転写因子とそのインターアクターの間でPPIネットワークを研究することは、転写調節の根底にある複雑さに関する洞察を提供する。
ニコチアナベンタミアナの一過性タンパク質発現は、生体内でタンパク質の局在化またはタンパク質とタンパク質の相互作用を研究する一般的なアプローチとなっています。BiFCとFRETは、蛍光レポーターシステム6を用いて生体内でタンパク質とタンパク質相互作用を研究するための方法である。これら2つの技術の組み合わせにより、3つのタンパク質7間の相互作用が明らかになったことが示されている。FRETはアクセクサフォトブリーチ、感作発光、蛍光寿命イメージング(FLIM)技術を用いて測定します。FLIMベースのFRETアッセイは、その蛍光寿命8に基づいて2つの分子間のエネルギー移動測定に正確な定量および時空間的特異性を提供するツールとして登場した。FLIMは、蛍光素が光子を放出する前に励起状態にとどまる時間を測定し、強度測定を単独で使用する技術よりも優れています9,10.ニコチアナベンタミアナやタマネギ表皮のような異種系のほかに、最近の報告では、シロイヌナズナや若い米の苗などの使用が実証されており、在来条件11,12の下でのタンパク質相互作用のインビボ解析に対して明示されている。
適切な発現系以外にも、BiFCおよびFRETアッセイの相互作用パートナーの選択も、この実験の成功にとって極めて重要である。BiFC 構成で使用されるパートナー間の PPI は、FRET 実験13で共役したパートナーとして使用する前に適切なコントロールを使用して検証する必要があります。BiFCは、蛍光タンパク質のN末端およびC末端部の構造的補引を利用する。BiFCアッセイで使用されるすべての蛍光タンパク質が2つの誘導非蛍光フラグメント間の自己集合性であったわけではない場合の最も一般的な制限は、2つの誘導性非蛍光フラグメント間の自己集合であり、偽陽性蛍光に寄与し、シグナル対雑音(S/N)比14を低下させる。最近の開発は、蛍光タンパク質を分割する点突然変異または位置を含む、強度の増加、特異性、高いS/N比15、16を有するBiFC対を生じさせた。これらの蛍光タンパク質は、実験の適性に応じてBiFCを実施するためにも使用できる。
従来、CFPとYFPは、FRET実験17においてドナー及びアクセプターペアとして使用されてきた。しかし、YFPまたはm-シトリンは、 シロイヌナズナ の根系における標的タンパク質の天然発現中の高い量子収率(QY)のために、より良いFRETドナー(アクセプターとしてRFPで使用される場合)であることが判明した。プロモーター(構成的対ネイティブ/内因性)およびフルオロフォアの選択は、BiFC-FRET-FLIM実験の成功を設計する上でも重要な役割を果たします。FRETドナーの効率とFRETペアの適合性は、発現に使用されるプロモーターおよび生物学的システムの変化に伴って変化する傾向があることに注意することが不可欠である。蛍光色素のQYは、その明るさに関連し、pH、温度、および使用中の生物学的システムに依存します。FRET実験用の蛍光素組を選択する前に、これらの基準を十分に考慮することを提案する。このプロトコルに使用される生物学的システム、プロモーター、およびタンパク質は、BiFC FRET-FLIM実験のためのCFP-YFPフルオロフォアとうまく機能しました。
本研究では、BiFCベースのFRETを用いて3つのタンパク質分子間の相互作用を可視化するFLIMの特徴を取り入れた。この技術では、2つのタンパク質が分割されたYFPタンパク質と3番目のタンパク質とCFPでタグ付けされます。MADS-boxタンパク質(M)ホモジマーとカルシウムセンサータンパク質(C)の相互作用を研究することに興味を持っていたので、これらのタンパク質はpSITE-1CAおよびpSITE-3CAベクター18において蛍光タンパク質でタグ付けされた。このアッセイでは、2人の相互作用パートナーが、pSPYNE-35SおよびpSPYCE-35Sベクトル19でYFPのNおよびC末端部分でタグ付けされ、その相互作用は、CFP(FRETドナーとして機能する)でタグ付けされた第3の相互作用パートナーにFRETアクセプターとして機能する機能的YFPの再構成をもたらす(図1).この特定の場合、2つのMモノマー間およびMとCの間のPPIは、酵母2ハイブリッド系と共に3つの異なるシステムでBiFCを行うことによって検証された。これらのベクターは、エレクトロポレーションにより アグロバクテリウム・トゥミファシエンス GV3101株に動員された。GV3101株は、ジェンタマイシン耐性20を有する武装解除されたTiプラスミドpMP90(pTiC58DT-DNA)を有する。p19 アグロバクテリウム 株は、遺伝子導入サイレンシング21を防ぐために、すべての浸潤と一緒に添加されました。三者間相互作用を検証するために、3つのタンパク質を反対の立体構造で使用することもお勧めします。
この技術では、まず、ドナーの蛍光寿命(非消痛ドナー生涯)がアクセプターの不在時に測定されるFLIMを採用した。その後、その寿命は、アクセンプター(クエンチドナー寿命)の存在下で測定される。このドナー蛍光寿命の差は、FRET効率の計算に使用され、これは蛍光寿命の減少を示す光子の数に依存する。以下に述べる詳細なプロトコルは、 ニコチアナベンタミ アナの蛍光タグ付きタンパク質を一時的に発現し、BiFC-FRET-FLIMによる相互作用をアッテルすることにより、任意の3つのタンパク質間の三元複合体の形成を決定する。
プロトコル
1. 遺伝子のクローン作成と終点ベクター (図 2)
- PCRによって目的の遺伝子(我々の場合はMおよびC遺伝子)のコード配列(CDS)を増幅し、適切なエントリベクター(例えば、pENTR/D-TOPOベクター)でそれらをクローン化する。この実験で使用したベクトルについては、表1を参照してください)。
- 抗生物質を含むプレート上のクローンを成長させます。制限消化とDNAシーケンシング22,23によって抗生物質で選択されたクローンを検証する。
- エントリクローンからターゲットベクター(pSPYNE-35S、pSPYCE-35S、pSITE-1CAおよびpSITE-3CA)への再構成されたCDSを動員し、制限酵素消化によってエントリから終先ベクターへの配列の伝達を確認する。
注: この実験で使用したすべてのベクトルを表 1に示します。 - 最後に、電気穿孔により先ベクトルを有するアグロバクテリウムGV3101(pMP90(ゲントR))細胞を変換する(図3)24。
2. ニコティアナベンタミアナ 植物の成長条件
注:コントロール条件で4-6葉のステージまで ニコチアナ 植物を育てます。
- ニコティアナ植物を栽培するには、2:1:1の比率でココピントと堆肥と市販の土壌混合物を混合することによって土壌ミックスを調製します。
- この土壌混合物の1インチの厚さの層をプラスチックトレイに広げて、土壌ベッドを作り、脱イオン水で飽和させます。この土壌ベッドに約200種を振りかけます。
- 1cmの立ち水を含む大きなトレイに移します。このトレイをラップで覆い、湿気チャンバーを作ります。
- このセットを16時間光、8時間暗サイクルで150-170 μmol/m2の光強度を有する23°Cに設定された成長室に移します。
- 2週間後、若い苗を水飽和土壌混合物を含む小さな3〜4インチのポットに移します。
- これらのポットをプラスチックトレイに入れ、さらに4週間成長室に移します。
3. アグロ浸潤用細菌株の準備
注:アグロ浸潤の場合、細菌株は、適切な比率でアグロバクテリウムのp19株と一緒に新たに副培養し、混合する必要があります。
- リフィンピシン(100 μg/mL)、ゲンタマイシン(25 μg/mL)、カナマイシン(50 μg/mL)を含む2xYT寒天プレートを、pSPYNE-35SおよびpSPYCE-35Sベクターを収容するアグロバクテリウム用に準備します。歪みを含むpSITEベクターの場合は、リバンピシン(100 μg/mL)、ゲンタマイシン(25 μg/mL)、スペクチノマイシン(50 μg/mL)を使用してください。
- これらのプレートにプラスミドを含むアグロバクテリウム株を、層流フードで無菌接種ループを使用してストリークする。
- 暗闇の中で48時間28°Cでこれらをインキュベートします。
- BiFCおよびFRETの構造を収容する アグロバクテリウム GV3101株を接種することによってこの手順を開始する(pSPYNE-35Sで調製 適切な抗生物質(リタンピシン(100 μg/mL)、ゲンタマイシン(25 μg/mL)、カナマイシン(50 μg/mL)またはスペキシノマイシン(50 μg/mL)を含む2xYTブロスの10 mLのストリークプレートからのpSPYCE-35SおよびpSITEベクター。
- さらに、リバンピシン(100 μg/mL)とカナマイシン(50 μg/mL)を含む2xYTブロスの10 mLを接種することにより、アグロバクテリアのp19株の培養を開始します。
注:p19株は、トランスジーンサイレンシングを防ぐために追加されます。 - フラスコをアルミホイルで覆い、暗闇の中で28°Cと170rpmに設定されたインキュベーターシェーカーに保管してください。
- 一晩成長した後、この培養物の1mLを使い捨てキュベットに移し、分光光度計を用いて600nmで培養物の光学密度(O.D.)を測定する。
- 各培養物の最終的なO.D.が0.5であり、p19のO.D.が2mLの総容積で0.3になるように、株を含む適切なBiFCおよびFRETパートナーの培養を混合する。
- これらの比率を達成するには、次の式を使用します。
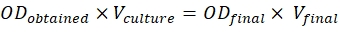
600 nmで測定した培養物のOD=O.D.
Vカルチャー =必要な文化の量
OD最終 = 0.5 コンストラクト、0.3 (p19)
V最終 = 2 mL の浸潤の最終容積
注: このスタディで使用する構成の組み合わせは 、表 2に示されています。 - 混合したアグロバクテリウム培養物を室温で5分間5分間遠心し、上清を慎重に捨てます。ペレットを2 mLで再懸濁して、2 mLの新たに調製した浸潤バッファー(10 mM MES、100 μMのアセトシリンギロン、および10 mM MgCl2)に入れます。ボルテックスミキサーを使用して、均質な細胞懸濁液を作ります。
- 再懸濁細胞を含むチューブを室温で暗所で3時間インキュベートする。
- 一方、各植木鉢に、浸潤するコンストラクト混合物をラベル付けする。各浸潤混合物に2つの植物を使用してください。
- 1 mL の無針注射器に農薬ミックスを充填します。緩やかに、しかししっかりと反対側から葉を支えながら、完全に展開された葉の腹側にシリンジを押し付ける。溶液がシリンジ先端の2〜3倍に相当する葉の領域を満たすまで、プランジャーをそっと押します。
- 図4 に示すように、葉の上に最大 4 つのスポット、植物ごとに 3~4 個の葉を浸します。
注:クロスコンタミネーションを防ぐために、サンプル間の70%のアルコールで手袋を交換するか、手袋を拭いてください。 - すべてのポットをトレイに移し、ステップ2で述べたように同じ条件で成長チャンバーでインキュベートします。
- 蛍光顕微鏡を用いて、異なる時点で、農浸潤葉の小さな部分を確認します。YFPとCFPの両方からの蛍光が細胞内で検出可能な場合は、BiFC-FRET FLIMアッセイ用の共焦点顕微鏡に進みます。本実験では、分析を、農浸潤後3日目に行った。
注:BiFC-FRET FLIMアッセイで使用されるキメラタンパク質の過剰発現を避けるために、プロモーターと遺伝子の組み合わせごとに、アグロインフィルレーション後のインキュベーション期間を個別に設定してください。パートナータンパク質の過剰発現は、偽陽性相互作用につながる可能性があります。
4. 蛍光可視化用スライドの準備
- 植物が視覚化の準備ができたら、角葉サンプルを、浸潤創傷から5〜8mm離れて切り、きれいなスライドに蒸留水に取り付けます。
注意:背景の蛍光を最小限に抑えるには、80%エタノールでスライドを洗浄し、その後に蒸留水を3〜4回洗浄し、空気乾燥し、吸収シートに保管してください。 - きれいなカバースリップで葉のサンプルを覆い、爪のエナメルを使用して密封します。
- これらのサンプルを共焦点レーザー走査顕微鏡で可視化します。
5. 共焦点レーザー走査顕微鏡を用いたFRET-FLIM解析
注:この手順では、2つのタンパク質間の相互作用を決定し、定量化する基礎は、FRETの効率を計算するために使用されるアクセクターとの相互作用時のFRET-ドナーパートナーの蛍光寿命の減少です。三者間相互作用の場合の複雑さは、FRETアクセプタ(この場合は)単一分子ではなく、機能的なFRETアクセプターフルオロフォアになるために 生体内で 再構成されるべきスプリットYFP-BiFCペアであるため、さらに増加する。FRET-FLIMを実施するには、ドナー分子の蛍光寿命を最初に決定し、次にFRETパートナーの存在下で決定する必要があります。
- FLIMアプリケーションを共焦点レーザー走査顕微鏡で開き、コンソールを起動し、パターン認識光子計を使用して蛍光寿命を測定します。標準の「すべてのフォトンカウント」測定モードを選択します。
- 2種類のアグロ浸潤植物のサンプルを分析します:1つはドナーのみ(C-CFP)、もう1つはドナーとアクセプター(M-YFPと共に)を持っています。
注:Mタンパク質の相互作用は、BiFCとY2Hを使用してCタンパク質とすでに検証されているため、この相互作用ペアで良好なFRET効率が期待されます。 - 次に、C-CFPの凝潤した葉をスキャンし、良好なCFP蛍光を示す細胞に焦点を当てます。レーザースキャンモードを開始し、CFP可視化およびFLIM測定用のシステムを設定します(λex 440 nmパルスレーザー、ハイブリッド検出器によるλem 480-520 nm、400Hzで512 x 512ピクセルのスキャン速度)。
- フォーカス、ズーム、スマートゲインを調整して、キャプチャする必要がある領域に焦点を当てます。
- 十分なレーザーパワーでサンプルを照らし、パルス当たり約0.1光子のキャプチャを実現します。可変蛍光強度のサンプルでは、50フレームをキャプチャして、寿命測定に必要な適切な光子を収集します。CFPは、その立体構造の適応に起因する2つの蛍光寿命を示す;したがって、n の値を 2 に維持しながら、n 指数再畳モデルを使用してデータを適合させます。
- これらの設定では、CFP は 1.0 と 3.2 ns の 2 つのライフタイムを示します。ここでは、より高い、3.2 ns、寿命は、すべての後続の計算25、26のために使用されます。
- 2つのタンパク質間の相互作用の度合いを測るFLIMを用いてFRET効率を計算するには、C-CFPおよびM-YFPと共浸透した葉試料を採取する。C-CFPとM-YFPの両方を発現するセルを探し、λ ex 440 nmパルスレーザー、λ em 480-520 nm、λex 514 nmの白色光レーザー(λem 526-550 nm)を使用して、それぞれの発光パターンを確認します。CFPとYFPの両方の蛍光を示す細胞を順次スキャンして同定する。
- 両方のタンパク質から蛍光を確認した後、FLIMコンソールに切り替えて、C-CFPの寿命を測定するために先ほど使用したのと同じ設定を使用してCFPの寿命を測定します(ステップ5.5)。
注: このセルは、C-CFP と対話する可能性があり、C-CFP の寿命を短縮する可能性がある M-YFP も表現しています。 - n指数再畳モデルを使用して得られたグラフをn = 2で合わせます。CFP寿命が3.2から2.6nsに減少することが観察され、CFPとYFPの間でのフェルスター共鳴エネルギー伝達を示す(図5A)。
- ソフトウェアでFRETコンソールを起動し、ソフトウェアで提供される方程式に手動で絶え間ないドナー寿命を入力してFRET効率を計算します。観察されたFRET効率は:56%である。
- 三者間相互作用
- 最後に、3つのパートナー間の相互作用を視覚化するために、C-CFP、M-YFPn、およびM-YFPcと共浸透した植物から葉のサンプルを採取します。
- 葉の外植をスキャンして、2つのMタンパク質間のBiFC相互作用から発せられるCFPと再構成されたYFP蛍光の両方を示す細胞を探します。以前使用したレーザーと発光波長を使用します。
- その後、514 nmレーザーの電源を切り、FLIMコンソールに移動します。
注: M-YFP ダイマーが C-CFP と相互作用する場合、M-YFP との相互作用の間に観察される C-CFP の寿命が減少するはずです。ただし、C-CFP が M-YFP ダイマーと相互作用しない場合、蛍光寿命は 3.2 ns にとどまります。 - 上記と同様の設定を使用して、再構成されたYFPの存在下でCFP寿命を測定します。n指数再畳モデルを使用して得られたグラフを n = 2 で適合させ、FRET コンソールに移動します。
注: CFP の有効期間は 3.2 から 2.3 ns に低下しています。上記のとおり、FRET効率を計算します。算出されたFRET効率は55%です。ドナーの寿命の低下と良好なFRET効率55%は 、生体内の 2つのMタンパク質とCタンパク質との三者間相互作用を確認する( 図5Bを参照)。
結果
このプロトコルは、 植物における生体三 個体タンパク質相互作用をインビボで研究するための最適化された方法を表します。このプロトコルの基本原則は、2つの蛍光タグ付きタンパク質相互作用技術、すなわちBiFCとFRETを組み合わせて、3つのタンパク質パートナー間の三元複合体形成を測定するアッセイを作成することです。ここでは、FRETアクセラシビサの存在下および不在中のFRE...
ディスカッション
本プロトコルは、MADS-boxタンパク質とカルシウムセンサータンパク質の2つのモノマー間の三元複合体の形成を確認するためのBiFCベースのFRET-FLIMアッセイの使用を実証する。このプロトコルは、Y. John Shyuらのレポートから、感作放出法7を用いてFos-Junヘテロ二量体とNFATまたはp65の間に形成された三次複合体を可視化するBiFCベースのFRET法を開発したところから適応される。以?...
開示事項
著者らは利益相反を宣言しない。
謝辞
NB, GG, SB, KCは、大学補助金委員会(UGC)、UGC-BSR、DBT-INSPIRE、科学産業研究評議会(CSIR)の研究フェローシップに心から感謝します。我々はありがたいことに、バイオテクノロジー省(DBT)、インド政府、科学技術省(DST-FIST)、インド政府の財政支援を認める。
資料
| Name | Company | Catalog Number | Comments |
| 1 ml Syringes without needles | Dispovan | - | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Gateway LR Clonase II Enzyme mix | Thermo Fischer Scientific | 11791020 | The vectors used in the study are Gateway based |
| Gentamycin Sulphate | Himedia | CMS461 | |
| Kanamycin Sulphate | Himedia | MB105 | |
| MES hydrate | Sigma-Aldrich | M2933 | |
| MgCl2 | Sigma-Aldrich | M2670 | |
| pENTR/D-TOPO Cloning Kit | Thermo Fischer Scientific | K240020 | The vectors used in the study are Gateway based |
| Phusion high fidelity Taq DNA polymerase | Thermo Fischer Scientific | F530-S | Any High fidelity Polymerase can work |
| Rifampicin | Himedia | CMS1889 | |
| SP8 FALCON Confocal laser scanning microscope | Leica | SP8 FALCON | Any CLSM with FLIM capabilities can be used for this analysis |
| Spectinomycin dihydrochloride pentahydrate | Himedia | TC034 |
参考文献
- Grove, C. A., Walhout, A. J. M. Transcription factor functionality and transcription regulatory networks. Molecular Biosystem. (4), 309-314 (2008).
- Amoutzias, G. D., Robertson, D. L., Van de Peer, Y., Oliver, S. G. Choose your partners: dimerization in eukaryotic transcription factors. Trends in Biochemical Sciences. 33 (5), 220-229 (2008).
- Arora, R., et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress. BMC Genomics. 8 (242), (2007).
- Theißen, G., Gramzow, L. Structure and evolution of plant MADS domain transcription factors. Plant Transcription Factors: Evolutionary, Structural and Functional Aspects. , 127-138 (2016).
- Schweiger, R., Schwenkert, S. Protein-protein interactions visualized by bimolecular fluorescence complementation in tobacco protoplasts and leaves. Journal of Visualized Experiments: JoVE. (85), (2014).
- Bracha-Drori, K., et al. Detection of protein-protein interactions in plants using bimolecular fluorescence complementation. Plant Journal. 40 (3), 419-427 (2004).
- Shyu, Y. J., Suarez, C. D., Hu, C. Visualization of ternary complexes in living cells by using a BiFC-based FRET assay. Nature Protocols. 3 (11), 1693-1702 (2008).
- Kwaaitaal, M., Keinath, N. F., Pajonk, S., Biskup, C., Panstruga, R. Combined bimolecular fluorescence complementation and förster resonance energy transfer reveals ternary SNARE complex formation in living plant cells. Plant Physiology. 152 (3), 1135-1147 (2010).
- Margineanu, A., et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Scientific Reports. 6, (2016).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (07), 1 (2020).
- Long, Y., et al. Optimizing FRET-FLIM labeling conditions to detect nuclear protein interactions at native expression levels in living Arabidopsis roots. Frontiers in Plant Science. 9, 1-13 (2018).
- Burman, N., Chandran, D., Khurana, J. P. A Rapid and highly efficient method for transient gene expression in rice plants. Frontiers in Plant Science. , 11 (2020).
- Kudla, J., Bock, R. Lighting the way to protein-protein interactions: Recommendations on best practices for bimolecular fluorescence complementation analyses. Plant Cell. 28 (5), 1002-1008 (2016).
- Shyu, Y. J., Hu, C. D. Fluorescence complementation: an emerging tool for biological research. Trends in Biotechnology. 26 (11), 622-630 (2008).
- Kodama, Y., Hu, C. D. An improved bimolecular fluorescence complementation assay with a high signal-to-noise ratio. BioTechniques. 49 (5), 793-803 (2010).
- Kodama, Y., Hu, C. D. Bimolecular fluorescence complementation (BiFC): A 5-year update and future perspectives. BioTechniques. 53 (5), 285-298 (2012).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends in Biochemical Sciences. 32 (9), 407-414 (2007).
- Chakrabarty, R., et al. pSITE vectors for stable integration or transient expression of autofluorescent protein fusions in plants: Probing Nicotiana benthamiana- Virus Interactions. Molecular Plant-Microbe Interactions. 20 (7), 740-750 (2007).
- Walter, M., et al. Visualization of protein interactions in living plant cells using bimolecular fluorescence complementation. Plant Journal. 40 (3), 428-438 (2004).
- Hellens, R., Mullineaux, P., Klee, H. A guide to Agrobacterium binary Ti vectors. Trends in Plant Science. 5 (10), 446-451 (2000).
- Van Der Hoorn, R. A. L., Rivas, S., Wulff, B. B. H., Jones, J. D. G., Joosten, M. H. A. J. Rapid migration in gel filtration of the Cf-4 and Cf-9 resistance proteins is an intrinsic property of Cf proteins and not because of their association with high-molecular-weight proteins. Plant Journal. 35 (3), 305-315 (2003).
- Xie, X., et al. Engineering SARS-CoV-2 using a reverse genetic system. Nature Protocols. 16, (2021).
- Xu, J., et al. Optimized plasmid construction strategy for Cas9. Cellular Physiology and Biochemistry. 48, 131-137 (2018).
- Mattanovich, D., et al. Efficient transformation of Agrobacterium spp. by electroporation. Nucleic Acids Research. 17 (16), 6747 (1989).
- Rizzo, M. A., Springer, G. H., Granada, B., Piston, D. W. An improved cyan fluorescent protein variant useful for FRET. Nature Biotechnology. 22 (4), 445-449 (2004).
- Tramier, M., et al. Picosecond-hetero-FRET microscopy to probe protein-protein interactions in live cells. Biophysical Journal. 83 (6), 3570-3577 (2002).
- Alvarez, L. A. J., et al. SP8 FALCON: a novel concept in fluorescence lifetime imaging enabling video-rate confocal FLIM. Nature Methods. 20, 2-4 (2019).
- Postma, M., Goedhart, J. Plotsofdata-a web app for visualizing data together with their summaries. PLoS Biology. 17 (3), 1-8 (2019).
- Galperin, E., Verkhusha, V. V., Sorkin, A. Three-chromophore fret microscopy to analyze multiprotein interactions in living cells. Nature Methods. 1 (3), 209-217 (2004).
- Waadt, R., Kudla, J. In plant visualization of protein interactions using bimolecular fluorescence complementation (BiFC). Cold Spring Harbor Protocols. 3 (4), (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved