このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
RNAアプタマーおよびリボスイッチをコードするDNAプラスミドを装填した多機能シルクベースのマイクロカプセルの調製
要約
このプロトコルは、堅牢で生体適合性のあるDNAを含むマイクロカプセルの形成を、いくつかのリガンドを追跡できるマルチプレックス 化されたin vitro バイオセンサーとして説明しています。
要約
犠牲球状コア上でのLayer-by-layer(LbL)アセンブリ法によるDNA含有絹フィブロインマイクロカプセルの調製のためのプロトコルを紹介します。プライム層とDNAプラスミドの吸着に続いて、単一のシルク層の急性脱水中にシルク二次構造にβシートを誘導することにより、堅牢なマイクロカプセルの形成を促進しました。したがって、層状化は複数の水素結合と疎水性相互作用を介して発生しました。多層シェルを吸着すると、コアシェル構造を金ナノ粒子(AuNP)および/または抗体(IgG)でさらに機能化して、リモートセンシングおよび/またはターゲットデリバリーに使用できます。ポリマープライマーの存在、DNAとシルクタンパク質の濃度、および多数の吸着層など、シリカコアへの主要な高分子の順次堆積中にいくつかの重要なパラメーターを調整すると、透過性とDNA負荷が変化する生体適合性のDNAを含むマイクロカプセルが得られました。シリカコアの溶解時に、プロトコルは、カプセル膜の内面に固定化されたDNAプラスミドを有する中空で堅牢なマイクロカプセルの形成を実証した。DNAプラスミドと外部環境との間に選択的に透過性の生体適合性膜を作成することで、長期保存中にDNAを保存し、空間的に閉じ込められたプラスミドからの出力応答の改善に重要な役割を果たしました。DNAテンプレートの活性とそのアクセシビリティを、 in vitro 転写および翻訳反応(無細胞系)中にテストしました。RNAライトアップアプタマーおよびリボスイッチをコードするDNAプラスミドは、卵殻膜における蛍光標識RNA転写物またはGFPa1タンパク質の局在化中に視覚化されたように、対応するアナライトで正常に活性化されました。
概要
合成生物学の分野は、微生物によって進化した自然のメカニズムを利用して環境と潜在的な脅威を監視することにより、センシング機能を開発するユニークな機会を提供します。重要なことに、これらのセンシングメカニズムは通常、これらの微生物を有害な曝露から保護し、遺伝子発現を調節して悪影響を軽減したり、有毒物質の摂取を防止したりする応答に関連しています。これらの微生物を操作して、これらの自然な応答を利用して全細胞センサーを作成するが、新しいターゲットを認識したり、定量目的で測定できる測定可能なシグナルを生成したりするようにリダイレクトする多大な努力がなされてきました(通常は蛍光)1,2。現在、遺伝子組み換え微生物(GMO)の使用に関する懸念は、特に環境または人体に放出する場合、ポリマーマトリックスにカプセル化されていても、細胞全体またはその遺伝物質の一部の漏出により、これらのセンシングアプローチを利用する別の方法が必要であることを示唆しています3。
GMOの展開を心配することなく微生物ベースのセンシングの利点を活用するための強力なアプローチは、in vitro転写/翻訳(IVTT)システムの使用です。実用的な観点から、IVTTシステムは、超音波処理、ビーズビートなど、さまざまな手段で細胞から「抽出」された活性状態の細胞成分のほとんどを含む混合物で構成されています4。このプロセスの最終生成物は、転写と翻訳を実行するためにすでに最適化された生化学反応混合物であり、細胞全体の使用に関連する制約(膜拡散、形質転換効率、細胞毒性など)なしに、「オープンベッセル」フォーマットでさまざまなセンサーをテストするために使用できます。重要なのは、さまざまなセンサーコンポーネントを定量的に追加でき、その効果をさまざまな光学的および分光的手法で研究できることです(5)。IVTTシステムのパフォーマンスに一貫性がない可能性があることがわかっています。しかし、最近の研究では、それらの準備と特性評価を標準化するアプローチが示されており、センサー設計6での性能を研究する際に非常に役立ちます。最近、重金属イオン、薬物、クォーラムセンシング素子などの検出を含む、紙マトリックス中の成分の凍結乾燥を通じて紙ベースのアッセイを作成するために使用するIVTTシステムの多くの例が実証されています7,8,9。IVTTベースのセンサのエキサイティングなアプリケーションスペースは、土壌、水、人体など、さまざまなタイプの環境でのセンシングアプリケーションでの使用です。これらのIVTTシステムをこれらの困難な環境に展開するには、IVTTコンポーネントを封じ込め、劣化から保護するためのカプセル化アプローチを実装する必要があります。
IVTTシステムのための最も一般的なカプセル化アプローチには、脂質カプセル、ミセル、ポリマーソーム、および他の緊密に密閉されたマイクロ容器の使用が含まれる10、11、12。このアプローチの欠点の1つは、外部環境との通信を可能にし、センシング機能を提供するために、容器の内外に材料を輸送するためのパッシブまたはアクティブメカニズムのいずれかを組み込む必要があることです。これらの問題のいくつかを克服するために、この研究では、IVTTシステムで表現されるさまざまなセンサー設計のエンコード材料をカプセル化するためのシンプルで効果的なアプローチを提供する方法を報告します。このアプローチは、目的のプラスミドの存在下でバイオポリマーをレイヤーバイレイヤー(LbL)堆積して、高い多孔性の中空マイクロカプセルを作成することに基づいており、保護された遺伝物質が選択したIVTTのさまざまなコンポーネントと相互作用できるようにします。この研究は、カプセル化されたプラスミドが、プラスミドコードされたアプタマーとリボスイッチの対応する標的への応答で示されるように、このポリマーマトリックス内で活性化されると転写と翻訳を指示できることを実証しました。さらに、このLbLコーティングは、特別な保存条件なしでプラスミドを数ヶ月間保護します。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. プラスミドベクターの構築
- pJ201:23976-RS-GFPa1ベクター(DNA2.0で設計・作成)からGFPa1と結合したテオフィリンリボスイッチ(ThyRS)のコード配列を増幅し、 大腸菌 発現ベクターpSAL13に挿入することにより、プラスミドベクター(pSALv-RS-GFPa1、3.4 kb)を構築します。フォワード(5'-CGTGGTACCGGTGATACCAGCATCGTCTTGATG-3')およびリバース(5'-CGTGCTCAGCTTAAGCCAGCTCGTAG-3')プライマーを使用して、GFPa1と結合したThyRSのコード配列を増幅し、メーカーのプロトコル14に従ってDNAポリメラーゼを使用して50μL容量のPCR反応を実行します。
- 0.5 gのアガロース、50 mLのTAEバッファー(40 mM トリスアセテート、1 mM EDTA、pH 8.0)、および3 μLのDNA染色剤から1%アガロースゲルを調製します。
- 5 μLのPCR増幅産物を5 μLのRNase/DNaseフリー水と2 μLの6xゲルローディング色素と混合し、アガロースゲル電気泳動で分析します。参照としてDNAラダー(0.1〜10.0 kb)をロードします。色素ラインがゲルの底部にほぼ到達するまで、ゲルを120 Vで実行します。
- UVトランスイルミネーターイメージングシステムを使用してDNA断片を視覚化し、DNA15の正しいサイズを確認します。
- 製造者のプロトコル16に従ってPCR精製キットを使用してPCR産物を精製する。
- 10 μLのPCR産物またはプラスミドベクター(濃度20-50 ng/μL)、1.5 μLの10x酵素バッファー、1 μLの各酵素、および1.5 μLのRNase/DNaseフリー水を含む15 μLの反応で、PCR産物およびpSAL発現ベクターをKpnIおよびBlpI制限酵素で37°Cで2時間消化します。
- 3 μLの6xゲルローディング色素を反応混合物に加え、ステップ1.3〜1.5の説明に従って、消化されたフラグメントを1%アガロースゲル上で分離します。
- 製造業者のプロトコル16に従ってゲル抽出キットを使用してDNA断片を精製する。
- T4 DNAリガーゼと補充リガーゼバッファーを使用して、3〜20 fmolの消化ベクター、9〜60 fmolの消化PCR産物、2 μLのリガーゼバッファー、1 μL(1ユニット)のT4 DNAリガーゼ、およびDNase/RNaseフリーの水を含む10 μLの反応で、消化PCR産物を消化された線状化プラスミドベクターpSALにリゲートします。ライゲーション反応を25°Cで3時間インキュベートします。
注:反応混合物中の総DNA含有量が0.01〜0.1μgであることを確認してください。 - 大腸菌DH5αコンピテントセルを10 ngのライゲーション反応混合物で製造者のプロトコール17に従って形質転換する。
- 形質転換細胞をアンピシリン(100 μg/mL)を添加したLB-寒天プレート上で37°Cで一晩増殖させます。
- プレートから3〜4個の細菌コロニーを選び、それぞれをアンピシリン(100 μg/mL)を添加した5 mLのLB培地に無菌的に移します。培養物を225rpmの振とうインキュベーター内で37°Cで一晩増殖させる。
- 室温で3分間11 x g で遠心分離することにより、一晩培養物をペレット化します。
- 精製キットを使用して、製造業者のプロトコル16に従ってプラスミドを精製する。
- 精製されたプラスミドの配列をDNAシーケンシングによって検証します。得られたコンストラクト(pSALv-RS-GFPa1)のプラスミドマップおよび配列を 図1に示す。
2.大規模なDNA精製。
- プラスミドベクターpSALv-RS-GFPa1(3.4 kb)(GFPa1レポーター遺伝子と結合したテオフィリンリボスイッチをコードする)またはpET28c-F30-2xブロッコリー(5.4 kb)(ブロッコリーアプタマーをコードする)を、製造元のプロトコル17に従って大腸菌DH5αコンピテントセルに形質転換します。
- pSALv-RS-GFPa1で形質転換した細胞の場合はアンピシリン(100 μg/mL)を添加したLB-寒天プレート上で、pET28c-F30-2xブロッコリーで形質転換した細胞の場合はカナマイシン(50 μg/mL)で形質転換細胞を37°Cで一晩増殖させます。
- プレートから3〜4個の細菌コロニーを選び、各コロニーを適切な抗生物質(100 μg/mLアンピシリンまたは50 μg/mLカナマイシン)を添加した5 mLのLB培地に無菌的に移します。培養物を225rpmの振とうインキュベーター内で37°Cで一晩増殖させる。
- 3 mLの一晩培養液を使用して、適切な抗生物質(100 μg/mLアンピシリンまたは50 μg/mLカナマイシン)を添加した150 mLのLBに接種し、225 rpmの振とうインキュベーターで37°Cで一晩培養物を増殖させます。
- 細胞を≥3400 x g で4°Cで10分間遠心分離してペレット化します。
- 精製キットを使用して、製造業者のプロトコル16に従ってプラスミドを精製する。
- 0.5 mL の純粋な DNase/RNase フリー水で DNA を溶出します。DNA濃度を測定し、1 mLのDNAストック溶液(100 ng/μL)を調製します。さらに使用するまで、DNAを含むチューブを4°Cで保管してください。
3.絹フィブロインの抽出と初期材料の調製。
- Silk−LiBr溶液18の10%を占めるように他の箇所に詳述した手順に従って、Bombyx moriカイコの繭から再構成された絹フィブロイン(SF)タンパク質の水溶液を調製する。
- SF水溶液の終濃度を決定する。60 mmシャーレに0.5 mLのシルク溶液をピペットで入れ、60°Cで乾燥させ、乾燥シルクフィルムの重量を測定した。乾燥重量を0.5mLで割って、体積パーセンテージあたりの重量を計算します。
- 血清学的ピペットを介してゆっくりと水を加えて、濃縮シルク溶液をDNase/RNaseフリーの蒸留水で希釈し、最終濃度1 mg/mLを得ます。将来の使用のために溶液を4°Cで保管してください。
- 抗体標識キットを用いて蛍光標識された絹フィブロインを調製する。1 mLの2 mg/mLシルクフィブロイン溶液を使用して、製造元のプロトコル19に従って、タンパク質のN末端αアミノ基をNHSエステル活性化誘導体色素と結合させます。
- 濃度6 mg/mLのポリエチレンイミン(PEI)水溶液50 mLを調製し、HCl(1 M)でpHを4に調整します。溶液を滅菌0.2μmメンブレンでろ過します。周囲条件で数ヶ月間保管が可能です。
- SiO2 コアを準備します。300 μLのSiO 2粒子を2 mLの微量遠心チューブにピペットで入れます。0.2 x g で1分間遠心分離することにより、1 mLのDNase/RNaseフリー水で微粒子を2回洗浄します。
4.プライム層、DNAプラスミド、およびシルク層の層ごとの堆積を実行します。
- PEIプライム層をSiO2 微粒子上に堆積させるには、ステップ3.6からスピンダウンしたペレットに1 mLのPEI溶液を加え、サーモミキサー上で周囲条件で混合物を800rpmで15分間攪拌します。粒子を1 mLのDNase/RNaseフリー脱イオン水で0.2 x g で1分間遠心分離することにより4回洗浄します。
- DNA層の堆積を行うには、ステップ2.7のDNAプラスミドの水溶液1 mLをPEIプライミング微粒子に加え、サーモミキサー上で4°Cで800 rpmで15分間穏やかに攪拌します。異なるDNA負荷でマイクロカプセルを調製するには、DNase/RNaseフリーの蒸留水を使用してDNAプラスミドの濃度を50〜200 ng / μLに調整し、これらの溶液1 mLを使用してDNAを堆積させます。0.2 x g で1分間遠心分離して微粒子を収集します。
- GFPa1と結合したテオフィリンリボスイッチをコードするDNAプラスミドをThyRS-GFPa1として、ブロッコリーアプタマーをコードするDNAプラスミドをBrocAptとしてチューブにマークします。
注意: 微量遠心チューブをDNAとともに氷上に置いてください。 - 上清を注意深く除去し、DNase/RNaseフリーの蒸留水1 mLで微粒子を4回洗浄し、そのたびに0.2 x g で1分間遠心分離した後、上清を廃棄します。特に指定がない限り、すべての実験を室温(RT)で実行します。
- 絹フィブロイン層の堆積を行うには、DNA吸着微粒子に工程3.3で再構成したSF水溶液1mLを加え、穏やかにボルテックスし、混合物をサーモミキサー上で10°Cで750rpmで15分間攪拌する。0.2 x g で4°Cで1分間遠心分離して微粒子を回収し、上清を除去した後、1 mLのDNase/RNaseフリー蒸留水で1回洗浄します。遠心分離を繰り返し、上清を捨てる。
注:実験中は、温度によるゲル化を避けるために、絹の溶液を氷の上に保管してください。 - 粒子をメタノールで徐々に処理して、シルクタンパク質構造にβシート形成を誘導します。まず、0.5 mLのDNase / RNase蒸留水を加え、マイクロ遠心管をボルテックスしてから、0.5 mLの100%メタノールを加えます。サーモミキサーで粒子を10°Cで5分間静かに振とうします。0.2 x g で1分間遠心分離して粒子を収集します。上清を取り除きます。
- 粒子をメタノールで処理して、βシートの形成を促進し、シルク層の強力な物理的吸着を確保します。1mLの100%メタノールを加える。サーモミキサーで粒子を750rpmで10°Cで10分間静かに振とうします。
- 粒子を0.2 x g で4°Cで1分間遠心分離して回収し、毎回1 mLのDNase/RNaseフリー蒸留水で2回洗浄し、上清を廃棄して穏やかにボルテックスしてから次の遠心分離を行います。
- 手順4.5〜4.8を20回繰り返して、シルク多層コアシェル構造を得る。最後の堆積ステップでは、ステップ3.4の蛍光標識シルク(Silk-DyLight550、1 mL)を使用します。
- 最後の洗浄ステップを実行し、周囲条件で1 mLのDNase/RNaseフリー蒸留水に微粒子を保持します。
注:シルク層の堆積中の粒子の凝集を避けるために、粒子懸濁液の目視検査を行い、1 mLピペットチップを使用して上下にピペットして、均一な粒子分布を促進します。 - 式1を用いて各マイクロカプセルに封入されたDNAプラスミドコピー数、 NDNA の数を計算する:
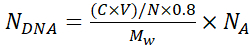 (1)
(1)
ここで、 N = 6.769 × 1011 - カプセル化に使用されるSiO2 コアの数。λ= 320 nmでの段階希釈と吸収A320 を使用して、既知の濃度のシリカ粒子の標準曲線から計算します。
C-吸着に用いるDNAの初期濃度
V-吸着に使用されるDNAの量
コアへの0.8-DNA吸着効率
Mw-DNAプラスミドの分子量
NA- アボガドロ数 (6.022 × 1023)
5.シルクマイクロカプセルを得るためのコアの溶解。
- 8%フッ化水素酸(HF)溶液(pH 5.5)を調製し、ストック溶液(48%)を蒸留水で希釈します。50 mL 遠心チューブを入手します。5 mLのHFを注意深くピペットし、25 mLの蒸留水を加えて8%HF溶液を得ます。
注意: HFは非常に腐食性の酸であり、組織に重度の火傷を引き起こす可能性があります。実験のためのHFの取り扱いおよび使用中は、細心の注意を払う必要があります。望ましくない流出事故を回避するために、組織が開発したHFの適切な使用と取り扱いについては、標準操作手順(SOP)を遵守してください。HF酸を希釈するためにガラス容器を使用しないでください。ケミカルフードを使用して、プロトコルのこの手順を実行します。 - ステップ4.10からペレット化されたコアシェル微粒子に1.5mLの8%HF溶液を加えてSiO2 コアを溶解します。穏やかにボルテックスし、450rpmで穏やかに振とうしながら、周囲条件でコアを一晩溶解させます。
注意: HFのこぼれを防ぐために、グラフトテープを使用してマイクロ遠心チューブをシールします。化学フードを使用して、プロトコルのこのステップを実行します。 - 2 Lの脱イオン水で満たされた2 Lのガラスビーカーを準備します。マイクロカプセル溶液を透析装置(50 kDa MWCO)に移し、3時間ごとに水を3日間繰り返し交換しながら脱イオン水に対して透析します。
注意: 最初の3回の水交換中に上清を収集し、有害廃棄物の確立されたプロトコルに従って溶液を廃棄します。 - 1 mLピペットを使用して、透析装置から新しい2 mLマイクロ遠心チューブに懸濁液を移し、マイクロカプセルを収集します。
注:マイクロカプセルの水溶液は、周囲条件で数年間保管してください。
6.共焦点レーザー走査顕微鏡(CLSM)を使用した絹フィブロインマイクロカプセルのイメージング。
- DNA色素を用いてDNA染色を行う。
- 300 μLの中空絹フィブロインマイクロカプセルを新しい1 mLマイクロ遠心チューブに移します。500 μLのRNase/DNaseフリー蒸留水を加えます。
- 5 μLのDNA染色色素を加え、短時間ボルテックスし、光から保護されたRTで2時間インキュベートします。
- 毎回0.1 x g で4°Cで20分間遠心分離し、400 μLの上清を注意深く除去し、400 μLのRNase/DNaseフリー蒸留水を補充することにより、4つの洗浄ステップを実行します。
- 100倍の油浸対物レンズ(NA 1.49)を使用して、3つの主要なレーザー(405 nm、488 nm、561 nm)を備えた倒立共焦点システムでシルクカプセルのイメージングを実行します。100 μLのカプセルサンプルを8ウェルチャンバーガラススライドの単一ウェルに移し、イメージング前にカプセルを20〜30分間沈降させます。
注:染料は光退色に非常に敏感です。スライドをアルミホイルで覆ってサンプルを保護します。
7. 分子量カットオフ(MWCO)法を用いた中空マイクロカプセルの透過性の推定
- 異なるMw(4 kDa、20 kDa、40 kDa、70 kDa、150 kDa、250 kDa、500 kDa、および2 MDa)のFITC標識デキストラン蛍光色素溶液(20 μM、diH2O)をそれぞれ2 mL調製します。
- カプセルの懸濁液100μLをチャンバー付きスライドガラスの単一ウェルにピペットで入れる。各マイクロカプセルの設計(PEIの濃度、DNAプラスミドの負荷数、絹フィブロインの濃度、および層数)を個別に分析します。
- 各ウェルに、最小M wから最高Mwまで300 μLの特定の蛍光色素溶液を添加し、各ウェルが特定の蛍光色素溶液に対応するようにします。上下にピペッティングして混合し、蛍光色素溶液の拡散が平衡に達するまでRTで1時間インキュベートします。
- スライドを共焦点レーザー走査型顕微鏡(CLSM)に移し、励起λ= 488 nmで100倍の油浸対物レンズを使用して各ウェルをイメージングします。
- 焦点面を調整して、カプセルが最大直径の円の形で表示されるようにして、関心のある領域を特定します。これは通常、カプセルが重力によって沈殿するときに、井戸の底に近いサンプルを表示するときに発生します。
- スライドをXY方向に動かしてマイクロカプセルサンプルの画像を複数収集します。サンプルごとに最大100〜150カプセルを占める画像をキャプチャします。
- ImageJソフトウェアを使用して、カプセルの内側と外側の蛍光強度を比較することにより、各MW 蛍光色素溶液中のカプセル膜の透過性を分析します。そのためには、関心領域(ROI)を円の形で描いてカプセルの円周の輪郭を描き、[ 分析/測定 ]をクリックして内部の蛍光強度を測定します。データをスプレッドシートに表にします。合計200〜300カプセルの各マイクロカプセルに対してこの操作を実行します。
- ROIの概要を説明し、カプセルから離れた強度を測定することにより、同じ方法で外部蛍光強度を評価します。統計分析のために3〜5回の測定を実行します。
- 統計解析を行うには、対応のある t検定(p < 0.05)を使用してカプセルの内側と外側の蛍光強度を比較します。
- 変換 表2 を使用して、可変 MwのFITC-デキストランの流体力学的半径に基づいてマイクロカプセルの透過性を推定します。
8. シルクマイクロカプセル中の合成テオフィリンリボスイッチの in vitro 活性化
- 1 mLのテオフィリンストック溶液(100 mM、DMSO)を調製します。氷上で成分を40分間解凍することにより、環状DNA用の 大腸菌 S30抽出物システムを調製します。
- 0.5 mL DNase/RNaseフリーのマイクロ遠心チューブを入手します。 in vitro 転写/翻訳反応を行い、無細胞成分をマイクロカプセルのサンプルと次の順序で組み合わせます(総容量50 μL):アミノ酸を含まないS30プレミックス(20 μL);S30抽出物、円形(15μL);完全アミノ酸混合物(5μL);ステップ4.10からのThyRS-GFPa1プラスミド(9μL)を含有する中空マイクロカプセル;テオフィリン、100 mM DMSO(1 μL)。
注:すべての成分を追加した後、チューブを短時間ボルテックスし、0.2 × g で数秒間短時間遠心分離中にサンプルを収集します。 - チューブを30°Cで4時間インキュベートし、λ=488 nmでの励起とGFP/FITCフィルター(510 nm±20 nm)の発光を使用して、プレートリーダーで蛍光を確認します。
- 488 nmおよび561 nmレーザーを使用して、任意のLCSMシステムでカプセルをイメージングします。100倍の油浸対物レンズと8ウェルチャンバースライドを使用して、最高品質の画像を取得します。
9. シルクマイクロカプセルにおけるブロッコリーアプタマーの in vitro 活性化
- DFHBI-1T色素(30 μM、diH2O)のストック溶液1 mLを調製します。PURE(組換え元素を用いたタンパク質合成)無細胞系反応キットを、成分を氷上で40分間解凍して調製します。
- 0.5 mL DNase/RNaseフリーのマイクロ遠心チューブを入手します。無細胞反応成分とマイクロカプセルのサンプルを次の順序で組み合わせることにより 、in vitro 転写反応を実行します(総容量50 μL):溶液A(20 μL);溶液B(15μL);ステップ4.10からのBrocAptプラスミド(14μL)を含む中空マイクロカプセル;DFHBI-1T色素(1μL)を使用します。
注:すべての成分を追加した後、チューブを短時間ボルテックスし、0.2 × g で数秒間短時間遠心分離中にサンプルを収集します。 - チューブを37°Cで6時間インキュベートし、λex = 470 nmでの励起とλem = 510 nm±20 nmでの発光を使用してプレートリーダーで蛍光を確認します。
- 488 nmおよび561 nmレーザーを使用して、任意のLCSMシステムでカプセルをイメージングします。100倍の油浸対物レンズと8ウェルカバーガラスチャンバースライドを使用して、最高品質の画像を取得します。
Access restricted. Please log in or start a trial to view this content.
結果
ここでは、絹タンパク質カプセルにカプセル化した後、さまざまなセンサー設計(2種類のRNA調節転写/翻訳要素)をコードするDNAテンプレートの機能について説明します。マイクロカプセルは、主要成分であるプライム層、センサー設計をコードするDNAプラスミド、および絹フィブロインバイオポリマーのテンプレート化されたレイヤーバイレイヤー(LbL)アセンブリを介して調製されました(
Access restricted. Please log in or start a trial to view this content.
ディスカッション
様々なタイプのDNAコードセンサー設計を装填した選択透過性ヒドロゲルマイクロカプセルは、このプロトコルに従って調製することができる。LbLアプローチの特徴の1つは、ボトムアップアセンブリ中にマイクロカプセルの複雑さを調整できることであり、これは通常、犠牲テンプレートへの分子種の吸着から始まります。初期成分の濃度、pH条件、および層数を注意深く調整することにより?...
Access restricted. Please log in or start a trial to view this content.
開示事項
ここに提示された見解および意見は著者のものであり、必ずしも国防総省またはそのコンポーネントの見解を表すものではありません。
謝辞
この研究は、空軍科学研究局からのLRIR 16RH3003J助成金、および米国国防次官研究工学事務所の軍事環境応用研究のための科学技術優先事項の進歩(ARAP)プログラムの支援を受けました。
ThyRSのプラスミドベクター配列(pSALv-RS-GFPa1、3.4 kb)は、J. Gallivan博士によって寛大に提供されました。 ボンビックスモリ のカイコの繭は、マサチューセッツ州タフツ大学のD.L.カプラン博士から寛大に寄贈されました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| (Z)-4-(3,5-difluoro-4-hydroxybenzylidene)-2-methyl-1-(2,2,2-trifluoroethyl)-1H-imidazol-5(4 H)-one (DFHBI-1T) | Lucerna | DFHBI-1T | |
| 5x T4 DNA Ligase Buffer | ThermoFisher Scientific | 46300-018 | |
| 6x Blue Gel Loading Dye | New England BioLabs | B7021S | |
| 96-well plates, black circular | Corning | 3601 | |
| Agarose | Sigma-Aldrich | A9539 | BioReagent, for molecular biology, low EEO |
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | powder or crystals, BioReagent, suitable for cell culture |
| BlpI restriction enzymes | New England BioLabs | R0585S | |
| Corning Disposable Vacuum Filter/Storage Systems | FisherScientific | 09-761-1 | |
| Dimethyl sulfoxide, DMSO | Sigma-Aldrich | 472301 | ACS reagent, ≥99.9% |
| DNA Plasmid, pET28c-F30-2x Broccoli (5.4 kb), BrocApt. | Addgene | Plasmid #66788 | |
| DyLightTM550 Antibody Labeling kit (Invitrogen) | ThermoFisher Scientific | 84530 | |
| E. coli S30 extract system for circular DNA | Promega | L1020 | |
| Falcon Conical centrifuge tubes, 15 mL | FisherScientific | 14-959-53A | |
| Falcon Conical centrifuge tubes, 50 mL | 14-432-22 | ||
| Fisherbrand Microcentrifuge tubes, 1.5 mL | FisherScientific | 05-408-129 | |
| Hydrofluoric acid, HF | Sigma-Aldrich | 695068 | ACS reagent, 48% |
| Kanamycin sulfate | Sigma-Aldrich | 60615 | mixture of Kanamycin A (main component) and Kanamycin B and C |
| KpnI restriction enzymes | New England BioLabs | R0142S | |
| LB agar plate supplemented with 100 µg/mL ampicillin | Sigma-Aldrich | L5667 | pre-poured agar plates with 100 µg/mL ampicillin |
| LB agar plate supplemented with 50 µg/mL kanamycin | Sigma-Aldrich | L0543 | pre-poured agar plates with 50 µg/mL kanamycin |
| LB broth (Lennox grade) | Sigma-Aldrich | L3022 | |
| Lithium bromide, LiBr | Sigma-Aldrich | 213225 | ReagentPlus, ≥99% |
| Max Efficiency DH5-α competent E. coli strain | ThermoFisher Scientific | 18258012 | |
| Methanol | MilliporeSigma | 322415 | anhydrous, 99.8% |
| MilliQ-water | EMD MilliPore | Milli-Q Reference Water Purification System | |
| MinElute PCR Purification Kit | Qiagen | 28004 | |
| N-(3-Dimethylaminopropyl)-N′-ethylcarbodiimide hydrochloride, EDC | Sigma-Aldrich | E1769 | |
| PBS (phosphate buffered saline) | ThermoFisher Scientific | 10010023 | 1x PBS, pH 7.4 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530S | |
| Polyethylenimine, branched | Sigma-Aldrich | 408727 | average Mw ~25,000 |
| PURExpress In Vitro Protein Synthesis Kit | New England BioLabs | E6800S | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| Quick-Load 2-Log DNA Ladder (0.1-10.0 kb) | New England BioLabs | N0469S | |
| SiO? silica microspheres, 4.0 µm | Polysciences, Inc. | 24331-15 | 10% aqueous solution |
| Slide-A-Lyzer G2 Dialysis Cassettes, 3.5K MWCO, 15 mL | ThermoFisher Scientific | 87724 | |
| Sodium carbonate, Na?CO? | Sigma-Aldrich | 222321 | ACS reagent, anhydrous, ≥99.5%, powder |
| Spectrum Spectra/Por Float-A-Lyzer G2 Dialysis Devices | FisherScientific | 08-607-008 | Spectrum G235058 |
| SYBR Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase (5 U/µL) | ThermoFisher Scientific | EL0011 | |
| Theophylline | Sigma-Aldrich | T1633 | anhydrous, ≥99%, powder |
| Tris Acetate-EDTA buffer (TAE buffer) | Sigma-Aldrich | T6025 | Contains 40 mM Tris-acetate and 1 mM EDTA, pH 8.3. |
| UltraPure DNase/RNase-Free Distilled Water | FisherScientific | 10-977-023 | |
| ZymoPURE II Plasmid MaxiPrep kit | ZymoResearch | D4202 |
参考文献
- Slomovic, S., Pardee, K., Collins, J. J. Synthetic biology devices for in vitro and in vivo diagnostics. Proceedings of the National Academy of Sciences of the United States of America. 112 (47), 14429-14435 (2015).
- Harbaugh, S. V., Goodson, M. S., Dillon, K., Zabarnick, S., Kelley-Loughnane, N. Riboswitch-based reversible dual-color sensor. ACS Synthetic Biology. 6 (5), 766-781 (2017).
- König, H., Frank, D., Heil, R., Coenen, C. Synthetic genomics and synthetic biology applications between hopes and concerns. Current Genomics. 14 (1), 11-24 (2013).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: An expanded repertoire of applications. Nature Reviews Genetics. 21, 151-170 (2020).
- Chushak, Y., et al. Characterization of synthetic riboswitch in cell-free protein expression systems. RNA Biology. , 1-12 (2021).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Grӓwe, A., et al. A paper-based, cell-free biosensor system for the detection of heavy metals and date rape drugs. PLoS One. 14 (3), 0210940(2019).
- Lin, X., et al. Portable environment-signal detection biosensors with cell-free synthetic biosystems. RSC Advances. 10 (64), 39261-39265 (2020).
- Caschera, F., Lee, J. W., Ho, K. K. Y., Liu, A. P., Jewett, M. C. Cell-free compartmentalized protein synthesis inside double emulsion templated liposomes with in vitro synthesized and assembled ribosomes. Chemical Communications. 52 (31), 5467-5469 (2016).
- Niederholtmeyer, H., Chaggan, C., Devaraj, N. K. Communication and quorum sensing in non-living mimics of eukaryotic cells. Nature Communications. 9, 5027(2018).
- Timin, A. S., Gould, D. J., Sukkhorukov, G. B. Multi-layer microcapsules: Fresh insights and new applications. Expert Opinion on Drug Delivery. 14 (5), 583-587 (2017).
- Bomati, E. K., Haley, J. E., Noel, J. P., Deheyn, D. D. Spectral and structural comparison between bright and dim green fluorescent proteins in Amphioxus. Scientific Reports. 4, 5469(2014).
- Frey, B. Amplification of Genomic DNA by PCR. Molecular Diagnosis of Infectious Diseases. Methods in Molecular Medicine. Reischl, U. 13, Humana Press. Totowa, NJ. 143-156 (1998).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923(2012).
- Zhou, Y., et al. Rapid regeneration and reuse of silica columns from PCR purification and gel extraction kits. Scientific Reports. 8, 12870(2018).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5α-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), 0137466(2015).
- Rockwood, D. N., et al. Materials fabrication from Bombyx mori silk fibroin. Nature Protocols. 6 (10), 1612-1631 (2011).
- Drachuk, I., et al. Silk macromolecules with amino acid-Poly(Ethylene Glycol) grafts for controlling layer-by-layer encapsulation and aggregation of recombinant bacterial cells. ACS Nano. 9 (2), 1219-1235 (2015).
- Antipov, A. A., Sukhorukov, G. B. Polyelectrolyte multilayer capsules as vehicles with tunable permeability. Advances in Colloid and Interface Science. 111 (1-2), 49-61 (2004).
- Drachuk, I., Harbaugh, S., Chávez, J. L., Kelley-Loughnane, N. Improving the activity of DNA-encoded sensing elements through confinement in silk microcapsules. ACS Applied Materials & Interfaces. 12 (43), 48329-48339 (2020).
- Melnikov, S., Ben-Shem, A., Garreau de Loubresse, N., Jenner, L., Yusupova, G., Yusupov, M. Structural basis for the inhibition of the eukaryotic ribosome. Nature Structural & Molecular Biology. 19 (6), 560-567 (2012).
- Zhao, S., et al. The future of layer-by-layer assembly: A tribute to ACS Nano associate editor Helmuth Möhwald. ACS Nano. 13 (6), 6151-6169 (2019).
- Main, K. H. S., Provan, J. I., Haynes, P. J., Wells, G., Hartley, J. A., Pyne, A. L. B. Atomic force microscopy-A tool for structural and translational DNA research. APL Bioengineering. 5, 031504(2021).
- Riera, R., Feiner-Gracia, N., Fornaguera, C., Cascante, A., Borrós, S., Albertazzi, L. Tracking the DNA complexation state of pBAE polyplexes in cells with super resolution microscopy. Nanoscale. 11 (38), 17869-17877 (2019).
- Bilokapic, S., Strauss, M., Halic, M. Cryo-EM of nucleosome core particle interactions in trans. Scientific Reports. 8, 7046(2018).
- Pritchard, E. M., Dennis, P. B., Omenetto, F., Naik, R. R., Kaplan, D. L. Physical and chemical aspects of stabilization of compounds in silk. Biopolymers. 97 (6), 479-498 (2012).
- Fritz, B. R., Jamil, O. K., Jewett, M. C. Implications of macromolecular crowding and reducing conditions for in vitro ribosome construction. Nucleic Acids Research. 43 (9), 4774-4784 (2015).
- Ge, X., Luo, D., Xu, J. Cell-free protein expression under macromolecular crowding conditions. PLoS One. 6 (12), 28707(2011).
- Cawte, A. D., Unrau, P. J., Rueda, D. S. Live cell imaging of single RNA molecules with fluorogenic mango II arrays. Nature Communications. 11, 1283(2020).
- Chen, X., et al. Visualizing RNA dynamics in live cells with bright and stable fluorescent RNAs. Nature Biotechnology. 37 (11), 1287-1293 (2019).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved