このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
スプライシング効率を監視するためのレポーターベースの細胞アッセイ
要約
このプロトコルは、スプライシングに対する5'-スプライス部位変異の影響をモニターするためのミニジーンレポーターアッセイを記述し、変異誘発スプライシング阻害のレスキューのためのサプレッサーU1 snRNAを開発する。レポーターおよびサプレッサーU1 snRNA構築物をHeLa細胞で発現させ、スプライシングをプライマー伸長またはRT-PCRによって分析する。
要約
遺伝子発現中、プレmRNAスプライシングの重要なステップは、スプライス部位の正確な認識と、成熟mRNAの細胞質的輸出前にエクソンを結合し、イントロンを除去するためのスプライソソーム複合体の効率的な組み立てを含む。スプライシング効率は、スプライス部位における変異の存在、トランス作用型スプライシング因子の影響、または治療薬の活性によって変化させることができる。ここで、任意の所与のエクソンのスプライシング効率をモニターするために適用することができる細胞アッセイのためのプロトコルを記載する。アッセイは、3-エクソン/2-イントロンミニジーンレポーターにコードされる適応性プラスミドを使用し、これは一過性トランスフェクションによって哺乳動物細胞で発現させることができる。トランスフェクション後、全細胞RNAが単離され、レポーターmRNAにおけるエクソンスプライシングの効率は、プライマー伸長または半定量的逆転写酵素 - ポリメラーゼ連鎖反応(RT-PCR)のいずれかによって決定される。我々は、5'スプライス部位変異に関連する疾患の影響が、レポーターにそれらを導入することによってどのように決定され得るかを記述します。これらの変異の抑制が、プレmRNAのエキソン-イントロン接合部の5'-スプライス部位と塩基対する5'領域に代償性変異を有するU1小型核RNA(snRNA)構築物との同時トランスフェクションによって達成され得る方法。したがって、レポーターは、変異体5'スプライス部位の認識を改善するための治療用U1粒子の設計に使用することができる。スプライシングエンハンサーまたはサイレンサー配列などの シス作用調節部位のレポーターへの挿入は、特定の選択的スプライシング因子によって媒介される調節におけるU1 snRNPの役割を調べるためにも使用され得る。最後に、レポーター発現細胞を小分子と共にインキュベートして、構成的プレmRNAスプライシングまたは変異体5'スプライス部位を有するエクソンに対する潜在的な治療薬の効果を決定することができる。全体として、レポーターアッセイは、基本的なスプライシングメカニズムおよびスプライシング関連疾患を研究するために、様々な条件下でのスプライシング効率をモニターするために適用することができる。
概要
プレmRNAスプライシングは、非コードイントロンを除去し、コードエクソンを正確にライゲーションして成熟mRNAを形成する重要なプロセシングステップです。5'-スプライス部位および3'-スプライス部位と呼ばれるエクソン-イントロン接合部におけるコンセンサス配列の認識は、スプライシング機構の構成要素によって開始される。U1小核リボヌクレオタンパク質(snRNP)は、U1 snRNAとプレmRNA1との塩基対形成によって5'-スプライス部位を認識する。5'スプライス部位配列を変化させる遺伝的に遺伝する変異は、多くの疾患と関連している2,3。U1 snRNAと変異型5'スプライス部位との塩基対形成が失われると、異常なスプライシングが発生し、影響を受ける転写産物の翻訳が損なわれる可能性があると予測されています。スプライシング欠損を補正するための潜在的な治療的アプローチは、5'-スプライス部位と塩基対するその5'-領域に代償性ヌクレオチド変化を有する修飾U1 snRNAによる変異の抑制を含む。エクソン特異的U1 snRNAとも呼ばれるこのような修飾U1 snRNAは、スプライシング欠陥を逆転させるのに有効であり、救助されたmRNA4、5、6、7、8からのタンパク質発現の増加をもたらすことが見出されている。
ここでは、エクソンのスプライシングに対する5'-ss変異の効果の評価を可能にし、エクソンインクルージョンのレスキューを可能にする修飾U1 snRNAの開発にも使用できるレポーターベースの細胞スプライシングアッセイであるU1 snRNP相補アッセイについて説明します。また、プライマー伸長およびRT-PCRによるスプライシングされたレポーター転写産物のモニタリング、およびプライマー伸長およびRT-qPCRによる修飾U1 snRNAの発現を決定するためのプロトコルも提供しています。
プロトコル
1. 試薬およびバッファー
注:真空フィルターを使用した滅菌はすべて、バイオセーフティキャビネット内の0.2μmポリエーテルスルホン(PES)膜で行う必要があります。
- 1.0Lの脱イオン水にジエチルピロカーボネート(DEPC)1.0mLを加えてRNase非含有水を調製し、室温(RT)で少なくとも1時間混合し、オートクレーブを2回、次いでRTに冷却してから使用前にRTに冷却する。
- DMEM粉末(13.4g)、炭酸水素ナトリウム3.7g、ウシ胎児血清(FBS)100mL、ペニシリン、ストレプトマイシン1パケットを約800mLの滅菌脱イオン水に混合して、ダルベッコの改変イーグル培地(DMEM)を調製する。DMEM中のペニシリンおよびストレプトマイシンの最終濃度は、それぞれ50U/mLおよび50μg/mLでなければならない。pHを7.4に調整し、滅菌脱イオン水で体積を1.0Lにします。ろ過により滅菌し、4°Cで保存する。
- 25.6gの水素二ナトリウム7水和物(Na2HPO4·7H2O)、リン酸二水素カリウム(KH2PO4)2g、塩化カリウム(KCl)2g、および塩化ナトリウム(NaCl)80gを800mLの脱イオン水に含む。混合して溶解し、体積を1.0Lに構成し、濾過によって滅菌し、4°Cで保存する。
- 186.1gのNa2•EDTA•2H2Oを約800mLの脱イオン水に溶解して、0.5Mエチレンジアミン四酢酸(EDTA)を調製する。pHを8.0に調整し、滅菌脱イオン水で体積を1.0Lにします。ろ過により滅菌し、4°Cで保存する。
- 100 mL の 10x トリプシン (2.5%)、2 mL の 0.5 M EDTA を混合して 1x トリプシン-EDTA 溶液を調製し、1 x PBS を 1.0 L まで加えます。4°Cで1~2週間保存するか、-20°Cで凍結して長期間使用してください。
- 14.4 mL のホルムアミドと 0.6 mL の 0.5 M EDTA を混合して、2x ホルムアミド DNA/RNA ローディング色素を調製し、最終容量を 15 mL にします。ブロモフェノールブルーとキシレンシアノール粉末を終濃度0.02%となるように加え、4°Cで保存した。
注:ホルムアミドは有毒で腐食性です。その他の安全に関する推奨事項については、製品安全データシートをお読みください。 - 54.0 g のトリス塩基、27.5 g のホウ酸、および 20 mL の 0.5 M EDTA を約 800 mL の脱イオン水に混合して、5x Tris/ホウ酸塩/EDTA (5x TBE) バッファーを調製します。混合して溶解し、脱イオン水で体積を1.0Lにします。
- 5x TBE200mL、40%19:1ビス/アクリルアミド250mL、および尿素450.5gを混合して、尿素-ポリアクリルアミドゲル電気泳動(urea-PAGE)溶液を調製する。その後、1.0Lまでの脱イオン水を加え、成分が完全に溶解するまで混合し、濾過により滅菌し、琥珀色のガラス瓶に4°Cで保存する。
注:ビス/アクリルアミドは有毒です。その他の安全手順については、製品安全データシートをお読みください。 - 10mLの脱イオン水に1gのAPSを溶解して10%過硫酸アンモニウム(APS)を調製し、4°Cで保存する。
2. HeLa細胞とレポーターおよびU1 snRNAプラスミドとのコトランスフェクション
注: Hela 細胞のトランスフェクションは、生物学的安全キャビネット内の無菌条件下で行う必要があります。すべての材料の外面には、生物学的安全キャビネットに導入する前に70%エタノールを噴霧する必要があります。
- 細胞が約80〜90%コンフルエントであるときに2〜3日ごとに継代することによって、5%CO2 を有する37°Cインキュベーター内のDMEM中のHela細胞を維持する。
- HeLa細胞を継代するために、使用済み培地を吸引し、次いで細胞を1mM EDTAを含む0.25%トリプシン3mLと共に37°Cで3分間インキュベートする。
- インキュベーション後、7mLの新鮮なDMEMを加える。細胞懸濁液を10mLチューブに移し、1,000 x g で5分間遠心分離する。
- 細胞ペレットを10mLの新鮮なDMEMに再懸濁し、次いで細胞を20%コンフルエントで新しい組織培養皿上にプレートする。
- 一過性トランスフェクションの場合は、クリーンな血球計数スライドで Hela 細胞をカウントし、密度 2.5 x 105 cells/mL の懸濁液を調製します。
- 12ウェルプレートの各ウェルに2.5 x 105 細胞/mLのHela細胞懸濁液1.0 mLをシードし、37°Cで一晩インキュベートする。
- 翌日、使用済み培地を吸引し、血清と共に0.8mLの新鮮なDMEMを加える。
- 0.2 μg の Dup51 または Dup51p レポータープラスミド、1.8 μg の pcDNA、pNS6U1、または pNS6U1-5a プラスミド、および 100 μL のトランスフェクション培地を新しい 1.5 mL マイクロ遠心チューブに添加して、溶液 I を調製します。
- サンプルあたり 100 μL のトランスフェクション培地と 4.0 μL のトランスフェクション試薬を添加して、トランスフェクションするすべてのサンプルの Solution II のマスターミックスを調製します。
- 溶液 I を含む各微量遠心チューブに 100 μL の溶液 II を加えてトランスフェクションミックスを調製します。
- トランスフェクションを 15 秒間ボルテックス混合し、卓上型微量遠心分離機で 3,000 x g で RT で 10 秒間遠心分離し、RT で 5 分間インキュベートします。
- トランスフェクションミックス 200 μL すべてを 12 ウェルの HeLa 細胞プレートの 1 ウェルに追加して、1 ウェルあたり 1.0 mL の最終容量を達成し、プレートを 37 °C で 48 時間インキュベートします。
- インキュベーション後、市販のチオシアン酸グアニジンおよびフェノール溶液でトランスフェクトしたHeLa細胞からRNAを抽出する。
注:この試薬にはフェノールが含まれており、この手順はヒュームフード内で行う必要があります。抽出されたRNAの再懸濁にはDEPC処理水の使用が推奨される。- 使用済みの培地を吸引し、各ウェルに500μLの試薬を加える。RTで5分間インキュベートする。
- 上下にピペッティングして均質化します。次いで、この溶液を新しい1.5mL微量遠心管に移す。
- 100 μLのクロロホルムとボルテックスを15秒間加える。
- RTで15分間、12,000 x g で遠心分離機。
- 200 μLのRNAを含む最上部水層を新しい1.5 mLマイクロ遠心チューブに移す。
- 各RNAサンプルに2μgのグリコーゲンおよび200μLのイソプロパノールを加える。チューブを反転させて混合します。
- RNA沈殿物を12,000 x g で4°Cで10分間遠心分離して回収する。
- RNAペレットを乱すことなく上清を除去して廃棄する。
- 1.0 mLの70%エタノールを加え、チューブを反転させ、ステップ2.13.7に記載したように遠心分離することによってペレットを2回洗浄する。
- ペレットをRTで約10~20分間空気乾燥させ、RNAを10~20μLのRNase非含有水に再懸濁します。
- DesjardinsおよびConklin9によって記述されているように260nmでの吸光度を測定することによってRNA濃度を決定する。
- プライマー伸長を進めるか、RNAサンプルを-20°Cで保存する。 単離されたRNAは、-20°Cで6〜12ヶ月間保存することができる。
オリゴヌクレオチドの 32P標識
注:32P-ATPおよび32P標識オリゴヌクレオチドの使用を含むステップは、認可された機関団体からの承認を得て、訓練を受けた個人によってアクリルシールドの後ろで実行されなければならない。以下に記載するプロトコールは、オリゴヌクレオチド、Dup3rおよびU17−26−R、ならびに尿素−PAGEのマーカーの標識に使用することができる。DEPC処理水の使用は、オリゴヌクレオチドおよびサイズ排除ビーズの再懸濁に推奨される。
- 1.5 mL マイクロ遠心チューブに、表 1 に記載したようにオリゴヌクレオチド、T4 ポリヌクレオチドキナーゼ (T4 PNK)、T4 PNK バッファー、および 32P-ATP を追加します。混合物に最後に32P-ATPを加える。これは重要です。
注:放射性溶液を添加する場合は、フィルターチップの使用をお勧めします。 - 37°Cの水浴中で30分間インキュベートする。
- 標識反応がインキュベートされている間、約10秒間穏やかにボルテックスすることによって遮断された分子量25kDaのサイズ排除ビーズを再懸濁する。
注:サイズ排除ビーズは、製造元の指示に従って調製し、4°Cで25%エタノール中の50%懸濁液として保存する必要があります。 - 1.5 mL 回収管に入れた使い捨てミニカラムに再懸濁ビーズ 600 μL を移し、ビーズストックを 4 °C に戻してカラムを作製します。
- RTで2,000 x g で1分間遠心分離し、流れを破棄します。
- カラムに 300 μL の RNase フリー水を加えてビーズを洗浄します。
- 手順 3.5 を繰り返します。および 3.6.ミニカラムを2回、新しい1.5 mL遠沈管に移します。
- キナーゼ反応混合物をサイズ排除ビーズカラムに加え、RTで5000 x g で1分間遠心分離します。
- 32P標識オリゴヌクレオチドを含むフロースルーを収集して保存し、すべてのチップとチューブをアクリル廃棄物箱に捨てます。
- RNase非含有水20 μLを加え、 32P標識オリゴヌクレオチドを最終濃度2.5 μMに希釈する。
注: 尿素-PAGEゲルへのローディングのために必要に応じて標識マーカーを希釈する。 - 標識オリゴヌクレオチドを-20°Cのアクリルマイクロチューブボックスに保存するか、プライマー伸長解析を続行する。
プライマー伸長によるスプライスされたレポーター転写産物の解析
注:使用前にRNase不活性化試薬で表面や機器を清掃することをお勧めします。
- Hela細胞から抽出した2.0 μgのRNAを別々の200 μLの微量遠心チューブに加え、RNaseフリーの水を加えて体積を6.55 μLにします。
- 表2に示すように希釈した32P-Dup3rおよびdNTPsでマスターミックスIを調製し、RNAサンプルを含む各PCRチューブに0.9 μLのミックスを加える。
- チューブを最初に65°Cで5分間、次に氷上で1分間インキュベートする。
- 表2に示すように、5xファーストストランドバッファー、ジチオスレイトール(DTT)、RNase阻害剤、および逆転写酵素でマスターミックスIIを調製します。
- RNAおよびマスターミックス1を含む各チューブに2.55 μLのミックスを加える。反応の全容量は10 μLでなければならず、チューブをRTに10分間保管する。
- チューブを乾燥浴またはサーマルサイクラーに移し、まず50°Cで60分間、次に70°Cで15分間インキュベートする。
- インキュベーション後、各サンプルに2xホルムアミドRNAローディング色素を10 μL加え、-20°Cのアクリルボックスに保存するか、長さ14 cmのゲルを使用して尿素-PAGEによるフラグメントの分離を進め、ステップ8で後述するようにゲル画像を視覚化します。
- 画像解析ソフトウェアでゲル画像の濃度測定スキャンを行い、含まれる生成物とスキップされた生成物のバンド強度を使用して、以下に示すようにエクソン2含有の割合を計算します。

5. 蛍光RT-PCRによるスプライスされたレポーター転写産物の解析
注: 以下に説明する RT-PCR 解析では、cDNA 合成にランダム 6 量体を使用し、スプライシングされた産物の PCR 増幅に非標識 Dup8f および Cy5 標識 Dup3r オリゴヌクレオチドを組み合わせて使用します。
- cDNA合成のために、トランスフェクトしたHela細胞から抽出した2.0 μgのRNAを別々の200 μLの微量遠心チューブに加え、RNaseフリーの水を加えて体積を11.0 μLにします。
- 表3に示すようにランダムヘキサマーとdNTPを含むマスターミックスIを調製し、各サンプルに2.0 μLのミックスを加えます。まず65°Cで5分間、次に氷上で1分間インキュベートする。
- 表3に示すように、ファーストストランドバッファー、RNase阻害剤、DTT、逆転写酵素を含むマスターミックスIIを調製し、RNAおよびマスターミックスIを含む各チューブに7.0 μLのミックスを加える。
- チューブをRTで10分間保持し、その後、50°Cで60分間、70°Cで15分間インキュベートします。
注:完了したcDNA反応は、-20°Cで保存することができる。 - PCR の場合は、各 cDNA サンプルの 1.0 μL (100 ng/μL) を新しい PCR チューブに移します。
- 表4に記載したように、Dup8f、Cy5-Dup3r、dNTP、Taq緩衝液、Taqポリメラーゼ、および水からなるマスターミックスを調製し、cDNAを含む各チューブに11.5 μLのミックスを加える。
- 初期変性ステップを94°Cで3分間行うサーマルサイクラーを用いてPCRを行う。続いて、変性(94°Cで30秒)、アニーリング(65°Cで30秒)、伸長(72°Cで15秒)、終了工程を72°Cで5分間20サイクル行った。
- 各チューブに12.5 μLの2xホルムアミドDNAローディング色素を加え、95°Cで5分間加熱する。
- PCR反応を-20°Cで保存するか、またはステップ8.1-8.4で後述するように尿素-PAGEで進行させる。
- 電気泳動後、ガラス板を電気泳動装置から取り出し、蛍光イメージャーを用いてスキャンし、ゲルを可視化した。
- ゲル画像の濃度測定スキャンを実行し、ステップ4.8で説明したように、含まれる生成物とスキップされた生成物のバンド強度を使用して、エクソン2包接の割合を計算します。
プライマー伸長による変異型U1 snRNA発現の解析
- Hela細胞から抽出した2.0 μgのRNAを別々の200 μLの微量遠心チューブに加え、RNaseフリーの水を加えて体積を4.325 μLにしてから、表5に示すようにdATPを加えます。
- 各チューブに10,000CPMの32P-U17-26-Rオリゴヌクレオチドを加える。
注:32P-U17-26-Rの10,000 cpm/μL溶液を調製するには(ステップ3から)、標識オリゴヌクレオチドを希釈し(1:20希釈)、シンチレーションカウンターを使用して1.0 μLでcpmを測定し、さらに脱イオン水で希釈して新しい1.5 mLマイクロ遠心分離管で10,000 cpm/μLの溶液を調製します。 - 65°Cで5分間インキュベートし、次いで氷上で1分間インキュベートする。
- 表5に示すように、5x First Strand Buffer、RNase 阻害剤、DTT、および逆転写酵素でマスターミックスを調製し、各サンプルに 1.8 μL のミックスを追加します。
- 最初にRTで10分間、次いで42°Cで10分間インキュベートする。
- インキュベーション後、各サンプルに10 μLの2xホルムアミドRNAローディング色素を添加し、-20°Cのアクリルボックスに保存するか、長さ38cmのゲルを使用して尿素-PAGEによるフラグメントの分離を進めます(ステップ8を参照)。
7. RT-qPCRによるバリアントU1 snRNA発現の解析
- 上記の手順5.1~5.4で調製したcDNAストックを0.2ng/μLの濃度に希釈する。
- 5.0 μL の希釈 cDNA を 96 ウェル qPCR プレートの個々のウェルに 3 連でピペットで送ります。非テンプレート制御(NTC)のためにcDNAの代わりに脱イオン水を加える。
- 表6に示すように、U1およびU2 snRNAsのフォワードプライマーおよびリバースプライマー、ならびに水からなる2つの別々のプライマーミックスを調製する。
注:U1およびU2 snRNA特異的プライマーの配列は 、表7に提供される。 - 5.0 μLのU1およびU2 snRNAプライマーミックスをサンプルおよびNTCウェルに加える。
- 10.0 μL のリアルタイム PCR ミックスを各ウェルに加えます。
- プレートを光学フィルムで密封し、RTで1,000 x g で2分間遠心分離し、ウェルの底部への反応を収集します。
- 標的アンプリコンの閾値定量サイクル(Cq)値を収集しながら、95°Cで10分間、変性(95°Cで15秒)とアニーリング/伸長(62°Cで60秒)からなる2ステッププロトコルを40サイクル実行してqPCRを実行します。
- U1およびU2 snRNA反応の解離曲線に単一のピークがないか確認して、qPCR反応を終了します。
- Cq値から、pcDNAコントロールと比較したU1およびU2 snRNAのデルタCq(ΔCq)を計算する。
- バリアント U1 snRNA 発現を、すべてのサンプルについて以下に示すように ΔΔCq 値を使用して、U2 と比較した U1 の相対量 (RQ) として決定します。

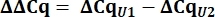
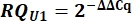
8. 尿素-PAGEゲルのセットアップと実行
注:ガラス板とゲル走行装置の組み立ては、製造元の指示に従って行う必要があります。10%尿素−PAGEゲルのキャスティングは、Summer et al.10によって以前に記載されたプロトコールに従って行うことができる。マーカーおよびサンプルの調製、ならびにゲルの実行および可視化を含むステップを以下に記載する。任意選択で、ゲルがガラス板に付着するのを防ぐために、内表面は、表面上に1mLの溶液を添加し、組織と共に全面に均等に広がることによって、シリコーン溶液でコーティングされ得る。乾燥したら、プレートを脱イオン水で洗浄し、再び乾燥させる必要があります。
警告: 未重合アクリルアミドは神経毒性があり、製品安全データシートで推奨されている保護具で取り扱う必要があります。
- プライマー伸長サンプルおよびマーカーを、95°Cで5分間加熱した後、卓上型微量遠心機で3,000 x g でRTで5秒間遠心分離することによって調製する。
- マーカーとサンプルをロードする前に、1x TBEバッファーでウェルを洗い流し、沈降した尿素を除去します。
- 10 μL/サンプル/ウェルをロードし、ゲルを300-500Vで2〜3時間、またはキシレンシアノールが底に達するまで実行します。
メモ: 約 1,000 cpm の 32P ラベルのマーカーをロードできます。 - 電気泳動後、ガラス板を電気泳動装置から取り出した。
- ゲルがいずれかのガラス表面に平らになるように2つのプレートを慎重に分離し、ゲルをろ紙に移し、プラスチックラップで覆います。
- ろ紙上のゲルをゲル乾燥機を用いて80°Cで30分間真空乾燥する。
- 乾燥ゲルを蛍光体イメージングカセットに入れ、RTで一晩保管する。
メモ:蛍光体の画面は、使用する前にライトボックスを使用して消去する必要があります。 - ゲル画像を視覚化するには、スクリーンを取り外し、蛍光体イメージャーを使用してスキャンします。
結果
スプライシングレポーターDup51は、3つのエクソン−2イントロンミニ遺伝子であり、ヒトβグロビン遺伝子に由来し、以前に記載した(図1A)11、12。我々は、プロトカドヘリン15(PCDH15)遺伝子のエクソン3に生じる5'-スプライス部位変異に関連するアッシャー症候群を導入することにより、変異レポーターDup51pを?...
ディスカッション
このアッセイは、HeLa以外の細胞株におけるスプライシング分析に適合させることができますが、細胞のコンフルエンシーやDNAの量など、トランスフェクション効率に影響を与える要因を最適化する必要がある場合があります。レポーター対U1構築物比は、他の細胞型で観察される発現レベルに応じて決定する必要があるかもしれない別の重要なパラメータである。抽出されたRNAの品質は、ス?...
開示事項
著者らは開示するものは何もありません。
謝辞
この研究は、国立衛生研究所(R21CA170786およびR01GM127464)および米国癌協会(Institutional Research Grant 74-001-34-IRG)からのS.S.およびW..Mへの資金と、Valley Research Partnership Program(P1-4009およびVRP77)からのS.S.およびW.への資金によって支援された。コンテンツは著者の責任であり、必ずしも国立衛生研究所の公式見解を表すものではありません。
資料
| Name | Company | Catalog Number | Comments |
| Reagent Grade Deionized Water | ThermoFisher Scientific | 23-751628 | |
| Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758-25ML | |
| Dulbecco's Modified Eagle Medium (DMEM) powder packet | Gibco | 12100-046 | |
| Sodium Bicarbonate | ThermoFisher Scientific | S233-500 | |
| Fetal Bovine Serum (FBS), Australian Source, Heat Inactivated | Omega Scientific | FB-22 | |
| Penicillin-Streptomycin (P/S) | Sigma-Aldrich | P4458-100ML | |
| Sodium Hydroxide, Standard Solution 1.0N | Sigma-Aldrich | S2567-16A | |
| Hydrochloric Acid, Certified ACS Plus, 36.5 to 38.0% | ThermoFisher Scientific | A144-500 | |
| Disposable PES Bottle Top Filters | ThermoFisher Scientific | FB12566510 | |
| EDTA Disodium Salt Dihydrate | Amresco | 0105-2.5KG | |
| 2.5% Trypsin (10x), no phenol red | ThermoFisher Scientific | 15090046 | |
| Sodium Chloride | Fisher Bioreagent | BP358-212 | |
| Potassium Chloride | Fisher Bioreagent | BP366-1 | |
| Disodium Hydrogen Phosphate Heptahydrate | Fisher Bioreagent | BP332-1 | |
| Potassium Dihydrogen Phosphate | Fisher Bioreagent | BP362-1 | |
| Transfection medium - Opti-MEM™ I Reduced Serum Medium, no phenol red | ThermoFisher Scientific | 11058021 | |
| Transfection Reagent - Lipofectamine™ 2000 | ThermoFisher Scientific | 13778150 | |
| TRIzol™ Reagent | ThermoFisher Scientific | 15596018 | |
| Chloroform (Approx. 0.75% Ethanol as Preservative/Molecular Biology) | ThermoFisher Scientific | BP1145-1 | |
| Ethanol, Absolute (200 Proof), Molecular Biology Grade, Fisher BioReagents | ThermoFisher Scientific | BP2818-4 | |
| Isopropanol, Molecular Biology Grade, Fisher BioReagents | ThermoFisher Scientific | BP2618-212 | |
| Glycogen (5 mg/ml) | ThermoFisher Scientific | AM9510 | |
| Direct-zol RNA Miniprep Kit | Zymo Research | R2052 | |
| ATP, [γ-32P]- 6000Ci/mmol 150mCi/ml Lead, 1 mCi | PerkinElmer | NEG035C001MC | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| Size exclusion beands - Sephadex® G-25 | Sigma-Aldrich | G2580-10G | |
| Size exclusion mini columns | USA Scientific | 1415-0600 | |
| pBR322 DNA-MspI Digest | New England Biolabs | N3032S | |
| Low Molecular Weight Marker, 10-100 nt | Affymetrix | 76410 100 UL | |
| Rnase inactivating reagents - RNaseZAP™ | Sigma-Aldrich | R2020-250ML | |
| dNTP Mix (10 mM ea) | ThermoFisher Scientific | 18427013 | |
| RNaseOUT™ Recombinant Ribonuclease Inhibitor | ThermoFisher Scientific | 10777019 | |
| Reverse Transcriptase - M-MLV Reverse Transcriptase | ThermoFisher Scientific | 28025013 | used for primer extension |
| Taq DNA Polymerase | ThermoFisher Scientific | 10342020 | |
| Random Hexamers (50 µM) | ThermoFisher Scientific | N8080127 | |
| Real time PCR mix - SYBR™ Select Master Mix | ThermoFisher Scientific | 4472903 | |
| SuperScript™ III Reverse Transcriptase | ThermoFisher Scientific | 18080093 | used for cDNA preparation |
| Dithiothreitol (DTT) | ThermoFisher Scientific | 18080093 | |
| 5X First-Strand Buffer | ThermoFisher Scientific | 18080093 | |
| Formamide (≥99.5%) | ThermoFisher Scientific | BP228-100 | Review Material Safety Data Sheets |
| Bromophenol Blue sodium salt | Sigma-Aldrich | 114405-5G | |
| Xylene Cyanol FF | Sigma-Aldrich | 2650-17-1 | |
| Tris Base (White Crystals or Crystalline Powder/Molecular Biology) | ThermoFisher Scientific | BP152-5 | |
| Boric Acid (Crystalline/Electrophoresis) | ThermoFisher Scientific | BP168-500 | |
| Acrylamide: Bis-Acrylamide 19:1 (40% Solution/Electrophoresis) | ThermoFisher Scientific | BP1406-1 | Review Material Safety Data Sheets |
| Urea (Colorless-to-White Crystals or Crystalline Powder/Mol. Biol.) | ThermoFisher Scientific | BP169-212 | |
| Ammonium peroxodisulphate (APS) ≥98%, Pro-Pure, Proteomics Grade | VWR | M133-25G | |
| Sigmacote | Sigma-Aldrich | SL2-100ML | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) ≥99%, Ultrapure | VWR | 0761-25ML | Review Material Safety Data Sheets |
| Adjustable Slab Gel Systems, Expedeon | VWR | ASG-400 | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 14.5cm | VWR | NGP-125NR | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 22.0cm | VWR | NGP-200NR | |
| Vertical Gel Wrap™ Glass Plate Sets, 16.5 x 38.7cm | VWR | NGP-400NR | |
| GE Storage Phosphor Screens | Sigma-Aldrich | GE28-9564 | |
| Typhoon™ FLA 7000 Biomolecular Imager | GE Healthcare | 28-9610-73 AB | |
| Beckman Coulter LS6500 Liquid Scintillation Counter | GMI | 8043-30-1194 | |
| C1000 Touch Thermal Cycler | ThermoFisher Scientific | ||
| QuantStudio 6 Flex Real-Time PCR Systems | ThermoFisher Scientific |
参考文献
- Zhuang, Y., Weiner, A. M. A compensatory base change in U1 snRNA suppresses a 5' splice site mutation. Cell. 46 (6), 827-835 (1986).
- Scotti, M. M., Swanson, M. S. RNA mis-splicing in disease. Nature Review Genetics. 17 (1), 19-32 (2016).
- Ward, A. J., Cooper, T. A. The pathobiology of splicing. Journal of Pathology. 220 (2), 152-163 (2010).
- Scalet, D., et al. Disease-causing variants of the conserved +2T of 5' splice sites can be rescued by engineered U1snRNAs. Human Mutatation. 40 (1), 48-52 (2019).
- Yamazaki, N., et al. Use of modified U1 small nuclear RNA for rescue from exon 7 skipping caused by 5'-splice site mutation of human cathepsin A gene. Gene. 677, 41-48 (2018).
- Yanaizu, M., Sakai, K., Tosaki, Y., Kino, Y., Satoh, J. I. Small nuclear RNA-mediated modulation of splicing reveals a therapeutic strategy for a TREM2 mutation and its post-transcriptional regulation. Science Reports. 8 (1), 6937 (2018).
- Balestra, D., et al. Splicing mutations impairing CDKL5 expression and activity can be efficiently rescued by U1snRNA-based therapy. International Journal of Molecular Sciences. 20 (17), 20174130 (2019).
- Donadon, I., et al. Exon-specific U1 snRNAs improve ELP1 exon 20 definition and rescue ELP1 protein expression in a familial dysautonomia mouse model. Human Molecular Genetics. 27 (14), 2466-2476 (2018).
- Desjardins, P., Conklin, D. NanoDrop microvolume quantitation of nucleic acids. Journal of Visualized Experiments. (45), e2565 (2010).
- Summer, H., Gramer, R., Droge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). Journal of Visualized Experiments. (32), e1485 (2009).
- Dominski, Z., Kole, R. Selection of splice sites in pre-mRNAs with short internal exons. Molecular Cell Biology. 11 (12), 6075-6083 (1991).
- Amir-Ahmady, B., Boutz, P. L., Markovtsov, V., Phillips, M. L., Black, D. L. Exon repression by polypyrimidine tract binding protein. RNA. 11 (5), 699-716 (2005).
- Le Guedard-Mereuze, S., et al. Sequence contexts that determine the pathogenicity of base substitutions at position +3 of donor splice-sites. Human Mutation. 30 (9), 1329-1339 (2009).
- Sharma, S., Wongpalee, S. P., Vashisht, A., Wohlschlegel, J. A., Black, D. L. Stem-loop 4 of U1 snRNA is essential for splicing and interacts with the U2 snRNP-specific SF3A1 protein during spliceosome assembly. Genes and Development. 28 (22), 2518-2531 (2014).
- Steitz, J. A., et al. . Functions of the abundant U-snRNPs. Structure and function of major and minor small nuclear ribonucleoprotein particles. , 115-154 (1988).
- Fortes, P., et al. Inhibiting expression of specific genes in mammalian cells with 5' end-mutated U1 small nuclear RNAs targeted to terminal exons of pre-mRNA. Proceedings of the National Academy of Sciences U.S.A. 100 (14), 8264-8269 (2003).
- Roca, X., et al. Widespread recognition of 5' splice sites by noncanonical base-pairing to U1 snRNA involving bulged nucleotides. Genes and Development. 26 (10), 1098-1109 (2012).
- Roca, X., Krainer, A. R. Recognition of atypical 5' splice sites by shifted base-pairing to U1 snRNA. Nature Structural Molecular Biology. 16 (2), 176-182 (2009).
- Taladriz-Sender, A., Campbell, E., Burley, G. A. Splice-switching small molecules: A new therapeutic approach to modulate gene expression. Methods. 167, 134-142 (2019).
- Hamid, F. M., Makeyev, E. V. A mechanism underlying position-specific regulation of alternative splicing. Nucleic Acids Research. 45 (21), 12455-12468 (2017).
- Martelly, W., et al. Synergistic roles for human U1 snRNA stem-loops in pre-mRNA splicing. RNA Biology. , 1-18 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved