このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
リアルタイム細胞代謝フラックス分析装置を使用して骨芽細胞生体エネルギーをモニタリング
要約
リアルタイム細胞代謝フラックスアッセイは、pHおよび酸素センサを使用して、ミトコンドリアおよび解糖系アデノシン三リン酸産生に対応する酸素消費速度および細胞外酸性化速度を測定する。原稿では、骨芽細胞のエネルギー状態と細胞の生体エネルギー状態の特徴付けと解釈を理解するための方法を説明しています。
要約
骨芽細胞による骨形成は、骨格の恒常性を維持し、最終的には骨折を防ぐために、適切な骨獲得と骨代謝回転に不可欠なプロセスです。ピーク骨量を最適化し、様々な筋骨格疾患(すなわち、閉経後骨粗鬆症、神経性食欲不振、1型および2型真性糖尿病)と戦うために、骨生物学の分野では、骨芽細胞の分化プロセス全体にわたって完全に特徴付けるための信じられないほどの努力がなされてきた。マトリックスタンパク質および石灰化小胞を分泌する成熟骨芽細胞の主な役割を考えると、これらのプロセスは信じられないほどの量の細胞エネルギー、またはアデノシン三リン酸(ATP)を必要とすることが注目されている。全体的な細胞エネルギー状態は、しばしば細胞生体エネルギー学と呼ばれ、細胞のニーズを満たすためにATPを導出するための基質の利用可能性を感知する一連の代謝反応を含む。したがって、現在の方法は、初代マウス骨髄間質細胞(BMSC)を単離し、骨芽細胞分化の様々な段階でリアルタイム細胞代謝フラックス分析装置を使用してそれらの生体エネルギー状態を監視するプロセスを詳述する。重要なことに、これらのデータは、代謝プロファイルが骨芽細胞分化を通して劇的に変化することを実証した。したがって、この生理学的に関連する細胞型を使用することは、細胞の生体エネルギー状態が全体的な機能を調節する方法を十分に理解するために必要である。
概要
骨芽細胞による骨の形成は、破骨細胞による骨の協調的な破壊または再吸収を伴う。骨芽球性骨形成と破骨細胞再吸収との間のバランスは、骨格恒常性維持に不可欠な骨代謝回転またはリモデリングを記述する結合プロセスである。骨芽細胞機能障害は、骨形成の障害をもたらし、骨粗鬆症を含む様々な疾患をもたらす1,2,3。骨髄間質幹細胞(BMSC)から骨芽細胞前駆体および成熟骨芽細胞へのエクスビボ/インビトロ分化は、経時的に培養容器内の石灰化骨マトリックスの形成および沈着をもたらす4,5,6。骨芽細胞によるこの骨形成は、かなりの量の細胞エネルギーを必要とする。具体的には、コラーゲン合成および分泌は、細胞ATP:ADP比に大きく依存することが示されており、おそらく、石灰化小胞の輸送および分泌は、追加のATP7、8、9、10、11を必要とする。多くの研究者は、骨芽細胞形成および骨芽細胞機能のプロセスが、骨形成の代謝需要を満たすために十分なエネルギー供給を必要とすることを実証している12,13,14,15,16。したがって、この方法の目標は、リアルタイム細胞代謝フラックス分析装置を用いて骨芽細胞分化全体を通して初代マウス間質細胞の生体エネルギー状態を特徴付けることである。これらの技術は、骨格恒常性の理解を深めるのに役立ち、最終的には骨格障害を改善することができる新規な治療選択肢の開発につながる可能性がある。
リアルタイム細胞代謝フラックス分析装置は、それぞれミトコンドリアおよび解糖系ATP産生に対応する生骨芽細胞の酸素消費速度(OCR)および細胞外酸性化速度(ECAR)を測定するために使用することができる。この方法論の基本的なことは、グルコースから乳酸への変換において解糖系中に乳酸塩あたり1つのH+イオンが放出され、ECAR値に反映された媒体pHを変化させるという事実である。逆に、TCA(トリカルボン酸)サイクルの間、ミトコンドリアを介した酸化的リン酸化は、酸素を利用または消費することによってCO2を生成するため、OCRのモニタリングはこの代謝プロセスを反映している。このアナライザは、細胞外微小環境におけるOCRとECARの両方を同時にリアルタイムで測定するため、細胞の生体エネルギーを研究する際に大きな可能性を秘めています6,17。さらに、これらのアッセイの実行は比較的簡単で、実験目標に応じて簡単にカスタマイズできます。同様の技術が、免疫系18、19、癌開始、および進行20のT細胞代謝調節を、メタボリックシンドローム21、22に寄与する複数の他の細胞型とともにさらに理解するために採用されている。
代替技術に対するリアルタイム代謝フラックス分析装置の利点には、(1)生細胞の細胞生体エネルギーをリアルタイムで測定する能力、(2)比較的少数の細胞でアッセイを行う能力(5,000細胞という低い細胞が必要)、(3)ハイスループット96ウェルシステムで複数の治療を並行して操作する注入ポート、(4)正規化のための放射性ラベルフリー自動細胞イメージャの使用18、23,24。以下の方法は、分析装置を用いた骨芽細胞分化全体を通してマウスBMSCにおける細胞生体エネルギー学のモニタリングに関する一般化されたが詳細な説明を提供することを目的としている。これには、日常的に行われるアッセイが含まれます。ただし、多くの手法や方法と同様に、個々のラボが実験の具体的な詳細を決定することを強くお勧めします。
アッセイの選択と利用可能なさまざまな種類のアッセイ:実験結果の信頼性と一貫性を確保しながら、細胞の生体エネルギー学を研究するために、多種多様なアッセイキットと試薬が利用可能です。さらに、デスクトップソフトウェアは、簡単にカスタマイズできるアッセイテンプレートも提供します。アッセイは、異なる代謝パラメータを測定するユーザのニーズに基づいて定義することができる。これらのアッセイは、実験目標および/または科学的質問に基づいて様々な方法で修正することができる。例えば、4つの注入ポートを用いて、複数の化合物をアッセイ培地に注入して、各代謝経路に特異的な細胞応答を分析することができる。
細胞エネルギー表現型試験:このアッセイは、生細胞の代謝表現型および代謝能を測定する。このアッセイは、経路特異的代謝の一般化されたアイデアを得るための第一歩としても推奨されます。ATP合成酵素の阻害剤であるオリゴマイシンA-と、ミトコンドリア脱共役剤であるカルボニルシアン化物4-(トリフルオロメトキシ)フェニルヒドラゾン(FCCP)-の混合物を注入し、細胞エネルギーポテンシャルを理解する。オリゴマイシンAの注射は、ATPの合成を阻害し、細胞がエネルギー需要を満たすことを可能にする解糖系(ECAR)の割合を増加させる。一方、FCCPの注射は、ミトコンドリア膜の脱分極のためにより高いOCRをもたらす。本質的に、このアッセイは、基礎代謝呼吸、および二重注射、プッシュ、またはストレスに続く代謝応答を描写する。これらのパラメータに基づいて、ソフトウェアは、細胞を経時的に好気的、静止状態、解糖系、またはエネルギー状態として分類することによって、細胞のOCRおよびECARをプロットする25,26。
ATPリアルタイム生産速度アッセイ: これは、解糖系およびミトコンドリア呼吸からの細胞ATP産生を同時に測定する。このアッセイは、2つのエネルギー経路からの代謝シフトを定量的に測定し、経時的なミトコンドリアおよび解糖系ATP産生速度に関するデータを提供する。アッセイは、基礎OCRおよびECARデータを取得し、続いて、オリゴマイシンAの注射によるミトコンドリアATP産生速度およびロテノン+アンチマイシンA混合物の注射による解糖系ATP産生速度(ミトコンドリア機能の総阻害)を計算し、ミトコンドリア酸性化をもたらす17、27。
細胞ミトコンドリアストレス試験(または細胞ミトストレス試験):これは、ATP結合呼吸を介してミトコンドリア機能を測定し、細胞の生体エネルギー学を定量化し、ミトコンドリア機能不全を同定し、ストレスに対する細胞の応答を測定する。基礎呼吸能力および予備呼吸能力、ATP結合呼吸、最大呼吸、および非ミトコンドリア酸素消費を含む様々なパラメータを、1つのアッセイで得ることができる。このアッセイは、オリゴマイシンA、FCCP(ミトコンドリア脱共役剤)、ロテノン/アンチマイシンA阻害剤の混合物の逐次注射を含み、ミトコンドリア機能に対するこれらの影響を効率的に分析する28。
柔軟性水戸燃料フレックステスト: これは、それらの阻害剤の有無による3つの主要なミトコンドリア燃料の酸化によるミトコンドリア呼吸数を測定する。グルコース、グルタミン、および脂肪酸の逐次阻害は、エネルギー需要を満たすために、細胞の依存性、容量、および柔軟性、ならびに様々な細胞経路における細胞の依存性を測定するのに役立つ。ミトコンドリアが他の燃料を酸化することによって目的の経路の遮断された要求を満たすことができないとき、細胞は依存状態に入る。細胞の容量は、他の2つの代替経路の阻害とそれに続く目的の経路の阻害によって計算される。細胞の柔軟性は、ミトコンドリアが阻害された経路の燃料ニーズを補償し、満たす能力を理解するのに役立ちます。これは、細胞の容量から細胞の依存性を差し引くことによって計算されます。3つの異なる阻害剤を独立して、または2つの混合物として使用して、アッセイパラメータを効果的に計算する。2-シアノ-3-(1-フェニル-1H-インドール-3-イル)-2-プロペン酸(UK5099)は、解糖系中のピルビン酸担体を遮断することによってグルコースの酸化を阻害する。ビス−2−(5−フェニルアセトアミド−1,3,4−チアジアゾール−2−イル)(BPTES)エチルスルフィドはグルタミン酸化経路を阻害し、エトモキシルは長鎖脂肪酸の酸化を阻害する29。
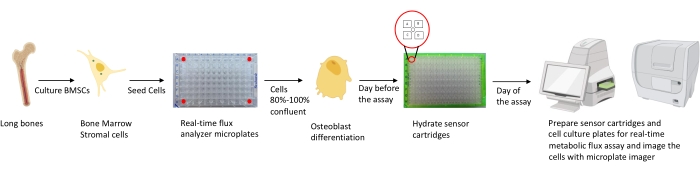
図1:分析のために骨芽細胞を培養および調製するための方法論の概略図。 マウスBMSCは長骨から単離され、培養され、25,000細胞/ウェル密度で96ウェルプレートに播種される。これらの細胞を骨芽細胞特異的培地中で培養することは、それらが80%〜100%コンフルエントに達したときに開始され、それらの分化を開始する。アッセイは、分化の異なる段階で行われる。カートリッジプレートをアッセイの1日前に水和させる。アッセイ当日、アッセイ要件に基づいて異なる阻害剤をセンサーカートリッジのポートに注入し、校正バッファーを96ウェル校正プレートに添加します。較正後、リアルタイム細胞代謝フラックスアッセイが行われ、続いてマイクロプレートイメージャを用いて細胞培養マイクロプレートを画像化することにより、リアルタイム細胞代謝フラックス分析計データを細胞数で正規化する。 この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
すべての手順は、ヴァンダービルト大学医療センターの施設動物ケアおよび使用委員会のガイドラインと承認に基づいていました。
1. 試薬の調製とアッセイのセットアップ
- 骨髄間質細胞の単離および培養(前項30も参照)。
- 10%FBS(ウシ胎児血清)、100 U/mLのペニシリン、および100 μg/mLのストレプトマイシンでアルファ修飾で最小必須培地を補充することにより、完全なアルファ最小必須培地(αMEM)細胞培養培地を調製する。
- 細胞が通過できるように0.6 mLの微量遠心管の端をトリミングし、100 μLの完全なαMEMを含む1.5 mLの微量遠心管に挿入して骨髄採取管を準備します。
- 以下のようにCO2 処理を用いてマウスを安楽死させる。動物をCO2チャンバーに2〜 3分間、または呼吸が止まるまで置く。動物が意識不明になってから少なくとも1分間待って、マウスをチャンバーから取り出し、子宮頸部脱臼させる。
- 滅菌鉗子および一対のはさみを用いて、安楽死させたマウスの下腹部を切り開き、小さな切開を行う。マウスの長骨(大腿骨、脛骨、および腸骨頂部)を単離する。
- すべての軟部組織を除去するために長骨をトリミングします。骨がきれいになったら、遠位端と近位端から約1〜2mm切り取って、骨髄が洗い流すための開口部を作ります。
注:この開口部は、骨髄が洗い流されるのを許しながら骨髄を失うのを避けるために、控えめにする必要があります。 - 100 μLの1x滅菌PBS(リン酸緩衝生理食塩水)を含む収集チューブに骨を入れて、骨髄全体を単離する。
- 室温で15〜20秒間、10,000 x g で遠心分離することによって骨髄を洗い流す。骨髄細胞ペレットをチューブの底に。
- 骨腔が白く見え、ほとんどの骨髄要素がなくなるまで遠心分離を繰り返します。骨髄の混合集団を上下に穏やかにピペッティングすることによって再懸濁する。
- 1匹の動物(大腿骨および脛骨の両方)由来の細胞を10mLの細胞培養培地中の75cm2細胞培養フラスコ中で培養し、5%CO2を有する細胞培養インキュベーター中で37°Cでインキュベートした。2〜3匹の動物由来の細胞をプールする場合は、150cm2細胞培養フラスコ(推奨)を使用する。
- 混合集団の24〜48時間のインキュベーションに続いて、培養培地内に含まれる非付着性造血細胞集団を吸引し、付着細胞を1x PBSで洗浄する。
- BMSCからの細胞播種と骨芽細胞分化
- フラスコ表面をわずかに覆うのに十分な0.25%トリプシン-EDTA(約3-4mL)を加えて付着細胞をトリプシン処理し、続いて37°Cで3分間インキュベートした。
- フラスコ/トリプシンに6~7mLの完全なαMEMを加え、注意深く上下にピペッティングすることにより、付着したBMSCを再懸濁させる。BMSC懸濁液を円錐遠心管に移す。
- BMSC懸濁液の50μLアリコートを除去し、それに50μLのトリパンブルー(1:1希釈)を加える。この混合物を血球計数器に10 μLピペッティングし、顕微鏡下で観察することによって、色素を除外した生細胞の総数をカウントする。青色に見える死んだ細胞や不健康な細胞(<10%の細胞)を数えないでください。
- 細胞数に基づいて、プレートあたり少なくとも10mLの総容量に対して、最終濃度2.4 x106 細胞/mLに必要な完全なαMEM中の細胞懸濁液の体積を計算します。

図2:分析装置用に特別に設計された細胞培養マイクロプレート 。(A)4つのバックグラウンド補正ウェルA1、A12、H1、H12が強調表示されています。これらのウェルには、細胞を含まないアッセイ培地のみが含まれています。(B)プレートの側面にあるバーコードを、イメージングリーダーおよびアナライザーを使用してプレートをスキャンする。 この図の拡大版を表示するには、ここをクリックしてください。
- 円錐管内の細胞を1,000 x g で5分間遠心分離し、細胞を所望の最終濃度2.4 x106 細胞/mLに再懸濁する。
- 細胞懸濁液をリザーバに移し、マルチチャネルピペットを使用して、細胞の均一な混合物を確実にするために細胞を慎重に再懸濁する。
- 80 μLの完全αMEMを有する96ウェル細胞培養マイクロプレートに1ウェルあたり2.5 x104 個の細胞をシードする。バックグラウンド補正ウェル(A1、A12、H1、H12)に細胞を播種しないでください。代わりに、これらの4つのウェルに培地を追加するだけです。
注: アッセイ用の BMSC は、センサーカートリッジと組み合わせて分析装置用に設計された 96 ウェル細胞培養マイクロプレートにプレートされます。これらのプレートの表面積は、通常の96ウェルプレートとは異なります。プレート内の各ウェルの表面積は0.106cm2であり、これは典型的な96ウェルプレート面積の約40%である。最適な細胞播種密度は、細胞型に基づいて選択される。典型的には、分析器は、ウェル当たり0.5〜4 x104個の細胞を検出することができる。骨芽細胞は効果的に区別するために接触する必要があります。この目的のために、80 μLの完全αMEM中の2.0 x 10 4-3.0 x 104 BMSC/ウェル間のめっきが選択されています。 - プレートを穏やかに攪拌してウェル内の細胞の均一な分布を確保し、37°C、5%CO2でインキュベートする。48時間後に顕微鏡下で細胞の成長および細胞コンフルエンシーを確認する。必要に応じて細胞培養培地を交換してください。
- アッセイの目的に応じて、BMSCが60%〜80%コンフルエント(典型的には48〜72時間)である場合、細胞培養培地を骨芽細胞分化培地(5mM βグリセロールリン酸および50μg/mLのL-アスコルビン酸を添加した完全なαMEM)に変更することによって骨芽細胞分化を開始する。
- 未分化間質細胞(Day 0)を解析する場合は、細胞を完全なαMEMの下に維持します。
- 骨芽細胞分化培地を1日おきに交換し、顕微鏡下で細胞を視覚化して、アッセイの日まで細胞が健康であることを確認します。好ましくは、スケジュールされたアッセイの24時間前に、培地を変更し、一貫した培地変更スケジュール(推奨)を維持する。
メモ: プレートを斜めに少し傾けて、メディアを慎重に交換してください。これにより、ピペットチップが細胞培養プレートに誤って接触し、細胞の単層の破壊が回避されます。
2. 細胞外フラックス校正用センサカートリッジの作製
- アッセイの日の前に細胞外アッセイキットからセンサーカートリッジを水和させる。センサーカートリッジ(緑色のプレート)を取り外し、センサーを裏返して置きます。
- マルチチャンネルピペットを使用して、ユーティリティプレートの各ウェルに200μLのH2Oを加える。センサーカートリッジをユーティリティプレートに慎重に置き、プレートを室温で一晩インキュベートします。
注:メーカーは、センサーカートリッジを非CO2 37°Cインキュベーターで一晩インキュベートすることをお勧めします。ただし、センサーカートリッジの著しい蒸発が発生する可能性があります。この場合、センサーカートリッジを室温でインキュベートすることができます。これらのプレートは、最小4時間、最大72時間インキュベートする必要があります。 - アッセイの日に、ユーティリティプレートからH2Oを捨て、200μLのキャリバートを加える。ユーティリティプレートをアッセイ前に少なくとも1時間インキュベートする。
3. リアルタイム細胞代謝フラックス分析装置培地調製
- フェノールレッドを含まない DMEM 培地を 7.4 (推奨) の事前調整済み pH で使用し、BMSC でアッセイを実行します。
- DMEM培地に1 mMピルビン酸ナトリウム、2 mMグルタミン、10 mMグルコース、200 nMインスリン、50-200 μMオレイン酸BSAを補充して、80 mLのアッセイ培地を調製します。
- 完全なアッセイ培地をウォーターバス中で37°Cでインキュベートする。
4. センサカートリッジ用コンパウンドの調製
- 氷上でオリゴマイシンA、ロテノン、およびアンチマイシンAを解凍する。使用前にピペットを上下にして化合物を可溶化します。
- 調製したアッセイ培地3 mLを各チューブに加え、続いて、それぞれの化合物チューブA:26.4 μLの2.5 mMオリゴマイシンAを添加し;チューブB:3.1 μLの12.67 mMロテノン+ 4.1 μLの9.4 mMアンチマイシンA + 30 μLのヘキスト染色。
- これらのインヒビターの 10 倍の濃度を対応するポートにロードします。必要な注射液の最終濃度は、2μMのオリゴマイシンA、1μMのロテノン、および4.1μMのアンチマイシンAである。
注:ヘキストは、イメージングおよび正規化の目的で核を蛍光染色するために最終注入口に添加される。これらの濃度は、細胞型に基づいて最適化することができる。 - これらの化合物 20 μL を 180 μL のアッセイ培地で細胞にロードします。
アッセイ用の細胞培養マイクロプレートの調製
- 細胞培養用マイクロプレートを37°Cの培養器から取り出し、顕微鏡下で細胞を観察した。
- アッセイ培地を水浴から除去する。
- 200 μL のアッセイ培地で細胞を 2 回穏やかに洗浄し、1 ウェルあたり 200 μL のアッセイ培地を加えます。
注: 最終的なアッセイ培地が細胞に添加されると、プレートが分析装置に入るまでの時間は非常に重要です。したがって、次の手順が 1 時間以内に実行されるまで、メディアの交換を開始しないでください。 - 顕微鏡下で細胞をチェックして、細胞がウェルに接着したままであることを確認します。
- D5およびE8の細胞が一貫した単分子膜で接着され、洗浄ステップ中に洗い流されなかったことを確認する。細胞イメージングソフトウェアは、オートフォーカスと自動露出を設定するためにこれら2つのウェルを使用します。
注:製造業者は、プレートを非CO2 37°Cインキュベーターで1時間インキュベートすることをお勧めします。自動イメージングが望ましい場合は、この手順をスキップできます。例えば、マイクロプレートイメージャは、密閉チャンバ内で同じ条件を維持し、細胞を明視野下で画像化することができる。
6. アッセイとイメージングのセットアップ
図3:コントローラソフトウェアソフトウェアは、機器が接続され、37°Cに設定されていることを確認します。 細胞外フラックス分析装置を用いて実施することができる異なるアッセイのためのテンプレートファイルを選択して、実験目標に基づいてアッセイをさらにカスタマイズすることができる。 この図の拡大版を表示するには、ここをクリックしてください。
- 機器の横にあるコンピューターでデスクトップソフトウェアを開きます。
- コントローラソフトウェアの左下隅にある接続ステータスを確認します。
- テンプレートに移動し、XF ATP レートアッセイテンプレートファイルまたは適切なアッセイテンプレートを選択します。
- 画面上部の [ グループ定義 ] を選択し、グループを定義します。
- プレートマップレイアウトを選択し、定義されたグループに応じてウェルを割り当てます。
- 機器のプロトコルを確認し、添加した化合物が正しくリストされていることを確認し、将来の参照のためにプロジェクト情報を含めます。
- [アッセイの実行]をクリックします。これにより、結果ファイルの保存場所の選択が促されます。
- 結果ファイルを保存する場所を選択します。
- アッセイの日付を含むファイルを保存し、[ 実行の開始]をクリックします。
- センサーカートリッジとユーティリティプレートをトレイに置き、[ 準備完了 ]をクリックしてキャリブレーションを開始します。
- キャリブレーションを開始する前に、カートリッジの蓋が取り外され、センサーカートリッジがユーティリティプレート上の正しい向きに置かれていることを確認します。この手順には10〜20分かかり、完了すると、ソフトウェアは ロードセルプレート ダイアログボックスを表示します。
7.明視野画像を取得する
メモ: この手順はオプションです。使用可能なイメージング機器がない場合は、手順 8 に進んでください。

図4:細胞イメージングソフトウェアは、コンピュータを介してイメージングリーダーと通信します。 マイクロプレート内の細胞はアッセイの前後に画像化することができ、アッセイ後に細胞数/ウェルを取得してデータを正規化することができます。 この図の拡大版を表示するには、ここをクリックしてください。
- コンピュータ上の細胞イメージングソフトウェアを開きます。
- マイクロプレートイメージャの電源が入っていて、ポートがコンピュータに接続されていることを確認します。
- 画面左下のステータスバーをチェックして、温度が37°Cに設定されていること、および接続ステータスが緑色で[準備完了]として強調表示されていることを確認します。
- プレートのバーコードをスキャンして、イメージングプロセスを開始します。
- セルプレートに名前を入力し、[保存]をクリックします(これは、明視野と蛍光画像の両方が 保存 される名前です)。[ 明視野スキャンの実行]をクリックします。
- 次の画面、プレート、およびスキャン メニューには、イメージングのオプションが表示されます。アッセイの前に、[ 明視野スキャンの開始] を選択します。
- 細胞培養マイクロプレートをプレートカバー/蓋とともにトレイホルダーに置き、A1をA1マークに合わせます。 [トレイを閉じる]をクリックします。
- 次の画面には、明視野画像取得、プレートマップが表示されます。[ すべてのウェルをスキャン]をクリックすると、システムの初期化プロセスが開始され、その後に30〜35分のスキャンが開始されます。
- 明視野スキャンの後、細胞培養マイクロプレートを取り出し、分析装置に入れてアッセイを行う。
8. アッセイの実行
- キャリブレーションが完了すると、ソフトウェアは ロードセルプレート ダイアログボックスを表示します。
- [トレイ を開く ]をクリックして、ユーティリティトレイを細胞培養マイクロプレートに置き換えます。蓋が取り外され、プレートの A1 が正しい向きに収まっていることを確認します。
- 次に、 ロードセルプレート をクリックしてアッセイを開始します。センサーカートリッジは、アッセイ注射のために分析装置の内部に残ります。
- アッセイが開始され、推定完了時間が表示されるまで待ちます。
- アッセイが完了すると、ソフトウェアはセンサー カートリッジのアンロード ダイアログボックスを表示します。 「取り出し 」をクリックし、細胞培養マイクロプレートを分析装置から取り外します。
- センサーカートリッジを慎重に取り外し、セルプレートの蓋を取り付けます。細胞は蛍光イメージングと細胞計数の準備が整いました。
- セルプレートとセンサーカートリッジを取り外すと、「 アッセイ完了」 ダイアログボックスが表示されます。
- [結果 の表示 ]をクリックしてアッセイ結果ファイルを開き、データをすぐに正規化するか、[ ホーム]をクリックします。
9. 蛍光画像を取得して正規化する
注:このステップは、BMSCおよび骨芽細胞の正常化のための任意であるが推奨される方法である。利用可能なイメージング機器がない場合は、タンパク質やDNAの単離や定量など、別の正規化方法を使用する必要があります。

図5:アッセイからのデータの正規化に使用される画像化ソフトウェアからの代表的な画像。(b)アッセイ結果を正規化するために細胞数を計数するために使用した骨芽細胞のヘキスト染色核を示すステッチ蛍光画像。これらは分化の7日後の骨芽細胞である。この図の拡大版を表示するには、ここをクリックしてください。
- アッセイ終了後、ハンドヘルドバーコードリーダーでプレートバーコードをスキャンします。プレートが既にイメージ化されている場合は、新しい名前は必要ありません。
- 蛍光と細胞数を選択し、トレイホルダーにセルプレートを置き、「トレイを閉じる」をクリックします。
- 画像取得ウィンドウで、[ すべてのウェルをスキャン ]を選択してイメージングを開始します。蛍光イメージングは、プレート全体をスキャンするのに約15〜20分かかります。スキャンが完了したことを示す緑色の目盛りを確認します。
- イメージングおよび細胞イメージングアプリケーションで蛍光画像と細胞数を確認するには、いくつかのウェルをランダムにクリックします。
メモ: 画面の右下にカウントされたセルを表示するオプションがあります。このオプションは、マスクされた画像を表示し、カウントされたオブジェクトを強調表示します。 - 蛍光イメージングが完了したら、追加の参照用に画像をエクスポートします。
- イメージングと細胞数が完了したら、 結果 ファイルを開き、[ 正規化]をクリックします。正規化画面には、プレートレイアウトとセル数をインポートするオプションが表示されます。
- インポートをクリックし、デスクトップソフトウェアの適用を選択して、アッセイを細胞数で自動的に正規化します。
結果
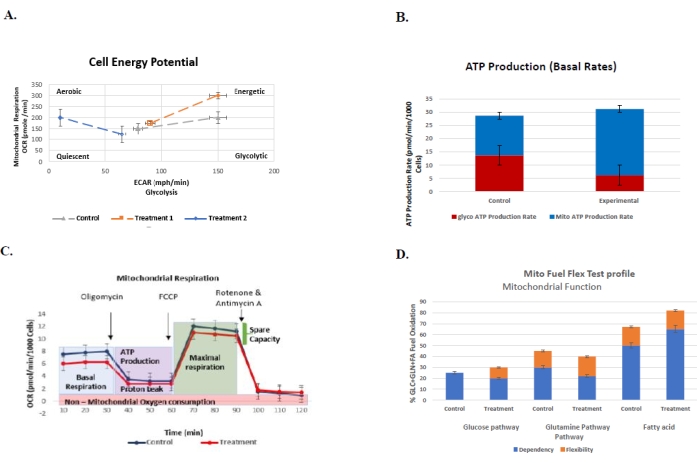
図6:対照対処置群の細胞生体エネルギープロファイルをそれらのそれぞれの標準誤差とともに理解するために日常的に実施されるアッセイのための代表的なグラフ。プロットは、対照対2つの治療群(n = 3)の解糖系(ECAR)対ミトコンドリア呼吸(OCR)を...
ディスカッション
リアルタイムの細胞代謝フラックス分析装置は、さまざまな条件下で細胞エネルギーを探索するために使用できます。このプロトコルは、BMSCの効率的な単離、適切な細胞培養プレートでの細胞の培養、および成熟骨芽細胞への分化を示しており、細胞外フラックス分析装置を使用したさまざまなアッセイに使用できます。さらに、センサーカートリッジの水和、注入ポートの装填、アッセイ...
開示事項
著者らは開示するものは何もありません。
謝辞
この研究は、国立衛生研究所(NIH)国立関節炎・筋骨格・皮膚疾患研究所(NIAMS)グラントAR072123および国立加齢研究所(NIA)グラントAG069795(ERRへ)の支援を受けた。
資料
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin EDTA | Sigma-Aldrich | T4049 | |
| 2-cyano-3-(1-phenyl-1H-indol-3-yl)-2-propenoic acid | Sigma - Aldrich | PZ0160 | UK5099 |
| Antimycin A | Sigma - Aldrich | A8674 | |
| Ascorbic acid | Sigma-Aldrich | A4544-100G | |
| Bis-2-(5-phenylacetamido-1,3,4-thiadiazol-2-yl)ethyl sulfide | Sigma - Aldrich | SML0601 | BPTES |
| Carbonyl cyanide 4-(trifluoromethoxy)phenylhydrazone | Sigma - Aldrich | C2920 | FCCP |
| Cytation 5 imaging reader | BioTek | N/A | Microplate imager |
| Etomoxir sodium salt hydrate | Sigma - Aldrich | E1905 | |
| Hoechst 33342 Solution (20 mM) | Thermo Scientific | 62249 | |
| Insulin | Sigma - Aldrich | I6634 | |
| Oleic Acid-Albumin from bovine serum | Sigma - Aldrich | O3008 | |
| Oligomycin A - 5 mg | Sigma - Aldrich | 75351 | |
| Rotenone | Sigma - Aldrich | R8875-1G | |
| Seahorse XF 1.0 M Glucose Solution | Agilent Technologies | 103577-100 | |
| Seahorse XF 100mM Pyruvate Solution | Agilent Technologies | 103578-100 | |
| Seahorse XF 200mM Glutamine solution | Agilent Technologies | 103579-100 | |
| Seahorse XF DMEM media | Agilent Technologies | 103575-100 | DMEM assay media eith 5mM HEPES, pH 7.4, without phenol red, sodium bicarbonate, glucose, pyruvate, and L-glutamine |
| Seahorse XFe96 Analyzer | Agilent Technologies | S7800B | Real- Time Metabolic flux analyzer |
| Seahorse XFe96 FluxPak | Agilent Technologies | 102416-100 | Includes XFe96 Sensor cartridges, Cell culture microplates, and Seahorse XF Calibrant solution |
| The Cell imaging 1.1.0.11 software | Agilent Technologies - BioTek | ||
| Wave software 2.6.1 | Agilent Technologies | ||
| β-glycerol phosphate | Sigma-Aldrich | G9422-50G |
参考文献
- Rodan, G. A. Bone homeostasis. Proceedings of the National Academy of Sciences of the United States of America. 95 (23), 13361-13362 (1998).
- Nakahama, K. I. Cellular communications in bone homeostasis and repair. Cellular and Molecular Life Sciences. 67 (23), 4001-4009 (2010).
- Kim, J. M., Lin, C., Stavre, Z., Greenblatt, M. B., Shim, J. H. Osteoblast-osteoclast communication and bone homeostasis. Cells. 9 (9), 2073 (2020).
- Gao, J., et al. SIRT3/SOD2 maintains osteoblast differentiation and bone formation by regulating mitochondrial stress. Cell Death and Differentiation. 25 (2), 229-240 (2018).
- Baron, R. Molecular mechanisms of bone resorption by the osteoclast. The Anatomical Record. 224 (2), 317-324 (1989).
- Tian, L., Rosen, C. J., Guntur, A. R. Mitochondrial Function and Metabolism of Cultured Skeletal Cells. Methods in Molecular Biology. 2230, 437-447 (2021).
- Zanotelli, M. R., et al. Regulation of ATP utilization during metastatic cell migration by collagen architecture. Molecular Biology of the Cell. 29 (1), 1-9 (2018).
- Gonzales, S., Wang, C., Levene, H., Cheung, H. S., Huang, C. Y. C. ATP promotes extracellular matrix biosynthesis of intervertebral disc cells. Cell and Tissue Research. 359 (2), 635-642 (2015).
- Kruse, N. J., Bornstein, P. The metabolic requirements for transcellular movement and secretion of collagen. Journal of Biological Chemistry. 250 (13), 4841-4847 (1975).
- Rendina-Ruedy, E., Guntur, A. R., Rosen, C. J. Intracellular lipid droplets support osteoblast function. Adipocyte. 6 (3), 250-258 (2017).
- Sinnott-Armstrong, N., et al. A regulatory variant at 3q21.1 confers an increased pleiotropic risk for hyperglycemia and altered bone mineral density. Cell Metabolism. 33 (3), 615-628 (2021).
- Esen, E., Lee, S. Y., Wice, B. M., Long, F. PTH promotes bone anabolism by stimulating aerobic glycolysis via IGF signaling. Journal of Bone and Mineral Research. 30 (11), 1959-1968 (2015).
- Borle, A. B., Nichols, N., Nichols, G. Metabolic studies of bone in vitro: I. Normal bone. Journal of Biological Chemistry. 235, 1206-1210 (1960).
- Borle, A. B., Nichols, N., Nichols, G. Metabolic studies of bone in vitro: II. The metabolic patterns of accretion and resorption. Journal of Biological Chemistry. 235, 1211-1214 (1960).
- Adamek, G., Felix, R., Guenther, H. L., Fleisch, H. Fatty acid oxidation in bone tissue and bone cells in culture. Characterization and hormonal influences. The Biochemical Journal. 248 (1), 129-137 (1987).
- Frey, J. L., et al. Wnt-Lrp5 signaling regulates fatty acid metabolism in the osteoblast. Molecular and Cellular Biology. 35 (11), 1979-1991 (2015).
- Romero, N., Rogers, G., Neilson, A., Dranka, B. P. . Quantifying cellular ATP production rate using agilent seahorse XF technology. , (2018).
- vander Windt, G., Chang, C., Pearce, E. Measuring bioenergetics in T cells using a Seahorse Extracellular Flux Analyzer. Current Protocols in Immunology. 113, 1 (2016).
- Traba, J., Miozzo, P., Akkaya, B., Pierce, S. K., Akkaya, M. An optimized protocol to analyze glycolysis and mitochondrial respiration in lymphocytes. Journal of Visualized Experiments:JoVE. (117), e54918 (2016).
- Noel, P., et al. Preparation and metabolic assay of 3-dimensional spheroid co-cultures of pancreatic cancer cells and fibroblasts. Journal of Visualized Experiments:JoVE. (126), e56081 (2017).
- Nicholls, D., et al. Bioenergetic profile experiment using C2C12 myoblast cells. Journal of Visualized Experiments: JoVE. (46), e2511 (2010).
- Sakamuri, S. S. V. P., et al. Measurement of respiratory function in isolated cardiac mitochondria using Seahorse XFe24 Analyzer: applications for aging research. GeroScience. 40 (3), 347-356 (2018).
- . What are the advantages of using Seahorse XF technology Available from: https://wwwagilent.com/en/support/cell-analysis/advantages-of-using-xf-tech (2018)
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 67 (10), 1022-1035 (2012).
- . XF cell energy phenotype test Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740884 (2021)
- Leung, D. T. H., Chu, S. Measurement of oxidative stress: Mitochondrial function using the seahorse system. Methods in Molecular Biology. 1710, 285-293 (2018).
- . XF ATP rate assay Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740889 (2021)
- . XF cell mito stress test Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740885 (2021)
- . XF cell mito fuel flex test Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-kits-reagents-cell-assay-media/seahorse-xf-cell-energy-phenotype-test-kit-740888 (2021)
- Maridas, D. E., Rendina-Ruedy, E., Le, P. T., Rosen, C. J. Isolation, culture, and differentiation of bone marrow stromal cells and osteoclast progenitors from mice. Journal of Visualized Experiments: JoVE. (131), e56750 (2018).
- Wei, J., et al. Glucose uptake and Runx2 synergize to orchestrate osteoblast differentiation and bone formation. Cell. 161 (7), 1576-1591 (2015).
- Zoch, M. L., Abou, D. S., Clemens, T. L., Thorek, D. L. J., Riddle, R. C. In vivo radiometric analysis of glucose uptake and distribution in mouse bone. Bone Research. 4, 16004 (2016).
- Divakaruni, A. S., Paradyse, A., Ferrick, D. A., Murphy, A. N., Jastroch, M. Analysis and interpretation of microplate-based oxygen consumption and pH data. Methods in Enzymology. 547, 309-354 (2014).
- Kam, Y., Jastromb, N., Clayton, J., Held, P., Dranka, B. . Normalization of agilent seahorse XF data by in-situ cell counting using a BioTek cytation 5 application note. , (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved