Method Article
アクチンダイナミクス、クラッチカップリング、成長コーンアドバンスの牽引力の解析
要約
前進するためには、成長コーンは外部環境に対して牽引力を発揮しなければならない。牽引力の生成はアクチンの動態およびクラッチの結合に依存する。本研究では、成長コーンアドバンスのためのアクチンダイナミクス、クラッチカップリングおよび牽引力を分析する方法について説明する。
要約
機能的なネットワークを確立するには、ニューロンは適切な目的地に移行し、そのターゲットセルに向かって軸索を拡張する必要があります。これらのプロセスは、神経突起の先端にある成長コーンの進歩に依存します。軸索成長コーンは、局所的な微小環境を感知し、細胞骨格ダイナミクスとアクチン接着結合(クラッチカップリング)を調節することによって駆動力を発生させます。数十年にわたる研究は、神経移動と軸索誘導を調節するための誘導分子、それらの受容体、および下流シグナル伝達カスケードの同定につながっている。しかし、成長コーンの前進と航行を促進するために力を発生させるために必要な分子機械は、解明され始めたばかりです。神経細胞増殖コーンのリーディングエッジでは、アクチンフィラメントは逆行流を受け、アクチン重合とアクトミオシン収縮によって動力を与えられる。F-アクチン逆行流と粘着基材のクラッチカップリングは成長コーン前進のための牽引力を発生させる。本研究では、単一スペックルイメージングによるF-アクチン逆行流をモニタリングするための詳細なプロトコルについて説明する。重要なことに、FアクチンマーカーLifeactと組み合わせると、この技術は1)F-アクチン重合率と2)F-アクチン逆行流と接着基材との間のクラッチカップリング効率を定量することができます。どちらも、成長コーンアドバンスとナビゲーションのための力を生成するための重要な変数です。さらに、本研究は、成長コーンによって生成される3)牽引力を定量化することができる牽引力顕微鏡の詳細なプロトコルを記述する。したがって、単一のスペックルイメージングと牽引力顕微鏡の解析を結合させることで、研究者は成長コーンの前進とナビゲーションの基礎となる分子力学を監視することができます。
概要
発達中の脊椎動物の脳では、ニューロンは精巧に組織化された移行を受け、適切なシナプスパートナーに向かって軸索を投影して機能的なニューロンネットワークを確立する1,2,3。神経突起の先端に位置する感覚および運動性構造である成長コーンは、ニューロンの移動と軸索の成長の速度と方向を決定する3,4,5。ニューロンは密集した環境に囲まれているので、成長コーンは6,7を前進させるために自分の環境に対して力を発揮しなければなりません。神経移動と軸索誘導の基礎となるメカニズムを理解するためには、成長コーンアドバンスのための分子力学の分析が不可欠です。
数十年にわたる分析の結果、成長コーンの前進を促進する牽引力は「クラッチ」メカニズムによって生成される。;このメカニズムは、軸索成長コーンだけでなく、ニューロンの移行の主要なプロセス成長コーンでも機能すると考えられている8,9,10,11,12.すなわち、成長コーンのアクチンフィラメント(F-アクチン)は、先端縁で重合し、近位で非重合し、先端膜13,14,15を押し出します。得られた力は、アクトミオシン収縮と併せて、逆行流7、11、16、17、18、19、20、21と呼ばれるF-アクチンの後方移動を誘導する。クラッチおよび細胞接着分子は、F-アクチン逆行流と接着基材との機械的結合を媒介し、Fアクチン流量の力を基質に伝達し、成長コーン前進のための牽引力を発生させる7,8,9,11,12,22 .同時に、アクチンと基質結合はF-アクチンの流速を減少させ、アクチン重合を力に変換して、最先端の膜9,10を突き出します。
軸索の成長コーンは、局所的な化学的手がかりを感知し、成長コーンナビゲーション3,23,24,25のための指向性の原動力にそれらを変換します。例えば、軸索誘導分子ネトリン-1は、大腸癌(DCC)で削除された受容体を刺激し、細胞分裂対照タンパク質42(Cdc42)およびRas関連C3ボツリヌス毒素基質1(Rac1)およびそれらの下流キナーゼp21活性化物1(Pak1)を活性化する。Cdc42及びRac1は1)アクチン重合を促進し、そしてPak1はクラッチ分子shootin122,26をリン酸化する。Shootin1はアクチン結合タンパク質コルクタクチン27を介してF-アクチン逆行流と相互作用する。Shootin1はまた、L1細胞接着分子(L1-CAM)20,24と相互作用する。Shootin1リン酸化は、コルタクチンおよびL1-CAMの結合親和性を増加させ、そしてshootin1媒介した2)クラッチカップリング24,27を増強する。成長コーン内では、ネトリン-1源の側のアクチン重合およびクラッチ結合の非対称活性化が3)牽引力を増加させ、成長コーン旋回のための指向性駆動力を発生させる(図1)24。神経遊び動と軸索誘導に関する過去数十年間の集中的な研究は、ガイダンス分子、それらの受容体、および関連する下流シグナル伝達カスケード2,10,28,29,30の理解を高めました。しかし、成長コーンアドバンスのための力を生成する分子機械は、ちょうど解明され始めています。これは、メカノ生物学的分析のためのプロトコルの限られた使用法に起因する可能性があります。
本研究は、単一のスペックルイメージング16,18によるF-アクチン逆行流をモニタリングするための詳細なプロトコルを説明する。F-アクチン逆行流のモニタリングは、超解像顕微鏡、回転ディスク共焦点顕微鏡、全干渉反射蛍光(TIRF)顕微鏡25,31,32,33,34,35,36,37,38を用いて広範囲に行われてきた。.しかしながら、本研究のプロトコルは、標準的な蛍光顕微鏡を用い、容易に採用可能である11,16,18,20,22,23,24,27,39,40,41,42。Lifeact43によるF-アクチンラベリングと組み合わせると、単一スペックルイメージングにより、アクチン重合速度の定量化と、F-アクチン逆行流と接着基材間のクラッチ結合効率39,42が可能になります。本研究は、蛍光ビーズ埋め込みポリアクリルアミド(PAA)gel11,22,23,24,27,39,41,42,44を用いたトラクション力顕微鏡の詳細なプロトコルについてさらに説明する。この方法は、力誘導ビーズの動き44,45を監視することにより、成長コーン下の牽引力を検出し、定量化する。オープンソースの牽引力分析コードが提供され、成長コーン移行時の牽引力を定量化する方法について詳しく説明します。単一のスペックルのイメージ投射および牽引力顕微鏡の助けによって、成長コーンの移動および運行の基礎となる分子力学を理解することは容易になる。これらの技術は、学習や記憶42において重要であることが知られている樹状脊椎の拡大の基礎となる分子力学を分析するためにも適用可能である。
プロトコル
実験動物を用いた実験は、奈良科学技術大学の動物医療・利用委員会で行われました。調査官は、実験動物のケアと使用のための彼らの制度的および国家動物規制委員会によって確立されたガイドラインに従う必要があります。
1. ソリューションとメディアの準備
- 補足ファイル 1 に要約したソリューションとカルチャ メディアを準備する
2. ポリD-リジン(PDL)/ラミニンコーティング基質の調製
- 直径14mmのガラス底皿に100μg/mL PDLをコーティングし、リン酸緩衝生理食塩水(PBS、pH 7.4)に溶解し、37°Cの加湿インキュベーターで一晩インキュベートします。
注:この手順の後、ガラス表面を乾かさないで下す - PDL溶液とピペット1 mLのPBSをガラス上で10回3回取り出します。
- 5 μg/mL ラミニンで皿をコーティングし、PBSに溶解し、37°Cの加湿インキュベーターで一晩インキュベートします。
- ガラスの上にラミニン溶液とピペット1mLのPBSを10回3回取り出します。
- PBSを取り出し、ガラス表面に0.5mLの神経基底培地を置きます。37 °Cの加湿インキュベーターに料理を保管してください。
注:PDL/ラミニンコーティングされた食器は、37°Cの加湿インキュベーターに2〜3日間保存することができます。
海馬の解剖と解離
- 子宮頸部脱臼により、妊娠中のマウスを安楽死させる。
注:本研究では市販のマウスを使用しています( 材料表を参照)。研究者は、殺菌された環境で飼育され、人道的に処理されたマウスを使用する必要があります。 - 胚性16日目(E16)マウス胚を解剖し、氷の上に置く。
- 層流フードで、はさみで脳を解剖し、10 mLの氷冷解剖液を含む無菌10cm皿の上に置きます。
- 解剖ステレオ顕微鏡では、慎重に大脳半球の髄液を剥がし、鉗子を使用して海馬を解剖する。
- 海馬を15mLチューブ内の氷冷消化液5mLに移します。海馬を水浴で37°Cで20分間インキュベートします。
- 消化液を取り除き、解剖液の3 mLを追加します。
- パスツールピペットでカバピピを4回軽くピペットします。
- 海馬を水浴で37°Cで20分間インキュベートします。
- 細胞懸濁液を50 mL遠心チューブに移します。解離されていない組織に3 mLの解剖液を加えます。
- 海馬が完全に解約されるまで、ステップ3.7-3.9を繰り返します。
- パスツールピペットで旋回し、吸引することによって、細胞懸濁液からDNAの浮遊凝集体を除去します。細胞懸濁液を180xgで4°Cで20分間遠心分離する。
注:損傷した細胞から得られたDNAは遠心分離プロセスを妨げるでしょう。ニューロンが上清に残っている場合は、DNAを慎重に除去した後に再び細胞懸濁液を遠心分離する。 - 10%のウシ胎児血清(FBS)、ペニシリン(100 U/mL)、ストレプトマイシン(100 μg/mL)を含む神経基底培地を、37°Cの加湿インキュベーターで5%CO2で平衡化した。
4. トランスフェクションと培養ニューロン
- 上清を取り除き、50 mLチューブに5mLの氷冷PBSを加えます。穏やかなピペットによって細胞ペレットを再懸濁する。
- 細胞懸濁液10μLをマイクロ遠心チューブに移し、0.5%のトリパンブルー溶液の10μLを加え、ヘモサイトメーターで細胞数を数えます。
- 50 mLチューブ内の細胞懸濁液を180 x g で4°Cで20分間遠心分離する。
- 一方、トランスフェクションのアリコート、pFN21A-ハロタガアクチン1μg、pmNeonGreen-N1-ビアクト3 μg/1 x 106 細胞、マイクロ遠心チューブに。
- 遠心分離後、上清を取り除き、100 μLのエレクトロポレーション媒体(トランスフェクションキットで提供)を50 mLチューブに加えます。ピペット処理により細胞ペレットを再懸濁する。
- 細胞懸濁液をアリクォートDNA溶液と混合し、キュベット(トランスフェクションキットによって提供される)に混合物を移す。
注:気泡がエレクトロポレーションを邪魔するので、細胞懸濁液からそれらを取り除きます。 - キュベットをエレクトロポレーション装置(材料表)に挿入し、プログラムO-005を用いてエレクトロポレーションを行います。
- 10%FBS、ペニシリン(100 U/mL)、ストレプトマイシン(100 μg/mL)を含む予温および平衡化された神経基底培地をすぐに1mL加えます。
- プラスチックピペット(トランスフェクションキットによって提供される)を使用して、セル懸濁液を15 mL遠心管に移します。
- 細胞懸濁液10μLをマイクロチューブに移し、0.5%のトリパンブルー溶液を10μL加え、ヘモサイトメーターで細胞数を数えます。
- インキュベーターからPDL/ラミニンコーティングガラス底皿を取り出し、神経基底培地を取り除きます。
- 1皿あたり2.0 x 105 細胞を含む細胞懸濁液のピペット0.5mLを、37°Cで加湿インキュベーターにインキュベートし、5%CO2 で3時間培養する。
- 培地を2%B-27サプリメント、グルタミン(1mM)、ペニシリン(100 U/mL)、ストレプトマイシン(100 μg/mL)を含む神経基底培地の0.5mLに交換してください。5%CO2で37°Cで3日間加湿インキュベーターでニューロンを培養します。
5. ニューロン成長コーンでのシングルスペックルイメージング
- イン ビトロ (DIV)3日に、テトラメチルロダミン(TMR)リガンドで、培養培地中の1:2000希釈の希釈でニューロンを治療する。5%CO2で37°Cで1時間のニューロンを維持します。
- TMRリガンドを、あらかじめ温めたPBSで3回洗います。
- 2%B-27サプリメント、グルタミン(1mM)、ペニシリン(100 U/mL)、ストレプトマイシン(100 μg/mL)を含む、温めたライボヴィッツのL-15培地のPBSおよびピペット0.5mLを取り除きます。
- 37°Cで1時間ニューロンを維持する。
- 蛍光顕微鏡をオンにし、ステージトップインキュベーターを37°Cに設定します。
注:本プロトコルは、相補的な金属酸化物半導体カメラ、100x/1.40 NA油浸し対物レンズ、水銀ランプ、画像取得ソフトウェアを搭載した蛍光顕微鏡を使用しています( 材料表参照)。同等の仕様を有する他の蛍光顕微鏡もこの分析に使用することができる。 - TMRリガンド処理したニューロンをガラス底皿に加えて、温めたステージトップインキュベーターに置きます。
- 画像取得パラメータを次のように設定します: 曝露時間、ライフアクトおよびハロタグアクチン蛍光チャネルの 500 ms;ビン、1 x 1 (0.065 μm ×ピクセルあたり 0.065 μm)時間間隔、3秒。期間、50フレーム。
- ライフアクトを強く表現し、ハロタグアクチンを弱く発現する成長コーンを選択します。
注: Lifeact の発現は、成長コーン形態を可視化するのに十分な強度を持つ必要があります。ハロタグアクチン発現は、検出するには弱く不十分であるべきである(図2A)。蛍光タンパク質のレベルは、トランスフェクション用のDNA量を変化させることで調整できます。 - ダイヤフラムのフィールドを閉じて、成長コーンを含む最小領域を照らします(図2B)。
注:最小領域の照明は、バックグラウンド信号を低減し、ノイズ(S/N)比に蛍光信号を増加させ、アクチン斑点の検出を可能にします(図2B)。S/N比をさらに高めるには、中性密度フィルタにより光を減らさずに成長コーンを照明することが推奨されます。 - タイムラプス画像(補足ファイル2)を取得します。マルチチャンネルタイムラプススタックイメージ(tiff形式)として保存します。
6. 画像処理および解析ソフトウェアフィジーを用いたF-アクチン流速と重合速度の定量化
メモ:F-アクチンの流速とアクチン重合率を定量化するための実践データについては 、補足ファイル3 を参照してください。
- フィジーでマルチチャネルタイムラプススタック画像を開きます。
- [ スケール>分析]を選択し、画像のピクセルサイズを設定します。
- フィロポディアまたはラメリポディアのFアクチンバンドル内の最低5つの時間枠のために逆行的に流れるアクチンスペックルを見つけます。
- イメージ >変形>回転を選択し、F-アクチンバンドルが上向きになるように画像の角度を調整します(図3A)。
- ツールバーの 矩形 をクリックし、アクチンの斑点とF-actinバンドルの先端を含むボックスを描きます(図3B、1、2)。
注:ハロタグアクチン斑点の信号は弱いです。さらに、成長コーンの遠位先端におけるLifeactシグナルは弱く、弱い(図2B)成長コーンの遠位先端が近位部に比べて薄いため(図2B)。研究者は、F-アクチンの流速と重合速度の定量を最適化するために信号を強化する必要があります(図3B)。近位成長コーンでのLifeactシグナルは飽和状態になるが、これはF-アクチンの遠位端の決定を妨げない(図3E、黄色の線)。 - [ イメージ>複製] を選択し、5つのタイムフレームを入力します(図3B、3-5)。
- [イメージ > スタック>モンタージュを作成] を選択し、パラメータを入力します。行、1;スケール係数、1。
- [OK] をクリックします。画像モンタージュが画面に表示されます。
- [イメージ>カラー>スプリットチャンネル] を選択します。これは2つの蛍光チャネルを分離する。
- Fアクチン流速の定量化
- [ 測定>分析]を選択し、[ 面積 ]および[ 外接する四角形]を選択します。
- ツールバーの オーバル をクリックし、アクチンのスペックルに円を描きます(図3C、1、2)。
- [ イメージ>オーバーレイ>選択を追加] を選択します。円はイメージモンタージュ上に重ねられます(図3C、3、および4)。
注: オーバーレイの色は、[ 色の編集] >オプション>変更できます。 - 残りの斑点について円のオーバーレイを繰り返します(図3C、5)。
注: アクチンスペックルの中心を正確に特定するには、調査員が斑点に円を重ねるようにしてください。 - ツールバーの [ 直線 ] をクリックし、円の中心をリンクする線を描画します(図 3D、1、2)。
- [ メジャー>分析] を選択します。結果が画面に表示されます(図3D、3)。
注: 結果に表示される 高さは 、観察中のアクチンスペックルの転座距離を示します(図3D、3)。 - 転座距離を観測時間で割ってFアクチン流速を計算します。
- F-アクチン重合率の定量化。
- F-アクチンバンドルの先端をリンクする線を描きます(図3E、1)。
- [ >測定の分析] を選択します。結果が画面に表示されます(図3E、2)。
注: 結果の 高さは 、観察中の F-アクチンバンドルの延長長さを示します(図 3E,2)。 - 延長長を観測時間で割ってFアクチン延長率を計算する。
- F-アクチンの流速と伸長速度の和としてFアクチン重合率を計算します(図3F)。
7. 牽引力顕微鏡とニューロン培養のためのPAAゲルの調製
- 牽引力顕微鏡に必要なPAAゲルの調製(図4)
- ピペット0.5mLのNaOH(100 mM)、蒸留H2O、直径27mmガラス底皿に溶解し、室温(RT)で15分間インキュベートする。
- 3-アミノプロピルトリメチオキシシラン(APTMS)の50 μLを皿のNaOH溶液に加え、穏やかなピペットで溶液を混合し、RTで15分間インキュベートします。
- ガラス底の食器を蒸留したH2Oで洗い、乾かします。
- ピペット0.5 mLの0.5%グルタルアルデヒド溶液は、PBSに溶解し、APTMS処理ガラス底皿に溶解し、RTで30分間インキュベートする。
- ガラス底の食器を蒸留したH2Oで洗い、乾かします。
- 蒸留H2O(412 μL)、30%(w/v)アクリルアミド溶液(63μL)、ビスアクリルアミド溶液(6μL)2.5%(6μL)、カルボンシル酸修飾マイクロスフィア(蛍光ビーズ)溶液(20μL)を含む溶液を調製します。
注:蛍光ビーズ溶液は、アクリルアミド溶液に添加する前に凝集ビーズを解体するボルテックス。 - RTで30分間真空チャンバーで溶液を脱気する。
- この溶液に、10%(w/v)の過硫酸アンモニウム(APS)の1μLを加え、蒸留H2Oに溶解し、1μLのN,N,N',N',N'-テトラメチルエチレンアミン(TEMED)を加え、穏やかなピペットで混合します。
- 15 mL 遠心管のキャップにガラスカバースリップ(直径18mm)を置きます。
- すぐに溶液の25 μLをカバースリップに加え、その後、蓋の上に逆ガラス底皿をそっと置きます。アクリルアミドをRTで1時間重合させる。
- 蒸留したH2Oをカバースリップに塗布し、曲がった先端を持つ注射針を使用してゲルから剥がします。
- H2Oを取り出し、硫化塩SANPAH(1mM)を加え、PBSに溶解し、ゲルに溶解させた。
- 滅菌条件下では、ゲルを紫外線に5分間照射します。ゲルをPBSで3回、15分間洗浄します。
- 微小球の字下げ法を用いたゲル剛性の決定(図5A)44。
- 直径と密度に指定されたマイクロスフィアをゲルに移し、レーザー走査共焦点顕微鏡の標本ステージに置きます。
- ゲル表面に焦点を合わせ、Z位置を記録します(図5B)。
- 同様に、微小球の底面に焦点を当て、z位置を記録します(図5C)。
- ゲルサーフェスのZ位置と微小球の底部の差によって決定されるインデント深度を計算します。
- ゲルのヤング率 E を計算します:
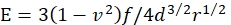
ここで、fは微小球の浮力補正重量、dはゲルのインデント深度、rは微小球の半径、vはポアソン比(値は46で決定された0.3)です。
- セクション2に記載されているように、PDLとラミニンでゲルをコーティングします。
- セクション3および4に記載されているように、E16マウス胚を解剖し、海馬を解離し、1.0 x 106 細胞あたり5μgのpEGFP-C1でトランスフェクトする。PDL/ラミニンコーティングゲルに2.0 x 105 細胞をシードします。
注:牽引力分析では、蛍光標識細胞の顕微鏡イメージングが成長コーン領域を決定するために必要です。
8. ニューロン成長コーンにおける牽引力顕微鏡
- DIV3では、培養培地を、2%B-27サプリメント、グルタミン(1mM)、ペニシリン(100 U/mL)、ストレプトマイシン(100μg/mL)を含む、0.5mLの温めたライボヴィッツのL-15培地に置き換えます。37°Cで1時間ニューロンを維持する。
- レーザー走査型共焦点顕微鏡をオンにし、ステージトップインキュベーターを37°Cに設定します。
注:本研究では、63x/1.2 NA水浸し対物レンズと画像取得および解析ソフトウェアを搭載したレーザー走査型共焦点顕微鏡を使用しています( 材料表参照)。 - 事前に温めたステージトップインキュベーターの上にガラス底皿にニューロンを置きます。
- 次のように画像取得パラメータを設定します: スキャンサイズ、512 ×512ピクセル;スキャン領域、1.5-3倍ズーム;スキャン速度、フレームあたり〜1秒。レーザー波長、561 nm(蛍光ビーズの励起波長)および488nm(強化された緑色蛍光タンパク質(EGFP)の励起波長)。時間間隔、3秒。期間、50フレーム。
- 成長コーン形態を可視化するには、EGFPを強く表現する成長コーンを選択します。
- ゲル表面に焦点を合わせ、タイムラプス画像を取得します(図6A および 補足ファイル4)。
注:トラクション分析には、2つの蛍光チャネル(蛍光ビーズとEGFP用)と明視野チャンネル(差動干渉コントラスト(DIC)または位相コントラスト画像のキャプチャが推奨されます)を使用してください。 - 画像処理ソフトウェア(フィジーなど)を使用して、単一チャンネルのタイムラプスRGB画像スタックを生成し、これらの画像をtiffファイルとして保存します。
注: 調査官が生データも保存することが重要です。 - 100 μLのドデシル硫酸ナトリウム(SDS)を、蒸留H2Oに溶解し、基質からニューロンを放出してゲル基板を弛緩させるガラス底皿に塗布する。37°Cで5分間インキュベートし、温度を安定させます。
注: 訓練されていない基板内のビーズの画像は、牽引力解析中に元のビーズの位置を参照するために使用されます(図6C)。SDS溶液の適用は、インキュベーターの熱変化および細胞誘導変形の緩和によるxyz位置を変化させる。したがって、調査官は、焦点面とx-y位置を修正する必要があります。 - ゲル表面に焦点を当て、訓練を受けていない基板内のビーズの画像を取得する(図6B)。
- 訓練されていない基板内のビーズの単一チャンネルRGB画像を生成し、tiffファイルとして保存します。
- フィジーを用いた非訓練基質におけるビーズ画像のx-y位置の補正
- フィジーでは、訓練されていない基板のビーズ画像と蛍光ビーズのタイムラプススタック画像を開きます。
- [ イメージ> スタック] を選択>ツール>連結します。画面にダイアログボックスが表示されます(図7A、1)。
- 非訓練基板内のビーズ画像と 、Image1 および Image2の蛍光ビーズのタイムラプススタック画像をそれぞれ選択します(図7A、2)。
- [OK] をクリックします。非訓練基板中のビーズ画像は、タイムラプススタック画像に追加される(図7A、3)。
- スクロールバー(図7B、1、赤枠)を使用して、スタック画像の2番目のフレーム(赤い矢印)を表示します。
- StackReg >プラグインを選択します(図7B、2)。画面にダイアログボックスが表示されます(図7B、3)。
- ドロップダウン リストから [リジッド ボディ ] を選択し (図 7B、3、赤枠)、[OK] をクリック します。x-y 位置の補正が開始されます。
- スクロール バーを使用して、x-y 位置補正スタック イメージの最初のフレームを表示します。
- [イメージ>複製]を選択します(図7C、1)。画面にダイアログボックスが表示されます(図7B、2)。
- 範囲に 1 を入力し、重複スタック (図 7C、2) の選択を解除して、「OK」をクリックします。訓練されていない基板内のx-y位置補正ビーズ画像が画面に表示されます(図7C、3)。このイメージを tiff ファイルとして保存します。
- 牽引力の定量化
注: トラクションフォース解析の方法では、MATLAB と 2 つの MATLAB ツールボックス 「イメージ処理ツールボックス」 と 「並列コンピューティング ツールボックス」を使用します。調査官は、分析の前にそれらをインストールする必要があります。牽引力分析コードはMATLABバージョン2018aに基づいて開発されました。したがって、分析には MATLAB バージョン 2018a (またはそれ以降) を使用する必要があります。牽引力分析に使用したアルゴリズムは、前に22を説明した。- 補足ファイル5からトラクションフォース分析コードTFM2021をダウンロードしてください。TFM 2021をMATLABで開く
- TFM2021で メイン.m を開き、実行します。グラフィカル・ユーザー・インターフェース (GUI) が画面に表示されます (図 8A)。
- [訓練されていない基板画像をロード]をクリックし、訓練されていない基板内のx-y位置補正ビード画像を選択します。
- 蛍光ビーズ画像をロードして、ビーズのタイムラプススタック画像を選択します。
- [明視野画像をロード]をクリックし、明視野のタイムラプススタック画像を選択します。
- GFP イメージの読み込みをクリックし、EGFP のタイムラプス スタック イメージを選択します。
- GUI のドロップダウン リストから GFP を選択します(図 8A、赤いボックス)。
- GUI に表示されるセル イメージ上の 2 つのポイントをクリックして、グロース コーンを含む目的の四角形領域 (ROI) を指定するには、[ ROI ] をクリックします(図 8B)。
- GUI の [保存 ]ボタンをクリックします(図8A、赤い矢印)。選択したスタック イメージと ROI は、.mat ファイル (MATLAB 形式ファイル) に保存されます。
- スポット検出をクリックします。画面にダイアログボックスが表示されます。
- ダイアログ ボックスに値 (通常は 50 ~ 150) を入力して、ビード検出のしきい値を決定します。[ OK] を クリックすると、計算が開始されます。
- 計算が終了したら、 プロットトラック をクリックしてステップ8.12.8で選択した領域を拡大し、検出されたビーズを白い点で表示します(図8C、1)。
注:ビード検出(白い点)が蛍光ビーズと重なっているかどうかを確認します。ビード検出は、バックグラウンドノイズを検出することもできます。この外部干渉を軽減するには、スポット検出でしきい値を変更します。さらに、ステップ 8.12.13 で正しいドットを手動で選択します。 - [ビーズを選択]をクリックし、成長円錐の下に正しいドットを含むポリゴン領域を区切ります。キーボードの Enter キーを押します。ポリゴン領域内の白い点は赤い色に変わります(図8C、2)。
- GUI の [推定力 ] をクリックします。次に、次のパラメータの入力値:ピクセルサイズ、μm/ピクセル;ヤング率、ステップ7.2.5で計算された値。ポアソンの比率、0.3。
- 計算を開始するために推定力を実行します。ソフトウェアは、計算結果をスプレッドシート形式のファイルに自動的に保存します。
注: スプレッドシートには、X 成分、y 成分、および各タイムフレームにおける力の大きさが表示されます(図 8D)。牽引力を定量化するための実践データについては 、補足ファイル6 を参照してください。
結果
アクチン重合率とクラッチカップリング効率を定量化する単一スペックルイメージング
高いライフアクトの表現は成長コーンの中でFアクチンの視覚化を可能にする;低いHaloTag-アクチン発現は、F-アクチン逆行流のモニタリングを可能にする(図3、 補足ファイル2)。アクチン斑点のトレースは、F-アクチンの流速の測定を可能にする(図3C、D)。F-アクチン逆行流と接着基材の機械的結合はF-アクチンの流速を低下させるため、クラッチカップリング効率は速度から推定できます。さらに、ライフアクトによるF-アクチン標識は、F-アクチン伸長の可視化を補助し、アクチン重合率を定量化するのに有用である(図3E、F)。
牽引力の定量的分析
ここで提示される方法論を厳密に遵守すると、成長コーン下の蛍光ビーズの動きが明らかになります(図6C、補足ファイル4)。基板上に逆行するF-アクチンの逆行流は、成長コーンの下で蛍光ビーズが後方に移動する原因となる牽引力を発生させる。牽引力分析コードは、蛍光ビーズ変位からの牽引力を推定し、算出された牽引力を力ベクトルとして表現する。牽引力の方向と大きさは、力ベクトルのx成分とy成分から決定されます(図8C,D)。図 8D の赤いボックスは、力ベクトルの x 成分と y 成分を表します。図8Cは、対応する成長コーンを示す。x-y 座標の点では、ベクトルは x 軸に対して -93.8° を指します。この方向は、成長コーンの背面に向いています(図8C)。牽引力Fの大きさは次のように計算した。
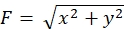
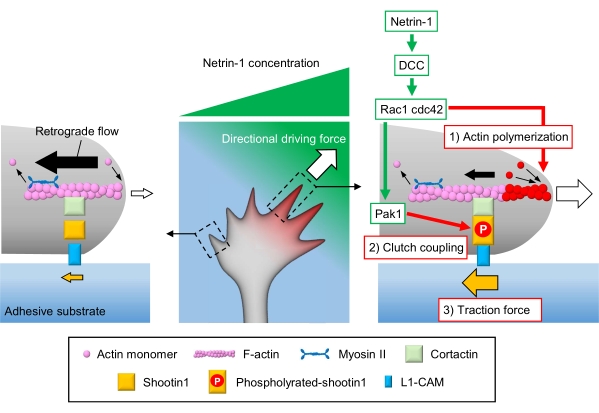
図1:力発生および成長コーンナビゲーションのための成長コーン機械。 ネトリン-1化学誘起剤勾配は、軸索成長コーン上の受容体DCCの非対称的な刺激を誘発する。これにより、Rac1 と Cdc42、および下流キナーゼ Pak1 がアクティブになります。Rac1およびCdc42は(1)アクチン重合を促進し、一方、Pak1はshootin1をリン酸化し、(2)クラッチカップリングを促進する。成長コーン内のアクチンダイナミクスとクラッチカップリングの非対称的な活性化は、ネトリン-1源の側の(3)牽引力を増加させ、それによって成長コーン引き寄せのための指向性駆動力を生成する。ここで示すプロトコルは、成長コーンナビゲーションのための主要変数(1)-(3)の定量化を可能にする。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図2:完全に開いた狭いダイヤフラムの下での神経成長コーンの蛍光画像。成長コーンは、ライフアクトとハロタグアクチンを発現する。 (A)高いライフアクト発現により、成長コーン形態の可視化が可能となる。一方、HaloTag-アクチン発現レベルは非常に低く、ダイヤフラムが完全に開かれたときには薄暗い信号が出ます。(B) 横隔膜が適切に狭くなると、バックグラウンド信号が減少し、成長コーンに単一のアクチン斑点が現れる。スケールバー:5 μm. この図の大きなバージョンを表示するには、ここをクリックしてください。

図3:画像処理および解析ソフトウェアフィジーを用いてF-アクチンの流速とアクチン重合速度を定量化するステップ(A)解析のために画像の角度を調整する。(1) F-アクチンバンドルに流れるアクチンスペックル(矢印頭)を見つけて選択します。(2) [イメージ>変換>回転を選択します。(3) Fアクチンバンドルが上向きになるように角度を設定します。(4) 画像の角度が変更されます。(B)アクチンスペックルとF-アクチンバンドルを含む領域をデマルケートする。(1) ツールバーの長方形をクリックします。(2) 画像上の領域をデリエーションする。画像の明るさとコントラストが増加し、アクチンスペックルとF-アクチンバンドルの先端を明確に視覚化できます。(3) 画像>複製を選択します。(4) F-アクチンバンドルにアクチンスペックルフローを示す5つのタイムフレームを入力します。(5) 選択したスタックイメージが画面に表示されます。(C) アクチンのスペックルに円を重ね合わせ(1) ツールバーのオーバルをクリックします。(2) アクチンのスペックルに円を描きます。(3) [画像>オーバーレイ>選択を追加] を選択します。(4) 円が重ねられます。5)残りのアクチン斑点に円を重ねて繰り返します。(D) 5つのタイムフレームの間にアクチン斑点の転座距離を測定する。(1) ツールバーの[直線]をクリックします。(2) 円の中心を結ぶ線を描きます。(3) [メジャー>分析]を選択します。パラメータ高さ(赤いボックス)で示される結果は、アクチンスペックル転座距離を中継する。(E) 5つの時間枠の間のF-アクチン突起の長さの変化を測定する。(1) F-アクチンの突起の先端をリンクする線を描きます。(2) [>測定を分析]を選択します。結果(赤いボックス)、高さ、F-アクチンバンドルの延長長さを示します。(F)アクチン重合率は、F-アクチン流速と伸長速度の和から算出される。補足ファイル 2 も参照してください。補足ファイル3は、調査官が上記の方法論を実践するのを助けます。この図の大きなバージョンを表示するには、ここをクリックしてください。
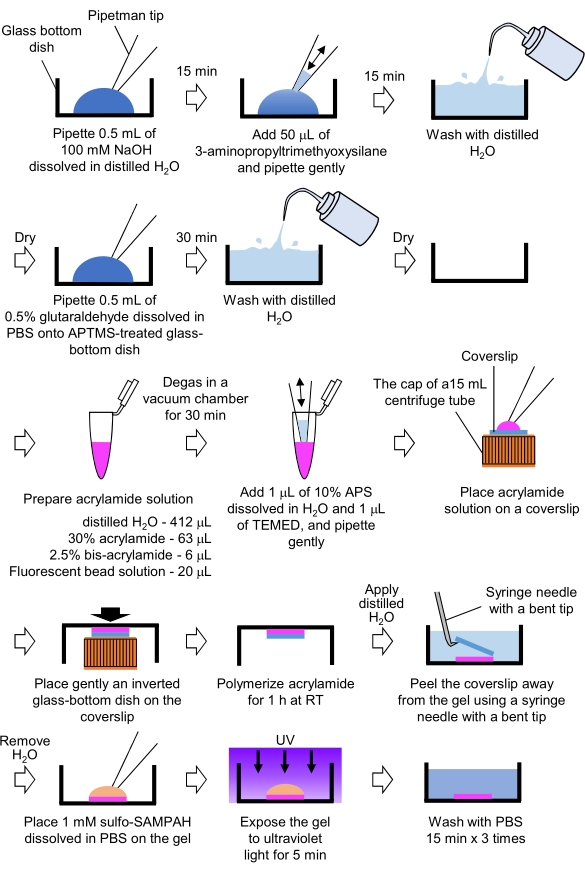
図4:PAAゲル調製のためのステップ。 詳細については、ステップ7.1を参照してください。 この図の大きなバージョンを表示するには、ここをクリックしてください。
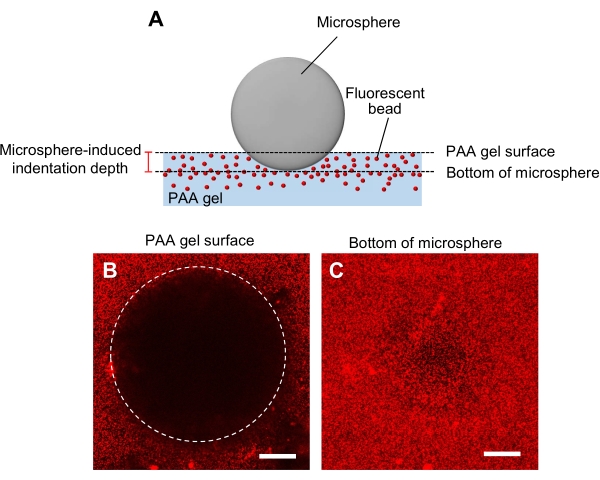
図5:PAAゲル剛性の測定(A) 微小球のインデント法。蛍光ビーズ埋め込みPAAゲルにマイクロスフィアを置くと、微小球の重量がゲルにくぼみを引き起こします。インデント深度は、微小球(B,C)の下面からPAAゲル表面のZ位置を微小球でインデントし、蛍光ビーズを含むPAAゲルの蛍光画像から差し引いて計算されます。レーザー走査共焦点顕微鏡を用いて、ゲル表面(B)と微小球の底部(C)の画像を撮影した。蛍光ビーズからのシグナルは、インデントされた領域(B、円)のゲル表面では見えません。しかし、それらは微小球の底部で観察することができる。スケールバー: 50 μm. この図の大きなバージョンを表示するには、ここをクリックしてください。
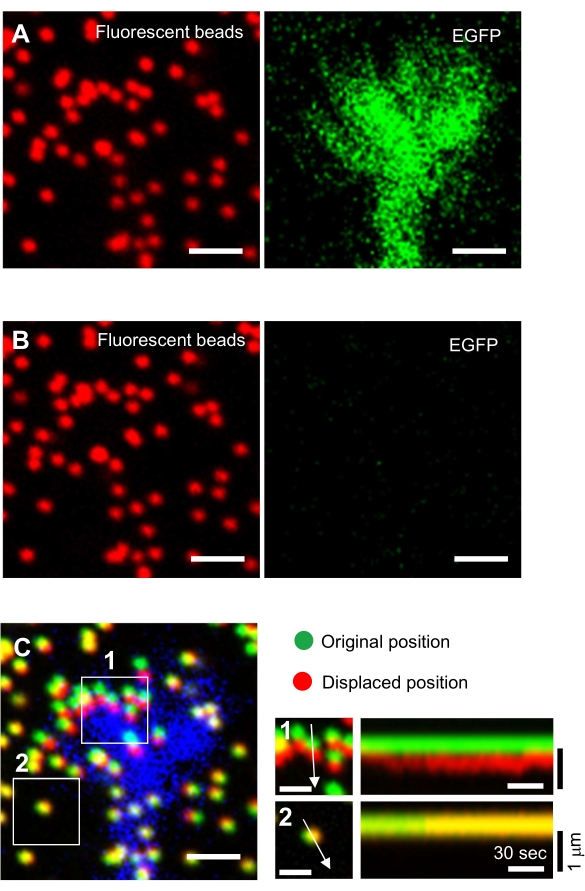
図6:神経成長コーンの強制マッピング (A-C)EGFPで可視化されたPAAゲルおよび神経成長コーンに埋め込まれたビーズの蛍光画像。タイムラプス画像(A)の取得後、SDS溶液を塗布してゲル基板からニューロンを放出し、未訓練の基質内のビーズの画像を取り込んだ(B)。(C) 非訓練基材のビーズの画像は、ビーズを元の(緑色)および変位(赤)位置に示しています。成長コーンのEGFP信号は青色で示される。キモグラフ(右)は、ボックス化された領域1と2の矢印で示される147 sの持続時間の3 s間隔でビーズの動きを示しています。エリア 2 のビードは参照ビードです。スケールバー:(A)、(B)、(C、左)の場合は2μm、(C、中央)は1μm。補足ファイル 4 も参照してください。この図の大きなバージョンを表示するには、ここをクリックしてください。
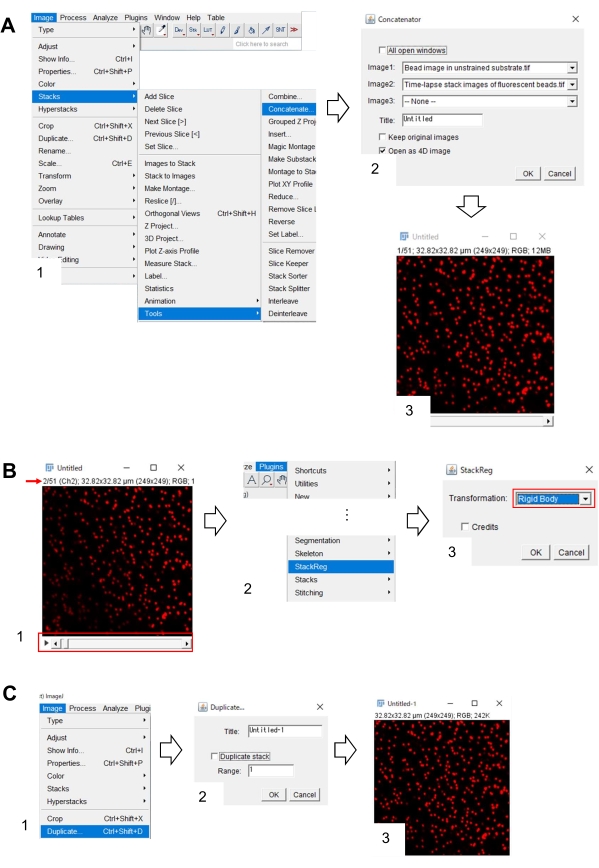
図7:フィジーを用いた不緊張化基質におけるビーズ画像のx-y位置を補正するためのステップ(A)蛍光ビーズのタイムラプススタック画像と非緊張基板内のビーズ画像を連結する。(1) [イメージ > スタック] を選択>[ツール]> [連結]を選択します。(2)画像1とImage2の蛍光ビーズの未訓練の基質内のビーズ画像と、蛍光ビーズのタイムラプススタック画像をそれぞれ選択します。[OK] をクリックします。(3)非訓練基板中のビーズ画像をタイムラプススタック画像に追加する。(B)蛍光ビーズ像のx-y位置を補正する。(1) スクロールバー(赤枠)を使用して、イメージスタックの2番目のフレーム(赤い矢印)を選択します。(2) StackReg >プラグインを選択します。(3) ドロップダウン リスト(赤枠)から[リジッド ボディ]を選択し、[OK]をクリックします。x-y 位置の補正が開始されます。(C) x-y 位置補正ビードイメージを保存します。x-y 位置補正スタックイメージの最初のフレームを選択した後、(1) [イメージ>複製]を選択します。(2) 範囲に 1 を入力し、「重複スタック」の選択を解除します。次に、[OK] をクリックします。(3)未訓練基板内のx-y位置補正ビーズ画像が画面に表示されます。このイメージを tiff ファイルとして保存します。補足ファイル6は、研究者が上記の方法論を実践するのに助けます。この図の大きなバージョンを表示するには、ここをクリックしてください。
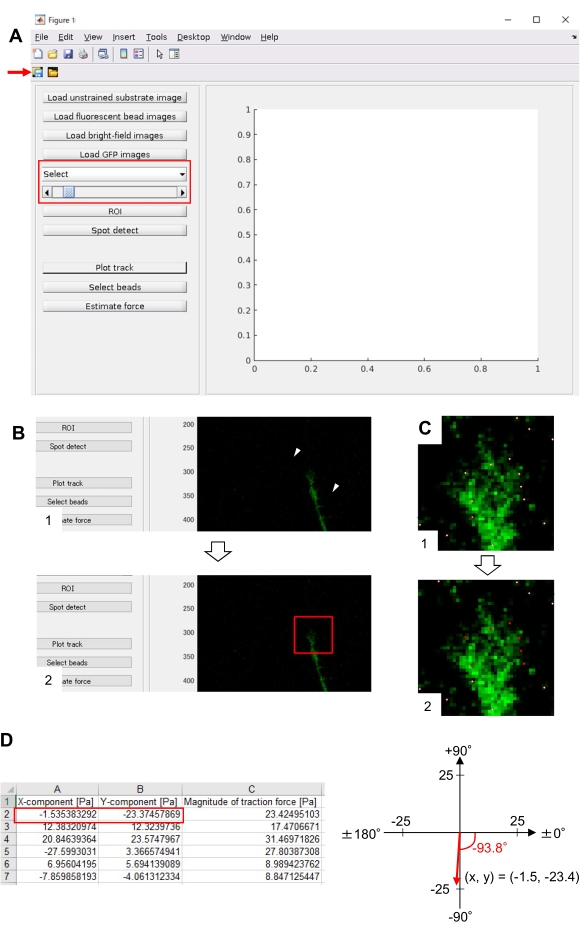
図8:オープンソースの牽引力分析コードを用いたニューロン成長コーン下の牽引力の分析。 (A)牽引力を分析するためのGUI。GUIで選択したタイムラプス画像は、ドロップダウンリストと表示されたスライダー(赤色のボックス)で確認できます。(B) 成長コーンを含むリージョンを選択します。(1) GUIの ROI をクリックします。マウスカーソルで、セルイメージ上の2つの点(矢印)を指定します。(2) セル画像に赤いボックスが表示されます。2 つのクリックで 2 つのコーナーの位置が決まります。(C) 成長円錐の下で検出されたビーズ(白い点)を選択します。(1) GUI で[ ビーズを選択]をクリックし、クリックして成長円錐を含むポリゴン領域を区切ります。 Enter キーを押します。(2) ポリゴン領域内の白い点が赤い色に変わります。(D) 動力力の方向と大きさの計算結果。スプレッドシートの赤いボックスは、力の x 成分と y 成分を表し、牽引力解析によって推定されます。右パネルの x-y 座標では、成長円錐によって生成される力ベクトルは x 軸に対して -93.8° を指します。この方向は、(C)の成長コーンの背面に向けられる。 補足ファイル6 は、研究者が上記の方法論を実践するのに助けます。 この図の大きなバージョンを表示するには、ここをクリックしてください。
補足ファイル1:この研究で使用されるソリューションとメディアのレシピ。 詳しい使用方法については、テキストを参照してください。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル2:神経成長コーンにおけるハロタグアクチンのライフアクトおよび蛍光スペックルイメージングの蛍光イメージング。 ライフアクト(緑)とハロタグアクチン(マゼンタ)。画像は合計147 sの間、3 sごとに取得されました。スケールバー:2 μm。 図 3. このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル3:F-アクチンの流速とアクチン重合速度を定量化するための実践データ。 ライフアクト(緑)とハロタグアクチン(マゼンタ)のマルチチャンネルタイムラプススタック画像。 図 3 も参照してください。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル4:ニューロン成長コーンでの牽引力を検出する力マッピングビデオ。 ビーズの元の(緑)と変位(赤)の位置。成長コーンのEGFPシグナルは青色で示されています。画像は147 sで3sごとに取得した。スケールバー:2 μm。 図 6 も参照してください。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル5:牽引力分析コード。 詳細な使用方法については、ステップ8.12を参照してください。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル6:牽引力を定量化するためのデータを練習します。 成長コーン、EGFP、および明視野下の蛍光ビーズの非訓練基板および単一チャネルタイムラプススタックRGB画像のビーズの単一チャネルRGB画像。 図 7 および 8 も参照してください。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
本研究で説明したプロトコルは、すべての研究所、研究所、大学で日常的に見られる市販の材料や顕微鏡検査装置を使用しています。したがって、研究者は、現在の単一のスペックルイメージングと牽引力顕微鏡を研究に容易に採用することができます。
スペックルイメージングはアクチン重合とクラッチカップリングを解析できます。さらに、スペックルイメージングは、F-アクチン逆行流と相互作用するshootin1やコルタクチンなどのクラッチ分子の逆行流を監視することができます。TIRF顕微鏡を用いることで、細胞接着分子L1-CAMの逆行流も23,41をモニターすることができる。L1-CAMはクラッチカップリング効率23,41を反映したグリップおよびスリップの挙動を受ける。本研究では、スペックルイメージングにTMR-HaloTagシステムを採用していますが、EGFPや単量体赤色蛍光タンパク質などの他の蛍光タンパク質も分析16,18,20,23,24,27,39で入手可能です。アクチンの斑点を可視化するための基本は、蛍光アクチンの発現量が低く、最小領域の照明である(図2)。このプロトコルでは、ライフアクトおよびハロタグアクチン信号が順次取得されます。アクチン逆行流は比較的遅いため(4.5±0.1 μm/min)24、F-アクチン逆行流量とアクチン重合の解析は、異なる蛍光チャネルの逐次画像取得(〜1秒間隔)の影響を受けません。ライフアクトは広く使用されているF-アクチンマーカーですが、アクチン結合タンパク質47と競合することができます。さらに重要なのは、Lifeactがアクチンダイナミクスを変化させ、それによってF-アクチン構造および細胞形態47,48,49に影響を及ぼす可能性がある。
牽引力顕微鏡は成長コーンの前進を促進する力を検出できる。細胞外マトリックス内にニューロンを取り付けることで、研究者は半3D環境11で発生する力を分析することもできる。高倍率イメージングは、成長コーンが弱い牽引力を生成するため、牽引力を正確に定量するために重要です7。また、ナノピラーやストレス感受性バイオセンサーを用いる他の方法も牽引力50,51を測定するために使用されますが、PAAゲルベースの方法は非常に適応性が高く、アクリルアミドとビスアクリルアミド41,44,52の濃度を変化させることによって基質剛性の調整を可能にします。このプロトコルでは、PAAゲルは3.75%アクリルアミドおよび0.03%ビスアクリルアミドの最終濃度で調製される。ヤング率は~270 Pa22であり、この剛性は脳組織の範囲内(100-10,000 Pa)53,54,55である。PAAゲルの厚さ(〜100 μm)のため、顕微鏡での高倍率レンズの使用を制限します。高倍率画像を得るためには、研究者はレーザー走査型共焦点顕微鏡でズーム機能を使用する必要があります。
結論として、本スペックルイメージングとトラクションフォース顕微鏡は、力世代における主要な事象の定量的分析を可能にする。この情報は、成長コーンの進歩とナビゲーションの根源となるメカニズムの理解を深める上で非常に貴重です。
開示事項
著者らは開示するものは何もない。
謝辞
この研究は、助成金番号21gm0810011h0005(N.I.)の下でAMEDによって部分的にサポートされました。 そしてY.S.)、JSPS KAKENHI(JP19H03223、N.I.)、JSPS早期研究者のための援助助成金(JP19K16258、T.M.)、大阪不治病医学研究財団(T.M.)、NAIST次世代学際研究プロジェクト(Y.S.)
資料
| Name | Company | Catalog Number | Comments |
| 0.5% trypan blue stain solution | Nacalai | 29853-34 | |
| 3-aminopropyltrimethyoxysilane | Sigma | 281778-100ML | |
| Acrylamide monomer | Nacalai | 00809-85 | |
| Ammonium persulphate | Cytiva | 17-1311-1 | |
| Axio Observer Z1 | Zeiss | 431007-9902-000 | Epi-fluorescence microscope (single speckle imaging) |
| B-27 supplement (50x) | Thermo Fisher Scientific | 17504-044 | |
| Bovine serum albmine | Sigma | A7906-10G | |
| C-Apochromat 63x/1.2 W Corr | Zeiss | 421787-9970-799 | Objective lens (traction force microscopy) |
| Coverslip (diameter 18 mm) | Matsunami | C018001 | |
| D-glucose | Nacalai | 16806-25 | |
| DNaseI | Sigma | DN25-100MG | |
| Fetal bovine serum | Thermo Fisher Scientific | 10270-106 | |
| Fiji | Open source software package | https://imagej.net/software/fiji/ | |
| FluoSpheres carboxylate-modified 0.2 mm, red (580/605), 2% solid | Thermo Fisher Scientific | F8810 | carboxylate-modified microspheres |
| Glass bottom dish (14 mm diameter) | Matsunami | D1130H | |
| Glass bottom dish (27 mm diameter) | Matsunami | D1140H | |
| Glutaraldehyde solution | Sigma | G5882-10X10ML | |
| HaloTag TMR ligand | Promega | G8251 | |
| HBO103 W/2 | Osram | 4050300382128 | Mercury lamp (single speckle imaging) |
| Image Processing Toolbox | MathWork | https://www.mathworks.com/products/image.html | |
| Laminin solution from mouse EHS tumor | Wako | 120-05751 | |
| Leibovitz’s L-15 medium | Thermo Fisher Scientific | 11415064 | |
| L-glutamine | Nacalai | 16919-42 | |
| LSM710 | Zeiss | N/A | Conforcal laser microscope (traction force microscopy) |
| MATLAB2018a | MathWork | https://www.mathworks.com/products/new_products/release2018a.html | |
| Mouse C57BL/6 | Japan SLC | N/A | |
| Mouse neuron nucleofector kit | Lonza | VPG-1001 | |
| N,N,N’,N’-tetramethylethylenediamine (TEMED) | Nacalai | 33401-72 | |
| N,N’-methylenebisacrylamide | Nacalai | 22402-02 | |
| Neurobasal medium | Thermo Fisher Scientific | 21103-049 | |
| Nucleofector I | Amaxa | AAD-1001 | Electroporation apparatus |
| ORCA Flash 4.0 V2 | Hamamatsu | C11440-22CU | CMOS camera (single speckle imaging) |
| Papain | Nacalai | 26036-34 | |
| Parallel Computing Toolbox | MathWork | https://www.mathworks.com/products/parallel-computing.html | |
| pEGFP-C1 | Clontech | 1528177 | |
| Penicillin-streptomycin (100x) | Nacalai Tesque | 26253-84 | |
| pFN21A-HaloTag-actin | (Minegishi et al., 2018) | N/A | |
| Phosphate buffered saline (PBS) pH 7.4 (10x) | Thermo Fisher Scientific | 70011-044 | |
| Plan-Apochromat 100x/1.4 Oil | Zeiss | 420790-9901-000 | Objective lens (single speckle imaging) |
| pmNeonGreen-N1-Lifeact | (Kastian et al., 2021) | N/A | |
| Poly-D-lysine hydrobromide | Sigma | P6407-5MG | |
| Slulfo-SAMPHA | Thermo Fisher Scientific | 22589 | |
| Sodium dodecyl sulfate | Nacalai | 08933-05 | |
| Sodium hydrate (NaOH) | Nacalai | 31511-05 | |
| Steel ball | Sako tekkou | N/A | Microshpere to determin PAA gel rigidity. 0.6 mm diameter, 7.87 g/cm3. |
| ZEN2009 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (traction force microscopy) |
| ZEN2012 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (single speckle imaging) |
参考文献
- TessierLavigne, M., Goodman, C. S. The molecular biology of axon guidance. Science. 274, 1123-1133 (1996).
- Marin, O., Valiente, M., Ge, X., Tsai, L. H. Guiding neuronal cell migrations. Cold Spring Harbor Perspectives in Biology. 2, 001834 (2010).
- Vitriol, E. A., Zheng, J. Q. Growth cone travel in space and time: the cellular ensemble of cytoskeleton, adhesion, and membrane. Neuron. 73 (6), 1068-1081 (2012).
- Cooper, J. A. Cell biology in neuroscience: mechanisms of cell migration in the nervous system. Journal of Cell Biology. 202, 725-734 (2013).
- Dupraz, S., et al. RhoA controls axon extension independent of specification in the developing brain. Current Biology. 29, 3874-3886 (2019).
- Suter, D. M., Miller, K. E. The emerging role of forces in axonal elongation. Progress in Neurobiology. 94, 91-101 (2011).
- Franze, K. Integrating chemistry and mechanics: the forces driving axon growth. Annual Review of Cell and Developmental Biology. 36, 61-83 (2020).
- Mitchison, T., Kirschner, M. Cytoskeletal dynamics and nerve growth. Neuron. 1, 761-772 (1988).
- Suter, D. M., Forscher, P. Substrate-cytoskeletal coupling as a mechanism for the regulation of growth cone motility and guidance. Journal of Neurobiology. 44, 97-113 (2000).
- Lowery, L. A., Van Vactor, D. The trip of the tip: understanding the growth cone machinery. Nature Reviews Molecular Cell Biology. 10, 332-343 (2009).
- Minegishi, T., et al. Shootin1b mediates a mechanical clutch to produce force for neuronal migration. Cell Reports. 25, 624-639 (2018).
- Minegishi, T., Inagaki, N. Forces to drive neuronal migration steps. Frontiers in Cell and Developmental Biology. 8, 863 (2020).
- Forscher, P., Smith, S. J. Actions of cytochalasins on the organization of actin filaments and microtubules in a neuronal growth cone. Journal of Cell Biology. 107, 1505-1516 (1988).
- Suter, D. M., Forscher, P. An emerging link between cytoskeletal dynamics and cell adhesion molecules in growth cone guidance. Current Opinion in Neurobiology. 8, 106-116 (1998).
- Pollard, T. D., Borisy, G. G. Cellular motility driven by assembly and disassembly of actin filaments. Cell. 112, 453-465 (2003).
- Waterman-Storer, C. M., Desai, A., Bulinski, J. C., Salmon, E. D. Fluorescent speckle microscopy, a method to visualize the dynamics of protein assemblies in living cells. Current Biology. 8, 1227-1230 (1998).
- Katoh, K., Hammar, K., Smith, P. J., Oldenbourg, R. Birefringence imaging directly reveals architectural dynamics of filamentous actin in living growth cones. Molecular Biology of the Cell. 10, 197-210 (1999).
- Watanabe, N., Mitchison, T. J. Single-molecule speckle analysis of actin filament turnover in lamellipodia. Science. 295, 1083-1086 (2002).
- Medeiros, N. A., Burnette, D. T., Forscher, P. Myosin II functions in actin-bundle turnover in neuronal growth cones. Nature Cell Biology. 8, 215-226 (2006).
- Shimada, T., et al. Shootin1 interacts with actin retrograde flow and L1-CAM to promote axon outgrowth. Journal of Cell Biology. 181, 817-829 (2008).
- He, M., Zhang, Z. H., Guan, C. B., Xia, D., Yuan, X. B. Leading tip drives soma translocation via forward F-actin flow during neuronal migration. Jounal of Neuroscience. 30, 10885-10898 (2010).
- Toriyama, M., Kozawa, S., Sakumura, Y., Inagaki, N. Conversion of a signal into forces for axon outgrowth through Pak1-mediated shootin1 phosphorylation. Current Biology. 23, 529-534 (2013).
- Abe, K., et al. Grip and slip of L1-CAM on adhesive substrates direct growth cone haptotaxis. Proceedings of the National Academy of Sciences of the United States of America. 115, 2764-2769 (2018).
- Baba, K., et al. Gradient-reading and mechano-effector machinery for netrin-1-induced axon guidance. Elife. 7, 34593 (2018).
- Nichol, R. I., Hagen, K. M., Lumbard, D. C., Dent, E. W., Gomez, T. M. Guidance of axons by local coupling of retrograde flow to point contact adhesions. Journal of Neuroscience. 36, 2267-2282 (2016).
- Shekarabi, M., et al. Deleted in colorectal cancer binding netrin-1 mediates cell substrate adhesion and recruits Cdc42, Rac1, Pak1, and N-WASP into an intracellular signaling complex that promotes growth cone expansion. Journal of Neuroscience. 25, 3132-3141 (2005).
- Kubo, Y., et al. Shootin1-cortactin interaction mediates signal-force transduction for axon outgrowth. Journal of Cell Biology. 210, 663-676 (2015).
- Huber, A. B., Kolodkin, A. L., Ginty, D. D., Cloutier, J. F. Signaling at the growth cone: ligand-receptor complexes and the control of axon growth and guidance. Annual Review of Neuroscience. 26, 509-563 (2003).
- Kolodkin, A. L., Tessier-Lavigne, M. Mechanisms and molecules of neuronal wiring: a primer. Cold Spring Harbor Perspectives in Biology. 3, 001727 (2011).
- Ayala, R., Shu, T., Tsai, L. H. Trekking across the brain: the journey of neuronal migration. Cell. 128, 29-43 (2007).
- Tatavarty, V., Kim, E. J., Rodionov, V., Yu, J. Investigating sub-spine actin dynamics in rat hippocampal neurons with super-resolution optical imaging. PLoS One. 4, 7724 (2009).
- Frost, N. A., Shroff, H., Kong, H., Betzig, E., Blanpied, T. A. Single-molecule discrimination of discrete perisynaptic and distributed sites of actin filament assembly within dendritic spines. Neuron. 67, 86-99 (2010).
- Chazeau, A., et al. Nanoscale segregation of actin nucleation and elongation factors determines dendritic spine protrusion. EMBO Journal. 33, 2745-2764 (2014).
- Garcia, M., et al. Two-tiered coupling between flowing actin and immobilized N-cadherin/catenin complexes in neuronal growth cones. Proceedings of the National Academy of Sciences of the United States of America. 112, 6997-7002 (2015).
- Swaminathan, V., et al. Actin retrograde flow actively aligns and orients ligand-engaged integrins in focal adhesions. Proceedings of the National Academy of Sciences of the United States of America. 114, 10648-10653 (2017).
- Tsai, T. Y., et al. Efficient front-rear coupling in neutrophil chemotaxis by dynamic myosin II localization. Developmental Cell. 49, 189-205 (2019).
- Zhang, X. F., et al. Regulation of axon growth by myosin II-dependent mechanocatalysis of cofilin activity. Journal of Cell Biology. 218 (7), 2329-2349 (2019).
- Reversat, A., et al. Cellular locomotion using environmental topography. Nature. 582, 582-585 (2020).
- Katsuno, H., et al. Actin migration driven by directional assembly and disassembly of membrane-anchored actin filaments. Cell Reports. 12, 648-660 (2015).
- Urasaki, A., et al. Shootins mediate collective cell migration and organogenesis of the zebrafish posterior lateral line system. Scientific Reports. 9, 12156 (2019).
- Abe, K., et al. Mechanosensitive axon outgrowth mediated by L1-laminin clutch interface. Biophysical Journal. 120, 3566-3576 (2021).
- Kastian, R. F., et al. Shootin1a-mediated actin-adhesion coupling generates force to trigger structural plasticity of dendritic spines. Cell Reports. 35, 109130 (2021).
- Riedl, J., et al. Lifeact: a versatile marker to visualize F-actin. Nature Methods. 5, 605-607 (2008).
- Chan, C. E., Odde, D. J. Traction dynamics of filopodia on compliant substrates. Science. 322, 1687-1691 (2008).
- Wang, Y. L., Pelham, R. J. Preparation of a flexible, porous polyacrylamide substrate for mechanical studies of cultured cells. Methods in Enzymology. 298, 489-496 (1998).
- Li, Y., Hu, Z., Li, C. New method for measuring poisson's ratio in polymer gels. Journal of Applied Polymer Science. 50, 1107-1111 (1993).
- Belyy, A., Merino, F., Sitsel, O., Raunser, S. Structure of the Lifeact-F-actin complex. PLoS Biology. 18, 3000925 (2020).
- Flores, L. R., Keeling, M. C., Zhang, X., Sliogeryte, K., Gavara, N. Lifeact-GFP alters F-actin organization, cellular morphology and biophysical behaviour. Scientific Reports. 9, 3241 (2019).
- Kumari, A., Kesarwani, S., Javoor, M. G., Vinothkumar, K. R., Sirajuddin, M. Structural insights into actin filament recognition by commonly used cellular actin markers. EMBO Journal. 39, 104006 (2020).
- du Roure, O., et al. Force mapping in epithelial cell migration. Proceedings of the National Academy of Sciences of the United States of America. 102, 2390-2395 (2005).
- Grashoff, C., et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 466, 263-266 (2010).
- Koch, D., Rosoff, W. J., Jiang, J., Geller, H. M., Urbach, J. S. Strength in the periphery: growth cone biomechanics and substrate rigidity response in peripheral and central nervous system neurons. Biophysical Journal. 102 (3), 452-460 (2012).
- Barnes, J. M., Przybyla, L., Weaver, V. M. Tissue mechanics regulate brain development, homeostasis and disease. Journal of Cell Science. 130, 71-82 (2017).
- Moore, S. W., Roca-Cusachs, P., Sheetz, M. P. Stretchy proteins on stretchy substrates: the important elements of integrin-mediated rigidity sensing. Developmental Cell. 19, 194-206 (2010).
- Tyler, W. J. The mechanobiology of brain function. Nature Reviews Neuroscience. 13, 867-878 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved