Method Article
考古学的遺跡から古代DNAを検索するための最適化された骨サンプリングプロトコル
要約
このプロトコルは、中世の個人からの5つの異なる骨格要素(放射性炭素年代測定)にわたる8つの推奨される解剖学的サンプリング場所(特定の骨格要素の特定の場所)から骨粉末を収集するための一連のベストプラクティスプロトコルを提示します西暦1040年から1400年頃、較正された2シグマ範囲)。
要約
ここで紹介する方法は、投入サンプル材料を制限しながら、古代の考古学的遺跡からヒトDNAを回収する可能性を最大化することを目指しています。これは、骨格全体のDNA回収率の比較分析において、最も多くの古代DNA(aDNA)を生成するように以前に決定された解剖学的サンプリング場所をターゲットにすることによって行われました。以前の研究では、これらのプロトコルが考古学的遺跡から古代のヒトおよび病原体DNAの回収に成功する可能性を最大化することが示唆されています。DNA収量は、ドイツ・パイセン近郊の放棄された中世の集落であるクラカウアーベルクの中世(西暦1040年から1400年頃の放射性炭素年代測定、較正された2シグマ範囲)墓地から回収された11人の個人からの複数の骨格要素にわたるaDNA保存の広範な調査で、Parkerらによって以前に評価されました。5つの骨格要素(ペトロサ、永久大臼歯、胸椎、遠位指節骨、距骨)にまたがるこれらの8つのサンプリングスポットは、すべての要素と個体の全体的な平均よりも収量が有意に高かった高品質の古代ヒトDNAを生成することに成功しました。収量は、ほとんどの一般的な下流集団遺伝子解析で使用するのに十分でした。私たちの結果は、考古学的遺跡からの古代の人間のDNAの分析を含むほとんどの研究で、これらの解剖学的サンプリング場所を優先的に使用することを支持しています。これらの方法の実装は、貴重な考古学的標本の破壊を最小限に抑えるのに役立ちます。
概要
DNAの回収と分析を目的とした古代の人間の遺体のサンプリングは、本質的に破壊的です1,2,3,4。サンプル自体は貴重な標本であり、形態学的保存は可能な限り保存する必要があります。そのため、かけがえのない材料の不必要な破壊を回避し、成功の確率を最大化するために、サンプリング方法を最適化することが不可欠です。現在のベストプラクティス技術は、法医学調査5,6、最適なサンプリングの開発が研究の直接の目的ではない古代標本の研究7、または人間以外の遺体8または非常に少数の解剖学的サンプリング場所(ここでは、骨粉の骨格要素の特定の領域を示すために使用されます、 ダウンストリームDNA分析で使用するために、生成されました)9,10。ここで提示されたサンプリングプロトコルは、複数の個人からの複数の骨格要素にわたるDNA保存に関する最初の大規模な体系的研究で最適化されました11。すべてのサンプルは、ドイツのザクセンアンハルト州パイセン近くのクラカウアーベルクの放棄された中世の集落の教会の墓地から発掘された11人の個人から回収された骨格要素に由来し(詳細なサンプル人口統計については表1を参照)、そのため、この地理的/時間的範囲外のサンプルで使用するには変更が必要な場合があります。
| 個体 | 性 | 推定死亡年齢 | 14名C 日付 (CE, Cal 2-シグマ) |
| KRA001 | 男性 | 25-35 | 1058-1219 |
| KRA002 | 女性 | 20-22 | 1227-1283 |
| KRA003 | 男性 | 25 | 1059-1223 |
| KRA004 | 男性 | 15 | 1284-1392 |
| KRA005 | 男性 | 10-12 | 1170-1258 |
| KRA006 | 女性 | 30-40 | 1218-1266 |
| KRA007 | 女性 | 25-30 | 1167-1251 |
| KRA008 | 男性 | 20 | 1301-1402 |
| KRA009 | 男性 | 不明 | 1158-1254 |
| KRA010 | 男性 | 25 | 1276-1383 |
| KRA011 | 女性 | 30-45 | 1040-1159 |
表1:サンプリングされた11人全員の遺伝的に決定された性別、考古学的に決定された推定死亡年齢、および放射性炭素年代測定(14 C Cal 2-シグマ)。この表は、Parker, C. et al. 202011から改作されたものである。
これらのプロトコルにより、実験室によるDNA汚染を制限しながら、5つの骨格要素(パースペトロサを含む)にわたる8つの解剖学的サンプリング場所から比較的簡単で効率的な骨粉末の生成が可能になります。これらの5つの骨格要素のうち、4つの骨格要素に見られる7つの解剖学的サンプリング位置は、錐体ピラミッド11,12の破壊的サンプリングの実行可能な代替手段であると決定されています。これらには、永久大臼歯のセメント質、象牙質、および歯髄室が含まれます。皮質骨は、上椎骨ノッチおよび胸椎の体から集まった。先端房の下面および遠位指骨のシャフトに由来する皮質骨。タリの外側部分に沿った密な皮質骨。パースペトロサ4、12、13、14、象牙質、および歯髄室1,2,15のサンプリングにはいくつかの広く適用されている方法がありますが、セメント質からの骨粉末の生成の成功を説明する公開された方法16、椎体、下椎骨ノッチ、距骨は入手が困難な場合があります。そのため、ここでは、ペトロスピラミッド用に最適化されたサンプリングプロトコルを示します(ステップ3.1)。成人大臼歯のセメント質(ステップ3.2.1)、象牙質(ステップ3.2.2)、および歯髄(ステップ3.2.3)。椎体の皮質骨(ステップ3.3.1)および上椎弓(ステップ3.3.2)。遠位指節骨(ステップ3.4);aDNAと法医学研究の両方のためにこれらの骨格要素を効果的に利用するために、距骨(ステップ3.5)をより広く利用できるようにします。
プロトコル
ここに提示されたすべての研究は、ドイツのイエナにあるマックスプランク人類史科学研究所が古代の人間の遺体を扱うために定めたガイドラインに準拠して実施されました。このプロトコルのステップを実行する前に、科学的研究の許可の取得と、お住まいの地域での破壊的なサンプリングのための人間の遺体の使用の両方に関連するすべての地方/州/連邦の倫理要件を順守するようにしてください。すべての手順/化学物質の保管は、個々の施設の安全ガイドラインに従って実行する必要があります。
1. サンプル処理前の考慮事項
- 古代の遺跡は複製不可能で有限のリソースであるため、サンプルは慎重に扱います(たとえば、サンプリングは可能な限り無駄を最小限に抑え、可能であればすべての遺骨をそれぞれの合法的なプロバイダーに返却する必要があります)。
- すべてのステップはクリーンルーム環境、できれば専用の古代DNA施設で実行してください17,18,19。フード付きの滅菌微孔性つなぎ服、滅菌手袋(2ペア)、サージカルマスク、保護眼鏡、滅菌カバー付きの滅菌ブーツまたは滑り止め靴で構成される個人用保護具(PPE)を使用してください(材料の表を参照)。特にサンプル間で手袋を頻繁に交換してください。
- 可能な場合は、漂白剤/DNA除染溶液/エタノールおよびUV照射(波長:254 nm)ですべての機器と表面を完全に洗浄および消毒します(例:ドリルビット、ドリル、バイス/クランプなど)。最後に、クリーンルーム環境による過度の疲労を避けるために、定期的に人間工学に基づいた休憩(可能であれば2〜3時間ごと)を取ることを強くお勧めします。
注:すべての骨格の遺体は、サンプリングの前に適切に文書化する必要があります(たとえば、写真撮影、計量、可能であればマイクロCTスキャン、3D画像化など)(適切な文書化のプロトコルはこの原稿ではカバーされていません)。すべてのサンプリングプロトコルは、サンプリング反復の間に一時停止することができ、サンプルは乾燥した温度制御された(25°C)無菌環境に無期限に保存できます。
2.前処理
- 骨粉を生成する前にすべての解剖学的サンプリング場所を除染して、汚染のリスクを最小限に抑えます18。
注:サンプル除染のための漂白剤および/または表面除去の有効性(表面除去手順についてはステップ3.3.2の注を参照)は、aDNA研究者の間でまだ議論の的となっています8,19,20,21,22,23,24,25は、特に高度に分解されたサンプルで全体的なDNA収量に影響を与える可能性があるためです。そのため、次の手順はオプションと見なされ、このホワイト ペーパーで示されている代表的な結果を生成するためにすべてのサンプルで使用されたため、ここに含まれています。これらの前処理プロトコルの使用は、各サンプルセットの分子用途、年齢、希少性、および形態学的劣化のレベルに基づいて、ケースバイケースで決定することをお勧めします。- UV光を備えたポリメラーゼ連鎖反応(PCR)フードまたはエアフローをオフにしたバイオセーフティキャビネットの下、専用のクリーンルームですべてのサンプリングを実行します。ベンチトップ全体に滅菌アルミホイルを広げて、浮遊骨粉/破片をキャッチします。
- ホイルを処分する前に、すべての骨片が(本国送還のために)回収されていることを確認してください。各骨格要素の処理間でホイルを変更します。使用済みのホイルは、オートクレーブ可能なバイオハザードバッグ/レセプタクルに入れて廃棄してください。
- 糸くずの出ない乾いた滅菌ワイプでその領域をそっと拭いて、解剖学的サンプリング場所からできるだけ多くの緩い汚れ/残骸を取り除きます( 材料の表を参照)。ワイプは、オートクレーブ可能なバイオハザードバッグまたはレセプタクルに入れて廃棄してください。
- 希釈した市販の漂白剤(~0.01% v/v、超高純度のDNase/RNaseフリー水で希釈)で湿らせた滅菌ワイプで拭いて洗浄した表面を除染し、5分間インキュベートします。ワイプは、オートクレーブ可能なバイオハザードバッグまたはレセプタクルに入れて廃棄してください。
注意: 漂白剤は非常に腐食性と反応性の化学物質です。したがって、使用前に適切な安全対策を講じる必要があります。 - 超高純度のDNase/RNaseフリー水で湿らせた滅菌ワイプを使用して、解剖学的サンプリング場所からできるだけ多くの残留漂白剤を取り除きます。ワイプは、オートクレーブ可能なバイオハザードバッグまたはレセプタクルに入れて廃棄してください。
- 洗浄したすべての解剖学的サンプリング場所をUV放射(波長:254 nm)に30分間さらした後、室温で完全に乾燥させます。骨粉の生成を容易にするだけでなく、サンプルのさらなる劣化(カビなど)を防ぐために、サンプリングを続行したり保管に戻したりする前に、解剖学的サンプリング場所が完全に乾いていることを確認してください。
注意: 紫外線にさらされると、目に害を及ぼす可能性があります。 - すぐにサンプリングに移動するか、乾燥した温度制御された(25°C)無菌環境に骨格要素を保管してください。
3.骨粉の生成
注:以下のプロトコルは、Dabney et al. 2019プロトコル26に従ったDNA抽出での使用を目的としています。
- のサンプリング pars petrosa
注:このプロトコルは、Pinhasi et al. 2019に記載されている手順から適応されています。4 使いやすさのためにここに提示されています。このプロトコルは、サンプリングのための現在の最も破壊的な方法を表すものではありません。 pars petrosa.そのため、Sirak et al. 2017で説明されているプロトコルを使用することをお勧めします。13 またはオルファノウら202014 形態学的保存が最も重要であるサンプル用。- UVライトを備えたPCRフードまたはバイオセーフティキャビネット(波長:254 nm)の下にある専用のクリーンルームで、エアフローをオフにしてすべてのサンプリングを実行します。ベンチトップ全体に滅菌アルミホイルを広げて、浮遊骨粉/破片をキャッチします。
- ホイルを処分する前に、すべての骨片とできるだけ多くの粉末が(本国送還のために)回収されていることを確認してください。各サンプリング間でフォイルを交換します。使用済みのホイルは、オートクレーブ可能なバイオハザードバッグ/レセプタクルに入れて廃棄してください。
- 滅菌されたクランプまたは万力を使用して、乾燥した除染された要素を固定します。
- 過熱を避けるために、0.6 mmの刃(材料表を参照)を備えた標準の宝石商のこぎりを使用して、上溝のペトロサス(図1を参照)に沿ってパースペトロサを中速で半分にカットします(手順3.1.6の下の注を参照)。
注意: パースペトロサ は非常に密度が高いため、切断が難しい場合があります。怪我をしないように、エレメントをしっかりと固定しておくように注意してくださいamp。壊れた鋸刃は、適切な鋭利物のレセプタクルに廃棄してください。 - クランプから錐体部分を取り外します。緩い/余分な材料を回収して保存します。
- 計量紙を滅菌計量ボートに入れます
- 錐体部分を計量紙の上にかざし、切断面を計量トレイに向かって傾けます。小さなゲージビット(材料の表を参照)を備えた歯科用ドリルを使用して、顔面管と乳様突起前庭の間の密な皮質骨(周囲の材料よりも光沢があるように見える、図1を参照)にドリルで穴を開け、中速、中トルクに設定して骨粉を生成します。
注意: 穴あけ/切断は、骨の過熱やDNAの破壊/損傷の可能性を避けるために、低速から中速で短いバーストで実行する必要があります。逸話的に、石油の密な部分が過熱し始めると、ベーコンを調理すると説明される臭いが観察されることがあります。すぐに穴あけ/鋸引きを中止し、再開する前に十分に冷えるまで骨を休ませます。 - 少なくとも50 mgの精度の密閉天びんを使用して測定された、約100〜0.01 mgの粉末が計量紙に収集されるまで、穴あけを繰り返します( 材料の表を参照)。
注:可能であれば、それぞれ50 mgの複製DNA抽出を2回行うために、100 mgの骨粉末を収集することをお勧めします。しかしながら、これは、解剖学的サンプリング位置自体の制限(例えば、遠位指節骨、歯髄室)または形態学的保存の必要性のいずれかに基づいて常に可能であるとは限らない。セメント質などの他の場所では、50mg未満の材料が利用できる場合があります。しかし、セメント質、歯髄室、および遠位指骨はすべて、抽出プロセスからの骨粉末の初期投入量が少ないにもかかわらず、有意な内因性DNA11、27、28を生成することが示されています。 - 計量紙から2 mLのラベル付きローバインドセーフロックチューブに粉末を移し、抽出または保管します。サンプルは-20°Cで無期限に保管してください。
- 残りの骨/余分な粉末は、返却/本国送還が完了するまで、乾燥した温度制御された(25°C)無菌環境に保管してください。
- すべての廃棄物は、オートクレーブ可能なバイオハザードバッグまたはレセプタクルに入れて処分してください。各サンプリングの間に、漂白剤/DNA除染溶液/エタノールおよびUV(波長:254 nm)曝露(該当する場合)を使用して、すべての再利用可能な機器(クランプ、ドリルビット、ドリル、のこぎりなど)を滅菌/除染します。
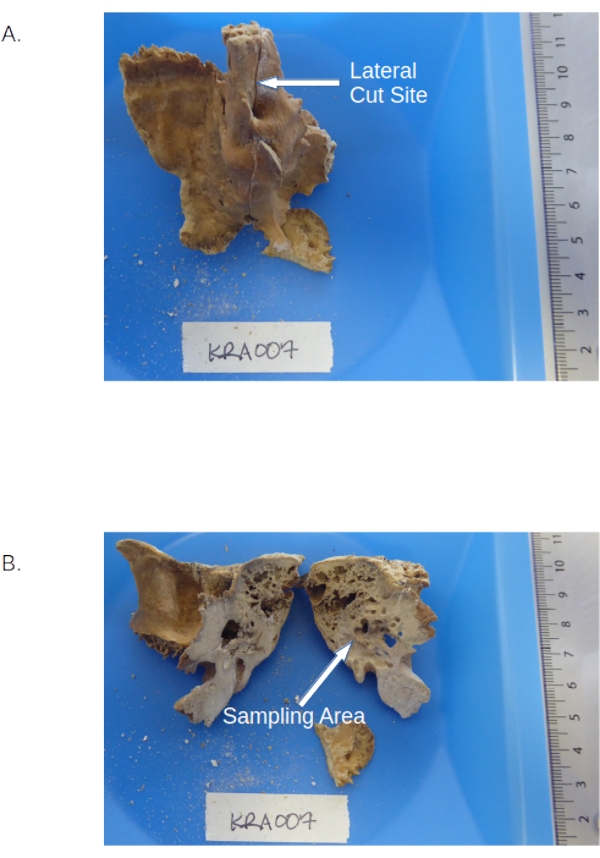
図1: ペトロサを含む側頭骨。 (A)錐体ピラミッドと 溝ペトロサ の位置を示すサンプルプレカット。(B)掘削する密集した領域を強調するペトラス部分の切断後。 この図の拡大版を表示するには、ここをクリックしてください。
- 永久大臼歯のサンプリング
注意: 永久大臼歯のサンプリングについては、事前に選択してください in situ 根が融合し、理想的には虫歯、エナメル質のひび割れ、または最良の結果を得るための過度の摩耗がない大臼歯。歯石のサンプリングをすべて取り除き、口腔微生物叢の将来の分析のために-20°Cで保管します(手順はここでは説明しません)。- セメント質のサンプリング
- UVライトを備えたPCRフードまたはバイオセーフティキャビネット(波長:254 nm)の下にある専用のクリーンルームで、エアフローをオフにしてすべてのサンプリングを実行します。ベンチトップ全体に滅菌アルミホイルを広げて、浮遊骨粉/破片をキャッチします。
- ホイルを処分する前に、すべての骨片とできるだけ多くの粉末が(本国送還のために)回収されていることを確認してください。各サンプリング間でフォイルを交換します。使用済みのホイルは、オートクレーブ可能なバイオハザードバッグ/レセプタクルに入れて廃棄してください。
- 計量紙のシートを滅菌計量トレイに入れます。
- 除染された大臼歯をエナメル質で保持/固定し、幅を下にして、モンキーレンチなどのハンドヘルドクランプを使用して計量トレイの上に置きます(材料の表を参照)。
- 歯科用ドリルにダイヤモンドエッジの円形カッティングホイールを装備します。ドリルを中速/トルク設定に設定した状態で、ビットの端を約-20°の角度で根元に軽く触れます。
- トレイに下向きにこすり落とし、根元(セメント質)から黄色の最も外側の材料を除去/収集します。象牙質の軽い(白い)材料が見えるようになったら、収集を停止します。
注意: 粉末がエアロゾル化され、トレイを完全に失うことでサンプルを無駄にする可能性があるため、収集トレイに対して切断ビットの回転方向を一致させることが重要です。セメント質は特にDNAが豊富です。ただし、材料の典型的な収量は、他の解剖学的サンプリング場所(~7-20 mg)よりもはるかに小さい11,27,28。 - 少なくとも0.01 mgの精度の同封天びんを使用して、計量紙に収集された粉末の質量を記録します( 材料の表を参照)。
- 計量紙から2 mLの低バインドで安全なロックチューブに粉末を移して抽出します。-20°Cで無期限に保管してください。
- パルプチャンバーのサンプリング
- セメント質が収集されたら(必要に応じて)、宝石商のこぎりを使用してセメントとエナメルの接合部に沿って大臼歯を切断し、リューズを取り外します( 図2を参照)。
- 新しい計量トレイに新しい計量用紙を置きます。
- クラウンセクションをハンドヘルドクランプまたは万力で計量トレイの上に固定します。切断面を下に傾けて保持し、クラウン部分内の歯髄チャンバーの端に沿って小さなゲージの穴あけビット( 材料の表を参照)を備えた歯科用ドリルを使用して、最初のパスとして材料をドリル/スクレイピングします( 図2を参照)。
注:歯髄室の内部の最初のパスのみが収集され、パルプ材料(5〜15 mgの典型的な収量)としてラベル付けされ、歯の奥深くにあるものはすべて象牙質と見なされます。 - 下部を下に向けて歯を回し、クランプをハンマーで叩き、遊離した粉末を計量紙に集めます。
- 少なくとも0.01mgの精度の同封天秤を使用して、計量紙に収集された粉末の重量を記録します(材料の表を参照)。
- 粉末を計量紙から2 mLの低バインドセーフロックチューブに移して抽出します。-20°Cで無期限に保管してください。
- 象牙質のサンプリング
- 新しい計量トレイに新しい計量用紙を置きます。
- クラウンセクションを計量トレイの上に保持し(ステップ3.2.2.3に従って)、ドリルアウトして、さらに象牙質サンプリングのために同じ方法で、0.01 mg( 材料の表を参照)に正確な同封天びんを使用して測定された50〜100 mgの象牙質を収集します( 図2を参照)。
- 骨粉末を計量紙から2 mLのローバインドセーフロックチューブに移して抽出します。-20°Cで無期限に保管してください。
- 残りの歯片/余分な粉末は、返却/本国送還が完了するまで、乾燥した温度制御された(25°C)無菌環境に保管してください。
- すべての廃棄物は、オートクレーブ可能なバイオハザードバッグまたはレセプタクルに入れて処分してください。各サンプリングの間に、必要に応じて漂白剤/DNA除染溶液/エタノールおよびUV(波長:254 nm)曝露を使用して、すべての再利用可能な機器(クランプ、ドリルビット、ドリル、のこぎりなど)を滅菌/除染します。
- セメント質のサンプリング
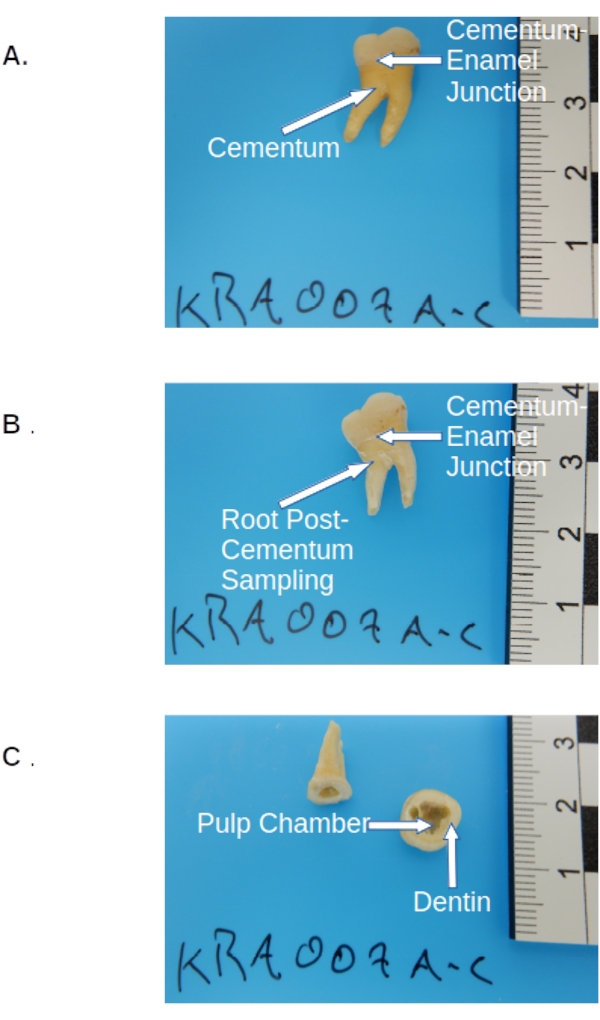
図2:永久大臼歯の事前 サンプリング。 (A)サンプリング前に前処理された大臼歯、クラウン、セメント質(根の黄色がかった層)、およびセメント-エナメル接合部の切断部位を示します。(B)同じモルポストセメントムコレクションで、セメント-エナメル接合部における切断部位を示す。(C)歯冠内の歯髄室と象牙質の解剖学的サンプリング位置を示す臼歯の切断後およびサンプリング。 この図の拡大版を表示するには、ここをクリックしてください。
- 胸椎のサンプリング
- 椎体のサンプリング
- UVライトを備えたPCRフードまたはバイオセーフティキャビネット(波長:254 nm)の下にある専用のクリーンルームで、エアフローをオフにしてすべてのサンプリングを実行します。ベンチトップ全体に滅菌アルミホイルを広げて、浮遊骨粉/破片をキャッチします。
- ホイルを処分する前に、すべての骨片とできるだけ多くの粉末が(本国送還のために)回収されていることを確認してください。各サンプリング間でフォイルを交換します。使用済みのホイルは、オートクレーブ可能なバイオハザードバッグ/レセプタクルに入れて廃棄してください。
- 計量用紙の小さなシートを標準の計量トレイに入れます。
- 椎体を外側にして、クランプまたはハンドバイスで椎骨を固定します。
- 椎体を下に傾けた状態で、椎骨を計量トレイの上にかざします。低速高トルクに設定された小さなゲージドリルビット( 材料表を参照)を備えた歯科用ドリルを使用して、椎体の海綿状内部組織を囲む皮質骨の最外縁(下および上)に沿ってドリルします( 図3を参照)。
- 0.01 mgの精度の同封天びんを使用して測定した50〜100 mgの材料が収集されるまで、標準の重み付けトレイで皮質層に対してビットをこすります( 材料の表を参照)。
- 骨粉末を計量紙から2 mLのローバインドで安全なロックチューブに移して抽出します。-20°Cで無期限に保管してください。
- 上椎弓のサンプリング
注: この手順はオプションです。上椎弓の皮質骨の最外層を、小さなゲージのドリルビット( 材料表を参照)を備えた歯科用ドリルを使用して、表面に沿って削り取って除去します19。皮質骨の層は一般的に非常に薄く、このプロセスによって完全に枯渇する可能性があるため、これは椎体からのサンプリングには推奨されません(セクション2の注を参照)。- 計量用紙の小さなシートを標準の計量トレイに入れます。
- 椎骨をハンドクランプ/万力で固定し、椎骨突起を外側に、上面を下に向けて固定します。
- 椎骨を上方面下に保持しながら、計量トレイ上で、低速および高トルクに設定された小さなゲージビット(材料表を参照)を備えた歯科用ドリルを使用して、棘突起とラメラ(図3を参照)の融合によって形成されたV字型のノッチの中央にドリルアップします。
- 抵抗が著しく低下した場合は、掘削を中止してください。穴あけ位置を少し変更し、0.01 mgの精度の密閉天びんを使用して測定した50〜100 mgの骨粉が収集されるまで繰り返します( 材料の表を参照)。
- 骨粉末を計量紙から2 mLの低結合チューブに移して抽出します。-20°Cで無期限に保管してください。
- 残りの骨/余分な粉末は、返却/本国送還されるまで、乾燥した温度制御された(25°C)無菌環境に保管してください。
- すべての廃棄物は、オートクレーブ可能なバイオハザードバッグまたはレセプタクルに入れて処分してください。各サンプリングの間に、漂白剤/DNA除染溶液/エタノールおよびUV(波長:254 nm)曝露(該当する場合)を使用して、すべての再利用可能な機器(クランプ、ドリルビット、ドリル、のこぎりなど)を滅菌/除染します。
- 椎体のサンプリング

図3:椎体と上椎弓皮質骨の胸椎の解剖学的サンプリング位置。 この図の拡大版を表示するには、ここをクリックしてください。
- 遠位指節骨のサンプリング
注: この手順はオプションです。シャフトおよび/または頂端房の皮質骨の最外層を、表面に沿って削り取ることによって小さなゲージの穴あけビットを備えた歯科用ドリルを使用して除去し、廃棄する19。これは、皮質骨が過度に薄いサンプルや若年遺体では不可能な場合があります(セクション2の注を参照)。- UVライトを備えたPCRフードまたはバイオセーフティキャビネット(UV波長:254 nm)の下で、エアフローをオフにして、専用のクリーンルームですべてのサンプリングを実行します。ベンチトップ全体に滅菌アルミホイルを広げて、浮遊骨粉/破片をキャッチします。
- ホイルを処分する前に、すべての骨片とできるだけ多くの粉末が(本国送還のために)回収されていることを確認してください。各サンプリング間でフォイルを交換します。使用済みのホイルは、オートクレーブ可能なバイオハザードバッグ/レセプタクルに入れて廃棄してください。
- 計量用紙の小さなシートを標準の計量トレイに入れます。
- サンプルをハンドヘルドクランプ/バイスで上側に固定します。
- サンプルを計量トレイにかざし、小さなゲージドリルビットを備えた歯科用ドリルを使用して最も外側の密な層( 図4を参照)をドリルで開けて、頂端の房とシャフトの下側から皮質骨から骨粉を収集します( 材料表を参照)。
- 抵抗が著しく減少した場合は、より軽くて海綿状の材料を意味するため、穴あけを中止してください。このプロセスを繰り返し、0.01 mgの精度の密閉天びんを使用して測定した、少なくとも50〜100 mgの骨粉が収集されるまで、最初の穴あけから外側に放射します( 材料の表を参照)。
- 骨粉末を計量紙から2 mLのローバインドセーフロックチューブに移して抽出します。-20°Cで無期限に保管してください。
- 残りの骨/余分な粉末は、返却/本国送還まで、乾燥した温度制御された(25°C)無菌環境に保管してください。.
- すべての廃棄物は、オートクレーブ可能なバイオハザードバッグまたはレセプタクルに入れて処分してください。各サンプリングの間に、必要に応じて漂白剤/DNA除染溶液/エタノールおよびUV曝露を使用して、すべての再利用可能な機器(クランプ、ドリルビット、ドリル、のこぎりなど)を滅菌/除染します。
注:より小さなサンプル(例:.、若年サンプル)の場合、サンプルに利用できる皮質骨の推奨50〜100mgよりもかなり少ない場合があります。しかし、少量であっても、この解剖学的サンプリング場所はDNA11が特に豊富であることが示されています。

図4:先端房のシャフトと下側に沿った密な皮質骨の位置を示す遠位指節骨。 この図の拡大版を表示するには、ここをクリックしてください。
- 距骨のサンプリング
- UVライトを備えたPCRフードまたはバイオセーフティキャビネット(波長:254 nm)の下にある専用のクリーンルームで、エアフローをオフにしてすべてのサンプリングを実行します。ベンチトップ全体に滅菌アルミホイルを広げて、浮遊骨粉/破片をキャッチします。
- ホイルを処分する前に、すべての骨片とできるだけ多くの粉末が(本国送還のために)回収されていることを確認してください。各サンプリング間でフォイルを交換します。使用済みのホイルは、オートクレーブ可能なバイオハザードバッグ/レセプタクルに入れて廃棄してください。
- 計量用紙の小さなシートを標準の計量トレイに入れます。
- サンプルをハンドヘルドクランプ/万力で固定し、ドームを上向きにします。
- 距骨、ドームを上向きに保持し、内側の表面をコレクターに向かって、計量トレイの上に置きます。低速および高トルクに設定された低ゲージビット(材料表を参照)を備えた歯科用ドリルを使用して、距骨の頸部から~1 mmの深さまで皮質骨をこすり落とします(図5を参照)。
- 穴あけ位置を少し変更し、0.01 mgの精度の密閉天びんを使用して測定した約50〜100 mgの骨粉が収集されるまで繰り返します( 材料の表を参照)。
- 骨粉末を計量紙から2 mLの低結合チューブに移して抽出します。-20°Cで無期限に保管してください。
- 残りの骨/余分な粉末は、返却/本国送還が完了するまで、乾燥した温度制御された(25°C)無菌環境に保管してください。.
- すべての廃棄物は、オートクレーブ可能なバイオハザードバッグまたはレセプタクルに入れて処分してください。各サンプリングの間に、漂白剤/DNA除染溶液/エタノールおよびUV(波長:254 nm)曝露(該当する場合)を使用して、すべての再利用可能な機器(クランプ、ドリルビット、ドリル、のこぎりなど)を滅菌/除染します。
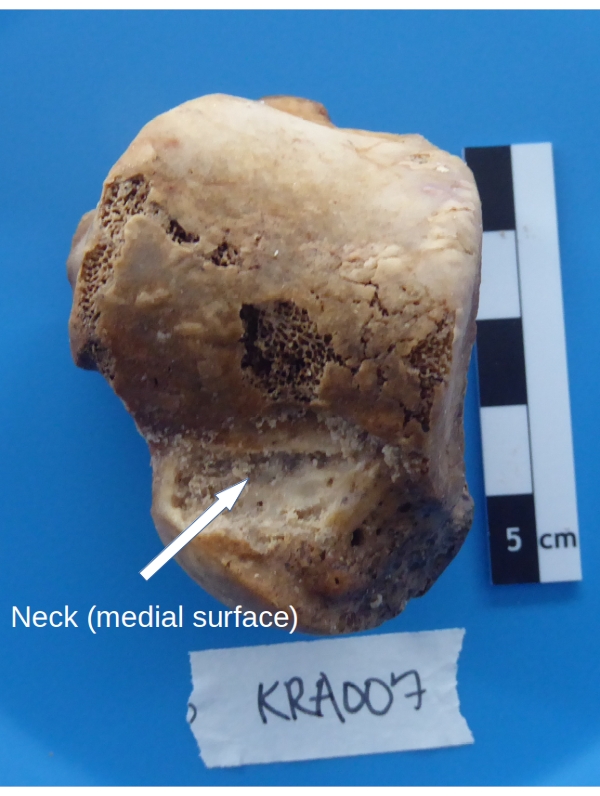
図5:皮質骨回復のための距骨のサンプリング領域。 この図の拡大版を表示するには、ここをクリックしてください。
注:距骨には皮質骨(薄い外層)がほとんどありません。材料は表面からだけでなく、海綿骨の下にある密な層からも収集する必要があります。
結果
別の研究11では、石灰化した組織からの短い断片に最適化された標準的なDNA抽出プロトコルを使用して、11人の各解剖学的サンプリング場所から生成された骨粉末からDNAを抽出しました2。次に、一本鎖ライブラリを28個生成し、HiSeq 4000(75 bpペアエンド)でサンプルあたり~20,000,000リードの深さまでシーケンスしました。次いで、得られた配列データを、EAGERパイプライン29を用いて内因性ヒトDNA含量について評価した(BWA設定:シード長32、0.1ミスマッチペナルティ、マッピング品質フィルタ37)。すべての代表的な結果は、一貫性を保つためにParker et al. 202011と同じ指標を用いて報告されている。パルスペトロサの粉末部分からのライブラリは、調査された他の23の解剖学的サンプリング場所のどれよりも平均して高い内因性DNAを生成しました(図6A-B)。このプロトコルで提示された7つの追加の解剖学的サンプリング位置(セメント質、歯髄室の最初の通過、永久大臼歯の象牙質、椎体からの皮質骨および胸椎の上椎弓、遠位指節骨の頂端房からの皮質骨、および距骨の頸部からの皮質骨)は、次に高い収量を生み出しました(これらの解剖学的サンプリング位置の間に統計的有意性はありません。図6A-B;補足ファイル1:内因性DNAPreCap)。これらの代替場所はすべて、ミトコンドリア分析や一塩基多型(SNP)分析などの標準的な集団遺伝学分析に適したDNA収量を一貫して生成しました。すべての解剖学的サンプリング場所に由来するライブラリの重複率は低かった(クラスター係数<平均1.2、一意のマッピングリードに対するすべてのマッピングリードの比率として計算、表2;補足ファイル1:ClusterFactor)、スクリーニングされたすべてのライブラリが非常に複雑であることを示しています。同様に、平均外因性ヒトDNA汚染推定値は低く、上椎弓を除くすべての解剖学的サンプリング場所で平均<2%(男性のX染色体汚染、n = 7、ANGSD30パイプラインによって報告された)でした(平均推定汚染:2.11%、1つのサンプルは外れ値として削除されました。KRA005:19.52%、表2を参照。補足ファイル1:X汚染)。平均フラグメント長(30 bp<すべての読み取り値を除去するためにフィルタリングした後)は、歯髄室および象牙質から収集された材料で最も低く、他の解剖学的サンプリング場所間で有意な変動はありませんでした(平均中央値62.87と比較して、それぞれ55.14 bpおよび60.22 bp、ペアワイズp値は0.019<、表2;補足ファイル 1: AvgFragLength)。さらに、歯と胸椎にはそれぞれ、高い内因性DNA回収が観察された複数の解剖学的サンプリング場所が含まれているため、ペトロサの代替品として特に適しています。
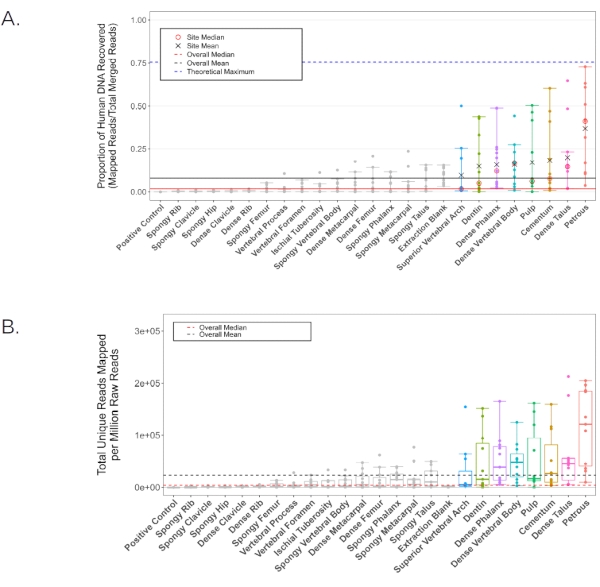
図6:スクリーニングされたすべてのサンプルのヒトDNA含有量。 黒い線は全体の平均を表し、赤い線は中央値を表します(実線:ヒトDNA比率、破線:生成された100万リードあたりのマッピングされたヒトリード)。平均ヒトDNA比率が全体平均(8.16%)よりも高い個々の解剖学的サンプリング場所は、すべての分析で色付けされています。(A)hg19リファレンスゲノムにマッピングするリードの割合。青い破線は、パイプラインのマッピングパラメータ(シミュレートされた損傷を伴うhg19参照ゲノムからの5,000,000リードのランダム分布をシミュレートするためにGargammel31 を使用して生成された)が与えられた理論上の最大値を表します。個々の平均(黒いX)と中央値(赤い円)は、全体の平均よりも平均ヒトDNA比率が高いサンプルについて報告されています。信頼区間は、統計的外れ値を除いた上限と下限を示します。(B)シーケンシング努力の100万リードあたりのhg19リファレンスゲノムにマッピングされた一意のリードの数(75 bpペアエンド)。信頼区間は、統計的外れ値を除いた上限と下限を示します。この図は、Parker, C. et al. 202011から改作されています。 この図の拡大版を表示するには、ここをクリックしてください。
表2:すべての解剖学的サンプリング場所の平均重複レベル(マッピングリード/ユニークリード)、フラグメント長の平均および中央値、およびX染色体汚染推定値。 誤差は平均の標準誤差として報告されます。この表は、Parker, C. et al. 202011から改作されたものである。
| サンプリング場所 | 平均重複係数 (# マップされた読み取り /# 一意のマップされた読み取り) | 平均フラグメント長 (bp 単位) | X染色体汚染の平均推定割合 |
| ペトラピラミッド | 1.188 ± 0.006 | 65.40 ± 1.36 | 0.000 ± 0.003 |
| セメント | 1.197 ± 0.028 | 67.28 ± 1.76 | ±0.011± ±0.003 |
| 象牙質 | 1.188 ± 0.061 | 60.22±2.37 | 0.002 ± 0.007 |
| パルプ | 1.179 ± 0.024 | 55.14 ± 2.90 | ±0.013± 0.006 |
| 遠位指節骨 | 1.191 ± 0.049 | 65.95±1.08 | 0.013± 0.005 |
| 椎体 | 1.194 ± 0.037 | 66.14 ± 1.03 | 0.008± 0.003 |
| 上椎弓 | 1.19 ± 0.017 | 63.02 ± 1.23 | 0.021 ± 0.009* |
| 距骨 | 1.198 ± 0.010 | 68.20±1.24 | ±0.011± ±0.003 |
| *サンプルKRA005は0.1952で外れ値として削除 |
コードの可用性
この原稿の分析に使用されたすべての分析プログラムとRモジュールは、それぞれの著者から無料で入手できます。すべてのカスタム R コードは、要求に応じて利用できます。
データの可用性
代表的な結果の計算に使用されるすべての生データは、欧州ヌクレオチドアーカイブENAデータリポジトリ(アクセッション番号PRJ-EB36983)またはParker、C.らの補足資料11で自由に入手できます。
補足ファイル 1.このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
古代人集団遺伝学における現在の慣行は、可能な限りパースペトロサ(ステップ2.1)から優先的にサンプリングすることです。しかし、パース・ペトロサは、無数の骨格評価(例えば、集団歴32、死亡時の胎児年齢の推定33、性決定34)で高く評価されているため、入手が困難なサンプルになる可能性があり、歴史的に、DNA分析のためのパース・ペトロサのサンプリングは非常に破壊的である可能性があります3,4(ここに提示されたプロトコルを含む、 新しい、低侵襲プロトコル13,14は、この懸念を軽減するために現在広く採用されています)。これは、ごく最近まで、骨格全体のヒトDNA回収の大規模で体系的な研究が試みられていなかったという事実によってさらに悪化し11、石油ピラミッドが利用できないときに適切なサンプリング戦略を見つけることを困難にしています。
ここで紹介するプロトコルは、pars petrosaを含む考古学的/法医学的骨格遺跡からのDNAサンプリングのための一連の最適化された手順と、4つの追加の骨格要素にわたる7つの代替解剖学的サンプリング場所を提供することにより、その課題を軽減するのに役立ちます。含まれている重要なステップはすべて、非効率的なサンプリング(ステップ2.1.6および3.2.1.3)または穴あけ/切断中のサンプルの過熱(ステップ3.1.6)によるDNAの損失/損傷の可能性を最小限に抑えることを目的としています。さらに、プロトコル全体を通して、高度に劣化したサンプルで最高のパフォーマンスを確保するために、前処理ステップを変更/省略する必要がある場合があることが指摘されています。また、ここで提示された選択された要素の中でも、いくつかの可能な代替サンプリング技術(特にpars petrosa13,14)と、ここで提示された十分に活用されていない解剖学的サンプリング場所(すなわち、距骨:ステップ2.5および椎骨:ステップ2.3)をさらに最適化するための十分な余地が残っていることにも留意する必要があります。
これらのプロトコルは、内因性のヒトDNA分析の目的で、高品質の(良好な形態学的保存)古代の幼体と成人の遺体を使用して設計およびテストされていることを覚えておくことも重要です。提示された結果は、追加の状況でのこれらのプロトコルの使用に関するさらなる調査が依然として必要であるため、より高度に分解された材料、他の保存状況、乳児の遺体、人間以外の遺体、または病原体または微生物叢の研究には及ばない可能性があります。さらに、ここに提示された代替骨格要素(歯、椎骨、遠位指節骨、およびタリ)は、混合された遺体の中で単一の個人に割り当てるのが難しい場合があり、単一の起源を確保するために複数の要素からのサンプリングが必要です。これらの制限にもかかわらず、これらのプロトコルを広く利用できるようにすることで、ヒト遺体に関する将来の幅広いaDNA/法医学研究で使用するための一般化および定量的に最適化されたフレームワークを提供することにより、サンプルの選択と処理を取り巻く異質性の一部を軽減するのに役立ちます。
開示事項
著者には、報告する利益相反はありません。
謝辞
著者らは、これらのプロトコルの開発と実施に協力してくれたマックスプランク人類史科学研究所の研究室スタッフに感謝したいと思います。この研究は、グイド・ブラント博士、エリザベス・ネルソン博士、アンティエ・ウィセゴット、フランツィスカ・アロンのインプットと努力なしには不可能でした。この研究は、マックスプランク協会、欧州連合のホライズン2020研究およびイノベーションプログラムの下で、助成金契約No.771234-PALEoRIDER(WH、ABR)および開始助成金番号805268 CoDisEASe(KIBへ)の下で、欧州研究会議(ERC)によって資金提供されました。
資料
| Name | Company | Catalog Number | Comments |
| #16 Dental Drill Bit | NTI | H1-016-HP | example drilling bit |
| 0.6 mm scroll saw blade | Fisher Scientific | 50-949-097 | blade for Jewellers Saw |
| 22mm diamond cutting wheel | Kahla | SKU 806 104 358 514 220 | Dremel cutting attachment |
| Commercial Bleach | Fisher Scientific | NC1818018 | |
| Control Company Ultra-Clean Supreme Aluminum Foil | Fisher Scientific | 15-078-29X | |
| DNA LoBind Tubes (2 mL) | Eppendorf | 22431048 | |
| Dremel 225-01 Flex Shaft Attachment | Dremel | 225-01 | Dremel flexible extension |
| Dremel 4300 Rotary Tool | Dremel | 4300 | Example drill |
| Dremel collet and nut kit | Dremel | 4485 | Adapters for various Dremel tool attachments/bits |
| Eagle 33 Gallon Red Biohazard Waste Bag | Fisher Scientific | 17-988-501 | |
| Eppendorf DNA LoBind 2 mL microcentrifuge tube | Fisher Scientific | 13-698-792 | |
| Ethanol (Molecular Biology Grade) | Millipore Sigma | 1.08543 | |
| FDA approved level 2 Surgical Mask | Fisher Scientific | 50-206-0397 | PPE |
| Fisherbrand Comfort Nitrile Gloves | Fisher Scientific | 19-041-171X | PPE |
| Fisherbrand Safety Glasses | Fisher Scientific | 19-130-208X | PPE |
| Granger Stationary Vise | Fisher Scientific | NC1336173 | benchtop vise |
| Invitrogen UltraPure DNase/Rnase free distilled water | Fisher Scientific | 10-977-023 | |
| Jewellers Saw | Fisher Scientific | 50-949-231 | |
| Kimwipes | Sigma-Aldritch | Z188956 | |
| Labconco Purifier Logic Biosafety cabinet | Fisher Scientific | 30-368-1101 | |
| LookOut DNA Erase | Millipore Sigma | L9042-1L | |
| Medium weighing boat | Heathrow Scientific | HS120223 | |
| MSC 10pc plier/clamp set | Fisher Scientific | 50-129-5352 | Miscellaneous clamps/vise grips for securely holding samples while drilling/cutting |
| Sartorius Quintix Semi-Micro Balance | Fisher Scientific | 14-560-019 | enclosed balance |
| Tyvek coveralls with hood | Fisher Scientific | 01-361-7X | PPE |
| Weigh paper | Heathrow Scientific | HS120116 |
参考文献
- Adler, C. J., Haak, W., Donlon, D., Cooper, A. Survival and recovery of DNA from ancient teeth and bones. Journal of Archaeological Science. 38 (5), 956-964 (2011).
- Dabney, J., Meyer, M. Extraction of highly degraded DNA from ancient bones and teeth. Ancient DNA: Methods and Protocols. , 25-29 (2019).
- Palsdottir, A. H., Bläuer, A., Rannamäe, E., Boessenkool, S., Hallsson, J. Not a limitless resource: ethics and guidelines for destructive sampling of archaeofaunal remains. Royal Society Open Science. 6 (10), 191059 (2019).
- Pinhasi, R., Fernandes, D. M., Sirak, K., Cheronet, O. Isolating the human cochlea to generate bone powder for ancient DNA analysis. Nature Protocols. 14 (4), 1194-1205 (2019).
- Latham, K. E., Miller, J. J. DNA recovery and analysis from skeletal material in modern forensic contexts. Forensic Sciences Research. 4 (1), 51-59 (2019).
- Mundorff, A. Z., Bartelink, E. J., Mar-Cash, E. DNA preservation in skeletal elements from the World Trade Center disaster: Recommendations for mass fatality management. Journal of Forensic Sciences. 54 (4), 739-745 (2009).
- Gamba, C., et al. Genome flux and stasis in a five millennium transect of European prehistory. Nature Communications. 5 (1), 1-9 (2014).
- Alberti, F., et al. Optimized DNA sampling of ancient bones using Computed Tomography scans. Molecular Ecology Resources. 18 (6), 1196-1208 (2018).
- Hansen, H. B., et al. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE. 12 (1), 0170940 (2017).
- Sirak, K., et al. Human auditory ossicles as an alternative optimal source of ancient DNA. Genome Research. 30 (3), 427-436 (2020).
- Parker, C., et al. A systematic investigation of human DNA preservation in medieval skeletons. Scientific Reports. 10 (1), 18225 (2020).
- Pinhasi, R., et al. Optimal ancient DNA yields from the inner ear part of the human petrous bone. PLoS ONE. 10 (6), 0129102 (2015).
- Sirak, K. A., et al. A minimally-invasive method for sampling human petrous bones from the cranial base for ancient DNA analysis. BioTechniques. 62 (6), 283-289 (2017).
- . Minimally-invasive sampling of pars petrosa (os temporale) for ancient DNA extraction. protocols.io Available from: https://www.protocols.io/view/minimally-invasive-sampling-of-pars-petrosa-os-tem-bqd8ms9w (2020)
- Damgaard, P. B., et al. Improving access to endogenous DNA in ancient bones and teeth. Scientific Reports. 5 (1), 1-12 (2015).
- Harney, &. #. 2. 0. 1. ;., et al. A minimally destructive protocol for DNA extraction from ancient teeth. Genome Research. 31 (3), 472-483 (2021).
- Cooper, A., Poinar, H. N. Ancient DNA: Do it right or not at all. Science. 289 (5482), 1139 (2000).
- Llamas, B., et al. From the field to the laboratory: Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era. STAR: Science & Technology of Archaeological Research. 3 (1), 1-14 (2017).
- Llamas, B., et al. From the field to the laboratory: Controlling DNA contamination in human ancient DNA research in the high-throughput sequencing era. STAR: Science & Technology of Archaeological Research. 3 (1), 1-14 (2017).
- Boessenkool, S., et al. Combining bleach and mild predigestion improves ancient DNA recovery from bones. Molecular Ecology Resources. 17 (4), 742-751 (2017).
- García-Garcerà, M., et al. Fragmentation of contaminant and endogenous DNA in ancient samples determined by shotgun sequencing; Prospects for human palaeogenomics. PLoS ONE. 6 (8), 24161 (2011).
- Malmström, H., et al. More on contamination: The use of asymmetric molecular behavior to identify authentic ancient human DNA. Molecular Biology and Evolution. 24 (4), 998-1004 (2007).
- Basler, N., et al. Reduction of the contaminant fraction of DNA obtained from an ancient giant panda bone. BMC Research Notes. 10, 754 (2017).
- Kemp, B. M., Smith, D. G. Use of bleach to eliminate contaminating DNA from the surface of bones and teeth. Forensic Science International. 154 (1), 53-61 (2005).
- Korlević, P., et al. Reducing microbial and human contamination in DNA extractions from ancient bones and teeth. BioTechniques. 59 (2), 87-93 (2015).
- Dabney, J., Meyer, M. Extraction of highly degraded DNA from ancient bones and teeth. Methods in Molecular Biology. 1963, 25-29 (2019).
- Hansen, H. B., et al. Comparing ancient DNA preservation in petrous bone and tooth cementum. PLoS ONE. 12 (1), 0170940 (2017).
- Gansauge, M. -. T., et al. Single-stranded DNA library preparation from highly degraded DNA using T4 DNA ligase. Nucleic Acids Research. 45 (10), 79 (2017).
- Peltzer, A., et al. EAGER: efficient ancient genome reconstruction. Genome Biology. 17 (1), 60 (2016).
- Korneliussen, T. S., Albrechtsen, A., Nielsen, R. ANGSD: analysis of next generation sequencing data. BMC Bioinformatics. 15, 356 (2014).
- Renaud, G., Hanghøj, K., Willerslev, E., Orlando, L. Gargammel: A sequence simulator for ancient DNA. Bioinformatics. 33 (4), 577-579 (2017).
- Ponce de León, M. S., et al. Human bony labyrinth is an indicator of population history and dispersal from Africa. Proceedings of the National Academy of Sciences of the United States of America. 115 (16), 4128-4133 (2018).
- Nagaoka, T., Kawakubo, Y. Using the petrous part of the temporal bone to estimate fetal age at death. Forensic Science International. 248, 188 (2015).
- Norén, A., Lynnerup, N., Czarnetzki, A., Graw, M. Lateral angle: A method for sexing using the petrous bone. American Journal of Physical Anthropology. 128 (2), 318-323 (2005).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved