このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
哺乳動物細胞溶解物におけるE3ユビキチンリガーゼによる基質ユビキチル化の評価
要約
我々は、哺乳動物細胞における特定の基質およびE3ユビキチンリガーゼのユビキチル化アッセイのための詳細なプロトコルを提供する。HEK293T細胞株をタンパク質過剰発現に使用し、ポリユビキチル化基質を免疫沈降によって細胞溶解物から精製し、SDS−PAGEで分解した。イムノブロッティングを用いて、この翻訳後修飾を可視化した。
要約
ユビキチル化は、真核細胞で起こる翻訳後修飾であり、細胞の生存、増殖、分化を含むいくつかの生物学的経路の調節に重要である。これは、E1(ユビキチン活性化酵素)、E2(ユビキチン結合酵素)、およびE3(ユビキチンリガーゼ酵素)からなる少なくとも3つの異なる酵素のカスケード反応を介して基質にユビキチンが共有結合することからなる可逆的プロセスである。E3複合体は、基質認識およびユビキチル化において重要な役割を果たしている。ここでは、選択された基質をコードするプラスミド、E3ユビキチンリガーゼ、およびタグ付きユビキチンの一過性同時トランスフェクションを使用して、哺乳動物細胞における基質ユビキチル化を評価するプロトコルが記載されている。溶解前に、トランスフェクトされた細胞をプロテアソーム阻害剤MG132(カルボベンゾキシ - ロイ - ロイ - ロイシナル)で処理して、基質プロテアソーム分解を回避する。さらに、細胞抽出物を小規模免疫沈降(IP)に提出し、ユビキチンタグに特異的な抗体を用いてウェスタンブロッティング(WB)によるその後の検出のためにポリユビキチル化基質を精製する。したがって、哺乳類細胞におけるユビキチル化アッセイのための一貫性のある複雑でないプロトコルは、特定の基質およびE3ユビキチンリガーゼのユビキチル化に対処する際に科学者を支援するために記載されている。
概要
翻訳後修飾(PTM)は、細胞の恒常性に不可欠なタンパク質調節に関する重要なメカニズムです。タンパク質ユビキチル化は、真核生物においていくつかの細胞転帰をもたらす異なるシグナルの集合体を作り出す動的で複雑な修飾である。ユビキチル化は、76個のアミノ酸を含むユビキチンタンパク質の基質への結合からなる可逆的なプロセスであり、3つの異なる反応1によって構成される酵素カスケードで起こる。第1のステップは、ユビキチンC末端とE1酵素の活性部位に存在するシステイン残基との間に高エネルギーチオエステル結合ユビキチンを形成するATP加水分解に依存するユビキチン活性化によって特徴付けられる。続いて、ユビキチンはE2酵素に転移し、ユビキチンとのチオエステル様複合体を形成する。その後、ユビキチンは、E2によって、またはより頻繁には、基質2,3を認識して相互作用するE3酵素によって基質に共有結合される。時折、E4酵素(ユビキチン鎖伸長因子)がマルチユビキチン鎖集合を促進するために必要である3。
ユビキチンは7つのリジン残基(K6、K11、K27、K29、K33、K48、およびK63)を有し、いくつかのエフェクタータンパク質によって認識されるであろう異なる三次元構造を生成するために別個の結合を生成するポリユビキチン鎖の形成を可能にする4,5。したがって、基質に導入されるポリユビキチン鎖の種類は、その細胞運命を決定するために不可欠である6、7、8。さらに、基板は、N-デグロンと呼ばれるN末端残基を介してユビキチン化することもできる。特定のE3ユビキチンリガーゼはN-degron認識に関与し、近くのリジン残基9のポリユビキチル化を可能にする。
今日では、40種類以上のSCF特異的基質が特徴付けられています。その中で、細胞の分化および発生ならびに細胞の生存および死を含むいくつかの生物学的経路の主要な調節因子が見出され得る10、11、12、13。したがって、各E3ユビキチンリガーゼの特異的基質の同定は、様々な生物学的事象の包括的なマップを設計するために不可欠である。真の基質の同定は生化学的に困難ですが、生化学ベースの方法の使用は、鎖特異性およびモノユビキチル化とポリユビキチル化の区別を評価するのに非常に適しています14。この研究は、基質UXT-V2(ユビキタス発現プレフォールディン様シャペロンアイソフォーム2)を過剰発現する哺乳動物細胞株HEK293TとE3ユビキチンリガーゼ複合体SCF(Fbxo7)を用いたユビキチル化アッセイのための完全なプロトコールを記載する。UXT-V2はNF-κBシグナル伝達に不可欠な補助因子であり、このタンパク質が細胞内でノックダウンされると、TNF-α誘導NF-κB活性化を阻害する11。したがって、ポリユビキチル化UXT-V2を検出するために、プロテアソーム複合体15の26Sサブユニットのタンパク質分解活性を遮断する能力を有することから、プロテアソーム阻害剤MG132が使用される。さらに、細胞抽出液を小規模IPに提出して基質を精製し、アガロース樹脂に固定化された特異的抗体を利用して、選択した抗体を用いたWBによるその後の検出を行う。このプロトコルは、細胞環境における基質ユビキチル化を検証するのに非常に有用であり、異なるタイプの哺乳動物細胞および他のE3ユビキチンリガーゼ複合体にも適合させることができる。しかし、両方のプロトコールが真の基質の同定に関して互いに補完し合うため、in vitroユビキチル化アッセイを通じて試験された基質を検証することも必要です。
プロトコル
注:哺乳動物細胞におけるユビキチル化アッセイプロトコルの概要を 図1に示します。
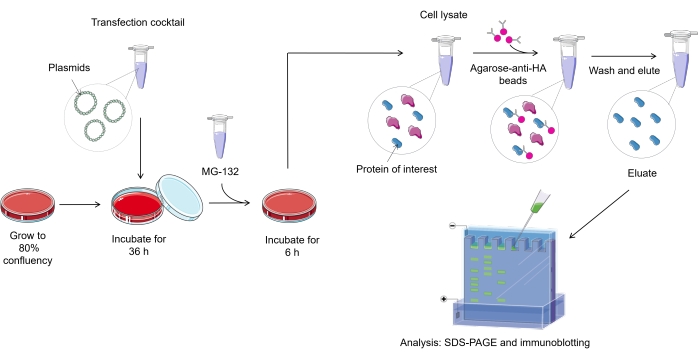
図1. ユビキチル化アッセイ手順の概要。 この図の拡大版を表示するには、ここをクリックしてください。
1. 細胞培養
- 100 mm TC 処理培養皿で HEK293T 細胞株を、増殖培地 (10% ウシ胎児血清 (FBS) およびペニシリン (100 単位)、ストレプトマイシン (100 μg) および L-グルタミン (0.292 mg/mL) を添加したダルベッコの改変イーグル培地 (DMEM) 高グルコース) 中で 80% ~ 90% のコンフルエントになるまで成長させます。5%CO2で加湿した細胞培養インキュベーター内で37°Cで培養物をインキュベートする。
- 細胞を継代するには、血清学的ピペットを用いて培養皿から培地を吸引する。滅菌した1xリン酸緩衝生理食塩水(PBS 1x)1mLで細胞を1回洗浄する。
- 1mLのトリプシン/EDTA(エチレンジアミン四酢酸)溶液を加えて細胞を剥離する。ディッシュを37°Cで5分間インキュベートする。血清学的ピペットを用いて細胞を2mLの増殖培地に再懸濁する。
- 細胞懸濁液を新鮮で清潔な15mLチューブに移し、500 x g で室温(RT)で5分間遠心分離する。上清を慎重に注いで取り除きます。細胞ペレットを3mLの成長培地に穏やかに再懸濁し、上下にピペッティングして均質な細胞懸濁液を得た。
- 細胞懸濁液1 mLを、9 mLの増殖培地を含む100 mm TC処理培養皿に移す。
注:細胞が良好な状態にある場合、HEK293Tの各培養皿は、コンフルエントが80%〜90%であり、継代後2日目に80%コンフルエントを有する3つの培養皿を生成することができる。
2. 細胞トランスフェクション
注:コンフルエントに達したものが80%未満の場合、細胞培養物をトランスフェクトすることは推奨されません。
- トランスフェクションの前に、細胞に汚染がなく、一過性トランスフェクションの適切なコンフルエントにあるかどうかを認証します。
- トランスフェクションサンプルごとに、DNA-ポリエチレンイミン(pH 7.2でPEI 1 μg/μL)複合体を次のように調製します。
- 各プラスミド3 μgを100 μLのopti-MEM I還元血清培地に無添加で希釈し、溶液を上下にピペッティングして穏やかに混合します。
注:ここでは、6xHis-myc-ユビキチンの有無にかかわらず、空のベクター(pcDNA3)またはFLAG-Fbxo7構築物およびUXT-V2-HAの各プラスミドを4 μgで細胞にトランスフェクトしました。全DNA含量は12μgであった。 - RTでPEIを解凍し、DNA1μgあたり3μLの割合のPEIに従って溶液に添加する。溶液を上下にピペッティングして均質化する。その後、RTで15分間インキュベートして、DNA-PEI複合体の形成を可能にします。
注:DNA量あたりのPEI体積の最適な割合は、選択した細胞株によって異なります。
- 各プラスミド3 μgを100 μLのopti-MEM I還元血清培地に無添加で希釈し、溶液を上下にピペッティングして穏やかに混合します。
- 細胞培養物を入れた各ディッシュにDNA-PEI複合体の全量を加え、プレートを前後に揺らして穏やかに混ぜる。5%CO2で加湿した細胞培養インキュベーター内で37°Cで細胞をインキュベートする。
- 5時間後に増殖培地を交換すると、長期間のPEI曝露がHEK293T細胞に有毒である可能性があるためである。5%CO2 の加湿細胞培養インキュベーター内で37°Cで細胞を36時間インキュベートする。
3. 細胞溶解と免疫沈降
- インキュベーション期間の後および細胞溶解の6時間前に、トランスフェクトされた細胞を10μMのプロテアソーム阻害剤MG−132で処理する。もう一度、5%CO2で加湿した細胞培養インキュベーター内で細胞を37°Cでインキュベートする。
- 血清学的ピペットを使用して各培養皿から培地を吸引し、1mLの1x PBSで1回洗浄する。1mLのトリプシンを加えて細胞を剥離し、ディッシュを37°Cで5分間インキュベートした。細胞を1mLの増殖培地に再懸濁する。
- 細胞懸濁液を新鮮で清潔な15mLチューブに移し、RTで5分間500 x g で遠心分離します。
- 上清を慎重に注いで取り除きます。細胞ペレットを200 μLの氷冷NP-40溶解バッファー(50 mM Tris-HCl pH 7.2、225 mM KCl、および1% NP-40)に穏やかに再懸濁し、プロテアーゼおよびホスファターゼ阻害剤カクテル(10 mM NaFおよび1 mMNa3VO4)を添加し、溶液を清潔な1.5 mLマイクロチューブに移します。
- 細胞溶解物を氷上で30分間インキュベートする。インキュベーション後、細胞溶解物を16,900 x g で4°Cで20分間遠心分離する。
- 一方、アガロース抗HAビーズを氷冷NP-40溶解バッファーで平衡化する。各サンプルに15 μLのアガロース-抗HAビーズを使用してください。ビーズを200 μLのNP-40溶解バッファーで洗浄し、3,000 x g の微量遠心管で4°Cで1分間パルスします。 ピペットで、上清を非常に慎重に吸引して捨てます。このプロセスを 3 回繰り返します。その後、ビーズを氷上で平衡化してから使用してください。
- 細胞溶解物を遠心分離した後、上清を回収する。ブラッドフォード法16を用いて全溶解物中のタンパク質含量を定量する。
- 免疫沈降に供する各サンプルが等量のタンパク質を示すことを確認します。UXT-V2-HAがアガロース抗HAビーズに結合することを可能にする4°Cの回転インキュベーター内で穏やかに回転させながら、平衡化されたアガロース - 抗HAビーズと細胞溶解物の必要量を4時間インキュベートする。
- アガロース-抗HAビーズを3,000 x g の微量遠心管で4°Cで1分間パルスして回収します。 慎重に吸引し、上清を捨てる。ビーズを氷冷NP-40細胞溶解バッファーで3回、氷冷FLAG/HAバッファー(10 mM Hepes pH 7.9、15 mM MgCl2、225 mM KCl、および0.1% NP-40)で2回洗浄します。
- 最終洗浄後、ピペットを使用してすべての上清を慎重に除去し、FLAG/HAバッファーで希釈したHAペプチド(300μg/mL)でポリユビキチル化タンパク質を溶出します。アガロース抗HAビーズをHAペプチドと共にロッキングシェーカープラットフォーム中で4°Cで1時間インキュベートする。
- ビーズを3,000 x g で4°Cで2分間スピンダウンし、ポリユビキチン化タンパク質を含む上清を慎重にピペットでピペットで送ります。必要に応じて、溶出液を-20°Cの新鮮で清潔なマイクロチューブに保存します。
- 溶出液および細胞溶解物を10%SDS−PAGE(ドデシル硫酸ナトリウム−ポリアクリルアミドゲル電気泳動)17 で解決し、イムノブロッティングした。
- 本研究では、湿式移送WBを実施した。この種の転写のために、濾紙 - ゲル - 膜 - 濾紙からなる転写サンドイッチにゲルを置き、パッドでクッションし、支持グリッドで一緒に押す。このシステムは、移送バッファで満たされたタンク内およびステンレス鋼/白金線電極の間に垂直に配置します。転写は、湿式移送バッファー(グリシン192 mM、トリス塩基25 mM、0.025% SDS、20%メタノール)中で150 Vで90分間行われます。
注:細胞抽出物を定量したので(ステップ3.7)、各サンプルに対して同じ量のタンパク質でSDS-PAGEを実行します。また、溶出液を沈殿させるために、各試料を等量に実行した。 - 選択された抗体 11を用いてイムノブロット膜をプローブする。野生型E3リガーゼ、基質、およびmyc-ubを含むサンプル中の抗myc抗体を使用して、IPプロセスで引き出されたポリユビキチル化基質からの溶出液にスミアシグナルが検出されることを確認します。細胞溶解物(インプット)において、選択された基質、Fbxo7タンパク質、ユビキチル化タンパク質、およびハウスキーピングタンパク質(例えば、GAPDHおよびβ-actin)からのシグナルが検出され、各レーンにおいて同じ量のタンパク質を保証することを確認する。
注:使用した各抗体の希釈液は、製造元の指示に従って調製した。
結果
UXT(ユビキタスに発現された転写産物)は、心臓、脳、骨格筋、胎盤、膵臓、腎臓、肝臓などのマウスおよびヒトの組織において遍在的に発現されたタンパク質折り畳み複合体を形成するプレフォールディン様タンパク質である18。UXT-V1およびUXT-V2と名付けられたUXTの2つのスプライシングアイソフォームが、別個の機能および細胞内位置を果たすことが記載されている。UXT-V1?...
ディスカッション
ユビキチル化は、いくつかのタンパク質のレベルを調節し、多くのシグナル伝達経路および生物学的プロセスにおいて重要な役割を果たし、健康な細胞内環境を保証する重要な翻訳後修飾である。ユビキチン-プロテアソーム系(UPS)は、最近の製薬研究の主な焦点の1つであり、腫瘍抑制因子を安定化させる可能性、または発癌性産物22の分解を誘導する可能性を提供する。例?...
開示事項
著者らは、利益相反は存在しないと宣言しています。
謝辞
F.R.Tは、FAPESP助成金番号2020/15771-6およびCNPq Universal 405836/2018-0によってサポートされています。P.M.S.P および V.S は CAPES によってサポートされています。C.R.S.T.B.CはFAPESP奨学金番号2019/23466-1によってサポートされました。資料サポートをしてくれたサンドラ・R・C・マルヤマ(FAPESP 2016/20258-0)に感謝いたします。
資料
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
参考文献
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved