このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
タンパク質と水和水のピコ秒ナノ秒ダイナミクスを研究するための高分解能中性子分光法
要約
中性子後方散乱分光法は、タンパク質とその水和水のps-nsダイナミクスへの非破壊でラベルフリーのアクセスを提供します。このワークフローには、アミロイドタンパク質に関する2つの研究、すなわち凝集中のリゾチームの時間分解ダイナミクスと、繊維形成時のタウの水和水ダイナミクスに関する研究が提示されています。
要約
中性子散乱は、重水素以外の標識なしに、非破壊的な方法で広範囲のエネルギーのサンプル内のダイナミクスをプローブする可能性を提供します。特に、中性子後方散乱分光法は、複数の散乱角度で同時に散乱信号を記録し、ps-nsタイムスケールでの生物系のダイナミクスの研究に適しています。D2Oおよび場合によっては重水素化緩衝液成分を使用することにより、この方法は、液体状態のタンパク質の重心拡散と骨格および側鎖運動(内部ダイナミクス)の両方をモニタリングすることを可能にする。
さらに、水和水の動態は、H2Oで水和された過重水素化タンパク質の粉末を使用することによって研究することができる。この論文では、タンパク質と水分補給の水動態を調査するために、ラウエランジュバン研究所(ILL)の装置IN16Bで採用されたワークフローを紹介します。蒸気交換を用いた溶液サンプルおよび水和タンパク質粉末サンプルの調製について説明する。タンパク質と水和水ダイナミクスの両方のデータ解析手順は、中性子後方散乱分光計で取得できるさまざまなタイプのデータセット(準弾性スペクトルまたは固定ウィンドウスキャン)について説明されています。
この方法は、アミロイドタンパク質を含む2つの研究で示されています。リゾチームのμmサイズの球状凝集体(微粒子と表記)への凝集は、IN16Bで調査された空間および時間範囲でのワンステッププロセスで発生することが示されていますが、内部ダイナミクスは変化しません。さらに、タウの水和水の動態を過重水素化タンパク質の水和粉末で研究した。水の並進運動はアミロイド線維の形成時に活性化されることが示されている。最後に、他の実験的生物物理学的方法に関するダイナミクスの研究に関して中性子散乱がどのように位置付けられているかに関して、プロトコルの重要なステップが議論されます。
概要
中性子は電荷のない巨大な粒子であり、基礎物理学から生物学まで、さまざまな分野のサンプルを調べるために長年にわたって成功裏に使用されてきました1。生物学的用途では、小角中性子散乱、非弾性中性子散乱、中性子結晶構造解析および反射率測定が広く使用されています2,3,4。非弾性中性子散乱は、特定の標識自体を必要とせずにダイナミクスのアンサンブル平均測定を提供し、サイズやタンパク質に依存しない信号品質を提供します5。測定は、重水素化細菌ライセートやin vivoなどの細胞内培地を模倣する研究中のタンパク質の非常に複雑な環境を使用して行うことができます3,6,7。ダイナミクス、すなわち、i)飛行時間によるサブps-psダイナミクスへのアクセス、ii)後方散乱によるps-nsダイナミクスへのアクセス、iii)nsから数百nsまでのダイナミクスへのアクセスなど、さまざまな実験セットアップを使用してダイナミクスを研究することができます。中性子後方散乱は、ブラッグの法則2d sinθ = nλを利用しており、dは結晶内の平面間の距離、θは散乱角、nは散乱次数、λは波長です。検出器への後方散乱に結晶を使用することで、エネルギーの高分解能(通常~0.8μeV)を達成することができます。エネルギー交換を測定するには、後方散乱で結晶を運ぶドップラードライブを使用して入射中性子波長8、9、10を定義および調整するか(図1)、エネルギー分解能の低下を犠牲にして飛行時間型セットアップを使用できます11。
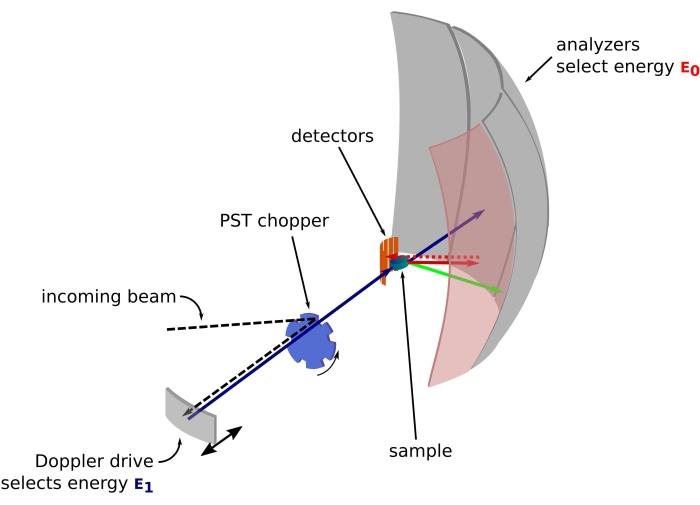
図1:ドップラー駆動の中性子後方散乱分光計のスケッチ。入射ビームは位相空間変換(PST)チョッパ42に当たり、試料位置における磁束を増加させる。次に、ドップラードライブによってサンプルに向かって後方散乱され、ドップラードライブはエネルギーE1(シアンの矢印)を選択します。次に、中性子はサンプルによって散乱され(矢印の色で表される異なるエネルギーで)、Si 111結晶で作られた分析装置は、特定のエネルギーE0(ここでは赤い色の矢印)の中性子のみを後方散乱します。したがって、運動量移動qは検出器アレイ上の中性子の検出位置から得られ、エネルギー移動は差E1-E0から得られます。PSTによって生成された中性子パルスに予想される飛行時間は、検出器管に向かって直接散乱された中性子からの信号を破棄するために使用されます。略語:PST =位相空間変換。この図の拡大版を表示するには、ここをクリックしてください。
後方散乱分光法では、タンパク質などの水素プロトンに富むサンプルからのシグナルへの主な寄与は、散乱強度Sinc(q、ω)が式(1)12で示されるインコヒーレント散乱から来ています
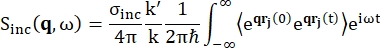 (1)
(1)
ここでσincは考慮される元素のインコヒーレント断面であり、k'は散乱波ベクトルのノルム、kは入射波ベクトルのノルム、q(= k - k')運動量移動、r j(t)時間tにおける原子jの位置ベクトル、ωは入射中性子と系との間のエネルギー移動に対応する周波数である。角括弧はアンサンブル平均を示します。したがって、インコヒーレント散乱は、原子位置のアンサンブル平均単粒子自己相関を時間とプローブし、システム内のすべての原子と異なる時間起源(アンサンブル平均)で平均化された自己ダイナミクスを提供します。散乱関数は、中間散乱関数 I(q, t) のときのフーリエ変換であり、式 (2) で示されるファンホーブ相関関数の空間でのフーリエ変換と見なすことができます。
 (2)
(2)
ここで、ρ(r,t)は、位置rと時間t13で原子を見つける確率密度です。
フィッキアン拡散過程の場合、自己拡散関数は、γ = Dq2で与えられる線幅のローレンツ分布からなる散乱関数の二重フーリエ変換の後に生じます(式(3)を参照)。
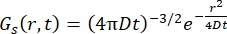 (3)
(3)
より洗練されたモデルが開発され、ps-ns内部タンパク質ダイナミクス14のSingwiとSjölanderによるジャンプ拡散モデルや、水和水15,16,17のSearsによる回転モデルなど、有用であることがわかりました。
フランスのグルノーブルにあるILLの中性子後方散乱(NBS)装置IN16B 8,9(補足図S1)では、タンパク質で一般的に使用されるセットアップは、入射波長を調整するためのドップラードライブを備えたアナライザー用のSi 111結晶で構成され(補足図S2A)、それによって運動量伝達範囲~0.2 Å-1 < q < ~2 Å-1および-30 μeVのエネルギー伝達範囲にアクセスできます< 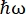 < 30μeV-数psから数nsの範囲のタイムスケールと数Åの距離に対応します。さらに、IN16Bは、固定エネルギー伝達でのデータ収集を含む弾性および非弾性固定ウィンドウスキャン(E/IFWS)10を実行する可能性を提供します。中性子を扱う場合、磁束が制限されるため、E/IFWSは1回のエネルギー伝達で磁束を最大化できるため、満足のいく信号対雑音比を得るために必要な取得時間を短縮できます。より最近のオプションは、後方散乱および飛行時間型分光計(BATS)モード11であり、ドップラー駆動よりも高い磁束で、広範囲のエネルギー移動(例えば、-150μeV<<
< 30μeV-数psから数nsの範囲のタイムスケールと数Åの距離に対応します。さらに、IN16Bは、固定エネルギー伝達でのデータ収集を含む弾性および非弾性固定ウィンドウスキャン(E/IFWS)10を実行する可能性を提供します。中性子を扱う場合、磁束が制限されるため、E/IFWSは1回のエネルギー伝達で磁束を最大化できるため、満足のいく信号対雑音比を得るために必要な取得時間を短縮できます。より最近のオプションは、後方散乱および飛行時間型分光計(BATS)モード11であり、ドップラー駆動よりも高い磁束で、広範囲のエネルギー移動(例えば、-150μeV<< 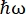 150μeV)の測定を可能にしますが、エネルギー分解能は低くなります(補足図S2B)。
150μeV)の測定を可能にしますが、エネルギー分解能は低くなります(補足図S2B)。
中性子散乱の重要な特性は、インコヒーレント断面積σincが水素に対して重水素よりも40倍高い値を持ち、生物学的サンプルに一般的に見られる他の元素に対しては無視できることです。したがって、液体環境におけるタンパク質の動態は、重水素化緩衝液を用いて研究することができ、粉末状態は、D2Oで水和した水素化タンパク質粉末によるタンパク質内部動態の研究、またはH2Oで水和した過重化タンパク質粉末の水和水の研究を可能にする。液体状態では、中性子後方散乱は通常、タンパク質の重心自己拡散(フィッキアン型拡散)とその内部ダイナミクスに同時にアクセスすることができます。後者は、通常、いわゆるジャンプ拡散モデルなどによって記述されるバックボーンおよびサイドチェーン運動です3,18。水素化タンパク質粉末では、タンパク質の拡散は存在せず、内部ダイナミクスのみをモデル化する必要があります。水和水の場合、水分子の並進運動および回転運動の寄与は、運動量移動qに対して異なる依存性を示し、これは、データ分析プロセスにおけるそれらの区別を可能にする17。
この論文は、β鎖のスタックからなる標準的な形に展開し、凝集し、いわゆるクロスβパターン19,20-そして細長い繊維を形成することができることが判明したタンパク質の研究による中性子後方散乱法を示しています。これはいわゆるアミロイド凝集であり、アルツハイマー病やパーキンソン病などの神経変性疾患における中心的な役割のために広く研究されています21,22。アミロイドタンパク質の研究は、それらが果たすことができる機能的役割23,24または新規生体材料の開発のためのそれらの高い可能性によっても動機付けられています25。アミロイド凝集の物理化学的決定要因は依然として不明であり、過去数年間の驚異的な進歩にもかかわらず、アミロイド凝集の一般理論は利用できません21,26。
アミロイド凝集は、タンパク質の構造と経時的な安定性の変化を意味し、その研究は当然、タンパク質の立体構造安定性、タンパク質機能、およびタンパク質エネルギーランドスケープに関連するダイナミクスを意味します27。ダイナミクスは、最速の運動28のエントロピー寄与を通じて特定の状態の安定性に直接関連しており、タンパク質の機能は、光感受性タンパク質29 のサブpsからドメイン運動のmsまで、ピコ秒-ナノ秒のダイナミクス30によって促進されるさまざまなタイムスケールの運動によって維持できます。
中性子後方散乱分光法を使用してアミロイドタンパク質を研究する2つの例、1つはタンパク質のダイナミクスを研究するための液体状態、もう1つは水和水のダイナミクスを研究するための水和粉末状態の2つの例を紹介します。第1の例は、リゾチームのμmサイズの球体(微粒子と呼ばれる)への凝集に関するものであり、続いてリアルタイムで5、第2の例は、ヒトタンパク質タウ31の天然状態および凝集状態における水動態の比較に関するものである。
リゾチームは免疫防御に関与する酵素で、129個のアミノ酸残基で構成されています。リゾチームは、pD10.5および90°Cの温度で重水素化緩衝液中で微粒子を形成することができます。 中性子散乱により、リゾチームの重心拡散係数の時間発展がチオフラビンT蛍光(アミロイド交差βパターンの形成をモニターするために使用される蛍光プローブ32)の単一の指数関数的速度論に従うことを示し、粒子形成上部構造と交差βパターンが同じ速度で1つのステップで発生することを示しています。さらに、内部ダイナミクスは凝集プロセス全体を通して一定であり、これはNBS装置では観察できない速い立体構造変化、または凝集時にタンパク質の内部エネルギーに大きな変化がないことによって説明できます。
ヒトタンパク質タウは、いわゆる2N4Rアイソフォームの441アミノ酸からなる天然変性タンパク質(IDP)であり、アルツハイマー病に特に関与しています33。過重水素化タンパク質タウの粉末に対する中性子後方散乱を用いて、繊維状態で水和水ダイナミクスが増加し、並進運動を受ける水分子の集団が多いことを示しました。この結果は、水和水エントロピーの増加がタウのアミロイド細動を引き起こす可能性があることを示唆している。
プロトコル
1. 液体状態のタンパク質用の重水素化バッファーを調製します
- 緩衝液のすべての成分を純粋なD2Oに溶解する。
- pH電極をH2Oで較正した場合、NaODまたはDCl34を用いて式pD = pH + 0.4に従ってpDを調整する。
注:H2Oの代わりにD2Oを使用すると、タンパク質の溶解度に影響を与える可能性があり、緩衝条件を適応させる必要がある場合があります(たとえば、塩濃度のわずかな変化によって)。
2.過重水素化タンパク質のH2O水和粉末を調製する
- サンプルホルダーを準備します。
- インジウムワイヤーシールとネジで平らなアルミニウムサンプルホルダーを水とエタノールで完全に洗浄し、乾燥させます。
注:粉末を表面全体に均一に分布させることができるように、平らなサンプルホルダーが使用されます。粉末の量は、壁の間に維持でき、サンプルホルダーを垂直に置いたときに落下しないように十分である必要があります。 - サンプルホルダーのさまざまな部分(底部、蓋、インジウムワイヤー)を精密天びんで別々に計量します。
- サンプルホルダーの底部の溝に1mmのインジウムワイヤーシールを配置し、両端が結合する場所に小さな重なりを残します(図2A)。
- 適量の凍結乾燥タンパク質(通常~100 mgのタンパク質)を、サンプルホルダーの底部の内面を満たすように配置します。
- インジウムワイヤーシールとネジで平らなアルミニウムサンプルホルダーを水とエタノールで完全に洗浄し、乾燥させます。
- プロテインパウダーを水和させます。
- サンプルホルダーをP2O5粉末を含むペトリ皿を備えたデシケーターに24時間置き、タンパク質粉末35を完全に乾燥させます(図2B)。インジウムシールと乾燥粉末とを含むサンプルホルダーの乾燥底部を秤量し、m乾燥を得る。
注意:P2O5 粉末は非常に腐食性があります。 - デシケーターからP 2 O5を取り出し、D2Oが入ったペトリ皿を中に入れる。粉末の質量を定期的に制御して、水和レベルh = mhyd / mdryを確認します(mhydとmdryはそれぞれ水和粉末と乾燥粉末の質量です)。
注:インスリンなどの疎水性の高いタンパク質の場合、デシケーター内の温度を上げて蒸気圧を高め、目的の水和レベルhに達する必要がある場合があります。 - 手順2.2.1と2.2.2を少なくとも3回繰り返して、交換可能なすべての水素を重陽子に正しく変換します。
注:あるいは、タンパク質がそれによって影響を受けない限り、凍結乾燥および純粋なD2O中での溶解のサイクルは、より良いH / D交換のために使用され得る。 - 粉末を所望のレベルよりわずかに上に水和し、インジウムワイヤーと水和粉末を有するサンプルホルダーの底部を精密天秤に残し、質量が所望の値までゆっくりと減少するのを待って目標hを得る(中型の球状タンパク質を1つの完全な水和層で覆う場合は通常0.2〜0.4)。
- 底部にすばやく蓋をし、最初に4本のネジでサンプルホルダーを閉じて蒸気交換を停止します(補足図S3A)。
- 底部と蓋の間に隙間が見えなくなるまで、残りのすべてのネジを配置して締めます(補足図S3B)。
- 密閉されたサンプルホルダーの重量を量って、中性子実験後の漏れによる潜在的な水和損失がないか確認します。
- サンプルホルダーをP2O5粉末を含むペトリ皿を備えたデシケーターに24時間置き、タンパク質粉末35を完全に乾燥させます(図2B)。インジウムシールと乾燥粉末とを含むサンプルホルダーの乾燥底部を秤量し、m乾燥を得る。
3. インコヒーレント中性子散乱実験を行う
- 実験に必要な装置の構成について、指定されたビームタイムの数週間前に現地の担当者と話し合い、再確認します。
- 液体状態サンプルを準備します。
- タンパク質を重水素化緩衝液に溶解します。
- 水を使用してサンプルホルダーに入れる液体の適切な量を決定します(サンプルホルダーを閉じたときにオーバーフローがないことを確認してください。 図2C)。
注:次の手順(3.3および3.4)は、低温炉をサンプル環境として使用して、ILL16,8,9のNBS分光計IN8,9Bで実施された実験について説明しています。機器の制御システムは機器ごとに変更されますが、動作原理は同じままです。
- サンプルを挿入します。
- サンプルスティックを完全に乾燥させ(図2D)、材料を取り扱う前に(ILLで)電離放射線量が100μSv / h未満であることを確認した後、前のサンプルがあればそれを取り除きます。
- サンプルを置き、ビームの中心に対して適切なセンタリングを確認し(補足図S4)、サンプルスティックをクライオ炉に挿入します(図2D)。真空ポンプをオンにして10〜3バール未満に到達し、次の3 回を繰り返してクライオ炉内の空気を洗い流します:大気圧に達するまでクライオファーネスにヘリウムガスを満たし、真空ポンプを使用してガスを再度除去します。
注意: フラットサンプルホルダーの場合、サンプルホルダーは入射ビームに対して45°の角度に向ける必要があります。有用な運動量伝達範囲は、セルによる吸収と散乱のために減少する可能性があります。カドミウムなどの強力な中性子吸収材を使用して、サンプルホルダーの特定の部分(ネジ、厚い部分など)をマスクできます。 - 圧力が~0.05バールになるように、クライオ炉にヘリウムガスを導入します。
- データを取得します(たとえば、ILLでIN16BでNOMADを使用すると、準弾性中性子スペクトル(QENS)スペクトルを取得する前に200 Kの温度を好むと仮定し、次に毎分0.5 Kで310 Kへの温度ランプ中にE/IFWS、最後に310 KでQENSを好むと仮定します)。
- NOMADを使用して、実行タブで、ファーネスクライオスタットコントローラーを発射台にドラッグアンドドロップします。温度を 200 K に設定します。高速モードと30分のタイムアウトを使用して、温度が安定する時間を確保します。回転矢印アイコンをクリックしてバックグラウンドで実行し、温度低下時にデータを取得できるようにします。
- IN16DopplerSettingsコントローラーをドラッグアンドドロップし、速度プロファイルを最大ΔEによって設定された正確な速度、0.00μeVの値、128チャンネルに設定して、EFWS構成を取得します。
- カウントコントローラーをドラッグアンドドロップし、データを簡単に識別できる名前を[字幕]フィールドに入力し、30秒のスキャンを60回繰り返して設定します(補足図S5A)。
- IN16DopplerSettings コントローラーをドラッグアンドドロップし、速度プロファイルを 4.5 m/s の値と 2,048 チャネルで速度によって設定されたサインに設定して、QENS 構成を取得します。
- カウント コントローラーをドラッグ アンドドロップし、 30分間のスキャンを4回繰り返し ます(補足図S5B)。
- 温度ランプの場合は、FurnaceCryostatコントローラーをドラッグアンドドロップし、温度を310 Kに設定し、ランプをΔ = 0.05 Kおよび6秒でSetPointに設定します。220分のタイムアウトを使用します(補足図S6A)。
- for ループを 65 回繰り返します。内部に、手順16のようにIN3.4.2DopplerSettingsコントローラーを挿入し、その後に30秒のシングルカウントを挿入します。続いて、前述のようにIN16DopplerSettingsを挿入しますが、1.5μeVのエネルギーオフセットと1,024チャンネルを使用し、その後に3分のシングルカウントを使用します(補足図S6B)。
- 310 Kで最後のQENSを取得するには、IN16DopplerSettingsとCountコントローラをそれぞれ手順3.4.4と3.4.5の説明に従ってドラッグアンドドロップします。
- スタートボタン(ウィンドウの下部にある直角三角形)を押して、スクリプトを実行します。
注:すべての実験では、キャリブレーションデータの取得が必要です。すなわち、減算または吸収補正のための空のセルと、バックグラウンドをモデル化するために用いられる異なる温度でのバッファーのみ、およびバナジウムの測定(または同等には、10K以下の温度のサンプル)の装置の分解能関数を得る。
4. データ分析 - QENS
- Python ソフトウェア nPDyn v3.x36 の 'IN16B_QENS.process()' メソッドを使用してデータセットをインポートします。
nPDyn.dataParsers import IN16B_QENS からの>>>>
>>>サンプル = IN16B_QENS(
...<データ ファイルへのパス>
...[detGroup=<整数または XML 内の検出器グループ化ファイル
...フォーマット>]
... ).プロセス()
>>> サンプル = sample.get_q_range(0.3, 1.8) - 次のコマンドを使用してデータ修正(オプション)を実行します(詳細については、nPDynのドキュメントを参照してください、 図3)。
#it、空のセル、バナジウム、およびバッファーのデータを想定しています
#は 'empty_cell'、 'バナジウム'というデータセットにすでにインポートされています。
#と 'バッファ'をそれぞれ。
#スケーリング係数を使用した空のセル減算の場合
# (エラーは自動的に伝播されます)
>>> サンプル = サンプル - 0.95 * empty_cell
# パールマン-Ping係数を用いた補正用
#(上記の例と相互に排他的)
>>> サンプル = サンプル.吸収補正(empty_cell)
# 正規化用
>>> サンプル = サンプル.正規化(バナジウム)
# 観測可能な軸に沿ってビニングする場合
#観測可能なのはここでの集計時間です
>>> サンプル = サンプル.bin(3、軸 = 0) - キャリブレーションデータを適合させます。データセット(空のセル、空のセル、重水素化バッファー(必要な場合)、およびバナジウム)は、組み込みモデルまたはユーザー定義モデルを使用して適合できます(nPDynのドキュメントを参照)。
>>> from nPDyn.models.builtins import (
...モデルPサイズ,
...モデル水,
...モデル校正済みD2O,
... )
# 組み込みモデルは運動量の列ベクトルを使用します
# q値を転送する
>>> q = バナジウム.q[:, なし]
#バナジウムは擬似フォークトプロファイルを使用して取り付けられています
>>> vanadium.fit(modelPVoigt(q)) - 「モデルウォーター」と呼ばれる水分補給水用の内蔵モデルを使用してください。このモデルは、式(4)17に示すように読みます
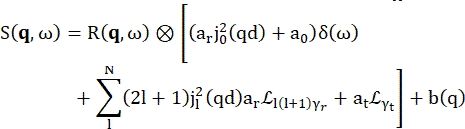 (4)
(4)
ここで、0、r、およびt は、それぞれ弾性信号、回転運動、および並進運動の相対的な寄与を説明するスカラーです。j1(qd) は l次の 球面ベッセル関数で、q は運動量伝達です。d水分子中のO-H距離;δ(ω)はディラックデルタであり、ここではEISFを掛けます。N は、使用される球面ベッセル関数の最高次 (通常は ~5) です。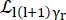 であり
であり 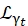 、 はそれぞれローレンツ回転運動と並進運動であり、b(q) は平坦な背景項である。球面ベッセル関数は、水分子の各角運動量状態の相対的な寄与を与え、数Nは運動量伝達q範囲に基づいて決定されます。典型的なNBS分光計の場合、N = 4までの項は信号をほぼ完全に説明しています(補足図S7)。
、 はそれぞれローレンツ回転運動と並進運動であり、b(q) は平坦な背景項である。球面ベッセル関数は、水分子の各角運動量状態の相対的な寄与を与え、数Nは運動量伝達q範囲に基づいて決定されます。典型的なNBS分光計の場合、N = 4までの項は信号をほぼ完全に説明しています(補足図S7)。
#ここで、式2は水和水に使用されます
#解像度関数と畳み込みと追加
#D2Oの背景は、
# 提供された引数
>>> sample.fit(modelWater(q),
...res=バナジウム、
...bkgd=バッファ、
...volume_fraction_bkgd=0.95
... )
注:回転運動と並進運動の寄与は、完全に厳密になるように複雑にする必要があります。加法モデルの成功は、タンパク質表面に異なる水の集団が存在し、アクセス可能なエネルギー範囲が限られていることに起因します。 - 以下を使用してデータをプロットします(図4)。
nPDyn.plotインポートプロットからの>>>
>>>プロット(サンプル)
5.データ分析-温度ランプ、弾性固定ウィンドウスキャン(EFWS)
- セクション4と同様の手順を使用して、最低温度(通常は10 K)での信号によって温度ランプデータを正規化します。
nPDyn.dataParsers import IN16B_FWS からの>>>
>>>サンプル = IN16B_FWS(
...<データファイルへのパス>、
...detGroup=[detGroup=<整数またはディテクタの XML 形式のグループ化ファイル>]
... ).プロセス()
#オブザーバブルの最初の5つのポイントによる正規化
温度に対応する#軸
>>> サンプル /= サンプル[:5].mean(0)
#ここで使用される運動量伝達Q範囲は小さいです
#使用されるモデルは低Qに対してのみ有効です
>>> サンプル = sample.get_q_range(0.2, 0.8) - 単純なガウスモデルを使用して開始し、その幅はいわゆる平均二乗変位(MSD)によって与えられます。モデルを構築し、適合するには、次のコマンドを使用します。
>>> numpy を np としてインポートする
nPDyn.models からの>>>は、パラメータ、モデル、コンポーネントをインポートします。
#aはスケール係数です
>>>パラメータ = パラメータ(
...a={'value': 1, 'bounds': (0, np.inf)},
...msd={'value': 1, 'bounds': (0, np.inf)}
... )
>>> モデル = モデル(パラメータ)
>>> model.addComponent(Component(
...'ガウス'、
...ラムダ x, a, msd: a * np.exp(-x ** 2 * msd / 6)
... ))
>>> sample.fit(model, x=sample.q[:, None])
>>>プロット(サンプル)
注:ガウス近似は、q2MSD << 1に対して常に成り立ちますが、サンプル間の相対比較には、より広い運動量伝達範囲を使用できます。ガウス近似を超えるより洗練されたモデルが開発されています37,38,39。
6.データ分析-弾性および非弾性固定ウィンドウスキャン(E / IFWS)
- 手順 4 と同様に、データセットをインポートしますが、'IN16B_FWS' クラスを使用します。
nPDyn.dataParsers import IN16B_FWS からの>>>
>>>サンプル = IN16B_FWS(
...<データ ファイルへのパス>
...[detGroup=<整数または XML 形式の検出器グループ化ファイル>]
... ).プロセス()
>>> サンプル = sample.get_q_range(0.3, 1.8) - キャリブレーションデータとサンプルデータを適合させます。
- 一般化されたMSD40 を使用するか、粗いQENSスペクトル(エネルギー軸上に少数のデータポイントしかない)と見なすことにより、E/IFWSデータを解析します。E/IFWSを粗QENSとして見ると、QENSに使用されるモデルを使用して、E/IFWSデータセット全体を一度に適合させます(エネルギー移動と運動量移動のグローバルフィット)。
注:後者の溶液(E/IFWSデータに対するQENSの溶液使用モデル)は、ここでは、重心拡散とタンパク質内部ダイナミクスの運動量移動依存性が課される場合に使用されます。 - 以下の式(5)を用いて液体中のタンパク質動態をモデル化する(nPDynの'modelProteinJumpDiff')。
 (5)
(5)
ここで、R(q,ω) は分解能関数です。β各運動量伝達qに対して独立したスカラーである。a0は弾性インコヒーレント構造係数(EISF)です。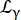 式(6)
式(6)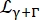 で与えられる幅を持つ重心拡散を説明するローレンツ。は、重心拡散と、内部ダイナミクスを説明するジャンプ拡散モデル14(式(7);サンプル中の体積分率によって再スケーリングされたD2Oからの適合信号である)
で与えられる幅を持つ重心拡散を説明するローレンツ。は、重心拡散と、内部ダイナミクスを説明するジャンプ拡散モデル14(式(7);サンプル中の体積分率によって再スケーリングされたD2Oからの適合信号である) に従う寄与を含むローレンツ分布である。
に従う寄与を含むローレンツ分布である。
γ = Dsq2 (6)
Dsは自己拡散係数である。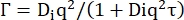 (7)
(7)
Diは内部ダイナミクスの見かけの拡散係数、 τ は拡散運動の緩和時間です。
>>> from nPDyn.models.builtins import (
...モデルPサイズ,
...モデルプロテインジャンプ差分、
...モデル校正済みD2O,
... )
# 組み込みモデルは運動量の列ベクトルを使用します
# q値を転送する
>>> q = バナジウム.q[:, なし]
#バナジウムは擬似フォークトプロファイルを使用して取り付けられています
>>> vanadium.fit(modelPVoigt(q))
#純粋なD2Oの場合、キャリブレーションされた線幅のモデル
#異なる温度のためにnPDynに含まれています
>>> buffer.fit(modelCalibratedD2O(q, temp=363))
#ここでは、式3は液体サンプルに使用されます
#解像度関数と畳み込みと追加
#D2Oの背景は、#指定された引数で自動的に行われます
>>> sample.fit(modelProteinJumpDiff(q),
...res=バナジウム、
...bkgd=バッファ、
...volume_fraction_bkgd=0.95
... )
- 一般化されたMSD40 を使用するか、粗いQENSスペクトル(エネルギー軸上に少数のデータポイントしかない)と見なすことにより、E/IFWSデータを解析します。E/IFWSを粗QENSとして見ると、QENSに使用されるモデルを使用して、E/IFWSデータセット全体を一度に適合させます(エネルギー移動と運動量移動のグローバルフィット)。
- 以下を使用して適合データをプロットします。
nPDyn.plotインポートプロットからの>>>
>>>プロット(サンプル)
結果
リゾチームの微粒子への凝集は、重水素化緩衝液(pD 10.5で0.1 M NaCl)中で50 mg/mLのタンパク質濃度で90°Cで実施しました。微粒子の形成は、90°Cへの温度上昇によって引き起こされ、6時間以内に発生します(補足図S8)。データ取得は、上記のプロトコルで説明されているように、IN16Bで実行されました(データはILLによって永続的にキュレーションされ、http://dx.doi.org/10.5291/ILL-DATA.8-04-811 ...
ディスカッション
中性子分光法は、重水素化を使用した場合のタンパク質のサイズや溶液の複雑さに関係なく、タンパク質サンプルのアンサンブル平均ps-nsダイナミクスをプローブできる唯一の方法です6。具体的には、溶液中でのタンパク質集合体の自己拡散を調べることにより、そのような集合体の流体力学的サイズを明確に決定することができる。それにもかかわらず、この方法は一般?...
開示事項
著者は、開示すべき利益相反はありません。
謝辞
著者らは、ドイツのガルヒングにあるハインツ・マイヤー・ライプニッツ・ツェントルムにあるユーリッヒ中性子科学センターのミカエラ・ザンポーニ氏に、SPHERES装置で行われた中性子散乱実験の一部に感謝している。この研究は、欧州連合がHPRI-2001-50065およびRII3-CT-2003-505925の契約に基づいて資金提供する重水素化研究所(DLAB)コンソーシアムの活動、および英国工学物理科学研究評議会(EPSRC)が資金提供する助成金GR/R99393/01およびEP/C015452/1に基づくラウエランジュバンEMBL研究所内の活動から恩恵を受けています。主要行動を通じた第7次枠組みプログラムに基づく欧州委員会による支援:欧州研究領域、研究インフラストラクチャの強化が認められています[契約226507(NMI3)]。ケビン・ポウノットとクリスチャン・ベックは、ポスドクフェローシップの資金提供について、連邦教育研究省(BMBF、助成金番号05K19VTB)に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| Aluminum sample holder | Not commercially available. Either the local contact on the instrument can provide them or they can be manufactured based on a technical drawing that can be provided by the local contact. | ||
| Deuterium chloride, 35 wt. % in D2O, ≥99 atom % D | Sigma-Aldrich | 543047 | |
| Deuterium oxide (D, 99.9%) | Eurisotop | DLM-4DR-PK | |
| Dow Corning high-vacuum silicone grease | Sigma-Aldrich | Z273554-1EA | |
| Ethanol 96%, EMSURE Reag. Ph Eur | Sigma-Aldrich | 1.5901 | |
| Glass dessicator | VWR | 75871-660 | |
| Glass dessicator plate, 140 mm | VWR | 89038-068 | |
| Indium wire, 1.0 mm (0.04 in) dia, Puratronic, 99.999% | Alfa Aesar | 00470.G1 | |
| Lysozyme from chicken egg white dialyzed, lyophilized, powder, ~100,000 U/mg | Sigma-Aldrich | 62970 | |
| nPDyn | v3.x | see github.com/kpounot/nPDyn, model functions fot fitting also included in the software | |
| OHAUS AX324 Adventurer balance, internal calibration | Dutscher | 92641 | |
| Phosphorus pentoxide, ReagentPlus, 99% | Sigma-Aldrich | 214701 | |
| Pipette ErgoOne 0.5-10 μL | Starlab | S7100-0510 | |
| Pipette ErgoOne 100-1,000 μL | Starlab | S7100-1000 | |
| Pipette ErgoOne 20-200 μL | Starlab | S7100-2200 | |
| Pipette tip TipOne 1,000 μL | Starlab | S1111-6001 | |
| Pipette tip TipOne 10 μL | Starlab | S1111-3200 | |
| Pipette tip TipOne 200 μL | Starlab | S1111-0206 | |
| Sodium deuteroxide solution, 40 wt. % in D2O, 99.5 atom % D | Sigma-Aldrich | 372072 |
参考文献
- Jacrot, B. Des neutrons pour la science: Histoire de l'Institut Laue-Langevin. Des neutrons pour la science. EDP Sciences. , (2021).
- Mahieu, E., Gabel, F. Biological small-angle neutron scattering: recent results and development. Acta Crystallographica Section D. 74 (8), 715-726 (2018).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Martel, A., et al. Membrane permeation versus amyloidogenicity: A multitechnique study of islet amyloid polypeptide interaction with model membranes. Journal of the American Chemical Society. 139 (1), 137-148 (2017).
- Pounot, K., et al. Tracking internal and global diffusive dynamics during protein aggregation by high-resolution neutron spectroscopy. The Journal of Physical Chemistry Letters. 11 (15), 6299-6304 (2020).
- Grimaldo, M., et al. Protein short-time diffusion in a naturally crowded environment. The Journal of Physical Chemistry Letters. 10 (8), 1709-1715 (2019).
- Jasnin, M., Stadler, A., Tehei, M., Zaccai, G. Specific cellular water dynamics observed in vivo by neutron scattering and NMR. Physical Chemistry Chemical Physics. 12 (35), 10154-10160 (2010).
- Frick, B. The neutron backscattering spectrometer IN16 at ILL-high energy resolution with high intensity and excellent signal-to-noise ratio. Neutron News. 13 (2), 15-22 (2002).
- Frick, B., Mamontov, E., van Eijck, L., Seydel, T. Recent backscattering instrument developments at the ILL and SNS. Zeitschrift für Physikalische Chemie. 224 (1-2), 33-60 (2010).
- Frick, B., Combet, J., van Eijck, L. New possibilities with inelastic fixed window scans and linear motor Doppler drives on high resolution neutron backscattering spectrometers. Nuclear Instruments and Methods in Physics Research Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 669, 7-13 (2012).
- Appel, M., Frick, B., Magerl, A. A flexible high speed pulse chopper system for an inverted neutron time-of-flight option on backscattering spectrometers. Scientific Reports. 8 (1), 13580 (2018).
- Squires, G. L. . Introduction to the theory of thermal neutron scattering. , (1996).
- Singwi, K. S., Sjölander, A. Diffusive motions in water and cold neutron scattering. Physical Review. 119 (3), 863-871 (1960).
- Sears, V. F. Theory of cold neutron scattering by homonuclear diatomic liquids: i. free rotation. Canadian Journal of Physics. 44 (6), 1279-1297 (1966).
- Sears, V. F. Theory of cold neutron scattering by homonuclear liquid: ii. hindered rotation. Canadian Journal of Physics. 44 (6), 1299-1311 (1966).
- Schirò, G., et al. Translational diffusion of hydration water correlates with functional motions in folded and intrinsically disordered proteins. Nature Communications. 6, 6490 (2015).
- Grimaldo, M., et al. Hierarchical molecular dynamics of bovine serum albumin in concentrated aqueous solution below and above thermal denaturation. Physical Chemistry Chemical Physics. 17 (6), 4645-4655 (2015).
- Eanes, E. D., Glenner, G. G. X-ray diffraction studies on amyloid filaments. Journal of Histochemistry & Cytochemistry. 16 (11), 673-677 (1968).
- Bonar, L., Cohen, A. S., Skinner, M. M. Characterization of the Amyloid Fibril as a Cross-β Protein. Proceedings of the Society for Experimental Biology and Medicine. 131 (4), 1373-1375 (1969).
- Chiti, F., Dobson, C. M. Protein Misfolding, Amyloid Formation, and Human Disease: A Summary of Progress Over the Last Decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews Molecular Cell Biology. 15 (6), 384-396 (2014).
- Maji, S. K., et al. Functional amyloids as natural storage of peptide hormones in pituitary secretory granules. Science. 325 (5938), 328-332 (2009).
- Li, J., et al. The RIP1/RIP3 necrosome forms a functional amyloid signaling complex required for programmed necrosis. Cell. 150 (2), 339-350 (2012).
- Knowles, T. P. J., Mezzenga, R. Amyloid fibrils as building blocks for natural and artificial functional materials. Advanced Materials. 28 (31), 6546-6561 (2016).
- Stephens, A. D., Kaminski Schierle, G. S. The role of water in amyloid aggregation kinetics. Current Opinion in Structural Biology. 58, 115-123 (2019).
- Adamcik, J., Mezzenga, R. Amyloid polymorphism in the protein folding and aggregation energy landscape. Angewandte Chemie International Edition. 57 (28), 8370-8382 (2018).
- Liu, Z., et al. Entropic contribution to enhanced thermal stability in the thermostable P450 CYP119. Proceedings of the National Academy of Sciences of the United States of America. 115 (43), 10049-10058 (2018).
- Coquelle, N., et al. Chromophore twisting in the excited state of a photoswitchable fluorescent protein captured by time-resolved serial femtosecond crystallography. Nature Chemistry. 10 (1), 31-37 (2018).
- Henzler-Wildman, K. A., et al. A hierarchy of timescales in protein dynamics is linked to enzyme catalysis. Nature. 450 (7171), 913-916 (2007).
- Fichou, Y., et al. Hydration water mobility is enhanced around tau amyloid fibers. Proceedings of the National Academy of Sciences of the United States of America. 112 (20), 6365-6370 (2015).
- Burns, J., Pennock, C. A., Stoward, P. J. The specificity of the staining of amyloid deposits with thioflavine T. The Journal of Pathology and Bacteriology. 94 (2), 337-344 (1967).
- Iqbal, K., Liu, F., Gong, C. -. X., Grundke-Iqbal, I. Tau in Alzheimer disease and related tauopathies. Current Alzheimer Research. 7 (8), 656-664 (2010).
- Krȩżel, A., Bal, W. A formula for correlating pKa values determined in D2O and H2O. Journal of Inorganic Biochemistry. 98 (1), 161-166 (2004).
- Dolman, M., Halling, P. J., Moore, B. D., Waldron, S. How dry are anhydrous enzymes? Measurement of residual and buried 18O-labeled water molecules using mass spectrometry. Biopolymers. 41 (3), 313-321 (1997).
- Pounot, K. kpounotnPDyn: v3.0.0. Zenodo. , (2021).
- Yi, Z., Miao, Y., Baudry, J., Jain, N., Smith, J. C. Derivation of mean-square displacements for protein dynamics from elastic incoherent neutron scattering. Journal of Physical Chemistry B. 116 (16), 5028-5036 (2012).
- Peters, J., Kneller, G. R. Motional heterogeneity in human acetylcholinesterase revealed by a non-Gaussian model for elastic incoherent neutron scattering. The Journal of Chemical Physics. 139 (16), 165102 (2013).
- Zeller, D., Telling, M. T. F., Zamponi, M., García Sakai, V., Peters, J. Analysis of elastic incoherent neutron scattering data beyond the Gaussian approximation. The Journal of Chemical Physics. 149 (23), 234908 (2018).
- Roosen-Runge, F., Seydel, T. A generalized mean-squared displacement from inelastic fixed window scans of incoherent neutron scattering as a model-free indicator of anomalous diffusion confinement. EPJ Web of Conferences. 83, 02015 (2015).
- Ortega, A., Amorós, D., García de la Torre, J. Prediction of hydrodynamic and other solution properties of rigid proteins from atomic- and residue-level models. Biophysical Journal. 101 (4), 892-898 (2011).
- Hennig, M., Frick, B., Seydel, T. IUCr Optimum velocity of a phase-space transformer for cold-neutron backscattering spectroscopy. Journal of Applied Crystallography. 44 (3), 467-472 (2011).
- Paalman, H. H., Pings, C. J. Numerical evaluation of X-ray absorption factors for cylindrical samples and annular sample cells. Journal of Applied Physics. 33 (8), 2635-2639 (1962).
- Ow, S. -. Y., Dunstan, D. E. The effect of concentration, temperature and stirring on hen egg white lysozyme amyloid formation. Soft Matter. 9 (40), 9692-9701 (2013).
- Tominaga, T., Sahara, M., Kawakita, Y., Nakagawa, H., Yamada, T. Evaluation of sample cell materials for aqueous solutions used in quasi-elastic neutron scattering measurements. Journal of Applied Crystallography. 54 (6), 1631-1640 (2021).
- Beck, C., et al. Following protein dynamics in real time during crystallization. Crystal Growth & Design. 19 (12), 7036-7045 (2019).
- Smith, A. A., Testori, E., Cadalbert, R., Meier, B. H., Ernst, M. Characterization of fibril dynamics on three timescales by solid-state NMR. Journal of Biomolecular NMR. 65 (3-4), 171-191 (2016).
- Wang, T., Jo, H., DeGrado, W. F., Hong, M. Water distribution, dynamics, and interactions with Alzheimer's β-amyloid fibrils investigated by solid-state NMR. Journal of the American Chemical Society. 139 (17), 6242-6252 (2017).
- Rezaei-Ghaleh, N., Giller, K., Becker, S., Zweckstetter, M. Effect of zinc dinding on β-amyloid structure and dynamics: Implications for Aβ aggregation. Biophysical Journal. 101 (5), 1202-1211 (2011).
- Vugmeyster, L., et al. Fast motions of key methyl groups in amyloid-β fibrils. Biophysical Journal. 111 (10), 2135-2148 (2016).
- Yang, X., Wang, B., Hoop, C. L., Williams, J. K., Baum, J. NMR unveils an N-terminal interaction interface on acetylated-α-synuclein monomers for recruitment to fibrils. Proceedings of the National Academy of Sciences of the United States of America. 118 (18), (2021).
- Tuttle, M. D., et al. Solid-state NMR structure of a pathogenic fibril of full-length human α-synuclein. Nature Structural & Molecular Biology. 23 (5), 409-415 (2016).
- Karamanos, T. K., Kalverda, A. P., Thompson, G. S., Radford, S. E. Mechanisms of amyloid formation revealed by solution NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 88-89, 86-104 (2015).
- Lai, Y. -. C., Kuo, Y. -. H., Chiang, Y. -. W. Identifying protein conformational dynamics using spin-label ESR. Chemistry - An Asian Journal. 14 (22), 3981-3991 (2019).
- Franck, J. M., Han, S. Overhauser dynamic nuclear polarization for the study of hydration dynamics, explained. Methods in Enzymology. 615, 131-175 (2019).
- Pavlova, A., et al. Protein structural and surface water rearrangement constitute major events in the earliest aggregation stages of tau. Proceedings of the National Academy of Sciences of the United States of America. 113 (2), 127-136 (2016).
- Lin, Y., et al. Liquid-liquid phase separation of tau driven by hydrophobic interaction facilitates fibrillization of tau. bioRxiv. , (2020).
- Decatur, S. M. Elucidation of residue-level structure and dynamics of polypeptides via isotope-edited infrared spectroscopy. Accounts of Chemical Research. 39 (3), 169-175 (2006).
- Chatani, E., Tsuchisaka, Y., Masuda, Y., Water Tsenkova, R. molecular system dynamics associated with amyloidogenic nucleation as revealed by real time near infrared spectroscopy and aquaphotomics. PLoS One. 9 (7), 101997 (2014).
- Goret, G., Aoun, B., Pellegrini, E. MDANSE: An interactive analysis environment for molecular dynamics simulations. Journal of Chemical Information and Modeling. 57 (1), 1-5 (2017).
- Fujiwara, S., et al. Internal dynamics of a protein that forms the amyloid fibrils observed by neutron scattering. Journal of the Physical Society of Japan. 82, (2013).
- Schiró, G., et al. Neutron scattering reveals enhanced protein dynamics in concanavalin a amyloid fibrils. Journal of Physical Chemistry Letters. 3 (8), 992-996 (2012).
- Pounot, K., et al. Zinc determines dynamical properties and aggregation kinetics of human insulin. Biophysical Journal. 120 (5), 886-898 (2021).
- Fujiwara, S., et al. Dynamic properties of human α-synuclein related to propensity to amyloid fibril formation. Journal of Molecular Biology. 431 (17), 3229-3245 (2019).
- Sanz, A., et al. High-pressure cell for simultaneous dielectric and neutron spectroscopy. Review of Scientific Instruments. 89 (2), 023904 (2018).
- Adams, M. A., et al. Simultaneous neutron scattering and Raman scattering. Applied Spectroscopy. 63 (7), 727-732 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved