このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
カエノラブディティス・エレガンスの健康に影響を与える微生物分離物のハイスループットスクリーニング
要約
腸内微生物は、特定のまたは保存されたメカニズム を介して 宿主の健康にプラスまたはマイナスの影響を与える可能性があります。 カエノラブディティスエレガンスは 、そのような微生物をスクリーニングするための便利なプラットフォームです。本プロトコルは、線虫の健康状態の代理として使用される、線虫ストレス耐性への影響についての48の細菌分離株のハイスループットスクリーニングを記載している。
要約
Caenorhabditis elegansは、サイズが小さく、寿命が短く、遺伝学が容易なため、微生物分離株が宿主生理機能に与える影響を研究するための便利なプラットフォームを提供します。また、死ぬと青色に蛍光を発し、死を特定する便利な手段を提供します。この特性を利用して、ハイスループットラベルフリーのC.エレガンス生存アッセイ(LFASS)を開発しました。これらには、マルチウェルプレートにセットされた線虫集団のタイムラプス蛍光記録が含まれ、そこから集団死亡時間の中央値を導き出すことができます。本研究では、LFASSアプローチを採用して、複数の微生物分離株を一度にスクリーニングし、 C.エレガンス 重度の熱および酸化ストレスに対する感受性。このような微生物スクリーニングパイプラインは、特にプロバイオティクスをプレスクリーニングするために使用でき、宿主の健康の代理として重度のストレス耐性を使用することがここで報告されています。このプロトコルでは、C. elegans腸内細菌叢分離コレクションと同期線虫集団の両方をマルチウェルアレイで増殖させてから、アッセイのためにそれらを組み合わせる方法を説明しています。提供された例は、2つのストレスアッセイを並行して行う2つのワーム株に対する47の細菌分離株と1つの対照株のテストをカバーしています。ただし、アプローチパイプラインは容易に拡張可能であり、他の多くのモダリティのスクリーニングに適用できます。したがって、C.エレガンスの健康に影響を与える生物学的および生化学的条件のマルチパラメトリックランドスケープを迅速に調査するための汎用性の高いセットアップを提供します。
概要
人体には、主に腸、皮膚、粘膜環境に見られる推定10〜100兆個の生きた微生物細胞(細菌、古細菌)が生息しています1。健康な状態では、これらはビタミン産生、免疫系の成熟、病原体に対する自然免疫応答および適応免疫応答の刺激、脂肪代謝の調節、ストレス応答の調節など、宿主に利益をもたらし、成長と発達、疾患の発症、および老化に影響を与えます2,3,4,5.腸内細菌叢も生涯を通じてかなり進化します。最も劇的な進化は乳児期と幼児期に起こりますが6、ビフィズス菌の存在量の減少やクロストリジウム、ラクトバチルス、腸内細菌科、 エンテロコッカス種の増加など、年齢とともに大きな変化も起こります7。ライフスタイルは腸内微生物の組成をさらに変化させ、腸内細菌叢症(有益な細菌の喪失、日和見細菌の異常増殖)を引き起こし、炎症性腸疾患、糖尿病、肥満などのさまざまな病状を引き起こす可能性があります5だけでなく、アルツハイマー病やパーキンソン病にも寄与します8,9,10,11。
この認識は、腸生理学(現在はその中の微生物を含む)と神経系の間の相互作用が動物の代謝と生理学的機能の主要な調節因子と考えられている腸脳軸(GBA)の概念の洗練に大きく貢献しています12。しかし、腸脳シグナル伝達における微生物叢の正確な役割とそれに関連する作用機序は完全には理解されていません13。腸内細菌叢が健康的な老化の重要な決定要因であるため、細菌が老化プロセスをどのように調節するかは、激しい研究と論争の対象となっています6,14,15。
回虫Caenorhabditis elegansが、他の種と同様に、バクテロイデス、ファーミキューテス、および放線菌によって優勢な正真正銘の腸内細菌叢を宿主としていることの実証により16,17,18,19,20、宿主と腸の共生相互作用を研究するための実験プラットフォームとしての急速な台頭21,22,23,24、25,26は、調査兵器26,27,28,29を大幅に拡大しました。特に、C.エレガンスが遺伝子-食事、遺伝子-薬物、遺伝子-病原体などの相互作用を研究するために利用できるハイスループット実験アプローチを適応させて、細菌分離株とカクテルがC.エレガンスの健康と老化にどのように影響するかを迅速に調査することができます。
本プロトコルは、健康の代理として C.エレガンスの ストレス耐性への影響についてマルチウェルプレートにセットされた細菌分離株または混合物のアレイを一度にスクリーニングするための実験パイプラインを説明し、プロバイオティクスを同定するために使用することができる。蛍光プレートリーダーを使用した自動ストレス耐性分析のためにワームを処理する前に、大規模なワーム集団を増殖させ、96ウェルおよび384ウェルプレートフォーマットで細菌アレイを処理する方法について詳しく説明します(図1)。このアプローチは、死に至る線虫が死の時間を特定するために使用できる青色蛍光のバーストを生成する死の蛍光31 の現象を利用するラベルフリー自動生存アッセイ(LFASS)30に基づいています。青色蛍光は、 C.エレガンス 腸顆粒(リソソーム関連オルガネラの一種)に貯蔵されたアントラニル酸のグルコシルエステルによって放出され、死亡時に線虫腸内で壊死カスケードがトリガーされると破裂します31。
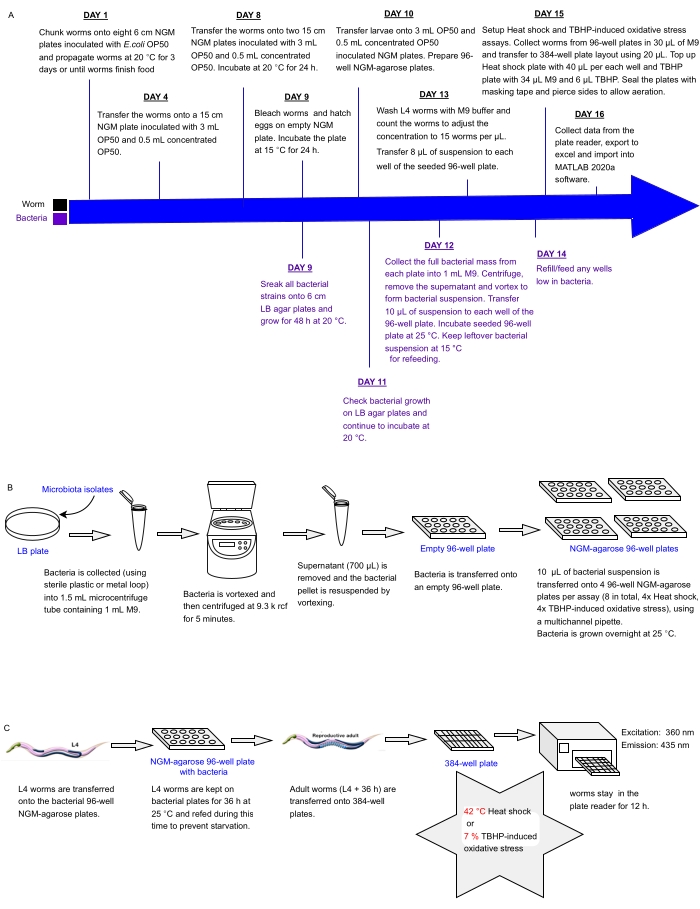
図1: ストレスに対するC.エレガンスの 耐性に影響を与える細菌分離株のハイスループットスクリーニングのための実験ワークフロー 。 (A)ワームとバクテリアのメンテナンスとアッセイのセットアップのタイムライン。(B)96ウェル細菌プレートアレイのセットアップと取り扱い。(C)384ウェルウォームプレートのセットアップ。 この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
本研究で並行して使用された2つの C.エレガンス 株は、ブリストルN2野生型とHT1890: daf-16(mgDf50)であり、これらは同様の速度で成長します。しかしながら、プロトコルは、類似した増殖速度を有する2つの株の任意の組み合わせで複製することができる。他の株(例えば、野生型と成長の遅い daf-2 変異体)を並行して試験する場合、異なる増殖速度を考慮する必要があり、それに応じてプロトコルを調整する必要があることに注意してください。以下のプロトコルのワームとバクテリアのタイムスケールと量は、テトラプリケートの2つのLFASSアッセイで2つのワーム株で48の細菌分離株を並行してテストするために最適化されています。より多くの条件を並行してテストする場合は、調整が必要になります。 大腸菌 OP50細菌株は、ミネソタ大学のカエノラブディティス遺伝学センター(CGC)から入手した。48の細菌分離株は、Schulenburgラボから取得され、LB寒天培地で維持されました。
1. OP50で培養する C.エレガンス (1日目から8日目)
注:現在のアプローチは、すべての段階で固体培地上でC.エレガンス雌雄同体を成長させることを目的としており、不必要な食事の変更を回避します(つまり、NA22などの代替のより急速に成長する大腸菌株または卵板などのより豊富な増殖培地を使用)まだ広く使用されている標準的な成長条件32,33に可能な限り近いままにします。線虫の成長温度(ここでは15°Cに設定)は、使用するC.エレガンス株に依存し、調整が必要な場合があります(たとえば、温度感受性表現型またはバイオマーカーの発現を回避またはトリガーするため)。ワーム飼育については、参考文献33を参照してください。
- 直径6cmのNGMプレート(線虫増殖培地寒天培地10mL、NGM、補足ファイル1)32,33を8枚用意し、室温で1日間乾燥させます。
- 50 mLコニカルチューブ内の25 mLのOP50培地(補足ファイル1)に、新たに成長したLysogeny Broth寒天培地(LB寒天培地、補足ファイル1)プレートから単一の細菌クローンを播種することにより、大腸菌OP50細菌の飽和液体培養を準備します。培養物をシェーカーインキュベーター内で37°Cで一晩増殖させる。
- 1株あたり8枚の6 cm NGMプレートに、プレートあたり100 μLの大腸 菌 OP50の飽和液体培養液を接種し、プレートを20°Cに2日間保ってから使用します。
- メスを使用して、最近飢餓したNGMプレートからワームを含む0.5 cmの正方形の寒天チャンクを切り取り、接種した8つの6 cm NGMプレートのそれぞれに移し、これらのプレートを20°Cで3日間(またはワームが餌を食べ終わるまで)インキュベートします。
- ワーム株ごとに5つの15 cm NGMプレートを準備し(プレートあたり30 mLのNGM培地)、3 mLのOP50を接種します。プレートを乾燥させてから、37°Cで一晩インキュベートします。後のステップで使用するまで、プレートを20°Cに保ちます。
- P-1000ピペットを使用して、最大3 mLの滅菌M9バッファー(補足ファイル1)を6 cm NGMプレート(ステップ1.1)に加えてワームを再懸濁し、1つの15 mLコニカルチューブで1株ごとに8つのプレートすべてからワーム溶液を回収します。
- 142 x g で 4 °C で 2 分間遠心分離します。 P-5000ピペットまたは滅菌パスツールピペットまたはチップを備えたウォーターポンプを使用して、上清を慎重に除去します。10 mLの滅菌M9バッファーを加えて、ワームペレットを洗浄します。2回繰り返します。
- 上清を(可能な限り)除去し、ピペットを使用してワームを15 cm OP50接種NGMプレートに移します(ステップ1.5)。0.5 mLの濃縮OP50培養液を追加します。
- 濃縮OP50を作るには、LBの1 Lボトル4本にそれぞれ2 mLのOP50スターター培養液(ステップ1.2で調製)を接種し、振とうインキュベーター内で37°C、160 x gで6時間増殖させます。細菌を3057 x g 、20°Cで15分間ペレット化します。上清を廃棄し、細菌ペレットを6 mLのOP50培地で再懸濁し、滅菌済みの50 mLコニカルチューブに回収します。
注:細菌は4°Cで最大1週間保存できます。
- 直径15 cmのNGMプレート上で、毎日0.5 mLの濃縮OP50をワームに再給餌することにより、15°Cで3〜4日間、各ワーム株を増殖させます。
- ワームが餌をほぼ食べ終わったら、M9バッファーで集めて洗浄し(ステップ1.6.1)、各ワーム株培養物を2つの15 cm NGMプレートに移し(ステップ1.5)、個体数の~95%が妊娠成虫になるまで20°Cでワームを繁殖させます(野生型ブリストルN2の場合は約24時間かかります)。
注:妊娠した成虫は、ワーム内に卵が存在することを特徴とし、理想的なプレートには、幼虫が多すぎずにプレート上に大量の孵化していない卵が置かれている必要があります33。
- ワームが餌をほぼ食べ終わったら、M9バッファーで集めて洗浄し(ステップ1.6.1)、各ワーム株培養物を2つの15 cm NGMプレートに移し(ステップ1.5)、個体数の~95%が妊娠成虫になるまで20°Cでワームを繁殖させます(野生型ブリストルN2の場合は約24時間かかります)。
2.腸内細菌叢分離コレクションの維持(9日目)
- 48個の細菌分離株を個々の6 cm LB寒天プレートにストリークし、20°Cで48時間増殖させます。
注:細菌は、必要に応じて25°Cで24〜36時間増殖させることができますが、20°Cの増殖が長いほど、潜在的な汚染物質を見つけることができます。 - 多数のC.エレガンスを同期させます。
- 標準的な卵の準備方法33 に従って妊娠した成虫を漂白し、卵を播種していない2つの15 cm NGMプレートに15°Cで24時間移して、すべてのL1幼虫が後続のステップで孵化して同期的に成長できるようにします。
注意: 漂白剤溶液を取り扱うときは注意してください。
- 標準的な卵の準備方法33 に従って妊娠した成虫を漂白し、卵を播種していない2つの15 cm NGMプレートに15°Cで24時間移して、すべてのL1幼虫が後続のステップで孵化して同期的に成長できるようにします。
3.大規模なC.エレガンス培養の成長(10日目)
- 孵化したら、L1幼虫(ステップ2.2.1から)をきれいな円錐形の15 mLチューブに3〜4 mLのM9で集めます。10 μLのワーム溶液4滴をスライドまたはプレートにピペットで移し、実体顕微鏡下で16倍の倍率で各滴中のワームの数をカウントします。ワーム溶液のすべての滴からの幼虫の数を平均することによって、溶液のワーム濃度を決定する。この値に残りの量を掛けて、各菌株の合計ワーム数を推定します。
注:この段階では、後で384ウェルプレートまたは2つのハーフプレートを満たすために、株あたり46,000〜50,000 L1幼虫が必要です。- 各菌株について、すべてのL1幼虫を2つの15 cm NGMプレート(プレートあたり23,000〜25,000 L1)に移し、3 mLのOP50を事前に接種し(ステップ1.5)、0.5 mLの濃縮OP50を再播種します。
- 15°Cでインキュベートし、ワームがL4段階に達するまで、必要に応じて毎日0.5 mLの濃縮OP50を補充します。
注:L4ステージは、わずかに暗い腸と、外陰部が最終的に32,33を形成する半円盤または三日月形の白いパッチが特徴です。 - 以下の手順に従って96ウェルNGM-アガロースプレートを準備します。
- 各ウェルに125 μLのNGMアガロースを充填することにより、8枚の96ウェルNGM-アガロースプレートを調製します(アッセイごとに4枚のプレート)。
注意: 後続の手順で一部が汚染された場合は、いくつかの追加のプレートをメッキすることをお勧めします。アッセイを並行して実行するには2つのプレートリーダーが必要ですが、わずか6時間で実行できるため、熱ストレスアッセイから連続して実行することもできます。これらのプレートでは、<4%の灰寒天がアガロースで置換されており( 材料の表を参照)、NGMプラグ全体でよりゆっくりと均一な乾燥を可能にし、ワームの穴掘りを減らしてより良い回収を可能にします。 - ウェルが均等に満たされ、気泡がないことを確認してください。プロセス中に混合物が固まらないように、70°Cに設定されたヒートブロックを使用します(マルチウェルプレートのプラスチックを介した熱伝達が遅いため、NGM-アガロースは約55〜60°Cまでしか加熱できません)。ウェル内の気泡を除去するには、滅菌火炎加熱針を使用します。
- 96ウェルプレートを無菌環境で室温にセットしてから反転させ(結露を防ぐために蓋を下ろします)、必要になるまでクリーンボックスに4°Cで保管します。
- 各ウェルに125 μLのNGMアガロースを充填することにより、8枚の96ウェルNGM-アガロースプレートを調製します(アッセイごとに4枚のプレート)。
- 11日目に、ステップ3.2のワームをチェックし、汚染が発生しておらず、ワームがまだいっぱいであることを確認します。
- 12日目に、ステップ3.1のワームをチェックし、汚染が発生しておらず、ワームがまだいっぱいであることを確認します。また、ワームの発生段階を確認してください。
注:使用されるL4またはL4 + 24時間のワームなど、ワームの性別/系統および発生段階は、ワームが受ける治療によって異なります。ここでは、野生型の雌雄同体をL4からの細菌分離株に36時間曝露した。
4.ワームを再給餌するための腸内細菌叢分離コレクションの準備
- ステップ2.1からLB寒天プレート上の細菌増殖を監視します。20°Cでインキュベートを続けます。
注:理想的ではありませんが、一部のクローンが成長しない、または汚染を明らかにしない場合は、細菌をクリーンストックから6 cm LBプレートに再ストリークし、25〜28°Cで24時間成長させて実験の準備をすることができます。 - テスト対象の細菌コレクションの96ウェルアレイレイアウトを定義し、後続のステップでの体系的なプレート播種とデータ分析を容易にします(補足表1)。
- 各6 cm細菌プレートから細菌塊を収集し(ステップ4.1)、1 mLのM9バッファーを含む標識された1.5 mLマイクロ遠心チューブに移します。これは、直径2mmの使い捨て滅菌プラスチックループまたは直径5mmの金属ループを使用して実行します。細菌株間の金属ループを100%エタノールに浸し、炎上させ、5秒間冷却して滅菌します。
- 細菌ペレットが完全に再懸濁されるまで、マイクロ遠心チューブをボルテックスします(細菌株によっては、~1〜10秒かかる場合があります)。
- 室温で9,300 x g で5分間スピンダウンし、700 μLの上清を除去し、ボルテックスによって細菌ペレットを再懸濁します。
- 各細菌懸濁液200 μLを、ステップ4.2に記載されているレイアウトに従って、空の滅菌96ウェルプレートの単一ウェルに移します。
- このプレートから、マルチチャンネルピペットを使用して8枚の96ウェルNGM-アガロースプレート(ステップ3.3で調製)に10 μLの細菌溶液を接種し、蓋をした状態で25°Cで24時間インキュベートします。プレートの乾燥と細菌の好気性増殖を可能にし、過剰な結露を避けるために、プレートを密封しないでください。
- ステップ4.6で準備した96ウェルサスペンションプレートを密封します。きれいな粘着シーリングフィルム( 材料の表を参照)を使用し、15°Cで最大5日間保管します。これは、必要に応じてワームの再給餌に使用されます。
5. LFASSヒートショックと酸化アッセイのセットアップ(13〜14日目)
- ステップ3.5のプレートを見て、ワームの発生段階を評価します。ワームの>90%がL434に達したら、15 mLコニカルチューブ内の最大10 mLの滅菌M9溶液にワームを収集します。
- 142 x g で4°Cで2分間スピンダウンし、上清を取り除き、各洗浄の間に10 mLの新鮮な滅菌M9を加えてOP50細菌を取り除くことにより、ワームを広範囲に(少なくとも4x)洗浄します。ワームペレットを10 mLのM9に再懸濁します。
- 50 μLのワーム溶液を、950 μLのM9を含む低表面結合チューブ( 材料の表を参照)に移します。ワームの沈降を避けるためにチューブの内容物を穏やかに混合した後、濡れた低結合ピペットチップを使用して、スライドガラスまたはNGMプレートに3〜4個の別々の10 μL滴を移し、実体顕微鏡( 材料の表を参照)でワーム数を16倍の倍率でカウントします。3〜4滴のカウントを平均し、ワーム溶液中のマイクロリットルあたりのワームの数を決定します(ステップ3.1を参照)。
- 10 mLチューブ内のワーム濃度を調整して、8 μLで~120ワームに到達します。ステップ5.2で調製した溶液の場合。十分に濃縮されていない場合は、ワームをスピンダウンし、それに応じてM9を除去して、8μLあたり120ワームに到達します。
- マルチチャンネルピペットまたはリピートピペットを使用して、ステップ4.7の8つの96ウェルNGMアガロースプレートの各ウェルに8 μLのワーム溶液(~120ワーム)を移します。ワームの損失を制限するために、保持期間の少ないヒントを使用してください。また、大きな成虫が成虫への機械的ストレスを制限できるように、先端を切る必要があるかもしれません。
注:このアッセイでは、確実に機能するために最低30匹の生きた健康なワームが必要ですが、ウェルあたり約100匹のワームで最適に機能します。 - ワームと細菌を播種した96ウェルNGMアガロースプレートを25°Cで36時間インキュベートします。
- 12〜24時間の間にプレートをチェックし、ワームが全体を通していっぱいのままであることを確認します。再給餌が必要な場合は、ステップ4.8で15°Cで保存した96ウェル細菌アレイプレート内に細菌を再懸濁し、36時間のインキュベーション期間が終了する前に、ワームが飢餓のリスクがある96ウェルNGM-アガロースプレートに対応する細菌溶液を最大10 μL追加します(飢餓状態のワームは大きく異なる結果をもたらします。 したがって、これは非常に重要です)。
注:次の手順は15日目に実行する必要があります。アッセイを開始する前に、読み取り高さを最適化する必要がある場合があります。最適な読み取り値は、ウェルの底から20〜50μm上で達成されます。これは、プレートリーダーのモデルによって異なります。Z-スキャンが可能なものもあれば、手動で高さを入力できるものもあります。最高の青色蛍光(365 nm/430 nm)シグナルが検出されるレベルで最適な高さを設定します。プレートリーダーの中には、接着細胞アッセイ用に最適化された固定高さで動作するものがあり、LFASSアッセイには理想的ではない場合があります。 - 36時間後、30 μLのM9を96ウェルプレートの各ウェルに分注します。
注意: 熱ストレスアッセイの場合、プレートリーダーはアッセイを実行するために必要な温度に達している必要があり、事前にオンにする必要があります。現在のプロトコルでは、殺傷速度を最大化するために42°Cを使用していますが、このアプローチは30°Cを超える他の温度にも適用されます。 - 低保持チップを使用して、設定されたレイアウトに従ってワーム(約20 μL)を384ウェルプレートに移します(大きなワームが成虫の機械的ストレスを軽減できるように、チップの端を切り取ることを検討してください)。
注:本研究では、ここで説明する2つのアッセイ(熱ストレスと酸化ストレス)に2つの異なるプレートリーダー設定が使用されるため、これら2つのアッセイを対象としたサンプルを同じ384ウェルプレートにプレーティングしないでください。 - プレートリーダーが正しくセットアップされていることを確認します(表1)。
- 384ウェルプレートにさらにM9を補充し、ウェルあたり60 μLの最終容量を目指します。熱ストレスアッセイの場合は40 μLのM9を加え、t-BHP誘発酸化ストレスの場合は6 μlのt-BHPに34 μlのM9を加えます(材料の表を参照)。
- t-BHPを追加してから2分以内にアッセイを開始します(理想的には、すべてのワームを同時にt-BHPに曝露する必要があり、アッセイ時間の分解能は2分です)。不可能な場合は、タイマーを使用してアッセイ開始前にt-BHPをピペッティングするのに費やした時間を推定し、後で死亡時間の中央値を調整できるようにします。
- 透明な蓋でプレートを閉じます。384ウェルプレートの端をマスキングテープ(プレートと蓋の上にテープで貼る)で密封し、テープが蓋の上やプレートの下に入らないようにします。メスを使用して蓋とプレートの間にテープを間隔を置いてスリットし、アッセイ中の蒸発を最小限に抑えながら空気交換を可能にします。
- プレートをプレートリーダーに挿入し( 材料表を参照)、実行を開始します。365 nmで励起し、435 nmで2分ごとに6〜12時間発光を検出することを目指します(表1)。
注:通常、42°Cの熱ストレスアッセイには6時間、7%t-BHP酸化ストレスアッセイには8時間で十分です。
6. プレートリーダーのデータ処理
- プレートリーダーからの生の蛍光データをカンマまたはタブ区切りの.txt、.csv、または.xls/.xlsx形式で保存し、xls /.xlsx形式に変換します。データ形式に応じて、LFASS分析に必要なExcelシートレイアウトに合わせて再編成します。参考文献30に記載されている詳細な指示に従ってください。
注:データは手動で分析し、各時系列を正規化し、死亡蛍光が半極大に達する時間を探すことができますが、LFASSルーチン30を実行するMatlabで自動分析を実行できます。 - https://github.com/ABA80/LFASS からMatlab(バージョン2014a以上)とLFASSソフトウェアパッケージをダウンロードしてインストールします。その中にあるガイドラインと注釈に従ってください。
メモ: 図 1C に、このアプローチの簡単な説明を示します。LFASSルーチンを実行するには、Matlabが必要です。あるいは、Matlabコードは、独自のフィッティング関数を除いて、Oracleに変換することもできます。新しいスムージング関数とシグモイド関数を書き直して、完全にオープンソースのプラットフォームで使用できるようにすることができます。 - LFASS解析ではデータフォルダ内のすべてのファイルが処理され、結果フォルダ内のファイルが上書きされるため、LFASS解析と結果を新しい場所に移動します。
7. データ検査
- Excelファイルを開き、384ウェルプレート上のウェル位置に従って行にラベルを付けます。 補足ファイル2 は、ヒートショックアッセイ用に生成された生の蛍光データのExcelファイルの例を示す。384ウェルプレートのウェル位置を使用して、ワームおよび細菌株を標識します。
- Matlab分析の前に、Excelでデータを視覚的に検査し、代表的なウェルの蛍光強度を経時的にプロットします。使用するプレートリーダーによっては、データにノイズが多い場合がありますが、明確なピークが表示されるはずです。特に:
- ピークがノイズと有意に異ならない蛍光値を決定します(LFASSでこのようなしきい値を設定すると、空のウェルを除外して分析が高速化されます)。
- 上昇する前に蛍光の変動が減衰する最も早い時点に注意してください(動物は最大30分間激しく叩きつけ、青色の蛍光測定値が急速に変動する可能性があります)。
注: ピークフィットは、カーブフィットウィンドウからこれらの初期の時点を除外することで改善される場合があります。 - カーブフィッティングに使用されるため、最小蛍光値と最大蛍光値が下がると予想される時点に注意してください(これらの範囲を特定するためにいくつかのウェルを見てください)。
- 蛍光ピークの振幅がウェル間で有意に異なるかどうかを確認し、次の式を使用してさらに分析する前にデータを正規化します。
正規化蛍光ウェルn(t)=(蛍光ウェルn[t]-最小蛍光ウェル[Dt])/(最大蛍光ウェル[Dt]-最小蛍光ウェル[Dt])
ここで、「n」は現在のウェル番号、「t」は時点、「Dt」はアッセイの一連の時点です。
8. LFASSデータ処理
注:詳細は、参考文献30の https://github.com/ABA80/LFASS および補足資料に記載されています。
- LFASS フォルダー内に 2 つのサブフォルダーを作成し、1 つは分析するデータ用、もう 1 つは結果 ("マイ データ" や "結果" など) 用です。
- データ検査後、アッセイエクセルデータファイルをLFASSサブフォルダー「マイデータ」にコピーします。
- MATLABを起動し、LFASSフォルダーに移動し、コマンドウィンドウ(補足ファイル3)にfitfolderと入力して実行します。次に、画面の指示に従います。
- 「fitfolder」と入力すると、システムはExcelファイルが配置されているフォルダーの名前(「マイデータ」など)を尋ねられます。データ フォルダーの名前を入力します (この例では "マイ データ")。
- 画面の指示に従って、要求されたさまざまなパラメーターを入力します。
- 現在のプロトコルで連続する測定間の時間間隔として「2」を入力します(これを指定すると、結果をタイムポイント単位ではなく分で表すことができます)。
注:時間間隔は、蛍光測定を多かれ少なかれ頻繁に実行するように変更でき(時間分解能を増減するため)、プレートリーダーの機能に応じて(つまり、十分な速度で測定を実行できないプレートリーダーの場合は時間間隔を増やす必要がある場合があります)。実験時間間隔を LFASS ルーチンと常に一致させてください。 - 「0.95」と入力して上側公差しきい値を割り当て(これはフィットを改善するために必要に応じて変更できます)、下側許容しきい値に「0.05」と入力して割り当て(これはフィットを改善するために必要に応じて変更できます)、シグモイドフィットを拘束します。
注: その他の時間パラメータは、データ インスペクションからのユーザ メモに基づいています (ステップ 7.2)。
- 現在のプロトコルで連続する測定間の時間間隔として「2」を入力します(これを指定すると、結果をタイムポイント単位ではなく分で表すことができます)。
- フィットおよびスムージングされたカーブを表示するかどうかを選択するには、プロンプトが表示されたら「はい」に「y」、NOに「n」と入力します。収束適合を視覚的に検査するには、前者を選択します。
注: 後者は、平滑化されたすべてのデータを視覚化するのに便利ですが、生成されるポップアップ グラフが多すぎるため、通常は選択されません。これに続いて、MatlabはLFASSルーチンを実行しますが、複数のExcelファイルが一度に処理されている場合は1〜10分かかる場合があります。曲線のあるポップアップウィンドウは、ステップ8.6の選択に従って表示されます。 補足ファイル4A は、フィット曲線の例を示しています。 - (1)ノイズとして識別されたカーブを解析するか、(2)[y/n]オプションを使用して適合不良のカーブを再修正するかを選択します。承認するには「 y 」、拒否するには 「n 」と入力します。
注記: 特に、適合不良または不適合のカーブが多数ある場合は、再フィットを承認することをお勧めします。これにより、ユーザーは画面に表示される各カーブに合わせたカーブフィットパラメータを提供し、シグモイドフィットの前後の境界のみを要求できます。必要な回数だけ試行できます。 - データが分析されたら、Matlabを閉じて LFASS フォルダーを開きます。
- LFASSサブフォルダをクリックします 私の結果、結果ファイルは.txtとして 結果フォルダに自動的に保存されます。
注: Matlab は、"バッチ適合.txt"、"バッチおよびノイズ適合.txt"、および "再適合.txt" の 3 つの.txtファイルを生成します。前者の2つは、再調整中にコンピューターがクラッシュしたりユーザーエラーが発生したりした場合の予防措置として保存されます。最も正確な完全な分析を含むファイルは「再適合.txt」です。 - Microsoft Excelでファイル再 調整.txt を開き、さらに処理するために.xlsとして保存します。 補足ファイル4B は、このような結果ファイルの例を示しています。
注:各ウェル(行に編成)について、ワーム集団の死亡時間の中央値の推定値を示す3つの値が列に提供されます。 「生」:実験データピークの半極大で交差する時間を報告します。「バッチフィット」: バッチフィット曲線の半値で交差する時間を報告します。「再フィット」: 再フィット曲線の半値で交差する時間を報告します。 - ファイルを.xls形式でコピーとして安全な場所に保存します。これを行わないと、LFASS ルーチンの次の実行時にファイルが上書きされるリスクがあります。
注:その後、結果はグラフ化または統計分析のためにさらに処理できます。
結果
LFASSアッセイは、ストレス耐性や老化に寄与する多数の遺伝的および微生物叢パラメータのスクリーニングなど、複数の試験条件の堅牢でハイスループットかつ迅速なスクリーニングを一度に提供します。実験が複数のテスト条件の広範なデータセットを取得するのに2〜3週間しかかかりません。L4 + 36時間の成体野生型ワーム集団は、48腸内微生物分離株で36時間培養した後、42°Cの熱ストレ?...
ディスカッション
C. elegansは、その小型、透明性、迅速な開発、短寿命、安価、取り扱いの容易さにより、一度に複数の実験パラメータを迅速にスクリーニングするための多くの利点を提供します。そのかなり単純なゲノム、ボディプラン、神経系、腸、およびマイクロバイオームでありながら、複雑で人間と十分に類似しているため、生物活性の有効性または毒性をテストしながらメカニズムの洞察を...
開示事項
著者は開示するものは何もありません。
謝辞
我々は、寄生虫株を提供してくれたCGCミネソタ州(米国マディソン、NIH - P40 OD010440)と、ここに描かれているすべての環境微生物分離株を提供してくれたOP50およびPr. Hinrich Schulenburg(CAU、キール、ドイツ)に感謝する。この研究は、ABへのUKRI-BBSRC助成金(BB / S017127 / 1)によって資金提供されました。JMは、ランカスター大学FHM博士号奨学金によって資金提供されています。
資料
| Name | Company | Catalog Number | Comments |
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
参考文献
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood - Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977 (2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer's disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98 (2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson's disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921 (2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095 (2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323 (2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364 (2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545 (2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849 (2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998 (2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613 (2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer's disease: a systematic review. Nutrients. 14 (1), 20 (2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson's disease. Ageing Research Reviews. 69, 101347 (2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -. E. H., Jung, Y., Lee, S. -. J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190 (2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652 (2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583 (2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398 (2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448 (2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved