このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
スフィンゴシン1-リン酸受容体の構造とシグナル伝達経路を調べるパイプライン
要約
S1Pは、S1P受容体(S1PR)サブファミリーを介して多様な生理効果を発揮する。ここでは、S1PR の構造と機能を説明するためのパイプラインについて説明します。
要約
リゾリン脂質(LPL)は、スフィンゴシン1-リン酸(S1P)、リゾホスファチジン酸などを含む生理活性脂質である。細胞膜におけるスフィンゴ脂質の代謝産物であるS1Pは、スフィンゴシン1-リン酸受容体(S1PR)によって媒介されるシグナル伝達経路 を介して 様々な細胞生理学的応答を調節する最も特徴的なLPLの1つである。これは、S1P-S1PRsシグナル伝達系が、多発性硬化症(MS)、自己免疫障害、癌、炎症、さらにはCOVID-19を含む障害の顕著な潜在的な治療標的であることを示唆した。クラスAのGタンパク質共役型受容体(GPCR)ファミリーの小さなサブセットであるS1PRは、S1PR1、S1PR2、S1PR3、S1PR4、およびS1PR5の5つのサブタイプで構成されています。しかし、詳細な構造情報の欠如は、S1PRを標的とする創薬を妨げる。ここでは、S1P-S1PRs複合体の構造を解明するためにクライオ電子顕微鏡法を適用し、細胞ベースの機能アッセイを用いて、活性化、選択的薬物認識、Gタンパク質結合のメカニズムを解明しました。他のリゾリン脂質受容体(LPLR)およびGPCRも、この戦略を用いて研究することができる。
概要
細胞膜におけるスフィンゴ脂質の代謝産物であるスフィンゴシン-1-リン酸(S1P)は、リンパ球輸送、血管発達、内皮完全性、および心拍数1,2,3を含む様々な生物学的活性を含むユビキタスなリゾホスファチド性シグナル伝達分子である。S1Pは、5つのS1P受容体サブタイプ(S1PRs1〜5)を介して多様な生理学的効果を発揮する。S1PRは様々な組織に見られ、下流のGタンパク質に対して独特の嗜好を示す4,5。S1PR1は主にGiタンパク質と結合し、その後cAMP産生を阻害する。S1PR2 および S1PR3 は Gi、Gq、および G12/13 と結合され、S1PR4 および S1PR5 は Gi および G12/13 を介して信号を伝送します。
S1P-S1PRシグナル伝達は、自己免疫疾患7、炎症8、癌9、さらにはCOVID-1910を含む複数の疾患の重要な治療標的である。2010年、フィンゴリモド(FTY720)は、再発性多発性硬化症(MS)を治療するためのS1PRを標的とするクラス初の薬物として認可されました11。しかしながら、S1PR2を除く全てのS1PRsに結合することができるが、S1PR3への非特異的結合は、大脳皮質の浮腫、血管および気管支狭窄、ならびに肺上皮漏出をもたらす12。治療選択性を高めるための代替戦略として、受容体に対するサブタイプ特異的リガンドが産生されている。シポニモド(BAF312)は、再発MS治療のために2019年に承認されました13;S1PR1およびS1PR5を効果的に標的とするが、S1PR3に対する親和性はなく、臨床現場でより少ない副作用を示す14。2020年、米国食品医薬品局(FDA)はオザニモドをMS治療に承認しました15。オザニモドは、S1PR516よりもS1PR1に対して25倍大きい選択性を保持することが報告されている。特に、現在のCOVID-19パンデミックの文脈では、S1PRを標的とするアゴニスト薬が、免疫調節療法技術を使用してCOVID-19を治療するために利用され得ることが発見されている17。フィンゴリモドと比較して、オザニモドはCOVID-19患者の症状を軽減する上で優位性を示しており、現在臨床試験を受けています10。S1PRの構造的基礎と機能を理解することは、S1PRsを選択的に標的とする薬剤を開発するための重要な基盤を築く18。
生体高分子の構造情報を調べるには、X線結晶構造解析、核磁気共鳴(NMR)、電子顕微鏡(EM)など、多くの技術が用いられています。2022年3月現在、タンパク質データバンク(PDB)には18万以上の構造が堆積しており、そのほとんどはX線結晶構造解析によって解明されています。しかし、2013年にYifan ChengとDavid Juliusによって報告されたTPRV1の最初の原子分解能に近い分解能構造(3.4 Å分解能)19では、クライオ電子顕微鏡(cryo-EM)がタンパク質構造の主流技術となり、EM PDB構造の総数は10,000を超えました。重要なブレークスルー分野は、直接電子検出カメラとして知られるイメージング用の新しいカメラと新しい画像処理アルゴリズムの開発です。Cryo-EMは、過去10年間で構造生物学と構造ベースの創薬に革命をもたらしました20。高分子複合体が生細胞内で複雑な役割を果たす方法を理解することは、生物科学の中心的なテーマであるため、クライオEMは、特に膜貫通タンパク質21について、動的分子複合体の立体構造を明らかにする可能性を秘めています。Gタンパク質共役型受容体(GPCR)は、膜貫通タンパク質の最大のスーパーファミリーであり、現在市販されている医薬品の30%以上の標的である22。クライオEMの開発は、GPCR-Gタンパク質複合体の高分解能構造のバーストに貢献し、薬物設計におけるX線結晶学的解析にはまだアクセスできない「難治性」標的の構造決定を可能にした23。したがって、クライオEMアプリケーションは、原子分解能24に近いネイティブに近い条件下でGPCRの3次元構造を決定する機会を提供します。クライオEMの進歩により、GPCRの刺激または阻害の機構的基盤を視覚化することが可能になり、GPCR標的薬作成のための新規結合部位の解明にさらなる利益をもたらす25。
クライオEM技術の驚異的な進歩に依拠して、我々は最近、苦悶のS1PR1-、S1PR3-、およびS1PR5-Giシグナル伝達複合体の構造を特定した26,27。ヒトでは、S1PRは様々な組織に見出され、下流のGタンパク質に対して独特の嗜好を示す4,5。S1PR1は主にGiタンパク質と結合し、その後、3',5'-環状アデノシン一リン酸(cAMP)産生を阻害する。S1PR3およびS1PR5は、Gi 6,28との結合も可能である。Gi共役型受容体活性化はcAMP29の産生を減少させるので、機能的変化を捕捉するためのcAMP阻害効果を測定するためにGi阻害cAMPアッセイが導入された26、27。cAMP結合タンパク質部分が挿入されたPhotinus pyralis lucifereraseの変異バージョンを使用して、このcAMPアッセイは、細胞内cAMP濃度の変化を介してGPCR活性をモニタリングするための簡単で信頼性の高い方法を提供する30。これは、高感度で非放射性の機能アッセイであり、創薬目的で広範囲のGPCRのリアルタイムの下流シグナル伝達をモニターするために適用することができる31。
ここでは、S1PRの活性化モードと薬物認識モードを解決する上で重要な方法の概要が提供され、主にクライオ-EM操作とGi阻害cAMPアッセイが含まれます。本稿は、GPCRの構造と機能をさらに探求するための包括的な実験的ガイダンスを提供することを目的としている。
プロトコル
1. S1PRs-Gタンパク質複合体の精製
- ヒトS1PRs-Gタンパク質複合体を精製するには、C末端残基338~382を欠くS1PR1のcDNA、C末端に345~398で切り詰めた野生型S1PR3、S1PR5、および野生型Gi1をpFastBac1ベクターに、野生型Gβ1およびGγ2のcDNAをpFastBacdualベクターにクローニングする(材料表)。
注:S1PRのすべての構成物には、ヘマググルチニン(HA)シグナル配列とそれに続くN末端のFlagエピトープタグとC末端の10倍のhisタグも含まれています。また、受容体の発現および精製を容易にするために、T4リゾチーム(T4L)を翻訳するための合成DNA配列(材料表)をS1PRのN末端に挿入した。 - S1PR、Gi1、およびGβ1γ2をコードするバキュロウイルスの調製
- -80°Cで1.5 mLチューブに保存したDH5αコンピテント大 腸菌(大腸菌) 50 μLに組換えベクターを添加し、氷上で30分間インキュベートした。
- 細胞を42°Cで90秒間ヒートショックし、すぐに氷に移し、2分間冷やします。
- 300 μLのライジェニーブロス(LB)培地を添加した後、チューブを37°Cで3〜5時間振盪する。細胞のプレート100 μLをLB寒天プレートに、48時間暗所に保ちながら37°Cでインキュベートした。
- 50 μg/mLのカナマイシン、10 μg/mLのテトラサイクリン、7 μg/mLのゲンタマイシンを含む5 mLのLB培地に白色コロニーを接種し、37°Cで16時間培養した。
- P0バキュロウイルスを製造するためのプラスミドミニプレップキット(材料表)で組換えバクミドを単離し、製造業者の指示に従ってください。
注:使用前に、精製バクミドをpUC/M13フォワードプライマーおよびリバースプライマーによるPCRによって分析しました。PCRの場合、サイクル数=30サイクル、融解温度=58°C、伸長時間=1分/1Kbとした。 - 以前のプロトコル32に記載されるようにP0バキュロウイルスを準備する。
- sf9細胞(ESF921培地)を6ウェルプレートで培養し、細胞が対数期(1.0-1.5 x106細胞/mL)にあることを確認します。
- 8 μL のバキュロウイルストランスフェクション試薬を 100 μL のグレースの培地に希釈し、室温で 30 分間インキュベートします。組換えバクミド10 μgをグレースの培地100 μLで希釈し、穏やかに混合する。希釈したバクミドを希釈したバキュロウイルストランスフェクション試薬と組み合わせ、穏やかに混合し、室温で30分間インキュベートする。
- 混合物(バクミドおよび試薬)を細胞上に加え(ステップ1.2.6.1)、それらを27°Cで3時間プレーティングする。
- グレースの培地を取り出し、2 mLのESF921細胞培養培地と交換してください。27°Cで6穴プレートをプレートし、トランスフェクション後5日後にESF921細胞培養培地を回収した。
- 4°Cで500 x g で10分間遠心分離し、細胞および破片を除去した。上清を2mLチューブに移す。これはP0バキュロウイルス株です。
- P1ウイルスストックを分離する
- 30 mLのsf9細胞を円錐形のボトルに移し、270 rpmで振とうしながら27°Cで培養し、細胞が対数期(1.0-1.5 x 106 cells/mL)に達することを確認します。
- 2mLのP0ウイルスストックをボトルに加え、270rpmで27°Cで4日間振盪する。
- 細胞を50 mLチューブに移し、1,800 x g で4°Cで10分間遠心分離して細胞および破片を除去し、上清を50 mLチューブに移した。これはP1バキュロウイルス株です。
- バキュロウイルス株を増幅する
- 対数期の 50 mL の sf9 細胞 (1.0-1.5 x106 cells/mL) と 1 mL の P1 ストックを使用して、ステップ 1.2.7 を繰り返します。
- 得られたP2バキュロウイルスストックを4°Cで保存し、光から保護する。
注:有害な変異体は各継代で産生されたため、バキュロウイルスを無期限に増幅しないでください。
- S1PRs-Gタンパク質複合体の発現
- sf9昆虫細胞を2.5 x106細胞/mL密度に達するまで培養し、S1PR、Gi1、およびGβ1γ2をコードするP2バキュロウイルスと1:2:1の体積比で共感染させ、27°Cで48時間培養する。
- 細胞を回収し、4°Cで700 x g で15分間遠心分離し、液体窒素中で凍結し、-80°Cで保存して使用する。
- タンパク質精製
- 工程1.3で得られた細胞ペレットを室温で解凍し、次いで、100μg/mLのベンズアミジン、100μg/mLのロイペプチン、100μg/mLのアプロチニン、25mU/mLのアピラーゼ、および10μMのアゴニストを添加した溶解緩衝液(20mM HEPES pH 7.5、50mM NaCl、50mM MgCl2、5mM CaCl2)に再懸濁した。細胞懸濁液を室温で2時間撹拌して、S1PRs−Gタンパク質複合体の形成を誘導する。
注:アピラーゼはATPジホスホヒドロラーゼである。これは、ATPからのガンマリン酸およびADPからのベータリン酸の除去を触媒する。 - 溶液をチューブに移し、70,000 x g で10分間遠心分離し、上清を慎重に除去する。ペレットを可溶化緩衝液(20 mM HEPES pH 7.5、100 mM NaCl、5 mM MgCl2、5 mM CaCl2、0.5% (w/v) LMNG、0.1% (w/v) CHS、1% (w/v) コール酸ナトリウム、10% (v/v) グリセロール)に再懸濁します。
- 懸濁液をガラスDounceに移し、ペレットを完全に均質化する。懸濁液に10 μMアゴニスト、scFv16 mg、ベンズアミジン100 μg/mL、ロイペプチン100 μg/mL、アプロチニン100 μg/mL、およびアピラーゼ25 mU/mLを加え、4°Cで2時間攪拌する。
注:ペレットを均質化するステップは、GPCR-Gタンパク質複合体の産生にとって極めて重要である。 - 溶液をチューブに移し、100,000 x g で4°Cで30分間遠心分離した。
- フラグ樹脂を洗浄バッファー (20 mM HEPES pH 7.5、100 mM NaCl、5 mM MgCl2、5 mM CaCl2、10 μM アゴニスト、0.0375% (w/v) LMNG、0.0125% (w/v) GDN、0.01% (w/v) CHS) で予め平衡化します。
- 上清をチューブに移し、フラグ樹脂とともに4°Cで2時間インキュベートした。
- 上記の混合物をガラスカラムにロードし、100 μg/mL ベンズアミジン、100 μg/mL ロイペプチン、および 100 μg/mL アプロチニンを添加した 50 mL の洗浄バッファーでカラムを洗浄します。
- 20 mM HEPES pH 7.5、100 mM NaCl、5 mM EDTA、200 μg/mL フラッグペプチド、10 μM アゴニスト、0.0375% (w/v) LMNG、0.0125% (w/v) GDN、0.01% (w/v) CHS、100 μg/mL ベンズアミジン、100 μg/mL ロイペプチン、および 100 μg/mL アプロチニンを含む 10 mL の溶出バッファーでカラムを溶出します。
- S1PRs-Gタンパク質複合体を回収し、4°Cで1,300 x g で100 kDaカットオフ濃縮器を使用して1 mLに濃縮します。 0.22 μM フィルターでろ過し、13,000 x g で 4 °C で 10 分間遠心分離して凝集体を除去します。
- S1PRs-G タンパク質複合体を、20 mM HEPES pH 7.5、100 mM NaCl、10 μM アゴニスト、100 Μm TCEP、0.00375% (w/v) LMNG、0.00125% (w/v) GDN、および 0.001% (w/v) CHS を含む SEC バッファーで予め平衡化したサイズ排除クロマトグラフィー (SEC) ゲルろ過カラムに、4 °Cで 0.5 mL/分の流速でロードします。
- ピーク画分を収集し、クライオEM用に4°Cで1,300 x g で100 kDaカットオフ濃縮器を使用して濃縮します。
- 工程1.3で得られた細胞ペレットを室温で解凍し、次いで、100μg/mLのベンズアミジン、100μg/mLのロイペプチン、100μg/mLのアプロチニン、25mU/mLのアピラーゼ、および10μMのアゴニストを添加した溶解緩衝液(20mM HEPES pH 7.5、50mM NaCl、50mM MgCl2、5mM CaCl2)に再懸濁した。細胞懸濁液を室温で2時間撹拌して、S1PRs−Gタンパク質複合体の形成を誘導する。
2. S1PRの構造を解く電子顕微鏡
- データ収集
- クライオEMグリッドを準備するには、グロー放電洗浄システムを使用して、300メッシュのAu R1.2/1.3グリッドを10秒間保持し、15mAで60秒間グロー放電します。
- 前述の33、34に記載したようにサンプルガラス化を行う。プランジ凍結コンソールで、チャンバーの作業環境の温度を4°C、相対湿度を100%に設定します。ブロット力を0、待機時間を0秒、ブロット時間を2~3秒、ドレイン時間を0秒にします。通常、単一のガラス化のために5-10 mg/mLの濃度で3 μLのサンプルしか必要としません。
- 前述のように、グリッドを自動グリッドアセンブリにクリップしてロードし、自動グリッドアセンブリをナノキャブにロードし、オートローダによってナノキャブを顕微鏡にロードします35。EPU2ソフトウェアによるスクリーンサンプル品質34.通常、適切な氷の厚さの地域で収集されたデータの方が優れていました。
- 前述の35で詳細に説明したようにクライオEMデータを収集する。S1P受容体の場合、デフォーカスオフセットを-1.0 μm~-1.8 μmに設定し、露光電子線量は50-65 e-/Å2です。S1PR1-Gi複合体の場合、EPU2ソフトウェアを使用して、K2検出器と計数モードで、合計露光時間2秒、記録速度56 e-/Å2 でムービースタックを自動的に収集し、スタックあたり35フレームを生成します。
注:通常、受容体構造を再構築するには5,000以上の映画が必要です。
- RELION の組み合わせを使用したデータの処理36 クライオスパーク37 理想的なクライオ-EM密度マップを得る。RELION-3.1_gpu_ompi4を使用して、前述のように同様の操作を伴うデータを最初に処理します。34.
- Linux システム端末で、データ・ストレージ・ディレクトリーの親ディレクトリーを入力します。
- 端末で relion コマンドを入力して、RELION グラフィカル・ユーザー・インターフェース (GUI) を開きます。
注: このディレクトリーで RELION GUI が初めてオープンされた場合は、プロンプト・ウィンドウがポップアップします。[ はい]をクリックします。 - RELION GUIの右側にある機能バーのインポートをクリックして、生データをRELIONにインポートします。
- 「ムービー/マイク」オプションで、「未加工のムービー/顕微鏡写真を読み込む」で「はい」を選択し、「未加工の入力ファイル」フィールドにデータパスを入力して(ワイルドカードを推奨)、および「これらのマルチフレームムービーですか?」で「はい」を選択します。 ピクセルサイズ(オングストローム)フィールドにムービーのピクセルサイズを、電圧(kV)フィールドに顕微鏡の動作電圧(kV単位)を、球面収差(mm)フィールドに顕微鏡の球面収差を入力します。これらは、データ収集時に記録されたパラメーターです。
- 「実行 中」 オプションで、プログラムが実行されているサーバーに従って キュー名 を変更します (他の関数もこのパラメーターを変更する必要があります)。その他のパラメーターは、RELION によって設定されたデフォルト値のままにしておきます。最後に、すべてのパラメータが正しく検出されたら、[ RUN !]をクリックしてプログラムを実行します。
- すべてのフレーム38のアライメントにモーション補正機能を使用します。
- I/Oオプションで、[参照]をクリックし、movies.starという名前のインポート機能の出力を入力ムービーSTARファイルの入力として選択します。[フレームあたりの線量(e/A2)]フィールドにフレームあたりの線量を入力します。これは、総線量をフレーム数で割った値に等しくなります。[検出力スペクトルの合計を保存する] で [いいえ] を選択します。
- モーションオプションで、Bファクターに250、パッチ数X、Yに5.5、グループフレームに2と入力します(グループ>3の線量を確認してください)。収集期間中にデータがゲイン参照されなかった場合は、空白のグリッド領域を取得することで取得できるゲイン参照画像が必要です。RELION独自の実装を使用するには「いいえ」を選択し、MOTIONCOR2 39の実行可能ファイルを含むディレクトリをMOTIONCOR2実行可能フィールドに入力します。
- 「実行中」オプションで、サーバーの計算能力に応じて適切な MPI とスレッド番号を選択します。ここでは、MPI = 8 とスレッド = 3 が使用されました。
- CTF推定機能を使用して、ガラス化標本40のクライオEM画像を変調する。I / Oオプションで、[参照]をクリックし、入力ムービーSTARファイルの入力として、修正されたmicrographs.starという名前のモーションコルの出力を選択します。Gctf オプションで、代わりに UseGctf に対して [はい] を選択します。
- サブセット選択機能を使用して、rlnCtfMaxResolutin >4の値を持つ顕微鏡写真を削除します。
- I/Oオプションで、OR micrographs.starの右側にある[参照]をクリックし、入力としてmicrographs_ctf.starという名前のCtfFindの出力を選択します。[サブセット] オプションで、[メタデータ値に基づいて選択] に [はい] を選択し、[メタデータの最大値] に 4 と入力して、不適切なデータを削除します。
- 手動ピッキング: 予備ピックと分類のために手動でいくつかの画像を選択します。
- I/Oオプションで、[参照]をクリックし、前の[ディレクトリの選択](ステップ2.2.6)から入力としてmicrographs_selected.starを選択します。
- RUN!をクリックします(ウィンドウがポップアップします)。新しいウィンドウの左上隅にある[ファイル]をクリックし、[選択を反転]をクリックしてすべての画像の選択をキャンセルします。各エントリの対応する行の左端の選択ボックスをチェックし、ピックをクリックして画像を確認し、〜500個の良い画像を選択します。[ファイル]>[選択範囲の保存]をクリックして、選択した画像を保存し、ウィンドウを閉じます。
- 自動ピッキング:自動パーティクルピッキングソフトウェアパッケージは便利で強力です41.
- I/O オプションで、自動ピック用の [入力顕微鏡写真] の右側にある [参照] をクリックし、前の ManualPick ディレクトリ (ステップ 2.2.7) から [micrographs_selected.star] を入力として選択します。ラプラシアン・オブ・ガウス・アルゴリズムが最初に使用されるため、OR:ラプラシアン・オブ・ガウス・アルゴリズムを使用 に「はい」を選択します。
- ラプラシアンオプションで、LoGフィルター(A)の最小直径を80に、LoGフィルター(A)の最大直径を130に設定します。[自動ピッキング] オプションで、[パーティクル間の最小距離 (A)] を 65 に設定し、[GPU にアクセスできる場合は GPU アクセラレーションを使用する] で [はい] を選択します。
- 次のステップのために粒子を抽出します。
- I/Oオプションで、顕微鏡写真STARファイルの右側にある[参照]をクリックし、ステップ2.2.6からmicrographs_selected.starを選択します。入力座標の右側にある[参照]をクリックし、ステップ2.2.8からcords_suffix_autopick.starを選択します。
- [抽出] オプションで、[パーティクルの再スケール] で [はい] を選択し、[再スケーリングされたサイズ (ピクセル)] を 128 に設定して、後の手順を高速化します。
- 粒子の予備分類のための2D分類
- I/O オプションで、入力イメージ STAR ファイルの右側にある [参照] をクリックし、ステップ 2.2.9 から particles.star を選択します。「最適化」オプションで、「クラス数」を「100」に、「マスク直径 (A)」を「140」に設定します。
- [計算] オプションで、[プールされたパーティクルの数] を 10 に設定し、[パーティクルをスクラッチ ディレクトリにコピー] フィールドに高速ローカル ドライブ(SSD ドライブなど)にあるディレクトリを入力し、[GPU アクセラレーションを使用する?] で [はい] を選択して処理速度を高速化します。
- パーティクルを選択するためのテンプレートとして良好な 2D 結果を選択するためのサブセット選択
- I/Oオプションで、model.starからクラスを選択の右側にある参照をクリックし、入力としてrun_it025_model.starという名前のステップ2.2.10の出力を選択します。「実行!」をクリックしてください。ポップアップウィンドウで、[画像を並べ替える]と[逆並べ替え]をオンにして、[表示]をクリックします。
- 自動ピッキング機能の参照の参照として、適切な代表的な 2D 結果を選択します。
メモ: 選択した結果は赤で囲まれています。良い 2D 分類結果と悪い 2D 分類結果は後で示されます。 - 右クリックして、[ 選択したクラスの保存] を選択します。
- このテンプレートは、自動ピッキングの第 2 ラウンドに使用します。I/Oオプションで、自動選択用の入力顕微鏡写真の右側にある[参照]をクリックし、ステップ2.2.6からmicrographs_selected.starを選択します。2D 参照の右側にある [参照] をクリックし、ステップ 2.2.11 から [class_averages.star] を選択し、[OR] に [いいえ] を選択します: ラプラシアン オブ ガウシア n を使用します。
- ステップ 2.2.12 の coord_suffix_autopick.star とステップ 2.2.6 の micrographs_selected.star を使用して、粒子抽出を再度実行します。
- ステップ 2.2.13 の particles.star を使用して、2D 分類を再度実行します。
- ステップ 2.2.14 から run_it025_optimiser.star を使用してサブセット選択を再度実行します。
注:明確な輪郭と正しい形状を持つすべての2D画像を選択する必要があります。 - 以下のようにして粒子抽出を行う。I/O オプションで、顕微鏡写真 STAR ファイルの右側にある [参照] をクリックし、ステップ 2.2.6 から micrographs_selected.star を選択し、[はい] を選択して [または精錬粒子を再抽出しますか?] を選択します。 [微細化されたパーティクル STAR ファイル] の右側にある [参照] をクリックし、ステップ 2.2.15 から Particles.star を選択します。
- 3D初期モデルと参照マップの生成:I/Oオプションで、入力画像STARファイルの右側にある参照をクリックし、ステップ2.2.16からparticles.starを選択します。[最適化] オプションで [クラス数] を 1 に設定し、[マスクの直径 (A)] を 140 に設定します。
- 3D分類と予備3Dマップの生成:I/Oオプションで、入力画像STARファイルの右側にある[参照]をクリックし、ステップ2.2.16からparticles.starを選択します。参照マップの右側にある [参照] をクリックし、ステップ 2.2.17 から initial_model.mrc を選択します。「最適化」オプションで「クラス数」を 4 ~ 6 に設定し、「マスク直径 (A)」を「140」に設定します。
- マスク生成: I/O オプションの入力として、ステップ 2.2.17 から適切な 3D マップを選択します。「マスク」オプションで「初期二値化しきい値」を「初期二値化しきい値」を 0.05 (出力に基づいて調整)に設定し、「この数のピクセルを 3 に拡張」および「この数のピクセルのソフトエッジを 8 に追加」に設定します。
- クライオスパークは、次の処理に使用します。
- 新しいワークスペースを作成し、最初の ジョブのジョブビルダー をクリックします。
- パーティクルスタックを読み込むには、「パーティクルメタパス」フィールドにパーティクルパス(ステップ 2.2.16)を入力し、「パーティクルデータパス」フィールドにムービーのパス(ステップ 2.2.16)を入力します。
メモ: 加速電圧(kV)、球面収差(mm)、および ピクセルサイズ(オングストローム) のパラメータは、以前と同じです。 振幅コントラスト (分数)値は0.1です。 - 3D ボリュームをインポートするには、ステップ 2.2.18 で最適な 3D ボリュームのパスをボリューム・データ・パスに入力し、「インポートするボリュームのタイプにマップ」を選択します。
- マスクをインポートするには、「ボリューム・データ・パス」にマスク・パス (ステップ 2.2.19) を入力し、インポートするボリュームのタイプで「マスク」を選択します。
- 不均一な絞り込み: ステップ 2.2.22、2.2.23、および 2.2.24 の出力を入力として使用します。
注:この機能は膜タンパク質に非常に有用です。- ステップ2.2.22(imported_particles)の出力を不均一な微細化の 粒子(粒子)の入力として、ステップ2.2.23(imported_volume_1)の出力を不均一な微細化の 体積(体積)の入力として、ステップ2.2.24 (imported_mask_1)の出力を不均一な微細化の マスク(マスク)の入力としてドラッグします。
注:マスクなしでより良い結果が得られる場合があります。 - 「 キュー 」をクリックして処理を開始します。
- ステップ2.2.22(imported_particles)の出力を不均一な微細化の 粒子(粒子)の入力として、ステップ2.2.23(imported_volume_1)の出力を不均一な微細化の 体積(体積)の入力として、ステップ2.2.24 (imported_mask_1)の出力を不均一な微細化の マスク(マスク)の入力としてドラッグします。
- より良い結果を得るために、ステップ2.2.18-2.2.25を実行します。以上の一連の処理により、解像度の良いS1PR-Gi 3Dマップが得られる。
S1PRs-Gi媒介性cAMP阻害アッセイ
注:S1PRs-Gi媒介性cAMP阻害実験はいくつかの部分に分かれており、以下は詳細な実験手順である。実験原理と一般的な実験過程をフローチャートの形で 図1に示す。
- プラスミド構築
- 野生型S1PR1、S1PR3、およびS1PR5のcDNAを、HAシグナル配列とそれに続くN末端のFlagタグでベクターpcDNA3.1+にサブクローニングします(材料表)。
注:すべての受容体の変異は、突然変異誘発キット(材料表)を使用して生成した。
- 野生型S1PR1、S1PR3、およびS1PR5のcDNAを、HAシグナル配列とそれに続くN末端のFlagタグでベクターpcDNA3.1+にサブクローニングします(材料表)。
- プラスミドの調製
- -80°Cの1.5 mLチューブに保存したDH5αコンピテント大腸菌(大腸菌)50 μLに組換えpcDNA3.1+ベクターまたはセンサープラスミド(材料表)を別々に加え、氷上で30分間インキュベートします。細胞を42°Cで90秒間ヒートショックし、すぐに氷に移し、2分間冷やします。
注:Gi阻害cAMPアッセイキット(材料表)によって提供されるセンサープラスミドは、cAMP結合ドメインと融合した修飾ルシフェラーゼ遺伝子を発現し、cAMPが結合すると発光活性を増加させます。 - 300 μLのリゾジェニーブロス(LB)培地を補充した後、チューブを37°Cで1時間振盪する。細胞のプレート100 μLをLB寒天プレートに、暗所に48時間保持して37°Cでインキュベートした。白色コロニーを100μg/mLアンピシリンを含む5mLのLB培地に接種し、37°Cで16時間培養した。
- プラスミドミニプレップキット(材料表)を使用して、製造元の指示に従ってDNAを単離します。プラスミドの濃度は350 ng/μLを超え、A260/A280 の値は1.7~1.9でした。
- -80°Cの1.5 mLチューブに保存したDH5αコンピテント大腸菌(大腸菌)50 μLに組換えpcDNA3.1+ベクターまたはセンサープラスミド(材料表)を別々に加え、氷上で30分間インキュベートします。細胞を42°Cで90秒間ヒートショックし、すぐに氷に移し、2分間冷やします。
- 細胞培養
- CHO-K1細胞を10cmシャーレに挽き、5%CO2を含む37°Cのインキュベーターで培養し、単層が80%〜90%のコンフルエントにあるときにそれらを回収する。
- CHO-K1細胞増殖培地を吸引し、37°Cで予め加温した0.05%トリプシン-EDTAを4 mLのシャーレ上に穏やかに添加し、15秒間インキュベートした。次いで、F12培地+10%FBSからなる4mLの増殖培地を加える。
- ペトリ皿の側面を軽く振って叩いて、ペトリ皿の表面から細胞を取り除きます。円錐形のチューブを細胞懸濁液で満たします。ゆっくりとかき混ぜてピペットで細胞集塊をやさしく取り除きます。
- 細胞を室温で5分間250 x g で遠心分離し、上清を吸引し、3mLのPBSで蘇生した。この手順を繰り返します。
- 血球計数器で細胞数を決定し、細胞を250 x g で室温で5分間遠心分離する。
- PBSバッファーを吸引し、F12培地および10%FBSからなる3mLの増殖培地でCHO−K1細胞を再懸濁する。
- F12培地と10%FBSからなる増殖培地2mLを6ウェルプレートの各ウェルに加え、各ウェルに細胞懸濁液150μLをシードして、細胞を1.5 x105 cells/mLの密度に保ちます。6ウェルプレートを5%CO2と共に37°Cの組織培養インキュベーター内で約24 時間インキュベートする。
- 一過性トランスフェクション
- 2 μg の DNA を 200 μL のトランスフェクション試薬バッファーに希釈します (工程 3.2.3)。使用前に10秒間ボルテックスし、短時間回転させて混合します。
注:ここでは、2 μgのDNAには、0.5 μgの受容体ベクター(S1PR1、S1PR3、またはS1PR5)と1.5 μgのセンサープラスミドが含まれています。 - 4 μL のトランスフェクション試薬 (材料表) を加え、10 秒間ボルテックスし、使用前に短時間スピンします。室温で15分間インキュベートする。
- 200 μLのトランスフェクションミックスを各ウェル(CHO-K1細胞を含む)にゆっくりと滴下し、均等に分配します。6ウェルプレートを静かに振って、完全に混合してください。
- 4〜6時間後にトランスフェクション培地をF12培地+10%FBSからなる細胞増殖培地と交換し、6ウェルプレートをインキュベーターに戻す。
- トランスフェクション後24〜48時間で細胞を回収する。
- ウェル上のCHO-K1細胞を500 μLの0.05%トリプシン-EDTA(37°Cで予熱)で15秒間消化し、F12培地+10%FBSからなる増殖培地1 mLを加える。ウェルの側面を揺らして静かに叩くことによって、ウェル表面から細胞を取り除きます。
- 細胞懸濁液を円錐形チューブに移し、室温で250 x g で5分間遠心分離した。上清を注ぎ、トランスフェクトした細胞を回収する。
注:蛍光シグナル測定の前に、前述のようにELISAによって受容体の細胞表面発現レベルを確認してください26。
- 2 μg の DNA を 200 μL のトランスフェクション試薬バッファーに希釈します (工程 3.2.3)。使用前に10秒間ボルテックスし、短時間回転させて混合します。
- D-ルシフェリン-カリウム塩との平衡化(材料表)
- 回収した細胞を直ちに3 mLのアッセイバッファー(すなわち、10 mM HEPES pH 7.4を含むハンクス平衡塩溶液(HBSS))で懸濁し、D-ルシフェリンカリウム塩をさらに3% v/v希釈します。
- マルチチャンネルピペットを用いて96ウェルプレートのウェルあたり90μLの細胞懸濁液を加え、穏やかに混合する。
- 室温で40分間インキュベートする。
- 蛍光シグナル測定
- DMSOに溶解したシポニモドの10mMストック溶液を予め調製し、リガンド刺激前に25μMフォルスコリンを含むHBSS緩衝液を用いて段階希釈を行う。
注:リガンドを含まない対照群を除き、残りは10-11-10-5 mol/Lの濃度勾配範囲を有する。 - 異なる濃度の10μL(ウェルあたり)のアゴニスト溶液で30分間刺激する。
- 以下のように、関連するソフトウェア(材料表)パラメータを使用して、マイクロプレートリーダー上の発光信号をカウントします。検出方法の場合はルミネッセンス、読み取りタイプはエンドポイント、光学タイプはルミネッセンスファイバーを選択します。 オプティクスゲインを255に設定します。
注:各測定は、少なくとも3つの独立した実験で、それぞれ3連で繰り返した。 - 蛍光シグナルの値を取得し、そのデータをスプレッドシートプログラムにインポートし、非線形回帰(カーブフィット)用量反応関数を使用してデータを処理します。
- DMSOに溶解したシポニモドの10mMストック溶液を予め調製し、リガンド刺激前に25μMフォルスコリンを含むHBSS緩衝液を用いて段階希釈を行う。
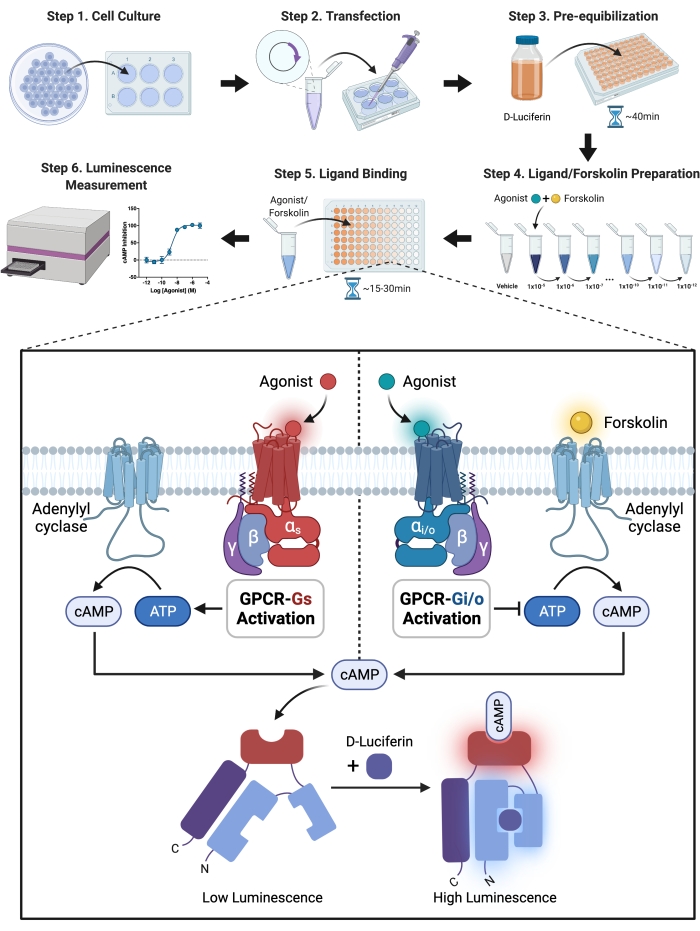
図1:実験の概略図。実験のセットアップと実行のための詳細なステップワイズガイド。簡単に言えば、受容体および修飾ルシフェラーゼを、トランスフェクション試薬を用いて受容体およびSensorプラスミドを細胞内にトランスフェクトすることによって、CHO−K1細胞において一過性に共発現させた。細胞をルシフェラーゼ基質であるD-ルシフェリンカリウム塩を含むHBSS溶液に懸濁し、24時間後に96ウェルプレートに播種した。細胞への透過を可能にするために、D-ルシフェリンは細胞と予め平衡化されなければならない。酸化酵素ルシフェラーゼは、ルシフェリンをオキシルシフェリンに変換し、発光する。一方、修飾ルシフェラーゼは、cAMPに結合した場合にのみ化学反応を介して光を生成し、光の強度は細胞内のcAMPレベルと正の関連を有する。cAMPのレベルは、アゴニストによって活性化されたGPCRで調節した。Gi共役型受容体はcAMPのレベルを低下させ、Gi阻害cAMP実験においてアデニリルシクラーゼを活性化するためにフォルスコリンを添加する必要があった。この図の拡大版を表示するには、ここをクリックしてください。
結果
S1PRs-Gi複合体のサンプルを凍結する前に、精製サンプルをサイズ排除クロマトグラフィー(SEC)で分離し、ゲルろ過クロマトグラフィーで分析する必要があります。 図2 は、S1PR3-Gi複合体を例に示している。均質なGPCR−Gタンパク質複合体のピーク画分は、通常、サイズ排除クロマトグラフィーの〜10.5mLに位置していた(図2A)。S1PR3-Gi複合体のSDSページ?...
ディスカッション
このプロトコルは、クライオEMによってS1PRの構造を決定し、Gi媒介cAMP阻害アッセイによってS1PRの活性化効力を測定するための一次パイプラインを記述する。いくつかのステップは、実験の成功に不可欠です。
S1PRs-Gi複合体を精製するためには、ウイルスの質およびsf9細胞の健康状態にもっと注意を払うべきである。受容体の発現は、貧弱なsf9細胞において?...
開示事項
著者には利益相反はありません。
謝辞
S1PRs-Gi複合体のデータは、四川大学の華西クライオEMセンターと南方科技大学(SUSTech)のクライオEMセンターで収集され、四川大学のDuyu高性能コンピューティングセンターで処理されました。この研究は、中国自然科学財団(32100965からL.C.、32100988がW.Y.、31972916がZ.S.)と四川大学の専任ポスドク研究基金(2021SCU12003からL.C.)の支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 0.05% trypsin-EDTA | GIBCO | Cat# 25300054 | |
| 0.22 µM filter | Thermo Fisher Scientific | Cat# 42213-PS | |
| 100 kDa cut-off concentrator | Thermo Fisher Scientific | Cat# 88533 | |
| 6-well plate | Corning | Cat# 43016 | |
| 96-well plate | Corning | Cat# 3917 | |
| Aprotinin | Sigma-Aldrich | Cat# 9087-70-1 | |
| Apyrase | NEB | Cat# M0398S | |
| Baculovirus transfection reagent | Thermo Fisher Scientific | Cat# 10362100 | For the preparation of P0 baculovirus |
| Benzamidine | Sigma-Aldrich | Cat# B6506 | |
| CHO-K1 | ATCC | N/A | |
| CHS | Sigma-Aldrich | Cat# C6512 | |
| CryoSPARC | Punjani, A., et al.,2017 | https://cryosparc.com/ | |
| DH5α competent E.coli | Thermo Fisher Scientific | Cat# EC0112 | |
| D-Luciferin-Potassium Salt | Sigma- Aldrich | Cat# 50227 | |
| DMSO | Sigma- Aldrich | Cat# D2438 | |
| EDTA | Thermo Fisher Scientific | Cat# S311-500 | |
| ESF921 cell culture medium | Expression Systems | Cat# 96-001 | |
| Excel | microsoft | N/A | |
| F12 medium | Invitrogen | Cat# 11765 | |
| FBS | Cell Box | Cat# SAG-01U-02 | |
| Flag resin | Sigma- Aldrich | Cat# A4596 | |
| Forskolin | APExBIO | Cat# B1421 | |
| Gctf | Zhang, 2016 | https://www.mrc-lmb.cam.ac.uk/kzhang/Gctf/ | |
| GDN | Anatrace | Cat# GDN101 | |
| Gel filtration column | GE healthcare | Cat# 28990944 | |
| Gen5 3.11 | BIO-TEK | N/A | |
| Gentamicin | Solarbio | Cat# L1312 | |
| GloSensor cAMP assay kit | Promega | Cat# E1291 | Gi-inhibition cAMP assay kit |
| GloSensor plasmid | Promega | Cat# E2301 | Sensor plasmid |
| Grace’s medium | GIBCO | Cat# 11595030 | |
| GraphPad Prism 8 | Graphpad | N/A | |
| HBSS | Thermo Fisher Scientific | Cat# 88284 | |
| HEPES | Sigma- Aldrich | Cat# H4034 | |
| jetPRIME Reagent | Polyplus Transfection | Cat# 114-15 | transfection reagent |
| Janamycin | Solarbio | Cat# K1030 | |
| LB medium | Invitrogen | Cat# 12780052 | |
| Leupeptin | Sigma-Aldrich | Cat# L2884 | |
| LMNG | Anatrace | Cat# NG310 | |
| MotionCor2 | (Zheng et al., 2017) | https://emcore.ucsf.edu/ucsf-software | |
| NanoCab | Thermo Fisher Scientific | Cat# 1121822 | |
| PBS | Invitrogen | Cat# 14190-144 | |
| pcDNA3.1-HA-FLAG-S1PRs | GenScript | N/A | |
| pFastBac1-Gαi | GenScript | N/A | |
| pFastBac1-HA-FLAG-T4L-S1PRs-His10 | GenScript | N/A | |
| pFastBacdual-Gβ1γ2 | GenScript | N/A | |
| PureLink HiPure Plasmid Miniprep Kit | Invitrogen | Cat# K210003 | For the preparation of plasmids and P0 baculovirus |
| Q5 site-Directed Mutagenesis kit | NEB | Cat# E0554S | For the preparation of plasmids |
| Quantifoil | Quantifoil | Cat# 251448 | |
| RELION-3.1 | (Zivanov et al., 2018) | https://www2.mrc-lmb.cam.ac.uk/relion | |
| S1PRs cDNA | addgene | N/A | |
| scFv16 | Invitrogen | Cat# 703976 | |
| Sf9 | Expression Systems | N/A | |
| Siponimod | Selleck | Cat# S7179 | |
| sodium cholate | Sigma-Aldrich | Cat# C1254 | |
| Synergy H1 microplate reader | BIO-TEK | N/A | |
| Synthetic T4L DNA (sequence) | N/A | N/A | Aacatcttcgagatgctgcgcatcgacgaagg cctgcgtctcaagatttacaagaataccgaagg ttattacacgattggcatcggccacctcctgaca aagagcccatcactcaacgctgccaagtctga actggacaaagccattggtcgcaacaccaac ggtgtcattacaaaggacgaggcggagaaac tcttcaaccaagatgtagatgcggctgtccgtgg catcctgcgtaatgccaagttgaagcccgtgt atgactcccttgatgctgttcgccgtgcagcctt gatcaacatggttttccaaatgggtgagaccgg agtggctggttttacgaactccctgcgcatgctcc agcagaagcgctgggacgaggccgcagtga atttggctaaatctcgctggtacaatcagacacc taaccgtgccaagcgtgtcatcactaccttccg tactggaacttgggacgcttac |
| TCEP | Thermo Fisher Scientific | Cat# 77720 | |
| Tetracycline | Solarbio | Cat# T8180 | |
| Vitrobot Mark IV | Thermo Fisher Scientific | N/A |
参考文献
- Verstockt, B., et al. Sphingosine 1-phosphate modulation and immune cell trafficking in inflammatory bowel disease. Nature Reviews: Gastroenterology & Hepatology. , 1-16 (2022).
- Rosen, H., Stevens, R. C., Hanson, M., Roberts, E., Oldstone, M. B. Sphingosine-1-phosphate and its receptors: structure, signaling, and influence. Annual Review of Biochemistry. 82, 637-662 (2013).
- Cartier, A., Hla, T. Sphingosine 1-phosphate: Lipid signaling in pathology and therapy. Science. 366 (6463), 5551 (2019).
- Jozefczuk, E., Guzik, T. J., Siedlinski, M. Significance of sphingosine-1-phosphate in cardiovascular physiology and pathology. Pharmacological Research. 156, 104793 (2020).
- Kihara, Y., Maceyka, M., Spiegel, S., Chun, J. Lysophospholipid receptor nomenclature review: IUPHAR Review 8. British Journal of Pharmacology. 171 (15), 3575-3594 (2014).
- Bryan, A. M., Del Poeta, M. Sphingosine-1-phosphate receptors and innate immunity. Cellular Microbiology. 20 (5), 12836 (2018).
- Pelletier, D., Hafler, D. A. Fingolimod for multiple sclerosis. New England Journal of Medicine. 366 (4), 339-347 (2012).
- Obinata, H., Hla, T. Sphingosine 1-phosphate and inflammation. International Immunology. 31 (9), 617-625 (2019).
- Pyne, N. J., Pyne, S. Sphingosine 1-phosphate and cancer. Nature Reviews: Cancer. 10 (7), 489-503 (2010).
- Abu-Farha, M., et al. The role of lipid metabolism in COVID-19 virus infection and as a drug target. International Journal of Molecular Sciences. 21 (10), 3544 (2020).
- Chun, J., Kihara, Y., Jonnalagadda, D., Blaho, V. A. Fingolimod: lessons learned and new opportunities for treating Multiple Sclerosis and other disorders. Annual Review of Pharmacology and Toxicology. 59, 149-170 (2019).
- Murakami, A., et al. Sphingosine 1-phosphate (S1P) regulates vascular contraction via S1P3 receptor: investigation based on a new S1P3 receptor antagonist. Molecular Pharmacology. 77 (4), 704-713 (2010).
- Cao, L., et al. Siponimod for multiple sclerosis. Cochrane Database of Systematic Reviews. 11, (2021).
- Scott, L. J. Siponimod: a review in secondary progressive Multiple Sclerosis. CNS Drugs. 34 (11), 1191-1200 (2020).
- Lamb, Y. N. Ozanimod: first approval. Drugs. 80 (8), 841-848 (2020).
- Scott, F. L., et al. Ozanimod (RPC1063) is a potent sphingosine-1-phosphate receptor-1 (S1P1) and receptor-5 (S1P5) agonist with autoimmune disease-modifying activity. British Journal of Pharmacology. 173 (11), 1778-1792 (2016).
- McGowan, E. M., Haddadi, N., Nassif, N. T., Lin, Y. Targeting the SphK-S1P-SIPR pathway as a potential therapeutic approach for COVID-19. International Journal of Molecular Sciences. 21 (19), 7189 (2020).
- O'Sullivan, C., Dev, K. K. The structure and function of the S1P1 receptor. Trends in Pharmacological Sciences. 34 (7), 401-412 (2013).
- Liao, M., Cao, E., Julius, D., Cheng, Y. Structure of the TRPV1 ion channel determined by electron cryo-microscopy. Nature. 504 (7478), 107-112 (2013).
- Bai, X. C., McMullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- Murata, K., Wolf, M. Cryo-electron microscopy for structural analysis of dynamic biological macromolecules. Biochimica et Biophysica Acta General Subjects. 1862 (2), 324-334 (2018).
- Zhang, M., et al. Cryo-EM structure of an activated GPCR-G protein complex in lipid nanodiscs. Nature Structural & Molecular Biology. 28 (3), 258-267 (2021).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews: Drug Discovery. 17 (7), 471-492 (2018).
- Ishchenko, A., Gati, C., Cherezov, V. Structural biology of G protein-coupled receptors: new opportunities from XFELs and cryoEM. Current Opinion in Structural Biology. 51, 44-52 (2018).
- Yang, D., et al. G protein-coupled receptors: structure- and function-based drug discovery. Signal Transduction and Targeted Therapy. 6 (1), 7 (2021).
- Yuan, Y., et al. Structures of signaling complexes of lipid receptors S1PR1 and S1PR5 reveal mechanisms of activation and drug recognition. Cell Research. 31 (12), 1263-1274 (2021).
- Zhao, C., et al. Structural insights into sphingosine-1-phosphate recognition and ligand selectivity of S1PR3-Gi signaling complexes. Cell Research. 32 (2), 218-221 (2022).
- Xu, Z., et al. Structural basis of sphingosine-1-phosphate receptor 1 activation and biased agonism. Nature Chemical Biology. 18, 281-288 (2022).
- Liu, Y. F., Ghahremani, M. H., Rasenick, M. M., Jakobs, K. H., Albert, P. R. Stimulation of cAMP synthesis by Gi-coupled receptors upon ablation of distinct Galphai protein expression. Gi subtype specificity of the 5-HT1A receptor. Journal of Biological Chemistry. 274 (23), 16444-16450 (1999).
- Buccioni, M., et al. Innovative functional cAMP assay for studying G protein-coupled receptors: application to the pharmacological characterization of GPR17. Purinergic Signalling. 7 (4), 463-468 (2011).
- Wang, F. I., Ding, G., Ng, G. S., Dixon, S. J., Chidiac, P. Luciferase-based GloSensor cAMP assay: Temperature optimization and application to cell-based kinetic studies. Methods. , (2021).
- Audet, M., et al. Small-scale approach for precrystallization screening in GPCR X-ray crystallography. Nature Protocols. 15 (1), 144-160 (2020).
- Sgro, G. G., Costa, T. R. D. Cryo-EM grid preparation of membrane protein samples for single particle analysis. Frontiers in Molecular Biosciences. 5, 74 (2018).
- White, J. B. R., et al. Single particle cryo-electron microscopy: from sample to structure. Journal of Visualized Experiments. (171), e62415 (2021).
- Thompson, R. F., Iadanza, M. G., Hesketh, E. L., Rawson, S., Ranson, N. A. Collection, pre-processing and on-the-fly analysis of data for high-resolution, single-particle cryo-electron microscopy. Nature Protocols. 14 (1), 100-118 (2019).
- Fernandez-Leiro, R., Scheres, S. H. W. A pipeline approach to single-particle processing in RELION. Acta Crystallographica Section D. 73 (6), 496-502 (2017).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Brilot, A. F., et al. Beam-induced motion of vitrified specimen on holey carbon film. Journal of Structural Biology. 177 (3), 630-637 (2012).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14 (4), 331-332 (2017).
- Zhang, K. Gctf: Real-time CTF determination and correction. Journal of Structural Biology. 193 (1), 1-12 (2016).
- Scheres, S. H. Semi-automated selection of cryo-EM particles in RELION-1.3. Journal of Structural Biology. 189 (2), 114-122 (2015).
- Liu, S., et al. Differential activation mechanisms of lipid GPCRs by lysophosphatidic acid and sphingosine 1-phosphate. Nature Communications. 13 (1), 731 (2022).
- Duan, J., et al. Cryo-EM structure of an activated VIP1 receptor-G protein complex revealed by a NanoBiT tethering strategy. Nature Communications. 11 (1), 4121 (2020).
- DiIorio, M. C., Kulczyk, A. W. A robust single-particle cryo-electron microscopy (cryo-EM) processing workflow with cryoSPARC, RELION, and Scipion. Journal of Visualized Experiments. (179), e63387 (2022).
- Pradelles, P., Grassi, J., Chabardes, D., Guiso, N. Enzyme immunoassays of adenosine cyclic 3',5'-monophosphate and guanosine cyclic 3',5'-monophosphate using acetylcholinesterase. Analytical Chemistry. 61 (5), 447-453 (1989).
- Jiang, L. I., et al. Use of a cAMP BRET sensor to characterize a novel regulation of cAMP by the sphingosine 1-phosphate/G13 pathway. Journal of Biological Chemistry. 282 (14), 10576-10584 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved