このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
レンチウイルス媒介CRISPR/Cas9遺伝子編集を用いたノックアウト筋細胞株の開発
要約
このプロトコルは、ガイドRNAの設計からノックアウトクローンの細胞クローニングおよび特性評価まで、CRISPR/Cas9を使用してノックアウト筋芽細胞を生成する方法を説明しています。
要約
クラスター化調節間隔短い回文反復(CRISPR)/Cas 9の重要な用途の1つは、特に遺伝子診断中に同定された疾患に関連する新しい遺伝子/タンパク質の機能を研究するためのノックアウト細胞株の開発である。このような細胞株の開発のためには、選択された細胞へのCRISPRツール(Cas9およびガイドRNA)の高効率の挿入、および選択された遺伝子の特異的欠失に対するCas9活性の制限の2つの主要な課題を解きほぐさなければならない。ここで説明するプロトコルは、筋肉細胞などのトランスフェクトが困難な細胞にCRISPRツールを挿入することに専念しています。このプロトコールは、一般に入手可能なプラスミドで産生されるレンチウイルスの使用に基づいており、すべてのクローニングステップが目的の遺伝子を標的とするために説明されています。Cas9活性の制御は、Cas9を標的とするガイドRNAをコードするレンチウイルスによる細胞の形質導入がCas9発現の漸進的廃止を可能にするKamiCas9と呼ばれる前述のシステムの適応を用いて行われている。このプロトコルは、筋肉細胞内カルシウム放出および励起収縮結合に関与するこの重要なカルシウムチャネルのノックアウトを確認するために、タンパク質および機能レベルでさらに特徴付けられた RYR1ノックアウトヒト筋細胞株の開発に適用されている。ここで説明する手順は、筋肉細胞または他のトランスフェクトが困難な細胞内の他の遺伝子に容易に適用することができ、ヒト細胞におけるこれらの遺伝子を研究するための貴重なツールを生成することができる。
概要
遺伝子シーケンシングの進歩や、特定の組織における機能未知の遺伝子の変異の同定に伴い、新たな標的遺伝子の機能を理解し、関連する病態生理学的機構への関与を確認するための関連細胞モデルの開発は不可欠なツールとなっています。さらに、これらのモデルは将来の治療開発にとって非常に重要であり1,2であり、実験における動物の使用を減らすための国際的な勧告と直線的にノックアウト動物モデルの開発に代わる興味深い代替手段を構成する。CRISPR/Cas9を用いた遺伝子編集は、現在利用可能な最も強力なツールの1つであり、多くのノックアウト/ノックインモデルの開発を可能にしており、CRISPR/Cas9を用いた標的遺伝子検証は、CRISPR/Cas93の最も広く使用されているアプリケーションの1つです。遺伝子編集の成功は、CRISPRツール(ガイドRNAおよびヌクレアーゼCas9)を標的細胞モデルに導入する能力にかかっており、筋肉細胞4などの多くのトランスフェクトが困難な細胞では課題になる可能性があります。この課題は、ウイルス、通常はレンチウイルスを使用することで克服することができ、これは効率的に多くの細胞型を形質導入し、その導入遺伝子を送達するという大きな利点を有する。しかし、その主な欠点は、宿主細胞ゲノムにおける導入遺伝子の組み込みであり、組み込み部位に局在する遺伝子の潜在的な変化および導入遺伝子の永久発現をもたらし、ヌクレアーゼCas9の場合、有害な結果をもたらすであろう5。Merienneら6は、Cas9遺伝子自体を標的とするガイドRNAを細胞に導入し、Cas9を不活性化させるスマートなソリューションを提案している。この戦略の適応は、トランスフェクトが困難な細胞内のほぼすべての遺伝子をノックアウトすることを可能にする、ユーザーフレンドリーで汎用性の高いプロトコルとしてここに提示されています。
ここで提示されたプロトコルの目的は、不死化筋肉細胞における目的遺伝子の不活性化を誘導することである。これは、異なるタイプの不死化細胞において、目的の任意の遺伝子をノックアウトするために使用することができる。ここで説明するプロトコルには、ガイドRNAとそのクローニングをレンチウイルスプラスミドに設計する手順、レンチウイルスベクターでCRISPRツールを製造する手順、異なるレンチウイルスで細胞を形質導入する手順、および細胞をクローニングして均質な編集細胞株を作製する手順が含まれています。
このプロトコールを用いて、細胞内カルシウム放出および筋肉収縮に関与する必須カルシウムチャネルである1型リアノジン受容体(RyR1)の欠失を伴う不死化ヒト骨格筋細胞が開発されている7。遺伝子のノックアウト(KO)は、ウェスタンブロットを用いたタンパク質レベルで、カルシウムイメージングを用いた機能レベルで確認されている。
プロトコル
筋肉生検は、欧州の勧告とフランスの法律に従って、研究のための組織銀行(Myobank、EUネットワークEuroBioBank、パリ、フランスのパートナー)から取得されました。書面によるインフォームドコンセントは、すべての個人から取得されました。不死化筋芽細胞は、V. Mouly博士(Myology Institute、パリ、フランス)によって親切に製造され、プロトコルはMyology Institute倫理委員会(MESRI、n AC-2019-3502)によって承認されました。
1. クリスパーガイドデザイン
- 欠失させる遺伝子の領域を特定する。ensembl.org や genome.ucsc.edu などのゲノムブラウザツールを使用してゲノム配列を検索し、2つの領域の染色体座標を決定して、削除する領域の両側にあるガイドRNA(gRNA)を探します。
- ここで用いた遺伝子について、 RYR1遺伝子のFASTA 配列およびエクソン101の配列を以下のようにして取得する。ensemblで、ヒトゲノムの最新バージョンで RYR1 を検索し、最初のエントリを選択して、タンパク質コード配列の転写産物をクリックします。次に、 Exons をクリックして、遺伝子のエクソンのリストにリダイレクトします。
- 「配列 のダウンロード 」をクリックし、「 ゲノム配列のみ」 を選択して、遺伝子全体の完全なコンセンサス配列をダウンロードします。遺伝子のエクソンとイントロンのリストを下にスクロールし、ターゲットのものを選択します。
- 遺伝子中の対応する塩基配列を見つける。欠失させるエクソンのすぐ上流および下流のイントロンの塩基配列を選択し、これをgRNAsの検索に使用する。
- ステップ1.1で特定した領域(削除する領域の上流と下流のイントロン)に、ガイド1とガイド2と呼ばれる2つのgRNAを、Crispor.tefor.net8などのオンラインツールを使用して設計します。数百塩基対(bp)で区切られた2つのgRNAを選択し、各gRNAの配列は、プロトスペーサー隣接モチーフ(PAM)なしで正確に20ヌクレオチド長である。オフターゲットを制限するために利用可能な最良のガイドを選択してください。ガイド設計の例については、 図 1 を参照してください。
- 2つのガイド間の配列は削除されることが予想され、そして目的の遺伝子のノックアウトをもたらすために、したがって、目的の遺伝子中の必須配列または必須エクソンを削除するように2つのガイドの位置を選択する。削除された配列/エクソンが遺伝子の代替転写産物にのみ存在しないこと、および/またはタンパク質の重要な部分をコードしているため、その欠失が機能的なノックアウトをもたらすことを確認してください。
注:Cas9切断部位はプロトスペーサー隣接モチーフ(PAM)の3bp上流で起こると予測されているが、より長い距離での切断も起こり得るため、良好な解決策は、イントロンでの切断などの切断部位の正確な局在化に依存しないことである。
- 2つのガイド間の配列は削除されることが予想され、そして目的の遺伝子のノックアウトをもたらすために、したがって、目的の遺伝子中の必須配列または必須エクソンを削除するように2つのガイドの位置を選択する。削除された配列/エクソンが遺伝子の代替転写産物にのみ存在しないこと、および/またはタンパク質の重要な部分をコードしているため、その欠失が機能的なノックアウトをもたらすことを確認してください。
- 以下の配列を有するように、PAMを含まない各gRNAの逆相補(RC)配列を決定する:ガイド1およびガイド1-RC、ガイド2、およびガイド2-RC。
- 表1に提示したプライマーを順に並べて、プラスミドのクローニングを行った。プロトコール全体を通して、滅菌H2O中で10nMの濃度でプライマーを使用する。
注:これらのプライマー(太字および下線付き)のgRNAに追加された配列は、それぞれガイド、プロモータ、およびトランス活性化クリスパーRNA(tracrRNA)の前後のプラスミドの配列に対応しており、プライマーとプラスミドとの間の良好なオーバーラップを確保するために改変すべきではない。
2. プラスミドクローニング
注:このステップでは、gRNAはレンチウイルス産生のためにプラスミド骨格に挿入されます。2つのgRNAをコードするカセットは、重なり合うプライマーを使用して、連続するポリメラーゼ連鎖反応(PCR)によって最初に産生される。次いで、新しいカセットをレンチウイルス骨格プラスミド#87919に挿入する。
- 以下のプラスミドを得る:レンチウイルスベクター中のCRISPRガイドRNAをコードするプラスミド#87919およびレンチウイルスベクター中のSpCas9配列をコードするプラスミド#87904。
- カセット構造
メモ: クローン作成プロトコルの概要は、 図2.- 2 μLのプラスミド#87919、2 μLのprimer_XmaIF、2 μLのprimer_Guide1R、25 μLのポリメラーゼミックス、および19 μLのH2Oを用いてPCR(A)反応を実行します。 60°Cで30秒、72°Cで1分45秒、72°Cで7分の最終伸びを示した。 工程1.4で説明したプライマーのTmは60°Cである。
注: プログラム 1 の伸び時間はかなり長く見えますが、この伸び時間は、適切なサイズの十分な材料を確実に生産するために選択されています。実際、プラスミド内の配列が繰り返されているため(RNAガイド後のtracrRNAの配列がプラスミド#87919で3回繰り返される)、予想されるDNAのPCR増幅は困難であり、連続したPCRではさらに小さなバンドが生成される。したがって、異なるPCR産物間の競合のために、伸長時間が延長されたか(最も長いものを優先し、最後に十分な精製材料を有するため)、またはタッチダウンPCR(プログラム2)が長い断片(ステップ2.2.6のPCR(最終)について説明したような)に使用された。 - PCR産物をトリスホウ酸塩-EDTAバッファー(TBE)中の1%アガロースゲル上で分離し、300 bp断片を切除して精製します。製造元の指示に従って専用キットを使用して精製を行い、最終容量 20 μL で溶出します。精製したフラグメントを直接使用するか、ステップ 2.2.5 では -20 °C で保存してください。
- PCR プログラム 1 を使用して、プラスミド #87919 2 μL、primer_Guide1F 2 μL、primer_Guide2R 2 μL、ポリメラーゼミックス 25 μL、H2O19 μL で PCR (B) 反応を実行します。TBE中の1%アガロースゲル上のPCR産物を分離し、400 bp断片を20 μLの溶出バッファーで切り取って精製する。精製断片を直接使用するか、ステップ2.2.5のために-20°Cで保存する。
- PCR プログラム 1 を使用して、プラスミド #87919 2 μL、primer_Guide2F 2 μL、primer_BlpIR 2 μL、ポリメラーゼミックス 25 μL、およびH2O 19 μL で PCR (C) 反応を実行します。PCR産物をTBEバッファー中の1%アガロースゲル上で分離する。600 bp フラグメントを 20 μL の溶出バッファーで切り取り、精製します。精製断片を直接使用するか、ステップ2.2.6のために-20°Cで保存する。
注:ステップ2.2.1の注に記載されるように、プラスミド中の反復領域上のプライマーのハイブリダイゼーションのために、900bpでの別の断片が見えることがある。存在する場合は、このバンドを破棄する必要があります。 - PCR プログラム 1 を使用して、2 μL の溶出 PCR A (ステップ 2.2.2 から)、2 μL の溶出 PCR B (ステップ 2.2.3 から)、2 μL のprimer_XmaIF、2 μL のprimer_Guide2R、25 μL のポリメラーゼミックス、および 19 μL のH2O で PCR (D) 反応を実行します。PCR産物をTBEバッファー中の1%アガロースゲル上で分離する;700 bp フラグメントを切除し、20 μL の溶出バッファーで精製します。精製断片を直接使用するか、ステップ2.2.6のために-20°Cで保存する。
注:他のバンドは、反復領域上のプライマーのハイブリダイゼーションのために、>1,000 bp、400 bp、および300 bpで見える場合があり、廃棄する必要があります。 - 4 μL の溶出 PCR C (ステップ 2.2.4 から)、4 μL の溶出 PCR D (ステップ 2.2.5 から)、4 μL のprimer_XmaIF、4 μL のprimer_BlpIR、50 μL のポリメラーゼミックス、および 34 μL のH2O を用いて、PCR (最終) 反応を実行します。使用されるプログラムは、以下のもの(PCRプログラム2)である:98°Cで5分間の初期変性;6サイクル:98°Cで30秒、66°Cで30秒(1サイクルあたり1°Cのハイブリダイゼーション温度の低下)、72°Cで1分45;35サイクル:98°Cで30秒、60°Cで30秒、72°Cで1分45秒、72°Cで5分の最終伸び。
注: この最終増幅の PCR プログラムはタッチダウン PCR であり、各ガイドの直後に 2 つの繰り返し tracrRNA 配列を含む最終増幅のサイズが大きいため、前のものとは異なります。 - PCR産物をTBEバッファー中の1%アガロースゲル上で分離する;最終カセットを切除して精製し、カセットは20 μLの溶出バッファー中で約1,300 bpで移行する。溶出生成物を定量する。精製された断片を直接使用するか、ステップ2.3.2.1のために-20°Cで保存する。これは、レンチウイルスプラスミドに挿入される最終産物である。
注: 他の断片は >1000 bp、400 bp、および 300 bp で見える場合があり、これは廃棄すべき不完全な PCR 断片に対応します。
- 2 μLのプラスミド#87919、2 μLのprimer_XmaIF、2 μLのprimer_Guide1R、25 μLのポリメラーゼミックス、および19 μLのH2Oを用いてPCR(A)反応を実行します。 60°Cで30秒、72°Cで1分45秒、72°Cで7分の最終伸びを示した。 工程1.4で説明したプライマーのTmは60°Cである。
- レンチウイルスプラスミド骨格へのgRNAsカセットの挿入。
- プラスミド#87919を制限酵素XmaIおよびBlpIで二重消化することによってプラスミドを線形化する。
- 15 μL の推奨バッファー、15 μL のプラスミド (1μg/μL)、7.5 μL の BlpI 酵素 (10 U/μL 時)、7.5 μL の XmaI 酵素 (10 U/μL 時)、および112.5μL の H 2 O で反応を調製します。 全量を1%アガロースゲルにロードし、〜10 kbプラスミドを切り出し、適切なキットで精製する。溶出は20μLの緩衝液中で行われ、溶出生成物は光学密度測定を用いて定量される。
注: 大規模な DNA 断片には、Sun および coll9 で説明されているプロトコルなど、適切な精製プロトコルを使用します。
- 15 μL の推奨バッファー、15 μL のプラスミド (1μg/μL)、7.5 μL の BlpI 酵素 (10 U/μL 時)、7.5 μL の XmaI 酵素 (10 U/μL 時)、および112.5μL の H 2 O で反応を調製します。 全量を1%アガロースゲルにロードし、〜10 kbプラスミドを切り出し、適切なキットで精製する。溶出は20μLの緩衝液中で行われ、溶出生成物は光学密度測定を用いて定量される。
- gRNAカセットおよびプラスミドをリゲートする。ステップ2.2.7のgRNAカセットおよびステップ2.3.1.1の直鎖状プラスミドで反応ミックスを調製し、2μLの酵素およびH2Oを加えp_guidesて最終容量が10μLになるようにする。
注:DNAカセット量は50〜100ng、プラスミド量は100〜200ngで、モル比は2:1でなければなりません。
- プラスミド#87919を制限酵素XmaIおよびBlpIで二重消化することによってプラスミドを線形化する。
- 新たに調製したプラスミド 2 μL を使用して、Stbl3 (50 μL) や XL10-Gold などの化学的に有能な 大腸菌 を形質転換し、抗生物質を使用せずに 37 °C で 1 時間増殖させた後、100 μg/mL アンピシリンを含む LB 寒天プレート上に広げます。37°Cで一晩インキュベートする。いくつかのコロニーを選び、メーカーの指示に従って市販のキットを使用してミニ準備を行います。
- PCRプログラム1を用いて、Primer_XmaIおよびPrimer_BlpRでミニプレップDNAに対してPCR増幅を行う( 図2参照)。PCR産物をTBEバッファー中の1%アガロースゲル上で分離する。約1300 bpの予想されるサイズのバンドを持ついくつかのコロニー(〜5)を選択します。
- 選択したコロニーのDNAシークエンシングを行い、Primer_XmaIまたはPrimer_BlpRを用いて、gRNAカセットの正しい挿入を確認した。
注:配列検証されたコロニーの1つが研究でさらに使用され、プラスミドはp_guidesと呼ばれます。 - ステップ2.2(カセット構築)からプライマーキラーFとR、およびプライマー-mCherry FとRを繰り返します。プラスミドはp_Killerと呼ばれます。
3. レンチウイルス産生
- エンドトキシンフリーのマキシプレップキットを使用して、必要なすべてのプラスミドを大量に製造および精製し、製造元の指示に従って精製します。アリコートを 2 μg/μL で調製し、-20 °C で保存する
- 細胞の調製(1日目)
- 10%ウシ胎児血清(FBS)および1%ペニシリン/ストレプトマイシンを添加したダルベッコ改変イーグル培地(DMEM)高グルコースピルビン酸からなる16mL培地に、プレートあたり1 x 106 HEK293細胞を播種した145cmの18プレートを調製する。細胞を37°Cで増幅し、5%CO2インキュベーター中で3日間培養する。
注:レンチウイルスの生産は、使い捨て防護服、保護キャップ、手袋などの適合保護具を使用して、バイオセーフティレベル2の実験室で注意して行う必要があります。すべての実験は、フィルターチップを備えた層流フード(BSLII安全キャビネット)の下で行う必要があります。レンチウイルスを含むすべての溶液およびすべての使用済みプラスチック/ガラス廃棄物は、エタノール70%または他のウイルス不活性化剤で不活性化されなければならない。
- 10%ウシ胎児血清(FBS)および1%ペニシリン/ストレプトマイシンを添加したダルベッコ改変イーグル培地(DMEM)高グルコースピルビン酸からなる16mL培地に、プレートあたり1 x 106 HEK293細胞を播種した145cmの18プレートを調製する。細胞を37°Cで増幅し、5%CO2インキュベーター中で3日間培養する。
- 細胞のトランスフェクション(4日目)
- セルの合流点をチェックして、60%〜65%の合流点に達していることを確認します。
- 目的のプラスミド 20.8 μg (p-guides、p-Killer、または pCas9 #87904)、エンベロープをコードするプラスミド 4.8 μg(VSV-G、#8454)、レンチウイルスパッケージング用のプラスミド psPAX2 (#12260) 20.8 μg、リン酸カルシウム 136 μL を含むトランスフェクション溶液を調製し、H2O で最終容量 1,000 μL に調整します。この溶液を滴下し、攪拌下で 1 mL の 2x HEPES 緩衝生理食塩水 (HBS) に添加します。
注:試薬の最適な調製を確実にするために、すべてのプレートの混合物を同時に調製しないでください。6枚のプレート用のミックスを同時に調製し、例えば、18枚のプレートに対して、6枚のプレート用のミックスを3回調製する。 - 室温(RT)で少なくとも10分間インキュベートし、2mLの溶液を細胞に滴下して加える。プレートを後方、前方、上方、下方に穏やかに攪拌しながらトランスフェクション試薬を均質化し、37°C、5%CO2 で少なくとも5時間インキュベートします。
注:この時点からレンチウイルスの生産が終了するまで、2組目の手袋、保護スリーブ、使い捨てプラストロンを含む追加の保護具を着用してください。 - トランスフェクションの 5 時間後、プレートから培地を取り出し、PBS ですすぎ、トランスフェクション試薬を取り除きます。12mLの新鮮な培地を加え、37°C、5%CO2で48時間インキュベートする。
- ウイルス粒子の採取(6日目)
- すべてのプレートから培地を集めてプールします。800 x g で遠心分離機で4°Cで5分間、細胞破片をペレット化した。上澄み液は0.45 μmフィルターでろ過します(複数のフィルターが必要です)。
- スイングバケットローター内で68,300 x g で4°Cで2時間遠心分離機。上清を取り出し、紙の上の安全キャビネットの下でチューブを逆さまにして5〜10分間、できるだけ多くの液体を除去し、ペレットあたり100μLのHEK増殖培地を加える。4°Cで少なくとも2時間後、ペレットを上下にピペッティングして再懸濁する。再懸濁したすべてのペレットをプールします。
注:ペレットは、分注する前に4°Cで一晩培地中に放置することができる。 - アリコートレンチウイルスを10 μLまたは25 μLのサンプルサイズ(用途に応じて)に組み込み、液体窒素でスナップフリーズします。-80°Cで保存してください。 解凍したアリコートを凍結しないでください。
- LVガイド、LV-KillerおよびLV-Cas9を製造するために、目的の他のプラスミドを用いてステップ3.2~3.4を繰り返します。
注:別の方法として、レンチウイルスは会社またはウイルス施設から購入することができます。
4. レンチウイルス滴定
注:ウイルス滴定はHEK293細胞に対して行われる。滴定は、目的の細胞について、細胞当たりの正確な数のレンチウイルス(レンチウイルスのバッチが何であれ)を後続のステップに組み込むために重要である。細胞を効率的に形質導入するウイルス粒子の数は、感染多重度(MOI)と呼ばれ、MOI 10は細胞あたり10個のウイルス粒子の導入に対応する。凍結/融解サイクルはレンチウイルスの生存率に影響を与えるため、滴定は凍結レンチウイルスアリコートで行われ、その後の各実験は同じプールの新しいアリコートで実行されます。ここでは1つの滴定方法を説明するが、他の方法も使用することができる。
- 1日目に、底面にガラスカバースリップとカバースリップのない2枚の35 mmプレートにプレートあたり1 x 105セルをシードします。
- 2日目に、カバースリップのない2枚のプレートから、トリプシン処理後の細胞数を採取・カウントし、プレート当たりの平均細胞量(N)を求めた。
- 増殖培地で1/10で希釈したレンチウイルス100μLを調製する。5つの培養プレートを、希釈ウイルスの異なる容量(1〜50μLの希釈ウイルス)で形質導入する。このプロトコールでは、次の容量を使用して 5 つのプレートを形質導入します: 1 μL、5 μL、10 μL、20 μL、50 μL。
- 4日目に、カバースリップをRTで20〜30分間4%パラホルムアルデヒド中でインキュベートすることによって細胞を固定する。LV-Cas9について、リン酸緩衝液生理食塩水(PBS)中の0.1%Triton X100でRTで10分間細胞を透過処理し、PBS-0.1%Triton X100-2%ヤギ血清-0.5%ウシ血清アルブミン(BSA)でRTで20分間飽和させ、一次抗体抗V5(希釈率1/400)でRTで45分間標識した後、蛍光二次抗体でRTで30分間インキュベートします。核をヘシュト(PBS中10μg/mL)でRTで10分間標識する。
- カバースリップをスライドに取り付け、20倍の対物レンズを備えた蛍光顕微鏡で観察します。LVガイドおよびLV-Killerの場合、固定後、核標識に直接進み、カバースリップを取り付けます。各カバースリップについて、視野内の核の総数(総細胞数)と標識された細胞数(V5またはmCherryのいずれか)を数え、各カバースリップに対して標識された細胞の割合を求める。
- 形質導入細胞の割合が少なくとも10%であり、かつ50%を超えないカバースリップを選択する。このカバースリップの形質導入効率(X)を決定し、この形質導入効率を得るために使用した希釈ウイルスの体積(V、μL単位)を書き留めます。
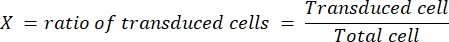
- ウイルスの力価(感染性粒子では、ip/mLとして表される)を以下の式に従って決定する:

希釈係数は、ステップ4.3で行われるレンチウイルスの希釈である。我々は、LV-Cas9で1 x109 ip/mL、LVガイドで1 x1010 ip/mLの最終力価を日常的に取得し、HEK細胞で測定した。
5. 筋芽細胞形質導入
注:不死化筋芽細胞は、以前に産生された3つのレンチウイルスと連続して形質導入される。それらは、20%FBS、2%ペニシリン/ストレプトマイシン、2%ウルトローザーGを添加したハムのF10からなる増殖培地中で50%未満の密度に維持され、37°C、5%CO2で培養される。
- LV-Cas9およびLVガイドの場合はMOI 10、LVキラーの場合はMOI 20で選択した細胞数を治療するために必要なレンチウイルスの量を、以下の式に従って決定します。
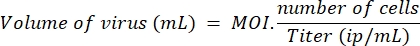
注:並行実験では、コントロールレンチウイルス(lenti-GFP)を用いて筋芽細胞とHEKの形質導入効率を比較し、HEK細胞と比較して筋芽細胞を効率的に形質導入するために5倍のレンチウイルスが必要であることを決定した。したがって、HEK上で測定されたMOI10は、筋芽細胞あたり2つのウイルス粒子に相当する。ここで使用されるMOIは、HEKセル上で計算される。 - 1日目に、1ウェルあたり100μLの増殖培地中に10,000個の細胞を含む96ウェルプレートをシードした。2日目に、ステップ5.1で計算したLVガイドおよびLV-Cas9の適切な容量を加えることによって、安全キャビネットの下の細胞を形質導入する。7日目まで細胞をインキュベーターに戻します。
注:特にLV-Cas9に対して25を超えるMOIを使用しても、高濃度のレンチウイルスで細胞死が多くなるため、編集された細胞数が改善されない可能性があります。 - 7日目に、トリプシン処理を行い、細胞をカウントする。新しいプレートに細胞を40%〜50%のコンフルエントで播種し、細胞をインキュベーターに戻します。5時間後、20のMOIでLV-Killerで細胞を形質導入する(ステップ5.1で計算された体積)。トランスフェクション後5〜10日間、少なくとも2回の継代で細胞を増幅し、常に<50%の低いコンフルエンシーに維持します。細胞は、その増殖が正常に戻ったとき(分裂時間によって推定される)、次のステップである細胞クローニングの準備ができている。
注:増殖は、形質導入後、少し遅くなることがあります。正常な細胞増殖は、細胞の分裂時間の推定によってこの実験手順の前に決定することができる。
6. 細胞クローニング
注:筋芽細胞の形質導入は困難であり、100%の効率に達することはないため、レンチウイルスを使用する場合でも、完全に補正された細胞株を得るためには細胞クローニングが必要です。これは、不死化細胞、または数週間/数ヶ月の間に培養および増幅することができる細胞でのみ可能である。
- トリプシン処理し、細胞をカウントします.増殖培地中の細胞を10細胞/mLで希釈し、100μL/ウェルの培地を含む96ウェルプレートに細胞を1細胞/ウェルで播種する。
注:播種されるプレートの数は、予想される遺伝子編集の確率に依存し、2〜10枚のプレートが日常的に使用される。 - 細胞の成長を監視し、筋芽細胞のコンフルエンシーを50%未満に維持しながら、少なくとも35mmプレートに達するまで、より大きなプレートで各ウェルを徐々に増幅する。このステップは、使用される細胞およびウェル内で単離されると増殖する能力に応じて、2〜6週間続くことができる。
7. クローンの選択
メモ: この手順は、どのクローンが適切に変更されているかを識別するために実行されます。
- 推定的に改変された配列を内包する領域を増幅するために、第1ガイド(Primer_BeforeGuide1F)の前に位置するプライマーと第2ガイド(Primer_AfterGuide2R)の後に位置する別のプライマーからなるプライマーのセットを設計する。ここで用いたプライマーについては 表1 を参照されたい。
- 各クローンから細胞を収集し、将来の増幅のために少なくとも300,000個の細胞を予備にし、残りの細胞の標準的なプロトコルを使用してゲノムDNAを抽出します。
- 多数のクローンが増殖している場合は、編集されていないクローンを破棄し、同じチューブ内の5つのクローンから細胞をプールして迅速な試験を行い、このプールからDNAを抽出し、PCRで試験する。必要な数のクローンで繰り返します。次に、編集された細胞を含むプールをさらに分離して、個々の分析を実行します。
- PCRによる編集を以下のように制御する。1 μL のPrimer_BeforeGuide1F、1 μL のPrimer_AfterGuide2R、12.5 μL のポリメラーゼミックス、3 μL のゲノム DNA、および 7.5 μL のH2Oで PCR 反応を調製します。1%アガロースゲル上で実行し、編集されたクローンを同定する。
- 以下のようにシーケンス処理によって編集を制御します。選択したクローンのサンガーシーケンシングを実行して、削除を確認し、各クローンで編集がどのように実行されたかを特定します。標的遺伝子のみが改変され、観察された生理学的効果に関与していることを確認するために、複数の編集済みクローンを保持し、その後の実験で対照クローン(CTRL)として使用される非編集クローンを保持する。
- 選択したクローンを展開します。各クローンのコンフルエント性が約50%に達したら、細胞をトリプシン処理し、より大きなディッシュに細胞をプレートし、生化学的および機能的特徴付け(通常、クローンあたり1 x106 以上)を実行するのに十分な細胞が産生されるまで、将来の使用のために各クローンの凍結アリコートを保存する。
8. 編集クローンの特性評価
注:DNAシーケンシングによって数クローンをピックアップして確認すると、標的遺伝子の欠失は、ウェスタンブロットを使用してタンパク質レベルで確認でき、この遺伝子に対して機能的な細胞アッセイが利用可能な場合は機能レベルで確認できます。RYR1-KOの場合、 RyR1がカルシウムチャネルであるように、培養細胞に対するカルシウムイメージングを用いて機能特性評価が行われている。
- 編集クローンにおけるタンパク質発現
注:RyR1は、分化した筋管細胞10においてのみ発現している。その発現は、ウェスタンブロットを用いて筋管細胞において評価され、RyR1のタンパク質レベルでの欠失、ならびにCas9タンパク質の欠失を確認した。- ラミニンでコーティングした35mmプレート中の約1.76cm2の表面上の増殖培地(上記、ステップ5)中の200,000細胞のプレート(カルシウムを含むPBS中の10mg/mLでのラミニンの200μL滴に対応する表面)。細胞をプレートに貼り付けたら、37°C、5%CO2で2〜3時間インキュベートした後、DMEM低グルコース+10%馬血清+1%ペニシリン/ストレプトマイシンからなる分化培地に培地をシフトし、細胞をインキュベーターに6日間戻した。
- 分化の6日後、プロテアーゼ阻害剤を添加した200μLのRIPAで細胞を収集し、溶解する。フォリン・ローリー法11を用いてタンパク質濃度を決定する。
- 15μgのタンパク質を、Laemmli変性緩衝液中でRTで30分間変性させた後、5%〜15%の勾配アクリルアミドゲル上にロードする。電気泳動分離後、イモビロンP上のタンパク質を0.8Vで4時間11時間転写する。
- 0.1% Tween 20および5%脱脂粉乳を含むPBS中でRTで30分間メンブレンを飽和させた後、同じ緩衝液で希釈した一次抗体と共にメンブレンをRTで2時間または4°Cで一晩インキュベートし、PBS-0.1% Tween 20でメンブレン5xを5分間洗浄し、メンブレンを二次抗体と共にRTで1時間インキュベートする。用いられる主な抗体は、Cas9を検出するためのV5タグに対する抗体(希釈率:1/5000)、ローディングコントロールとしての抗GAPDH(希釈率:1/1000)、抗RyR1抗体12、13 (希釈率:1/10.000)、DHPRのα1サブユニットに対する抗体(希釈率:1/1000)、およびミオシン重鎖MF20に対する抗体(希釈率:1/1000)である。
- メンブレン5xをPBS-0.1%Tween 20で5分間洗浄し、過剰の液体を乾燥させ、化学発光基質を加える。化学発光シグナルを検出するために、基質提供者の推奨に従って進める。
- 編集されたクローンの機能的特徴付け
注:RyR1の機能は、CTRLまたはKOクローン14から産生された分化筋管におけるカルシウムイメージングを用いて評価した。- ラミニンでコーティングした35mmディッシュの中央にある0.2cm2 の表面(50μLのラミニン滴で覆われた表面、カルシウムを含むPBS中の10mg/mL)に50,000個の細胞をプレートし、ステップ8.1.1に記載されるように6日間分化誘導する。各刺激について3枚のプレートを用意し、生物学的に3連持させる。
- 筋管に50 μLのフルオ4直接を負荷し、分化培地で1:1に希釈し、37°Cで30分間インキュベートした。 1mg/mLのグルコースを添加したKREBS緩衝液で細胞を2回すすいでください。
- 10倍の対物レンズを使用して、倒立蛍光顕微鏡または共焦点顕微鏡で蛍光変動を測定します。プレートを顕微鏡のステージに取り付け、毎秒1フレームで90秒間取得を開始します。
- 残りのKREBSを除去し、膜脱分極のために2mLのKCl(140mM最終濃度)またはRyR1直接刺激のために2mLの4CmC(500μM最終濃度)を加えることによって、フレーム25で細胞を刺激する。記録されたフィールドに少なくとも10本の筋管が存在することを確認してください。
- 各筋管における蛍光変動を、専用のソフトウェアを用いて定量化する。分析のために、ディッシュあたり少なくとも10本の筋管(理想的には1皿あたり20〜30本の筋管)を選択し、各筋管の長軸に線(または関心領域(ROI))を描き、すべてのフレームについてこの線に沿って蛍光Fを収集します。
- フレーム 1 ~ 24 に対応する初期蛍光値 F0 を決定します。蛍光変動(F-F0)/F0を0~90秒の時間関数としてプロットします。実験を3回繰り返して、3つの異なる培養物からの少なくとも90個の筋管細胞からの蛍光変動を得た。90本の筋管のすべての結果をプールし、各時間枠における(F-F0)/F0の平均±SEMを計算します。各刺激および各クローンについてのカルシウム放出のピーク振幅を定量化する。
結果
このプロトコールは、RyR1タンパク質をコードするRYR1遺伝子をノックアウトするために、RyR1が以前に特徴付けられていた健常者15由来の不死化筋芽細胞15(いわゆるHM細胞、ヒト筋芽細胞用)に適用された。ガイドRNAの設計は、遺伝子のエクソン101およびイントロン101の一部を包含する配列を削除するためになされた。エクソン101の一部の欠失は、リー?...
ディスカッション
病態に関与する未知の機能の遺伝子の特性評価への道のりにおける主要なステップは、これらの遺伝子の機能を研究するための関連する細胞モデルの開発である。CRISPR/Cas9を用いた遺伝子編集の利用は指数関数的に成長している研究分野であり、ここで紹介するノックアウトモデルの開発は、その最も広く使用されているアプリケーションの1つです。この文脈において、我々はここで、関心?...
開示事項
著者らは、開示する利益相反はありません。
謝辞
この研究は、Association Française contre les myopathies(AFM-Téléthon)とAuvergne-Rhône Alpes Région(AURA)からの助成金によって資金提供されました。
資料
| Name | Company | Catalog Number | Comments |
| Anti-CACNA1S antibody | Sigma-Aldrich | HPA048892 | Primary antibody |
| Blp I | NE BioLabs | R0585S | Restriction enzyme |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | Transfection kit |
| Easy blot anti Mouse IgG | GeneTex | GTX221667-01 | HRP secondary antibody |
| Easy blot anti Rabbit IgG | GeneTex | GTX221666 | HRP secondary antibody |
| Fluo-4 direct | Molecular Probes | F10472 | Calcium imaging |
| GAPDH(14C10) Rabbit mAb | Cell Signaling Technology | #2118 | Primary antibody |
| HindIII | Fermentas | ER0501 | Restriction enzyme |
| InFusion HD Precision Plus | Takara | 638920 | Ligation kit |
| MasterMix Phusion High Fidelity with GC | ThermoFisher Scientific | F532L | Mix for PCR reaction with High fidelity Taq polymerase and dNTPs |
| Myosin Heavy Chain antibody | DHSB | MF20 | Primary antibody |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | REF 740424 | Maxipreparation kit for purification of plasmids |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609 | DNA purification |
| NucleoSpin Tissue | Macherey-Nagel | 740952 | Kit for DNA extraction from cell |
| One Shot Stbl3 Chemically Competent E. coli | ThermoFisher Scientific | C737303 | Chemically competent cells |
| Plasmid #87904 | Addgene | 87904 | Lentiviral plasmid encoding the SpCas9 (for LV-Cas9) |
| Plasmid #87919 | Addgene | 87919 | Lentiviral backbone for insertion of cassette with guides (for LV-guide-target) |
| Plasmid #12260 | Addgene | 12260 | Lentiviral plasmid encoding lentiviral packaging GAG POL |
| Plasmid #8454 | Addgene | 8454 | Lentiviral plasmid encoding envelope protein for producing lentiviral and MuLV retroviral particles |
| V5 Tag Monoclonal Antibody | Invitrogene | R96025 | Primary antibody |
| XL10-Gold Ultracompetent Cells | Agilent | 200317 | Chemically competent cells |
| Xma I | NE BioLabs | R0180S | Restriction enzyme |
参考文献
- Claussnitzer, M., Susztak, K. Gaining insight into metabolic diseases from human genetic discoveries. Trends in Genetics. 37 (12), 1081-1094 (2021).
- Fuster-García, C., García-Bohórquez, B., Rodríguez-Muñoz, A., Millán, J. M., García-García, G. Application of CRISPR tools for variant interpretation and disease modeling in inherited retinal dystrophies. Genes. 11 (5), 473 (2020).
- Modell, A. E., Lim, D., Nguyen, T. M., Sreekanth, V., Choudhary, A. CRISPR-based therapeutics: current challenges and future applications. Trends in Pharmacological Sciences. 43 (2), 151-161 (2022).
- Olson, E. N. Toward the correction of muscular dystrophy by gene editing. Proceedings of the National Academy of Sciences of the United States of America. 118 (22), (2021).
- Wu, X., Kriz, A. J., Sharp, P. A. Target specificity of the CRISPR-Cas9 system. Quantitative Biology. 2 (2), 59-70 (2014).
- Merienne, N., et al. The self-inactivating KamiCas9 system for the editing of CNS disease genes. Cell Reports. 20 (12), 2980-2991 (2017).
- Marty, I., Fauré, J. Excitation-contraction coupling alterations in myopathies. Journal of Neuromuscular Diseases. 3 (4), 443-453 (2016).
- Concordet, J. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Research. 46, 242-245 (2018).
- Sun, Y., Sriramajayam, K., Luo, D., Liao, D. J. A quick, cost-free method of purification of DNA fragments from agarose gel. Journal of Cancer. 3, 93-95 (2012).
- Flucher, B. E., Conti, A., Takeshima, H., Sorrentino, V. Type 3 and type 1 ryanodine receptors are localized in triads of the same mammalian skeletal muscle fibers. The Journal of Cell Biology. 146 (3), 621-630 (1999).
- Hess, H. H., Lees, M. B., Derr, J. E. A linear Lowry--Folin assay for both water-soluble and sodium dodecyl sulfate-solubilized proteins. Analytical Biochemistry. 85 (1), 295-300 (1978).
- Garibaldi, M., et al. Dusty core disease' (DuCD): expanding morphological spectrum of RYR1 recessive myopathies. Acta Neuropathologica Communications. 7 (1), 3 (2019).
- Marty, I., et al. Biochemical evidence for a complex involving Dihydropyridine receptor and Ryanodine receptor in triad junctions of skeletal muscle. Proceedings of the National Academy of Sciences of the United States of America. 91 (6), 2270-2274 (1994).
- Oddoux, S., et al. Triadin deletion induces impaired skeletal muscle function. Journal of Biological Chemistry. 284 (50), 34918-34929 (2009).
- Mamchaoui, K., et al. Immortalized pathological human myoblasts: towards a universal tool for the study of neuromuscular disorders. Skeletal Muscle. 1, 34 (2011).
- Cacheux, M., et al. Functional characterization of a central core disease RyR1 mutation (p.Y4864H) associated with quantitative defect in RyR1 protein. Journal of Neuromuscular Diseases. 2 (4), 421-432 (2015).
- Luis, A. The old and the new: Prospects for non-integrating lentiviral vector technology. Viruses. 12 (10), 1103 (2020).
- Leenay, R. T., Beisel, C. L. Deciphering, communicating, and engineering the CRISPR PAM. Journal of Molecular Biology. 429 (2), 177-191 (2017).
- Salmon, P., Trono, D. Production and titration of lentiviral vectors. Current Protocols in Neurosciences. , (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved