このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
モデル線虫の天然微生物叢の分離と特性評価
要約
Caenorhabditis elegans は生物学の主要なモデル種の1つですが、ほとんどすべての研究は、その自然に関連する微生物の不在下で行われています。ここで説明する方法は、将来の機能的 C.エレガンス 研究の基礎として、関連する微生物の多様性の理解を深めるのに役立ちます。
要約
線虫カ エノラブディティスエレガンスは 、自然界の多種多様な微生物と相互作用します。一般に、 C.エレガンスは 腐った植物、特にリンゴのような腐った果物や堆肥の山によく見られます。また、ナメクジやウッドスライスなどの特定の無脊椎動物の宿主にも関連しています。これらの生息地は微生物が豊富で、 C.エレガンスの 餌として機能し、線虫の腸に持続的にコロニーを形成することもできます。今日まで、生息地や地理的な場所を超えた在来の C.エレガンス 微生物叢の正確な多様性と一貫性は完全には理解されていません。ここでは、 C.エレガンスを 自然から分離し、ワームの微生物叢を特徴付けるための適切なアプローチについて説明します。線虫は、堆肥材料、腐ったリンゴ、ナメクジから簡単に分離したり、リンゴを堆肥の山に置くことによって引き付けることができます。北半球で C.エレガンス を見つけるのに最適な時期は9月から11月です。ワームは、基質を緩衝液に浸し、続いて線虫を捕集し、その後の分析のために線虫増殖培地またはPCR緩衝液に移すことによって、収集された基質材料から洗い流すことができます。さらに、サンプルを使用してワーム関連微生物を分離および精製し、微生物叢群集組成の16SリボソームRNA分析のためにワームを処理する方法を示します。全体として、記載された方法は、生息地および地理的位置にわたる C.エレガンス 微生物叢の特性評価に関する新しい研究を刺激し、それによって将来の機能研究の基礎として線虫の微生物叢の多様性と安定性の包括的な理解を得るのに役立つ可能性があります。
概要
自然界では、C.エレガンスは腐った植物、特にリンゴのような腐った果物や堆肥の山によく見られます1。また、ナメクジやウッドスライスなどの特定の無脊椎動物の宿主にも関連しています2,3。これらの生息地は微生物が豊富で、ワームの餌として役立つだけでなく、それと安定した関係を形成する可能性もあります。天然関連微生物の多様性に関する情報は、2016年にのみ公開されました4,5,6。それ以来、これらおよびほんの数件の最近の研究により、C.エレガンスはさまざまな細菌および真菌と関連していることが明らかにされており、最も一般的にはシュードモナス属、エンテロバクター属、オクロバクトラム属、エルウィニア属、コマモナス属、グルコノバクター属、および他のいくつかの細菌が含まれます6,7,8。いくつかの関連細菌は、すべてではありませんが、ワーム腸に安定してコロニーを形成することができます6,9,10,11,12。それらは、栄養を提供し、病原体やおそらく他のストレッサーから保護し、生殖率、発達、行動反応などの中心的な生活史形質に影響を与える可能性があるため、C.エレガンスの生物学を理解する上で非常に重要である可能性があります。
一例として、シュードモナス属、オクロバクトラム属、およびエンテロバクター属またはグルコノバクター属の天然会合分離株は、異なる方法で病原体感染および死滅からワームを保護することができる5、6、11、13、14。コマモナス属の特定の分離は、線虫の食事反応、発達、寿命、および生殖能力に影響を与えます15,16,17。プロビデンシア属細菌は神経調節物質チラミンを産生し、それによって宿主の神経系の活動とその結果生じる行動反応を調節します18。一連の異なる天然関連細菌が、集団成長率、生殖能力、および行動反応に影響を与えることが実証されました5、6、9、11、19。
今日まで、生息地や地理的な場所にわたる在来のC.エレガンス微生物叢の正確な多様性と一貫性は完全には理解されておらず、ワームとその環境からの微生物とのさらなる関連はまだ明らかにされていません。以前のいくつかの研究では、いくつかの土壌環境、自然のC.エレガンスの生息地、またはC.エレガンスの実験室株4,5,20を使用したメソコズム実験(つまり、自然の生息地を再現するラボベースの環境)から分離された細菌株を使用していました。これらの研究は、特定の線虫の形質(例えば、線虫代謝21)に対する微生物の影響に関する新しい洞察を得たが、これらの相互作用が自然界のC.elegans生物学との関連性は不明である。したがって、この原稿は、C.エレガンスを自然から直接分離し、単一のワームとワームのグループの両方から自然に関連する微生物を分離し、その後特徴付ける方法を説明しています。記載された方法は、天然のC.エレガンスおよびその天然微生物叢の単離および特性評価のために以前に使用された手順の更新および改良版である2,6,7。C.エレガンスが世界中の植物物質の分解に広く見られることを考えると(特に腐った果物、温帯地域、および秋)1,2,22,23,24,25、このプロトコルは、C.エレガンスの関連に関心があるときはいつでも、どのラボでも適用できます。 自然に関連する微生物への形質、したがってより自然に関連するコンテキスト。後者は、関連する微生物叢が多様な生活史特性に影響を与える可能性があることが他の宿主システムの多様性から知られているため、線虫の生物学を完全に理解するために極めて重要です26、現在、ほぼすべての生命科学分野にわたる多数のC.エレガンス研究でほとんど無視されている側面。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. バッファーと培地の調製
- 5.85 gのNaCl、1.123 gのK2HPO 4、5.926 gのKH2 PO4、および1 Lの脱イオン化H2Oをフラスコとオートクレーブに加え、121°Cで20分間、Sバッファーを調製します。
- 1.2%(w/v)ヒドロキシメチルセルロース(培地の粘度の原因となる物質)、5 mg/mLコレステロール、1 mM MgSO4、1 mM CaCl2、および0.1%(v/v)アセトンを含むSバッファーを加えて粘性培地を調製します。オートクレーブし、粘性媒体が完全に均質になるまで撹拌する。
注: これには数時間かかる場合があります。また、S-バッファーは、事前の滅菌なしで粘性媒体に直接調製して添加することができます。 - 3 gのKH 2 PO 4、6 gのNA 2 HPO4·2 H 2 O、5 gのNaCl、および1 Lの脱イオン化H2Oを1 Lフラスコに加えることにより、M9バッファーを調製します。溶液をオートクレーブし、冷却後、1mLの1M MgSO 4(500mLの脱イオンH 2 O中に123.24gのMgSO4・7H2O、滅菌済み)を加える。
- 5 mLのTriton X-100と45 mLのM9バッファーを混合して、10%(v/v)のTriton X-100ストック溶液を調製します。0.2 μmフィルターを使用して溶液をフィルター滅菌します。
- オートクレーブ処理後、10% (v/v) Triton X-100 ストック溶液 2.5 mL を M9 バッファー 1 L に添加して Triton X-100 (M9-T) を含む M9 バッファーを調製し、0.025% (v/v) M9-Tを得ます。
- 50 mLチューブで15 mLの滅菌100%グリセロールと35 mLの滅菌Sバッファーを混合することにより、Sバッファー中の30%(v/v)グリセロールを調製します。
2. 環境サンプルの作製(図1)
- 堆肥や腐った果物などの環境サンプルを収集し、各サンプルを個別のビニール袋、チューブ、またはその他の清潔な容器に入れます。
注:線虫を引き付けるために、サンプリングの数週間前にリンゴを堆肥に置くことができます。 - 環境サンプルの断片を空の滅菌済み9 cmのペトリ皿に均等に広げます。
注:崩壊レベルが高いサンプルには 、C.エレガンス が含まれている可能性が高くなります。オプションで、ペプトンフリー寒天培地(PFM)を充填したペトリ皿を使用してコントラストを高めることができます。 - サンプルを約20 mLの滅菌粘性培地で注意深く覆います。
注:線虫は1〜2時間以内に表面に浮かびます。粘性媒体はワームの動きを遅くし、それらをサンプリングするのを容易にします。滅菌M9バッファーを代替として使用することができますが、ワームの移動は速くなります。
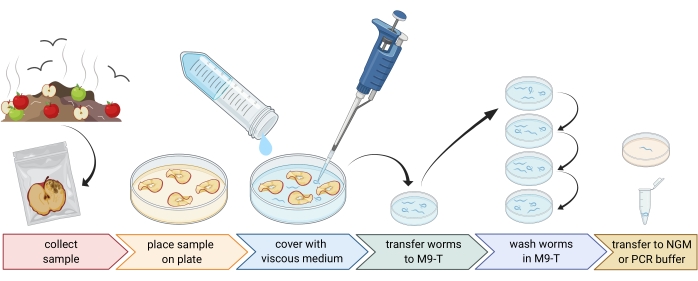
図1:基質からの線虫の分離。 基質サンプルを空のペトリ皿に入れ、粘性培地で覆って線虫を洗い流します。線虫をM9-Tに移し、洗浄を繰り返して外部から細菌を除去します。個々の線虫は、DNA単離、関連細菌の単離、または寒天プレート上に配置してワーム集団を培養するために使用することができる。BioRender.com で作成された図。 この図の拡大版を表示するには、ここをクリックしてください。
3. カエノラブディティス 線虫の分離(図1)
- BarrièreとFélix27によるC.エレガンスおよび関連する線虫の分離に関するWormBookの章で提示されたガイドラインに従って、解剖顕微鏡を使用してCaenorhabditis線虫を検索します。
注: カエノラブディティス雌 雄同体/女性は、(透過照明下で)薄茶色の腸を持っています。さらに、腸細胞には大きな細胞核があり、白い点として見えます。対照的に、他の線虫種はしばしば暗褐色の腸を持ち、色素強度において前後の非対称性を示し、通常は目に見える核を示さない。成体の カエノラブディティス 雌雄同体/雌の外陰部は、動物の中央にあります。この外陰部の局在は、他の線虫分類群によって常に示されるわけではありません。 Caenorhabditis 線虫は2つの咽頭球根を有し、それらは両方とも十分な倍率および透過照明および他の多くの線虫分類群とのコントラストで見える。 カエノラブディティス 線虫は尖った尾を持っていますが、他のいくつかの線虫は丸い尾を持っています。 - カエノラブディティス線虫を解剖顕微鏡下で、20〜100 μLのピペットを使用してできるだけ少ない液体で収集します。採取した線虫を滅菌3cmのペトリ皿に入れた滅菌M9-T1-3mLに直接移し、ワームを洗浄して付着していない微生物を除去するか(ステップ3.3)、あるいは個々の線虫を線虫増殖培地(NGM)を含むプレートに移して線虫集団を確立します(ステップ3.4)。
- 線虫を洗って、線虫の外側から微生物を取り除きます。
注:このプロトコルは腸内細菌を濃縮しますが、ワームキューティクルに付着する細菌を完全に排除するわけではありません。- 線虫をM9-Tで10〜15分間インキュベートします。
- 線虫をできるだけ少ない液体で、新しい滅菌3 cmペトリ皿に入れて1〜3 mLの新鮮なM9-Tにピペットで入れます。
- インキュベーションを繰り返し、線虫をさらに2回新鮮なM9-Tに移します。
注: 次の手順は、個々のワームまたはワーム集団のいずれかで実行できます。ワームは、 C.エレガンス および関連する微生物の特性評価、および細菌の分離に使用できます(ステップ4および5)。あるいは、それらをNGMプレートに個別に移して、ワーム集団を確立することもできます(ステップ3.4)。
- 線虫集団を得るには、個々の線虫をNGMプレートにピペットで貼り付けます。
注:C. エレガンス や C.ブリグサエ などの雌雄同体線虫だけが、単一のワームから子孫を産むことができます。ただし、他の種の単一のワームは、すでに交配されている場合でも子孫を産む可能性があります。- 自然に分離された線虫は通常、腸内に細菌を含み、それをNGMプレートに流し、そこでこれらの細菌が増殖し、 C.エレガンスの食物として利用できます。標準的な実験用食品生物である 大腸菌 株OP50のような個別の食品生物を追加しないでください。
注:プレート上でワームを飼育することは、関連する細菌群集の組成に影響を与えますが、それでも組成は天然のC.エレガンス分離株の組成に匹敵します6,7。 - 線虫を適切な温度で最大10日間増殖させます(たとえば、温帯の場所では15〜20°C)。これらの線虫を凍結するか(ステップ3.5)、 C.エレガンス および関連する微生物の特性評価、および微生物の分離(ステップ4および5)に使用します。
- 自然に分離された線虫は通常、腸内に細菌を含み、それをNGMプレートに流し、そこでこれらの細菌が増殖し、 C.エレガンスの食物として利用できます。標準的な実験用食品生物である 大腸菌 株OP50のような個別の食品生物を追加しないでください。
- 長期保存のための凍結線虫
- 食用細菌が消えるまで線虫をプレート上に置いておき、プレート上には主に小さな幼虫期があります。1.5 mLのSバッファーでワームをプレートから洗い流します。
- 500 μLのワーム含有Sバッファーと500 μLの30%(v / v)グリセロールをSバッファー中で滅菌2 mLチューブで完全に混合します。.長期保存のために-80°Cでチューブを速やかに凍結してください、さもなければグリセロールは線虫に害を及ぼすかもしれません。
4. C.エレガンス および微生物の分子同定のためのワームの調製
- 線虫関連微生物を公平に同定するには、滅菌済み1 mmビーズ3個、PCRバッファー19.5 μL、およびウェルあたり0.5 μLのプロテイナーゼK(20 mg/mL)を含む96ウェルプレートを調製します。個々の洗浄した線虫を各ウェルにピペットで入れ、できるだけ少ない液体を移します。
- ワーム集団も使用できます。このために、M9バッファーでプレートからワームを洗浄し、~300 μLのワーム含有M9バッファーを10〜15個のビーズを含む2 mLチューブに移します。
- ビーズホモジナイザーを使用して線虫を分解します(例:30Hzで3分間ビーズを叩きます)。プレートまたはチューブを短時間遠心分離して、液体を底に運びます(例:室温[RT]で8000 x g で10秒間)。
- C.エレガンスの同定
- サンプルをPCRサイクラーで50°Cで1時間、95°Cで15分間加熱することにより、個々の線虫のDNAを分離します。 任意の単離方法を用いて、線虫集団のDNAを単離する(市販キット7、9を用いた異なる単離方法のプロトコルの例)。長期保存のためにDNAを-20°Cで凍結します。
- C. elegansの同定には、選択したTaqサプライヤーの指示に従って、DNAとプライマーペアnlp30-F(表1、5'-ACACATACAACTGATCACTCA-3')およびnlp30-R(表1、5'-TACTTTCCCCATCCGTATC-3')をPCRで使用します。
- 次のPCR条件を使用します:95°Cで2分間の初期変性ステップ、続いて95°Cで45秒間、55°Cで30秒間、72°Cで1分間、および72°Cで5分間の最終伸長ステップを35サイクルします。 エレ ガンスは 154 bpのPCR産物を生成します。
- 単離されたDNAを使用して、V3-V4領域の16Sアンプリコンシーケンシング を介して 線虫関連細菌を特徴付けます。
- 選択した16Sプライマーを使用して16Sライブラリを調製し、ライブラリ調製キットのプロトコルに従います。1つの選択肢は、16S rRNA遺伝子のV3-V4領域をカバーするプライマー341F(表1、5'-CCTACGGGNGGCWGCAG-3')および806R(表1、5'-GACTACHVGGGTATCTAATCC-3')を使用することであり、その結果、良好な分解能7の標準データベースで分類できる配列が得られる。
注:このステップでは、入力DNAの量が重要です。単一のワームから取得されたDNAは、ワーム集団から取得されるDNAよりもはるかに少なくなります。単一のワームの場合、入力DNAの量を増やすか、PCRサイクルの量を増やす必要がある場合があります7。 - ライブラリは、適切なシーケンシングキットを使用してシーケンシングプラットフォーム上でシーケンシングできます。
注:ここでは、MiSeqプラットフォームを適切なMiSeq試薬キットとともに使用します。試薬は絶えず改良されており、最新の基準に合わせて選択する必要があります。
- 選択した16Sプライマーを使用して16Sライブラリを調製し、ライブラリ調製キットのプロトコルに従います。1つの選択肢は、16S rRNA遺伝子のV3-V4領域をカバーするプライマー341F(表1、5'-CCTACGGGNGGCWGCAG-3')および806R(表1、5'-GACTACHVGGGTATCTAATCC-3')を使用することであり、その結果、良好な分解能7の標準データベースで分類できる配列が得られる。
5. 線虫関連細菌の単離と培養(図2)
- 細菌を単離するには、3つの滅菌1 mmビーズ、ウェルあたり20 μLのM9バッファーを含む96ウェルプレートを準備し、個々の洗浄した線虫を各ウェルにピペットで移し、できるだけ少ない液体を移します。
注:あるいは、M9バッファーでプレートからワーム集団を洗浄し、~300 μLのワーム含有M9バッファーを10〜15個のビーズを含む2 mLチューブに移すことができます。すべての場合において、ワームは、手順4.1〜4.4を使用して C.エレガンス であると特定された培養物から来ている必要があります。 - ビーズホモジナイザーを使用して線虫を分解します(例:30Hzで3分間ビーズを叩きます)。プレートまたはチューブを短時間遠心分離して、液体を底に運びます(たとえば、RTで8000 x g で10秒間)。
注:この方法を使用すると、16S rDNAアンプリコンシーケンシング7で明らかになったものと同様の細菌分類群が単離され、記載されたビーズビートがほとんどの細菌細胞を無傷のままにすることを示唆しています。 - 上清を回収し、1:10で連続希釈し、9cm寒天プレートに最大100μLをプレートします。
- ほとんどの細菌を確実に培養できるようにするには、希釈トリプチカーゼ大豆寒天培地(TSA、1:10希釈)、マッコンキー寒天培地、サブローグルコース寒天培地、ジャガイモデキストロース寒天培地、酵母ペプトンデキストロース寒天培地など、さまざまな栄養組成のさまざまな代替培地を使用します。
- サンプリング場所の平均温度条件(たとえば、温帯の場所では15〜20°Cの温度)でプレートを24〜48時間インキュベートします。
- 標準的な3ストリーク技術と滅菌ループを使用して、純粋な細菌培養物を取得します(図2)。
- 滅菌ループまたはつまようじを使用してプレートから単一のコロニーを選択し、精製に使用したのと同じ寒天培地を含む新しい寒天プレートにストリークします。プレートの約1/3のみを使用してください。
- 新しい滅菌ループを使用するか、再利用可能なループを滅菌して最初のストリークにドラッグし、同じプレートの別の1/3に2番目のストリークを作成します。
- 滅菌ループを2番目のストリークにドラッグして、この手順を繰り返します。
- 単離に使用したのと同じ増殖条件でプレートをインキュベートします。この技術は、3番目の縞の領域で単一のコロニーを成長させる結果に違いありません。
注:天然の分離株はバイオフィルムや凝集体を形成する傾向があるため、精製ステップを複数回繰り返す必要がある場合があります。
- 上記と同じ温度と成長時間を使用して、液体培地(寒天培地と同じタイプ)で純粋なコロニーを成長させます(ステップ5.4)。
- 300 μLの細菌培養物を200 μLの86%(v/v)グリセロール(TSBなどのそれぞれの増殖培地中)に加え、ピペットで上下に適切に混合して、細菌ストックを準備します。あるいは、50 μLの細菌培養物と50 μLの7%(v/v)DMSOを混合してDMSOストックを調製します。長期保存のために-80°Cで凍結してください。
- 完全な16SリボソームRNA遺伝子のシーケンシングを使用した細菌の特性評価
- 適切な技術を使用して純粋な液体培養物から細菌DNAを抽出する(例えば、DNA抽出キット;経験から、CTABベースの抽出プロトコルは非常にうまく機能する22)。
- プライマー27F(表1、5'-ガガットガトガトグCTGGCTCAG-3')および1495R(表1、5'-CTACGGCTACCTTGTTACGA -3')28を用いて16S rRNA遺伝子を増幅し、以下のPCR条件:95°C、2分、22倍(95°C、30秒;55°C、30秒;72°C、100秒)、および72°C、5分での最終伸長期間。
- 完全な配列を取得するには、701F(表1、5'-GTGTAGCGGTGAAATGCG-3')および785R(表1、5'-GGATTAGATACCCTGGTAGTCC-3')などの2つの内部シーケンシングプライマーを追加で使用します6。
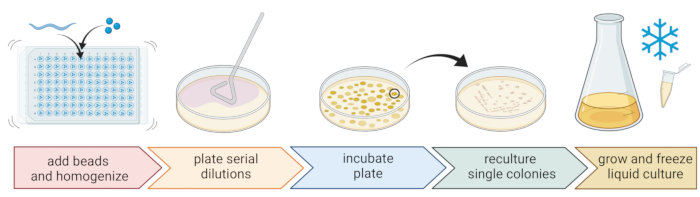
図2:個々の細菌の種の同定と分離。 個々の線虫はビーズホモジナイザーを使用して分割され、DNAはPCRまたはシーケンシング による 種決定のために単離されます。あるいは、分割された線虫材料を連続的に希釈し、成長培地プレート上に播種する。プレートは細菌のコロニーが現れるまでインキュベートされ、単一のコロニーは純粋な培養物を得るために新しいプレートにストリークされます。純粋培養物の単一コロニーは、-80°Cでの長期保存のための細菌ストックの調製のための液体細菌培養物を増殖させるために使用されます。 BioRender.com で作成された図。 この図の拡大版を表示するには、ここをクリックしてください。
Access restricted. Please log in or start a trial to view this content.
結果
線虫C.エレガンスは、リンゴなどの果物の分解や堆肥サンプルに頻繁に見られます。北ドイツでは、主に9月から11月2日まで、C. elegansと同属種(特にC. remaneiだけでなくC. briggsae)が見られます。線虫は、植物物質、特にリンゴやナシなどの腐った果物、および堆肥、特に高グレードの分解を示す材料に最も一般的に見られます。果物と堆肥のサンプル?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
線虫カエノラブディティスエレガンスは、生物学研究で最も集中的に研究されているモデル生物の1つです。これは、もともと神経系の発達と機能を理解するために、1960年代にシドニーブレナーによって導入されました29。それ以来、C. elegansは、行動生物学、神経生物学、老化、進化生物学、細胞生物学、発生生物学、免疫学など、すべての生物学分野にわた?...
Access restricted. Please log in or start a trial to view this content.
開示事項
私たちは、利益相反がないことを宣言します。
謝辞
我々は、ドイツ科学財団(メタ生物の起源と機能に関する共同研究センター1182のプロジェクトA1.1及びA1.2)からの財政的支援を認める。シュレンブルク研究所のメンバーのアドバイスとサポートに感謝します。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| COMSOL | COMSOL | multiphysics simulation software |
参考文献
- Schulenburg, H., Félix, M. -A. The natural biotic environment of Caenorhabditis elegans. Genetics. 206 (1), 55-86 (2017).
- Petersen, C., Dirksen, P., Prahl, S., Strathmann, E. A., Schulenburg, H. The prevalence of Caenorhabditis elegans across 1.5 years in selected North German locations: the importance of substrate type, abiotic parameters, and Caenorhabditis competitors. BMC Ecology. 14 (1), 4(2014).
- Petersen, C., et al. Travelling at a slug's pace: possible invertebrate vectors of Caenorhabditis nematodes. BMC Ecology. 15 (1), 19(2015).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38(2016).
- Johnke, J., Dirksen, P., Schulenburg, H. Community assembly of the native C. elegans microbiome is influenced by time, substrate and individual bacterial taxa. Environmental Microbiology. 22 (4), 1265-1279 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485(2017).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3 Genes|Genomes|Genetics. 10 (9), 3025-3039 (2020).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2019).
- Kissoyan, K. A. B., et al. Exploring effects of C. elegans protective natural microbiota on host physiology. Frontiers in Cellular and Infection Microbiology. 12, 775728(2022).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10 (1), 604(2019).
- Kissoyan, K. A. B., et al. Natural C. elegans microbiota protects against infection via production of a cyclic lipopeptide of the viscosin group. Current Biology. 29 (6), 1030-1037 (2019).
- Watson, E., MacNeil, L. T., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Integration of metabolic and gene regulatory networks modulates the C. elegans dietary response. Cell. 153 (1), 253-266 (2013).
- Watson, E., et al. Interspecies systems biology uncovers metabolites affecting C. elegans gene expression and life history traits. Cell. 156 (4), 759-770 (2014).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Diet-induced developmental acceleration independent of TOR and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- O'Donnell, M. P., Fox, B. W., Chao, P. -H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Snoek, B. L., et al. A multi-parent recombinant inbred line population of C. elegans allows identification of novel QTLs for complex life history traits. BMC Biology. 17 (1), 24(2019).
- Avery, L., Shtonda, B. B. Food transport in the C. elegans pharynx. Journal of Experimental Biology. 206 (14), 2441-2457 (2003).
- Zhang, J., et al. A delicate balance between bacterial iron and reactive oxygen species supports optimal C. elegans development. Cell Host & Microbe. 26 (3), 400-411 (2019).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Félix, M. -A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10 (1), 59(2012).
- Barrière, A., Félix, M. -A. Temporal dynamics and linkage disequilibrium in natural Caenorhabditis elegans populations. Genetics. 176 (2), 999-1011 (2007).
- Dolgin, E. S., Félix, M. -A., Cutter, A. D. Hakuna Nematoda: genetic and phenotypic diversity in African isolates of Caenorhabditis elegans and C. briggsae. Heredity. 100 (3), 304-315 (2008).
- Douglas, A. E. Simple animal models for microbiome research. Nature Reviews Microbiology. 17 (12), 764-775 (2019).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and related nematodes. WormBook. , ed. The C elegans research community 1-19 (2014).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. Molecular Ecology. 31 (8), 2327-2347 (2022).
- Haber, M. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: Evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2004).
- Watson, E., et al. Metabolic network rewiring of propionate flux compensates vitamin B12 deficiency in C. elegans. eLife. 5, 17670(2016).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved