このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
イメージングフローサイトメトリーと人工知能を用いた小核アッセイの自動化
要約
小核(MN)アッセイは、DNA損傷を定量化するための確立されたテストです。ただし、手動顕微鏡や特徴ベースの画像分析などの従来の手法を使用してアッセイをスコアリングすることは、面倒で困難です。この論文では、イメージングフローサイトメトリーデータを使用してMNアッセイをスコアリングするための人工知能モデルを開発する方法論について説明します。
要約
小核(MN)アッセイは、化学物質の遺伝毒性を評価するために世界中の規制機関によって使用されています。アッセイは2つの方法で実施することができる:一度分裂した、細胞質分裂がブロックされた二核細胞または完全に分裂した単核細胞におけるMNをスコアリングすることによって。歴史的に、光学顕微鏡はアッセイをスコアリングするためのゴールドスタンダードの方法でしたが、それは面倒で主観的です。フローサイトメトリーは、近年、アッセイのスコアリングに使用されていますが、細胞画像の重要な側面を視覚的に確認できないため、制限されています。イメージングフローサイトメトリー(IFC)は、ハイスループット画像キャプチャと自動画像解析を組み合わせたもので、MNアッセイのすべての重要なイベントの画像を迅速に取得し、スコアリングするために成功裏に適用されています。最近、畳み込みニューラルネットワークに基づく人工知能(AI)手法を使用して、IFCが取得したMNアッセイデータをスコアリングできることが実証されました。このホワイトペーパーでは、AIソフトウェアを使用してディープラーニングモデルを作成し、すべての主要なイベントをスコアリングし、このモデルを適用して追加データを自動的にスコアリングするためのすべての手順について説明します。AIディープラーニングモデルの結果は、手動顕微鏡と比較して優れているため、IFCとAIを組み合わせることでMNアッセイの完全に自動化されたスコアリングが可能になります。
概要
小核(MN)アッセイは、ヒト用の化粧品、医薬品、および化学物質の開発におけるDNA損傷を評価するための遺伝毒物学の基本です1,2,3,4。小核は、分裂後に核に取り込まれず、核とは別の小さな円形体に凝縮する染色体全体または染色体断片から形成されます。したがって、MNは、遺伝毒性試験1におけるDNA損傷を定量するためのエンドポイントとして使用できます。
MNを定量するための好ましい方法は、サイトカラシン-B(Cyt-B)を用いて分裂をブロックすることによって一回分裂した二核細胞(BNC)内である。このバージョンのアッセイでは、細胞毒性は、単核(MONO)および多核(POLY)細胞のスコアリングによっても評価されます。このアッセイは、ブロックされていないMONO細胞におけるMNをスコアリングすることによっても実行することができ、これはより速く、より簡単にスコアリングすることができ、増殖を評価するために曝露前および曝露後の細胞カウントを使用して細胞毒性を評価する5,6。
アッセイの物理的スコアリングは、すべての重要なイベントを視覚的に確認できるため、歴史的に手動顕微鏡によって行われてきました。しかし、手動顕微鏡は困難で主観的です1。そのため、顕微鏡スライドスキャンやフローサイトメトリーなどの自動化技術が開発されていますが、それぞれに独自の利点と制限があります。スライドスキャン法では重要なイベントを視覚化できますが、スライドは最適なセル密度で作成する必要があり、達成が困難な場合があります。さらに、この手法は細胞質の可視化を欠いていることが多く、MONO細胞とPOLY細胞のスコアリングを損なう可能性があります7,8。フローサイトメトリーはハイスループットなデータキャプチャを提供しますが、細胞を溶解する必要があるため、Cyt-B型のアッセイを使用することはできません。さらに、非イメージング技術として、従来のフローサイトメトリーは重要な事象の視覚的検証を提供しない9、10。
そのため、MNアッセイを行うためにイメージングフローサイトメトリー(IFC)が検討されている。ImageStreamX Mk IIは、従来のフローサイトメトリーの速度と統計的堅牢性を、顕微鏡の高解像度イメージング機能を単一のシステムで組み合わせた11。IFCを使用することにより、すべての主要なイベントの高解像度画像をキャプチャし、特徴ベースの12,13または人工知能(AI)技術14,15を使用して自動的にスコアリングできることが示されています。IFCを使用してMNアッセイを行うことにより、顕微鏡検査と比較して、より短時間でより多くの細胞を自動的にスコアリングすることができます。
この作業は、前述の画像分析ワークフロー16 から逸脱し、Amnis AIソフトウェア(以下「AIソフトウェア」と呼ぶ)を使用してランダムフォレスト(RF)および/または畳み込みニューラルネットワーク(CNN)モデルを開発およびトレーニングするために必要なすべてのステップについて説明します。AI支援タグ付けツールを使用したグラウンドトゥルースデータの入力、モデルトレーニング結果の解釈、追加データを分類するためのモデルの適用など、必要なすべてのステップが説明されており、遺伝毒性と細胞毒性の計算が可能になります15。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. イメージングフローサイトメトリーを用いたデータ取得
注:Rodrigues et al.16 を参照し、次の変更を加えて、IFCを使用する取得領域を最適な画像キャプチャのために変更する必要があるかもしれないことに注意してください。
- Non-Cyt-B法の場合は、培養直前および回収期間直後の各培養液について、製造元の説明書( 材料表参照)に従って市販のセルカウンターを用いて細胞カウントを行います。
- シングルカメライメージングフローサイトメーターでサンプルを実行する場合は、明視野(BF)をチャンネル4に配置します。M01 を M04 に、M07 を M01 に置き換えます。
注意: 「M」はIFCのカメラチャンネルを指します。 - 集録時には40倍の倍率を使用してください。
- 集録時のBF面積対BFアスペクト比プロットで、次の領域座標を使用します。
X座標:100と900。Y 座標: 0.7 および 1 (Cyt-B 法)
X座標:100と600。Y 座標: 0.7 および 1 - ヘキスト強度プロットで、次の領域座標を使用します。
X座標:55と75。Y 座標: 9.5 および 15 (Cyt-B 法)
X座標:55と75。Y 座標: 13 および 21 (非 Cyt-B 法) - アポトーシスおよび壊死物体の画像をデータから削除するには、IDEAS 6.3ソフトウェアパッケージを起動します(以下、「画像解析ソフトウェア」と呼びます。
注:AIソフトウェアは、最新バージョンの画像解析ソフトウェアを使用して処理された.dafファイルで動作するように設計されています。画像解析ソフトウェアが最新であることを確認します。 - この作業をテンプレート (.ast) ファイルとして保存します。
2. すべての .rif ファイルに対して .daf ファイルを作成する
- AIソフトウェアは.dafファイルのインポートのみを許可します。バッチ処理を使用して、実験内のすべての .rif ファイルの .daf ファイルを作成します。
- [ ツール ] メニューで、[バッチ データ ファイル ] をクリックし、[ バッチの追加] をクリックします。
- 新しいウィンドウで、[ ファイルの追加 ] を選択し、バッチに追加する .rif ファイルを選択します。[ テンプレートまたはデータ分析ファイル (.ast、.daf) の選択] オプションで、前に作成した .ast ファイル を選択します。
- 必要に応じてバッチ名を割り当て、[ OK ]をクリックして、ロードされたすべての.rifファイルの.dafファイルを作成します。
3. AIソフトウェアで実験を作成する
- AIソフトウェアを使用してディープラーニングモデルを作成するプロセスを説明する 図1 のフローチャートを参照してください。
- AIソフトウェアを起動し、ウィンドウの左下隅にある[ バージョン情報 ]をクリックして、最新バージョンがインストールされていることを確認します。最新バージョンがインストールされていない場合は、support@luminexcorp.com に連絡して入手してください。
- ソフトウェアの既定の画面は [新しい実験 ] 画面です。 [フォルダー ] アイコンを使用して実験を保存する場所を選択し、実験の名前を入力します (例: "MN モデル")。
- [実験の種類] で、[トレーニング] の横にあるラジオ ボタンをクリックしてトレーニング実験を開始し、CNN モデルの構築を開始します。[次へ]をクリックします。
- オプション:モデルが以前にトレーニングされている場合は、新しいAIモデルのテンプレートとして使用でき、[テンプレートモデルの選択]画面から新しい モデルを作成するためのテンプレート として選択できます。テンプレート モデルが存在しない場合は、[ 次へ] をクリックしてこの手順をスキップします。
- 次の画面は、[ 新しいモデルの定義 ]画面です。[ モデル] で、手順 3.3 でモデルに付けられた名前が自動的に入力されます。
- [ 説明] にモデルの説明を入力し (オプション)、最大画像サイズは 150 ピクセルのままにします。
- [チャネル]で、[BFの追加]をクリックして、明視野チャネルをリストに追加します。[名前]で[ブライトフィールド]をダブルクリックし、このチャンネルの名前をBFに変更します。[FLの追加]をクリックして、蛍光チャンネルをリストに追加します。[名前]で[蛍光]をダブルクリックし、このチャネルの名前をDNAに変更します。
- [クラス名] で [追加] をクリックします。ポップアップウィンドウで、単核文字を入力し、[OK]をクリックします。これにより、単核クラスがクラス名のリストに追加されます。このプロセスを繰り返して、次の 6 つのクラスがリストに定義されていることを確認します。
単核
MNによる単核化
二核
MNで二核化
多核
不規則な形態
[ 次へ]をクリックします。
注:これらの6つのグラウンドトゥルースモデルクラスは、スコアリングされる主要なイベントと、受け入れられているスコアリング基準5とは異なる形態の画像を表します。- オプション: 必要に応じて、1.7 の解析テンプレートを組み込んで、画像解析ソフトウェアの機能を使用できます。これらの機能を AI モデルに含める場合は、.ast ファイルを参照し、チャネル固有のドロップダウンから、含める機能のサブセットを選択します。
- [ファイルの選択]で、[ファイルの追加]をクリックし、AIソフトウェアに追加してグラウンドトゥルースデータを構築する目的のファイルを参照します。[次へ]をクリックします。
注:十分な数のすべてのキーイベントを含む複数のデータファイル(ポジティブコントロールデータとネガティブコントロールデータなど)を追加することが重要です。
- 次に、[ ベース集団の選択 ] 画面で、母集団階層から 非アポトーシス 集団を見つけます。 非アポトーシス 集団を右クリックし、 一致するすべての集団を選択を選択します。[ 次へ]をクリックします。
注:分類すべきではない集団(ビーズ、破片、ダブレットなど)を除外することが重要です。 - この画面は、 真理母集団の選択 画面です。
- 主要なイベントのタグ付けされた真理値母集団が画像解析ソフトウェアで作成されていない場合は、[ 次へ]をクリックします。
- タグ付き真理値母集団が画像解析ソフトウェアで作成されている場合は、適切なモデルクラスに割り当てます。
- MNを持つMONO細胞のタグ付けされた真理値集団を割り当てるには、左側のモデルクラスの下にある単核MNクラスをクリックします。次に、これらのイベントを含む右側の適切なタグ付けされた真理値母集団をクリックします。
- タグ付けされた真理値母集団が複数のデータ・ファイルに作成されている場合は、真理値母集団の1つを右クリックし、 一致する母 集団をすべて選択を選択して、複数のファイルから適切なクラスにタグ付けされた母集団を追加します。
- すべての適切な真理値母集団が割り当てられたら、 次へをクリックします。
- [ チャネルの選択] 画面で、実験に適したチャネルを選択します。ここでは、BFをチャンネル1に、ヘキストをチャンネル7に設定します。チャネルを右クリックし、[ すべてに適用] を選択します。[ 次へ]をクリックします。
- 最後に、[ 確認 ] 画面で [ 実験の作成] をクリックします。
- AI ソフトウェアはデータ ファイルから画像を読み込み、手順 3.7 で割り当てられたグラウンド トゥルース画像を使用して、手順 3.5.3 で定義されたモデル クラスを作成します。[ 完了]をクリックします。
- 実験が作成されると、次の 5 つのオプションが表示されます。
実験: 読み込まれたデータファイル、選択されたチャネル、定義されたグラウンドトゥルースモデルクラスなど、実験の詳細を提供します。
タグ付け: ユーザーがグラウンドトゥルースデータを入力できるタグ付けツールを起動します。
トレーニング: グラウンドトゥルースデータに基づいてモデルをトレーニングします。
分類: トレーニング済みのモデルを使用してデータを分類します。
結果: トレーニング実験と分類実験の両方の結果を提供します。
4. AI支援タグ付けツールを使用したグラウンドトゥルースデータの入力
- [タグ付け]をクリックして、 タグ付け ツールのインターフェイスを起動します。
- ズームツール(虫眼鏡アイコン)をクリックして、見やすくするために画像をトリミングします。
- スライダーバーをクリックして画像サイズを調整し、ギャラリーに表示される画像の数を変更します。
- [ 表示設定 ]オプションをクリックし、すべての主要なイベントを識別するのに最適なコントラスト画像を提供する [最小-最大]を選択します。
- [ギャラリーディスプレイのセットアップ]をクリックして、DNA画像の色を黄色または白に変更すると、小さなオブジェクト(MNなど)の視覚化が向上します。
- [ クラスター ]をクリックしてアルゴリズムを実行し、類似した形態を持つオブジェクトをグループ化します。クラスタリングが完了すると、クラスタあたりのオブジェクト数を持つ個々のクラスタが [不明な母集団] の下のリストに表示されます。個々のクラスターを選択してクラスター内のオブジェクトを表示し、これらのオブジェクトを適切なモデル クラスに割り当てます。
- 各モデル クラスに少なくとも 25 個のオブジェクトが割り当てられると、 Predict アルゴリズムが使用可能になります。[ 予測] をクリックします。
注: どの母集団にもうまく適合しないオブジェクトは、 不明として分類されたままになります。より多くのオブジェクトが真理値母集団に追加されるにつれて、予測精度が向上します。 - 各クラスのオブジェクト数が十分に達するまで、グラウンドトゥルース モデル クラスに適切な画像を設定し続けます。
- 各モデルクラスに最低100個のオブジェクトが割り当てられたら、画面上部の[ トレーニング ]タブをクリックします。 [トレーニング ] ボタンをクリックして、ランダム フォレストと CNN アルゴリズムを使用してモデルを作成します。
注:AIソフトウェアはランダムフォレストとCNNアルゴリズムの両方を使用してモデルを作成し、チェックボックスはCNNアルゴリズムのランダムフォレストのみを使用してモデルを作成できます。
5. モデルの精度の評価
- モデルのトレーニングが完了したら、[ 結果の表示] をクリックします。
- 結果画面を使用して、モデルの精度を評価します。プルダウン メニューを使用して、ランダム フォレストと CNN を切り替えます。
注: 真理母集団を更新したり、モデルを再トレーニングしたり、そのまま使用して追加のデータを分類したりできます。- 真理の母集団を更新するには、上部の タグ付け をクリックし、セクション4に従います。
6. モデルを使用したデータの分類
- AIソフトウェアを起動します。既定の画面は [新しい実験 ] 画面です。 [フォルダー ] アイコンを使用して、実験を保存する場所を選択し、実験の名前を入力します。
- [実験タイプ] で、[分類] の横にあるラジオ ボタンをクリックして、分類実験を開始します。[次へ]をクリックします。
- 分類に使用するモデルをクリックし、[ 次へ] をクリックします。
- [ ファイルの選択 ] 画面で、[ファイルの追加] をクリックし、CNN モデルで分類する ファイル を参照します。[ 次へ]をクリックします。
- 次に、[ベースポピュレーションの選択]画面で、ロードされたファイルの1つで非アポトーシスポピュレーションの横にあるチェックボックスをクリックします。非アポトーシス集団を右クリックし、一致するすべての集団を選択をクリックして、ロードされたすべてのファイルからこの母集団を選択します。[次へ]をクリックします。
- オプション: 分類するデータに真理値の母集団が含まれている場合は、「真理値母集団の選択」画面で適切なモデル・クラスに割り当てることができます。それ以外の場合は、[次へ] をクリックしてこの手順をスキップします。
- チャンネル選択画面で、明視野にはチャンネル1、DNA染色にはチャンネル7を選択します。チャンネルを右クリックし、[すべてに適用]をクリックします。次に、[次へ]をクリックします。
- 最後に、[ 確認 ] 画面で [ 実験の作成] をクリックします。AIソフトウェアは、選択したデータファイルから選択したモデルとすべての画像をロードします。[ 完了]をクリックします。
- [分類] をクリックして、分類画面を起動します。[分類]ボタンをクリックします。これにより、RF および CNN モデルを使用して追加データを分類し、指定されたモデル クラスに属するすべてのオブジェクトを識別するプロセスが開始されます。
注:チェックボックスを使用して、RFモデルやCNNモデルを選択できます。 - 分類が完了したら、[ 結果の表示] をクリックします。
- [DAF の更新] ボタンをクリックして、[分類結果で DAF を更新] ウィンドウを表示します。[OK] をクリックして .daf ファイルを更新します。
7. 分類結果のレポートの生成
- [結果] 画面で、[レポートの生成] をクリックします。入力 DAF ごとに個別のレポートが必要な場合は、[入力 DAF ごとにレポートを作成] の横にあるチェックボックスをオンにします。OKをクリックします。
- 完了したら、レポートファイルが保存されているフォルダを開きます。フォルダー内には、実験.pdfレポートと リソース フォルダーがあります。
- .pdfを開いてレポートを表示します。レポートには、モデルと実験の情報、入力 .daf ファイルの一覧、表形式とヒストグラム形式のクラス数とクラスの割合、およびすべての入力 .daf ファイルの予測確率の中央値を要約した混同行列が含まれています。
- [リソース] フォルダーを開き、[CNN] フォルダーを開きます。このフォルダ内には、クラス数とパーセンテージ棒グラフの.pngファイル、および混同行列があります。さらに、各入力ファイルのクラス数とパーセンテージを含む.csvファイルがあります。
8.MN頻度と細胞毒性の決定
- MN 周波数の計算
- 非 Cyt-B 法: MN 頻度を決定するには、ステップ 7.4 の class_count.csv ファイルを開きます。各入力ファイルについて、「MNで単核」母集団のカウントを「単核」母集団のカウントで割り、100を掛けます。

- Cyt-B 法: MN 頻度を決定するには、ステップ 7.4 の class_count.csv ファイルを開きます。各入力ファイルについて、"MNで二核化" 母集団のカウントを "二核" 母集団のカウントで割り、100 を掛けます。
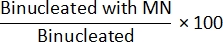
- 非 Cyt-B 法: MN 頻度を決定するには、ステップ 7.4 の class_count.csv ファイルを開きます。各入力ファイルについて、「MNで単核」母集団のカウントを「単核」母集団のカウントで割り、100を掛けます。
- 細胞毒性の計算
- 非サイトB法:
- 初期細胞数と処理後の細胞数を使用して、最初に各サンプルの母集団倍増(PD)を計算します2:
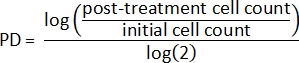
- 次に、相対人口倍増2を計算します。

- 最後に、各サンプルの細胞毒性2 を計算します。
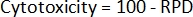
- 初期細胞数と処理後の細胞数を使用して、最初に各サンプルの母集団倍増(PD)を計算します2:
- サイトBメソッド:
- 細胞質分裂ブロック増殖指数(CBPI)2を計算するには、 class_count.csv ファイルの各サンプルの単核、二核、および多核クラスのカウントを使用します。
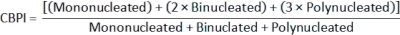
- 細胞毒性2を計算するには、対照培養物(C)および暴露培養物(T)からのCBPIを使用します。
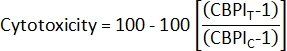
- 細胞質分裂ブロック増殖指数(CBPI)2を計算するには、 class_count.csv ファイルの各サンプルの単核、二核、および多核クラスのカウントを使用します。
- 非サイトB法:
Access restricted. Please log in or start a trial to view this content.
結果
図1は、AIソフトウェアを使用してMNアッセイのモデルを作成するためのワークフローを示しています。ユーザーは、目的の.dafファイルをAIソフトウェアにロードし、AI支援クラスター(図2)と予測(図3)タグ付けアルゴリズムを使用して、オブジェクトをグラウンドトゥルースモデルクラスに割り当てます。すべてのグラウンドト...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
ここで紹介する研究では、MNアッセイのスコアリングを自動化するためのディープラーニングアルゴリズムの使用について説明しています。最近のいくつかの出版物は、直感的でインタラクティブなツールが、深い計算知識を必要とせずに画像データを分析するための深層学習モデルの作成を可能にすることを示しています18,19。ユーザーインター?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは、ImageStreamイメージングフローサイトメーターと本研究で使用したAmnis AIソフトウェアのメーカーであるDiaSorin CompanyであるLuminex Corporationに雇用されています。
謝辞
何一つ。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Falcon | 352096 | |
| Cleanser - Coulter Clenz | Beckman Coulter | 8546931 | Fill container with 200 mL of Cleanser. https://www.beckmancoulter.com/wsrportal/page/itemDetails?itemNumber=8546931#2/10//0/25/ 1/0/asc/2/8546931///0/1//0/ |
| Colchicine | MilliporeSigma | 64-86-8 | |
| Corning bottle-top vacuum filter | MilliporeSigma | CLS430769 | 0.22 µm filter, 500 mL bottle |
| Cytochalasin B | MilliporeSigma | 14930-96-2 | 5 mg bottle |
| Debubbler - 70% Isopropanol | MilliporeSigma | 1.3704 | Fill container with 200 mL of Debubbler. http://www.emdmillipore.com/US/en/product/2-Propanol-70%25-%28V%2FV%29-0.1-%C2%B5m-filtred,MDA_CHEM-137040?ReferrerURL=https%3A%2F%2Fwww.google.com%2F |
| Dimethyl Sulfoxide (DMSO) | MilliporeSigma | 67-68-5 | |
| Dulbecco's Phosphate Buffered Saline 1X | EMD Millipore | BSS-1006-B | PBS Ca++MG++ Free |
| Fetal Bovine Serum | HyClone | SH30071.03 | |
| Formaldehyde, 10%, methanol free, Ultra Pure | Polysciences, Inc. | 04018 | This is what is used for the 4% and 1% Formalin. CAUTION: Formalin/Formaldehyde toxic by inhalation and if swallowed. Irritating to the eyes, respiratory systems and skin. May cause sensitization by inhalation or skin contact. Risk of serious damage to eyes. Potential cancer hazard. http://www.polysciences.com/default/catalog-products/life-sciences/histology-microscopy/fixatives/formaldehydes/formaldehyde-10-methanol-free-pure/ |
| Guava Muse Cell Analyzer | Luminex | 0500-3115 | A standard configuration Guava Muse Cell Analyzer was used. |
| Hoechst 33342 | Thermo Fisher | H3570 | 10 mg/mL solution |
| Mannitol | MilliporeSigma | 69-65-8 | |
| MEM Non-Essential Amino Acids 100X | HyClone | SH30238.01 | |
| MIFC - ImageStreamX Mark II | Luminex, a DiaSorin company | 100220 | A 2 camera ImageStreamX Mark II eqiped with the 405 nm, 488 nm, and 642 nm lasers was used. |
| MIFC analysis software - IDEAS | Luminex, a DiaSorin company | 100220 | "Image analysis sofware" The companion software to the MIFC (ImageStreamX MKII) |
| MIFC software - INSPIRE | Luminex, a DiaSorin company | 100220 | "Image acquisition software" This is the software that runs the MIFC (ImageStreamX MKII) |
| Amnis AI software | Luminex, a DiaSorin company | 100221 | "AI software" This is the software that permits the creation of artificial intelligence models to analyze data |
| Mitomycin C | MilliporeSigma | 50-07-7 | |
| NEAA Mixture 100x | Lonza BioWhittaker | 13-114E | |
| Penicllin/Streptomycin/Glutamine solution 100X | Gibco | 15070063 | |
| Potassium Chloride (KCl) | MilliporeSigma | P9541 | |
| Rinse - Ultrapure water or deionized water | NA | NA | Use any ultrapure water or deionized water. Fill container with 900 mL of Rinse. |
| RNase | MilliporeSigma | 9001-99-4 | |
| RPMI-1640 Medium 1x | HyClone | SH30027.01 | |
| Sheath - PBS | MilliporeSigma | BSS-1006-B | This is the same as Dulbecco's Phosphate Buffered Saline 1x Ca++MG++ free. Fill container with 900 mL of Sheath. |
| Sterile water | HyClone | SH30529.01 | |
| Sterilizer - 0.4%–0.7% Hypochlorite | VWR | JT9416-1 | This is assentually 10% Clorox bleach that can be made by deluting Clorox bleach with water. Fill container with 200 mL of Sterilzer. |
| T25 flask | Falcon | 353109 | |
| T75 flask | Falcon | 353136 | |
| TK6 cells | MilliporeSigma | 95111735 |
参考文献
- Fenech, M., et al. HUMN project initiative and review of validation, quality control and prospects for further development of automated micronucleus assays using image cytometry systems. International Journal of Hygiene and Environmental Health. 216 (5), 541-552 (2013).
- OECD. Test No. 487: In Vitro Mammalian Cell Micronucleus Test. Section 4. OECD Guidelines for the Testing of Chemicals. , OECD Publishing. Paris. (2016).
- Fenech, M. The in vitro micronucleus technique. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 455 (1), 81-95 (2000).
- Bonassi, S., et al. An increased micronucleus frequency in peripheral blood lymphocytes predicts the risk of cancer in humans. Carcinogenesis. 28 (3), 625-631 (2007).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nature Protocols. 2 (5), 1084-1104 (2007).
- Fenech, M. Commentary on the SFTG international collaborative study on the in vitro micronucleus test: To Cyt-B or not to Cyt-B. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 607 (1), 9-12 (2006).
- Seager, A. L., et al. Recommendations, evaluation and validation of a semi-automated, fluorescent-based scoring protocol for micronucleus testing in human cells. Mutagenesis. 29 (3), 155-164 (2014).
- Rossnerova, A., Spatova, M., Schunck, C., Sram, R. J. Automated scoring of lymphocyte micronuclei by the MetaSystems Metafer image cytometry system and its application in studies of human mutagen sensitivity and biodosimetry of genotoxin exposure. Mutagenesis. 26 (1), 169-175 (2011).
- Bryce, S. M., Bemis, J. C., Avlasevich, S. L., Dertinger, S. D. In vitro micronucleus assay scored by flow cytometry provides a comprehensive evaluation of cytogenetic damage and cytotoxicity. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 630 (1), 78-91 (2007).
- Avlasevich, S. L., Bryce, S. M., Cairns, S. E., Dertinger, S. D. In vitro micronucleus scoring by flow cytometry: Differential staining of micronuclei versus apoptotic and necrotic chromatin enhances assay reliability. Environmental and Molecular Mutagenesis. 47 (1), 56-66 (2006).
- Basiji, D. A. Principles of Amnis imaging flow cytometry. Methods in Molecular Biology. 1389, 13-21 (2016).
- Rodrigues, M. A. Automation of the in vitro micronucleus assay using the Imagestream® imaging flow cytometer. Cytometry Part A. 93 (7), 706-726 (2018).
- Verma, J. R., et al. Investigating FlowSight® imaging flow cytometry as a platform to assess chemically induced micronuclei using human lymphoblastoid cells in vitro. Mutagenesis. 33 (4), 283-289 (2018).
- Wills, J. W., et al. Inter-laboratory automation of the in vitro micronucleus assay using imaging flow cytometry and deep learning. Archives of Toxicology. 95 (9), 3101-3115 (2021).
- Rodrigues, M. A., et al. The in vitro micronucleus assay using imaging flow cytometry and deep learning. Npj Systems Biology and Applications. 7 (1), 20(2021).
- Rodrigues, M. A. An automated method to perform the in vitro micronucleus assay using multispectral imaging flow cytometry. Journal of Visualized Experiments. (147), e59324(2019).
- Lovell, D. P., et al. Analysis of negative historical control group data from the in vitro micronucleus assay using TK6 cells. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 825, 40-50 (2018).
- Berg, S., et al. ilastik: interactive machine learning for (bio)image analysis. Nature Methods. 16 (12), 1226-1232 (2019).
- Hennig, H., et al. An open-source solution for advanced imaging flow cytometry data analysis using machine learning. Methods. 112, 201-210 (2017).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved