このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
グルカンホスファターゼの基質結合を測定するためのコンカナバリンAベースの沈降アッセイ
要約
この方法は、グルカンホスファターゼとアミロペクチンの結合親和性を定量化するためのレクチンベースの in vitro 沈降アッセイを記載する。この共沈降アッセイは、グルカンホスファターゼ基質結合の測定に信頼性が高く、さまざまな可溶化グルカン基質に適用できます。
要約
グルカンホスファターゼは、動物のグリコーゲンや植物のデンプンなどのグルカン基質を脱リン酸化する二重特異性ホスファターゼ(DSP)のより大きなファミリーに属します。モデルグルカン基質を有するグルカンホスファターゼの結晶構造は、DSPと糖鎖結合ドメインからなる明確なグルカン結合界面を明らかにする。しかし、グルカン-グルカンホスファターゼと生理学的に関連する基質との相互作用の定量的測定は、グルカンホスファターゼファミリーの酵素の生物学的理解とエネルギー代謝の調節にとって基本的なものです。この原稿は、異なるグルカン基質に対するグルカンホスファターゼの基質結合親和性を検出するために設計されたConcanavalin A(ConA)ベースのin vitro沈降アッセイを報告しています。概念実証として、グルカンホスファターゼシロイヌナズナデンプン過剰4(SEX4)とアミロペクチンの解離定数(KD)を測定しました。SEX4変異体およびグルカンホスファターゼファミリーの酵素の他のメンバーの特性評価は、タンパク質-炭水化物相互作用の差次的結合を評価するためのこのアッセイの有用性をさらに実証しています。これらのデータは、広範囲のデンプンおよびグリコーゲン相互作用タンパク質の特性評価に対するこのアッセイの適合性を実証する。
概要
グルカンホスファターゼは、タンパク質チロシンホスファターゼ(PTP)スーパーファミリー1内の二重特異性ホスファターゼ(DSP)の機能的に多様なサブファミリーのメンバーです。それらは、広く異なる光合成生物、人間、脊椎動物、および一部の無脊椎動物や原生生物を含むほとんどの生命体で発見されています2,3,4。植物には、デンプン過剰4(SEX4)、ライクセックスフォー1(LSF1)、およびライクセックスフォー2(LSF2)5,6,7の3つの既知のグルカンホスファターゼが含まれています。グルカンホスファターゼを欠く植物は、一時的なデンプンの分解と葉のデンプンの蓄積の速度が低下します8,9。ラフォリンは、脊椎動物およびヒトのグリコーゲンを脱リン酸化するグルカンホスファターゼファミリーの創設メンバーです3,10。ラフォリンの変異は、てんかんの致命的な常染色体劣性形態である神経変性ラフォラ病をもたらします11。グルカンホスファターゼはグリコーゲンとデンプンの代謝に必要であり、植物のデンプン含有量を調節し、神経変性ラフォラ病を治療するための重要な酵素として浮上しています12,13。モデルグルカン基質を用いたグルカンホスファターゼに関する最近のX線結晶構造解析研究は、基質結合とグルカン脱リン酸化の触媒機構を明らかにしています14,15,16,17。しかし、グルカンホスファターゼがそれらの生理学的基質にどのように結合するかについての現在の理解は不完全である。
デンプンは、80%〜90%のアミロペクチンと10%〜20%のアミロースからなるグルコースの不溶性ポリマーです18。植物グルカンホスファターゼの基質は、グリコーゲンやデンプン顆粒などのリン酸化炭水化物分子です。リン酸化グルコシル残基は、1:600のリン酸:グルコシル残基比で存在します。興味深いことに、リン酸塩はアミロペクチン分子19上にのみ存在する。主な植物グルカンホスファターゼSEX4は、デンプン顆粒に作用してアミロペクチン分子を脱リン酸化します。構造誘導突然変異誘発研究と組み合わせたSEX4のX線結晶構造は、グルカン構造内のさまざまな位置に対するSEX4のユニークな基質特異性を実証しました15。我々は最近、SEX4の生物学的に関連する活性は、その可溶化アミロペクチン基質20に作用するときにのみ観察できることを示した。しかし、グルカン-SEX4相互作用を理解することは、基質の構造の複雑さ、より広い結合特異性、およびタンパク質とその基質間の結合親和性が低いため、困難であることが証明されています。これらの問題により、等温滴定熱量測定(ITC)、核磁気共鳴(NMR)分光法、酵素結合免疫吸着アッセイ(ELISA)ベースのアッセイなど、タンパク質-リガンド相互作用で一般的に使用される方法を利用する能力が妨げられてきました。
興味深いことに、炭水化物とタンパク質の相互作用に関する私たちの理解の多くは、レクチンの研究から来ています。コンカナバリンA(ConA)は、もともとジャックビーンから抽出されたタンパク質のマメ科レクチンファミリーです。ConAは炭水化物に高い特異性で結合するため、薬物の標的化および送達用途での使用に有利です。非還元性α-D-マンノシルおよびα-D-グルコシルを含む様々な基質へのConAの結合は、広く研究されている19,20。市販のConA結合セファロースビーズは、糖タンパク質および糖脂質を精製するために一般的に使用されている21。ConAは、グルコース残基のC3、C4、およびC6ヒドロキシル基を介してこれらのグルカンに結合します。ConA-セファロースビーズは、グリコーゲン-タンパク質およびデンプン-タンパク質相互作用の結合を測定するためにも成功裏に使用されています22,23。本研究では、ConA-Sepharoseビーズを用いて、グルカンホスファターゼ-アミロペクチン相互作用の結合特異性を測定するための結合アッセイを開発しました。
以前は、グルカンホスファターゼ基質結合能を評価するためにConAベースの沈降アッセイが使用されていました14、20、24。この研究では、同じ戦略を使用して、グルカン-グルカンホスファターゼと炭水化物相互作用の結合親和性を決定するための新しい方法を開発しました。この方法は、様々な可溶化糖質-タンパク質相互作用を調べるための利点もある。
プロトコル
1. ConA-セファロースビーズの調製
- 67 mM HEPES (pH 7.5)、10 mM MgCl 2、および 0.2 mM CaCl2 を含む結合バッファー 250 mL を作製します。1 M NaOH溶液を使用してpHを調整します。
- 250 μLのConA-セファロースビーズ懸濁液を1.5 mLのマイクロ遠心チューブにピペットで入れます。内容物を10,000 x g で4°Cで30秒間遠心分離します。 上清を捨てる。
注:アッセイに使用するアミロペクチン濃度ごとに、1.5 mLマイクロ遠心チューブに250 μLのConA-セファロースビーズが必要です。 - 250 μLのConA-セファロースビーズを含む各チューブに750 μLの結合バッファーを追加します。チューブを10,000 x g で4°Cで1分間遠心分離します。 上清を除去する。このステップを2回繰り返して、ビーズが適切に洗浄され、結合バッファーで平衡化されていることを確認します。
2.アミロペクチン溶液の調製
- 10 mg/mLポテトアミロペクチンの原液を作ります。アミロペクチンは水不溶性であり、熱によって可溶化することができる。可溶化するには、0.1 gのジャガイモアミロペクチンを10 mLの蒸留水に加えます。懸濁液を水浴中で80°Cで1時間、または溶液が濁らなくなるまで加熱する。
- 溶液を室温(RT)に戻して、凝集を避けるためにボルテックスを繰り返します。
- アルコールアルカリ処理は、アミロペクチン基質を可溶化するための代替方法です。この方法を使用して可溶化するには、以下の手順に従います。
- 0.5 gのアミロペクチン基質を5 mLの20%エタノールおよび5 mLの2 M NaOHに懸濁します。内容物をRTで15〜20分間激しく攪拌します。
- 次に、10 mLの水を加え、2 M HClを加えて溶液のpHを6.5に調整し、得られた溶液の容量を蒸留水で50 mLにして、10 mg/mLのアミロペクチン溶液を作ります。
- 10 mg/mLの可溶化アミロペクチン溶液を希釈して、一連の2 mLの希釈アミロペクチン溶液を作ります。例えば、10 mg/mLの半希釈を行い、一連のアミロペクチン濃度(5 mg/mL、2.5 mg/mL、1.25 mg/mL、0.625 mg/mL、0.3125 mg/mL、0.156 mg/mL、0.078 mg/mL、0.039 mg/mL、0.019 mg/mL、0 mg/mL)を調製します。
3. ConA-セファロースの調製:アミロペクチンビーズ
- 希釈した各アミロペクチン溶液250 μLを、結合バッファーで事前に平衡化した250 μLのConA-セファロースビーズを含む1.5 mLマイクロ遠心チューブに加えます。内容をよく混ぜます。対応するアミロペクチン濃度でチューブにラベルを付けます。
- 内容物を回転ホイール上で4°Cで30分間インキュベートします。
注:ConA-セファロース:アミロペクチン結合複合体は、20分後に経時的に変化はありません。.30分のインキュベーション時間は、平衡に達することを保証するために、インキュベーション時間を10分から1時間に変更することによって選択されました。 - チューブを10,000 x g で1分間遠心分離します。新しくラベル付けした1.5 mLマイクロ遠心チューブに上清を収集します。これらの上清画分を保存して、D−グルコースアッセイ12 (アミロペクチンの酸加水分解とそれに続く酵素アッセイ による グルコースのUV定量)を行う。このステップは、すべてのアミロペクチンがビーズに結合していることを確認するために必要です。
- 750 μLの結合バッファーをConA-セファロース:アミロペクチンビーズに加えます。チューブを10,000 x g で1分間遠心分離します。上清を捨てて、結合していないアミロペクチン分子を除去します。
- 手順3.4を繰り返して、十分に洗浄します。各チューブには、さまざまな量のアミロペクチン基質に結合したConA-セファロースビーズが含まれています。
4. SEX4をConA-セファロース:アミロペクチンビーズでインキュベートする
- 250 μLのConA-セファロース:アミロペクチンビーズを、10 μgのSEX4タンパク質、10 mMジチオスレイトール(DTT)、および10 μMのプロテアーゼ阻害剤カクテル(PIC)を含む100 μLの結合バッファーと混合します。各チューブの総容量は350μLであることに注意してください。
注:プロテアーゼ阻害剤カクテルは、不必要なSEX4分解を回避するための予防措置として追加されます。これはオプションの手順です。このアッセイでは、組換えタンパク質シロイヌナズナSEX4(AtSEX4)が使用される。精製されたタンパク質は、化学発光を介してタンパク質を検出するために必要なN末端ヒスチジンタグを含有する。グルカンホスファターゼ精製に関する詳細な情報は、以前の刊行物14、20、24に記載されている。 - タンパク質とConA-セファロース:アミロペクチンビーズ懸濁液を4°Cで45分間、穏やかな回転でインキュベートします。
注:45分のインキュベーション時間は、複合体の平衡に達することを確実にするために選択されます。 - チューブを10,000 x g で1分間遠心分離します。ゲルローディングチップを使用して上清50 μLを新しい1.5 mLマイクロ遠心チューブに注意深くピペットで入れます。20 μLの4x SDS-PAGE色素と10 μLの水を、50 μLの上清画分を含む各チューブに加えます。サンプルを95°Cで10分間加熱します。SDS-PAGEゲルを実行するためにこれらのサンプルを保存してください。「上清(S)」とラベル付けされた10本の新しいチューブが対応する基質濃度を持っていることを確認してください。
- 750 μLの結合バッファーをConA-セファロース:アミロペクチン:SEX4ビーズに加えて、ビーズから未結合タンパク質を除去します。チューブを10,000 x g で1分間遠心分離します。この手順をもう一度繰り返して、適切に洗浄します。上清を捨てる。
- 洗浄したConA-セファロース:アミロペクチン:SEX4ビーズを含むチューブに20 μLの4x SDS-PAGE色素と80 μLの蒸留水を加えます。サンプルを95°Cで10分間加熱し、10,000 x g で1分間遠心分離します。
- ペレットを廃棄し、SDS-PAGEゲルを実行するための上清を保存します。上清80 μLを新しいチューブにピペットで入れ、「ペレット(P)」とラベル付けします。
5. SDS-PAGEゲルの実行
- 40 μLの未結合タンパク質サンプル(ステップ2.3で作成、ラベルS)を4%〜12%のプレキャストポリアクリルアミドゲルウェルに最低基質濃度から最高濃度までロードしますが、最初のレーンはタンパク質分子量マーカーをロードするために自由に保ちます。2番目のゲルを使用して、ステップ2.5で作成した10個の結合タンパク質サンプル(Pとラベル付け)をロードします。
- 新たに調製した1x SDS-PAGEランニングバッファーを装置の両方のチャンバーに追加します。ゲルを150 Vで35分間、または色素前面がゲルの底に達するまで実行します。
- 装置からランゲルを取り外し、スペーサーとガラス板を取り外します。分離したゲルを使用して、ウェスタンブロット分析を実行します。
6. 化学発光検出のためのウェスタンブロッティング14,15
注:この方法は、ユーザーがラボで使用しているウェスタンブロッティング装置に応じて簡単に変更/適合できます。
- 5.8 gのトリス塩基、2.9 gのグリシン、0.37 gのSDS、および200 mLのメタノールを含む1 Lの転写バッファーを作成します。
- サイズ分離したタンパク質をポリアクリルアミドゲルからニトロセルロース膜に移します。スポンジ、ろ紙、ゲル、およびニトロセルロース膜をウェスタン転写プロトコル14,15に従って簡単に組み立てます。70 Vで1時間運転します。
- 非特異的タンパク質結合を防ぐために、1%〜5%ウシ血清アルブミン(BSA)または乳タンパク質のタンパク質溶液を含むニトロセルロース膜を50 mLのTBSTバッファー(20 mM Tris [pH 7.5]、150 mM NaCl、0.1%トゥイーン20)中で1時間インキュベートします。TBSTバッファーを使用してメンブレンを3回洗浄し、未結合のブロッキング溶液を除去します。
- Hisタグ付きタンパク質に特異的な西洋ワサビペルオキシダーゼ(HRP)結合抗体でメンブレンを1時間インキュベートします。メンブレンをTBSTバッファーで3倍洗浄し、未結合の抗体を除去します。最適な再現性と感度を得るために、TBSTに対する抗体の1:2,000希釈を使用してください。
- HRP酵素結合抗体は、SEX4タンパク質のヒスチジンタグに特異的に結合し、化学発光試薬の存在下でバンドを生成します。デジタルイメージングの場合は、等量の化学発光基質溶液(各750 μL)を1.5 mLチューブに入れた溶液を作ります。メンブレンを溶液中で少なくとも5分間インキュベートします。
- 膜タンパク質面を下にしてブロットスキャナーに置き、取得ソフトウェアを実行して、ペレット画分と上清画分の両方のタンパク質を定量します。
7.データ分析
- ブロットスキャナーで取得ソフトウェアを使用して定量信号測定を実行します。上清およびペレット画分中のすべての定量的測定値を、負荷された総タンパク質に正規化します。
注:このソフトウェアでは、上清およびペレット画分中の各タンパク質バンドの強度を定量化することができます。 - 飽和結合実験において、タンパク質結合対アミロペクチン濃度のパーセンテージをプロットする。データ分析ソフトウェアを使用してKDを計算し、データをY = Bmax x X/(KD + X)に適合させます。
注:Bmaxは最大特異的結合であり、Y軸は結合したタンパク質の割合であり、X軸はアミロペクチン濃度である。
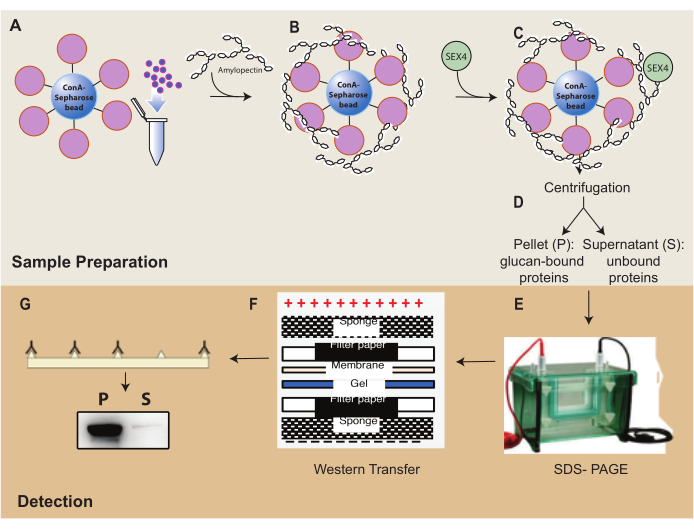
図1:ConA-セファロース沈降アッセイワークフローの概要 。 (A)ConA-セファロースビーズの調製。(B)アミロペクチン基質とのインキュベーション。(C)SEX4タンパク質とのインキュベーション。(D)遠心分離による結合タンパク質画分と非結合タンパク質画分の分離。(E)SDS-PAGEによるタンパク質の分離。(F)ウェスタンブロット分析。(g)Hisタグ付きSEX4タンパク質の化学発光検出。 この図の拡大版を表示するには、ここをクリックしてください。
結果
グルカンホスファターゼファミリーのタンパク質の重要な特徴の1つは、グルカン基質に結合する能力です。まず、SEX4のConA-セファロース:アミロペクチンビーズへの結合能をSDS-PAGEを用いて分析した(図2A)。ウシ血清アルブミン(BSA)は、ConA-Sepharose:アミロペクチンビーズへのタンパク質の非特異的結合を検出するためのネガティブコントロールとして機能しました。タンパ...
ディスカッション
この研究は、グルカン-グルカンホスファターゼ相互作用の結合親和性の決定を可能にする新しい in vitro 沈降アッセイの開発に成功したことを示しています。アッセイデザインでは、グルコースのヒドロキシル残基 を介した グルカンへのレクチンConAの特異的結合を利用して、可溶化された炭水化物基質をセファロースビーズ上に間接的に捕捉します。これにより、遠心分離 ...
開示事項
著者は利益相反を宣言しません。
謝辞
この研究は、国立科学財団賞MCB-2012074によってサポートされました。著者らは、貴重な議論と支援を提供してくれたフロリダ大学生化学分子生物学部のクレイグW.ヴァンダークーイ博士に感謝します。著者らはまた、フロリダ大学生化学・分子生物学部のマシュー・S・ジェントリー博士の支援に感謝している。スキッドモア大学神経科学プログラムの議長であるサラ・ラガルワー博士に、ウェスタンブロットイメージングにLICORC桁ブロットスキャナーの使用を許可してくださったことに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 6x-His Tag monoclonal antibody (HIS.H8), HRP | Therm Fisher Scientific | MA1-21315-HRP | |
| Biorad gel electrophoresis and Western blot kit | Biorad | 1703930 | |
| Calcium chloride | Sigma-Aldrich | 208291 | |
| C-Digit blot scanner | LICOR | 3600-00 | Blot scanner |
| Complete protease inhibitor cocktail | Sigma-Aldrich | 11836170001 | |
| Concanavalin A-sepharose beads | Sigma-Aldrich | C9017 | This product contains in 0.1 M acetate buffer, pH 6, containing 1 M NaCl, 1 mM CaCl2, 1 mM MnCl2, and 1 mM MgCl2 in 20% ethanol |
| Centrifuge | Eppendorf | 5425R | |
| Glycine | Fisher Scientific | BP381-5 | |
| GraphPad Prism 8.0 software | GraphPad | Version 8.0 | Data analysis software |
| HEPES | Sigma-Aldrich | H8651 | |
| Image Studio | LICOR | 3600-501 | Acquisition Software |
| Magnesium chloride | Sigma-Aldrich | M2670 | |
| Methanol | Fisher Scientific | A452SK-4 | |
| Sodium dodecyl sulfate | Fisher Scientific | PI28312 | |
| Potato amylopectin | Sigma-Aldrich | A8515 | |
| Precast SDSPAGE Gels | Genscript | M00653S | |
| Tris base | Fisher Scientific | BP154-1 | |
| Tween 20 | Fisher Scientific | MP1TWEEN201 | |
| Westernsure premium chemiluminescence substrate | LI-COR | 926-95000 |
参考文献
- Meekins, D. A., Vander Kooi, C. W., Gentry, M. S. Structural mechanisms of plant glucan phosphatases in starch metabolism. The FEBS Journal. 283 (13), 2427-2447 (2016).
- Gentry, M. S., et al. The phosphatase laforin crosses evolutionary boundaries and links carbohydrate metabolism to neuronal disease. The Journal of Cell Biology. 178 (3), 477-488 (2007).
- Worby, C. A., Gentry, M. S., Dixon, J. E. Laforin, a dual specificity phosphatase that dephosphorylates complex carbohydrates. The Journal of Biological Chemistry. 281 (41), 30412-30418 (2006).
- Gentry, M. S., Pace, R. M. Conservation of the glucan phosphatase laforin is linked to rates of molecular evolution and the glucan metabolism of the organism. BMC Evolutionary Biology. 9, 138 (2009).
- Niittyla, T., et al. Similar protein phosphatases control starch metabolism in plants and glycogen metabolism in mammals. The Journal of Biological Chemistry. 281 (17), 11815-11818 (2006).
- Kotting, O., et al. STARCH-EXCESS4 is a laforin-like Phosphoglucan phosphatase required for starch degradation in Arabidopsis thaliana. The Plant Cell. 21 (1), 334-346 (2009).
- Comparot-Moss, S., et al. A putative phosphatase, LSF1, is required for normal starch turnover in Arabidopsis leaves. Plant Physiology. 152 (2), 685-697 (2010).
- Zeeman, S. C., Northrop, F., Smith, A. M., Rees, T. A starch-accumulating mutant of Arabidopsis thaliana deficient in a chloroplastic starch-hydrolysing enzyme. The Plant Journal: For Cell and Molecular Biology. 15 (3), 357-365 (1998).
- Kotting, O., et al. Identification of a novel enzyme required for starch metabolism in Arabidopsis leaves. The phosphoglucan, water dikinase. Plant Physiology. 137 (1), 242-252 (2005).
- Tagliabracci, V. S., et al. Laforin is a glycogen phosphatase, deficiency of which leads to elevated phosphorylation of glycogen in vivo. Proceedings of the National Academy of Sciences. 104 (49), 19262-19266 (2007).
- Gentry, M. S., Guinovart, J. J., Minassian, B. A., Roach, P. J., Serratosa, J. M. Lafora disease offers a unique window into neuronal glycogen metabolism. The Journal of Biological Chemistry. 293 (19), 7117-7125 (2018).
- Brewer, M. K., et al. Targeting pathogenic lafora bodies in lafora disease using an antibody-enzyme fusion. Cell Metabolism. 30 (4), 689-705 (2019).
- Santelia, D., Zeeman, S. C. Progress in Arabidopsis starch research and potential biotechnological applications. Current Opinion in Biotechnology. 22 (2), 271-280 (2011).
- Raththagala, M., et al. Structural mechanism of laforin function in glycogen dephosphorylation and lafora disease. Molecular Cell. 57 (2), 261-272 (2015).
- Meekins, D. A., et al. Phosphoglucan-bound structure of starch phosphatase Starch Excess4 reveals the mechanism for C6 specificity. Proceedings of the National Academy of Sciences. 111 (20), 7272-7277 (2014).
- Vander Kooi, C. W., et al. Structural basis for the glucan phosphatase activity of Starch Excess4. Proceedings of the National Academy of Sciences. 107 (35), 15379-15384 (2010).
- Meekins, D. A., et al. Structure of the Arabidopsis glucan phosphatase like sex four2 reveals a unique mechanism for starch dephosphorylation. The Plant Cell. 25 (6), 2302-2314 (2013).
- Smith, A. M., Zeeman, S. C. Starch: A flexible, adaptable carbon store coupled to plant growth. Annual Review of Plant Biology. 71, 217-245 (2020).
- Jane, J., Kasemuwan, T., Chen, J. F., Juliano, B. O. Phosphorus in rice and other starches. Cereal Foods World. 41 (11), 827-832 (1996).
- Mak, C. A., et al. Cooperative kinetics of the glucan phosphatase starch excess4. Biochemistry. 60 (31), 2425-2435 (2021).
- Campbell, K. P., MacLennan, D. H. Purification and characterization of the 53,000-dalton glycoprotein from the sarcoplasmic reticulum. The Journal of Biological Chemistry. 256 (9), 4626-4632 (1981).
- Campbell, K. P., MacLennan, D. H., Jorgensen, A. O., Mintzer, M. C. Purification and characterization of calsequestrin from canine cardiac sarcoplasmic reticulum and identification of the 53,000 dalton glycoprotein. The Journal of Biological Chemistry. 258 (2), 1197-1204 (1983).
- Davey, M. W., Sulkowski, E., Carter, W. A. Binding of human fibroblast interferon to concanavalin A-agarose. Involvement of carbohydrate recognition and hydrophobic interaction. Biochemistry. 15 (3), 704-713 (1976).
- Meekins, D. A., et al. Mechanistic insights into glucan phosphatase activity against polyglucan substrates. The Journal of Biological Chemistry. 290 (38), 23361-23370 (2015).
- Wilkens, C., et al. Plant α-glucan phosphatases SEX4 and LSF2 display different affinity for amylopectin and amylose. FEBS Letters. 590 (1), 118-128 (2016).
- Atanasova, M., Bagdonas, H., Agirre, J. Structural glycobiology in the age of electron cryo-microscopy. Current Opinion in Structural Biology. 62, 70-78 (2020).
- Doyle, M. L. Characterization of binding interactions by isothermal titration calorimetry. Current Opinion in Biotechnology. 8 (1), 31-35 (1997).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved