Method Article
蛍光アッセイまたはフローサイトメトリーを使用したカスパーゼ活性の測定
要約
本プロトコルは、フローサイトメトリーまたは分光蛍光光度計を使用して蛍光発生基質を介してカスパーゼ活性を測定する2つの方法を記載する。
要約
カスパーゼとして知られるシステインプロテアーゼの活性化は、複数の形態の細胞死において依然として重要なプロセスです。カスパーゼは、プログラムされた細胞死の最も研究されている形態であるアポトーシスの重要な開始者および実行者です。アポトーシスは発生過程で起こり、組織の恒常性において必要な事象である。パイロトーシスは、カスパーゼを利用する別の形態の細胞死であり、インフラマソームの活性化を通じて免疫系を活性化する重要なプロセスであり、その結果、インターロイキン-1(IL-1)ファミリーのメンバーが放出されます。カスパーゼ活性を評価するために、標的基質を評価することができます。ただし、単一細胞または低レベルの活性を調べる場合、感度が問題になる可能性があります。蛍光発生基質を集団ベースのアッセイまたはフローサイトメトリーによるシングルセルアッセイでどのように使用できるかを示します。適切なコントロールでは、異なるアミノ酸配列を使用して、どのカスパーゼが活性であるかを識別できます。これらのアッセイを用いて、腫瘍壊死因子(TNF)刺激によるアポトーシスタンパク質の阻害剤の同時消失が確認されており、これは主に他の形態の細胞死ではなくマクロファージにおいてアポトーシスを誘導する。
概要
カスパーゼは、いくつかの形態のプログラム細胞死に関与しています。アポトーシスは、プログラムされた細胞死の最も研究されている形態であり、カスパーゼ活性に関連しています1。すべてのカスパーゼは、大小の触媒サブユニットを持っています。カスパーゼ-1、カスパーゼ-4、カスパーゼ-5、カスパーゼ-9、およびカスパーゼ-11はカスパーゼ活性化およびリクルートメントドメイン(CARD)を有し、カスパーゼ-8およびカスパーゼ-10はデスエフェクタードメイン(DED)2,3,4,5を含む(表1)。アポトーシスは、外因性経路と内因性経路の2つの主要な経路によって開始できます。外因性アポトーシス経路は、腫瘍壊死因子スーパーファミリー(TNFSF)の一部である死受容体によって引き起こされます。死の受容体はDEDドメインを有し、カスパーゼ-8活性を促進します6。内因性アポトーシス経路は、アポトソームの形成後のカスパーゼ-9の活性化を含み、シトクロムcおよびApaf-17の放出を必要とする。イニシエーターカスパーゼ、カスパーゼ-8またはカスパーゼ-9のいずれかの活性化は、カスパーゼ-3、カスパーゼ-6、およびカスパーゼ-7である実行者カスパーゼの切断とその後の活性化につながります。実行者カスパーゼが活性であることを特定することは、細胞がアポトーシスを受けていることを示しており、この活性化は細胞死の様式を定義する上で重要な要素と考えられています。
カスパーゼ活性化は、炎症を調節し、プログラムされた細胞死の代替形態を誘導するための重要な分岐点でもあります。例えば、カスパーゼ-1の活性化は、インターロイキン-1ファミリーの炎症誘発性サイトカインの成熟をもたらす8。このファミリーからのサイトカイン、特にIL-1βおよびIL-18の放出および活性化は、原形質膜におけるガスダーミンD切断および孔形成から生じる9,10。ガスダーミンD孔の不十分な膜修復は、パイロトーシス11として知られる一種の細胞死を引き起こす可能性があります。さらに、カスパーゼ-8活性は、ネクロプトーシス12として知られるカスパーゼ非依存性細胞死の阻害をもたらす。受容体相互作用セリン/スレオニンプロテインキナーゼ1(RIPK1)は、ネクロトーシスおよびNF-kBによって調節される炎症の促進における重要な因子の1つです。 モデルは、RIPK1がカスパーゼ-8によって切断され、NF-kBシグナル伝達、アポトーシス、およびネクロトーシスを制限することを示しています13,14。したがって、異なるカスパーゼの活性を特定することは、結果として生じる炎症および細胞死のモダリティを理解するのに役立ちます。
細胞死様式の調節におけるカスパーゼの機能とは無関係に、カスパーゼ活性は、感染に応答して、インターフェロン(IFN)などの他のサイトカインファミリーも調節することができる15,16。さらに、カスパーゼは、細胞運命の決定、組織の修復と再生、DNA修復による腫瘍形成、ニューロンのシナプス機能など、非細胞死機能に関与しています。これらの非致死的役割におけるカスパーゼの活性は、細胞局在およびカスパーゼの量によって制限されると考えられている。したがって、カスパーゼ活性のレベルを定量化することは、細胞が細胞死を受けるかどうか、またはカスパーゼが非細胞死機能において役割を果たすかどうかを適切に定義することができる4,17,18。
カスパーゼ活性は、複数の方法で評価することができる。切断されたカスパーゼとその基質のウェスタンブロッティングは活性の指標として使用されてきましたが、これらのアッセイはせいぜい定性的です。カスパーゼ活性が細胞死と関連しているかどうかを判断するには、定量的測定が理想的です。カスパーゼは4つのアミノ酸からなる認識部位で基質を切断するため、比色法、発光法、蛍光法が開発されている。しかしながら、カスパーゼは、それらの基質認識において可塑性を有するように見える19,20。認識配列はタンパク質ドメインと会合していない(表1)。しかしながら、テトラペプチド配列DEVDは、カスパーゼ−3およびカスパーゼ−7活性を検出するために使用することができる20,21。
Smac模倣体は、アポトーシスタンパク質(IAP)の阻害剤を標的とする化合物です。がん細胞のサブセットでSmac模倣体を使用すると、細胞はTNF誘発性細胞死に敏感になります22。一次マクロファージでは、Smac模倣体はTNF23,24の外因性添加なしに細胞死を引き起こす。Smac模倣体誘導分解によるcIAP1の喪失は、TNFの産生をもたらす。カスパーゼ活性が検出された場合、これは細胞がネクロトーシスによって死滅したのではなく、アポトーシス様式で死滅したことを意味します。この方法では、切断されたDEVD基質の検出を使用して、カスパーゼ-3/カスパーゼ-7活性を同定します。アポトーシス細胞死を確認するためのさらなる実験が以前に発表されている24。
プロトコル
本研究は、チューリッヒ大学の動物倫理委員会(#ZH149/19)の承認を得て、ガイドラインに従って実施されました。本研究では、特定の病原体フリー(SPF)条件で飼育および飼育された、8〜16週齢の雄C57Bl / 6Jマウスを使用しました。無傷の骨は、2%熱不活化ウシ胎児血清(FBS)を含む滅菌ハンク緩衝生理食塩水(HBSS)で氷上に保つことができます。骨髄は分化当日にマウス25 の大腿骨及び脛骨から採取した。カスパーゼ活性を評価するための両方の方法は、初代および形質転換の両方を含む他の細胞型に使用することができる。
1. 骨髄由来マクロファージ(BMDM)の鑑別
注:組織培養層流フードですべての手順を実行し、滅菌無菌技術を使用します。
- 21 Gの針が付いた1 mLのシリンジを準備します。
- 5 mLのHBSS + 2%FBSを15 mLチューブに加えます。
- 滅菌鉗子を使用して、切除した大腿骨を取り、大腿骨の開口部に針を挿入します。大腿骨をHBSS + 2%FBSに保持し、骨が白くなるまで骨髄を洗い流します。同じ方法で脛骨を洗い流します。溶液を洗い流し続けて、単一細胞懸濁液を得る。
- 単一細胞懸濁液を200 x g で室温(RT)で4分間遠心分離します。
- 真空吸引器を使用して上清を除去します。チューブをタップして、ペレットを静かに再懸濁します。赤血球溶解バッファー1 mL( 材料の表を参照)を加え、P1,000ピペットで穏やかに混合します。RTで1分間インキュベートします。
- 10 mLのHBSS + 2% FBSを加え、200 x g でRTで4分間遠心分離します。
- 上清を除去し、10mLの骨髄由来マクロファージ(BMDM)培養液に再懸濁する。
注:BMDM培地は、10%FBS、20 ng / ml M-CSF、ペニシリン(50 U / mL)、およびストレプトマイシン(50 μg / mL)を添加した低グルコースDMEMで構成されています( 材料の表を参照)。あるいは、20%L929馴化培地をM−CSFの代わりに用いることができる。 - 15 mLのBMDM培地を2つの15 cmペトリ皿に加えます。5 mLの単一細胞溶液を各プレートに追加します。
注:組織培養処理プレートは使用しないでください。組織培養処理プレートは分化を制限する。 - 皿を37°C、5%CO2に6日間置きます。
注意: BMDMは5〜7日後に収穫できます。分化のためのインキュベーション時間が長くなると、抗アポトーシスタンパク質発現が増加します26。
2.細胞の収穫、播種、処理
注:組織培養層流フードですべての手順を実行し、滅菌無菌技術を使用します。完全に分化したマクロファージはプレートに接着し、容易に分離でき、浮遊細胞は廃棄することができます。リン酸緩衝生理食塩水(PBS)は、Ca2+ およびMg2+の有無にかかわらず使用できます。
- 6日間のインキュベーション後、アスピレーターを使用してプレートから浮遊細胞と培地を取り除きます。
- 各15 cmプレートに5 mLのPBSを追加します。アスピレーターを使用してプレートからPBSを取り外します。
- 各プレートに2 mLのトリプシン( 材料表を参照)を追加します。プレートを軽くたたくと細胞がはがれるまでプレートをインキュベートします。5 mLのBMDM培地を取り、プレートから細胞を回収します。
- 2枚目の15cmプレートに移し、プレートから細胞を回収する。細胞懸濁液をプレートから取り出して50 mLチューブに入れます。さらに5 mLのBMDM培地を取り、両方のプレートを洗浄して、すべての細胞が回収されたことを確認します。細胞懸濁液を同じ50 mLチューブに入れます。
- 細胞懸濁液を10 μL採取し、トリパンブルーで1:1希釈した血球計算盤を用いて計数します。
注:自動セルカウンターは、マクロファージのサイズに合わせて校正された場合にも使用できます。 - マクロファージを1 x 106 細胞/mLの密度で播種します。6ウェルプレートに2 x 106 細胞/ウェルで播種したBMDMは、約2 mg/mLの総タンパク質を提供します。処理前に、細胞をプレートに最低6時間接着させます。
注:他の細胞タイプについては、最適な濃度を決定する必要があります。 - マクロファージをSmac模倣体(化合物A、 材料表を参照)22で250 nMおよび500 nMで16時間処理します。
注:アッセイが機能していることを確認するために、アポトーシスとカスパーゼ-3切断を誘導するポジティブコントロールを含めます。一般的なアポトーシス誘導物質には、スタウロスポリンおよびエトポシドが含まれる。多くの細胞株がアポトーシスを起こすためには、16時間の刺激に対して0.1〜10μMで十分です。
3. 処理細胞からの細胞ライセートの調製
注:このステップは氷上で実行する必要があり、試薬と材料は事前に冷却する必要があります。
- 処理した細胞を含むプレートを氷上に移します。細胞培養液から培地を1.5 mLチューブに回収し、300 x g で4°Cで5分間遠心分離し、培地を吸引し、チューブを氷上に置きます。これにより、プレートから剥離した細胞の収集が可能になります。
- 細胞を洗浄するための細胞培養プレートに1 mLの冷PBSを加え、すべてのPBSを吸引します。100 μLのトリプシンを細胞に加えます(6ウェルディッシュを使用する場合)。トリプシンで細胞をプレートから持ち上げ、1.5 mLチューブに回収します。1 mLのコールドPBSを使用して、すべての細胞が収集されていることを確認します。
注:浮遊細胞を扱う場合は、培地と細胞を1.5 mLチューブに静かに移します。 - 細胞を300 x g で4°Cで5分間遠心分離します。 上清を除去し、100 μL DISC 溶解バッファー (150 mM 塩化ナトリウム、2 mM EDTA、1% Triton X-100、10% グリセロール、20 mM トリス、pH 7.5、 材料表を参照) に再懸濁します。
- サンプルを氷上で20分間インキュベートします。
- ライセートを~12,000 x g で4°Cで10分間遠心分離し、不溶性画分をペレット化します。
- カスパーゼ-3/カスパーゼ-7活性アッセイ(ステップ5)のために、25 μLのライセートを白色平底96ウェルプレートに移します。
注意: ペレットを乱さないでください。これは細胞ライセートの不溶性画分です。 - 残りのライセート10 μLを、ビシンコニン酸(BCA)アッセイ用の透明な平底96ウェルプレートに移します(ステップ4)。これは、サンプルの正規化に使用されます。
- さらなる処理のために両方のプレートを氷の上に置いてください。
注:この段階で、プレートは粘着カバーで密封し、-20°Cで約4週間保存できます。
4. BCAアッセイを用いたタンパク質定量
注:他の試薬またはアッセイを使用して、各サンプル中のタンパク質量を定量できます。ポピュレーションベースのアッセイでは、アッセイで使用されるタンパク質の量を正規化することによってサンプルを比較できます。
- ウシ血清アルブミン(BSA)を使用して、0 μg/mLから2,000 μg/mL(0 μg/mL、25 μg/mL、125 μg/mL、250 μg/mL、500 μg/mL、750 μg/mL、100 μg/mL、1,500 μg/mL、2,000 μg/mL)の標準タンパク質濃度を調製します。溶解バッファーのみでブランクを調製します。
- ステップ3.7で説明したサンプルを含む平底96ウェルプレートに各標準物質10 μLを追加します。
- BCA試薬1とBCA試薬2を50:1の比率で混合します( 材料表を参照)。200 μLの混合BCA試薬を各サンプルと標準物質に加えます。
- 37°Cで30分間インキュベートします。
- 蛍光測定装置で562 nmの吸光度を測定し、検量線でタンパク質濃度を定量します。
5.カスパーゼ-3/カスパーゼ-7活性の集団ベースのアッセイ
注意: プレート内の細胞溶解物を氷の上に3時間以上置かないでください。カスパーゼ活性が存在する場合、サンプルが氷上にあるにもかかわらず、これは時間とともに増加します。サンプルが凍結している場合は、氷上で解凍し、ライセートが解凍したらすぐに進めます。
- 蛍光測定装置( 材料表を参照)を起動し、機械を37°Cに加熱します。 前述のようにスクリプトを準備します。
- 反応の速度論を決定するために、毎分40分間個々の読み取りを実行します。
- 励起を360 nm、発光を465 nmに設定します。ウェルあたり10回のフラッシュで十分です。
- 組換えカスパーゼ-3のポジティブコントロールを準備します( 材料の表を参照)。組換えカスパーゼ-3酵素1Uを50 μLの溶解バッファー(チューブ1)に混合します。25 μLの溶解バッファーを追加の3本のチューブに加えます。チューブ1からチューブ2に25 μLを移します。ピペッティングで混ぜる。
- チューブ3とチューブ4についても繰り返します。チューブ5には50 μLの溶解バッファーのみが含まれます。ステップ3.6で説明したカスパーゼ-3活性アッセイ用の白い平底96ウェルプレートに各標準物質25 μLを追加します。
- 氷上でのカスパーゼ活性アッセイ用のマスター反応ミックスを調製します。1回の反応で、50 μLの2xカスパーゼ切断バッファー(0.2M HEPES pH 7.5;20%スクロースまたはPEG;0.2%チャップス)、5 μLの1 mM DEVD-AMC(カスパーゼ-3テトラペプチド基質)、2 μLの500 mM DTT、および18 μLの脱イオン水を混合します(材料の表を参照)。
- 75 μLの反応ミックスを各サンプルと標準物質に加え、総反応量100 μLを得ました。
- ステップ5.1で設定した蛍光測定器を使用して、すぐに蛍光を測定します。
- 蛍光測定ごとに、サンプルの蛍光からブランク(溶解バッファーのみ)の蛍光測定値を差し引きます。サンプルのタンパク質濃度(ステップ4.5で計算)で割って読み取り値を正規化します27:
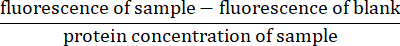
- y軸に正規化された蛍光の傾き、x軸に時間を決定することにより、カスパーゼ活性率を計算します。
6. カスパーゼ-3/カスパーゼ-7活性のシングルセルアッセイ(フローサイトメトリー解析)
- セクション2の説明に従ってセルをシードします。
- 細胞を5 mLのポリスチレンチューブに回収します。接着細胞を扱う場合は、培地を5 mLチューブに回収します。1 mLの冷たいPBSを細胞培養プレートに加え、PBSを5 mLチューブに回収します。細胞にトリプシンを加えます(6ウェルディッシュで作業する場合は100 μL)。トリプシンで細胞をプレートから持ち上げ、5 mLチューブに集めます。1 mLのコールドPBSを使用して、すべての細胞が収集されていることを確認します。
注:浮遊細胞を使用する場合は、培地と細胞を5 mLチューブに静かに移します。 - サンプルを300 x g で5分間、4°Cで遠心分離します。 真空吸引器を使用して上清を除去します。
- 染色ミックスを準備します。最適な染色濃度は、50 μL中の1 x 10 6-2 x 106細胞です。製造元の指示に従って蛍光発生基質を希釈します(材料表、フローサイトメトリーを参照)。ストック基質を1 μL取り、PBS150 μLで希釈します。サンプルあたり50 uLを追加します。
- サンプルを光から保護された37°Cで30分間インキュベートし、15分ごとに混合します。
- この研究で使用したフローサイトメーターを使用して、640 nmの赤色レーザーを使用し、675/25 nmを使用して検出します。最初に染色されていないコントロールサンプルを実行し、目的の母集団の少なくとも10,000イベントを取得します。FSC-AおよびSSC-Aドットプロット上のゲート(P1)を使用して破片を除外します。
- P1ゲートのイベントとカスパーゼ基質の検出(y軸)を表示するヒストグラムを使用して、蛍光強度の中央値(MFI)を決定します。
注:この方法に記載されているカスパーゼ基質は、590 nmでの励起を必要とし、628 nmで発光します。
- P1ゲートのイベントとカスパーゼ基質の検出(y軸)を表示するヒストグラムを使用して、蛍光強度の中央値(MFI)を決定します。
結果
初代マウスマクロファージを6日間分化させた。6日後、細胞を回収し、カウントし、播種した。以下の処理を使用しました:無処理およびSmac模倣体(化合物A)22を250 nMおよび500 nMで16時間使用しました(図1)。カスパーゼ-3/カスパーゼ-7の活性化を集団ベースのアッセイまたはフローサイトメトリーを用いた単一細胞分析のいずれかで評価できるように、実験を二重に実施しました。
細胞溶解物のタンパク質濃度をBCAアッセイを用いて定量した(補足表1)。これは、カスパーゼ-3/カスパーゼ-7活性アッセイで使用されるタンパク質の量がサンプル間で同じであることを確認するために必要です。この集団ベースのアッセイでは、データは2つの方法で提示できます。1つ目は、調整された蛍光(y軸)と時間(x軸)をプロットすることにより、動態を示すことです(図2A)。あるいは、傾きを計算してサンプルを直接比較することもできます(図2B)。500 nM Smac模倣処理時の傾きの増加は、多重比較による通常の一元配置分散分析に基づいて有意ではありませんでした(Dunnettの多重比較検定28)。
フローサイトメトリーを用いたカスパーゼ-3/カスパーゼ-7活性の分析のために、細胞および上清を回収した。未染色細胞または蛍光マイナス1を陰性対照として使用し、ならびに未処理細胞とした。フローサイトメトリーによって収集された細胞のヒストグラムは、未処理の細胞と比較して、Smac模倣体で処理された細胞の蛍光のシフトを示しました(図2C)。データは、蛍光強度の中央値(図2D)または未処理細胞の倍率変化(図2E)のいずれかとして示すことができます。

図1:研究のフローチャート 。 (A)大腿骨と脛骨をC57Bl/6マウスから切除した。骨をフラッシュし、20 ng / mL M-CSFで6日間分化させました。(B)6日目に、マクロファージを回収し、処理のために再播種した。1セットの細胞をライセート用に回収し、蛍光発生活性によって評価し、もう1セットを回収し、蛍光発生基質とともにインキュベートし、フローサイトメトリーで評価しました。 この図の拡大版を表示するには、ここをクリックしてください。
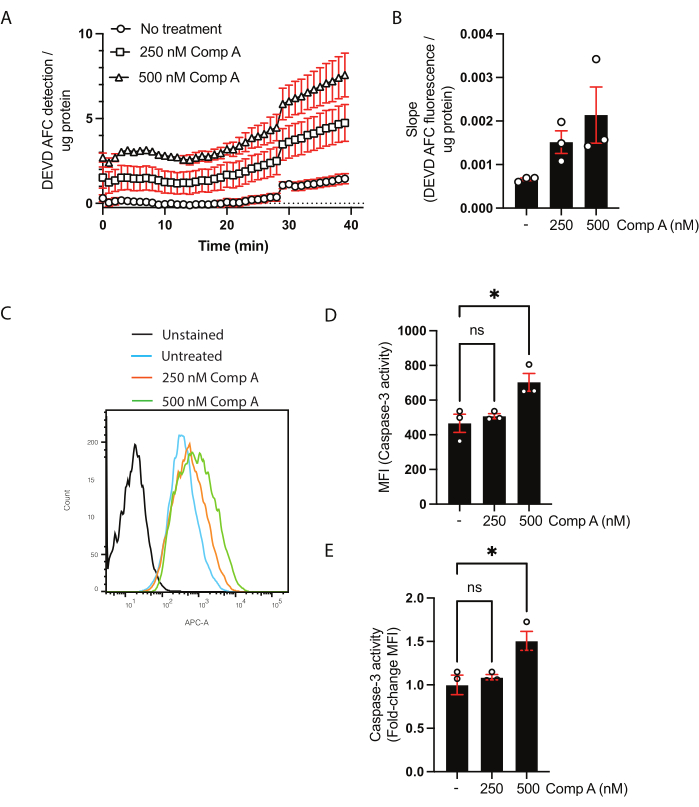
図2:フローサイトメトリーによるカスパーゼ-3/カスパーゼ-7活性および基質切断の速度論的アッセイ。 (A,B)フローサイトメトリーによるカスパーゼ-3/カスパーゼ-7活性(C-E)および基質切断の速度論的アッセイの代表的なデータ。マクロファージを2つの濃度のSmac模倣体(化合物A;250nMおよび500nM)で16時間処理した。 (A)切断されたDEVD AFC基質の経時的な検出。データは、サンプル中のタンパク質濃度に対して標準化した。(B)各サンプルのDEVD AFCの切断速度(スロープ)を未処理のスロープに正規化し、未処理の細胞に対するフォールド変化として提示しました。(C)カスパーゼ-3基質と共にインキュベートした細胞のフローサイトメトリーヒストグラム。(D,E)未処理サンプルと比較したMFIの比較および倍率変化。各データポイントは独立したサンプルを表します。平均±平均の標準誤差が表示されます。*p < 一元配置分散分析と多重比較検定(ダネットの多重比較検定)を用いて0.05。この図の拡大版を表示するには、ここをクリックしてください。
| カスパーゼ | 種 | 基板配列 | タンパク質ドメイン | ||
| カスパーゼ-1 | 高さ、ミリメートル | (長さ)ティッカー | CARD, 大規模ドメイン, 小触媒ドメイン | ||
| カスパーゼ-2 | 高さ、ミリメートル | デックス | CARD, 大規模ドメイン, 小触媒ドメイン | ||
| カスパーゼ-4 | ティッカー | (長さ)ティッカー | CARD, 大規模ドメイン, 小触媒ドメイン | ||
| カスパーゼ-5 | ティッカー | (長さ)ティッカー | CARD, 大規模ドメイン, 小触媒ドメイン | ||
| カスパーゼ-9 | 高さ、ミリメートル | (入出力/対面/左)E(H/T)D | CARD, 大規模ドメイン, 小触媒ドメイン | ||
| カスパーゼ-11 | ミリメートル | (長さ)ティッカー | CARD, 大規模ドメイン, 小触媒ドメイン | ||
| カスパーゼ-12 | ミリメートル | ティッカー | CARD, 大規模ドメイン, 小触媒ドメイン | ||
| カスパーゼ-8 | 高さ、ミリメートル | (入出力/対面/左)E(H/T)D | DED、大規模ドメイン、小触媒ドメイン | ||
| カスパーゼ-10 | ティッカー | (入出力/対面/左)E(H/T)D | DED、大規模ドメイン、小触媒ドメイン | ||
| カスパーゼ-3 | 高さ、ミリメートル | デックス | 大ドメイン、小触媒ドメイン | ||
| カスパーゼ-6 | 高さ、ミリメートル | (入出力/対面/左)E(H/T)D | 大ドメイン、小触媒ドメイン | ||
| カスパーゼ-7 | 高さ、ミリメートル | デックス | 大ドメイン、小触媒ドメイン | ||
| カスパーゼ-14 | HS、MM | (長さ)ティッカー | 大ドメイン、小触媒ドメイン | ||
| カード | カスパーゼ活性化およびリクルートメントドメイン | ||||
| デッド | デスエフェクタードメイン | ||||
| ティッカー | ホモサピエンス | ||||
| ミリメートル | ムス・ムスクルス | ||||
表1:カスパーゼの基質特異性とタンパク質ドメイン。 この表はMcStayら20から改作されています。シャリーニら3;ヴァン・オプデンボッシュとラムカンフィ4.
補足表1:DEVD速度論的分析。この表をダウンロードするには、ここをクリックしてください。
ディスカッション
この方法では、蛍光発生基質を集団ベースのアッセイまたはシングルセル分析に使用して、カスパーゼ-3/カスパーゼ-7活性を測定します。どちらの方法も、基質の切断に基づいて定量的な方法でカスパーゼ活性を測定します。1つの利点は、これらの方法を多数のサンプルに利用できることです。これらの方法では、カスパーゼ-3/カスパーゼ-7活性がSmac模倣体で処理された一次マクロファージで検出されます。
ポピュレーションベースの蛍光測定アッセイの重要な側面は、溶解から蛍光の読み取りまでの時間です。サンプルは、手順全体を通して、特にアッセイを「読み取る」前に氷上に保管する必要があります。これにより、基質の早期切断および蛍光が防止される。ポピュレーションベースのアッセイを使用すると、必要な最適化が少なくて済みます。アッセイで使用されるタンパク質の量は正規化され、サンプルを直接比較することができます。1つの注意点は、アポトーシス細胞死の後期段階で、総タンパク質量が減少することです。したがって、カスパーゼ活性の検出が不可能な場合があります。この問題を回避するために、異なる動態または異なる治療用量が推奨される。さらに、この方法に記載されるソフトウェア以外の他のソフトウェアを使用して、カスパーゼ活性の速度を正確に評価することができる。
フローサイトメトリーアッセイでは、集団を確実にゲートするのに十分なイベントまたは細胞が必要です。さらに、基質と細胞数の最適な比率を達成するために、フローベースのアッセイにおけるさらなる最適化が必要になる場合があります。ただし、フローサイトメトリーでは、この方法は、細胞型識別のための細胞表面マーカーなどの追加パラメーターの測定に役立ちます。
ポピュレーション法とシングルセル法の両方が他のカスパーゼに使用することができます。ただし、認識シーケンスは他のカスパーゼに対してあまり識別されないことを覚えておくことが重要です。そのため、カスパーゼ活性のための他の方法を使用しなければならない。これには、カスパーゼ活性の阻害、特定のカスパーゼのCRISPRまたはノックダウン、および既知の基質の切断を検出するためのウェスタンブロッティングが含まれます。
カスパーゼ活性を検出するための代替方法の1つは、タイムラプスイメージングです。同じ透過性カスパーゼ基質を、アネキシンVなどの他の生存マーカーとともに使用して、細胞死の動態に関する情報を提供することができます。イメージングはまた、カスパーゼ活性と細胞生存を分離し、細胞集団における致死量のカスパーゼ活性の検出を可能にする。カスパーゼ-3/カスパーゼ-7の非致死機能は、自然免疫細胞29における抗ウイルス調節、特にミトコンドリアDNA放出を介したI型IFNの活性化に関連しています15,16。したがって、カスパーゼ活性を測定するためのこれらのアッセイは、細胞死の異なるモードを同定するために重要であり、非細胞死機能の評価に有用であり得る。
開示事項
著者は開示する利益相反を持っていません。
謝辞
WWWはClöetta Medical Research Fellow grant、S.R.はCanDoc UZH Forschungskredit、J.T.はChina Scholarship Councilの支援を受けています。
資料
| Name | Company | Catalog Number | Comments |
| 1.5 mL microfuge tubes | Sarstedt | 72.706.400 | |
| 15 cm Petri plates | Sarstedt | 82.1184.500 | |
| 37 degree incubator shaker | IKA shaker KS 4000i | 97014-816 | distributed by VWR |
| 6-well cell culture dish | Sarstedt | 83.392 | |
| 96 well flat bottom, white polystyrene, non-sterile | Sigma | CLS3600 | |
| 96 well flat plate | Sarstedt | 82.1581 | |
| Ac-DEVD-AFC | Enzo Life Sciences | ALX-260-032-M005 | Caspase-3 substrate |
| BD Fortessa | BD | any flow cytometer with the appropriate excitation and emission detector will work | |
| b-glycerolphosphate | Sigma | G9422-10G | |
| caspase-3 recombinant | Enzo Life Sciences | ALX-201-059-U025 | |
| CHAPS | Sigma | 1.11662 | |
| DMEM, low glucose, pyruvate | Thermoscientific | 31885023 | |
| DMSO | Sigma | D8418-250ML | |
| EDTA | Sigma | 03685-1KG | |
| EGTA | Sigma | 324626-25GM | |
| Etoposide | MedChem Express | HY-13629 | |
| FBS | Thermoscientific | 26140 | |
| Flow cytometry tubes | Falcon | 352008 | |
| Flowjo | Flowjo | A license in required but any program that can analyze .fcs files will suffice | |
| Glycerol | Sigma | G5516-500ML | |
| HEPES | Sigma | H4034 | |
| Magic Red caspase-3/7 assay kit; flow cytometry or imaging | Immunochemistry Technologies | 935 | |
| M-CSF | ebioscience | 14-8983-80 | now a subsidiary of Thermoscientific |
| M-Plex | Tecan | any fluorometric reader will work with the appropriate excitation and emission detectors | |
| NaCl | Roth | 3957.1 | |
| PBS pH 7.4 | Thermoscientific | 10010023 | |
| Penicillin-Streptomycin-Glutamine (100X) | Thermoscientific | 10378016 | |
| Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23225 | protein concentration assay |
| protease inhibitors | Biomol | P9070.100 | |
| Smac mimetic, Compound A | Tetralogics | also known as 12911; structure shown in supplementary figures of Vince et al., Cell 2007 | |
| Sodium Fluoride | Sigma | S7920-100G | |
| sodium orthovanadate | Sigma | S6508-10G | |
| sodium pyrophosphate | Sigma | P8010-500G | |
| Staurosporine | MedChem Express | HY-15141 | |
| sucrose | Sigma | 1.07687 | |
| Tris Base | Sigma | T1503-1KG | |
| Triton X100 | Sigma | T8787-50ML | |
| TrypLE | Thermoscientific | A1285901 | In the protocol, it is listed as Trypsin |
参考文献
- Ramirez, M. L. G., Salvesen, G. S. A primer on caspase mechanisms. Seminars in Cell and Developmental Biology. 82, 79-85 (2018).
- Nicholson, D. W., Thornberry, N. A. Caspases: Killer proteases. Trends in Biochemical Sciences. 22 (8), 299-306 (1997).
- Shalini, S., Dorstyn, L., Dawar, S., Kumar, S. Old, new and emerging functions of caspases. Cell Death and Differentiation. 22 (4), 526-539 (2014).
- Van Opdenbosch, N., Lamkanfi, M. Caspases in cell death, inflammation, and disease. Immunity. 50 (6), 1352-1364 (2019).
- Kesavardhana, S., Malireddi, R. K. S., Kanneganti, T. -. D. Caspases in cell death, inflammation, and pyroptosis. Annual Review of Immunology. 38, 567-595 (2020).
- Fu, T. -. M., et al. Cryo-EM structure of caspase-8 tandem DED filament reveals assembly and regulation mechanisms of the death-inducing signaling complex. Molecular Cell. 64 (2), 236-250 (2016).
- Jiang, X., Wang, X. Cytochrome c promotes caspase-9 activation by inducing nucleotide binding to Apaf-1. Journal of Biological Chemistry. 275 (40), 31199-31203 (2000).
- Christgen, S., Place, D. E., Kanneganti, T. -. D. Toward targeting inflammasomes: Insights into their regulation and activation. Cell Research. 30 (4), 315-327 (2020).
- Shi, J., et al. Cleavage of GSDMD by inflammatory caspases determines pyroptotic cell death. Nature. 526 (7575), 660-665 (2015).
- Sborgi, L., et al. GSDMD membrane pore formation constitutes the mechanism of pyroptotic cell death. EMBO Journal. 35 (16), 1766-1778 (2016).
- Rühl, S., et al. ESCRT-dependent membrane repair negatively regulates pyroptosis downstream of GSDMD activation. Science. 362 (6417), 956-960 (2018).
- Declercq, W., Takahashi, N., Vandenabeele, P. Dual face apoptotic machinery: From initiator of apoptosis to guardian of necroptosis. Immunity. 35 (4), 493-495 (2011).
- Newton, K., et al. Cleavage of RIPK1 by caspase-8 is crucial for limiting apoptosis and necroptosis. Nature. 574, 428-431 (2019).
- Fritsch, M., et al. Caspase-8 is the molecular switch for apoptosis, necroptosis and pyroptosis. Nature. 575, 683-687 (2019).
- White, M. J., et al. Apoptotic caspases suppress mtDNA-induced STING-mediated type I IFN production. Cell. 159 (7), 1549-1562 (2014).
- Rongvaux, A., et al. Apoptotic caspases prevent the induction of type I interferons by mitochondrial DNA. Cell. 159 (7), 1563-1577 (2014).
- Nakajima, Y. -. I., Kuranaga, E. Caspase-dependent non-apoptotic processes in development. Cell Death and Differentiation. 24, 1422-1430 (2017).
- Kumar, S., Dorstyn, L., Lim, Y. The role of caspases as executioners of apoptosis. Biochemical Society Transactions. 50 (1), 33-45 (2022).
- Agniswamy, J., Fang, B., Weber, I. T. Plasticity of S2-S4 specificity pockets of executioner caspase-7 revealed by structural and kinetic analysis. FEBS Journal. 274 (18), 4752-4765 (2007).
- McStay, G. P., Salvesen, G. S., Green, D. R. Overlapping cleavage motif selectivity of caspases: implications for analysis of apoptotic pathways. Cell Death and Differentiation. 15 (2), 322-331 (2008).
- Thornberry, N. A., et al. A combinatorial approach defines specificities of members of the caspase family and granzyme B. Functional relationships established for key mediators of apoptosis. Journal of Biological Chemistry. 272 (29), 17907-17911 (1997).
- Vince, J. E., et al. IAP antagonists target cIAP1 to induce TNFalpha-dependent apoptosis. Cell. 131 (4), 682-693 (2007).
- McComb, S., et al. cIAP1 and cIAP2 limit macrophage necroptosis by inhibiting Rip1 and Rip3 activation. Cell Death and Differentiation. 19 (11), 1791-1801 (2012).
- Wong, W. W. -. L., et al. cIAPs and XIAP regulate myelopoiesis through cytokine production in an RIPK1- and RIPK3-dependent manner. Blood. 123 (16), 2562-2572 (2014).
- Liu, X., Quan, N. Immune cell isolation from mouse femur bone marrow. Bio-Protocol. 5 (20), e1631 (2015).
- Lin, H., Chen, C., Chen, B. D. Resistance of bone marrow-derived macrophages to apoptosis is associated with the expression of X-linked inhibitor of apoptosis protein in primary cultures of bone marrow cells. Biochemical Journal. 353 (Pt 2), 299-306 (2001).
- . Caspase activity assay buffer. Cold Spring Harbor Protocols. , (2015).
- Mackridge, A., Rowe, P. One-way analysis of variance (ANOVA) - Including Dunnett's and Tukey's follow up tests. Practical Approach to Using Statistics in Health Research: From Planning to Reporting. , 93-103 (2018).
- Chen, H., Ning, X., Jiang, Z. Caspases control antiviral innate immunity. Cellular and Molecular Immunology. 14 (9), 736-747 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved