Method Article
PyDesigner v1.0: 拡散磁気共鳴イメージングのためのDESIGNERパイプラインのPythonic実装
* これらの著者は同等に貢献しました
要約
ここでは、Pythonベースの拡散磁気共鳴イメージング(dMRI)処理パイプラインであるPyDesignerを紹介します。これは、典型的なdMRIアーティファクトを補正し、拡散テンソルイメージング(DTI)、拡散尖度イメージング(DKI)、ファイバーボールイメージング(FBI)、微細構造モデリング(白質完全性[WMTI]およびファイバーボール白質[FBWM])、およびトラクトグラフィー出力を生成することができます。
要約
PyDesignerは、dMRIの前処理とテンソル推定のためのオリジナルのDiffusion parameter EStImation with Gibbs and NoisE Removal(DESIGNER)パイプライン(Dv1)に基づくPythonベースのソフトウェアパッケージです。このソフトウェアは非営利の研究のために公開されており、臨床ケアに使用することはできません。PyDesignerは、FSLとMRtrix3のツールを組み合わせて、ノイズ除去、ギブスリンギング補正、渦電流運動補正、ブレインマスキング、画像スムージング、およびライシアンバイアス補正を実行して、複数の拡散測定の推定を最適化します。Windows、Mac、Linux のプラットフォーム間で使用でき、DKI、DTI、WMTI、FBI、FBWM データセット、トラクトグラフィー ODF および .fib ファイルから一般的に使用されるメトリクスを正確に導き出すことができます。また、ファイル形式に依存せず、.nii、.nii.gz、.mif、および dicom 形式の入力を受け入れます。ユーザーフレンドリーでインストールが簡単なこのソフトウェアは、S/N比グラフ、外れ値ボクセル、ヘッドモーションを示す品質管理メトリックも出力し、データの整合性を評価します。さらに、このdMRI処理パイプラインは、複数のエコータイムデータセット処理をサポートし、パイプラインのカスタマイズ機能を備えているため、ユーザーはさまざまなユーザーのニーズを満たすために、どのプロセスを採用し、どの出力を生成するかを指定できます。
概要
拡散MRI(dMRI)は、脳内の微細構造特性の非侵襲的研究に広く適用されています。多くのdMRI法が提案されていますが、一般的に使用されるのは、拡散テンソルイメージング(DTI)と拡散性尖度イメージング(DKI)の2つです。これらの手法は密接に関連しており、DKI は拡散性非ガウス性1 の定量化を含む DTI の拡張です。どちらもさまざまなスカラー拡散測定を提供し、白質繊維トラクトグラフィーの構築を可能にします。DTIとDKIの重要な利点は、拡散物理学の強固な基盤を持っているため、その有効性が組織の微細構造に関する詳細な仮定に依存しないことです2,3。これにより、DTIとDKIを脳と全身に適用し、あらゆる年齢や病状の参加者に適用することができます。
生の拡散強調画像(DWI)は、信号ノイズ、動き、ギブスリンギング、渦電流歪みなどの複数の要因によって劣化するため、拡散量を計算する前に前処理を行う必要があります4。DWIの前処理は、現在高度に発展している活発な研究分野です。しかし、必要な処理ステップを1つのパイプラインに結合して一貫した結果を得ることは、dMRI取得の詳細に応じていくつかのユーザー定義の設定を調整する必要があり、前処理ステップの実行順序が結果に影響を与えるため、困難です。このため、DWI5 の前処理を最適化、標準化、合理化するために、2016 年に Diffusion パラメータ EStImation with Gibbs and NoisE Removal (DESIGNER, GitHub: NYU-DiffusionMRI/DESIGNER) パイプラインが最初に提案されました。DESIGNER Dv1は、FSL、MRtrix3、MATLAB、Pythonに組み込まれたソフトウェアツールに依存して、前処理による画像補正と拡散/尖度テンソル推定5を含むシームレスで完全なDWIプロセスを作成します。前処理ステップのオンとオフを切り替える制御フラグにより、DWI修正を選択的に実行できます。DESIGNERは、(i)Marchenko-Pastur主成分分析(MP-PCA)ノイズ除去6、 (ii)ギブスリンギング補正7、(iii)エコー平面イメージング(EPI)歪み補正8、渦電流補正9、モーション補正10、および外れ値置換11、(iv)B1バイアスフィールド補正、(v)ブレインマスク生成、(vi)スムージング、(vii)ライシアンノイズバイアス補正、および(viii)b0正規化。この特定の順序で前処理を行うことで、精度と実効S/N比(SNR)5の両方が向上します。PyDesignerの各ステップはオプションであり、ユーザーの好みに基づいて使用またはスキップできることに注意してください。たとえば、スムージングは、一部のデータセットでは必要な前処理手順ではない場合があります。フィルタリングエラーを軽減するのに役立ちますが、超高品質のデータセットには必要ないかもしれません。そのため、ユーザーはデータに必要な手順のみを使用することを選択できます。
プラットフォーム間でのDESIGNERの実装は、オペレーティングシステムや環境設定の違いにより困難です。特に、DESIGNER Dv1 が主に MATLAB で記述されているという事実は、Python-MATLAB インターフェースを有効にするために必要な複雑な構成要件から生じる移植性の重大な問題を引き起こします。さらに、MATLAB、Python、および依存関係のバージョンのさまざまな組み合わせにより、このパイプラインの再現性が損なわれます。そのため、私たちは完全にPythonベースのPyDesignerを開発しました。このdMRI処理パイプラインは、シームレスな前処理を可能にするだけでなく、パイプラインをDockerコンテナに組み込むこともできるため、移植性と再現性が大幅に向上します。さらに、MATLAB コードを置き換えることで、PyDesigner はすべてのライセンス料を回避し、アクセシビリティを向上させます。
このdMRI処理パイプラインは、DESIGNERによって導入されたハンズフリーアプローチを強化し、いくつかの新機能を追加し、前処理を実行するためのFSLおよびMRtrix3のツールを組み込んでいます。Numpy12、SciPy13、CVXPY14,15 などの標準的な数学 Python ライブラリを使用して、DESIGNER の MATLAB 部分を Python コードに置き換えました。このソフトウェアは、Github16で公開されており、入手できます。このホワイトペーパーの目的は、当社のソフトウェアを検証したり、類似のソフトウェアと比較したりすることではなく、ユーザーが PyDesigner でデータを処理することを選択した場合に、その手順を追ったガイドを提供することです。
プロトコル
ソフトウェアの開発とテストに使用されたすべてのデータは、施設内審査委員会が承認した研究プロトコルの下で収集されました。
注: PyDesigner は MacOS 10.14 上に構築されており、Windows で実行するには Windows Subsystem for Linux (WSL) が必要です。同じコマンドをLinux/Macシステムで使用できます。
1.オペレーティングシステムの互換性
- Windows システムの場合は、リンクに従って WSL17 をインストールし、手順 2.6 に進みます。
- Unix ベースのシステム (Linux や Mac など) の場合は、セクション 2 に進みます。
注: Unix ベースのシステムでは、PyDesigner とその依存関係をネイティブにインストールできます。
2. FSLのインストール
注: すべての PyDesigner テストは FSL バージョン 6.0.2 で行われており、v6.0.2 以降を使用することをお勧めします。
- FSL wiki18 に移動し、[ ダウンロード/インストール] を選択します。Windows ユーザーの場合は、手順 2.6 に進みます。
- [FSL のインストール] 見出しの下にある [FSL のダウンロード ] を選択します。次に、 FSL Install Software License を選択し、契約を読み、 同意する を選択します。
- 必須フィールドに入力し、[ 登録] を選択します。
- [FSL のダウンロード] リンク (ページの最初のリンク) を選択します。次に、インストール手順のリンク (ページ上の 2 番目のリンク) を選択します。次の手順に従って、各オペレーティングシステムのFSLをダウンロードします。
注:FSLのインストールには約10分かかります。 - インストールが成功したことを確認するには、新しいターミナル・ウィンドウに次のように入力します。
$ flirt -version.これにより、バージョン番号が返されます。ステップ 3 に進みます。 - Windowsユーザーの場合は、FSL19のWSLインストールガイドに従ってください。
3. Conda 環境を作成する
- conda パッケージをインストールして、Python 環境を管理します。ユーザーはMinicondaまたはAnacondaのいずれかを選択できます。
- Miniconda / Anaconda20をダウンロードします。MacおよびLinuxユーザーは、手順3.2に進みます。
- Windows の場合は、WSL21 内で conda を設定します。
注: 次の手順は Miniconda 用ですが、同じコマンドが Anaconda でも機能します。conda は Python 3.7 をサポートしなくなりましたが、Python 3.7 を使用して conda 環境を作成することは可能であり、このプロトコルを説明どおりに正確に実装できます。
- ターミナルに「
$ conda create -n mri python=3.7」と入力して、conda環境を作成します。この conda 環境は、PyDesigner 関連のすべてのアクティビティに使用します。Proceed ([y]/n)?が表示されたら、「y」と入力します。
注: -n フラグ: これを使用して、conda 環境の名前を示します。ビデオでは、conda 環境の名前は mri です。
注:python=: PyDesigner はこのバージョンでビルドおよびテストされているため、バージョン 3.7 をお勧めします。
4. MRtrix3 のインストール
- 次のコマンドを入力して、conda環境をアクティブ化します:
$ conda activate mri - MRtrix322 のダウンロードページを開き、適切なオペレーティングシステムのダウンロード手順に従います。インストールコードは、特定の依存関係の確認を求めます。「
y」と入力して、ダウンロードプロセスを続行します。- MRtrix3のドキュメントを印刷してインストールを確認するには、ターミナルに次のように入力します。
$ mrinfo -h
- MRtrix3のドキュメントを印刷してインストールを確認するには、ターミナルに次のように入力します。
5. PyDesignerをインストールする
- 新しいターミナル・ウィンドウを開き、ターミナルに次のように入力してconda環境をアクティブ化します:
$ conda activate mri - 次のコマンドを実行します。
$ pip install PyDesigner-DWI - インストールが成功したことを確認するには、ターミナルに次のように入力してバージョン番号を印刷します。
$ pydesigner -v- 「module not found」というエラーが表示された場合は、プロトコルの最後にある「トラブルシューティング」セクション(セクション10)を参照してください
注: $ pydesigner -h は、ソフトウェアのコマンドと構文を説明するヘルプドキュメントを出力します。PyDesignerはGithubからダウンロードすることもできます。Webサイト23のインストール手順を参照してください。
- 「module not found」というエラーが表示された場合は、プロトコルの最後にある「トラブルシューティング」セクション(セクション10)を参照してください
6. データの準備
注: PyDesigner は、.nii、.nii.gz、.mif、DICOM などの複数のファイルタイプで動作します。このビデオでは、このdMRI処理パイプラインを使用する前に、DICOMをソートしてNIfTIに変換しています。NIfTI ファイルは匿名化されており、シーケンスごとに 1 つのファイルがあるため、操作が簡単です。
- DICOM を並べ替えるには、DicomSort ツール24 を使用します。
注:同じタスクを実行する他のDICOMソートツールを使用できます。 - DICOMをソートした後、dcm2niix25を使用してNIfTIファイルに変換します。
- dcm2niixをインストールするには、ターミナルに次のコマンドを入力します。
$ sudo apt installdcm2niix - ダウンロードが完了したら、ターミナルに次のように入力して、dcm2nii のドキュメントを印刷してインストールを確認します。
$ dcm2niix -h
- dcm2niixをインストールするには、ターミナルに次のコマンドを入力します。
- dcm2niixを実行するには、ターミナルに次のコマンドを入力します。
$ dcm2niix -f %s_%p_%d -o pathtosavefolder/ pathtorawdicoms/
注: -f: 出力ファイルのファイル名構造を指定するには、このタグの後に任意のファイル名構造を指定します。ビデオでは -f %s_%p_%d を使用しています。%s、%p、および %d は、それぞれシリーズ、プロトコル番号、および説明の表記です。どのファイル構造でも機能します。dcm2niix から出力されるのは、.bval、.bvec、.json、および .nii ファイルです。TOPUPデータを取得した場合は、.jsonファイルと.nii TOPUPファイルがあります。
7. PyDesigner の基本的な使い方
注: PyDesigner は、処理パラメータとコンピューターの機能によっては、1 つのデータ セットに最大 1.5 時間かかる場合があります。
- 標準処理を実行するには、フラグ
-sまたは-standardを使用します。このコマンドは、推奨される前処理パイプラインを次の順序で実行します: ノイズ除去6、ギブスリンギング補正7、EPI 歪み補正8、渦電流運動補正9、脳および脳脊髄液 (CSF) マスクの作成、スムージング、ライシアン バイアス補正、外れ値検出11、テンソル推定、パラメトリック マップの作成。 - 基本的な PyDesigner コマンドを実行するには、まずターミナルに次のコマンドを入力して conda 環境をアクティブにします。
$ conda activate mri - ソフトウェアのヘルプ・ドキュメントを印刷するには、次のコマンドを入力します。
$ pydesigner -h。このドキュメントでは、PyDesigner で使用可能なすべてのコマンドと構文について説明します。 - 次の例を使用して、コマンドを構造化します。
$ pydesigner -s --verbose --rpe_pairs 1 -o ./output_folder/ ./input_folder/input1.nii ./input_folder/input2.nii - 次の重要なフラグに注意してください。
--verbose: これを使用して、PyDesigner コンソールがデータを処理しているときに表示します (必須ではありません)。--rpe pairs #: TOPUP シーケンスを使用しているユーザーは、TOPUP を使用して EPI 歪み補正8 プロセスを高速化できます。# は、TOPUP シーケンスの B0 ボリュームがいくつ使用されるかを示します。#=1 を使用すると、歪みフィールドの過大評価を防ぎ、このフィールドの作成に必要な時間を短縮できます。ユーザーは日常的に #=1 を使用しており、信頼性の高い結果が得られています。#=1 を使用すると、PyDesigner はデフォルトで最初のボリュームを取ります。
8. PyDesignerの高度な使用法:ユーザーの個々のニーズに合わせてソフトウェアを調整する
注: PyDesigner を調整する場合は、 -s フラグを省略します。このフラグは自動前処理のコマンドです。
- ターミナルに次のコマンドを入力して、conda環境をアクティブ化します。
$ conda activate mri - ソフトウェアのWebサイト23 にフラグのリストを参照してください。each フラグを使用すると、ユーザーは PyDesigner の各部分を個別に実行できます。
- コマンドを構造化します。ユーザーが実行したい各シーケンスのフラグから開始します。次に、PyDesigner を入力ファイルと出力ファイルに誘導します。例えば:
$ pydesigner flag1 flag2 flag3 -o ./output_folder/ ./input_folder/input1.nii - 例 1: テンソル フィッティングを単独で実行します (追加の前処理は不要)。
- ターミナルに次のコマンドを入力して、conda環境をアクティブ化します。
$ conda activate mri - フラグのリストについては、PyDesignerのWebサイト23 を参照してください。この例では
-mを使用します。 -m: このフラグを追加して、テンソル フィッティングを脳内ボクセルのみに制限します。これにより、テンソルフィッティング+外れ値の検出が高速化されます。- 次の例を使用して、コマンドを構造化します。
$ pydesigner -m -o ./output_folder/ ./input_folder/input1.nii
注:テンソルフィッティングの入力は、前処理された.niiファイルです。
- ターミナルに次のコマンドを入力して、conda環境をアクティブ化します。
- 例 2: ノイズ除去を単独で実行します (追加の前処理は行いません)。
- ターミナルに次のように入力して、conda環境をアクティブ化します。
$ conda activate mri - フラグのリストについては、PyDesignerのWebサイト23 を参照してください。この例では
-nを使用します。 - -n: このフラグを追加して、ノイズ除去手順を完了します。各被験者の出力フォルダにあるDWI_preproccessed.niiというラベルの付いた、以前に前処理されたDWIファイルを入力します。
- 次の例を使用して、コマンドを構造化します。
$ pydesigner -n -o ./output_folder/ ./input_folder/input1.nii
- ターミナルに次のように入力して、conda環境をアクティブ化します。
9. 今後の PyDesigner のアップデート
注:更新はWebサイト23で見つけることができます。
- PyDesigner を更新するには、新しいターミナルウィンドウに次のように入力します。
$ pip install --upgrade PyDesigner-DWI
注: PyDesigner が Github16 を通じてインストールされた場合は、git を使用して更新する必要もあります。
10. インストールのトラブルシューティング
- PyDesignerをダウンロードするときに、Pythonモジュールが不足していると「モジュールが見つかりません」というエラーコードが表示されます。
- 欠落しているモジュールを修正するには、ターミナルに次のように入力します。
$ pip install nameofmodule - 次に、ターミナルに次のように入力して、最初のインストールコマンドを繰り返します。
$ pip install PyDesigner-DWI - これで、PyDesignerが完全にインストールされたはずです。手順5.3に戻ってください。同じエラーが発生した場合は、欠落しているすべてのPythonモジュールがインストールされるまでこのプロセスを繰り返します。
- 欠落しているモジュールを修正するには、ターミナルに次のように入力します。
11. 出力エラーのトラブルシューティング
- 問題が発生した場合は、 log_command.json ファイルを確認して、前処理エラーを特定します。中間ファイルを確認して、特定のエラーを特定します。
- トラブルシューティングのヘルプについては、GitHub ページ16 でリクエストを送信してください。
12. サンプルデータセットを使用したPyDesignerの実行
- OpenNeuroからサンプルデータセットをダウンロードする26.
注: このサンプル データセットには、生データと前処理済みデータの両方が含まれています。生データは、PyDesigner のサンプル処理の入力として意図されています。前処理されたデータは、iMac OS v12.4 の dMRI 処理パイプラインを使用して生データを処理した後に生成された出力で構成されます。ユーザーは、生データをダウンロードし、PyDesigner を使用してこれらのデータを処理し、その出力を提供された出力例と比較して、PyDesigner が正しく実行されていることを確認できます。生データはメインフォルダ(/PyDesigner Example Dataset/sub-01)に、前処理されたデータはderivativesフォルダ(/PyDesigner Example Dataset/derivatives/sub-01)に収録されています。特に指定がない限り、OpenNeuroデータセットはデフォルトのユーザーフォルダにダウンロードされます。 - Mac / Linuxで新しいターミナルを開くか、WindowsでWSLを開きます。
注: 標準の PyDesigner 処理パイプラインを実行するには、以下のコマンド構造の例に従ってください。以下のコマンドをターミナル/WSL に入力します。この例では、 "./user_download_folder/" はサンプル データ フォルダのパスを示します。このパスは、個々のユーザーのコンピューター上のサンプル データが格納されている場所に固有です。 - 出力用の新しいフォルダを作成するには、ターミナルに次のように入力します。
$ mkdir ./user_download_folder/ ds004945-download /PyDesigner_Outputs - conda環境をアクティブ化するには、ターミナルに次のように入力します。
$ conda activate mri - サンプル データセットで PyDesigner を実行するには、ターミナルに次のように入力します。
$ pydesigner - o ./user_download_folder/ds004945-download/PyDesigner_Outputs -s ./user_download_folder/ds004945-download/sub-01/dwi/sub-01_dwi.nii
注: この例では、./user_download_folder/ はサンプル データ フォルダーのパスを示します。このパスは、各ユーザーのコンピューターに固有です。ステップ 12.3 から 12.5 のコマンドは、ステップ 8.3 の構造に従います。このコマンドは、PyDesigner の標準処理フラグである -s フラグを使用して、ノイズ除去6、ギブスリンギング補正7、歪み除去8、9、ブレインマスキング、スムージング、およびライシアンバイアス補正の順に処理ステップを実行します。 - ソフトウェアが正しくインストールされていれば、新しく作成されたPyDesigner_Outputsフォルダにすべての出力が入力されます。PyDesigner によって生成された前処理ファイルを、OpenNeuro からダウンロードした "derivatives" フォルダーにあるサンプルデータセット前処理フォルダーと比較します。
- 代表的な結果のセクションを参照して、メトリクスマップをデータセットの結果の例のメトリクスマップと比較してください。
結果
PyDesignerソフトウェアは、生の拡散データに複数の画像補正ステップを適用し、分析を行う際に生ファイルの精度を向上させるために使用される出力を生成します。パイプラインで利用可能な各ステップは、序論で説明したように、査読済みの出版物5、6、7、8、9、10、11を通じて以前に検証されています。ソフトウェアからの出力は、トラクトグラフィープロファイル、接続性マトリックス、ボクセルワイズ解析、ROI解析、fODF解析、TBSS、フィクセルベースの解析などの解析に使用できます。
ソフトウェアウェブサイト23 は、前処理パイプライン中に生成されたすべての出力ファイルをリストアップしている。各プロンプトが実行されると、コンソールは完了したすべてのプロセスの説明を出力します。出力ファイルには、処理ファイル、メトリック、品質管理の 3 つのタイプがあります。出力ディレクトリー構造を 図 1 に示します。これらのファイルは、標準の前処理を使用する場合に使用できます(プロトコルのセクション7を参照)。ユーザーがより高度な使用法を必要とする場合(プロトコルのセクション8を参照)、使用可能な出力ファイルは、完了したプロセスによって異なります。
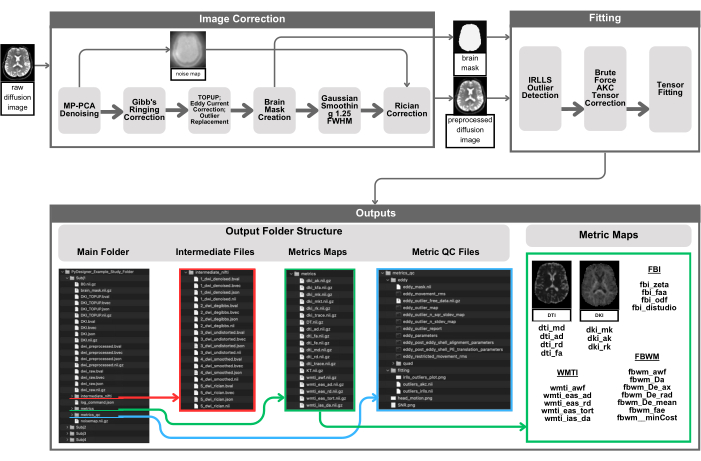
図 1: PyDesigner パイプラインの視覚的表現。 前処理は、入力された4D DWIをPyDesigner(左上)に提供することから始まり、次にMP-PCAノイズ除去が行われ、ノイズ除去された4D DWIと3Dノイズマップが生成されます。ノイズ除去された4D DWIは、その後、ギブスリンギング補正、ライシアンバイアス補正、TOPUP、渦流補正、および外れ値補正を受けます。その後、ブレインマスクは、後続のステップ、外れ値検出、およびテンソルフィッティングに対して計算され、ブレインマスク内でのみ実行することで計算を高速化します。出力は、メインのサブジェクト処理フォルダー (intermediate_nifti、メトリック、metrics_qc) のサブディレクトリにあります。PyDesigner の出力のスクリーンショットは、すべての可能な出力の完全なリストではなく、ユーザーが何を期待できるかの概略的な視覚的な例を提供することを意図していることに注意してください。出力 (最終出力と中間ファイルの両方) は、ユーザー入力データと使用される処理フラグによって異なります。 この図の拡大版を表示するには、ここをクリックしてください。
加工
処理ファイルは PyDesigner パイプライン中に使用され、ルート出力ディレクトリに格納されます。前処理の各ステップで、 図1に示すように、中間DWIファイルが出力フォルダ「intermediate_nifti」に保存されます。これらのファイルは、パイプラインの各ステップを個別に評価するために、処理または出力に関する問題に対処するときに参照する必要があります。
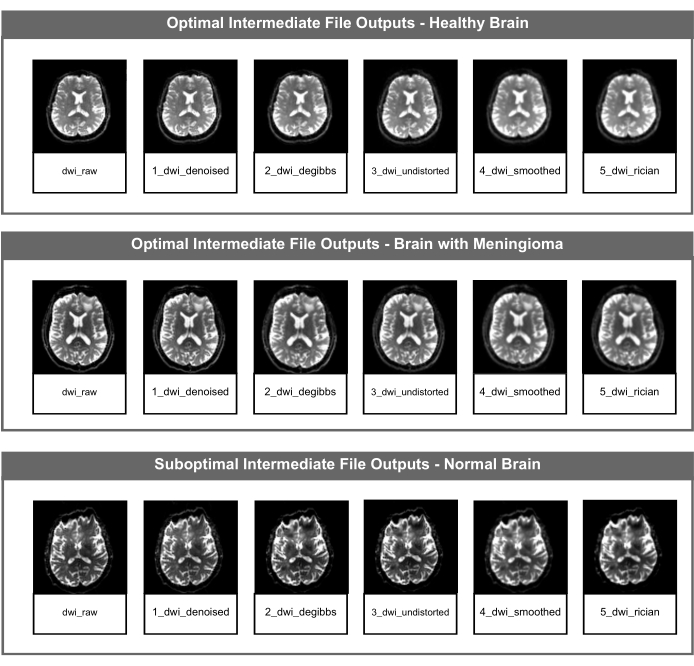
図2:最適および最適でない中間DWI NifTIファイル。 この図は、PyDesigner パイプラインの各画像補正ステップの中間 NIfTI ファイルを示しています。上の行は健康な成人の脳からのデータを使用した最適な中間ファイル出力の例、中央の行は病的な脳(髄膜腫)からのデータを使用した最適な中間ファイル出力の例、下の行は、脳の構造や健康とは無関係の感受性アーチファクトを持つ健康な成人の脳からのデータを使用した最適でない中間ファイル出力を示しています。 この図の拡大版を表示するには、ここをクリックしてください。
メトリック
このフォルダには、PyDesigner が計算するすべてのパラメトリック マップが含まれています ( 図 1 を参照)。これには、DTI/DKI、ファイバーボールイメージング(FBI)/ファイバーボール白質(FBWM)、および白質路完全性(WMTI)メトリックのパラメトリックマップが含まれます(表1)16。

表 1: DTI/DKI および FBI/FBWM メトリックの予想される値の範囲。 この表には、PyDesigner によって生成された堅牢な DTI、DKI、FBI、FBWM メトリクスと、それらの期待値範囲のリストが含まれています。各メトリックを導出するために必要なシーケンスと b 値 (s/mm2) もリストされています。リストされているDTIメトリックは、FA、MD、AD、およびRDです。リストされているDKIメトリックは、MK、AK、RK、およびKFAです。リストされているFBIの指標はFAAです。リストされているFBWMメトリックは、AWF、DA、DE_AX、DE_RAD、およびFAEです。
ユーザーは、平均拡散率(MD)、分数異方性(FA)、および平均尖度(MK)メトリックの視覚的および価値品質管理(QC)を実施して、最適でない結果を特定できます。これらのメトリクスが以下で説明する基準で最適でない場合、ユーザーは 図 2 に示す各中間ファイルを調べて、失敗した前処理ステップを特定する必要があります。
Visual QCは、最適でない結果(テンソルフィッティングの問題やアーティファクトなど)を特定するために使用されます。視覚的なQCにはImageJを使用して、ソフトウェアのデフォルトによる画像の操作が行われないようにすることをお勧めします。 図3 の一番上の行は、それぞれ0-3 μm2/ms、0-1 μm2/ms、0-2 μm2/msの生物学的にもっともらしい閾値を使用した典型的なMD、FA、およびMKメトリックマップを示しています(図3 [上段])。MDマップは、心室で最も高い値を持ち、皮質灰白質で高い値を持つ必要があります(図3A [上段])。FAマップには、脳全体に明確な明らかな白質路があるはずです(図3B [上段])。MKマップは、WMで高い値を持ち、灰白質とCSFで低い値を持つ必要があります(図3C [上段])。 図 3D [上段] は、テンソル近似の問題があるメトリック マップの例であり、その結果、ゼロ値のボクセルのクラスターが生成されます。問題が発生した場合は、log_command.json ファイルを確認して、前処理エラーがないか確認します。中間ファイルを確認して、特定のエラーを特定します。トラブルシューティングのヘルプについては、PyDesigner GitHub ページでリクエストを送信してください。
値の品質管理は、特定のメトリック マップのボクセルが各データセットの被験者間で比較的均一であるかどうかを識別するために使用されます。各マップとデータセットの期待値の範囲は、データと PyDesigner パラメーターによって異なります。私たちのテストデータセットでは、FSLeyes のデフォルトのヒストグラムビニングを使用して、MD、FA、MK の 8000 から 10,000、2500 から 4000、5000 から 13,000 の範囲で一貫したピークが見られました。 図 3 の一番下の行は、ヒストグラムの変動の例を示しています。 表 1 には、これらのメトリックで想定される X 軸の値が含まれています。ボクセルが高いか低いかは、データセットの特性であるか、アーティファクトまたは前処理の問題を示しています(図3D [下の行])。
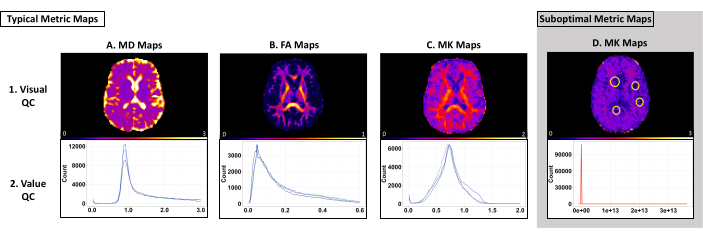
図3:PyDesignerのメトリックマップとヒストグラムの例(最適および非最適の結果)。上の行は、ビジュアルQCに使用される単一被験者のMD、FA、およびMKマップの例を示しています。一番下の行は、値QCに使用されるマルチサブジェクトヒストグラムを示しています。(A-C)視覚的で価値ある品質管理に合格するメトリックマップとヒストグラムの典型的な例。各メトリクスタイプのヒストグラム上の各青い線は、個々のデータセットを表しています。各データセットは同様の曲線をたどり、同様の範囲内に収まることに注意してください。(D)視覚的または価値の品質管理に合格しないメトリックマップの例。ヒストグラムの赤い線が A-C の曲線とは異なる曲線を示していることに注意してください。このメトリック マップで囲まれたゼロ ボクセルは、前処理中のテンソル フィッティングの問題によるものです (パネル D、上段)。このヒストグラムは、データセット内のボクセルが一般的に予想よりも高いまたは低い例です (パネル D、下の行)。この図の拡大版を表示するには、ここをクリックしてください。
品質管理
PyDesigner でデータが処理されたら、metrics_qc フォルダー ( 図 1 参照) を使用して、最適でないデータセットを特定する必要があります。PyDesigner は、データセットごとに、品質管理に使用される 3 つのプロットを出力します。
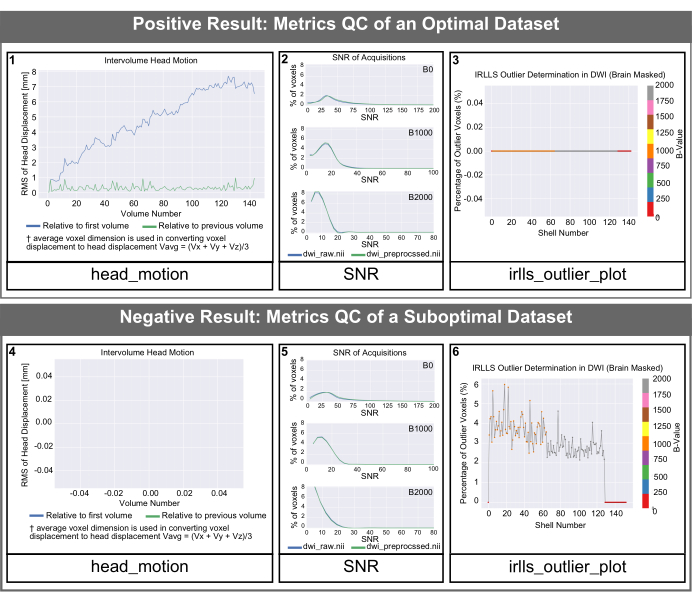
図4:PyDesignerで生成された最適なデータセットと最適でないデータセットのQCヒストグラム。 PyDesigner によって生成された SNR、ボリューム間ヘッド モーション、および外れ値のヒストグラム。どちらの行も、健康な成人の脳からのデータを表しています。一番上の行は、最適なデータセットの品質管理ヒストグラムの例です。一番下の行は、脳の構造や健康とは無関係の感受性アーティファクトを持つ最適でないデータセットの品質管理出力を示しています。PyDesigner からのデフォルト出力のラベルのテキストサイズは、この図に表示されるサイズよりも小さいことに注意してください。この図では、読みやすくするためにテキストサイズを大きくしています。 この図の拡大版を表示するには、ここをクリックしてください。
図 4 のhead_motionグラフは、最初の体積と前の体積に対する頭部の変位を示しています。図 4 (パネル 1) に示すように、ヘッドの変位は通常小さく、PyDesigner は、FSL プログラム Eddy とモーション用の TOPUP および渦電流モーション補正9 を使用して、標準処理パイプラインでこれらのモーション アーティファクトを調整します。最適でないデータセットの場合、図 4 (パネル 4) に示すように、head_motionグラフが空で表示されることがあります。これは、渦流動補正が失敗したため、PyDesignerがグラフを出力できなかったことを示しています。渦電流補正ログファイルは、metrics_qcフォルダ内のeddyサブフォルダにあります(図1を参照)。信号対雑音比(SNR)グラフには、3つのプロットが表示されます。各プロットは異なる b 値を対象としており、前処理されたデータと生データの両方が表示されます。最適なデータセットを得るには、生データの SNR ピークを ≥5 にする必要があります(図 4 [パネル 2])。最適でないデータセットでは、生データの SNR ピークは ≤3 になります (図 4 [パネル 5])。理想的には、すべての b 値の SNR ピークがわずかに増加するが、劇的には増加しないことをユーザーが確認する必要があります。外れ値プロットは、metrics_qc内のfitingフォルダにあり、データセット内の外れ値の割合を示しています(図4[パネル3および6])。優れたデータセットは、外れ値の割合が低く、通常は 5% 未満である必要があります (図 4 [パネル 3])。最適でないデータセットには、図 4 (パネル 6) に示すように、外れ値の割合が高くなります。
データセットの結果の例
PyDesigner がサンプルデータセットの処理を終了したら、すべての出力は "PyDesigner_Outputs" フォルダ内に含まれるはずです。これらの出力は、OpenNeuro からダウンロードしたサンプル データセット (MacOS 12.4 で処理) にパッケージ化された "derivatives" フォルダーにある出力と比較できます。ソフトウェアが正しく動作すれば、「PyDesigner_Outputs」と「派生物」のファイル構造はまったく同じになります。同様に、サブフォルダ「metrics_qc」にあるSNR、頭の動き、および外れ値のプロットは、 図5Aのプロットと一致する必要があります。メトリックマップ(サブフォルダ「metrics」にあります)は、FSLeyes、MRIcron、ImageJなどのイメージングソフトウェアを介して比較できます。前処理済み/メトリックからのFA、MD、およびMK値のヒストグラムを 図5Bに示します。 図 5B に示されているすべてのメトリック ヒストグラムは、 表 1 の推奨メトリック値スケールに基づいてスケーリングされることに注意してください。
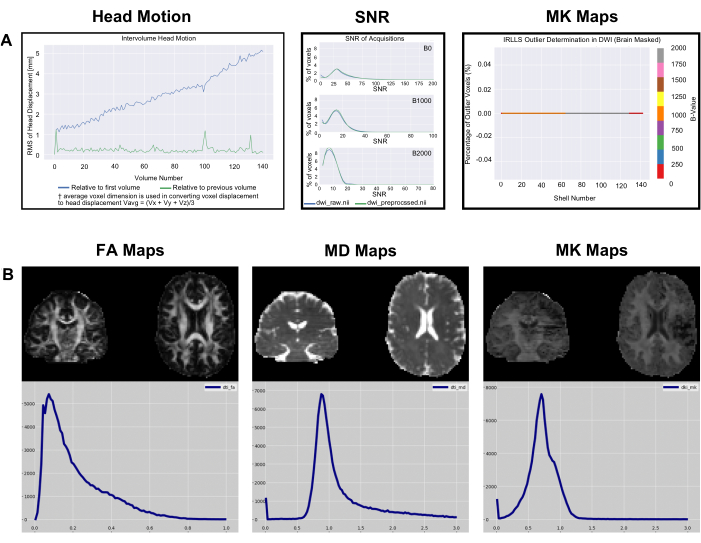
図5:データメトリックとメトリックQCヒストグラムの例(A)OpenNeuroからダウンロードしたサンプルデータについてPyDesignerによって生成されたSNR、ボリューム間ヘッドモーション、および外れ値のヒストグラム。この図のプロットでは、読みやすくするためにテキスト サイズが大きくなっていることに注意してください。(B) 単一被験者メトリックは、FSLeyes v6.0 を通じて視覚化された、同じデータセットの FA、MD、および MK のボクセル値カウントのヒストグラムをマッピングします。この図の拡大版を表示するには、ここをクリックしてください。
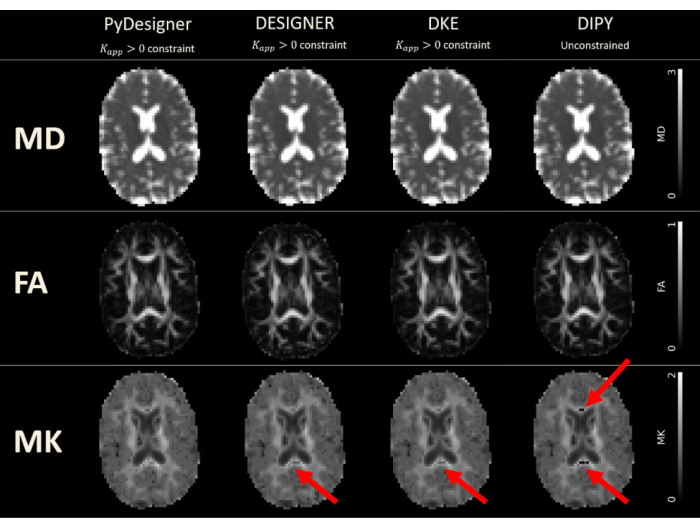
図6:PyDesigner、DESIGNER、DKE、およびDIPYから派生したDTIマップとDKIマップ。 テンソル フィッティングは、PyDesigner、DESIGNER5、および Diffusional Kurtosis Estimator (DKE)27 では Kapp > 0 制約を使用して実行されましたが、ソフトウェアの制限により、Diffusion Imaging in Python (DIPY)28 では制約なしフィッティングが使用されました。MDの単位はミリ秒あたりの二乗マイクロメートル(μm2 / ms)ですが、他のメトリックは無次元です。ギブスリンギング補正の一般的な問題によるドロップアウトは、DESIGNER、DKE、および DIPY によって作成された MK マップで確認できます。この図は、Dhimanらの許可を得て複製されています29。 この図の拡大版を表示するには、ここをクリックしてください。
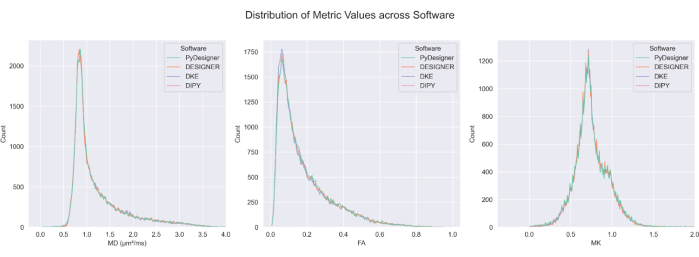
図7:パイプライン間のFA、MD、MKの比較。 PyDesigner、DESIGNER5、DKE27、および DIPY28 の FA、MD、および MK の計算値の CSF 除外脳の分布は、ほとんどのボクセルで類似しています。メソッド間でのメトリックマップの計算は同等です。この図は、Dhimanらの許可を得て複製されています29。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
PyDesigner を開発した主な動機は、DESIGNER の主要な要素を実装しながら、すべての MATLAB コードを Python に置き換えて、移植性とアクセシビリティを向上させることでした。PyDesigner と DESIGNER Dv1 は、ほぼ同じ出力を生成します29。それにもかかわらず、PyDesignerにはいくつかの追加オプション、デフォルト設定、およびマイナーなバグ修正が含まれています。オンラインのPyDesignerドキュメント16 には、これらが詳細に説明されています。
PyDesignerは、一般的に使用される拡散尖度推定器(DKE)27およびPythonでの拡散イメージング(DIPY)28、DKI分析ツール29(図6および図7を参照)と同様の結果ももたらしますが、DESIGNERで前処理されたdMRIに基づいて生成されたマップは、Ades-Aronらによって実証されているように、フィッティングアルゴリズムによりより正確であると考えられています.5。PyDesigner、DESIGNER5、DKE27、および DIPY28 の MD、FA、および MK メトリック マップの比較については、図 6 を参照してください。図7は、各パイプラインのMD、FA、MKヒストグラムの比較を示しています。制約付きテンソル フィッティング、外れ値検出、および見かけの尖度係数補正を組み合わせると、PyDesigner と DESIGNER5 に見られるように、より堅牢で正確なテンソル フィッティングが得られます。
DESIGNER Dv1 に対する PyDesigner の利点は、NeuroDock Docker container30 を介して利用できるため、移植性が大幅に向上し、インストールが簡素化されることです。このコンテナは、Windows、Mac OS、さまざまなLinuxディストリビューションなど、Dockerと互換性のあるすべての主要なOSプラットフォームで動作します。ユーザーからのフィードバックに基づき、PyDesigner v2.0 には PyDesigner Docker コンテナのアップデートが含まれる予定です。PyDesignerの新しいバージョンによって導入された改善と、マルチステージのDockerfileの導入により、ユーザーが直面している既存の問題はすべて解決されます。インストールに問題がある場合は、PyDesigner のディスカッションページ16 に質問を提出することをお勧めします。また、Docker のコンテナ テクノロジにより、ハイパフォーマンス クラスター (HPC) へのデプロイが容易になり、Docker と互換性のあるローカル クラスターで DWI を迅速にバッチ処理できます。
PyDesigner には、WMTI3、FBI、FBWM など、DKI を超える微細構造モデリング計算も含まれています。WMTIの場合、標準のDKIデータセットが適切であり、関連する微細構造パラメータがデフォルトで計算されます。ただし、WMTIの有効性は、FAが高い白質領域(つまり、FA≥0.4)に限定されていることを強調する必要があります。一部のWMTIメトリクスは、任意の所与のボクセル31における軸索の平行整列の仮定のために、精度が制限されている。FBI 32,33,34 は、大脳白質全体に適用可能な明確な dMRI 法であり、高い b 値 (つまり、b ≥ 4000 s/mm 2) と、少なくとも 64 の拡散符号化方向 (および b = 0 のデータ) でサンプリングされた dMRI データが必要です。FBIの主な出力は、各白質ボクセルの繊維配向密度関数(fODF)であり、これは白質トラクトグラフィーに使用でき、FBWMの入力として機能するほか、軸索内分数異方性(FAA)にもなります。FBWMは、DKIとFBIの両方からのdMRIデータを利用して、WMTIと同じパラメータを推定しますが、精度が向上しており、FA値に関係なく白質全体に適用できます。したがって、この追加データが利用可能な場合、FBWMの推定値はWMTI35の推定値よりも優先されます。FBIと同様に、FBWMは成人の脳白質でのみ検証されています。
PyDesigner によって提供される回転不変量 (RAS 方向) に加えて、DSIstudio (LPS 方向) に固有の .fib ファイルは、DTI、DKI、FGI 用に個別に生成されます。.fib ファイルには、各手法のトラクトグラフィー プロファイルを生成するための ODF 方向情報が含まれています。得られたトラクトグラフィープロファイルを使用して、被験者およびグループレベルで接続性マトリックスを生成できます。DKI および FBI の .fib ファイルには、DTI トラクトグラフィーでは利用できない交差ファイバー情報を中継する多方向の情報が含まれています。さらに、各 .fib ファイルには、各方法のさまざまな回転不変量が含まれており、トラクトグラフィーのシード、実行、および停止の基準としてさまざまな組み合わせで使用できます。DSIstudioの詳細については、彼らのウェブサイト36をご覧ください。
PyDesignerのもう1つの注目すべき機能は、NifTi(.nii)、圧縮NifTi(.nii.gz)、DICOM(.dcm)、MRtrixファイル形式(.mif)など、さまざまなファイル入力を処理できるマルチファイル入力です。PyDesignerは、入力形式に関係なく、ヘッダーメタデータから取得情報を自動的に識別し、それに応じて修正を実行するため、ハンズオフアプローチをサポートします。プロトコルの違いに関係なく、同じコマンド(上記参照)を使用して、さまざまなDWIを処理できます。したがって、このソフトウェアは、手動の前処理ステップとコマンドを最小限に抑えることで、時間と労力を節約します。最近リリースされたアップデート(v1.0-RC10)では、複数のエコータイム(マルチTE)データセットの互換性を導入することで、これが強化されました。これにより、PyDesigner は、TE から大きく独立する画像前処理ステップをマルチ TE DWI で実行し、ノイズやアーティファクトを最小限に抑えた画像を生成できます。次に、TE依存のテンソル計算が各TEで個別に実行され、拡散または尖度メトリックが生成されます。
PyDesigner v1.0 は、画像前処理パイプラインの設計におけるエンドポイントを表していないことに注意してください。画像処理ツールの開発と検証は、活発な研究分野です。特に、この原稿の準備中に、画像のノイズ除去とギブスリンギングの除去に関する新しい開発が提案され、その結果、Partial Fourier acquisitions35 で取得した dMRI の Gibbs アーティファクトのノイズ除去と補正の改善を含む改訂された Designer パイプライン Designer Dv237 がリリースされ、DESIGNER Github38 で入手可能になりました。同様に、BIDS39の組織コンプライアンスは、あらゆるMRI分析パイプラインの重要な側面です。BIDS 形式では、すべての MRI データ型に対して特定のセット構造を確立することで、より効率的なデータ共有とパイプラインの実装が可能になります。PyDesigner は現在 BIDS に準拠していませんが、PyDesigner の次期バージョンには BIDS 準拠の出力が含まれる予定です。さらに、PyDesigner はもともと Python バージョン 3.7 を使用して書かれていました。この公開の時点では、Python 3.7 は古くなっていると考えられています。ただし、この原稿に含まれる情報はPythonバージョンとは無関係です。すなわち、重要な前処理機能である本明細書に記載の手順(例えば、ノイズ除去、ギブスリンギング補正など)は、設置手順の変更に関係なく、同じ運用上および概念的なワークフローに従うことになる。重要なのは、すべてのPythonベースのソフトウェアと同様に、将来のPyDesignerバージョンは新しいバージョンのPythonに更新されるということです。私たちは、このような開発を時間をかけてさらに検証し、統合するよう努めています。
Pythonのバージョン更新に関連する新しいドキュメントを含む、PyDesignerの最新バージョンについては、読者は新しい分析を開始する前にWebサイトを参照し、PyDesignerに関する質問を送信できるディスカッションフォーラムに参加することをお勧めします16。移植性のための Docker 実装は NeuroDock30 と呼ばれ、PyDesigner とその依存関係が含まれており、さまざまなプラットフォームでの処理を可能にします。
開示事項
何一つ。
謝辞
Olivia Horn氏、Daniel Lench氏、Graham Warner氏との有益なディスカッションに感謝します。
本書で報告された研究は、米国国立衛生研究所(NIH)の助成金R01AG054159、R01AG057602、R01AG055132、R01DC014021、R01NS110347、R21DA050085、F31NS108623、P20GM109040、P50DC000422、T32GM008716、およびT32DC014435によって部分的に支援されました。内容は著者の責任であり、必ずしも国立衛生研究所の公式見解を表すものではありません。追加の資金はLitwin Foundationから提供されました。
資料
| Name | Company | Catalog Number | Comments |
| Python version 3.7 or above | Python Software Foundation | https://www.python.org/ | |
| FMRIB Software Library (FSL) verison 6.0.2 or above | University of Oxford Centre for Integrative Neuroimaging | https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/ | |
| MRtrix3 version 3.0_RC3 or above | numerous contributors | https://www.mrtrix.org/ | |
| Anaconda | Anaconda | https://anaconda.org/ | |
| Computer | Apple | Mac OS 10.14 | Built on Mac OS 10.14; tested on Mac OS 12.4, Mac OS 13, Windows 11 via WSL |
参考文献
- Jensen, J. H., Helpern, J. A. MRI quantification of non-Gaussian water diffusion by kurtosis analysis. NMR Biomed. 23 (7), 698-710 (2010).
- Basser, P. J. Relationships between diffusion tensor and q-space MRI. Magn Reson Med. 47 (2), 392-397 (2002).
- Fieremans, E., Jensen, J. H., Helpern, J. A. White matter characterization with diffusional kurtosis imaging. Neuroimage. 58 (1), 177-188 (2011).
- Le Bihan, D., Poupon, C., Amadon, A., Lethimonnier, F. Artifacts and pitfalls in diffusion MRI. J Magn Reson Imaging. 24 (3), 478-488 (2006).
- Ades-Aron, B., et al. Evaluation of the accuracy and precision of the diffusion parameter EStImation with Gibbs and NoisE removal pipeline. Neuroimage. 183, 532-543 (2018).
- Veraart, J., Novikov, D. S., Christiaens, D., Ades-aron, B., Sijbers, J., Fieremans, E. Denoising of diffusion MRI using random matrix theory. Neuroimage. 142, 394-406 (2016).
- Kellner, E., Dhital, B., Kiselev, V. G., Reisert, M. Gibbs-ringing artifact removal based on local subvoxel-shifts. Magn Reson Med. 76 (5), 1574-1581 (2016).
- Holland, D., Kuperman, J. M., Dale, A. M. Efficient correction of inhomogeneous static magnetic field-induced distortion in Echo Planar Imaging. Neuroimage. 50 (1), 175-183 (2010).
- Andersson, J. L. R., Sotiropoulos, S. N. An integrated approach to correction for off-resonance effects and subject movement in diffusion MR imaging. Neuroimage. 125, 1063-1078 (2016).
- Godenschweger, F., et al. Motion correction in MRI of the brain. Phys Med Biol. 61 (5), 32-56 (2016).
- Andersson, J. L. R., Graham, M. S., Zsoldos, E., Sotiropoulos, S. N. Incorporating outlier detection and replacement into a non-parametric framework for movement and distortion correction of diffusion MR images. Neuroimage. 141, 556-572 (2016).
- Harris, C. R., et al. Array programming with NumPy. Nature. 585 (7825), 357-362 (2020).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nat Methods. 17 (3), 261-272 (2020).
- Agrawal, A., Verschueren, R., Diamond, S., Boyd, S. A rewriting system for convex optimization problems. J Control Decis. 5 (1), 42-60 (2018).
- Diamond, S., Boyd, S. CVXPY: A Python-embedded modeling language for convex optimization. , Available from: http://arxiv.org/abs/1603.00943 (2016).
- PyDesigner. , Available from: https://github.com/muscbridge/PyDesigner (2023).
- How to install Linux on Windows with WSL. , Available from: https://learn.microsoft.com/en-us/windows/wsl/install (2023).
- FSL Wiki. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki (2024).
- Fsl Instillation. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FslInstallation/Windows (2024).
- Installing conda. , Available from: https://conda.io/projects/conda/en/stable/user-guide/install/download.html (2024).
- Steps to install Anaconda on Windows Ubuntu terminal. , Available from: https://gist.github.com/kauffmanes/5e74916617f9993bc3479f401dfec7da (2024).
- Install MRtrix3. , Available from: https://www.mrtrix.org (2024).
- PyDesigner Read the Docs. , Available from: https://pydesigner.readthedocs.io/en/latest/ (2024).
- dicomSort. , Available from: https://github.com/muscbridgelab/dicomSort (2024).
- dcm2niix. , Available from: https://github.com/rordenlab/dcm2niix (2024).
- PyDesigner Example Dataset. , Available from: https://openneuro.org/datasets/ds004945 (2024).
- Tabesh, A., Jensen, J. H., Ardekani, B. A., Helpern, J. A. Estimation of tensors and tensor-derived measures in diffusional kurtosis imaging. Magn Reson Med. 65 (3), 823-836 (2011).
- Garyfallidis, E., et al. Dipy, a library for the analysis of diffusion MRI data. Front Neuroinform. 8, 8(2014).
- Dhiman, S., et al. PyDesigner: A Pythonic implementation of the DESIGNER pipeline for diffusion tensor and diffusional Kurtosis imaging. bioRxiv. , (2021).
- Neurodock. , Available from: https://hub.docker.com/r/dmri/neurodock (2024).
- Jelescu, I. O., et al. One diffusion acquisition and different white matter models: How does microstructure change in human early development based on WMTI and NODDI. Neuroimage. 107, 242-256 (2015).
- Jensen, J. H., Russell Glenn, G., Helpern, J. A. Fiber ball imaging. Neuroimage. 124, 824-833 (2016).
- Moss, H. G., Jensen, J. H. High fidelity fiber orientation density functions from fiber ball imaging. NMR Biomed. 35 (1), e4613(2022).
- Moss, H. G., McKinnon, E. T., Glenn, G. R., Helpern, J. A., Jensen, J. H. Optimization of data acquisition and analysis for fiber ball imaging. Neuroimage. 200, 690-703 (2019).
- McKinnon, E. T., Helpern, J. A., Jensen, J. H. Modeling white matter microstructure with fiber ball imaging. Neuroimage. 176, 11-21 (2018).
- DSIstudio. , Available from: https://dsi-studio.labsolver.org (2024).
- Chen, J., et al. Optimization and validation of the DESIGNER dMRI preprocessing pipeline in white matter aging. ArXiv. , arXiv:2305.14445v2 (2024).
- DESIGNER. , Available from: https://github.com/NYU-DiffusionMRI/DESIGNER (2024).
- Gorgolewski, K. J., et al. The brain imaging data structure, a format for organizing and describing outputs of neuroimaging experiments. Sci Data. 3, 160044(2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved