このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
心血管疾患に関連する変動パターンと分子プロセスの解明のための教師なしマルチオミクス因子解析の応用
要約
複雑なマルチオミクスデータセットの教師なし分析のための柔軟で拡張可能なJupyterラボベースのワークフローを紹介します。これは、さまざまな前処理ステップ、マルチオミクス因子分析モデルの推定、およびいくつかのダウンストリーム分析を組み合わせたものです。
要約
病気のメカニズムは通常複雑で、いくつかの異なる分子プロセスの相互作用によって支配されています。複雑な多次元データセットは、これらのプロセスに関するより多くの洞察を生成するための貴重なリソースですが、このようなデータセットの分析は、たとえば、さまざまな病状、時点、さまざまな解像度でプロセスをキャプチャするオミクスなど、高次元性のために困難な場合があります。
ここでは、急性および慢性冠症候群の免疫応答を捕捉する血液サンプルから生成されたデータセットにマルチオミクス因子分析(MOFA)を適用することにより、このような複雑なマルチオミクスデータセットを教師なしの方法で分析および探索するアプローチを紹介します。このデータセットは、サンプルレベルのサイトカインデータ、血漿プロテオミクスおよび好中球prime-seq、シングルセルRNA-seq(scRNA-seq)データなど、異なる分解能の複数のアッセイで構成されています。患者ごとにいくつかの異なる時点といくつかの患者サブグループを測定することで、さらに複雑さが増します。
分析ワークフローでは、(1)データの前処理と調和、(2)MOFAモデルの推定、(3)ダウンストリーム分析など、いくつかのステップでデータを統合および分析する方法を概説しています。ステップ 1 では、さまざまなデータ型の特徴を処理し、低品質の特徴を除外し、それらを正規化して分布を調和させ、さらに分析する方法について概説します。ステップ2では、MOFAモデルを適用し、すべてのオミクスと特徴にわたってデータセット内の分散の主な原因を調査する方法を示します。ステップ3では、捕捉されたパターンの下流分析のためのいくつかの戦略を示し、それらを疾患の状態とそれらの状態を支配する潜在的な分子プロセスに関連付けます。
全体として、複雑なマルチオミクスデータセットの教師なしデータ探索のワークフローを提示し、他のコンテキストやマルチオミクスデータセット(例示的なユースケースで提示された他のアッセイを含む)にも適用できる、異なる分子特性で構成される変動の主軸の同定を可能にします。
概要
病気のメカニズムは通常複雑で、いくつかの異なる分子プロセスの相互作用によって支配されています。特定の疾患につながる、または疾患の進化を支配する複雑な分子メカニズムを解読することは、疾患の理解と治療のための新たな洞察を明らかにする可能性があるため、医学的関連性が高い課題です。
近年の技術の進歩により、これらのプロセスをより高い分解能(シングルセルレベルなど)で、またさまざまな生物学的層(DNA、mRNA、クロマチンアクセシビリティ、DNAメチル化、プロテオミクスなど)で同時に測定することが可能になりました。これにより、大規模な多次元生物学的データセットの生成が増加し、それらを共同で分析して、基礎となるプロセスについてより多くの洞察を得ることができます。同時に、生物学的に意味のある方法で異なるデータソースを組み合わせて分析することは、依然として困難な課題です1。
異なる技術的限界、ノイズ、および異なるオミクス間のばらつきの範囲が異なることが、1つの課題を提起します。例えば、シングルセルRNAシーケンシング(scRNA-seq)のデータは非常にまばらで、大きなテクニカルエフェクトやバッチエフェクトの影響を受けやすいです。さらに、フィーチャスペースは多くの場合非常に大きく、数千の測定された遺伝子またはタンパク質にまたがっていますが、サンプルサイズは限られています。これは、いくつかの病状、交絡因子、時点、および解決策を含む可能性のある複雑な設計によってさらに複雑になります。たとえば、提示されたユースケースでは、シングルセルレベルまたはサンプル(バルク)レベルのいずれかで異なるデータ型が使用可能でした。それに加えて、データが不完全である可能性があり、分析されたすべての被験者がすべての測定値を利用できるとは限りません。
これらの課題により、統合解析を行うことでプロセスの全体像が得られるだけでなく、1つのオミクスからの生物学的および技術的なノイズが他のオミクス3,4によっても補償される可能性があるにもかかわらず、異なるオミクスと含まれる特徴は依然として別々にのみ解析されることがよくあります2。マルチオミクスデータの統合解析を行うためには、ベイズ法、ネットワークベース法5,6、マルチモーダル深層学習7、行列因数分解8,9による次元削減法など、いくつかの異なる方法が提案されている。後者については、大規模なベンチマーク研究10の結果により、MOFA9(マルチオミクス因子分析)法は、データを臨床アノテーションにリンクする必要がある場合により適したツールの1つであることが示されています。
特に複雑な設定では、教師なし行列因数分解法は、複雑さを軽減し、さまざまなデータソースや特徴から共有シグナルと相補的なシグナルを抽出するための有用なアプローチです。複素空間を低ランクの潜在表現に分解することにより、データ内の分散の主要な原因を迅速に探索し、既知の共変量に関連付けることができます。同じパターンの変動が複数の特徴(遺伝子やタンパク質など)で共有されている場合、ノイズが減少する一方で、これはいくつかの要因に集約される可能性があります。正則化を使用してモデル係数のスパース性を増加させることができるため、このアプローチは、特徴空間が大きく、サンプル数が限られている設定に適しています9。
このプロトコルは、MOFAモデルを使用して、複雑なマルチオミクスデータセットを迅速に探索し、このデータセットを特徴付ける主要な変動パターンを抽出する方法を示す柔軟な解析ワークフローを提供します。ワークフローは、主に 3 つのステップで構成されています。最初のステップである「データの前処理と調和」では、さまざまな入力データタイプ(scRNA-seq、プロテオミクス、サイトカイン、臨床データ)に基づくデータ前処理のさまざまな戦略が提示されます。このプロトコルでは、さまざまな入力データセットの特徴を処理し、低品質の特徴を除外し、それらを正規化して分布を調和させる方法について詳しく説明します。また、これらの前処理の決定が下流の結果にどのように影響するかについても説明します。2番目のステップでは、MOFAモデルをデータに適用し、結果として生じる分散分解を使用して、異なるデータセットの統合を評価できます。3番目のステップでは、捕捉した因子を共変量にリンクし、それらの因子を定義する分子プログラムを明らかにする方法を示します。提示されたワークフローにより、冠症候群に罹患している患者のデータセットで臨床共変量に関連するいくつかの潜在因子を抽出し、以前のプロジェクト11から潜在的な基礎となる多細胞免疫プログラムを特定することができました。ここではこのデータセットを使用しますが、このプロトコルは他のオミクスを含む他のコンテキストにも簡単に適用できます。
このデータセットは、安定した慢性冠症候群(CCS)、急性冠症候群(ACS)、および健康な冠動脈症候群(非CCS)の対照群からのサンプルで構成されています(図1)。ACSは、既存のCCSのプラーク破裂によって引き起こされ、心筋への血流の急性の混乱とそれに続く心臓の虚血性損傷につながります。この損傷は、免疫系による炎症反応を引き起こし、その後修復段階を引き起こし、それは急性イベントの数日後まで続きます12。ACS患者のこの免疫応答を特徴付けることができるように、血液サンプルは4つの異なる時点で採取されました:急性(TP1);再開通後(14 [± 8] h)(TP2);60 [± 12] h後(TP3);退院前(6.5 [±1.5]日)(TP4)(図1A)。CCSおよび健康な冠状動脈の患者の場合、利用可能なタイムポイントは1つだけでした-(TP0)。すべての患者と時点について、血液サンプルに基づくさまざまなアッセイが測定されました:炎症の臨床マーカー(クレアチンキナーゼ(CK)、CK-MB、トロポニン、C反応性タンパク質(CRP))、末梢血単核細胞(PBMC)のscRNA-seq、サイトカイン分析、血漿プロテオミクス、好中球のprime-seq13 データ。
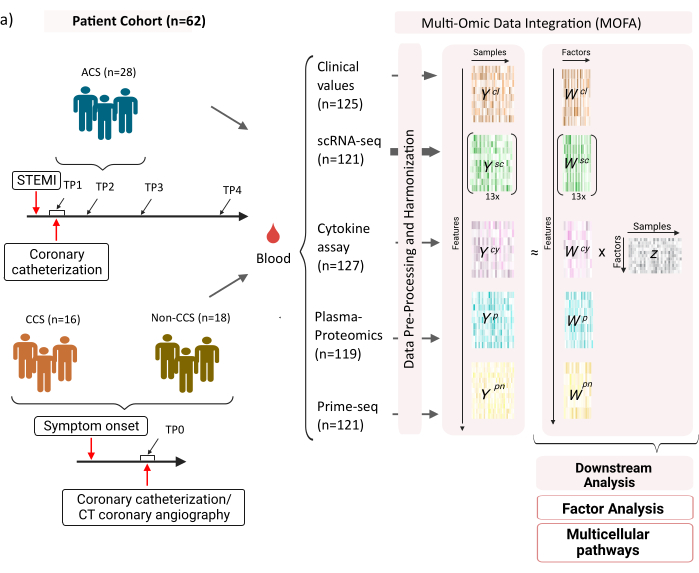
図1:心筋梗塞マルチオミクス入力データセット。 入力データセット: 分析されたデータには、急性冠症候群 (ACS)、慢性冠症候群 (CCS)、および健康な冠動脈 (非 CCS) の患者 (n = 62) の血液サンプルが含まれます。ACS 患者の場合、血液サンプルは 4 つの異なる時点 (TP1-4) に含まれ、CCS 患者と非 CCS 患者の場合は 1 つの時点 (TP0) に含まれていました。各患者と時点の組み合わせは、分析で個別のサンプルとして扱われます。サンプルでは、臨床血液検査(n = 125)、scRNA-seq(n = 121)、血漿プロテオミクス(n = 119)、サイトカインアッセイ(n = 127)、好中球prime-seq(n = 121)など、さまざまなオミクスアッセイがサンプルで測定されました。その後、記載されたプロトコルを適用して、すべてのオミクスにわたるデータを統合し、MOFAモデルを用いて探索し、さらに下流の分析(因子分析、パスウェイエンリッチメント)を行いました。 この図の拡大版を表示するには、ここをクリックしてください。
ここで紹介するワークフローの入力として、例えばscanpy14 の前処理チュートリアルで概説されているように、cellrangerおよび品質管理(QC)で処理した後のscRNA-seqデータから生のカウントを取ります。細胞タイプのアノテーションには、自動化されたAzimuth15 パイプラインを使用しました。次に、カウントは、各サンプルとセルタイプのすべてのセルの平均を取ることにより、各セルタイプのサンプルレベルで集計されます(疑似バルク集計)。プラズマプロテオミクスは正規化された強度と中央中心の強度として含まれ、好中球については、prime-seqからumi unique molecular identifier(UMI)エクソンカウントを取得します。サイトカインおよび臨床値については、以前の前処理は適用されていません。(実験的)データ生成に関するさらなる詳細は、対応する原稿11に概説されている。ここで紹介した結果は、scRNA-seqデータ中の細胞型に対して自動Azimuthアノテーションを使用した結果に基づいており、参照された論文で使用されたマーカーベースの戦略と比較した結果であるため、ここで提示された結果は、論文で提示されたものと類似していますが、まったく同じではありません。この原稿では、細胞型アノテーション戦略は分析の主要なパターンと生物学的解釈を変更しないが、モデルから生じる正確な値のわずかな変化は異なる可能性があることを示すことができます。全体として、入力データは、10,000を超える異なる特徴(遺伝子、タンパク質、臨床値)のさまざまな時点と測定レベル(単一細胞とバルク)を含む複雑な多次元データセットでした。MOFA分析に続く厳密な前処理とデータ調和戦略は、データを探索し、関連する免疫プログラムを抽出するための有用で迅速なツールであることが示されています。各時点と患者の組み合わせは、MOFA 分析では独立したサンプルとして扱われます。各データ型とセルの種類は、MOFA 分析では個別のビューと見なされます。
このプロトコルでは、ワークフローの入力データの準備、さまざまなワークフローステップの実行、構成のカスタマイズ、結果の数値の解釈、および解釈に基づく構成の反復調整を行う手順を提供します。プロトコルのさまざまなステップの概要、各ステップで必要な入力データセット、および結果として得られる図とデータセットは、テクニカルワークフローの概要に記載されています(図2)。
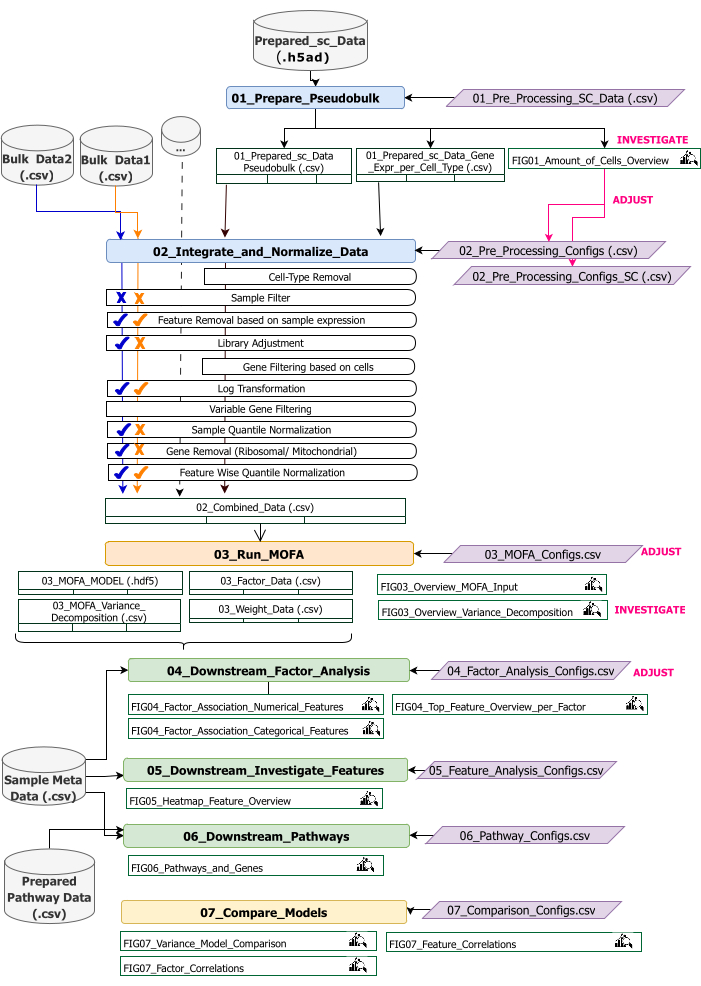
図2:テクニカルワークフローの概要。 マルチオミクスデータセットの解析ワークフローの概要。さまざまな要素が、さまざまな色と記号で強調表示されます。データの前処理と調和 (1) のステップに属する Jupyter Notebook は青色で色分けされています。「MOFA Model」(2)ステップに属するJupyter Notebookはオレンジ色で色づいています。「Downstream Analysis」(3)ステップに属するJupyter Notebookは緑色で色付けされています。結果の比較に使用する 1 つの Jupyter Notebook は、黄色で色付けされています。ワークフローの実行パラメータを変更できる設定ファイルは、紫色で強調表示されます。ワークフローの実行に必要な入力データセットは、データセット記号で示され、グレーでハイライト表示されます。ワークフローの実行中に生成されるすべての Figure 出力は、虫眼鏡記号で示されます。ワークフローの実行中に生成されたデータセットは、テーブルとして示されます。一般に、ワークフローは順番に実行されます:(1)データの前処理と調和は、scRNA-seq入力データに基づく疑似バルクテーブルの第一生成(01_Prepare_Pseudobulk)と、その後のこのデータと他のすべてのサンプルレベル(バルク)入力(02_Integrate_and_Normalize_Data)の統合と正規化の2つのステップで構成されます。このステップでは、設定ファイルを使用して、指定された前処理ステップと正規化ステップ(サンプルフィルターなど)をデータセットごとに個別に構成することができます。(2) 「MOFAモデル」:設定ファイル(03_MOFA_configs.csv)で指定された構成を使用して、最初のステップで生成された入力に対してMOFAモデルを実行します (3) 「ダウンストリーム分析」:生成されたMOFA結果に対する洞察を生成し、それらを「Sample Meta Data.csv」ファイルを介して入力として提供されるサンプルメタデータ(共変量)に関連付けるために、互いに独立して実行できる3つの異なるノートブックで構成されています。(4) 「モデル比較」:ステップ2で生成された異なるモデルを比較するために使用できる小さな別個のステップです。 この図の拡大版を表示するには、ここをクリックしてください。
ワークフローは、R と Python で記述された複数の Jupyter Notebook で構成されています (ワークフローの実行に R と Python 言語の知識は必要ありませんが、エラーが発生した場合に役立つ場合があります)。プロトコルのさまざまなステップで、パラメータは設定ファイル(名前に接尾辞「_Configs」を含む「.csv」ファイル)を介して変更されます。プロトコル内では、デフォルト設定から変更する必要があるパラメータのみを概説します。
他のいくつかのパラメータも、たとえば前処理をカスタマイズするために変更できます。これらのパラメータと説明のドキュメントは、ダウンロードしたリポジトリに含まれているファイル 'Documentation_Config_Parameter' に記載されています。
プロトコル
1.準備:技術的なセットアップとインストール
注: このプログラムを実行するには、wget、git、および Apptainer がデバイスにプリインストールされています。さまざまなシステム(Linux、Windows、Mac)にApptainerをインストールするためのガイドは、ここにあります:https://apptainer.org/docs/admin/main/installation.html。git のインストール情報は、https://git-scm.com/book/en/v2/Getting-Started-Installing-Git にあります。さまざまな入力データセットのサイズに応じて、適切なマシン(16 CPU、64 GB メモリ)でワークフローを実行することをお勧めします。提供されたサンプルデータを使用したスモークテストは、ローカルマシンで実行できます。サンプル データでプロトコルを実行した場合の手順と予想される出力は、補足ファイル 1 に記載されています。上記のデータセットで実行されるプロトコルの重要な手順については、 補足ビデオファイル1 を参照してください。
- コンソールを開き、すべての解析コードと出力を保存するフォルダを選択または作成します。ターミナルでコマンドcd path_to_folderを入力して、フォルダに移動します。
- Github(https://github.com/heiniglab/mofa_workflow)からコードリポジトリをダウンロードまたはクローンするには、ターミナルウィンドウにgit clone https://github.com/heiniglab/mofa_workflow.git と入力します。
- 必要なすべてのインストールを含むイメージを Zenodo からダウンロードするには、ターミナル ウィンドウに 「wget https://zenodo.org/records/11192947/files/mofa_image.sif」と入力します。
- ターミナルウィンドウに 「mkdir results 」と入力して、すべての結果データが保存されるフォルダを生成します。
- 解析で使用するすべての入力データを追加するフォルダを生成するには、ターミナルウィンドウに mkdir input_data と入力します。
- JupyterLab セッションを開始するコンテナを実行するには、ターミナルで次のコマンドを入力します。 apptainer run mofa_image.sif。コマンドによって返された URL をブラウザーにコピーすると、Jupyter-lab セッションが開きます (Jupyter-lab の詳細については、ソフトウェアのドキュメント16 を参照してください)。
注: ワークフローがラップトップ上でローカルに実行される場合は、代わりに コマンド apptainer exec mofa_image.sif jupyter-lab を使用することをお勧めします。これにより、ローカル ホスト アドレスが直接返されます。コンテナがクラスタ化されたコンピューティング環境内で実行される場合、 ssh 経由で実行できるポートフォワーディングの設定が必要になることがあります。
2. 初期化とデータ準備
- Jupyter-Lab セッションでは、左側のナビゲーション メニューを使用します。input_dataをダブルクリックしてinput_dataフォルダに移動します。
- 解析の入力として使用するすべてのデータセットを、 ドラッグ&ドロップを使用してinput_dataディレクトリにコピーします。現在配置されているフォルダからファイルをドラッグし、Jupyter-lab セッションの input_data フォルダの下の領域にドロップします。
注: すべてのデータセットは、.csv または .h5ad (シングルセル データの場合) 形式である必要があります。すべての.csvファイルには、一致する sample_id 列が含まれている必要があります (データセット全体で同一の ID を使用する必要があります)。他のすべての列はフィーチャとして使用されます。h5ad- ファイル内では、セル注釈には、sample_idとcluster_idを指定する 2 つの識別子が含まれている必要があります。これらは、集計と照合に使用されます。他の形式のオミクスデータセットは、使用する前に指定された.csv形式に変換する必要があります(図3)。 .h5seurat 形式で与えられた scRNA-seq データセットは、Jupyter-notebook: 00_Data_Conversion.ipynb を実行して .h5ad に変換できます。 - フォルダの記号をクリックし、フォルダmofa_workflow、スクリプト、および構成をダブルクリックして、構成フォルダに移動します。フォルダ内で、ファイルData_configs.csvをダブルクリックして開きます。
- [値] 列に、input_data (data_path) フォルダーと結果 (result_path) フォルダーのフォルダーへのパスを追加します。configuration_nameの値列に、保存されているすべてのファイルにファイル拡張子として追加される名前を追加します (このプロトコルは [Myocardial Infarction version1] MI_v1使用されます) (図 4)。
- 上部のメニューで[ ファイル]>[CSVファイルを保存 ]をクリックして、変更を保存します。
- 左側のナビゲーションメニューを使用して、scriptsをクリックしてscriptsフォルダに移動します。00_Configuration_Update.ipynb をダブルクリックして、初期化ノートブックを開きます。スクリプトを実行するには、上部にある[Restart kernel and run all cells]ボタンをクリックし、ポップアップで[Restart]をクリックします(図5)。
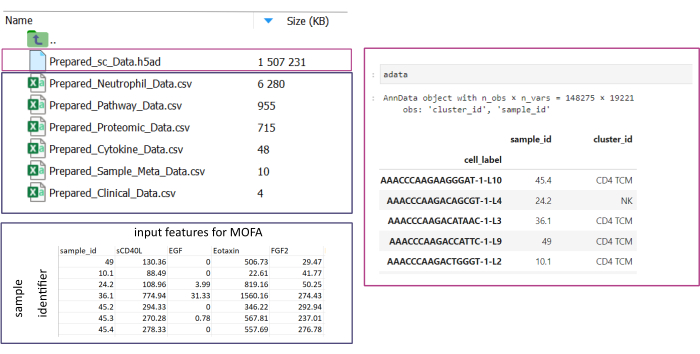
図3:データ入力とセットアップ。 ワークフローを実行するには、すべてのデータを指定したinput_dataフォルダーに保存する必要があります。入力データセットごとに、個別のファイルを用意する必要があります。シングルセルデータは、cluster_id上の細胞アノテーション(例えば、以前の細胞タイプのアノテーションステップから得られる)とsample_id列(分析すべき各サンプルを一意に識別する)を含む.h5adとして指定する必要があります。他のすべての入力データセットは、sample_idを指定する1つの列(単一セルデータの対応する列と一致する)と、他のすべての列のMOFA分析で使用される特徴を含む「.csv」形式で指定する必要があります。 この図の拡大版を表示するには、ここをクリックしてください。
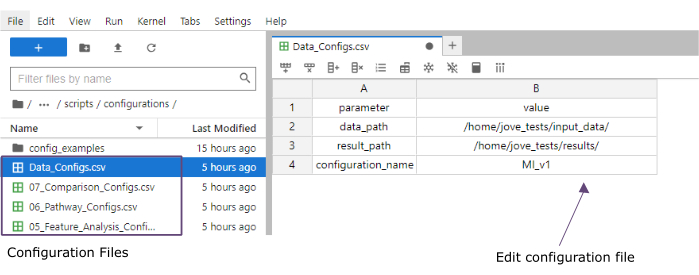
図4:Jupyter-lab設定ファイル。 ワークフローの実行中、パラメータの変更(フィルタリングオプションの調整など)は、「.csv」設定ファイルを介して指定されます。クローニングされたリポジトリ内には、各ステップのデフォルトの設定ファイルが含まれています。これらは、スプレッドシートと同様に、jupyter-lab コンソールで直接編集できます。 この図の拡大版を表示するには、ここをクリックしてください。
図5:Jupyter-notebooksスクリプト。 完全なワークフローは、対応する設定ファイルが変更された後に順番に実行される一連の Jupyter Notebook で構成されています。左側のJupyterノートブックをダブルクリックすると、対応するファイルが右側で開きます。ファイルの完全な実行は、上部で強調表示されているボタンから開始できます。 この図の拡大版を表示するには、ここをクリックしてください。
3. データの前処理と調和
- 前処理 - scデータを擬似バルクに変換します。
注:この手順は、分析でシングルセルデータを使用する場合にのみ実行する必要があります。- 左側のナビゲーション メニューを使用して、 構成をダブルクリックして構成フォルダーに移動します。ダブルクリックして 01_Preprocessing_SC_Data.csv ファイルを開きます。ファイルに自動的に入力された値を確認し、必要に応じて、[ data_name ] 列の値を、解析に使用される input_data フォルダー内の単一セル データセットのファイル名に対応するように調整します。
注 : デフォルトでは、入力データ フォルダ内のすべての .h5ad ファイルの名前が初期化スクリプトの設定ファイルに追加されます。一部のデータセットを分析に使用しない場合は、ここで削除できます。 - 上部のメニューで[ ファイル]>[CSVファイルを保存 ]をクリックして、変更を保存します。
- 左側のナビゲーションメニューを使用して、scriptsをクリックしてscriptsフォルダに移動します。ノートブック 01_Prepare_Pseudobulk.ipynb をダブルクリックして開きます。スクリプトを実行するには、上部にある[カーネルを再起動してすべてのセルを実行]ボタンをクリックし、ポップアップで[再起動]をクリックします。
- 左側のナビゲーション メニューを使用して figures フォルダーに移動するには、最初に figures をダブルクリックしてから 01_figuresをダブルクリックします。新しく生成されたプロット FIG01_Amount_of_Cells_overview をダブルクリックして開きます。
注: ノートブックの実行には数分かかる場合があります。 ノートブックが正常に実行されると、ポップアップが表示され、ファイル FIG01_Amount_of_Cells_Overview ノートブックによって更新されるか、新しく生成されます。 [最終変更日 ] 列は、ファイルが新しいファイルか古いファイルかを評価するために、ファイルが生成された日時を示すことができます。 - プロットを調査して、サンプルあたりの細胞数が非常に少ない細胞型クラスターを特定します。これらのcluster_idsの名前を書き留めて、後続の手順で除外します(図6)。
- 左側のナビゲーションメニューを使用して、をクリックして構成フォルダーに戻ります...次に、[ 構成]をダブルクリックします。ファイル 02_Preprocessing_Configs_SC.csv をダブルクリックして開きます。
- configuration_name列とdata_name列の値を確認し、必要に応じて調整します。
注 : 初期化スクリプト内では、これらの値には、入力データ フォルダ内のすべての .h5ad ファイルの名前と、以前に Data_Configs.csv ファイル内で設定された configuration_name 値が事前に入力されています。ファイルを分析から除外する必要がある場合、またはファイル名の別の拡張子を使用する必要がある場合は、ここで調整できます。 - cell_type_exclusion列の値を調整し、前の手順で除外するように特定されたすべてのcluster_idを「,」で区切って追加します。
- 上部のナビゲーションバーで[ ファイル]>[CSVファイルを保存 ]をクリックして、変更を保存します。
- 左側のナビゲーション メニューを使用して、 構成をダブルクリックして構成フォルダーに移動します。ダブルクリックして 01_Preprocessing_SC_Data.csv ファイルを開きます。ファイルに自動的に入力された値を確認し、必要に応じて、[ data_name ] 列の値を、解析に使用される input_data フォルダー内の単一セル データセットのファイル名に対応するように調整します。
- 前処理 - 他のオミクスデータソースを調和させ、統合します。
- ファイル 02_Preprocessing_Configs.csv をダブルクリックして開き、 data_input フォルダーに含まれて保存される各データセットの前処理構成を調整します (データセットごとに 1 行)。
- configuration_name列とdata_name列の値を確認し、必要に応じて調整します。
- 列内の他のパラメータは、適用する前処理ステップに応じて、それに応じて調整します。
注: デフォルト値は、input_dataset フォルダ内にあるデータセットごとに追加されますが、データの 1 つのデータ型に固有ではありません。そのため、調整が必要になります。パラメータの詳細なドキュメントは、 Documentation_Config_Parameter.doc ファイルに記載されています。 - [ ファイル] > [CSV ファイルの保存] をクリックして、変更を保存します。
- 左側のナビゲーションメニューを使用して、scriptsをクリックしてscriptsフォルダに移動します。ノートブック 02_Integrate_and_Normalized_Data_Sources.ipynb をダブルクリックして開きます。スクリプトを実行するには、上部にある[カーネルを再起動してすべてのセルを実行]ボタンをクリックし、ポップアップで[再起動]をクリックします。
- 左側のナビゲーション メニューを使用して、生成された 02_results フォルダーに移動するには、 フォルダー 記号をクリックし、 結果 と 02_resultsをダブルクリックします。結合された前処理されたデータ入力ファイルを含むファイル 02_Combined_data_'configuration_name'_Integrated.csv が含まれているかどうかを確認します。
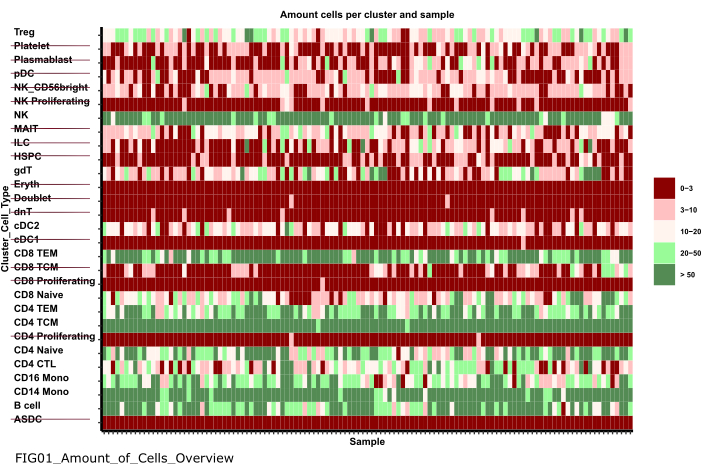
図6:データの前処理と調和 '01_Prepare_Pseudobulk' ステップの出力の 1 つは、プロット 'Fig01_Amount_of_Cells_Overview' です。ここでは、各cluster_id(y軸は前の細胞型アノテーションステップの細胞タイプを示す)について、サンプルあたりの細胞数('sample_id')が与えられます。提示された結果の中で、サンプルあたりの細胞量が少ない細胞タイプは、その後の分析から除外されます(取り消し線で示されています)。 この図の拡大版を表示するには、ここをクリックしてください。
4. MOFAの実行
- Jupyter-Lab では、左側のナビゲーション メニューを使用して、 フォルダー 記号をクリックしてから mofa_workflow をダブルクリックし、 次にスクリプト と 構成をダブルクリックして、構成フォルダーに移動します。ファイル 03_MOFA_Configs.csv をダブルクリックして開きます。
- configuration_name列とmofa_result_name列のエントリを確認し、別の名前を使用する必要がある場合はエントリを調整します。
注: mofa_result_name は、MOFAに基づいて生成されたすべての結果ファイルにファイル拡張子として追加されます。これは、同じ入力データで異なるMOFAセットアップが実行される可能性があるため、 configuration_name 値とは異なる場合があります(このプロトコルは MI_v1_MOFAを使用します)。 - MOFAモデルで推定する係数の量(amount_of_factors 列)を入力し、ファイル内の値を調整して、重み付けとスケーリングを適用するかどうか(weighting_of_views 列と scale_views 列)を定義します。
- [ ファイル] > [CSV ファイルの保存] をクリックして、変更を保存します。
- 左側のナビゲーションメニューを使用して、「scripts」をクリックしてscriptsフォルダに移動します。ノートブック 03_Run_MOFA.ipynb をダブルクリックして開きます。スクリプトを実行するには、上部にある[カーネルを再起動してすべてのセルを実行]ボタンをクリックし、ポップアップで[再起動]をクリックします。
- 図をダブルクリックしてから03_figuresをクリックして、03_figuresフォルダに移動します。生成されたプロットFIG03_Overview_Variance_Decomposition_を開きmofa_result_nameモデル結果を調査します(図7A)。
- 左側のナビゲーション メニューを使用して、生成された 03_results フォルダーに移動するには、 フォルダー 記号をクリックし、 results と 03_resultsをダブルクリックします。サンプル係数値ファイル 03_Factor_Data_'mofa_result_name'.csv と特徴因子重みファイル 03_Weight_Data_'mofa_result_name'.csv が含まれているか確認してください。
5. ダウンストリーム解析
- 因子の解釈。
- 左側のナビゲーションメニューを使用して、フォルダ記号をクリックし、次にinput_dataをダブルクリックしてinput_dataフォルダに移動します。
- 生成された因子に関連して分析されるサンプルのすべてのメタデータ(共変量)を含む.csvファイル(Prepared_Sample_Meta_Data.csv)を準備します。ドラッグ&ドロップでinput_dataフォルダにファイルをコピーし、input_dataフォルダのフォルダ概要にファイルをドロップします。
注: 以前に使用したデータと照合するための sample_id 列と、分析する必要がある各機能の追加の列が含まれている必要があります。 - Jupyter-Lab では、左側のナビゲーション メニューを使用して、 フォルダー 記号をクリックし、 次に [mofa_workflow] をダブルクリックし、 次に [スクリプト と 構成] をクリックして、構成フォルダーに戻ります。ファイル 04_Factor_Analysis.csv をダブルクリックして開きます。
- configuration_nameとmofa_result_nameのエントリに、スクリプトで分析される構成とMOFAの結果の名前が含まれていることを確認し、必要に応じて調整します。
- [numeric_covariates] 列に、MOFA 係数に関連して調査される Prepared_Sample_Meta_Data.csv ファイル内のすべての数値列の名前をコンマで区切って追加します (このプロトコルでは CRP,CK を使用します)。
- categorical_covariates' 列に、MOFA 要因に関連して調査される Prepared_Sample_Meta_Data.csv ファイル内のすべてのカテゴリ列の名前をコンマで区切って追加します (このプロトコルでは測定を使用します)。
- [ ファイル]>[CSVファイルを保存]をクリックして変更を保存します。
- 左側のナビゲーション メニューを使用して、 スクリプトをクリックして 'scripts' フォルダーに移動します。ノートブック 04_Downstream_Factor_Analysis.ipynb をダブルクリックして開きます。上部にある [カーネルを再起動してすべてのセルを実行する ]ボタンをクリックし、ポップアップで[再起動]をクリックして、スクリプトを実行します。
- 左側のナビゲーションメニューを使用して、図をダブルクリックしてから04_figuresし、04_figuresフォルダに移動します。生成されたプロットをダブルクリックして開き、興味深いパターンと関連性の要因を調査します。FIG04_Factor_Association_with_numeric_features_
'mofa_result_name.pdf (図 7B)。FIG04_Factor_Association_
with_categorical_features_mofa_result_name.pdf (図 7C)。FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf (図 8A)。
- 特徴解析
- 左側のナビゲーションメニューを使用して、... 次に、[ 構成]をダブルクリックします。ファイル 05_Feature_Analysis_Configs.csv をダブルクリックして開きます。
- configuration_name列とmofa_result_name列のエントリが、ダウンストリーム解析に使用されるコンフィギュレーションと生成されたMOFA結果の名前に対応していることを確認し、必要に応じて調整します。
- [係数] 列に、次のスクリプト内で上位の特徴がプロットされる係数を追加します。
- 列 faceting_variableで、プロット内のサンプルをグループ化するために使用される Prepared_Sample_Meta_Data.csvカテゴリ列の列名を追加します(このプロトコルは 測定を使用します)
- [ ファイル]>[CSVファイルを保存]をクリックして変更を保存します。
- 左側のナビゲーション メニューを使用して、スクリプト フォルダーに移動し 、 スクリプトをクリックします。ノートブック 05_Downstream_Investigate_Features_Heatmap.ipynb をダブルクリックして開きます。スクリプトを実行するには、上部にある [カーネルを再起動してすべてのセルを実行 ]ボタンをクリックし、ポップアップで [再起動 ]をクリックします。
- 左側のナビゲーション メニューを使用して 05_figures フォルダーに移動するには、最初に figures をダブルクリックしてから 05_figures をダブルクリックします。'mofa_result_name' FIG05_Heatmap_Feature_Overview__生成されたプロットを開いて調査します。pdf ファイルをダブルクリックして貼り付けます (図 8B)。
注: プロットに表示されるフィーチャの量によっては、05_Feature_Analysis_Configs.csv内のパラメータ plot_width と plot_height を調整し、スクリプトを再実行して、すべてがプロットに収まるようにする必要がある場合があります。
- パスウェイ解析
- 左側のナビゲーションメニューを使用して、フォルダ記号をクリックし、次にinput_dataをダブルクリックしてinput_dataフォルダに移動します。
- エンリッチメントについてテストするパスウェイのリストを含む .csv ファイル (Prepared_Pathway_Data.csv) を準備します。ドラッグ&ドロップでinput_dataフォルダにファイルをコピーし、input_dataフォルダのフォルダ概要にファイルをドロップします。
注: ID (パスウェイの一意の識別子)、 gene (パスウェイに属する遺伝子名(SYMBOL)によって与えられる遺伝子、遺伝子ごとに1行)、 pathway_name (パスウェイの名前/テキストによる説明)の3つの列を含める必要があります。 - Jupyter-Lab セッションでは、左側のナビゲーション メニューを使用して、...次に、[ 構成]をダブルクリックします。ファイル 06_Pathway_Configs.csv をダブルクリックして開きます。
- mofa_result_name列のエントリを確認し、パスウェイエンリッチメントの計算に使用される生成されたMOFA結果の名前に対応していることを確認します。
- types 列のエントリを確認し、Prepared_Pathway_Data.csv ファイルの gene 列に一致する特徴を含まない types 列内のエントリを削除します。
注: デフォルトでは、MOFA モデル内で使用されているすべての異なるビューは、ワークフローの実行中にこのファイルに追加されます。少なくとも 1 つの経路に一致するフィーチャが含まれていないビューがある場合は、それらを削除する必要があります。そうしないと、実行は失敗します。たとえば、パスウェイファイルには遺伝子のパスウェイアノテーションのみが含まれていますが、タンパク質名を含むビューがあります。 - [ ファイル]>[CSVファイルを保存]をクリックして変更を保存します。
- ナビゲーションメニュー を使用して、 scriptsフォルダに移動するには、 scriptsをクリックします。ノートブック 06_Downstream_Pathways.ipynb をダブルクリックして開きます。上部にある [カーネルを再起動してすべてのセルを実行する ]ボタンをクリックし、ポップアップで [再起動 ]をクリックして、スクリプトを実行します。
- 左側のナビゲーションメニューを使用して 06_figures フォルダに移動するには、最初に 図 をダブルクリックしてから 06_figuresをダブルクリックします。生成されたプロット FIG06_Pathways_and_Genes_ 'mofa_result_name をダブルクリックして開き、視覚化された経路を調査します (図 8C)。
注:視覚化された経路の選択方法は、設定ファイルを使用して構成できます。詳細については、パラメーターのドキュメントを参照してください。 - 左側のナビゲーション メニューを使用して、生成された 06_results フォルダーに移動するには、 フォルダー 記号をクリックし、 結果 と 06_resultsをダブルクリックします。エンリッチされたパス ウェイ 06_Pathway_enrichment__'mofa_result_name' を含むファイルが含まれているかどうかを確認します。
6. 異なる構成とバージョンの比較(補足図1、補足図2、補足図3、補足図4)
- ワークフロー全体で異なるパラメーター/構成を使用した場合の影響を比較するには、セクション 3 から 5 を再実行し、構成ファイル内のパラメーターを変更し、異なる configuration_name 識別子と mofa_result_name 識別子を使用します。
注:新しい結果は、異なる実行の比較に使用されるために、これらの名前で保存されます。 - Jupyter-Lab で、左側のナビゲーション メニューを使用して configurations フォルダーに移動します。ファイル 07_Comparison_Configs.csv をダブルクリックして開きます。
- [mofa_result_name] 列に、比較する以前のすべての MOFA 実行の名前を追加します (名前/構成ごとに 1 行 (例: MI_v1_MOFA、MI_v2_MOFA)。
- 「compare_factors」列に、モデル間で比較する因子を追加します。デフォルトでは、Factor1,Factor2,Factor3 です。(補足図2A)。
注:このスクリプトでは、さまざまなモデルの特徴値と因子値を関連付けることによって比較されます。これは、同じサンプル( sample_idで示される)と同じフィーチャセットに基づくモデルでのみ機能します。サンプルまたは特徴が比較バージョン間で一致しない場合、それらは比較から除外されます。 - [ ファイル]>[CSVファイルを保存]をクリックして変更を保存します。
- 左側のメニューを使用して、スクリプトをクリックしてスクリプトフォルダに移動します。ノートブック 07_Compare_Models.ipynb をダブルクリックして開きます。スクリプトを実行するには、[カーネルを再起動してすべてのセルを実行]ボタンをクリックし、ポップアップで[再起動]をクリックします。
- 左側のメニューを使用して、最初に図をダブルクリックしてから06_figuresをクリックして06_figuresフォルダーに移動します。生成されたプロットを開くには、ファイルをダブルクリックして、異なるバージョンの類似性を分析します。
FIG07_Variance_Model_Comparison.pdf(補足図2B)
FIG07_Factor_Correlations.pdf
(補足図2C)
FIG07_Feature_Correlations.pdf
(補足図3C)
7. ワークフローの拡張:他のパラメーターと設定の追加
注: 設定ファイルで現在設定可能なパラメータの他に、コードや他のパラメータに他の調整が含まれている場合があります。例えば、MOFAモデル自体は、コード内で直接変更することも、設定ファイルを通じて調整することもできる他のいくつかの訓練パラメータ17 を提供する。プロトコルの次のセクションでは、追加のMOFAモデルのトレーニングパラメータに対してこれを行う方法の例を概説します。この部分では、Rプログラミングの知識が必要です。
- Jupyter-Lab で、左側のナビゲーション メニューを使用して scripts フォルダーに移動します。ノートブック 03_Run_MOFA.ipynb をダブルクリックして開きます。
- 左側の [目次 ]タブをクリックし、[ 4.3 MOFAトレーニングオプションの設定 ]サブセクションに移動し、クリックしてモデルトレーニングを実行します。下にスクロールして、ノートブック内の構成可能なパラメーターのMOFAモデルの印刷出力を確認します。
- 見出しの下のコードの R for ループ内では、MOFA データ、モデル、およびトレーニング オプションがすべて設定されます。 model_opts$num_factors = mofa_configs$amount_of_factors[i] の行の下に、次のコードを含む別の行を追加します
model_opts$likelihoods['data_type'] = 'ポアソン'.
注: これにより、すべての MOFA 実行の data_type という名前で指定されたビューの入力としてモデルが受け取る分布が変更されます。データ型にポアソンを指定すると、このデータ型の特徴が整数 (RNA-seq からの読み取りカウントなど) の場合にのみモデルが実行されます。MOFAデータ、トレーニング、およびモデルオプションの詳細については、MOFAのチュートリアルとドキュメント17も参照してください。 - ノートブックの変更を保存するには、上部にある [保存 ] ボタンをクリックします。
- .csv設定ファイルを介して新しいパラメータを引き渡すには、左側のナビゲーションを使用して、configurationsをダブルクリックしてconfigurationsフォルダに移動し、ダブルクリックしてファイル03_MOFA_Configs.csvを開きます。
- パラメータ名を指定する新しい列( 例:number_iterations )を追加し、 値(例:1000)を入力します。[ ファイル]>[CSVファイルの保存]をクリックして変更を保存します。
- ナビゲーションメニューを使用して、 scriptsをクリックしてscriptsフォルダをナビゲートします。ノートブック 03_Run_Mofa.ipynb をダブルクリックして開きます。左側の [目次 ]タブをクリックし、[ 4.3 MOFAトレーニングオプションの設定 ]サブセクションに移動し、クリックしてモデルトレーニングを実行します。
- 行 train_opts$maxiter = 50000 を train_opts$maxiter = mofa_configs$column_name[i] に置き換えます (追加された列の名前が number_of_iterations の場合は train_opts$maxiter = mofa_configs$number_of_iterations[i]]) です。
注: このノートブックで03_MOFA_Configs.csv設定ファイルは、ノートブックの先頭 (サブセクション: 前提条件 設定とパラメータ) でセッションの data.frame として読み取られmofa_configしたがって、このコード行では、このオブジェクトと対応する新しく生成された列が参照されます。複数の構成を同時に実行できるため、i は data.frame の行を識別し、モデル推定は .csv ファイル内のすべての異なる行に対して for ループで実行されます。ノートブックの冒頭にある「前提条件、構成とパラメータ」のセクションにある設定ファイルを読み込む原則は、すべてのノートブックで同じであり、このようにさらに変更を加えることができます。 - [ 保存 ] ボタンをクリックして、ノートブックの変更を保存します。
結果
ワークフローが正常に実行されると、 図 2 に示すように、いくつかのテーブルと図が生成されます。図は /figures フォルダ (図 6、 図 7、 図 8、 補足図 1、 補足図 2、 補足図 3、 補足図 4) に配置され、テーブルは指...
ディスカッション
概説されたプロトコルでは、複雑なマルチオミクスデータセットを迅速に探索するために使用できる、モジュール式で拡張可能なJupyter-notebookベースのワークフローが提示されます。ワークフローの主要な部分は、前処理とデータ調和の部分(データのフィルタリングと正規化のためのさまざまな標準ステップを提供)、MOFA9 モデルの推定、およびいく...
開示事項
著者は、利益相反を宣言しません。
謝辞
C.L.は、共同研究学校「Munich School for Data Science - MUDS」の下でヘルムホルツ協会の支援を受けています。
資料
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
参考文献
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31 (2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535 (2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125 (2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22 (2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806 (2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124 (2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124 (2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88 (2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15 (2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121 (2022).
- . Get Started - JupyterLab 4.1.0a4 documentation Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024)
- . MOFA2: training a model in R Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020)
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272 (2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68 (2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111 (2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA Available from: https://biofam.github.io/MOFA2/ (2024)
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved