このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
クマムシHypsibius exemplarisにおけるqRT-PCRによる相対転写レベルの比較のための単一動物、単一チューブRNA抽出
要約
この研究は、クマムシ Hypsibius exemplaris の遺伝子発現を解析するための迅速なRNA抽出および転写産物レベルの比較方法を示しています。物理的溶解を用いたこのハイスループット法では、出発物質として単一のクマムシが必要であり、定量的逆転写ポリメラーゼ連鎖反応(qRT-PCR)用のcDNAの堅牢な産生が得られます。
要約
クマムシ Hypsibius exemplaris は、極端な環境を生き抜く能力で有名な新興のモデル生物です。このような極限耐性の分子メカニズムと遺伝的基盤を探るために、多くの研究は、大規模なコホートから個々の動物までの集団で実施できるRNAシーケンシング(RNA-seq)に依存しています。その後、逆転写ポリメラーゼ連鎖反応(RT-PCR)とRNA干渉(RNAi)を使用して、それぞれRNA-seq所見を確認し、候補遺伝子の遺伝的要件を評価します。このような研究では、定量的RT-PCR(qRT-PCR)によるRNA抽出と相対転写産物レベルの測定に、効率的で正確、かつ手頃な価格の方法が必要です。この研究は、個々のクマムシからRNAを確実に単離するだけでなく、各抽出に必要な時間とコストを削減する効率的なシングルクマムシ、シングルチューブRNA抽出法(STST)を示しています。このRNA抽出法では、定量的PCR(qRT-PCR)による複数の転写産物の増幅および検出に使用できる量のcDNAが得られます。この方法は、熱ショックタンパク質70 β2(HSP70 β2)と熱ショックタンパク質90α(HSP90α)の2つの熱ショック制御タンパク質をコードする遺伝子の発現の動的変化を解析することで検証されており、熱曝露された個人におけるそれらの相対的な発現レベルをqRT-PCRを用いて評価することが可能となります。STSTは、既存のバルクおよび単一クマムシRNA抽出法を効果的に補完し、qRT-PCRによる個々のクマムシの転写レベルの迅速かつ手頃な価格の検査を可能にします。
概要
クマムシは、他のほとんどの生命体にとって致命的な極限状態を生き抜く能力で有名な小さな多細胞動物です1。例えば、これらの動物は、人間に致命的な電離放射線のほぼ1000倍の線量を生き残ることができます2,3,4,5,6,7,8,9,10、ほぼ完全な乾燥11,12,13,14,15、添加物がない場合に凍結します凍結保護剤16,17,18、そして乾燥状態では、宇宙の真空19,20でさえも。極限環境での生存のための彼らのユニークな能力のために、これらの動物は、複雑な多細胞生物1,21,22,23の極限耐性を理解するための基礎モデルとなっています。
トランスジェネシスや生殖細胞系遺伝子改変を含む、これらの注目すべき動物の安定した遺伝子操作は、最近までとらえどころのないままでした24,25。そのため、極限耐性の分子メカニズムを明らかにするためのほとんどの実験は、RNAシーケンシングによる転写プロファイリングを通じて行われます。放射線8,9,26,27,28、熱ストレス29、凍結ストレス12、乾燥27,30,31,32,33など、さまざまな極限条件下でのクマムシに関する多くの貴重で有益なRNAシーケンシングデータセットが存在します.これらの研究の一部は、バルクRNAの抽出および精製方法を利用して、極限耐性の分子的理解を明らかにしています。しかし、多くの動物からRNA転写産物をバルク抽出すると、個体間の遺伝子発現のばらつきの解析が妨げられ、より精緻なデータセットの潜在的な豊富さが失われます。重要なことに、これらの研究は、環境ストレス要因を生き残る動物と生き残らない動物の両方を含む動物の不均一な集団を分析することがよくあります。そのため、これらの研究は、複数の、潜在的に劇的に異なる応答状態からの発現データを平均化することによって交絡します。この問題に対処するために、Arakawa et al., 201634は、RNA抽出キットとそれに続く、単一の34,35,36または複数の30,37,38匹の動物をインプットとして用いた線形PCR増幅ステップを適用する、洗練された低インプットRNA-seqパイプラインを開発した。これらの研究は、クマムシの極限耐性22を理解するための基礎となっています。興味深いことに、このプロトコルは、出発物質24として7匹の動物を用いたqRT−PCRにも適用されている。
ほとんどのモデル生物では、RNA-seqによって潜在的な標的を特定した後、qRT-PCRを実施して、RNA-seqによって同定された転写変化を確認し、候補遺伝子の発現時間経過を高分解能で評価します。同定された遺伝子の機能を調べるために、このような研究の後には、RNAiを介した分子標的のノックダウン39,40と極限耐性能の分析12,41がしばしば行われる。各RNAiノックダウンの有効性は、通常、転写産物存在量の減少を直接モニタリングすることにより、qRT-PCRによって確認されます。しかし、RNAiはクマムシでは労働集約的なプロセスであり、各dsRNAは個々の39,40の手動マイクロインジェクションによって送達されなければならない。この戦略はスループットが低いため、単一動物からのqRT-PCRに適した迅速で低コストのRNA抽出法は、クマムシの研究に非常に価値があります。単一のクマムシからRNAを抽出するための以前の方法が開発されてきたが、これらのプロトコルは、それらの抽出をqRT-PCRと組み合わせておらず、代わりに光学密度ベースの方法12,40,41に依存している。これらの課題に動機付けられて、私たちは、単一のH. exemplarisからのqRT-PCRに使用できるRNAの量と質を確実に得るプロトコルの開発を目指しました。
線虫Caenorhabditis elegans42用に開発された単一動物RNA抽出プロトコルから適応されたSTSTは、H. exemplarisに最適化されています。抽出方法は、キューティクルを物理的に破壊する6つの急速凍結融解ステップで構成され、RNA抽出とその後のcDNA合成を可能にします。STST法は、Boothby, 201843で説明されているように、バルクRNA抽出法と比較して抽出時間を24倍以上短縮し、Arakawa et al., 201634で説明されているように、単一のクマムシRNA抽出キットと比較して30%短縮します。さらに、サンプルと実験者の相互作用の数は、RNA抽出キットの調製物と比較して5回からわずか1回に減少するため、外因性リボヌクレアーゼによる汚染のリスクが軽減されます。高発現遺伝子を解析する場合、STST法では、クマムシ1回あたり25回の定量的RT-PCR反応に十分なcDNAが生成され、1回の反応で合計25μLのcDNA容量のうち1μLしか必要としません。ただし、存在量が少ない転写産物については、テンプレート濃度を経験的に決定する必要があります。
STST法による遺伝子発現の動的変化の解析は、35°Cで20分間の短時間熱ショックを受けた場合、熱ショックタンパク質-90α(HSP90α)と熱ショックタンパク質70β2(HSP70β2)をコードする遺伝子の発現差を調べることで評価した。ほとんどの真核生物におけるHSP70β2とHSP90αは、短期間の熱ショック曝露(20分)後に急速にアップレギュレーションされる42。 H. exemplaris での解析により、単一の熱処理されたクマムシから抽出されたHSP70β2およびHSP90αをコードするRNAは、短期間の熱曝露後に統計的に有意な発現増加を示したことが明らかになった。これらの知見は、STSTプロトコルを用いて、個々の動物における遺伝子発現の経時的な動的変化を解析できることを示しています。
STST抽出法は、RNA-seqなどの既存の実験法を補完するもので、迅速かつ安価なRNA抽出とその後のqRT-PCRによる転写産物レベルの比較を容易にする必要があります。この方法は、手動で注入した個人のRNAiの効率と浸透度を、光学密度のみの場合よりも定量的に評価するのにも役立ちます。最後に、それらの類似したクチクラ構造および物理的特性のために、この方法は他のクマムシ種における遺伝子発現の解析にも有効である可能性が高い44。
プロトコル
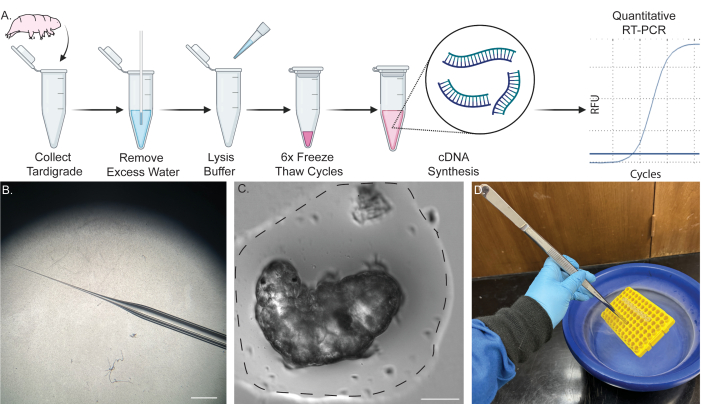
図1:単一のクマムシからのRNA抽出のためのシングルチューブパイプライン。 (A)6回の凍結融解サイクルとその後のcDNA合成を含む、単一のクマムシからのRNA抽出のプロトコルを示すスキーム。その後、サンプルをRT-PCRおよびqRT-PCRに使用できます。(B)水分の除去に用いるマイクロピペットテーパーのイメージ。スケールバー:2 mm.(C)少量の水(点線)中のクマムシの明るい視野画像。ほとんどの水は、示されている範囲で除去する必要があり、抽出を成功させ、溶解バッファーの希釈を防ぎます。スケールバー:50μm。(D)長い鉗子を使用してサンプルを液体窒素に浸し、サンプルを安全に急速に凍結融解する様子を示す画像。一部のコンテンツはBioRenderで作成されました。(2022) BioRender.com/d93s511 この図の拡大版を表示するには、ここをクリックしてください。
注:図1Aは、手順の概略図を示しています。クマムシおよび藻類の詳細な培養手順については、以前に公開されたレポート45,46,47を参照してください。
1.湧き水の殺菌
- 2ガロンの水差しから5Lの湧き水を注ぎます(詳細については 材料の表 を参照)2Lのオートクレーブに安全なガラス瓶に入れます。
- オートクレーブセーフボトルにキャップをかぶせ、少量のオートクレーブテープで密封します。ボトルを締めないでください。キャップを上に置きます。
- 湧き水を乾燥ステップなしで湿式サイクルで50分間オートクレーブします。
- 水を室温(RT)に戻し、キャップをしっかりと密封してからRTで保管してください。
2.ガラスマイクロピペット引き(ピペット引き手付き)
- ガラス製マイクロピペット(外径:1mm、内径:0.58mm、長さ:10cm)をマイクロピペットプーラーに固定します。加熱フィラメントとの接触は、ピペットの形状が変わり、フィラメントが損傷するため、避けてください。
- 各フィラメントとピペットプーラーについて、ピペットの引き込みを経験的に決定します。ただし、最適化の出発点として、78°Cを使用し、182.2gの引張重量の1つの引張ステップを使用します。
- フィラメントを加熱し、重力に任せて、ガラスマイクロピペットを鋭利な先端を持つ2つのガラスマイクロピペットに分離します(図1B)。
- これらの引っ張ったガラスマイクロピペットは、ワックスまたは粘土を入れた閉じた100mmのペトリ皿に保管して所定の位置に保持し、鋭い先端が壊れるのを防ぎます。
3.ガラスマイクロピペットプーリング(ピペットプーラーなし)
- ブンゼンバーナーまたはその他の制御された火炎源を低い設定で点灯させます。
- 片方の端を両手に持ったガラス製のマイクロピペットを取ります。
- ガラスが溶け始めるまで、ガラスマイクロピペットの中央を炎の上に保持します。次に、両端をすばやく引き離します。これにより、非常に繊細でシャープな先端が2つ作成されます。
- 滅菌した細い鉗子で先端を軽く折ってください。
- これらの引っ張ったガラスマイクロピペットは、ワックスまたは粘土を入れた閉じた100mmのペトリ皿に保管して所定の位置に保持し、鋭い先端が壊れるのを防ぎます。
4. RNA抽出
- 0.5 Lの液体窒素をクライオセーフ容器に入れて入手します。
注意: 液体窒素は極低温であり、皮膚や目にさらされると火傷を負う可能性があります。取り扱いの際は、防護服、スプラッシュゴーグル、ニトリル手袋、クライオ手袋、白衣、つま先が閉じた靴を使用してください。液体を輸送する前に、容器が液体窒素に安全であることを確認してください。このステップにエタノールドライアイスバスを使用することも可能です。 - cDNA合成マスターミックスを作製します:ランダムヘキサマープライマー1 μL、DNase2 μL、5x RT Buffer4 μL、酵素ミックス1 μL、H2O1 μL、および10 mM dNTPs1 μLを含む10 μL溶液。この溶液を氷上に保存します。
- クマムシ溶解バッファー(5 mM Tris (pH = 8)、0.5% (v/v) Detergent 1、0.5% (v/v) Detergent 2、0.25 mM EDTA を滅菌ヌクレアーゼフリー水溶液で調製します。
注:この溶液はベンチトップに6か月間保存できます。ただし、無菌性を維持し、潜在的なRNAse汚染源を避けてください。 - 抽出に十分な溶解バッファー(2 μL/クマムシ)を分注します。
- RNAse阻害剤をクマムシ溶解緩衝液に添加し、最終濃度を4 U/μLにします。
- 溶液をボルテックスし、室温でベンチトップ遠心分離機で2000 x g の速度で5秒間スピンダウンしてから、溶液を氷上に保存します。
- 無菌フィルターチップ付きP1000ピペットを使用して、実験に必要な数のクマムシを培養から取り除き、無菌の35 mmペトリ皿に入れます。
注:この方法では、クマムシをいくつでも処理できます。通常、条件ごとに3つのクマムシが抽出のために処理されます。 - オートクレーブ滅菌湧き水1 mLと滅菌フィルターチップ付きP1000ピペットを使用して、クマムシを3回洗浄します。ゆっくりとピペッティングで上下させることで、藻類の汚染物質を取り除くことができます。
- 25倍から50倍の倍率で解剖顕微鏡を使用し、この洗浄した培養物から単一のクマムシを、滅菌フィルターチップ付きP10ピペットを使用して新しい滅菌35 mmシャーレに移します。
- 滅菌フィルターチップ付きP200ピペットを使用して、100 μLの滅菌ヌクレアーゼフリー水で1つのクマムシを洗浄します。
注:この洗浄ステップは、リボヌクレアーゼなどの汚染物質をさらに除去するために使用されます。 - 洗浄したクマムシを、滅菌フィルターチップ付きP10ピペットを使用して、1〜2 μLの滅菌ヌクレアーゼフリー水中の清潔な滅菌PCRチューブの底に移し、クマムシが先端の側面に付着しないように注意深く確認します。
- クマムシを解剖顕微鏡で25倍の倍率で視覚化します。
- 水分の除去を容易にするために、引っ張ったガラスマイクロピペットの先端をチューブの外側で軽く折ってください。ボアが水を引き上げるのに十分な大きさであることを確認してくださいが、クマムシは引き上げないでください。
- 引っ張ったガラスマイクロピペットの毛細管現象を使用して、動物が直径約2クマムシの長さの小さな水の泡に囲まれるまで水を取り除きます。
- 解剖スコープで水分除去プロセスを監視し、水位が適切で、クマムシが水分補給されたままであることを確認します。
注意: 図1C は、除去する水の量の例を示しています。これは重要なステップです。クマムシが乾燥するのを防ぐために、クマムシを小さな水の泡で囲みますが、溶解バッファーの希釈を防ぐために、できるだけ多くの余分な水を取り除く必要があります。例についてはamp残りの水位のファイル、 図1Cを参照してください。 - 水を取り除いた直後に、2 μLのクマムシ溶解バッファーをチューブの底に加え、短時間ボルテックスし、卓上遠心分離機でチューブをRTで5秒間、2000 x g で遠心分離します。
- クマムシを含むサンプルを直ちにPCRチューブラックに入れ、ラックにしっかりと保持されていることを確認してください。
- 一対の長い粗い鉗子を使用してラックをつかみ、サンプルが入ったラックを完全に凍結するまで液体窒素にゆっくりと浸します(図1D)。
- 液体窒素からラックを取り出し、すぐに氷の上に置きます。サンプルを解凍します(~45秒から1分かかります)。サンプルを氷から取り出し、目視で検査することにより、15秒ごとにサンプルを監視します。サンプルが目に見える形で透明になったら、次のステップに進みます。
- 手順 4.18 から 4.19 をさらに 5 回繰り返します。最大限の溶解と抽出には、合計6回の凍結融解サイクルが必要です(図2A、B)。
- 凍結融解が完了したら、サンプルを氷の上に置き、すぐに次のステップに進みます。この時点でサンプルを凍結して保存すると、cDNA調製に利用可能なRNAが減少するため、保管しないでください。
5. cDNA合成
- クマムシ溶解物を含むPCRチューブに2 μLのcDNA合成マスターミックスを加えます。チューブを短時間フリックし、卓上遠心分離機で室温2000 x g で5秒間回転させてから、氷上のサンプルを交換します。
- サンプルをサーモサイクラーに入れ、25°Cで10分間インキュベートしてプライマーをアニールし、55°Cで30分間インキュベートして逆転写を行い、最後に酵素を85°Cで5分間熱不活性化します。
- インキュベーション後、すぐにチューブを氷の上に置き、21 μLの滅菌ヌクレアーゼフリー水を加えてサンプルを総容量25 μLに希釈します。コピー数が少ない転写産物の場合は、経験的に決定されたようにこの希釈ステップを変更します。
6. qPCRの
- 大量のクマムシから調製した全RNAを用いてプライマーセットのアニーリング温度を決定する、例えば、Boothby, 201843で提示されたバルク抽出法。
- qRT-PCRを実行する前に、PCR温度グラジエントを実行して最適なアニーリング温度を決定します(このプロトコルで使用されるすべてのPCR設定については、 表1 および 表2を参照してください)。
- インジケーター染料スーパーミックスの1本のチューブを氷上で解凍し、光から隔離します。96ウェルqPCRプレートを氷上に配置し、5 μLのスーパーミックス、2 μLの水、各プライマー1 μL(10 μM)、および1 μLのcDNA産物を目的のウェル数に配置します。
- PCRプレートをプレートシールでシールし、プライマーセットに適したアニーリング温度でqRT-PCRを実行します(このホワイトペーパーで使用したすべてのqRT-PCR設定については、 表3を参照)。
7. 定量化と結果の解釈
- 結果を、課せられた条件で発現が一定であると予想される1つ以上の対照ハウスキーピング遺伝子と定量的に比較します。この研究では、アクチン遺伝子を使用しました。
- 各ウェルのCt値またはサイクル閾値を取得し、コントロールハウスキーピング遺伝子反応のCt 値と比較します。次の式を使用して、遺伝子発現の倍率変化を計算します。
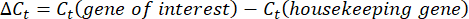
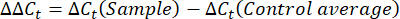
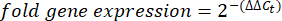
注:フォールド遺伝子発現は、各転写産物およびクマムシについて2-(ΔΔCt)48としてプロットされます。 - Ct値から転写産物数の大まかな推定値を取得するには、次の式を使用します。
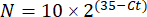
ここで、Nは転写産物の数であり、2は想定されるPCR効率またはPCR48のサイクル当たりの蛍光の倍増である。
結果
一本鎖RNA抽出法の開発と最適化
クマムシのRNA抽出に関するLy et al., 201542 のプロトコルを適応させると、STSTシステムは調製物の量と質を最大化するように最適化されています(図1A)。アクチン転写産物に対してRT-PCRを行い、エクソン1およびエクソン2にまたがる527 bp領域を増幅することにより転写産物の収量を定?...
ディスカッション
この研究は、単回クマムシqRT-PCRのためのRNA抽出のための効率的な方法を提示します。STST の手法を既存のクマムシ 1 種の RNA 抽出キットと直接比較すると、STST の RNA 抽出により >200 倍高い量のアクチン RNA 転写産物が得られ、サンプルあたり 1 ドル未満にコストが削減され、抽出に必要な時間が 30% 短縮されることが明らかになりました。STSTを関連する生物学的問題?...
開示事項
著者は、開示する利益相反を宣言しません。
謝辞
これらの研究活動を支援したNIH Ruth Kirschstein Fellowship #5F32AG081056-02、Molly J. Kirk博士を支援したErrett Fisher Post-Doctoral Fellowship、Chaoming Xuを支援したCrowe Family Fellowship、カリフォルニア大学サンタバーバラ校アカデミック上院助成金、NIH助成金 #R01GM143771 と #2R01HD081266 に感謝します。また、著者らは、カリフォルニア大学サンタバーバラ校とカリフォルニア大学学長室が支援するカリフォルニアナノシステム研究所内の生物学的ナノ構造研究所が使用されていることを認めています。
資料
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
参考文献
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890 (2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884 (2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333 (2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621 (2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330 (2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495 (2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245 (2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, &. #. 3. 2. 1. ;. The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983 (2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522 (2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120 (2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298 (2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405 (2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143 (2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266 (2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663 (2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168 (2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063 (2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24 (2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888 (2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508 (2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved