Method Article
多様な環境および基質における真菌ハイウェイカラムを用いた細菌-真菌相互作用の調査
要約
このプロトコルは、さまざまな環境基質から真菌ハイウェイを介して相互作用する細菌 - 真菌ペアを濃縮するために、真菌ハイウェイカラムを構築、滅菌、組み立て、利用、および再利用する方法についての詳細な指示を提供します。
要約
細菌と真菌の相互作用(BFI)は、微生物群集の構成、生物地球化学的機能、空間ダイナミクス、および微生物の分散の形成に不可欠な役割を果たします。糸状菌類または他の糸状微生物(卵菌類など)によって形成された菌糸体ネットワークは、細菌が不均一な環境全体を輸送するために利用できる「真菌ハイウェイ」として機能し、細菌の移動性を大幅に促進し、自分では到達が困難または不可能な領域へのアクセスを可能にします(たとえば、土壌内のエアポケットのため)。これらの真菌ハイウェイを研究するために、真菌ハイウェイのコラムを含むいくつかの装置と実験プロトコルが作成されています。当グループが設計した真菌ハイウェイカラムは、 in situ または in vitro の様々なアプリケーションに使用できるほか、環境や宿主に関連する様々なサンプルタイプにも使用できます。ここでは、これらのカラムを使用して実験を行う方法(デバイスの設計、印刷、滅菌、調製など)について説明します。これらのデバイスの使用から得られたデータを分析するためのオプションについてもここで説明し、真菌ハイウェイカラムを使用した実験に関連する潜在的な落とし穴に関するトラブルシューティングのアドバイスを提供します。これらのデバイスを使用して、真菌ハイウェイBFIの多様性、メカニズム、およびダイナミクスをより包括的に理解し、複雑な環境(土壌など)内および細菌と真菌が共存する多様な生息地全体の構造的および機能的ダイナミクスに関する貴重な洞察を得ることができます。
概要
細菌と真菌の相互作用(BFI)は、環境微生物叢の構造的、空間的、機能的特性を形成する上で非常に重要です。例えば、糸状菌または他の真菌様糸状微生物の成長および拡大は、細菌などの他の微生物の移動を促進するための「ハイウェイ」として機能することができる生物学的ネットワークを生成する。環境基質内の不均一性と一貫性のない飽和は、細菌の運動性を妨げる可能性があります。しかし、バクテリアはこれらの高速道路を使用して、環境の別の領域へのアクセスを容易にすることができます1,2。これらの相互作用は、微生物群集の空間動態を理解するために重要です。真菌ハイウェイを調べるためにいくつかの技術と方法が使用されてきましたが、それらは主に実験室ベースの調査に限定されています3,4。
1つのプレートベースの方法では、ペトリ皿の中央から寒天の大部分が除去され、2つの寒天島の間に隙間ができます。真菌菌糸はこのギャップを横断することができ、適合する細菌が1つの寒天島から他の5島に渡る手段を提供します。他の改変されたペトリ皿法には、土が蓋に置かれる逆プレートが含まれ、真菌菌糸が垂直に成長し、直接接触することなく培地にコロニーを形成し、細菌輸送の手段を提供する5,6。最近開発された増殖培地液滴ベースの方法は、特定の栄養プロファイル7に対する細菌の選択的な菌糸輸送を評価するために使用できます。バクテリアブリッジおよびトレイルデバイスは、バクテリアの動きに対する非生物的要因の影響を調査するためにも使用されています8。真菌ハイウェイの調査にはいくつかの方法と技術が利用されてきましたが、糞、土壌、根圏などの複雑な環境基質からの真菌ハイウェイの確立を促進しながら、無菌の微小環境を維持する標準化されたデバイスの必要性が残っています。
私たちのグループは、菌類が一方の端からもう一方の端に細菌を輸送できる菌類の高速道路の柱の3Dプリントバージョンを設計しました9。これらのデバイスは、砂時計形状と複雑な内部格子構造の柱自体、ねじ付きリング、2つのキャップ(大きなキャップと小さなキャップ)、および滅菌されたナイロンメッシュの4つの印刷コンポーネントから組み立てられます(図1)。組み立てられたカラムは、目的の環境基板に直接追加されます。次に、カラムは、微生物がカラムの底部にあり、メッシュを介して環境基質と接触している「ベイト」培地プラグとして知られる寒天増殖培地プラグにコロニーを形成することを可能にします。このナイロンメッシュのサイズは、バクテリアを輸送できる他の土壌居住者を除外しているため、柱内のバクテリアの動きが真菌の高速道路に制限されます。このベイトプラグがコロニー形成されると、糸状菌類は、土壌(または他の不飽和媒体)に似た不飽和システムを作成し、ベイト媒体からの潜在的な汚染を最小限に抑えるように設計されたカラムの中心内の内側の格子を通って伸びて成長することができます。その後、菌類はカラムの上部にあるターゲットミディアムプラグに向かって成長し、コロニーを形成します。カラムには、特定の真菌分離株を接種して細菌を輸送する能力をテストするか、または接種せずに放置して、基質からどの真菌が細菌を輸送できるかを特定することができます。標的培地に到達した生物は、さらに培養、単離、シーケンシング解析(純粋培養物から、またはアンプリコンまたはメタゲノムシーケンシングアプローチを使用した混合コミュニティから)を行うことができます。全体として、これらのカラムは、さまざまな基質中の真菌ハイウェイを調べるための標準化された、再現性のある、再利用可能で直感的な方法を提供します。これらの装置は、研究や教室での教育ツールとして利用することができますが、ここでは、過去に行われた実験に基づいて、それらを使用するための指導手順を提供します。この方法によりプロトコルの標準化が容易になりますが、デバイスの設計と構造は、他のアプリケーションや追加の基板に合わせて変更できます。
プロトコル
本試験で使用した試薬および機器の詳細は、 資料表に記載されています。
1. カラムの設計、材料、パラメータの変更
- 公開されているカラム設計9 をダウンロードしてそのまま使用するか、互換性のあるCAD(Computer Aided Design)ソフトウェアでカラム設計を変更します。
- 歯科用サージカルガイドレジンを入手するか、他の感光性透明レジンなどの代替3Dプリント材料を選択します。
- 選択した3Dプリンター、3Dプリンティング技術、または3Dプリンティング材料が以前に使用されていたものから変更された場合は、必要に応じてコラムの仕様を調整する9。
列の2. 3D印刷
- 0.05 mmのスライス厚さと0.8秒の露光時間を使用するように印刷パラメータを設定するか、選択したプリンタ、印刷材料、および印刷ソフトウェアに応じて印刷パラメータを調整します。
- 互換性のある3Dプリンタを使用して、真菌の高速道路の柱、ネジ付きリング、およびキャップを印刷します(図1)。

図1:菌類ハイウェイコラムのコンポーネント(A、B)スモールキャップ、ネジ付きリング、ラージキャップの上面図と側面図(左から右)。(C,D)スモールキャップの上面図と側面図。(E,F)ネジ付きリングの上面図と側面図。(G,H)大型キャップの上面図と側面図。(I)ナイロンメッシュフィルター(25μm)片をカラムの端に配置し、環境基板に挿入して、微小動物がカラムに侵入するのを防ぎます。(J)組み立てられていないカラム。(K)組み立てられたカラム:「ベイト」の端は基板に入り、「ターゲット」の端は覆われず、基板から出たままになります。この図の拡大版を表示するには、ここをクリックしてください。
3. 3Dプリントされたカラムコンポーネントのクリーニング
- プリントしたカラム、キャップ、ネジ付きリングを99%イソプロピルアルコールに室温で15分間浸し、1分ごとに手でバスを前後に動かして撹拌し、余分な樹脂を取り除きます。
- 成分を新鮮な99%イソプロピルアルコール浴に移します。5分間沈め、手順3.1と同じ方法で手で攪拌します。
- 部品を純水で満たされた超音波洗浄機に移し、部品を沈め、中速設定で2分間撹拌します。水を温めないでください。コンポーネントを取り外します。
- すべてのコンポーネントを少なくとも30分間風乾します。
- レジンの二次硬化を行うには、すべての3Dプリント部品を405nmの光に60°Cで30分間さらします。
4. カラムの滅菌
- 選択した材料がオートクレーブ可能な場合は、25 μmナイロンメッシュフィルターのカラム、ネジ付きリング、キャップ、シートを個別に、または大きなビーカー内で121°C、1気圧で20〜30分間オートクレーブします。
注:カラムのコンポーネントは、オートクレーブ後に形状や色が変わる場合がありますが、目的の材料特性は維持されます。オートクレーブ後のカラムコンポーネントの最終的なサイズ、形状、および色を 図 1 に示します。
5. カラムのメディアの準備
- 滅菌した寒天ベースの培地の90 mmシャーレを準備します:カルボキシメチルセルロースナトリウム培地(CMC)、麦芽エキス寒天培地(MEA)、ジャガイモデキストロース寒天(PDA)、またはReasoner's 2A寒天(R2A)9,10のいずれか。製造元の指示に従ってメディアを準備します。
- 121°C、1気圧で21分間またはメーカー推奨時間で培地をオートクレーブ滅菌し、寒天がペトリ皿の側面の上部に近づくまで90 mmのペトリ皿に注ぎます(図2)。この手順を生物学的安全キャビネットで実行して、無菌性を高めます。
- 寒天を固化させて乾かします メーカーの指示に従って。

図2:菌類高速道路の支柱の組み立てプロセス。 カラム自体の開放端を使用して、プラグを切り取って挿入し、研究者がカラムをメディアから取り外すときにカラムをひねって、プラグがカラムの端内に留まるようにします。その端は小さなキャップピースで覆われています。次に、同じ方法でメディアプラグをカラムのもう一方の端に追加します。次に、メッシュピースをこの端に置き、ネジ付きリングで固定します。次に、大きなキャップがねじ付きリングの上のこの「餌」の端に使用されます。メッシュのある面は、環境基板に配置されます。 この図の拡大版を表示するには、ここをクリックしてください。
6.真菌高速道路の支柱の準備
注:このステップは、カラム成分と培地の無菌性を維持するために、生物学的安全キャビネットで実施します。 図2 は、真菌ハイウェイカラムの組み立てプロセスを示しています。
- カラムの端自体(キャップなし)を使用して、カラムの一方の端にしっかりと収まるメディアプラグを取り出します。カラムをメディアから持ち上げるときにねじる動作を使用して、寒天がカラムの端内に留まるようにします。または、カラムの端をテンプレートとして使用し、培地を切り出し、ピペットチップを使用してカラムに移します。
- 最初のプラグをカラムの端に追加した後、この端に小さなキャップを追加して、ターゲット培地の無菌微小環境を維持します。
- カラムを裏返し、カラムのもう一方の端で手順6.1を繰り返します。
- 滅菌したハサミを使用して、オートクレーブ処理したナイロンメッシュ(孔径25μm)の直径~2cmの円形ピースを切断します(図1)。柱の露出した端にメッシュを置きます。ねじ山のリングをこのベイトメディアの端にねじり、メッシュをねじ山内に固定します。
- 大きなキャップをカラムの底部に置き、メッシュとネジ付きリングのもう一方の端の上に置き、カラムを保管または輸送するときはこのキャップを着用したままにします。
7. 基質またはベイト培地に目的の真菌を事前に接種する
注: この手順はオプションです。
- 目的の真菌(例えば、真菌ハイウェイを作成することが知られている真菌)を所望の基質上に接種するには、まず、固形の真菌増殖培地(MEA、PDAなど、ステップ5で説明したように調製)で真菌を増殖させ、目に見える真菌の増殖を伴う寒天の小さな(~1cm)部分を基質に移します。
注:以前の実験9 では、コロニー形成済みの糞基質を Coprinopsis cinerea と共に10日間使用してから、コロニー形成済みの基質にカラムを添加しました。 - 基質に事前にコロニーを形成する代わりに、少量の真菌菌糸体を使用して、餌菌をペトリ皿に直接添加するか、カラム内の餌培地の底に直接加えます(例:滅菌ループのスワイプから)。
- ペトリ皿については、ペトリ皿の大部分(覆われたプレートの約50%〜75%)を覆う目に見える成長があるまで待ち、次に、真菌の成長の外縁に最も近いコロニー化されたペトリ皿から直接餌媒体セクションを打ち抜きます(ステップ6.1で説明)。
- ベイト培地を直接プレコロニー化する場合は、ベイト培地全体に明確な増殖が見られるまで待ってから、カラムを基質に添加します(これには数日かかる可能性があります)。
注:以前の実験9 では、 C. cinerea を事前にコロニー化した14日齢の培地を餌培地として使用しました。コロニー形成前の時間は、真菌の成長率、培地の種類、およびインキュベーション条件によって異なります。
8. コントロール処理と複製の準備
- ネガティブコントロールカラムを調製し(ステップ1〜6で説明)、これらのカラムを基質に接種したり、配置したりしないでください。これらを使用して、後続の分析のベースラインを提供し、調製プロセスでの汚染を評価します。
- 実験には、列の反復を少なくとも 3 回含めます。
9. 基質へのカラムの追加
- 実験室の小宇宙を設定します(例:箱、ポット、またはチューブ内の土壌。 図3A)または、列を関心のあるフィールド サイトに移動します。
- コラムの底からラージキャップを取り外して、ネジ付きリングとメッシュを露出させます。
- 目的の基質(図 3)にカラムを添加し、以下で説明する各基質タイプの特定の考慮事項に従ってください。必要に応じて、メッシュへの損傷を最小限に抑えるために、カラムを基板に挿入する前に、手袋をはめた手または小さなツール(小さなこてなど)を使用して基板にくぼみを作ります。
- 糞:この柱は、以前は新鮮な馬の糞にうまく使用されていました。使用前に糞を4°Cで保存し、カラムを追加する前にマゼンタの箱などのチャンバーに加えてください。カラムを基板に挿入して、カラム全体の高さの1〜2 cmをカバーします。
- 土壌:底部全体が土壌内(土壌の深さの1~2 cm)になるように柱を挿入します(図3B)。柱は土の上に置くことも、ターゲット側の小さなキャップの真下まで埋めることもできます。
- 根圏:ステップ9.3.2の指示に従って、柱を特定の根の近くに角度を付けて配置しながら、植物の根の周囲の土壌に柱を挿入して、根圏微生物を捕獲する可能性を高めます(図3C)。
- チューブ内の基質:目的の基質を50 mLのコニカルチューブなどに~10 mL加え、必要に応じて(たとえば、非常に低湿度の環境で)純水を使用して基質を湿らせます。底が埋まり、ネジ付きリングやメッシュの一部が見えないように、カラムをチューブに挿入します(図3A)。50 mLチューブの蓋をねじ込み、50 mLチューブラックに入れて直立させます。
10. カラムを基板に残す
- カラムを基板に3〜21日間放置します。
注:カラムを基質に残す必要がある時間は、使用する餌菌の成長率、餌培地にコロニーを形成する菌類の成長率、および環境条件(たとえば、乾燥状態は培地の乾燥が速く、長く使用することはできません)によって異なります。 - 実験に必要な明暗サイクルを確立します。以前の実験では、実験室内のカラムは暗闇に保たれていましたが、フィールド内のカラムは局所的な昼夜条件にさらされていました9。
11. カラムを基板から取り外す
- カラムを基板に所望の時間接触させた後、カラムを取り外し、余分な基板を慎重に振り落とし、ラージキャップをメッシュの下のカラムの底部に戻してカラムを輸送します。
- カラムを滅菌ビーカーまたは50 mLチューブに入れて輸送します。輸送中はコラムを直立させてください。
12. カラムの標的培地からの分離株の培養
- 生物学的安全キャビネットなどの無菌環境では、ベイトメディアのラージキャップ、ネジ付きリング、メッシュを取り外します。メッシュを破棄します。
注:研究者が餌培地にコロニーを形成した生物に興味がある場合は、ターゲット培地プラグに加えて餌培地プラグを使用して手順12.3〜12.4に従ってください。 - ターゲット培地を含むカラムエンドからスモールキャップを取り外します。
- ターゲットミディアムプラグを取り外すには、カラムを裏返してターゲットプラグをカラムから落下させるか、滅菌した鉗子またはピペットチップを使用してプラグを抜きます。ターゲット培地プラグを、選択した寒天培地で満たされた90mmシャーレの中央に直接置きます(ステップ5で準備)。
注:ここでは、真菌または細菌特異的培地を使用して、どちらの王国からのメンバーの成長を促進することができます。プラグは、滅菌されたハサミまたはピペットチップを使用して2つ以上の部分に分割することもでき、複数の培地タイプにめっきしたり、DNA抽出用のターゲット培地を保存したりすることができます(ステップ14を参照)。 - 標的培地プラグを接種したペトリ皿を、暗所で25°Cで少なくとも72時間インキュベートします。
13. 微生物を分離するための継代培養
注: この手順はオプションです。
- 細菌の場合:滅菌接種ループを使用して、ペトリ皿の他の領域との接触を最小限に抑えながら、目的の単一のコロニーをスワイプします。R2Aなどの好ましい細菌培地(ステップ5で調製)を含む新鮮な90 mmシャーレにコロニーをストライクし、単一のコロニーを形成できる任意の方法(例:4つのゾーンにストリークする)を使用します。ペトリ皿を25-37°Cで24-72時間インキュベートし、コロニーの成長を確認します。
- 真菌の場合:滅菌された接種ループ、滅菌されたカミソリ、または滅菌されたピペットチップを使用して、菌糸の成長を含む寒天の最小(1 mm2)セクションを切り開きます。ステップ5で調製したように、MEAやPDAなどの好ましい真菌培地を含む新鮮な90mmのシャーレに寒天の小片を置きます。ペトリ皿を25°Cの暗所で最大1週間インキュベートし、過成長を防ぐために毎日チェックします。
- 必要に応じて、生物の形態が均一になるまでステップ1またはステップ2を繰り返します。
14. プレートから、またはターゲット培地から直接DNAを抽出する
- 1.5 mL 遠心分離チューブ内のカラムから抽出した寒天プラグ全体(ターゲットおよび/またはベイト)を -20 °C で凍結するか、抽出前にプラグの一部を 1.5 mL 遠心分離チューブの防腐剤に浸します。
- ターゲットおよび/またはベイト培地からの培養が行われ、その後の継代培養ステップが取られた場合は、真菌または細菌の増殖(またはその後の単離ステップが取られなかった場合は両方の混合物)を含む~1 cmの寒天をスタンプまたは切り出します。
- 単離された細菌コロニーの抽出には、滅菌接種ループを使用してプレートからコロニーをスワイプし、市販のDNA抽出キットに関連付けられた抽出バッファー内で直接渦巻きます(ステップ14.4)。
- 乳鉢と乳棒を使用して寒天片を液体窒素で別々に粉砕し、粉砕した組織を抽出チューブに移します。
- 土壌または細菌や真菌に最適化された市販のDNA抽出キットを使用し、製造元の指示に従ってDNAを抽出します( 材料の表を参照)。
- 得られたDNAを蛍光光度計または同等のシステムを使用して定量します。
15. アンプリコンまたはメタゲノムシーケンシングアプローチを用いた標的および/またはベイト培地中の微生物分類学的多様性の評価
- アンプリコンシーケンシング(16Sおよび/または内部転写スペーサー[ITS])またはメタゲノムシーケンシングのいずれかを以下の手順に従って実行します。
- アンプリコンシーケンシング:プライマーBakt 341F(5′-CCT ACG GGN GGC WGC AG-3′)およびBakt 805R(5′-GAC TAC HVG GGT ATC TAA TCC-3′)を使用して、V3-V4 16S rRNA遺伝子領域を増幅します。プライマーITS3 KYO2(5′-GAT GAA GAA CGY AGY RAA-3′)およびITS4(5′-TCC GCT GCT TGA TAT GC-3′)9を使用して、真菌ITS2領域を増幅します。
- 選択したシーケンサーと互換性のある市販のライブラリ調製キットを使用して、アンプリコンライブラリを調製します。ショートリードシーケンサーを使用してアンプリコンをシーケンシングし、シーケンシングプラットフォームの製造元のローディング濃度の指示に従って、十分なカバレッジで150または250の塩基対ペアエンドリードを生成します。
- メタゲノムシーケンシング:選択したシーケンサーと互換性のある市販のメタゲノムライブラリ調製キットを使用して、抽出したDNAからメタゲノムライブラリを作成します。ショートリードシーケンサーセットを使用してメタゲノムライブラリをシーケンシングし、シーケンシングプラットフォームの製造元のローディング濃度に関する指示に従って、十分なカバレッジ(メタゲノムあたり~10-20Gb)で150または250の塩基対ペアエンドリードを生成します。
16. シーケンシングデータの解析
- アンプリコンデータの分析: QIIME2 プラットフォーム11 と DADA212 を利用して、結果を視覚化する前にアンプリコンデータを分析します13。以下の手順に従って、Empowering the Development of Genomics Expertise (EDGE)14 Web ベースのバイオインフォマティクスプラットフォームで QIIME2 を実行します。QIIME2 は、オンラインで提供されている "Moving Pictures" ガイド (https://docs.qiime2.org/2024.2/tutorials/moving-pictures/) など、公開されているチュートリアルを使用して実行することもできます。
- https://edgebioinformatics.org/ に移動してログインするか、アカウントを作成します。
- ホームページから [RUN QIIME2 ] を選択します。左側のメニューバーで「 Upload Files 」を選択して、amplicon シーケンシングランからファイルをアップロードします。
- 「メタデータマッピングファイル」の横の「i」にカーソルを合わせたときに表示される指示を使用して、メタデータマッピングファイルを作成します。
- 必要な情報(プロジェクト/実行名、読み取りタイプ、パラメータなど)をすべて追加し、アップロードされた正しい入力データを選択します。DADA2が品質管理方法12として選択されていることを確認します。他のパラメータは、他の変更が必要でない限り、デフォルト値のままにしておくことができます。
- アンプリコンの種類として、細菌アンプリコンには 16S Greengenes (http://greengenes.lbl.gov)を、真菌アンプリコンには真菌ITSのいずれかを選択します。細菌(16S)と真菌(ITS)のデータを独立して分析します。
- 送信を押し、実行が終了するのを待って結果を表示します。
- アンプリコン データの視覚化を生成する (省略可能): GitHub で提供されているガイダンスに従って、および R Studio で利用可能な R パッケージ ヘルプに従って、phyloseq (https://github.com/joey711/phyloseq)、VEGAN (https://github.com/vegandevs/vegan)、APE (https://emmanuelparadis.github.io/) などの一般的なパッケージを使用して、R で追加のコミュニティ データ分析を実行します。
- メタゲノムデータの解析
- NMDC EDGEサイト15(https://nmdc-edge.org/home)に移動し、ORCiDアカウント(https://orcid.org/)を使用してログインします。
- 画面左側のメニューバーで 「ファイルのアップロード 」を選択し、正しい入力FASTQファイルをドラッグアンドドロップまたは参照します。
- 「メタゲノミクス」を選択し、画面左側のメニューバーで「複数のワークフローを実行」オプションを選択し、すべてのワークフローを「オン」に設定します。プロジェクト名と説明(オプション)を追加します。
- アップロードした raw 読み取り (fastq) ファイルを選択し、適切なファイル形式 (インターリーブまたはペア) を選択します。
- 実行を開始するには、[ 送信 ] をクリックし、実行が完了したら、画面上部の [マイ プロジェクト ] タブでプロジェクトを選択して、概要テーブルと視覚化を表示します。
17. アンプリコンおよび/またはメタゲノムの結果から分類データの追加の視覚化を作成する
- GitHub で提供されている手順に従って、GraPhlAn (https://github.com/biobakery/graphlan) パッケージを使用して円形のクラドグラムを生成します。
注:National Center for Biotechnology Information(NCBI)のタクソノミー(https://www.ncbi.nlm.nih.gov/taxonomy)識別子は、代表的な配列割り当てから取得し、Entrez Direct E-utilitiesの「eftech」プログラムに渡して、GraPhlAn16に必要なタクソノミーロールアップ情報を収集することができます。
18. 列の再利用
- カラムがまだ組み立てられている場合は、キャップ、ネジ付きリング、およびメッシュを緩めて取り外します。メッシュを破棄します。残っている寒天プラグをすべて取り外し、99%イソプロピルアルコールと精製水でカラムを洗浄し、ステップ3.1〜3.4の説明に従って成分を洗浄して乾燥させます。
- オートクレーブ滅菌する新しいナイロンメッシュのシートを準備します。
- 手順4.1で提供されたガイダンスに従って、コンポーネントをオートクレーブします。
結果
完全に組み立てられた菌類ハイウェイの支柱の長さは約5cmです(図1)。カラムはどの部分でも壊れてはならず、キャップとネジ付きリングは、カラム内に微小環境を作り出すために、簡単かつしっかりと組み合わされている必要があります。フィルターメッシュは、ねじ山付きリングを超えて延長することも( 図1 および 図2に示すように)、滅菌はさみでトリミングすることもできます。寒天プラグは、カラムの両端にぴったりと収まる必要があります。基板に配置すると、フィルターメッシュが基板に接触し、カラムが完全に埋まらないようにする必要があります。
カラムは以前に馬の糞9でテストされました。また、研究現場のバルク土壌と根圏土壌、および実験室の50 mLチューブ内の少量の土壌にもカラムを配置しました(図3)。真菌ハイウェイカラムを基質から取り除き、分解した後、ベイトとターゲットメディアプラグの両方で微生物の増殖が見られました( 図4Aに示す例)。細菌と真菌は、継代培養技術 によって 標的培地とベイト培地から単離され(図4B)、培地プラグ上に存在する微生物は、アンプリコンシーケンシングを使用して分類学的に同定されました(図4C、D)。 図4C、D は、複数の実験にわたるアンプリコンシーケンシングの結合結果を示しており、ウマ糞9に添加したカラムからどの微生物が標的培地プラグに到達できたかを示しています。この細菌および真菌データの視覚化は、ステップ 17 で概説したように生成されました。結果は、分類群の相対的な存在量として表示することもできます。
カラムを極端に低い湿度の環境に加え、培地プラグが数日で完全に乾燥し、コロニー形成された微生物の回収につながらなかった場合、最適ではない結果が得られました(図5A)。また、ターゲットメディアプラグから微生物が増殖しない場合(図5B)や、ターゲットメディアプラグから十分なシーケンシングデータを回収できず、有意義な解析ができない場合も確認されています。また、菌類がカラムから生い茂るケースもあり、実験をやり直す必要に迫られました(図5C)。

図3:実験室および野外で環境サンプルにカラムをセット した例(A)ラボで湿らせた土壌を入れた50 mLチューブ内にカラムをセットした状態。また、スケール用の定規と一緒に表示されます。(B)圃場の土に柱を埋め込んだもの。(C)フィールド内の植物の根ネットワークに配置された柱。 この図の拡大版を表示するには、ここをクリックしてください。
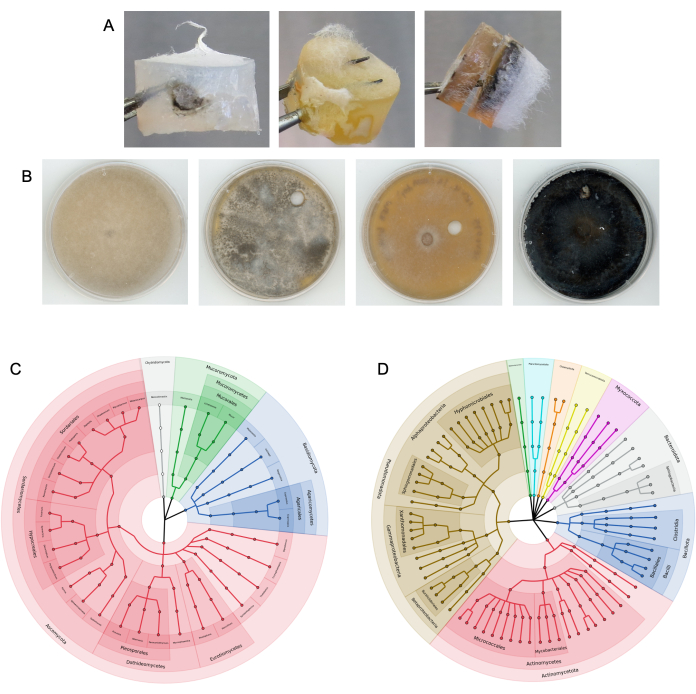
図4:成功したカラム実験の代表的な結果(A)カラムから抽出したコロニー形成された培地プラグの例。(B)標的培地から継代培養された真菌分離株の例。基材は土でした。上 ITS 配列 NCBI BLAST の同定 左から右へ: Rhizopus azygosporous、Aspergillus novofumigatus、Curvularia subpapendorfii、および Phaeomycocentrospora cantuariensis. (C,D) 馬糞を使用した複数の真菌ハイウェイ実験後に標的培地から回収された (C) 真菌 ITS および (D) 細菌 16S 配列の系統学的多様性を示す円形クラドグラム。セクションは門によって色分けされ、ラベル付けされ、エンドノードは一意の属を表します。この図の拡大版を表示するには、ここをクリックしてください。
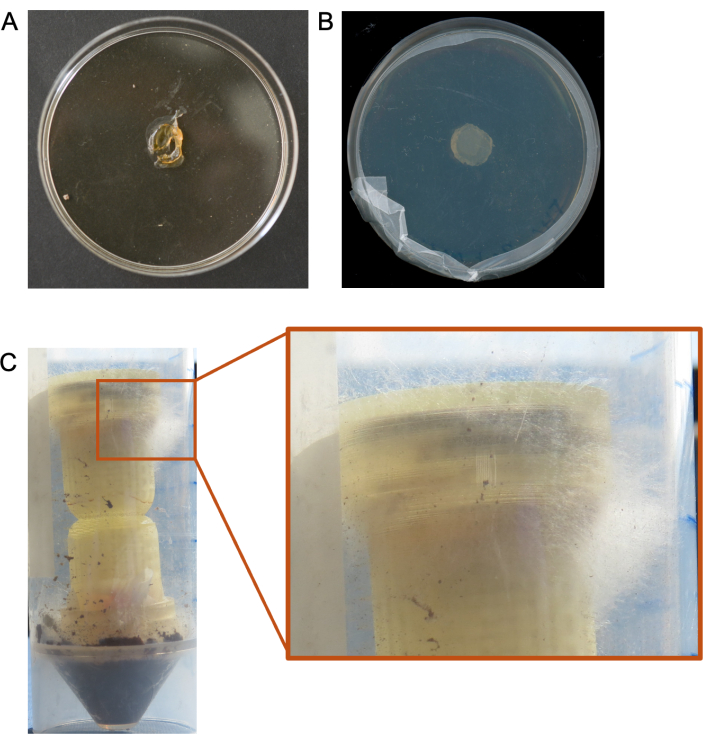
図 5.カラム実験の結果が最適ではない。 (A)低湿度の環境条件に起因する乾燥媒体プラグの例。(B)カラム培地プラグからの微生物増殖がない場合の例。(C)カラムの上部(ターゲット培地)を通る真菌の異常増殖の例。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
カラムコンポーネントを生成するとき、3Dプリンタとプリンティング材料の選択は、入手可能性と所望の材料特性に基づいて変更することができる17、18。生体適合性、表面の質感、オートクレーブ性、微細なディテールをプリントする能力、相対的な透明性など、すべて私たちのグループの材料選択で考慮されました。多孔性、疎水性、印刷パラメータなどの他の機能も考慮する必要があります。最終選考に先立って、さまざまな樹脂が試験され( 材料表を参照)、これらのカラムの印刷には多くの生体適合性材料が機能します。カラムコンポーネントの構造に選択された材料によって、どの洗浄、二次硬化、および滅菌アプローチを使用すべきかが決まります。すべての材料がオートクレーブ可能であるわけではなく、材料メーカーの指示に従って、紫外線、漂白剤、またはその他の滅菌技術が必要になる場合があります。一部の滅菌または洗浄技術も損傷したり、選択した材料と互換性がなかったりする可能性があるため、材料メーカーからのこの情報には特に注意を払う必要があります。3Dプリンターの場合、プリント時間、材料の互換性、ビルドプラットフォームのサイズ、プリント技術、コスト19などの考慮事項があります。3Dプリントされたカラムの部品は壊れやすく、力を入れすぎると壊れる可能性がある。リングとキャップのねじ山は必ずしも正確に位置合わせされているとは限らないため、組み立てステップの前に追加のコンポーネントを印刷して滅菌するか、選択したパラメータと材料がねじ山にどのように影響するかをテストするために予備印刷を行うことをお勧めします。キャップとリング内のねじ切りの設計仕様は、選択した3Dプリント材料に応じて調整する必要がある場合があります。寸法、格子の複雑さ、およびその他の物理的特徴はすべて、印刷前にCAD設計ソフトウェアで変更できます。設計通り、柱自体の高さは4cmで、柱の中心内の格子構造は2mmサイズのユニットセル、支柱径0.5mm、全体の格子高さは22mm9です。これらのパラメータは、研究者がたとえば、より大きなまたはより複雑な格子構造を必要とする場合に調整できます。全体として、これらのデバイスの3Dプリント製造は、設計の柔軟性を可能にすると同時に、単一の設計を組織やグループ間で標準化された方法で使用したり、教室の教育ツールとしても使用したりできるようにします9。
プロトコルのいくつかのステップでは、環境や実験のセットアップによってはトラブルシューティングが必要になる場合があります。真菌ハイウェイカラムは、低湿度条件下ではあまり効果的ではなく、メディアプラグが真菌の増殖を促進する前に急速に乾燥し、これらの環境での実験期間が制限される可能性があります(図5A)。低湿度環境でのカラムの有効性を向上させた技術には、基板に水分を添加して人工的に湿度を上げる方法や、カラムと基板を水源(純水の小さな容器など)で二次容器に密封する方法などがあります。砂時計の形状と格子構造は、高湿度環境で結露が発生した場合に細菌の移動のみを防ぐために組み込まれました(真菌の高速道路を確立することなく)。増殖の早い真菌は、ターゲットとベイト培地の表面積を過剰に増殖させ、カラムの上部または下部から伸びることがあります(図5C)。餌菌のインキュベーション時間または実験の期間を短くすると、この過成長を最小限に抑えるか排除できます。さらに、これらのデバイスの限界は、目的の基質で急速に成長する真菌が、成長の遅い真菌による餌および標的媒体のコロニー形成を制限する可能性があり、どのハイウェイ相互作用が観察されるかをバイアスする可能性があることです。一部の菌類、特に成長の遅い菌類は、寒天栓を通って格子構造に成長することを可能にする方法で餌培地にコロニーを形成しない場合があります。環境内に十分な湿度がある場合は、ベイト寒天プラグのコロニー形成後に、より薄い寒天プラグを使用して格子への成長を促進することができます。培地は、研究者が真菌または細菌の増殖のために選択したいかどうかに基づいて選択できますが、これにより、その培地タイプ20を好む生物にサブカルチャリングを制限することもできます。標的培地で増殖が見られない場合は、真菌ハイウェイを作成することが知られている真菌を餌培地または基質に接種する必要があるかもしれません。
メタゲノムシーケンシングまたはアンプリコンシーケンシングは、これらの実験の一部として行うことができ、これらの戦略は両方とも、それぞれに限界と長所を与える21。メタゲノムシーケンシングは、微生物に関する追加のゲノム情報を取得するのに理想的です。しかし、標的培地から直接回収可能な核酸の量は非常に少ない場合があるため、シーケンシングの前にアンプリコンシーケンシングやその他の増幅法を利用する必要がある場合があります。Amplicon シーケンシングライブラリは別々に調製する必要があり (16S と ITS)、この方法では分類学的分解能が不足しており、メタゲノムシーケンシングを使用して達成できるゲノムの特徴や機能的可能性に関する評価が制限されます。プラグからの直接シーケンシング法は、微生物を継代培養できない場合に好ましい場合があります。プラグを複数のセクションに分割して、培養アプローチとシーケンシングアプローチの両方を可能にすることをお勧めします。
これらのデバイスの利点は、実験室と現場の両方で使用できることです。フィールド内の柱が直立したままで、その配置を妨げる可能性のある動物や環境の摂動から保護されるように、特別な注意を払う必要があります。カラムは、基板で完全に覆われた水平位置ではまだテストされておらず、大量の降雨や雪にさらされる環境でのテストも行われていません。前述のように、格子構造は、高湿度環境下で細菌が目的の媒体に移動できる可能性を最小限に抑えるように設計されています。ただし、カラムが大量の水にさらされ、この水がカラムを完全に飽和させた場合、細菌の移動は、存在する真菌のハイウェイとは無関係にカラム全体で促進される可能性があります。ラボベースの実験では、カラムは50 mLの円錐管内、基質の小さな小宇宙内、鉢植えの植物を囲む土壌、箱内、またはその他の制御された実験システム内で使用できます。このカラムは、土壌、根圏、糞で成功裏に利用されており、その有用性は、落葉、汚泥、砂、雪、堆肥などの他の基質に拡大することができます。
真菌ハイウェイカラムにより、さまざまなサンプルタイプ内でこのBFI表現型を理解するための多くの比較が可能になります。餌と標的培地との間の群集組成を比較することで、どの細菌が真菌ハイウェイを利用できるか、そしてどの真菌が潜在的なハイウェイとして役立つことができるかを示すことができる9。メタゲノムシーケンシングを使用する場合、ベイトとターゲット培地の生物を区別するゲノム特徴も調べることができます。また、異なる基質(土壌と糞など)に配置したカラムや、同じ基質に異なる条件(温度や湿度など)で配置したカラムのターゲット培地を比較することも可能です。全体として、真菌ハイウェイカラムは、この形式のBFIを調査するための以前の方法の機能を拡張し、複雑な環境マイクロバイオームの空間ダイナミクスを形作るこれらの相互作用の広範な調査を可能にします。
開示事項
著者には、開示すべき利益相反はありません。
謝辞
この研究は、米国エネルギー省(DOE)、生物環境研究(BER)、生物システム科学部門(BSSD)の科学重点分野助成金(助成金番号LANLF59T)によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 50 mL tubes | Greiner BIO-ONE | 5622-7261 | 50 mL tubes for performing column experiments in the lab |
| 90 mm Petri dishes | Thermo Scientific Nunc | 08-757-099 | Petri dishes for preparation of agar and for microbial growth |
| Asiga Freeform Pico Plus 39 digital light processing (DLP) 3D printer | Asiga Germany | Freeform Pico Plus 39 | 3D printer used to generate batches of the columns; other 3D printers can be used |
| Autoclave | Fisher Scientific | LS40F20 | Benchtop autoclave to sterilize the column components |
| Beaker | Fisher Scientific | FB100600 | 600 mL beaker for various uses throughout the protocol |
| Dental LT Clear Resin V2 | Formlabs | RS-F2-DLCL-02 | Alternative resin for 3D printing that was tested |
| Dental Surgical Guide Resin | Formlabs | RS-F2-SGAM-01 | Was used to generate the columns discussed in manuscript; Other photosensitive resins can be used in place of this material |
| DNA Low Bind 1.5 mL tubes | Eppendorf | 13-698-791 | Tubes used for various preparations including nucleic acid extractions |
| DNA/RNA shield preservative | Zymo Research | R1100-50 | Preservative used prior to nucleic acid extractions |
| EDGE Bioinformatics | Open source; Developed by the Los Alamos National Laboratory (LANL) | n/a | Bioinformatics platform for processing amplicon data |
| FastDNA spin kit for soil | MP Biomedicals LLC | 116560200-CF | DNA extraction kit option for soil |
| Forceps | Fisher Scientific | 10-300 | Forceps that can be sterilized |
| Formlabs BioMed Clear Resin | Formlabs | RS-F2-BMCL-01 | Alternative resin for 3D printing that was tested |
| Formlabs Form 3B+ stereolithography (SLA) 3D printer | Formlabs | Form 3B+ | Alternative 3D printer |
| Formlabs IBT Resin | Formlabs | RS-F2-IBCL-01 | Alternative resin for 3D printing that was tested |
| Inoculating Loops | Fisher Scientific | 22-363-598 | Used to isolate/transfer microbes |
| Malt Extract Agar (MEA) | Criterion | 89405-654 | A media type used in columns |
| MiSeq sequencer + MiSeq sequencing kit | Illumina | SY-410-1003 | Can use other sequencers |
| Mortar & Pestle | Fisher Scientific | FB961K; FB961A | Can use any common mortar & pestle that can be sterilized between uses |
| NEBNext Ultra II DNA Library Prep Kit for Illumina | New England Biolabs | E7805S | Library prep kit for metagenomic sequencing |
| Nextera XT DNA Library Preparation Kit (24 samples) | Illumina | FC-131-1024 | Library prep kit for amplicon sequencing |
| NMDC EDGE | Open source: Developed by the National Microbiome Data Collaborative (NMDC) | n/a | Bioinformatics platform for processing metagenomic data |
| Nylon mesh | Sefar | 03-25/19 | The mesh used as part of the column construction |
| Pipette tips | Rainin | 30807966 | Can use many different sterilized pipette tips for the protocol steps |
| Potato Dextrose Agar | Cole Parmer | EW-14200-28 | A media type used in columns |
| QIIME2 | Open source | n/a | Software for processing amplicon data |
| Qubit dsDNA HS assay kit | Thermo Fisher Scientific | Q32851 | Used to quantify DNA after extractions |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Used to quantify DNA after extractions |
| Quick-DNA Fungal/Bacterial Miniprep Kit | Zymo Research | D6005 | DNA extraction kit option that works with both bacteria and fungi |
| R2A agar | BD Difco | 218263 | A media type used in columns (bacterial media) |
| Rack for 50 mL tubes | Fisher Scientific | 03-448-11 | Rack to hold 50 mL tubes upright |
| Scissors | Fisher Scientific | 12-000-155 | Fine precision scissors that can be sterilized |
| Sodium carboxymethyl cellulose medium | Aldrich | 419273-100G | A media type used in columns |
| SolidWorks CAD software | SolidWorks | n/a | Software used to design the columns |
| Trowel scoop | Fisher Scientific | S41701 | To make a depression in the substrate prior to adding the column |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen: ThermoFisher Scientific | 10977015 | Water for the ultrasonicator water bath |
| Ultrasonicator | Fisher Scientific | FB-11201 | Ultrasonicator for cleaning the columns |
参考文献
- Or, D., Smets, B. F., Wraith, J. M., Dechesne, A., Friedman, S. P. Physical constraints affecting bacterial habitats and activity in unsaturated porous media - A review. Adv Water Resour. 30 (6), 1505-1527 (2007).
- Kohlmeier, S., et al. Taking the fungal highway: Mobilization of pollutant-degrading bacteria by fungi. Environ Sci Technol. 39 (12), 4640-4646 (2005).
- Simon, A., Hervé, V., Al-Dourobi, A., Verrecchia, E., Junier, P. An in situ inventory of fungi and their associated migrating bacteria in forest soils using fungal highway columns. FEMS Microbiol Ecol. 93 (1), 217 (2017).
- Wick, L. Y., et al. Effect of fungal hyphae on the access of bacteria to phenanthrene in soil. Environ Sci Technol. 41 (2), 500-505 (2007).
- Bravo, D., et al. Isolation of oxalotrophic bacteria able to disperse on fungal mycelium. FEMS Microbiol Lett. 348 (2), 157-166 (2013).
- Furuno, S., Remer, R., Chatzinotas, A., Harms, H., Wick, L. Y. Use of mycelia as paths for the isolation of contaminant-degrading bacteria from soil. Microb Biotechnol. 5 (1), 142-148 (2012).
- Buffi, M., et al. Fungal drops: A novel approach for macro- and microscopic analyses of fungal mycelial growth. Microlife. 4, 042 (2023).
- Kuhn, T., et al. Design and construction of 3D printed devices to investigate active and passive bacterial dispersal on hydrated surfaces. BMC Biol. 20 (1), 203 (2022).
- Junier, P., et al. Democratization of fungal highway columns as a tool to investigate bacteria associated with soil fungi. FEMS Microbiol Ecol. 97 (2), 003 (2021).
- Reasoner, D. J., Geldreich, E. E. A new medium for the enumeration and subculture of bacteria from potable water. Appl Environ Microbiol. 49 (1), 1-7 (1985).
- Bolyen, E., et al. Reproducible, interactive, scalable, and extensible microbiome data science using QIIME 2. Nat Biotechnol. 37 (8), 852-857 (2019).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nat Methods. 13 (7), 581-583 (2016).
- Vázquez-Baeza, Y., Pirrung, M., Gonzalez, A., Knight, R. EMPeror: A tool for visualizing high-throughput microbial community data. Gigascience. 2 (1), 16 (2013).
- Li, P. -. E., et al. Enabling the democratization of the genomics revolution with a fully integrated web-based bioinformatics platform. Nucleic Acids Res. 45 (1), 67-80 (2017).
- Eloe-Fadrosh, E. A., et al. The National Microbiome Data Collaborative Data Portal: An integrated multi-omics microbiome data resource. Nucleic Acids Res. 50 (1), D828-D836 (2022).
- Entrez Direct: E-utilities on the Unix Command Line. Entrez Programming Utilities Help Available from: https://www.ncbi.nlm.nih.gov/books/NBK179288/ (2024)
- Palmara, G., Frascella, F., Roppolo, I., Chiappone, A., Chiadò, A. Functional 3D printing: Approaches and bioapplications. Biosens Bioelectron. 175, 112849 (2021).
- Guttridge, C., Shannon, A., O'Sullivan, A., O'Sullivan, K. J., O'Sullivan, L. W. Biocompatible 3D printing resins for medical applications: A review of marketed intended use, biocompatibility certification, and post-processing guidance. Ann 3D Print Med. 5, 100044 (2022).
- Yao, L., et al. Comparison of accuracy and precision of various types of photo-curing printing technology. J Phys Conf Ser. 1549 (3), 032151 (2020).
- Basu, S., et al. Evolution of bacterial and fungal growth media. Bioinformation. 11 (4), 182-184 (2015).
- Liu, Y. -. X., et al. A practical guide to amplicon and metagenomic analysis of microbiome data. Protein Cell. 12 (5), 315-330 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved