Method Article
オフターゲット遺伝子編集の検討のためのCIRCLE-Seq
要約
CRISPRのような技術に対する大きな障壁は、重要な遺伝子を破壊する可能性のあるオフターゲットイベントです。「Circularization for In Vitro Reporting of Cleavage Effects by Sequencing」(CIRCLE-seq)は、意図しない切断部位を特定するために設計された技術です。この方法は、CRISPR-Cas9のゲノムワイドな活性を高感度かつバイアスなくマッピングします。
要約
Circularization for In Vitro Reporting of Cleavage Effects by Sequencing (CIRCLE-seq) は、CRISPR-Cas9 切断された DNA のターゲットシーケンシングを通じて、CRISPR-Cas9 の意図しない切断部位を公平に同定するために開発された新しい手法です。このプロトコルには、ゲノムDNA(gDNA)の環状化が含まれ、その後、Cas9タンパク質と目的のガイドRNA(gRNA)で処理されます。処理後、切断されたDNAは精製され、Illuminaシーケンシング用のライブラリーとして調製されます。シーケンシングプロセスでは、ペアエンドリードが生成され、各切断部位に関する包括的なデータが提供されます。CIRCLE-seqは、Cas9切断gDNAのシーケンシング深度要件が最小限であること、バックグラウンドが低いこと、高濃縮であることなど、他の in vitro 法に比べていくつかの利点があります。これらの利点により、意図的な切断イベントと意図しない切断イベントの両方を識別する際の感度が向上します。この研究では、CIRCLE-seqを使用してCRISPR-Cas9のオフターゲット活性を調べるための包括的で段階的な手順を提供します。一例として、このプロトコルは、 AAVS1 遺伝子座の修飾中にCRISPR-Cas9のゲノムワイドな意図しない切断部位をマッピングすることによって検証されます。CIRCLE-seqの全プロセスは2週間で完了し、細胞増殖、DNA精製、ライブラリ調製、およびIlluminaシーケンシングに十分な時間を確保できます。シーケンシングデータをCIRCLE-seqパイプラインに入力することで、切断部位の解釈と解析を効率化できます。
概要
ゲノム工学は過去20年間で大きな進歩を遂げており、大きな節目は2012年にクラスター化された規則的に間隔を空けた短い回文反復配列(CRISPR)-Cas9の発見です1。CRISPR-Cas9テクノロジーは、細菌のDNAエンドヌクレアーゼのプログラム可能な性質を活用して、ほぼすべてのDNA配列の正確な標的化と修飾を可能にします。創業以来、このシステムは、Cas9エンドヌクレアーゼとガイドRNA(gRNA)のみに依存して特定のゲノム領域を編集するように最適化されてきました。CRISPR-Cas9の治癒療法としての可能性は、レーバー先天性黒内障、トランスサイレチンアミロイドーシス、鎌状赤血球貧血などのさまざまな疾患の臨床試験で実証されています2,3,4。
CRISPR-Cas9は二本鎖切断(DSB)を誘導しますが、これは通常、エラーが発生しやすい非相同末端結合(NHEJ)またはより正確な相同組換え修復(HDR)の2つのメカニズムのいずれかによって解決されます(テンプレートDNAが利用可能である場合)。CRISPR-Cas9は、意図しないゲノム部位での切断とともに、NHEJ関連の挿入および欠失(インデル)を引き起こす傾向があり、臨床現場でのその適用を制限します5,6,7,8,9,10。さらに、意図しないゲノム改変は、いくつかのゲノム編集試験で観察されたように、不可解なスプライス部位、ナンセンスまたはミスセンス変異を作り出し、染色体裂を誘導し、または細胞に発がん性の可能性を与える可能性があります 11,12,13,14,15.結論として、CRISPR-Cas9のオフターゲット活性を正確に特定することは、その臨床応用、特に数十億の細胞を変化させる可能性のある全身性遺伝子治療にとって非常に重要です。
CRISPR-Cas9のオフターゲット切断部位を同定するためには、二本鎖オリゴデオキシヌクレオチドを使用して生細胞のDSBをタグ付けするGenome-wide Unbiased Identification of Double-stranded breaks (GUIDE)-seq 16など、さまざまな方法を採用することができます。それにもかかわらず、この方法に対する批判は、偽陽性がランダムなDSBまたはPCRアーティファクトから発生する可能性があり、オンターゲットサイトとの類似性が低いキャプチャーされたサイトを除外することによって破棄する必要があるというものです。インテグラーゼ欠損レンチウイルスベクター(IDLV)の使用に基づく方法は、感度が低く、多くのオフターゲット部位を見逃す可能性があります17。DSBCapture、BLESS、BLISS 18,19,20などの他のin situ法は、固定細胞と標識DSBを直接関与させますが、即時のDSB捕捉に依存し、外因性DNAが存在しないという制約があります。in vitro法であるDigenome-seq21とSelective enrichment and Identification of Tagged genomic DNA Ends by sequencing (SITE-seq)22は、どちらもシーケンシングソリューションを提供しますが、それぞれバックグラウンドノイズとシングルエンド解析に制限があります。in situ Casオフターゲットの発見とシーケンシング(Discover-Seq)23による検証は、MRE11結合を介したCas9活性のin vivoおよびin situ同定を提供するが、サンプル調製時に存在するDSBのみを検出する24。最後に、Inference of CRISPR Edits(ICE)は、バイオインフォマティクスアプローチを使用して、サンガーデータ25を使用してCRISPR編集をロバストに解析します。
この記事では、Circularization for In Vitro Reporting of Cleavage Effects by Sequencing (CIRCLE-seq): 目的のgRNAとの複合体におけるCas9ヌクレアーゼのゲノムワイドなオフターゲット活性を高感度かつ公平にマッピングする in vitro技術について説明する26。このアプローチは、目的の細胞を培養してDNAを単離することから始まり、続いて集束超音波処理によるランダムせん断、次にエキソヌクレアーゼとリガーゼの処理が行われます。このプロセスにより、最終的に環状二本鎖DNA分子が生成され、プラスミドセーフDNase処理によって精製されます。次に、この環状DNAはCas9-gRNA複合体に曝露され、意図された切断部位と意図しない切断部位の両方で切断され、Illuminaアダプターライゲーションの基質として機能する露出したDNA末端が残ります。このプロセスにより、各ヌクレアーゼ誘導DSBの両端を含む多様なゲノムDNA(gDNA)ライブラリが生成され、各リードが各切断部位に必要なすべての情報を持つようになります。これにより、シーケンシングカバレッジ要件を低く抑えたIlluminaシーケンシングを使用することができ、CIRCLE-seqは上記の他の同様の方法とは一線を画しています。CIRCLE-seqは in vitro 法として他のプロトコルよりも高いオフターゲット感度を持っていますが、これにはGUIDE-seq16などの他の方法に存在するエピジェネティックなランドスケープがないため、偽陽性が高くなることに注意することが重要です。さらに、DSB DNA修復とそれに関連する機構はCIRCLE-seqには存在せず、そうでなければ観察されるであろうインデルや適切な修復を抑制します。
CIRCLE-seqを実行するための段階的なプロトコールを説明するだけでなく、このプロトコールは、 AAVS1 遺伝子座の修飾中に発生するCRISPR-Cas9のゲノムワイドな意図しない切断部位を一例として特定することによって検証されます。このわかりやすいプロトコールでは、人工多能性幹細胞(iPSC)の培養やgDNAの単離から、gDNAの環状化、Cas9-gRNAの切断、ライブラリ調製、シーケンシング、パイプライン解析まで、詳細な手順を説明します。シーケンシングカバレッジの要件が低いため、CIRCLE-seqは次世代シーケンシングにアクセスできるすべてのラボで利用できます。
プロトコル
本試験に使用した試薬、消耗品、機器の詳細は、 資料表に記載されています。
1. 細胞培養(5日間)
- このプロトコル全体にネガティブコントロールを含めます。追加の 25 μg の gDNA (~2.0eサンプルあたり 7 細胞) に十分な細胞を調製します。
- 確立されたプロトコル27に従ってiPS細胞を培養します。細胞を採取し、10mLのPBSに再懸濁します。細胞の6 μLサンプルをピペットで移し、トリパンブルーと1:1の比率で再懸濁します。自動セルカウンターを使用してサンプルをカウントします。
- チューブあたり2 x 107 個の細胞を分注し、次いで300 x g で25°C(室温(RT))で3分間スピンダウンします。これは、複数の反復には十分です。ピペットでオフにし、上清を捨てます。
2. ゲノムDNAの単離(1日)
- 市販のDNA精製キットを使用して、製造元の指示に従ってgDNAを単離します。
- 細胞ペレットが入った15 mLのコニカルチューブに200 μLのPBSを加え、再懸濁します。次に、3 mLの細胞溶解バッファーと15 μLのプロテイナーゼKをチューブにピペットで移します。チューブを25回反転させて完全に混合します。チューブを55°C、150rpmに設定したウォーターバスシェーカーに3時間、または一晩置くと、最適なDNA収量が得られます。
- 15 μLのRNase Aをピペットで入れ、25回反転します。37°Cの水浴に1時間入れます。
- サンプルを氷の上で5分間冷やします。次に、1 mLのタンパク質沈殿溶液を添加し、高速で20秒間ボルテックスし、2000 x g でRTで10分間遠心分離します。ペレットが見えない場合は、サンプルを氷上でさらに5分間インキュベートし、再度遠心分離します。
- 3 mLの100%イソプロパノールを新しい15 mLコニカルチューブに加えます。ステップ2.1.3の上清を慎重にチューブにピペットで移します。チューブを50回反転させて混合し、2000 x g 、室温で3分間遠心分離します。DNAペレットを乱さないように、真空トラップに接続されたパスツールピペットを使用して上清を慎重に吸引し、清潔で糸くずの出ないワイプでチューブを反転させます。
- 70%(v / v)エタノール3mLをDNAペレットにピペットで移し、10回反転させて洗浄します。2,000 x g でRTで3分間遠心分離します。次に、上澄みを慎重に注ぎます。
- チューブを開いたままにして、得られたDNAペレットを30分間乾燥させ、すべてのエタノールが完全に蒸発したことを確認します。50 μLのDNA水和液にピペットで移し、穏やかなピペッティングで十分に混合します。
- サンプルを65°Cのウォーターバスシェーカーに1時間入れてDNAを溶解し、サンプルを室温で一晩放置します。サンプルをRTおよび2,000 x g で1分間遠心分離し、dsDNA BR Assay Kitと関連チューブを使用して、蛍光光度計で単離されたDNAを定量します。
3. gRNAの調製(7日間)
- 目的の合成gRNAを市販のソースから注文してください( 資料表を参照)。このプロトコールは、crRNA/tracrRNAとも互換性があります。
4. gRNA のin vitro 切断試験
注:ここでは、 AAVS1 遺伝子のターゲットが使用されます。他の目的の遺伝子を標的とするには、標的領域を増幅するプライマーを設計し(表1)、次のステップのプライマーをカスタムプライマーに置き換えます。
- PCR反応液の準備:Phusion Hot Start Flex 2x Master Mix 25 μL(最終濃度1x)、 AAVS1 F Primer 0.5 μL(最終濃度0.1 μM)、 AAVS1 R Primer 0.5 μL(最終濃度0.1 μM)、gDNA 5 μL(ステップ2.1.6から100 ng、20 ng/μL)、Nuclease-free-H2O(総容量: 50μL)。
- 次のサーモサイクラーパラメータを使用します:変性:98°Cで2分間(1サイクル)、変性:98°Cで10秒(10サイクル)、アニーリング:72-62°C(-1°C/サイクル)で15秒(10サイクル)、延長:72°Cで30秒(10サイクル)、変性、98°Cで10秒(30サイクル)。アニーリング:65°Cで15秒(30サイクル)、伸長:72°Cで30秒(30サイクル)、最終伸長:72°Cで5分間(1サイクル)、保持:4°C無期限。
- AMPure XP ビーズを使用して、PCR 反応産物を精製します。まず、1.8倍容量(90 μL)のXPビーズをPCR産物にピペットで移します。ピペットで10回動かしてよく混ぜます。混合物を室温で5分間放置してインキュベートします。
- 磁気ラックを使用して、PCR反応プレートを磁石に3分間置き、ビーズを溶液から分離します。透明化した溶液をピペットで取り出し、廃棄します。ビーズに200 μLの80%エタノール(v / v)を加え、30秒間インキュベートした後、エタノールを除去します。この洗浄ステップを2回繰り返して、エタノールが完全に除去されるようにします。
- サンプルを磁石の上に置いて、サンプルを3分間自然乾燥させます。プレートを磁石から取り出し、40 μL の TE バッファー (pH 8.0) を加えます。ピペッティングで10回上下させて混合します。サンプルを室温で2分間放置します。
- PCR反応プレートを磁石の上にさらに1分間置きます。1分後、上清を新しいプレートに移します。分光光度計を使用して精製されたPCR収量を測定し、メーカーの指示に従って、光学チューブストリップキャップ付きの光学チューブストリップ、高感度D1000スクリーンテープおよび高感度D1000試薬(ラダーおよびバッファー)を使用してTapeStationで分析します。調製したサンプルは、-20°Cで最大数か月間保存します。
- Cas9ヌクレアーゼタンパク質を1 μMに希釈します:2 μLの10x Cas9バッファー(最終濃度1x)、1 μLのCas9 Nuclease、 S. pyogenes (最終濃度1 μM)、および17 μLのヌクレアーゼフリーH2Oを総容量20 μLで混合します。
- RNaseフリーの手順を実行して、gRNAの分解を防ぎます。gRNA(ステップ3.1から)をH2Oで3 μMに希釈し、総容量を10 μLにします。
注:gRNAの分子量を推定するには、次の式を使用します:ssRNAの分子量(g / mol)=(ssRNAの長さ(nt)x 321.47 g / mol)+ 18.02 g / mol。参考までに、3 μMの104 nt長gRNAは約100 ng/μLです。
5. DNAシャーリング(3時間)
- 最初にコントロールアームをホーミングしてME220を準備します。次に、精製された脱イオンH2Oをリザーバーに充填します。Control Stationのラップトップで、 Water Works にアクセスし、[ Fill]をクリックします。温度を4.5°Cに調整します。
- 25 μg の gDNA をマイクロチューブ (microtube-130 AFA ファイバー スクリュー キャップ) に移します。次に、1x TEでチューブを総容量130μLまで充填します。次の条件を使用して、DNAを平均長約300 bpにせん断します: Duration を 10秒に設定します。 ピーク電力 は 70まで。 デューティファクター% を 20に。 サイクル/バースト は 50まで。これらすべてにより、 平均電力 が自動的に 14.0に設定されます。
6. 剪断されたゲノムDNAの精製(1時間)
- 剪断したゲノムDNAをそれぞれ65μLの2つの部分に分割します。ステップ4.3-4.6で概説した手順に従って、1.8倍の容量のXPビーズ(117μL)を使用して精製します。上清を新しいPCRプレートに移し、分光光度計を使用して量を測定します。
- 溶出した剪断したgDNAを1 μLのTapeStationで、製造元の指示に従って、gDNAが約300 bpの広範囲に剪断されていることを確認します。必要に応じて、せん断したgDNAを-20°Cで最大数か月間保存します。
7. CIRCLE-seqライブラリーの準備(3日間)
- ヘアピンアダプターのアニーリング
- ヘアピンアダプターであるoSQT1288(表1)を1x TEで最終濃度100μMまで再懸濁します。
- アダプターのアニールは、oSQT1288 40 μL (最終濃度 40 μM)、10 μL の 10x STE (最終濃度 1x)、ヌクレアーゼフリー H2O 50 μL を混合して、合計容量 100 μL にします。
- 次のアニーリングパラメータを使用します:95°Cで5分間、-1°C/分で70サイクル、4°Cで無期限に保持します。
- 修復の終了を実行します。PCRフリーHTPライブラリ調製キットを使用し、End-Repair Master Mixを調製します。
- 8 μL のヌクレアーゼフリー H2O、7 μL の 10x 末端修復バッファー (最終濃度 1x)、5 μL の末端修復酵素ミックス (最終容量 20 μL の Total End-Repair Master Mix) を混合します。
- ステップ4.3-4.6から剪断したgDNAサンプルに、エンドリペアマスターミックス20μLをピペットで入れます。20 μLのEnd-Repair Master Mixと50 μLのせん断されたgDNAを混合し、最終容量を70 μLにします。
- 混合物を20°Cのサーモサイクラーに30分間置き、その後4°Cで無期限に保持します。
- 1.7倍容量(120 μL)のXPビーズを添加し、ステップ4.3〜4.6の精製手順に従います。42 μL の TE、pH 8.0 で溶出します。次のステップのために、ビーズが溶液に残っていることを確認してください。
- Aテーリングを実行します。HTP Library Preparation(PCR フリー)キットを使用して、A-tailing Master Mix を調製します。
- 5 μL の 10x A-テーリングバッファー (最終濃度 1x) と 3 μL の A-テーリング酵素 (最終容量合計 8 μL の A-テーリングマスターミックス) を混合します。
- ステップ7.2.4のビーズを含む各DNA試料に8 μLのA-tailing Master Mixをピペットで移し、8 μLのA-tailing Master Mixをビーズを含む42 μLの末端修復DNAに混合します(最終容量の合計は50 μL)。サーモサイクラーに30°Cで30分間置きます。4°Cで無期限に保持します。
- PEG/NaCl SPRI溶液(HTPライブラリ調製キット(PCRフリー、96反応)の成分)をAテールDNAに1.8容量または90 μLピペットで注入します。AテールDNAをステップ4.3-4.6に従って精製します。A-tailed DNAを30 μLのTE溶液(pH 8.0)で溶出します。次のステップのためにビーズを溶液に保持します。
- アダプターのライゲーションを実行します。HTP Library Preparation(PCR フリー)キットを使用して、Adapter Ligation Master Mix を調製します。
- ステップ7.1.3から、5xライゲーションバッファー(最終濃縮1x)10 μL、DNAリガーゼ5 μL、アニーリングヘアピンアダプター(40 μM)5 μLを混合します。アダプターライゲーションマスターミックスの合計が20 μLの場合、最終濃度は4 μMであることを確認してください。
- ステップ 7.3.3 で抽出したビーズを含む各 DNA 試料に Adapter Ligation Master Mix 20 μL をピペットで注入します (サンプルあたり最終容量合計 50 μL)。
- 20°Cのサーモサイクラーに1時間置きます。4°Cで無期限に保持します。
- 1倍容量または50 μLのPEG/NaCl SPRI溶液をアダプターライゲーションDNAに移し、ステップ4.3-4.6に従って精製します。30 μL の TE、pH 8.0 で溶出し、上清を新しいセミスカート PCR プレートにデカントします。dsDNA BRアッセイを使用してDNAを組み合わせ、定量します。必要に応じて、精製したアダプターライゲーションDNAを-20°Cで最大1か月間保存します。
- Lambda Exonuclease/Exonuclease I (E. coli) Master Mix(両端にライゲーションされたアダプターなしで一本鎖または二本鎖DNAを除去させる機能)を調製します。
- ステップ7.4.4から1 μgのAdapter Ligated DNAを取り出し、40 μLに希釈します。10x Exonuclease I 反応バッファー 5 μL (最終濃度 1x)、エキソヌクレアーゼ Lambda 4 μL (最終濃度 0.4 U/μL)、および Exonuclease I (大腸菌) 1 μL (最終濃度 0.4 U/μL) を混合して、Lambda Exonuclease/Exonuclease I Master Mix の総容量 10 μL にします。
- Lambda Exonuclease/Exonuclease I Master Mixの10 μLを40 μL(1 μg)のアダプターライゲーションDNA(総容量50 μL)にピペットで移します。サーモサイクラーに37°Cで1時間、次に75°Cで10分間置きます。4°Cで無期限に保持します。
- 1.8倍の容量または90 μLのXPビーズをラムダエキソヌクレアーゼ/エキソヌクレアーゼI処理DNAにピペットで取り付けます。手順4.3〜4.6の指示に従って精製します。40 μL の TE、pH 8.0 で溶出します。ビーズが次の酵素ステップのために溶液中に残っていることを確認してください。
- USER酵素とT4ポリヌクレオチドキナーゼ(PNK)で治療します。USER/T4 PNK Master Mixを調製します(4 bpのオーバーハングを放出し、その後のライゲーション反応に必要なライゲーション準備完了DNA末端を調製するために必要)。
- 5 μL の 10x T4 DNA リガーゼバッファー (最終濃度 1x)、3 μL の USER 酵素 (最終濃度 0.05 U/μL)、および 2 μL の T4 PNK (最終濃度 0.4 U/μL) を混合し、USER/PNK マスターミックスの総容量 10 μL を得ます。
- USER Enzyme/T4 PNK Master Mix 10 μL をステップ 7.5.3 のビーズを含む Lambda and Exonuclease I 処理 DNA 試料 40 μL にピペットで移し、総容量 50 μL にします。サーモサイクラーに37°Cで1時間置きます。4°Cで無期限に保持します。
- USER/T4 PNKで処理したDNAにPEG/NaCl SPRI溶液を1.8倍容量または90 μLにピペットで移し、ステップ4.3-4.6に従って精製します。35 μL の TE、pH 8.0 で溶出します。上清を新しいセミスカートPCRプレートにデカントします。dsDNA HSアッセイを使用してDNAを組み合わせ、定量します。
- 分子内環状化を行います。Circularization Master Mixを調製します。
- ヌクレアーゼフリーH2Oを8 μL、10x T4 DNAリガーゼバッファー(最終濃度1x)を10 μL、T4 DNAリガーゼを2 μL(最終濃度8 U/μL)を混合し、Circularization Master Mixの総容量を20 μLにします。
- ステップ 7.6.3 で USER/PNK で処理した DNA 500 ng に Circularization Master Mix 20 μL をピペットで移します。USER/PNKで処理したDNA500 ngを80 μLで希釈し、Circularization Master Mix(総容量100 μL)を20 μL加えます。サーモサイクラーで16°Cで16時間(一晩)インキュベートします。
- 環状DNAに1倍容量または100 μLのXPビーズを加え、ステップ4.3-4.6に従って精製します。38 μL の TE、pH 8.0 で溶出します。上清を新しいセミスカートPCRプレートにデカントします。
- プラスミドセーフATP依存性DNaseで処理します。プラスミドセーフATP依存性DNaseマスターミックスを調製します(残留直鎖DNAの分解に必要)。
- 5 μL の 10x Plasmid-Safe Reaction Buffer (Final Concentration 1x)、2 μL の ATP (Final Concentration 1 mM)、および 5 μL の Plasmid-Safe ATP-Dependent DNase (Final Concentration 1 U/μL) を混合し、Plasmid-Safe Master Mix の総容量を 12 μL にします。
- ステップ 7.7.3 で ATP 依存性 DNase Master Mix 12 μL を 38 μL の環状化 DNA にピペットで移します (総容量 50 μL)。サーモサイクラーで37°Cで1時間インキュベートし、次に70°Cで30分間インキュベートします。4°Cで無期限に保持します。
- プラスミドセーフATP依存性DNaseで処理したDNAにXPビーズを1倍量または50 μLピペットで注入し、ステップ4.3-4.6に従って精製します。15 μL の TE、pH 8.0 で溶出します。上清を新しいセミスカートPCRプレートにデカントします。
- DNAを組み合わせ、dsDNA HSアッセイを使用して定量します。必要に応じて、環状DNAを-20°Cで最大数ヶ月間保存します。
8. 酵素精製された環状化gDNAを in vitro で切断(2時間)
- Cas9:gRNA複合体を用いて in vitro で切断を行います。 in vitro Cleavage Master Mixを調製します。5 μL の 10x Cas9 バッファー (最終濃度 1x)、4.5 μL の 化膿性ブドウ球菌 Cas9 (最終濃度 90 nM)、1.5 μL の gRNA (最終濃度 90 nM) を混合すると、Cleavage Master Mix の総容量は 11 μL になります。
- 切断マスターミックスを室温で10分間保持し、Cas9:gRNP複合体を形成します。
- ステップ 7.8.3 で 125 ng のプラスミドセーフ DNase 処理 DNA を最終容量 39 μL まで希釈します。次に、11 μLのCleavage Master Mixを39 μLのプラスミドセーフDNase処理DNAに加え、総容量を50 μLにします。
注:このステップには、Cas9:gRNA複合体を含まない、Cas9バッファーと混合された環状DNAを含むネガティブコントロール標本を含めます。 - サーモサイクラーで37°Cで1時間インキュベートします。 4°Cで無期限に維持します。 in vitroで切断したDNAに50 μL (1x容量) のXPビーズを添加し、ステップ4.3-4.6に従ってDNAを精製します。42 μL の TE バッファー (pH 8.0) で溶出します。次のステップのために、ビーズが溶液中に留まることを確認してください。
9. 次世代シーケンシングライブラリーの調製 (4 - 6 時間)
- Aテーリングを実行します。A-tailing Master Mixを調製します。
- 5 μL の 10x A-tailing Buffer (最終濃度 1x) と 3 μL の A-tailing Enzyme (A-tailing マスターミックスの総容量は 8 μL) を混合します。
- A-tailing Master Mix 8 μL をステップ 8.4 のビーズを含む溶出 DNA サンプル 42 μL にピペットで移します (総容量 50 μL)。サーモサイクラーに30分間、30°Cで置きます。 4°Cで無期限に保持します。
- 1.8倍容量のPEG/NaCl SPRI溶液(90 μL)をAテールDNAにピペットで移し、ステップ4.3〜4.6に従ってDNAを精製します。25 μL の TE、pH 8.0 で溶出します。次のステップのために、ビーズを溶液に保持してください。
- アダプターのライゲーションを実行します。アダプターライゲーションマスターミックスを調製します。
注:NEBアダプターのシングルユースアリコートは、3'T'の凍結融解による加水分解によって引き起こされるアダプターダイマーの形成を防ぐために準備する必要があります。- 5x Ligation Buffer (Final Concentration 1x) 10 μL と DNA Ligase 5 μL と 10 μL のシーケンシング用アダプター (Final concentration is 3 μM) を合計 25 μL 混合します。
- アダプターライゲーションマスター25μLをピペットで、ステップ9.1.3のビーズを含むAテールDNAサンプル25μLにミックスします。サーモサイクラーに20°Cで1時間置きます。 4°Cで無期限に保持します。
- アダプターライゲーションされたDNAにPEG/NaCl SPRI溶液を1倍容量または50 μLでピペットで移し、ステップ4.3-4.6に従ってDNAを精製します。47 μL の TE、pH 8.0 で溶出します。ビーズが次の酵素ステップのために溶液中に残っていることを確認してください。
- USER Enzymeで処理を行います(ウラシル残基に1つのヌクレオチドギャップを生成します)。
- ステップ9.2.3のビーズを含むアダプターライゲーションDNA試料に、3 μLのUSER酵素(Dual Index Primers Kitに付属)を加えます。37°Cで15分間インキュベートします。
- USER酵素処理DNAに35 μL (0.7倍容量) のPEG/NaCl SPRI溶液を加え、ステップ4.3-4.6に従って精製します。20 μL の TE バッファー (pH 8.0) で溶出します。上清を新しいセミスカートPCRプレートに移し、dsDNA HSアッセイを使用してDNA濃度を測定します。予想される濃度は約2〜5 ng /μLです。
- (オプション)次のステップに進む前に、PippinHTを使用してDNAサイズの選択を行うことができます。1.5% PippinHTカセットを250-850 bpのサイズ範囲でご使用ください。得られたサンプルは、次のステップのPCRで直接利用できます。
- バーコード添加のためのPCRの実施
注:各サンプルで選択したプライマー配列の組み合わせが一意であることを確認してください。可能であれば、各サンプルには一意の i5 バーコードと i7 バーコードが必要です。- PCR Master Mixを調製して、paired-indexバーコードを追加します。ヌクレアーゼフリーのH2Oを5 μL、2x HotStart Ready Mix(最終濃度1x)を25 μL、i5 Primer(最終濃度1 μM)を5 μL、i7 Primer(最終濃度1 μM)を5 μL(マスターミックスの総容量40 μL)を混合します。
- ステップ9.3.2からUSER酵素(約20 ng)で処理した精製DNA10 μLにPCR Master Mix40 μLをピペットで移します(総容量50 μL)。
- 以下のPCRサーモサイクル条件を選択してください:変性:98°C、45秒、1サイクル、変性:98°C、15秒、20サイクル、アニーリング:65°C、30秒、20サイクル、延長:72°C、30秒、20サイクル、最終延長:72°C、1分間、1サイクル、保持:4°C無期限。
- 0.7倍容量(35 μL)のXPビーズをPCR産物に加え、ステップ4.3〜4.6に従って精製します。30 μL の TE、pH 8.0 で溶出します。上清を新しいセミスカートPCRプレートにデカントします。必要に応じて、環状DNAを-20°Cで最大数ヶ月間保存します。
注:TapestationでPCRのサンプルを解析し、ライブラリの品質を制御し、アダプターダイマーの形成を評価します。アダプター・ダイマーが検出された場合は、ステップ 9.5.4 を繰り返します。
10. Droplet Digital PCR (dd_PCR) によるCIRCLE-seqライブラリの定量 (6時間)
注:定量は、qPCR、Tapestation、または同様の方法を使用して行うこともできます。
- まず、ライブラリーから採取した 5 μL の DNA (PCR ステップ 9.5.4) を 45 μL のヌクレアーゼフリー TE とよく混合し、次に各サンプルを 50 μL の容量で 10-10 から 10-8 希釈の範囲で 1:10 の段階希釈を行います。
- dd_PCR Master Mixストック溶液をセットアップします。プローブ用2x dd_PCRミックス 11 μL (最終濃度 1x)、プローブ oSQT1310 0.055 μL (最終濃度 250 nM)、プローブ oSQT1311 0.055 μL (最終濃度 250 nM)、プライマー oSQT1274 0.099 μL (最終濃度 450 nM)、プライマー oSQT1275 (最終濃度 450 nM)、およびヌクレアーゼフリー H2O 6.292 μL を混合します。 合計dd_PCRマスターミックスの容量は17.6μLです。すべてのサンプルのマスターミックスを調製して、正確なピペッティングに十分な容量を確保します。
- 最も低い3つの希釈液(10-6、10-7 、10-8)を二重にアッセイします(96ウェルプレート中)。非テンプレート コントロール (NTC) を使用する必要があります。
- 次のように、各試料に17.6μLのdd_PCRマスターミックスをピペットで入れます:17.6μLを4.4μLのサンプルと混合します(ヌクレアーゼフリーのH2OをNTCでウェルに加えます)、総容量は22μLです。プレートを密封し、2000 x g で室温で1分間遠心分離します。
- 液滴生成、サーモサイクル、および分析を実行します。Droplet Reader PCR システムを使用して、DG8 カートリッジ (8 ウェル) をカートリッジホルダーに移します。カートリッジのオイル列に、プローブ用の液滴発生オイル70μLを分注します。
注:ステップ10.1.2からサンプルを20μL取ります。そして、それを8ウェルカートリッジのサンプル列に追加し、カートリッジをDG8ゴムガスケットで覆い、液滴発生器に入れ、次にそれを閉じてプロセスを開始します(自動)。完了したら、カートリッジを取り外し、8ウェルカートリッジの液滴列から40 μLをセミスカート付きの96ウェルPCRプレートに移し、ゆっくりとピペットで移動させます。- ヒートブロックをPX1 PCRプレートシーラーに入れます。シーラーは、電源を入れると180°Cまで加熱を開始します。ホイルヒートシールをプレートに置き、赤い線が上にくるようにします。プレートをPX1にセットし、 シールを押します。
- 次のサーモサイクラー条件を選択してください: 酵素活性化: 95 °C で 10 分間 1 サイクル、変性: 94 °C で 30 秒間 40 サイクル、アニーリング/伸長: 60 °C で 1 分間 40 サイクル、酵素不活性化: 98 °C で 10 分間 1 サイクル、保持: 4 °C 無期限。
- 液滴リーダーで、互換性のあるソフトウェアを開き、読み取るウェルを選択します。実験タイプとして ABS を選択し、プローブ dd_PCR Supermix を選択します。[ターゲット 1] に [Ch1 不明 ] を選択し、[ターゲット 2] に [Ch2 不明 ] を選択します。 [適用] を選択し、[ OK] を選択します。プレートを液滴リーダーに入れます。染料セットの場合は、 FAM/HEX を選択し、 Runをクリックします。
- dd_PCR結果を分析します。ネガティブコントロールを参照として使用して、二重の陽性の液滴集団をゲートします。重複する値の平均を計算し、次にdd_PCRの希釈係数と5倍希釈係数を掛けます。
注:マイクロリットル当たりの総コピー数は、次のように計算される:マイクロリットル当たりの総コピー数=平均値×5×希釈係数、ここで「平均値」はCh1およびCh2からの平均定量値を表す。 - すべてのサンプルを等モル濃度で1つのライブラリに結合します。1xプールライブラリーには、約4.5 x 109 分子が含まれ、総容量は5 μLである必要があります。
11. 次世代シーケンシング
- シーケンシング用のサンプルを外部機関に提出し、正しいアダプター配列が記録されていることを確認します。
12. CIRCLE-seqデータ解析(1〜3時間)
- Pythonバージョン2.7、Burrows-Wheeler Aligner(BWA)、およびSAMtoolsをインストールします。http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz から参照ゲノム(hg38など)をダウンロードします。
注:対象生物種のゲノムが入手できない場合は、CIRCLE-seq計算ワークフローを参照に依存しないモードで実行できます。この状況では、この手順をスキップできます。 - 次のコマンドを使用して、CIRCLE-seq パイプラインをダウンロードしてインストールします: (1) git clone https://github.com/tsailabSJ/circleseq.git、(2) cd circleseq、(3) pip install -r requirements.txt。
- YAML 形式 (.yaml) でマニフェスト ファイルを作成します。以下は、CIRCLE-seqソフトウェアで提供されているサンプルデータセットと併用してワークフローをテストできるサンプルマニフェストです。
注:(1)参照ゲノム:data / input / CIRCLEseq_test_genome.fa;(2)analysis_folder:データ/出力;(3)BWA:BWA;(4)サムツールズ:サムツールズ;(5)read_threshold:<価値>;(6)window_size:<価値>;(7)mapq_threshold:<価値>;(8)start_threshold:<価値>;(9)gap_threshold:<価値>;(10)mismatch_threshold:<価値>;(11)merged_analysis:本当です。(12)サンプル:U2OS_EMX1;(13)ターゲット:GAGTCCGAGCAGAAGAANGG;(14)読み取り1:データ/入力/EMX1.r1.fastq.gz;(15)読み取り2:データ/入力/EMX1.r2.fastq.gz;(16)ControlRead1:データ/入力/EMX1_control.r1.fastq.gz;(17)controlread2:データ/入力/EMX1_control.r2.fastq.gz;(18)説明:U2OS。マニフェスト値 read_threshold: 4、window_size: 3、mapq_threshold: 50、start_threshold: 1、gap_threshold: 3、mismatch_threshold: 6 が使用されました。 - 参照ゲノム FASTA ファイル、解析用の出力ディレクトリ、 BWA コマンドと SAMtools コマンドへのパスを定義します。ヌクレアーゼ切断サンプルとコントロールサンプルの両方について、ターゲット配列と逆多重化FASTQファイルへのパスを定義します。複数の実験をバッチモードで同時に処理するには、すべての実験を 1 つのマニフェストファイルに含める必要があります。
- 標準の参照ベースの分析に対して、次のコマンドを実行します: (1) python /path/to/circleseq.py all - manifest; (2) /path/to/manifest.yaml。
- または、標準の非参照ベースの解析に対して次のコマンドを実行します: (1) Python /path/to/circleseq.py reference-free - manifest;(2) /path/to/manifest.yaml です。
- 完全なパイプラインを実行する場合は、各ステップの出力結果を、その特定のステップに指定された個別の output_folder で見つけます。
結果
ここでは、CIRCLE-seqを使用して、人工多能性幹細胞(iPSC)から単離されたDNAを使用して、アデノ随伴ウイルス統合部位1(AAVS1)を標的とするように設計されたgRNAとの複合体におけるCas9のヌクレアーゼ誘導切断部位を調査します。このgRNAは、以前に私たちの出版物27で説明されました。約25μgのgDNAをiPS細胞から単離し、焦点超音波処理により剪断し、AMPure XPビーズ精製を用いてサイズを選択したことにより、約300bpsのフラグメントを得ました。この25μgのDNAから、約2-5 ngのDNAを in vitro Cas9:gRNA切断のために正常に環状化しました。手順全体を 図 1 に示します。
CIRCLE-seqの手順と計算ワークフローを使用した解析に続いて、検出されたすべてのオンターゲットおよびオフターゲット切断部位の可視化を 図2Aに示します。また、CIRCLE-seqパイプラインは、R統計ソフトウェアを介して解析された「マージリード」を提供し、検出されたヌクレアーゼ誘発性切断部位が各染色体に沿ってマッピングされたマンハッタンプロットを生成しました(図2B)。
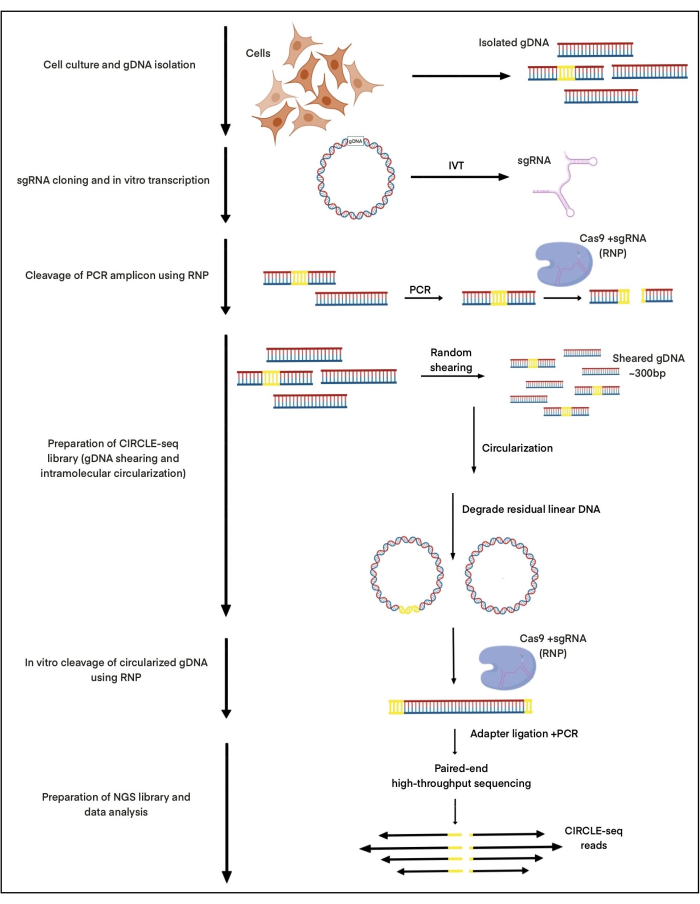
図1:CIRCLE-seqワークフローの概略図 プロトコルの主要なステップが示されています。 この図の拡大版を表示するには、ここをクリックしてください。
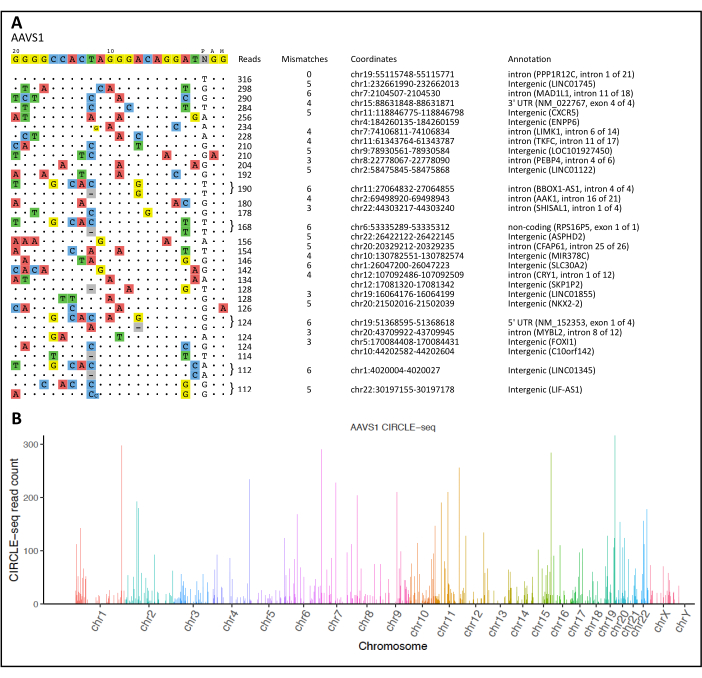
図2:CIRCLE-seqの可視化とマンハッタンプロット (A)AAVS1遺伝子座の意図したターゲットに対するオフターゲット部位のアライメント。ターゲット・シーケンスは上部に表示され、オフ・ターゲットはリード・カウントの降順でランク付けされます。元の標的配列の違いは、色付きのヌクレオチドで示されています。AAVS1遺伝子座の意図しない切断部位の上位のサンプルが示されています。(B)AAVS1遺伝子座の意図しない切断部位の検出を示すマンハッタンプロット。バーの高さは、各染色体位置の読み取りカウントを表します。 この図の拡大版を表示するには、ここをクリックしてください。
| 入門 | シーケンス (5'-3') | コメント/説明 |
| AAVS1 シングルガイドRNA(sgRNA) | GGGGCCACCAGGGACAGGAU | AAVS1遺伝子座の蛍光タンパク質ノックイン用 |
| AAVS1フォワードプライマー | GCTCTGGGCGGAGGAATATG | gRNAのin vitro切断試験用 |
| AAVS1リバースプライマー | ATTCCCAGGGCCGGTTAATG | gRNAのin vitro切断試験用 |
| オシフタ1288 | /5Phos/CGGTGGACCGATC /ideoxyU/ATCGGTCCACCGaT | CIRCLE-seqヘアピンアダプター |
| オスクワット1274 | AATGATACGGCGACCACCGAGの | TruSeq F1 |
| オスクエアT1275 | CAAGCagaagacggカタッガガット | TruSeqF2 (トゥルーシークF2) |
| オSQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | TruSeqプローブ |
| オSQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | TruSeqプローブ |
表1: AAVS1 遺伝子座のCIRCLE-seq解析に使用したgRNAとプライマーの配列。
ディスカッション
本研究では、CIRCLE-seqが、iPS細胞由来のgDNAの AAVS1 遺伝子座を標的とすることにより、ゲノム全体でヌクレアーゼ誘導性DSBを同定するための偏りのない高感度な手法であることが実証されました。iPS細胞内の AAVS1 部位は、CRISPR-Cas9を用いた外因性遺伝子の統合部位としてよく用いられるセーフハーバー遺伝子座としてよく知られている28。私たちの最近の報告では、構成的に発現したEGFPレポーターを AAVS1 部位にCRISPRで介在させることにより、EGFP標識iPS細胞の可能性を研究しました。これにより、細胞の系統全体にわたるEGFPの持続性により、iPS細胞と分化したiPS細胞の両方の標識と追跡が可能になります27。このiPS細胞株は、移植後のiPS細胞由来細胞の生体分布を評価するために in vivo で使用できます。この細胞株はCRISPR修飾されており、iPS細胞の臨床応用試験にも使用されているため、安全性と有効性を確保するためには、 AAVS1 のオフターゲット部位の可能性を既知化し、調査することが不可欠であり、CIRCLE-seqの試験に理想的な遺伝子座となっています。
以前に発表されたButterfieldらの研究27と今回の研究との顕著な違いは、AAVS1遺伝子座を標的とするために修飾されたgRNAを使用したことです。gRNAは、ゲノム編集の精度を向上させるように設計することができる24。ガイドシーケンスは、オンターゲットとオフターゲットの効率に影響を与える最も重要な要素です。したがって、選択したgRNAは、他のいくつかのガイドに対してテストされ、優れた忠実度を持つことがわかりました。さらに、ヒト線維芽細胞のリプログラミングに関する方法の記事は、修飾核酸塩基を含む合成キャップ付きmRNAが抗ウイルス応答の低活性化から利益を得るという発見を実証しました29,30。低免疫原性はin vitroアッセイには関係ないかもしれませんが、生細胞で使用できる臨床的に関連性のある治療法を開発することが最終的な目標である場合、それは重要になります。
CIRCLE-seqは、同様の方法に比べて多くの利点があります。例えば、Digenome-seqは、~4億リード21を用いて、ヌクレアーゼ切断されたgDNAと切断されていないgDNAの両方を配列決定します。その結果、バックグラウンドが高くなり、低周波の真正なカット部位をフィルタリングするのが難しくなります。CIRCLE-seqは、ヌクレアーゼ切断されたgDNAの濃縮により、~300万〜500万リードしか使用せず、バックグラウンドが低くなっています。さらに、Digenome-seqおよび同様の方法であるSITE-seqは、単一のヌクレアーゼ切断されたDNA末端のシーケンシングに依存しています。対照的に、CIRCLE-seqリードはカット部位の両端を含むため、リファレンス21,22,26を必要とせずにオフターゲット部位の同定が可能になります。
CIRCLE-seqの利点は、GUIDE-seqなどの細胞培養に依存する方法と比較して感度が高いことです。2つの方法を比較したところ、CIRCLE-seqはGUIDE-seqが検出したすべてのオフターゲット部位を捕捉し、GUIDE-seqが見逃していた意図しない切断部位をさらに発見することができました。ただし、顕著な違いは、GUIDE-seqはエピジェネティックなランドスケープによって妨げられる可能性があるのに対し、CIRCLE-seqは全ゲノムにアクセスできることです。
in vitroアッセイとして、CIRCLE-seqにはいくつかの制限があり、その最初のものは偽陽性の検出です。エピジェネティクスはin vivoの特定の部位でヌクレアーゼ活性を阻害しますが、超音波処理はin vitroでこれらの障害を取り除き、細胞の状況では通常はアクセスできない場所でのオフターゲット活性を可能にします。さらに、Cas9はこのin vitroアッセイに高濃度で存在し、他の方法ではin vivoでは不可能な切断を可能にします。また、このアッセイには比較的大量の開始gDNAが必要であり、利用可能なリソースによっては、このプロトコルの使用を無効にすることができます。最後に、現在の次世代シーケンシング技術の限界により、一部のオフターゲット部位が検出できない可能性があります。
最近の研究では、 インシリコ アプローチを使用し、そのアルゴリズムにより、CIRCLE-seqやGUIDE-seq31など、さまざまなヌクレアーゼ特性評価法を比較するためのいくつかの関連パラメータが特定されました。関連するパラメータのうちの 2 つは、「カットサイト濃縮」と「偽陽性率」でした。興味深いことに、CIRCLE-seqの偽陽性率は88%と計算されましたが、そのカットサイト濃縮は他の in vitro 法よりも高かった。すべての方法の比較解析により、GUIDE-seqは、中程度の偽陽性率32で最大のオンターゲット特異性を示したため、最高のパフォーマンスを発揮したことが明らかになりました。これはCIRCLE-seqを否定するものではなく、CIRCLE-seqとGUIDE-seqを併用する可能性を示唆しており、GUIDE-seqは前者の方が感度が高く、後者はカットサイト濃縮度の高い細胞ベースの方法であるため、GUIDE-seqでCIRCLE-seqの知見を検証することができます。データはまた、アンプリコンベースの次世代シーケンシング(NGS)が、潜在的な候補部位における真のオフターゲット修飾を同定するための好ましい方法であるべきであることを示している31。このデータは、オフターゲット効果を調べるために、CIRCLE-seq、GUIDE-seq、アンプリコンベースのNGSを使用するという潜在的な戦略を示唆しています。
開示事項
著者は何も開示していません。
謝辞
国立衛生研究所(R01AR078551およびT32AR007411)、オーストリアのジストロフィー性表皮水疱症研究協会(DEBRA)、ゲイツ・グラブステーク基金、ゲイツ・フロンティア基金からの資金援助に深く感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
参考文献
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185 (2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856 (2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786 (2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058 (2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157 (2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474 (2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745 (2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved