Method Article
종양 미세환경의 미세스케일 프로테오믹 분석을 위한 산업화된 인공 지능 유도 레이저 미세 해부
* 이 저자들은 동등하게 기여했습니다
요약
이 프로토콜은 레이저 미세 해부를 사용하여 조직학적으로 해결된 세포 집단의 농축을 위해 염색된 얇은 조직 섹션 이미지로부터 병리학적으로 확인된 관심 영역의 인공 지능 기반 세분화를 위한 고처리량 워크플로우를 설명합니다. 이 전략에는 관심 있는 세포 집단을 나타내는 경계선을 레이저 현미경으로 직접 전송할 수 있는 새로운 알고리즘이 포함되어 있습니다.
초록
종양 미세환경(TME)은 종양, 스트로마 및 면역 세포 집단을 포함한 수십 개의 별개의 세포 유형으로 구성된 복잡한 생태계를 나타낸다. 프로테옴 수준 변이와 종양 이질성을 대규모로 특성화하기 위해서는 고형 종양 악성종양에서 분리된 세포 집단을 선택적으로 분리하기 위해 높은 처리량 방법이 필요합니다. 이 프로토콜은 인공 지능 (AI)에 의해 활성화 된 고처리량 워크 플로우를 설명하며, 헤마톡실린 및 에오신 (H & E) 염색 된 얇은 조직 절편의 이미지를 레이저 미세 해부 (LMD)를 사용하여 조직학 해결 세포 집단의 선택적 수확을 위해 병리학 확인 관심 영역으로 분할합니다. 이 전략에는 디지털 이미지 소프트웨어를 사용하여 주석을 달고 관심있는 세포 집단을 나타내는 영역을 레이저 현미경으로 직접 전송할 수있는 새로운 알고리즘이 포함되어있어 더 쉬운 수집이 가능합니다. 이 워크플로우의 성공적인 구현이 수행되었으며, 고분해능 질량 분광법에 의한 정량적이고 다중화된 프로테오믹 분석을 위해 TME로부터 종양 세포 집단을 선택적으로 수확하는 이 조화된 방법의 유용성을 입증하였다. 이 전략은 일상적인 조직병리학 검토와 완전히 통합되며, 디지털 이미지 분석을 활용하여 관심있는 세포 집단의 농축을 지원하며 완전히 일반화 가능하여 다오믹 분석을 위해 TME에서 세포 집단의 조화 된 수확을 가능하게합니다.
서문
TME는 복잡한 세포외 매트릭스1과 함께 종양 세포, 기질 세포, 면역 세포, 내피 세포, 다른 중간엽 세포 유형 및 지방세포와 같은 매우 다양한 세포 유형의 어레이에 의해 채워지는 복잡한 생태계를 나타낸다. 이 세포 생태계는 서로 다른 질병 기관 부위 내외에서 다양하며, 복잡한 종양 이질성 2,3을 초래한다. 최근 연구에 따르면 이질적인 종양과 낮은 종양 세포성 (낮은 순도)을 가진 종양은 종종 가난한 질병 예후 2,3과 관련이 있습니다.
TME 내의 종양과 비종양 세포 집단 사이의 분자 상호 작용을 대규모로 이해하려면, 하류 다중종 분석을 위해 관심있는 별개의 세포 집단을 선택적으로 수확하기 위해 표준화되고 높은 처리량 전략이 필요하다. 정량적 프로테오믹스는 암 생물학에 대한 이해를 증진시키기 위해 빠르게 진화하고 점점 더 중요한 기술을 나타낸다. 현재까지, 프로테오믹스를 채용하는 연구의 우세는 전체 종양 조직 제제로부터 추출된 단백질(예를 들어, 동결분쇄)으로 이루어졌으며, 이는 TME 4,5,6에서의 프로테옴 수준 이질성에 대한 이해에 있어서 불충분성을 초래하였다.
임상 병리학 워크플로우의 정보와 원활하게 통합되고 활용되는 샘플 수집 전략의 개발은 황금 표준의 진단 병리학 워크플로우를 매우 보완하는 새로운 세대의 조직학 해결 프로테오믹스를 가능하게 할 것입니다. LMD는 조직학적으로 염색된 조직 얇은 절편(7)의 현미경 검사를 통해 세포 하위집단 또는 관심 영역(ROI)의 직접적이고 선택적인 수집을 가능하게 한다. 디지털 병리학 및 AI 활성화 분석의 최근 주요 발전은 자동화 된 방식으로 TME 내의 고유 한 구성 특징 및 ROI를 식별 할 수있는 능력을 입증했으며, 그 중 많은 부분이 분자 변경 및 임상 질병 특징, 예를 들어 치료 및 질병 예후와 같은 임상 질환 특징과 상관 관계가 있습니다8.
여기에 제시된 프로토콜에 설명된 워크플로우는 상용 소프트웨어 솔루션을 활용하여 디지털 조직병리학 이미지 내에서 종양 ROI에 선택적으로 주석을 달고, 사내에서 개발된 소프트웨어 도구를 활용하여 이러한 종양 ROI를 레이저 현미경으로 전송하여 다운스트림 다중종 분석 워크플로우와 원활하게 통합되는 관심 있는 개별 세포 집단의 자동 수집을 수행합니다. 이 통합 전략은 LMD 작업자 시간을 크게 줄이고 조직이 주변 온도에서 유지되어야 하는 기간을 최소화합니다. 자동화된 특징 선택 및 LMD 수확과 고처리량 정량적 단백체학의 통합은 두 가지 대표적인 상피 난소암 조직학적 아형, 고급 장액성 난소암(HGSOC) 및 난소 맑은 세포 암종(OCCC)으로부터 TME의 차등 분석을 통해 입증된다.
프로토콜
모든 연구 프로토콜은 미국 연방 규정 45 CFR 46.102 (f)에 따라 면제 된 것으로 간주되는 서부 IRB 승인 프로토콜 "임상 정보 바이오 마커를 확인하고 검증하기위한 자궁 내막 및 난소 암의 통합 분자 분석"에 따라 사용하도록 승인되었습니다. 이 연구에서 인간 데이터와 관련된 모든 실험 프로토콜은 헬싱키 선언에 따른 것이었다. 정보에 입각한 동의는 연구에 참여한 모든 피험자로부터 얻어졌다.
주의: 프로토콜 전반에 걸쳐 사용되는 다음 시약은 발암 물질로 알려져 있거나 의심되는 물질이며 / 또는 유해 물질을 포함합니다 : 에탄올, DEPC 물, 메이어의 헤마톡실린 용액, Eosin Y 용액, 메탄올, 아세토니트릴 및 포름산. 각 안전 데이터 시트 (SDS)에 설명 된대로 적절한 취급 및 적절한 개인 보호 장비 (PPE)의 사용은 필수입니다.
1. 교정기 신탁을 포함하는 기본 형상 목록 데이터(.sld) 파일 생성
참고: 이 섹션에서 설명하는 프로토콜 단계는 반전 레이저 현미경 및 관련 소프트웨어와 함께 사용하도록 규정되어 있습니다(재료 표 참조). 기본 .sld 파일을 만드는 것은 레이저 현미경 당 한 번만 필요합니다. 결과 파일은 그 이후에 사용되는 모든 PEN 슬라이드로 신탁을 절단하는 데 사용할 수 있습니다. 대략적인 시간 : 5 분 (한 번만 해당).
- LMD 소프트웨어를 열고 폴리에틸렌 나프탈레이트(PEN) 멤브레인 슬라이드를 LMD 스테이지에 아래로 향하게 하중시키고 라벨을 사용자에게 가장 가깝게 부착합니다. 프로그램 창의 오른쪽에 있는 닫기 줄 상자의 선택을 취소합니다.
- 고배율(63x)에서 PtoP (포인트 투 포인트) 함수를 사용하여 세 개의 "V" 화살표를 그려 교정 신탁 역할을 합니다. V의 한 외부 지점에서 시작하여 V의 중간 지점에 선을 그리고 한 번의 클릭으로 선을 그립니다. 그런 다음 V의 중심점에서 두 번째 외부 V 점의 끝까지 두 번째 선을 그리고 두 번 클릭하여 두 선에서 단일 닫히지 않은 V 모양을 만듭니다.
참고: 이러한 교정 신탁은 슬라이드의 세 모서리(오른쪽 앞, 오른쪽 후면, 왼쪽 후면)에 배치해야 합니다. - 절단하기 전에 AF(자동 초점) 옵션을 선택합니다. 슬라이드를 위치 1로 자르고 나머지 각 슬라이드 위치로 이동한 다음 보정 컷 위로 정확하게 추적합니다.
- .sld 파일을 저장하고 팝업 대화 상자에서 교정 없이 저장 옵션을 선택하여 교정 신탁이 멤브레인으로 절단되지 않도록 합니다.
참고: 네 개의 슬라이드 위치에 대한 표준 교정기 신탁을 포함하는 대표적인 .sld 파일이 보충 파일 1에 제공됩니다.
2. LMD 슬라이드 준비
참고: 이 섹션에서 설명하는 프로토콜 단계는 반전 레이저 현미경 및 관련 소프트웨어와 함께 사용하도록 규정되어 있습니다(재료 표 참조). 대략적인 시간 : 5 분.
- 기준 교정 신탁을 절단하기 전에 슬라이드가 완전히 건조되었는지 확인하십시오. LMD 소프트웨어를 열고 셰이프 가져오기 옵션에서 기본 보정 .sld 파일을 엽니다.
- 절단하기 전에 AF(자동 초점) 옵션을 선택합니다. 티슈가 아래쪽을 향하도록 슬라이드를 로드하고 레이블이 작업자에게 더 가까이 슬라이드되어 LMD 스테이지의 슬라이드 홀더에 로드합니다.
- 레이저 현미경 및 기본 교정 .sld 파일을 사용하여 교정 신탁을 PEN 멤브레인으로 자릅니다.
- 선택 사항: 교정 신탁을 PEN 멤브레인으로 잘라 조직 섹션이 슬라이드에 배치되기 전이나 후에 잘라냅니다. 조직 배치 전에 교정 신탁이 절단되는 경우, 조직이 단계 2.5에서 슬라이드에 배치될 때 조직 및/또는 고정제가 교정기와 겹치지 않는지 확인하십시오. 조직 배치 후 교정 신탁이 절단되면 단계 2.4 완료 후 중지하고 섹션 3으로 진행하십시오.
- 모든 교정기를 개별적으로 검토하여 각 컷이 완전하고 보이는지 확인합니다.
참고: 이동 및 잘라내기 기능을 사용하여 PEN 멤브레인을 완전히 절단하지 않은 교정 신탁 위에 레이저를 수동으로 지시할 수 있습니다. - 냉동 또는 포르말린-고정된, 파라핀 포매(FFPE) 조직 섹션을 교정 피두시알을 함유하는 슬라이드 상에 놓는다.
3. 조직 염색
참고: 대략적인 시간: 30분
- 동결된 LMD 조직 슬라이드를 포스파타제 억제제 칵테일 시약을 함유하는 70% 에탄올(EtOH)에 5분 동안 고정시켰다.
- 슬라이드를 포스파타제 억제제 칵테일 시약을 함유하는 디에틸 피로카르보메이트(DEPC) 물에서 1분 동안 세척한다.
- 슬라이드를 DEPC 물에서 1 분 동안 씻으십시오.
- 슬라이드를 메이어의 헤마톡실린 용액에서 3분 동안 인큐베이션한다.
- 슬라이드를 DEPC 물에 3 분 동안 헹구십시오.
- 슬라이드를 DEPC 물을 1 분 동안 신선한 교환으로 헹구십시오.
- 슬라이드를 수성 Eosin Y 용액에서 1초 동안 인큐베이션한다.
- 슬라이드를 95% EtOH로 2 x 5초 헹구십시오.
- 슬라이드를 100% EtOH에서 3 x 10초 헹구십시오.
- 슬라이드 뒷면에서 과도한 EtOH를 닦아내고 슬라이드를 공기 건조시킵니다.
- LMD가 즉시 수행되지 않도록 하는 경우 슬라이드를 -80°C에서 보관한다.
4. 슬라이드 이미징
참고: 이 섹션에서 설명하는 프로토콜 단계는 스캔한 슬라이드( 자료 표 참조)와 .svs 파일로 저장된 결과 이미지에 따라 다릅니다. 이미지 분석 소프트웨어( 자료표 참조)가 열 수 있는 형식으로 이미지 파일을 생성하는 스캐너 및 관련 소프트웨어를 사용하십시오. 지원되는 피라미드형 티프를 사용하는 파일 형식에는 JPG, TIF, MRXS, QPTIFF, 구성 요소 TIFF, SVS, AFI, SCN, LIF, DCM, OME이 포함됩니다. TIFF, ND2, VSI, NDPI, NDPIS, CZI, BIF, KFB 및 ISYNTAX. 대략적인 시간 : 5 분.
- 스캐너를 켜고 슬라이드 스캐너 소프트웨어를 엽니다. 조직이 위쪽을 향하도록 슬라이드를 스캐너의 단일 슬라이드 스테이지에 로드합니다. 슬라이드가 완전히 건조되었는지 확인하고 커버슬립을 티슈 위에 부드럽게 놓습니다. 커버슬립 아래에 에탄올이나 침지 오일을 사용하지 마십시오.
- 유리 배경 대신 PEN 멤브레인을 조정하고 제조업체의 지침에 따라 배경 멤브레인 염색을 무시하도록 보정된 설정을 사용하여 현미경 사진 이미지를 캡처합니다.
- 필요에 따라 내부 녹색 둘레를 드래그 및 크기 조정하여 이미징 영역을 조정하여 전체 PEN 멤브레인 영역을 캡처합니다. 스냅샷 개요 이미지를 두 번 클릭하여 조직에 네 개의 초점 포인트를 추가하고 교정 신탁 근처의 멤브레인에 세 개의 초점 포인트(세 개의 교정 신탁 각각당 하나의 초점 포인트)를 추가합니다.
참고: 네 개의 초점 포인트는 조직 섹션의 거의 모든 위치에 배치할 수 있지만, 너무 어둡게 얼룩져 검게 보이는 조직에 배치하면 스캔이 실패할 수 있습니다. - 보기 메뉴에서 비디오 모니터를 선택합니다. 필요에 따라 LMD 조직 주변의 각 지점에 대해 미세 및/또는 매크로 초점 슬라이더를 사용하여 초점을 수동으로 조정합니다. 이미지 스캔을 고배율(20x)로 캡처합니다. 저장된 이미지에서 모든 교정 신탁이 표시되고 명확한지 확인합니다.
5. 이미지 분석 소프트웨어를 사용한 자동 기능 선택
- 전체 종양 수집의 경우 (대략적인 시간 : 5 분, 대소 면적에 따라 다름) :
- 이미지 분석 소프트웨어를 엽니다( 자료표 참조). 이미지 열기 를 선택하고 팝업 창에서 AT2 스캐너의 슬라이드를 스캔할 때 생성된 .svs 이미지 파일을 선택합니다.
참고: 대표적인 .svs 이미지 파일은 보충 파일 2에 제공됩니다. - 주석 탭으로 이동합니다. 주석 도구 모음에서 펜 도구를 선택하고 조직 주위에 도형을 그립니다.
- 셰이프를 선택하고 이미지를 마우스 오른쪽 단추로 클릭합니다. 고급 드롭다운 메뉴에서 분할(타일링)을 선택합니다. 타일 크기와 공간을 각각 500과 40 사이로 설정하고 확인을 선택하여 타일을 생성합니다. 5.1.2단계에서 타일을 생성하는 데 사용되는 경계 셰이프를 선택하고 삭제합니다.
- 레이어 작업 드롭다운 메뉴를 선택| 바둑판식 주석을 .annotation 파일로 저장하려면 내보내기합니다.
참고: 전체 종양 조직 컬렉션에 대한 대표적인 .주석 파일은 보충 파일 3에 제공됩니다. - 세션 또는 프로젝트에 대한 폴더를 만들고 .annotation 파일을 슬라이드의 고유 식별자로 레이블이 지정된 하위 폴더에 저장합니다.
- 주석 탭으로 이동합니다. 레이어 작업 드롭다운 |를 선택합니다. 모든 레이어를 삭제하여 이미지에서 모든 주석을 제거합니다. 펜 도구를 선택하고 각 교정 신탁에 대해 화살촉의 내부 끝에서 짧은 선을 그립니다. 표시에서 선을 왼쪽 위, 오른쪽 위, 오른쪽 아래 순서로 그립니다.
- 레이어 작업 드롭다운 메뉴를 선택하| 내보내기를 통해 줄 주석을 .annotation 파일로 저장합니다. 파일 이름에 _calib을 추가하고 타일 모양의 좌표가 들어 있는 하위 폴더에 파일을 배치합니다.
참고: 대표적인 _calib.주석 파일은 보충 파일 4에 제공됩니다. - 기본 프로젝트 또는 세션 폴더의 주소를 복사합니다. IDLE 통합 개발 환경을 사용하여 XML 가져오기 생성 스크립트 "Malleator"(https://github.com/GYNCOE/Mitchell.et.al.2022 를 통해 사용 가능)를 열고 스크립트 맨 아래에 있는 따옴표 사이에 프로젝트 폴더 주소를 붙여 넣습니다.
- 실행 드롭다운 메뉴를 선택하| 모듈을 실행하여 스크립트를 실행합니다.
참고: LMD 가져오기 파일.xml은 이미지/슬라이드용으로 만든 하위 폴더 내에서 생성됩니다. 대표 .xml 파일은 보충 파일 5에 제공됩니다.
- 이미지 분석 소프트웨어를 엽니다( 자료표 참조). 이미지 열기 를 선택하고 팝업 창에서 AT2 스캐너의 슬라이드를 스캔할 때 생성된 .svs 이미지 파일을 선택합니다.
- LMD가 강화된 컬렉션의 경우에만 해당(대략적인 시간: 15분, 대/소문자를 구분함):
- 이미지 분석 소프트웨어를 엽니다( 자료표 참조). 이미지 열기 를 선택하고 팝업 창에서 슬라이드 스캔에서 생성된 .svs 이미지 파일을 선택합니다.
- 주석 탭으로 이동합니다. 사각형 주석 도구를 선택하고 사용하여 조직 주위에 상자를 그립니다.
- 상자 주석을 선택하고 이미지를 마우스 오른쪽 단추로 클릭합니다. 고급 드롭다운 메뉴를 선택하| 파티셔닝(타일링) 옵션. 타일 크기와 공간을 각각 500과 40 사이로 설정하고 확인을 선택하여 타일을 생성합니다. 5.2.2단계에서 타일을 생성하는 데 사용되는 경계 상자 주석을 선택하고 삭제합니다.
- 레이어 작업 드롭다운 메뉴를 선택하| 바둑판식 주석을 .annotation 파일로 저장하려면 내보내기합니다.
- AI 분류 주석 레이어를 병합하기 위해 개발된 Python "Dapọ"(https://github.com/GYNCOE/Mitchell.et.al.2022 를 통해 사용 가능) 알고리즘의 저장된 복사본을 타일 주석 파일과 동일한 폴더에 배치합니다.
- 타일로 묶인 주석 파일의 이름을 복사합니다. IDLE 통합 개발 환경을 사용하여 Python 프로그램을 열고 프로그램 하단의 인용문 사이에 타일 주석 파일의 이름을 붙여 넣습니다.
- 실행 드롭다운 메뉴를 선택하| 모듈 실행. 모든 타일 주석이 단일 레이어 아래에 병합되는 새 파일이 생성될 때까지 기다립니다.
- 이미지 분석 소프트웨어를 열고 주석 탭으로 이동합니다. 레이어 작업 드롭다운 메뉴를 선택하십| 모든 레이어를 삭제 하여 이미지에서 모든 주석을 제거합니다.
- 레이어 작업 드롭다운 메뉴를 선택하| 로컬 주석 파일을 가져옵니다. 팝업 창에서 스크립트에 의해 생성된 병합된 .annotation 파일을 선택합니다. 가져온 모든 타일이 동일한 주석 레이어 아래에 있는지 확인합니다.
- 분류자 탭으로 이동하여 제조업체의 지침에 따라 ROI에 대한 셰이프를 생성합니다. 분류자를 실행하기 전에 주석 탭의 고급 분류기 옵션 아래의 ROI 상자 또는 상자를 선택하여 원하는 주석 레이어(들)(즉, 종양 또는 스트로마 층)를 선택합니다. 분류자 작업 메뉴의 주석 레이어 옵션을 사용하여 분류자를 실행합니다.
- 분류자 분석이 완료되면 주석 탭으로 이동하여 분석에서 생성된 주석 레이어를 선택합니다. 레이어 작업 드롭다운 메뉴를 선택하십| 모든 레이어 그러나 현재를 삭제 하여 이미지에서 다른 모든 주석 레이어를 제거합니다.
- 레이어 작업 드롭다운 메뉴를 선택하| 주석을 .annotation 파일로 저장하도록 내보냅니다. 세션 또는 프로젝트의 폴더를 만들고 .annotation 파일을 슬라이드의 고유 식별자로 레이블이 지정된 하위 폴더에 저장합니다.
참고: 분류된 LMD 농축 조직 수집을 위한 대표적인 .주석 파일은 보충 파일 6에 제공됩니다. - 주석 탭으로 이동하여 레이어 작업 드롭다운을 선택한 | 모든 레이어를 삭제하여 이미지에서 모든 주석을 제거합니다. 펜 도구를 선택하고 각 교정 신탁에서 짧은 선을 그립니다. 표시에서 선을 왼쪽 위, 오른쪽 위, 오른쪽 아래 순서로 그립니다.
- 레이어 작업 드롭다운 메뉴를 선택하| 내보내기를 통해 줄 주석을 .annotation 파일로 저장합니다. 파일 이름에 _calib 추가하고 타일 모양의 좌표가 들어 있는 하위 폴더에 파일을 배치합니다.
- 기본 프로젝트 또는 세션 폴더의 주소를 복사합니다. IDLE 통합 개발 환경을 사용하여 XML 가져오기 생성 스크립트 "Malleator"(https://github.com/GYNCOE/Mitchell.et.al.2022 를 통해 사용 가능)를 연 다음 스크립트 하단의 따옴표 사이에 프로젝트 폴더 주소를 붙여 넣습니다.
- 실행 드롭다운 메뉴를 선택하| 모듈을 실행하여 스크립트를 실행합니다.
참고: LMD 가져오기 파일.xml은 이미지/슬라이드용으로 만든 하위 폴더 내부에 생성됩니다.
6. 레이저 미세 해부
참고: 이 섹션에서 설명하는 프로토콜 단계는 반전 레이저 현미경 및 관련 소프트웨어와 함께 사용하도록 규정되어 있습니다(재료 표 참조). 대략적인 시간: 2 시간; 대/소문자를 구분합니다.
- 조직이 아래를 향하도록 표시된 멤브레인 슬라이드(교정 신탁 포함)를 로드하고 라벨 측면이 작업자에게 더 가까이 있는 상태에서 레이저 현미경 스테이지의 슬라이드 홀더에 로드합니다.
- 파일 드롭다운 메뉴에서 셰이프 가져오기를 선택합니다. 슬라이드에 대해 생성된 LMD 가져오기 파일.xml을 선택합니다. 팝업 창에서 아니요를 선택하여 파일에서 참조 지점을 로드하지 않도록 하고 두 번째 팝업 창에서 아니요를 선택하여 보정을 위해 이전에 저장된 참조점을 사용하지 않도록 합니다.
- LMD 응용 프로그램의 프롬프트에 따라 교정 십자가를 슬라이드의 세 가지 교정 신탁 각각에 맞춥니다. 현미경 스테이지의 반전된 LMD 슬라이드의 앞쪽, 오른쪽 뒤 및 왼쪽 후면 모서리에 있는 기준점에 해당하는 이미지 분석 소프트웨어에서 슬라이드 이미지의 왼쪽 위, 오른쪽 위 및 오른쪽 아래에 나타나는 보정 신탁을 찾습니다. 5x 대물 렌즈를 사용하여 위치를 찾는 것과 63x 목표를 사용하여 각 교정 신탁을 정렬하는 것으로 전환합니다. 팝업 창에서 아니요 를 선택하여 참조 지점을 파일에 저장하지 않도록 하고 두 번째 팝업 창에서 확인을 선택하여 슬라이드가 삽입되었는지 확인합니다.
- 5x 대물 렌즈를 위치로 이동하고 팝업 창에서 예를 선택하여 실제 배율을 사용합니다. 가져온 도형이 나타나면 카메라를 티슈에 초점을 맞춥니다.
- 셰이 프 목록 창에서 모든 도형을 강조 표시하고 선택하고, 시야 내에서 하나 또는 두 개의 주석을 참조로 사용하여 제자리로 드래그한 다음, 레이저로 절단하기 위해 세로 z축을 정렬합니다.
- 가져온 도형을 검토하고 수집을 위해 적절한 튜브 위치에 할당합니다. 컷 시작 을 눌러 레이저를 시작합니다.
참고: .xml 파일에서 가져온 셰이프는 셰이프 목록 창의 "뚜껑 없음" 위치에 자동으로 할당됩니다. 조직을 수확하려면 가져온 모양을 적재 된 튜브가 포함 된 위치에 다시 할당해야합니다.
7. 압력 사이클 기술 (PCT)에 의한 단백질 소화
참고 : 대략적인 시간 : 4 시간 (진공 원심 분리기 건조 시간 없이 3 시간).
- 수확된 조직을 20 μL의 100 mM TEAB/10% 아세토니트릴에 함유하는 캡핑된 PCT 마이크로튜브를 함유하는 0.5 mL 튜브를 열순환기에 넣고 99°C에서 30분 동안 가열한 다음, 50°C로 10분 동안 냉각시킨다.
- 튜브를 4,000 × g에서 30초 동안 회전시킨 다음 0.5 mL 튜브에서 MicroTube를 분리합니다. MicroCap 도구를 사용하여 PCT 마이크로튜브에서 MicroCaps를 제거하고 버립니다. 트립신(재료 표 참조)을 30mm2 조직 당 1μg의 비율로 첨가하고 MicroCap 도구를 사용하여 MicroPestle을 MicroTube에 삽입합니다.
- MicroTubes를 바로사이클러 카트리지로 옮기고 전체 카트리지를 조립합니다. 카트리지를 바로사이클러 압력 챔버에 넣고 뚜껑을 고정시킵니다. 45,000 psi에서 50 s 동안 기압에서, 대기압에서 50 °C에서 60 사이클 동안 10 초.
- 바로사이클링이 완료되면, 마이크로튜브를 0.5 mL 마이크로원심분리 튜브로 옮기고 4,000 × g에서 2분 동안 원심분리한다.
- 캡 도구를 사용하여 0.5 mL 마이크로 원심분리 튜브에서 MicroTube를 분리합니다. 뚜껑 도구를 사용하여 페슬을 조심스럽게 제거하고 20 μL의 액체 크로마토그래피 - 질량 분광법 (LC-MS) 등급 물로 페슬의 하단 절반을 헹구고 세척을 깨끗한 0.5 mL 마이크로 원심분리 튜브에 수집하십시오.
- 벤치 상단의 MicroTube를 부드럽게 탭하여 액체를 바닥으로 옮기고 MicroTube의 모든 용액을 0.5mL 마이크로 원심분리 튜브로 옮깁니다.
- 20μL의 LC-MS 등급 물을 MicroTube에 넣고 벤치 상단에서 부드럽게 두드려보십시오. 세척 용액을 0.5 mL 튜브로 옮기고 이 세척 단계를 다시 한 번 반복한다.
- 진공 원심분리하여 샘플을 ∼2 μL로 건조시키고 100 mM TEAB, pH 8.0의 100 μL를 첨가한다.
- 제조자의 프로토콜에 따라 비색 분석법(비인치코닌산(BCA) 분석법; 물질 표 참조)을 사용하여 펩티드 농도를 결정한다.
8. 탠덤 질량 태그 (TMT) 라벨링 및 EasyPep 정리
참고 : 대략적인 시간 : 7 시간 20 분 (진공 원심 분리기 건조 시간 없이 2 시간 20 분).
- 등압 TMT 라벨링 시약을 개봉하기 전에 주변 온도로 가져 오십시오. 500 μL의 100% 아세토니트릴을 각 TMT 바이알(5 mg)에 첨가한다. 가끔 소용돌이 치면서 10 분 동안 배양하십시오.
- 5 μg의 펩티드 샘플을 100 μL의 100 mM TEAB, pH 8.0에 용해시키고, 주어진 TMT 시약 10 μL를 첨가한다. 다수의 TMT 멀티플렉스9에 걸친 샘플의 정량화를 용이하게 하기 위해 실험에서 각각의 개별 샘플을 나타내는 기준 풀을 구성하고 포함한다. 가끔씩 흔들거나 두드리면서 주변 온도에서 1 시간 동안 반응을 배양하십시오.
- 10 μL의 5% 히드록실아민을 첨가하여 TMT 라벨링 반응을 켄칭하고, 가끔 탭핑하면서 주위 온도에서 30분 동안 인큐베이션한다. 켄칭 후, TMT 표지된 샘플을 하나의 튜브에 결합하고 약 200 μL로 건조시킨다.
- 1,800 μL의 0.1% 포름산을 첨가한다. pH 종이로 pH를 확인하십시오 : pH ~ 3 인 경우 0.1 % 포름산 1 mL를 첨가하십시오. pH가 >3인 경우, pH∼3이 될 때까지 5% 포름산 10-20 μL를 첨가한다. 0.1% 포름산을 첨가하여 3 mL의 최종 부피로 만든다.
- 펩티드 클린업 컬럼 하단의 탭을 제거하고 뚜껑을 제거한 다음 15mL 원뿔형 튜브에 넣습니다. TMT 라벨이 붙은 샘플을 컬럼으로 옮기고 제조업체의 프로토콜에 따라 정리를 진행하십시오.
- 진공 원심분리하여 용출된 펩티드를 ∼20 μL로 건조시키고, 최종 부피 80 μL의 25 mM 중탄산암모늄을 사용하여 LC 바이알로 옮기고, 오프라인 분획을 진행한다.
9. TMT 멀티플렉스 샘플 분별 및 풀링
참고 : 대략적인 시간 : 3 시간 30 분 (진공 원심 분리기 건조 시간 없이 1 시간 30 분).
- 이동상 B(아세토니트릴)의 증가된 선형 구배(0.69% min-1)를 이동상 A(10 mM NH4HCO3, pH 8.0)로 현상함으로써 염기성 역상 크로마토그래피에 의해 TMT 표지된 펩티드 멀티플렉스를 96개의 분획으로 분획한다.
- 샘플 웰을 풀링하여 36개의 연결된 분획을 생성합니다. 분획을 ∼2 μL로 건조시키기 위해 진공 원심분리하고, 25 mM 중탄산암모늄 (최종 농도 1.5 μg/10 μL)에 재현탁시키고, 15,000 × g 에서 10-15분 동안 원심분리하고, MS 분석을 위해 LC 바이알로 옮긴다.
10. 액체 크로마토그래피 탠덤 질량 분광법 (LC-MS/MS)
참고 : 대략적인 시간 : 계측기 방법 및 실험 설계에 따라 다릅니다.
- 제조업체의 지침/프로토콜에 따라 질량 분광계를 교정합니다.
- 새로운 이동상 및 표준을 준비하고 적절한 LC 사전 실행 준비를 수행합니다(참조된 계측기에 대한 용매 제거, 플러시 공기 및 누출 테스트 스크립트를 포함하되 이에 국한되지 않음[ 재료 표 참조]). 분석을 시작하기 전에 사전 및 분석 컬럼과 샘플 루프를 평형화합니다.
- 직렬 TMT 멀티플렉스 분석 전과 그 사이에 LC-MS 시스템이 품질 보증/품질 관리(QA/QC) TMT 표지된 펩티드 다이제스트 및 (예: MSPE( 재료 표 참조), HeLa)를 사용하여 이전에 벤치마킹된 성능 메트릭을 충족하는지 검증합니다.
- LC 자동 시료 주입기의 적절한 위치에 자동 시료 주입기 바이알을 로드합니다. 적절한 그라디언트/MS 방법으로 개별 분수를 분석합니다. 크로마토그래피 및 질량 스펙트럼 성능을 평가하기 위해 대략 하루에 한 번 펩티드 표준(예를 들어, 펩티드 체류 시간 교정[PRTC])과 함께 "세척" 실행을 인터스퍼스한다. 각 TMT 멀티플렉스 샘플 분획 시리즈를 분석한 후 QA/QC TMT 벤치마크 표준을 실행하여 시스템 성능을 평가합니다.
- QA/QC TMT 벤치마크 표준 이후에 질량 분광계 평가 루틴을 실행하여 사후 샘플 성능을 평가한 다음 다음 샘플 세트를 위해 10.1단계와 같이 시스템을 교정합니다.
11. 생물정보자료 분석
참고: 대략적인 시간: 실험 설계에 따라 다릅니다.
- 모든 샘플 데이터(예: .raw 파일)를 적절한 네트워크 스토리지/컴퓨터 드라이브로 전송합니다.
- 펩티드 스펙트럼 매칭 (PSMs) 및 추출 TMT 리포터 이온 신호 강도를 생성하기 위해 종 특이적 단백질 참조 데이터베이스에 대해 적절한 파라미터9를 사용하여 원하는 데이터 분석 애플리케이션 (예를 들어, 프로테옴 디스커버, 마스코트)을 사용하여 모든 분획을 함께 검색한다. 적절한 품질 관리 메트릭에 기초하여 PSM을 필터링하고, 앞서 기술한 바와 같이 정규화된, 중앙값 로그2-형질전환된 TMT 리포터 이온 비 풍부도를 글로벌 단백질 수준 풍부도로 응집한다 3,9.
- 원하는 차등 분석 소프트웨어를 사용하여 관심 조건의 단백질 변화를 비교하십시오.
결과
두 HGSOC 및 두 OCCC 환자로부터의 신선-동결 조직 얇은 절편을 이 통합된 AI 구동 조직 ROI 식별, 세분화, LMD 및 정량적 프로테오믹 분석 워크플로우를 사용하여 분석하였다(그림 1). 각 종양에 대한 대표적인 H&E 염색된 조직 절편은 보드 인증 병리학자에 의해 검토되었다; 종양 세포성은 70% 내지 99% 범위였다. Tissues는 PEN 멤브레인 슬라이드(보충 파일 2)에 얇게 단면화되고 교정기 신탁(보충 파일 1)으로 프리컷되어 이미지 분석 소프트웨어에서 생성된 주석의 위치 방향 데이터( 재료 표 참조)를 LMD 소프트웨어의 데카르트 좌표 방향과 통합할 수 있습니다. H&E 염색 후, 조직과 교정기가 포함된 PEN 슬라이드의 고해상도 이미지(20x)가 캡처되었습니다.
현미경 사진에서 종양 및 기질 세포 집단은 LMD에 의한 선택적 수확을 위해 이미지 분석 소프트웨어 (물질 표 참조)를 사용하여 분절되었고, 전체 조직 얇은 단면 (예를 들어, 전체 종양 조직)을 나타내는 수확과 함께 (도 1). 전체 종양 조직 수집에 대한 비차별적 주석은 전체 조직 섹션을 500μm2의 타일로 분할하여 생성되었으며, 타일 사이에 40μm 간격을 남겨 PEN 멤브레인 무결성을 유지하고 LMD 동안 멤브레인이 컬링되는 것을 방지합니다. 조직학 해결 LMD 농축을 위한 슬라이드에서, 이미지 분석 소프트웨어(자료 표 참조)의 AI 분류자는 빈 유리 슬라이드 배경과 함께 종양과 기질 세포를 구별하도록 훈련되었다. 대표적인 종양, 스트로마 및 빈 유리 영역이 수동으로 강조 표시되었고, 분류기 도구를 사용하여 전체 조직 섹션에 걸쳐 이러한 ROI를 세그먼트화했습니다. 전체 조직, 종양 상피 및 스트로마를 나타내는 세그먼트 된 층은 개별 .annotation 파일 (보충 파일 3 및 보충 파일 6)으로 별도로 저장되었습니다. 분할된 ROI 주석이 없는 이미지 파일의 별도 복사본에서는 세 개의 신탁 보정기 각각의 맨 가운데 끝에서 짧은 줄에 주석을 달고 각 LMD 주석 레이어 파일과 동일한 파일 이름을 사용하지만 접미사 "_calib"(보충 파일 4)가 추가된 .annotation 파일로 저장했습니다. 이 선들은 이미지 분석 소프트웨어에 그려진 주석 형상 목록 데이터와 함께 PEN 멤브레인 교정기의 위치를 공동 등록하는 데 사용되었습니다.
본 연구는 파이썬에서 "Malleator"와 "Dapọ"라는 두 가지 알고리즘을 제공하여 https://github.com/GYNCOE/Mitchell.et.al.2022 에서 사용할 수있는이 AI 기반 LMD 워크 플로우를 지원합니다. Malleator 알고리즘은 쌍을 이룬 .annotation 파일에서 모든 개별 주석(조직 ROI 및 교정기)에 대한 특정 데카르트 좌표를 추출하여 단일 확장 마크업 언어(XML) 가져오기 파일(보충 파일 5)로 병합합니다. 특히 Malleator 알고리즘은 상위 폴더의 디렉토리 이름을 입력으로 사용하여 모든 하위 디렉토리 폴더를 검색하고 .xml 병합 파일이 아직 없는 하위 폴더에 대해 .xml 파일을 생성합니다. Malleator 알고리즘은 이미지 분석 소프트웨어( 재료 표 참조)의 모든 주석 레이어를 단일 레이어로 병합하고 독점 .주석 파일 유형으로 저장되는 AI 생성 형상 목록 데이터를 LMD 소프트웨어와 호환되는 .xml 형식으로 변환합니다. 주석 및 교정기 파일을 병합한 후 알고리즘에서 생성된 .xml 파일이 저장되고 LMD 소프트웨어로 가져옵니다. 주석의 정렬을 수동으로 조정하려면 약간의 조정이 필요하며, 이는 또한 레이저 현미경 상에 슬라이드 스테이지의 수직(z-plane) 위치를 등록하는 역할을 한다. Dapọ 알고리즘은 LMD가 풍부한 컬렉션에 특별히 사용됩니다. 분할된 타일은 이미지 분석 소프트웨어에 의해 개별 주석 레이어에 자동으로 할당됩니다. Dapọ 알고리즘은 분류자 도구를 사용하기 전에 분할된 모든 타일을 단일 주석 계층으로 병합하여 LMD 보강 컬렉션의 분류자 분석 런타임을 줄입니다.
전체 종양 및 LMD-농축된 조직 샘플을 소화시키고, TMT 시약으로 표지하고, 다중화하고, 오프라인으로 분획하고, 앞서 기술된바와 같이 정량적 MS-기반 프로테오믹스를 통해 분석하였다. 이 AI 기반 워크플로우를 사용하여 수확한 샘플에 대한 평균 펩티드 수율(43-60 μg) 및 회수율(0.46-0.59 μg/mm2)은 이전 보고서 9,10과 비교되었습니다. 총 5,971개의 단백질을 모든 샘플에 걸쳐 공동-정량하였다(보충 표 S1). 100개의 가장 가변적인 단백질을 사용한 무감독 계층적 클러스터링은 LMD 농축 및 전체 종양 샘플로부터 HGSOC 및 OCCC 조직형의 분리를 초래하였으며(도 2A), 앞서 기술된11과 유사하다. 대조적으로, HGSOC 및 OCCC 둘 다로부터의 LMD 농축 스트로마 샘플은 LMD 농축 종양 및 전체 종양 샘플로부터 함께 그리고 독립적으로 클러스터링되었다. 5,971개의 정량화된 단백질 중에서, HGSOC와 OCCC 표본으로부터의 전체 종양 수집물 사이에서 215개가 유의하게 변경되었다(LIMMA adj. p < 0.05). 이들 변경된 단백질은 Hughes et al.11에 의해 HGSOC 및 OCCC 종양 조직을 분화시키는 것으로 확인된 단백질과 비교되었다. Hughes et al.에 의해 정량화된 76개의 시그니처 단백질 중에서, 57개는 이 데이터세트에서 공동-정량화되었고, 고도로 상관관계가 있었다(Spearman Rho = 0.644, p< 0.001)(도 2B).
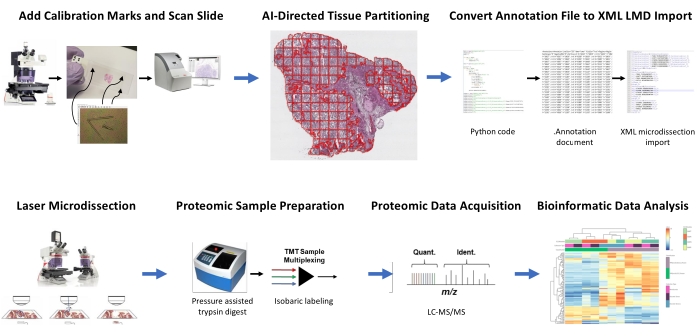
그림 1: 다운스트림 정량적 프로테오믹스를 위한 레이저 미세 해부를 위한 관심 조직 선택 자동화를 위한 통합 워크플로우 요약. 캘리브레이션 신탁은 PEN 멤브레인 슬라이드로 절단되어 LMD 현미경에 수평 위치와 함께 이미지 분석 소프트웨어인 HALO에서 AI 파생 조직 ROI 세그먼트의 위치 방향 데이터를 공동 등록합니다. Malleator 알고리즘은 슬라이드에 대한 모든 주석 레이어에서 주석이 달린 세그멘테이션 데이터를 _calib 참조 파일과 병합하고 LMD 소프트웨어와 호환되는 .xml 파일로 변환하는 데 사용됩니다. 프로테오믹 분석을 위해 LMD-수확된 조직은 앞서 기술된 바와 같이 고처리량 정량적 프로테오믹스에 의해 소화되고 분석된다(9). 약어: LMD = 레이저 미세 해부; ROI = 관심 지역; TMT = 탠덤 질량 태그; 수량. = 정량화; 식별. = 신원 확인; LC-MS/MS = 액체 크로마토그래피-탠덤 질량 분광법. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
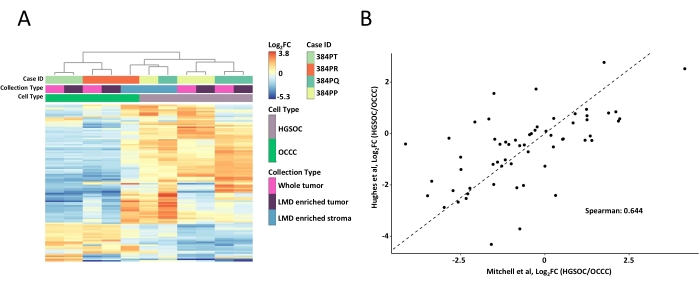
도 2: LMD 농축 및 전체 종양 샘플에서의 단백질 분석. (A) HGSOC 및 OCCC LMD 농축 및 전체 종양 샘플에서 가장 가변적으로 풍부한 100개의 단백질에 대한 무감독 계층적 클러스터 분석. (b) 본 연구에서 HGSOC와 OCCC 전체 종양 수확 사이의 log2 배 변화 단백질 풍부도의 상관관계 (Mitchell et al., x-axis) 및 Hughes et al. (y-axis)11에 의한 유사한 연구. 약어: LMD = 레이저 미세 해부; HGSOC = 고급 장액성 난소암; OCCC = 난소 맑은 세포 암종; log2 FC = log2-형질전환된 프로테오믹 풍부도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 표 S1: HGSOC 및 OCCC 조직 표본으로부터의 모든 LMD 농축 및 전체 종양 샘플에 걸쳐 공동-정량화된 5,971개의 단백질의 풍부함. 약어: LMD = 레이저 미세 해부; HGSOC = 고급 장액성 난소암; OCCC = 난소 맑은 세포 암종. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
보충 표 S2: HGSOC 대 OCCC(LIMMA adj. p < 0.05)로부터의 전체 종양 수집물에서 차등적으로 발현된 단백질(215). 약어: HGSOC = 고급 장액성 난소암; OCCC = 난소 맑은 세포 암종. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
보충 파일 1: 네 개의 슬라이드 위치에 대한 표준 교정기 신탁을 포함하는 대표적인 모양 목록 데이터(.sld) 파일입니다. 이 파일은 LMD 소프트웨어로 가져올 수 있습니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 2 : H & E 염색 된 고해상도 (20x) 조직 섹션의 대표적인 .svs 이미지 파일. 이미지 분석 소프트웨어 또는 LMD 소프트웨어를 사용하여 파일을 열고 볼 수 있습니다. 약어: H&E = 헤마톡실린 및 에오신; LMD = 레이저 미세 해부. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 3 : 분할 된 전체 종양 세그먼트의 대표적인 .annotation 파일. 파일을 이미지 분석 소프트웨어로 가져올 수 있습니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 4 : 교정기 신탁 세그먼트의 대표 _calib.annotation 파일. 좌표 정보는 각 화살촉 신탁으로부터 그려진 짧은 교정기 선의 동양 위치를 나타냅니다. 파일을 이미지 분석 소프트웨어로 가져올 수 있습니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 5: Malleator 알고리즘에 의해 생성된 대표적인 확장 가능한 마크업 언어(.xml) 파일입니다. 이 파일은 레이저 미세 해부 소프트웨어로 가져올 수 있습니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 6: LMD가 풍부한 컬렉션에 대해 분할된 AI 분류 세그먼트의 대표적인 .주석 파일입니다. 파일을 이미지 분석 소프트웨어로 가져올 수 있습니다. 약어: AI = 인공 지능; LMD = 레이저 미세 해부. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
FFPE 및/또는 신선 냉동 조직으로부터 표적 세포 하위집단의 농축을 위한 워크플로우를 개발 및/또는 개선하려는 여러 연구 선례가 있었지만,9,12,13,14,15를 처리하는 동안 샘플 품질을 유지하기 위한 방법론이 있지만, 가변성을 감소시키기 위해 분자 분석을 위한 임상 조직 표본을 제조하기 위한 자동화된 전략을 개발해야 할 상당한 필요성이 존재하며, 재현성을 높입니다. 이 워크플로우는 임상 조직 표본에서 LMD에 의한 이산 세포 집단의 조직학적으로 해결된 수확을 위해 기존의 이미지 분석 소프트웨어 도구(자료 표 참조)를 통합하는 표준화된 반자동화 프로토콜을 설명합니다.
분리된 세포 집단을 포획하는 ROI의 공간적으로 해결된 LMD 농축은 분자 특성화 및 식별을 개선하고 세포 선택적 바이오마커 발견을 촉진하기 위해 다중종 분석 이전의 차세대 조직 처리 단계를 나타낸다. 이 프로토콜은 조직 학자에 의한 ROI의 수동 세분화와 관련된 주변 환경에 대한 조직 절편의 종종 긴 노출을 줄임으로써 기존 방법론을 개선합니다 (LMD 수집 전에 >1-2 시간이 걸릴 수 있음). 대신 이 워크플로우를 통해 AI 기반 분류 및 세분화를 통해 ROI를 미리 식별할 수 있습니다. 조직 체류 시간을 제한하면 포스포펩타이드 및 mRNA와 같은 매우 불안정한 분자 표적의 평가에서 또는 검출을 위해 그것의 본래 입체 형태에 있는 표적 단백질에 의존하는 항체 기반 분석 기술에 대한 스퓨리어스 변이를 감소시킬 것이다.
스캔한 슬라이드 이미지에서 명확하게 볼 수 있는 PEN 멤브레인 슬라이드에 깔끔한 교정기 신탁을 절단하는 것은 LMD 워크플로우와 이미지 분석 소프트웨어( 자료 표 참조)를 통합할 수 있는 핵심 구성 요소 중 하나입니다. 교정기가 "V" 형상의 하단에 정밀한("깨끗한" 점)을 갖도록 보장하면 단계 5.1.6 및 5.2.13에 설명된 대로 교정기 선에서 그려질 이미지 분석 소프트웨어에서 정확한 점을 선택할 수 있습니다. LMD 소프트웨어로 가져오는 동안 이러한 지점의 정렬은 주석("Malleator" 및/또는 "Dapọ" 알고리즘을 사용하여 호환 가능한 .xml 파일 생성을 통해 촉진됨)을 물리적 LMD 슬라이드의 관련 조직 ROI에 적절하게 오버레이하는 데 중요합니다. LMD 소프트웨어로 가져올 때 정렬이 정확할 때에도 모든 모양을 강조 표시하고 공동으로 "드래그 앤 드롭"하여 레이저 현미경에 슬라이드 스테이지의 수직 (z-plane) 위치를 등록해야합니다. 필요한 경우 조직 ROI에 대한 주석의 위치를 약간 조정할 수도 있습니다.
Malleator 알고리즘의 현재 버전의 한계는 이미지 분석 소프트웨어에서 제공하는 사전 정의 된 주석 모양 도구 ( 자료 표 참조)와 호환되지 않는다는 것입니다.하지만 알고리즘의 향후 업데이트 / 버전은이 호환성을 향상시키는 것을 목표로합니다. 이러한 도구를 사용하여 그려진 셰이프의 .annotation 파일에는 각 주석에 대해 쌍을 이룬 x 및 y 좌표 집합이 두 개만 포함되어 있으며 이러한 점 주위의 전체 공간 방향은 포함되지 않습니다. 현재 이러한 도구를 사용하면 주석이 가져오기 프로세스 중에 두 점만 정의된 직선으로 변환됩니다. 조직 ROI 세그먼트의 수동 정의는 XML 형식 및 LMD 가져오기로의 성공적인 변환을 위해 필요합니다. 이는 대상 영역에 특정한 개별 자유형 다각형 주석으로 각 ROI를 수동으로 정의하거나 원하는 경우 모든 조직 ROI 세그먼트에 대략적인 원형 또는 직사각형 주석을 적용하여 수행할 수 있으며 이 워크플로와 호환됩니다.
여기에 제시된 워크 플로우는 신선하게 얼어 붙은 인간 암 조직 표본의 프로테오믹 분석을 위해 입증되었지만,이 AI 기반 LMD 워크 플로우는 FFPE 조직, 비 암성 조직 유형 및 비인간 소스의 것과 동등하게 사용될 수 있습니다. 또한 전사체, 게놈 또는 포스포프로테오믹 분석을 포함한 다른 다운스트림 분자 프로파일링 워크플로우를 지원할 수 있습니다. 이 워크플로우는 또한 "Multiplex IHC" 모듈 또는 "Tissue Microarray (TMA) Add-on"을 포함한 세포 계수 또는 기타 분석 모듈과 관련된 기능을 포함하여 이미지 분석 소프트웨어( 자료 표 참조)의 다른 용도를 활용할 수 있습니다. 이 워크플로우의 향후 적용은 ROI 세그먼트당 세포 수를 미리 정의하여 여러 컬렉션에 걸쳐 동등한 세포 입력을 보장하거나 면역조직화학 또는 세포 사회학과 같이 관심있는 세포 ROI를 정의하기 위한 대체 방법을 사용하여 이점을 얻을 수 있습니다.
공개
T.P.C.는 ThermoFisher Scientific, Inc SAB 회원이며 AbbVie로부터 연구 자금을 받습니다.
감사의 말
이 프로젝트에 대한 기금은 국방 건강 프로그램 (HU0001-16-2-0006 및 HU0001-16-2-00014)에 의해 부인과 암 센터 우수성을위한 Uniformed Services University에 부분적으로 제공되었습니다. 스폰서는 연구의 설계, 실행, 해석 또는 작성에 아무런 역할을하지 못했습니다. 면책 조항: 여기에 표현 된 견해는 저자의 견해이며 육군 / 해군 / 공군, 국방부 또는 미국 정부의 공식 정책을 반영하지 않습니다.
자료
| Name | Company | Catalog Number | Comments |
| 1260 Infinity II System | Agilent Technologies Inc | Offline LC system | |
| 96 MicroCaps (150uL) in bulk | Pressure Biosciences Inc | MC150-96 | |
| 96 MicroPestles in bulk | Pressure Biosciences Inc | MP-96 | |
| 96 MicroTubes in bulk (no caps) | Pressure Biosciences Inc | MT-96 | |
| 9mm MS Certified Clear Screw Thread Kits | Fisher Scientific | 03-060-058 | Sample vial for offline LC frationation and mass spectrometry |
| Acetonitrile, Optima LC/MS Grade | Fisher Chemical | A995-4 | Mobile phase solvent |
| Aperio AT2 | Leica Microsystems | 23AT2100 | Slide scanner |
| Axygen PCR Tubes with 0.5 mL Flat Cap | Fisher Scientific | 14-222-292 | Sample tubes; size fits PCT tubes and thermocycler |
| Barocycler 2320EXT | Pressure Biosciences Inc | 2320-EXT | Barocycler |
| BCA Protein Assay Kit | Fisher Scientific | P123225 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Easy-nLC 1200 | Thermo Fisher Scientific | Liquid Chromatography | |
| EasyPep Maxi Sample Prep Kit | Thermo Fisher Scientific | NCI5734 | Post-label sample clean up column |
| EASY-SPRAY C18 2UM 50CM X 75 | Fisher Scientific | ES903 | Analytical column |
| Eosin Y Solution Aqueous | Sigma Aldrich | HT110216 | |
| Formic Acid, 99+ % | Thermo Fisher Scientific | 28905 | Mobile phase additive |
| ggplot2 version 3.3.5 | CRAN | https://cran.r-project.org/web/packages/ggplot2/ | |
| HALO | Indica Labs | Image analysis software | |
| IDLE (Integrated Development and Learning Environment) | Python Software Foundation | ||
| iheatmapr version 0.5.1 | CRAN | https://cran.r-project.org/web/packages/iheatmapr/ | |
| iRT Kit | Biognosys | Ki-3002-1 | LC-MS QAQC Standard |
| limma version 3.42.2 | Bioconductor | https://bioconductor.org/packages/release/bioc/html/limma.html | |
| LMD Scanning stage Ultra LMT350 | Leica Microsystems | 11888453 | LMD stage model outfitted with PCT tube holder |
| LMD7 (software version 8.2.3.7603) | Leica Microsystems | LMD apparatus (microscope, laser, camera, PC, tablet) | |
| Mascot Server | Matrix Science | Data analysis software | |
| Mass Spec-Compatible Human Protein Extract, Digest | Promega | V6951 | LC-MS QAQC Standard |
| Mayer’s Hematoxylin Solution | Sigma Aldrich | MHS32 | |
| PEN Membrane Glass Slides | Leica Microsystems | 11532918 | |
| Peptide Retention Time Calibration Mixture | Thermo Fisher Scientific | 88321 | LC-MS QAQC Standard |
| Phosphatase Inhibitor Cocktail 2 | Sigma Aldrich | P5726 | |
| Phosphatase Inhibitor Cocktail 3 | Sigma Aldrich | P0044 | |
| Pierce LTQ Velos ESI Positive Ion Calibration Solution | Thermo Fisher Scientific | 88323 | Instrument calibration solution |
| PM100 C18 3UM 75UMX20MM NV 2PK | Fisher Scientific | 164535 | Pre-column |
| Proteome Discoverer | Thermo Fisher Scientific | OPTON-31040 | Data analysis software |
| Python | Python Software Foundation | ||
| Q Exactive HF-X | Thermo Fisher Scientific | Mass spectrometer | |
| R version 3.6.0 | CRAN | https://cran-archive.r-project.org/bin/windows/base/old/2.6.2/ | |
| RColorBrewer version 1.1-2 | CRAN | https://cran.r-project.org/web/packages/RColorBrewer/ | |
| Soluble Smart Digest Kit | Thermo Fisher Scientific | 3251711 | Digestion reagent |
| TMTpro 16plex Label Reagent Set | Thermo Fisher Scientific | A44520 | isobaric TMT labeling reagents |
| Veriti 60 well thermal cycler | Applied Biosystems | 4384638 | Thermocycler |
| Water, Optima LC/MS Grade | Fisher Chemical | W6-4 | Mobile phase solvent |
| ZORBAX Extend 300 C18, 2.1 x 12.5 mm, 5 µm, guard cartridge (ZGC) | Agilent Technologies Inc | 821125-932 | Offline LC trap column |
| ZORBAX Extend 300 C18, 2.1 x 150 mm, 3.5 µm | Agilent Technologies Inc | 763750-902 | Offline LC analytical column |
참고문헌
- Motohara, T., et al. An evolving story of the metastatic voyage of ovarian cancer cells: cellular and molecular orchestration of the adipose-rich metastatic microenvironment. Oncogene. 38 (16), 2885-2898 (2019).
- Aran, D., Sirota, M., Butte, A. J. Systematic pan-cancer analysis of tumour purity. Nature Communications. 6, 8971 (2015).
- Hunt, A. L., et al. Extensive three-dimensional intratumor proteomic heterogeneity revealed by multiregion sampling in high-grade serous ovarian tumor specimens. iScience. 24 (7), 102757 (2021).
- Dou, Y., et al. Proteogenomic characterization of endometrial carcinoma. Cell. 180 (4), 729-748 (2020).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Gillette, M. A., et al. Proteogenomic characterization reveals therapeutic vulnerabilities in lung adenocarcinoma. Cell. 182 (1), 200-225 (2020).
- Silvestri, A., et al. Protein pathway biomarker analysis of human cancer reveals requirement for upfront cellular-enrichment processing. Laboratory Investigation. 90 (5), 787-796 (2010).
- Echle, A., et al. Deep learning in cancer pathology: a new generation of clinical biomarkers. British Journal of Cancer. 124 (4), 686-696 (2021).
- Lee, S., et al. Molecular analysis of clinically defined subsets of high-grade serous ovarian cancer. Cell Reports. 31 (2), 107502 (2020).
- Xuan, Y., et al. Standardization and harmonization of distributed multi-center proteotype analysis supporting precision medicine studies. Nature Communications. 11 (1), 5248 (2020).
- Hughes, C. S., et al. Quantitative profiling of single formalin fixed tumour sections: proteomics for translational research. Scientific Reports. 6 (1), 34949 (2016).
- Espina, V., et al. A portrait of tissue phosphoprotein stability in the clinical tissue procurement process. Molecular & Cellular Proteomics. 7 (10), 1998-2018 (2008).
- Espina, V., Heiby, M., Pierobon, M., Liotta, L. A. Laser capture microdissection technology. Expert Review of Molecular Diagnostics. 7 (5), 647-657 (2007).
- Havnar, C. A., et al. Automated dissection protocol for tumor enrichment in low tumor content tissues. Journal of Visualized Experiments. (169), e62394 (2021).
- Mueller, C., et al. One-step preservation of phosphoproteins and tissue morphology at room temperature for diagnostic and research specimens. PLoS One. 6 (8), (2011).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유