나노포어 기술을 사용한 광견병 바이러스의 신속한 특성 분석을 위한 전체 게놈 염기서열 분석
요약
여기에서는 나노포어 기술을 사용하여 광견병 바이러스(RABV) 게놈을 특성화하기 위한 빠르고 비용 효율적인 워크플로우를 제시합니다. 이 워크플로우는 지역 수준에서 유전체학 정보 감시를 지원하기 위한 것으로, 광견병 통제 조치를 안내하기 위해 순환하는 RABV 계통 및 지역 계통 발생 내 배치에 대한 정보를 제공합니다.
초록
게놈 데이터는 전염병의 전염 및 지리적 확산을 추적하는 데 사용할 수 있습니다. 그러나 개 매개 광견병 및/또는 흡혈박쥐와 같은 야생 동물에 의해 전염되는 광견병이 주요 공중 보건 및 경제적 문제를 제기하는 많은 저소득 및 중간 소득 국가(LMIC)에서는 게놈 감시에 필요한 염기서열 분석 능력이 여전히 제한적입니다. 여기서는 나노기공 기술을 사용하여 빠르고 저렴한 샘플-시퀀스-해석 워크플로우를 제시합니다. 시료 채취 및 광견병 진단을 위한 프로토콜에 대해 간략하게 설명한 후 다중 중합효소 연쇄 반응(PCR)을 위한 프라이머 설계 및 최적화, 수정된 저비용 염기서열 분석 라이브러리 준비, 실시간 및 오프라인 염기 서열 분석을 통한 염기서열 분석, 유전자 계통 지정 및 계통 발생 분석을 포함한 최적화된 전체 게놈 염기서열 분석 워크플로우에 대한 세부 정보가 뒤따릅니다. 워크플로우의 구현이 시연되며, 파이프라인 검증, 프라이머 최적화, 음성 대조군 포함, 지역 및 글로벌 계통 발생 내 분류 및 배치를 위한 공개적으로 사용 가능한 데이터 및 게놈 도구(GLUE, MADDOG) 사용과 같은 로컬 배포를 위한 중요한 단계가 강조됩니다. 워크플로우의 소요 시간은 2-3일이며 비용은 96개 샘플 실행의 경우 샘플당 $25에서 12개 샘플 실행의 경우 샘플당 $80입니다. 우리는 중저소득국에서 광견병 바이러스 게놈 감시를 설정하는 것이 실현 가능하며, 2030년까지 개 매개 인간 광견병 사망자 제로라는 글로벌 목표를 향한 진전을 지원할 수 있을 뿐만 아니라 야생 광견병 확산에 대한 모니터링을 강화할 수 있다고 결론지었습니다. 또한 이 플랫폼은 다른 병원체에 적용할 수 있어 전염병 및 팬데믹 대비에 기여하는 다재다능한 게놈 역량을 구축하는 데 도움이 됩니다.
서문
광견병 바이러스(RABV)는 포유류에 치명적인 신경 질환을 일으키는 Rhabdoviridae 과에 속하는 리사바이러스입니다1. 광견병은 백신 접종으로 100% 예방할 수 있지만, 풍토병 국가에서는 여전히 주요 공중 보건 및 경제적 문제입니다. 매년 발생하는 것으로 추정되는 60,000명의 인간 광견병 사망자 중 95% 이상이 개가 주요 저장소인 아프리카와 아시아에서 발생합니다2. 이와는 대조적으로, 개 예방 접종은 서유럽, 북미 및 라틴 아메리카 대부분의 지역에서 개 매개 광견병을 퇴치하는 데 기여했습니다. 이 지역에서 광견병 저수지는 현재 박쥐, 너구리, 스컹크, 야생 갯과 동물과 같은 야생동물로 제한되어 있다3. 라틴 아메리카 전역에서 흔히 볼 수 있는 흡혈박쥐는 밤마다 피를 먹는 동안 박쥐로부터 인간과 가축에게 정기적으로 전염되기 때문에 광견병의 문제가 되는 원인이 된다4. 광견병이 연간 세계 경제에 미치는 영향은 86억 달러로 추산되며, 가축 손실은 6%를 차지합니다5.
바이러스 병원체의 염기서열 데이터와 감염 시기 및 출처에 대한 메타데이터를 결합하면 강력한 역학적 통찰력을 제공할 수 있다6. RABV의 경우, 염기서열 분석은 발병의 기원을 조사하고7,8 야생동물또는 개와의 숙주 연관성을 식별하고8,9,10,11,12 인간 사례의 원인을 추적하기 위해 사용 되었다 13,14. 계통발생학적 분석을 이용한 발병 조사에 따르면 광견병은 이전에 광견병이 없던 인도네시아 발리 지방에서 칼리만탄이나 술라웨시의 인근 풍토병 지역에서 한 번의 유입을 통해 출현한 것으로 나타났다15. 한편 필리핀에서는 롬블론주 타블라스섬에서 발생한 발병이루손섬 본섬에서 유입된 것으로 확인됐다. 바이러스 게놈 데이터는 지리적으로 표적 제어 조치에 필요한 병원체 전파 역학을 더 잘 이해하는 데에도 사용되었습니다. 예를 들어, RABV의 게놈 특성분석은 군집(clade)17,18,19, 계통(20,21,22)의 공동 순환, 인간 매개 바이러스 이동(human-mediated viral movement)17,23,24 및 메타개체군 동태(metapopulation dynamics)25,26)의 지리적 군집을 보여준다.
질병 모니터링은 SARS-CoV-2 팬데믹에 대응하여 전 세계적으로 염기서열 분석 능력이 증가함에 따라 강화된 게놈 감시의 중요한 기능 중 하나입니다. 게놈 감시는 우려되는 SARS-COV-2 변종의 실시간 추적을 지원했다27,28 및 관련 대책6. 나노포어 기술과 같은 접근 가능한 시퀀싱 기술의 발전은 인간 29,30,31,32와 동물 33,34,35 병원체의 신속한 시퀀싱을 위한 개선되고 더 저렴한 프로토콜로 이어졌습니다. 그러나 많은 광견병 풍토병 국가에서는 SARS-CoV-2 염기서열 분석 능력의 글로벌 격차에서 알 수 있듯이 병원체 게놈 감시를 운영하는 데 여전히 장벽이 있습니다36. 실험실 인프라, 공급망 및 기술 지식의 한계로 인해 게놈 감시의 구축 및 루틴화가 어려워지고 있습니다. 이 백서에서는 리소스가 제한된 환경에서 RABV 감시를 위해 최적화되고 신속하며 저렴한 전체 게놈 염기서열 분석 워크플로우를 배포할 수 있는 방법을 보여줍니다.
프로토콜
이 연구는 미국 국립의학연구소(NIMR/HQ/R.8a/vol. IX/2788), 탄자니아의 지역 행정 및 지방 정부부(AB.81/288/01) 및 Ifakara Health Institute Institutional Review Board(IHI/IRB/No:22-2014); 케냐의 나이로비 대학교 열대 및 전염병 연구소(P947/11/2019) 및 케냐 의학 연구소(KEMRI-SERU, 프로토콜 번호 3268); 필리핀 보건부(2019-023) 열대의학 연구소(RITM). 나이지리아에서 유래한 샘플의 염기서열 분석은 국가 감시의 일환으로 수집된 보관된 진단 자료에서 수행되었습니다.
참고: 섹션 1-4는 전제 조건입니다. 섹션 5-16은 RABV 나노포어 시퀀싱을 위한 샘플-시퀀스-해석 워크플로우를 설명합니다(그림 1). 펄스 원심분리가 필요한 프로토콜의 후속 단계에서는 10-15,000 x g 에서 5-15초 동안 원심분리합니다.
1. 염기서열분석 및 데이터 분석을 위한 연산 환경 설정
- 옥스포드 나노포어 기술(ONT) 웹사이트37 을 열고 나노포어 특정 리소스에 액세스할 수 있는 계정을 만듭니다.
- 로그인하여 ONT 시퀀싱 및 베이스콜링 소프트웨어설치 38.
- GitHub39 를 열고 계정을 만듭니다.
- artic-rabv40 및 MADDOG 리포지토리41 로 이동하여 설치 지침을 따릅니다.
2. 멀티플렉스 프라이머 체계 설계 또는 업데이트
참고: 기존의 RABV 스킴은 artic-rabv 저장소(40)에서 입수할 수 있다. 새로운 지리적 영역을 타겟팅할 때는 새로운 체계를 설계하거나 기존 체계를 수정하여 추가적인 다양성을 통합해야 합니다.
- 연구 영역의 다양성을 나타내기 위해 게놈 참조 세트를 선택합니다. 이는 일반적으로 공개적으로 사용 가능한 염기서열(예: NCBI GenBank) 또는 예비 사내 데이터 세트입니다. 2.1.1단계에 따라 RABV 시퀀스 데이터 리소스인 RABV-GLUE42를 사용하여 NCBI 시퀀스 및 관련 메타데이터를 필터링하고 다운로드합니다.
참고: 완전한 게놈(즉, 갭 및 마스킹된 염기가 없는)이 있는 참조 염기서열을 선택하십시오. primer design을 위한 reference set으로 최대 10개의 염기서열을 선택하는 것이 좋습니다. 사용 가능한 서열 데이터가 불완전하거나 연구 영역을 대표하지 않는 경우 보충 파일 1의 조언43,44,45를 참조하십시오.- RABV-GLUE의 Sequence Data(시퀀스 데이터) 드롭다운 메뉴에서 NCBI RABV Sequences by Clade 페이지로 이동합니다. 광견병 바이러스(RABV) 링크를 클릭하여 사용 가능한 모든 데이터에 액세스하거나 관심 있는 특정 분류를 선택합니다. 필터 옵션을 사용하여 원하는 기준(예: 원산지, 시퀀스 길이)에 맞는 필터를 추가합니다. 시퀀스 및 메타데이터를 다운로드합니다.
- PrimalScheme 46에 제공된 지침에 따라 다중 중합효소 연쇄 반응(PCR)에 대한 프라이머 계획을 생성합니다. 50bp 겹침이 있는 400bp 체계는 저품질 샘플의 염기서열을 분석하는 데 권장됩니다. 모든 출력을 다운로드하고 저장합니다(파일 또는 프라이머 이름을 편집하지 마십시오).
알림: 이 체계는 입력 fasta의 첫 번째 시퀀스에 인덱싱되며, 이후 '인덱스 참조'라고 합니다(그림 2). 보충 파일 1 에서 프라이머 성능을 최적화하기 위한 옵션을 참조하십시오.
3. RAMPART 및 ARTIC 생물정보학 파이프라인 설정
- RAMPART 및 ARTIC 생물정보학 파이프라인에 대한 입출력 파일을 관리하기 위한 디렉토리 구조를 설정하려면 보충 파일 2 를 참조하십시오.
4. 생물 안전 및 실험실 설정
- 생물 안전 수준(BSL) 2 또는 3 조건에서 잠재적으로 광견병 양성 샘플을 취급합니다.
- 실험실 직원이 광견병 노출 전 예방 접종을 완료하고 세계보건기구(WHO) 권장 사항에 따라 면역 모니터링을 받도록 합니다3.
- 국내 또는 국제 지침에 따라 실험실에 대한 전용 표준 운영 절차 및 위험 평가가 마련되어 있는지 확인합니다.
- 필수 실험실 설정: PCR 전후 영역 간의 물리적 분리를 유지하여 오염을 최소화합니다. 공간이 제한된 실험실이나 현장 실험실 환경에서는 휴대용 글로브 박스 또는 임시 실험실 스테이션을 사용하여 오염을 최소화하십시오.
- 이 프로토콜에서는 다음에 대해 별도의 영역을 지정해야 합니다.
- 샘플 추출: BSL2/3 캐비닛/글로브 박스를 설정하여 생물학적 물질을 처리하고 불활성화 및 RNA 추출을 수행합니다.
- 템플릿 영역: 사전 준비된 반응 마스터 믹스에 템플릿(RNA/cDNA)을 추가하기 위해 BSL1 캐비닛/글로브 박스를 설정합니다.
- 마스터 믹스 영역: 시약 마스터 믹스 준비를 위해 지정된 세척 영역(BSL1 캐비닛/글로브 박스)을 설정합니다. 이 영역에는 템플릿이 없어야 합니다.
- Post-PCR 영역: 앰플리콘 및 염기서열분석 라이브러리 준비 작업을 위한 별도의 영역을 설정합니다.
알림: 모든 영역은 사용 전후에 표면 오염 제거제로 청소하고 자외선(UV)을 살균해야 합니다.
5. 현장 시료 채취 및 진단
알림: 샘플은 개인 보호 장비를 착용하고 참조된 표준 절차47,48,49에 따라 교육을 받고 예방 접종을 받은 직원이 수집해야 합니다.
- Mauti et al.50에 자세히 설명된 바와 같이 구멍 매그넘(foramen magnum)(즉, 후두부 경로)을 통해 샘플을 수집합니다.
- 현장에서 신속 진단 검사로 광견병을 진단하고 직접 형광 항체 검사(DFA), 직접 신속 면역조직화학 검사(DRIT)51,52 또는 실시간 역전사(RT)-PCR(53)과 같은 권장 절차(47)를 사용하여 실험실에서 확인합니다.
- RNA 추출을 위해 확인된 양성 뇌 샘플을 사용하거나 -20°C에서 2-3개월 또는 -80°C에서 장기간 냉동실에 보관합니다. 적절한 DNA/RNA 안정화 배지를 사용하여 저장 및 운송을 위해 RNA를 보존합니다.
6. 시료 전처리 및 RNA 추출(3시간)
NOTE: 시료 유형에 적합한 스핀 컬럼 기반 바이러스 RNA 추출 키트를 사용하십시오.
- 2mL PCR 튜브에 1.4mm 세라믹 비드로 가득 찬 약 200μL 튜브를 채워 두 개의 세라믹 비드 튜브를 준비하고 튜브에 라벨을 붙입니다.
- RNA 추출 키트에 제공된 권장 용량의 용해 완충액을 라벨링된 PCR 튜브에 추가합니다.
- 나무 어플리케이터를 사용하여 광견병 감염이 확인된 뇌 샘플에서 약 3mm 큐브를 채취하고 샘플 ID와 100μL의 뉴클레아제가 없는 물이 있는 라벨이 붙은 튜브에 음성 대조군으로 표시된 튜브에 넣습니다.
참고: 시료 노출을 제한하기 위해 폐쇄 튜브 비드 기반 균질화를 사용하십시오. 가능하지 않은 경우 다른 적절한 기계적 교란기(예: 로터 기반) 또는 수동 마이크로 유봉을 사용하십시오. 그러나 이는 조직을 파괴하기 위해 단단한 표면에서 비드 치팅보다 덜 효과적일 수 있습니다(조직 샘플은 특정 저장 매체에서 굳어질 수 있음). - 나무 어플리케이터 스틱을 사용하여 수동으로 뇌 조직을 파괴한 다음 완전한 조직 균질화가 이루어질 때까지 최대 속도로 소용돌이칩니다.
- 제조업체의 지침에 따라 용해물을 원심분리하고 피펫을 사용하여 상층액을 라벨이 부착된 새 마이크로 원심분리기 튜브로 옮깁니다. 후속 단계에서만 이 상층액을 사용하십시오.
- RNA 추출 키트의 스핀 컬럼 지침에 따라 정제된 RNA를 얻습니다.
- 여기에 네거티브 추출 제어(NEC)를 포함하고 염기서열분석 단계까지 진행합니다.
7. cDNA 준비(20분)
- 마스터 믹스 영역에서, 처리할 샘플 및 대조군의 수에 따라 첫 번째 가닥 cDNA 합성을 위한 마스터 믹스를 준비합니다(적절한 시약을 보장하기 위해 10%의 초과 부피; 표 1). 이 단계에서는 NTC(No-Template Control)를 포함해야 합니다.
- 라벨 0.2mL PCR 스트립 튜브와 마스터 혼합물 5μL를 분취합니다.
- 준비된 튜브를 템플릿 영역으로 가져갑니다. NEC를 포함하여 라벨링된 각 튜브에 5μL의 RNA를 추가합니다. NTC에 5μL의 뉴클레아제가 없는 물(NFW)을 추가합니다.
- 표 1에 언급된 조건에 따라 열 순환기에서 배양합니다.
참고: 선택적 일시 중지 지점: cDNA는 필요한 경우 최대 1개월 동안 -20°C에서 보관할 수 있지만 PCR로 진행하는 것이 좋습니다.
8. 프라이머 풀 스톡 준비(1시간)
알림: 이 단계는 개별 프라이머에서 새 스톡을 만드는 경우에만 필요하며, 그 후에는 미리 준비된 스톡 용액을 사용할 수 있습니다.
- 마스터 믹스 영역에 100μM 스톡의 프라이머 풀을 준비합니다.
- 동결건조된 프라이머를 각각 100μM 농도의 1x tris-EDTA(TE) 완충액 또는 NFW에 재현탁시킵니다. 소용돌이를 철저히 하고 회전하십시오.
참고: 다음 단계에서 개별 프라이머는 앰플리콘 겹침 측면에 있는 프라이머 간의 상호 작용을 피하기 위해 홀수(풀 A)와 짝수(풀 B)의 두 프라이머 풀로 분리됩니다. 이러한 프라이머 풀은 표적 게놈에 걸쳐 겹치는 400bp 앰플리콘을 생성합니다. - 홀수 번호의 모든 프라이머를 튜브 랙에 배열합니다. 각 프라이머에서 5μL를 "프라이머 체계 이름 - Pool A(100μM)"라고 표시된 1.5mL 마이크로 원심분리 튜브에 추가하여 프라이머 풀 스톡을 생성합니다.
- 모든 짝수 번호의 프라이머에 대해 이 과정을 반복하고 "프라이머 체계 이름 - 풀 B(100μM)"로 레이블을 지정합니다.
- 각 프라이머 풀을 분자 등급 물에 1:10으로 희석하여 10μM 프라이머 스톡을 생성합니다.
참고: 10μM 프라이머 희석액을 여러 개 분취하여 분해 또는 오염된 경우 동결합니다.
9. 멀티플렉스 PCR(5시간)
- 마스터 믹스 영역의 각 primer pool에 대해 하나씩 두 개의 PCR 마스터 믹스를 준비합니다.
- 프라이머당 0.015μM의 최종 농도를 사용합니다. 다음 공식을 사용하여 PCR 반응에 필요한 primer pool 부피(표 2)를 계산합니다.
프라이머 풀 부피 = 프라이머 수 x 반응 부피 x 0.015/프라이머 스톡 농도(μM)
- 프라이머당 0.015μM의 최종 농도를 사용합니다. 다음 공식을 사용하여 PCR 반응에 필요한 primer pool 부피(표 2)를 계산합니다.
- Pool A 마스터 믹스 및 Pool B 마스터 믹스를 각각 10μL씩 템플릿 영역의 표지된 PCR 스트립 튜브에 분취합니다. 모든 샘플에 대해 2.5μL의 cDNA(3단계에서)를 해당 표지된 프라이머 Pool A 및 B 반응 각각에 추가합니다. 과잉 cDNA는 -20 °C에 저장될 수 있다.
- 부드럽게 튕기고 원심분리기를 펄스하여 혼합합니다.
- 표 2에 언급된 조건으로 샘플을 PCR 기계에서 배양합니다.
참고: 이 프로그램에는 5분의 긴 어닐링 시간(프라이머 수가 많기 때문에 필요)과 확장에 충분한 앰플리콘의 짧은 길이(400bp)로 인해 특정 확장 단계가 포함되지 않습니다.
10. PCR 청소 및 정량화(3.5시간)
- PCR 후 영역에서 이 시점부터 모든 작업을 수행합니다.
- 분취액 고체상 가역적 고정화(SPRI) 비드를 메인 병에서 마이크로 원심분리기 튜브로 주입합니다. 4 °C에서 보관하십시오.
- SPRI 비드 부분 표본을 실온(RT, ~20°C)으로 예열하고 비드가 용액에 완전히 재현탁될 때까지 완전히 와류합니다.
- 1.5mL 튜브에서 각 샘플에 대해 프라이머 Pool A와 프라이머 Pool B PCR 산물을 결합합니다. 필요한 경우 물을 추가하여 부피를 25μL로 만듭니다.
- 각 샘플에 25μL의 SPRI 비드를 추가합니다(1:1 비드:샘플 비율). 피펫팅을 위아래로 하거나 튜브를 부드럽게 두드려 혼합합니다.
- 실온에서 10분 동안 배양하고 때때로 튜브를 뒤집거나 튕깁니다.
- 비드와 용액이 완전히 분리될 때까지 마그네틱 랙에 놓습니다. 비드 펠릿을 방해하지 않도록 주의하면서 상층액을 제거하고 폐기하십시오.
- 80% 에탄올로 두 번 세척합니다(RT로 데우기).
- 펠릿에 200μL의 에탄올을 추가합니다. 비드가 제대로 세척될 때까지 30초 동안 기다리십시오.
- 비드 펠릿을 만지지 않도록 상층액을 조심스럽게 제거하고 폐기합니다.
- 10.8.1-10.8.2단계를 반복하여 펠릿을 두 번째로 세척합니다.
- 10μL 팁을 사용하여 에탄올의 흔적을 모두 제거합니다. 미량 에탄올이 증발 할 때까지 공기 건조 (작은 비드의 경우 ~ 30 초가 빠르게 발생함). 이런 일이 발생하면, 펠릿은 광택에서 무광택으로 바뀌어야 합니다. 과도하게 건조하지 않도록 주의하십시오(펠릿이 갈라지면 너무 건조합니다), 이는 DNA 회복에 영향을 미칩니다.
- 비드를 15μL의 NFW에 재현탁시키고 RT(마그네틱 랙에서 분리)에서 10분 동안 배양합니다.
- 마그네틱 랙으로 돌아가 상층액(세척된 제품)을 새 1.5mL 튜브로 옮깁니다.
- 별도의 튜브(제품 2μL + NFW 18μL)에 각 샘플을 1:10 희석하여 준비합니다.
알림: 이 단계에서 매우 주의하십시오.tage 교차 오염을 피하기 위해. 한 번에 하나의 앰플리콘 튜브만 열어야 합니다. 먼저 18μL의 물을 튜브에 분취합니다(깨끗한 마스터 믹스 영역에서). - protocols.io54,55에 설명된 대로 고감도의 특이적 형광측정기를 사용하여 희석된 각 샘플의 DNA 농도를 측정합니다.
11. 정규화(30분)
- 정규화 템플릿(보충 파일 3) 및 각 샘플의 DNA 농도(ng/μL)를 사용하여 총 5μL 부피에서 각 샘플의 200fmol에 필요한 희석된(또는 순수한) 샘플의 부피를 계산합니다.
- 새 PCR 튜브에 라벨을 붙이고 계산된 부피의 NFW 및 샘플을 추가하여 정규화된 DNA를 얻습니다.
- 200fmol을 얻기 위해 희석된 샘플의 5μL 이상이 필요한 경우 희석되지 않은(순수한) 샘플에 대해 계산된 부피를 사용합니다.
참고: 선택적 일시 중지 지점: 이 시점에서 세척된 PCR 산물은 4°C에서 최대 1주일 동안 보관하거나 필요한 경우 장기 보관을 위해 -20°C에 둘 수 있습니다.
12. 엔드 프렙 및 바코드(1.5시간)
참고: 다음 단계에서는 나노포어 특이적 바코드 및 결찰 시퀀싱 키트의 특정 시약을 사용한다고 가정합니다(자세한 내용은 재료 표 참조). 이 프로토콜은 다양한 화학 버전으로 이전할 수 있지만 사용자는 제조업체 지침에 따라 호환 가능한 키트를 사용하도록 주의해야 합니다.
- 끝 수리 및 dA-tailing
- 표 3에 언급된 각 샘플에 대한 최종 분취 반응을 설정합니다. 샘플 수에 따라 마스터 믹스를 준비합니다(초과 10% 추가). 피펫팅할 때는 시약이 점성이 있으므로 주의하십시오.
- 정규화된 DNA(5μL)의 각 튜브에 마스터 믹스 5μL를 추가합니다. 총 반응 혼합물은 10μL여야 합니다. 매번 팁을 교체하고 한 번에 하나의 튜브만 열도록 하십시오.
- 표 3에 언급된 조건 하에서 열 순환기에서 배양한다.
- 바코드
- 바코드 키트의 바코드를 1.25μL/튜브의 PCR 스트립 튜브로 분주하고 각 샘플에 할당된 바코드를 기록합니다.
- 0.75μL의 최종 준비된 샘플을 할당된 바코드 부분 표본에 추가합니다.
- 샘플 수(10% 초과 추가)에 따라 ligation master mix를 준비합니다(표 4).
- 8 μL의 ligation master mix를 말단 전처리 샘플 + 바코드에 추가하여 총 10 μL의 반응을 제공합니다.
- 표 4에 언급된 조건을 사용하여 열 순환기에서 배양합니다.
- SPRI 비드 세척 및 DNA 정량
- 짧은 단편 완충액(SFB)을 RT에서 해동하고 와류, 펄스 원심분리기로 혼합하고 얼음 위에 놓습니다.
- 바코드가 부착된 모든 샘플을 1.5mL 로빈드 마이크로 원심분리기 튜브에 함께 모읍니다. 클린업 부피가 너무 커서 사용할 수 없도록 각 기본 바코드 반응에서 12-24개 샘플(10μL/샘플), 최대 48개 샘플(5μL/샘플) 또는 최대 96개 샘플(2.5μL/샘플)을 풀링합니다.
- 0.4x 부피의 SPRI 비드를 바코드 풀에 추가합니다. 부드럽게 혼합(튕기기 또는 피펫팅)하고 실온에서 5분 동안 배양합니다.
- 비드가 펠릿화되고 상층액이 완전히 투명해질 때까지(~2분) 샘플을 자석에 놓습니다. 상층액을 제거하고 폐기하십시오. 구슬을 방해하지 않도록 주의하십시오.
- 250μL의 SFB로 두 번 세척합니다.
- 자석에서 튜브를 제거하고 펠릿을 250μL의 SFB에 완전히 재현탁시킵니다. 30초 동안 배양하고 원심분리기를 펄스하고 자석으로 돌아갑니다.
- 상층액을 제거하고 폐기하십시오.
- 12.3.5단계를 반복하여 두 번째 SFB 세척을 수행합니다.
- 원심분리기를 펄스하고 잔류 SFB를 제거합니다.
- 200 μL의 80 % (RT) 에탄올을 첨가하여 펠릿을 목욕시킵니다. 비드 펠릿을 방해하지 않도록 주의하면서 에탄올을 제거하고 폐기하십시오. 30초 동안 또는 펠릿이 광택을 잃을 때까지 자연 건조합니다.
- RT에서 22μL의 NFW에 10분 동안 재현탁합니다.
- 자석에 놓고 ~2분 동안 그대로 둔 다음 용액을 조심스럽게 제거하고 깨끗한 1.5mL 미세 원심분리기 튜브로 옮깁니다.
- 앞에서 설명한 대로 1μL를 사용하여 DNA 농도를 얻습니다(단계 10.13).
참고: 선택적 일시 중지 지점: 이 시점에서 라이브러리는 최대 4주일 동안 1°C에서 보관하거나 장기 보관을 위해 -20°C에서 보관할 수 있지만 어댑터 결찰 및 시퀀싱을 계속하는 것이 좋습니다.
13. 시퀀싱(최대 48시간)
- 컴퓨터를 준비합니다(필수 구성 요소 섹션 1-4 참조).
- 새 데이터를 저장할 수 있는 충분한 공간(최소 150GB)이 있는지, 삭제하기 전에 이전 실행의 데이터를 서버에 백업/이동했는지, 최신 버전의 MinKNOW가 설치되어 있는지 확인하십시오.
- 저장된 플로우 셀을 냉장고에서 꺼내 RT에 도달하도록 합니다.
- 어댑터 결찰(1시간)
- 어댑터 혼합물과 리가아제를 펄스 원심분리하고 얼음 위에 놓습니다.
- 용출 완충액(EB), SFB 및 결찰 완충액을 RT에서 해동합니다. 와류, 펄스 원심분리기로 혼합하고 얼음 위에 놓습니다.
- 어댑터 결찰 마스터 믹스(표 5)를 준비하고, 낮은 결합 튜브에서 지정된 순서로 시약을 결합합니다.
참고: 어댑터 결찰 마스터 믹스 시약(표 5)의 대안은 실험실의 가용성에 따라 사용할 수 있습니다. 대안 목록은 보충 파일 3 및 재료 표를 참조하십시오. 보충 파일 3 워크시트의 계산을 사용하여 200fmol에 해당하는 DNA 라이브러리의 부피를 얻습니다. 20μL 미만을 계산하는 경우 NFW를 추가하여 최대 20μL를 만듭니다. - 부드럽게 튕기고 펄스 원심분리기로 혼합합니다. 상온에서 20분 동안 배양합니다.
참고: 배양 중에 플로우 셀(flow cell) 준비를 시작합니다(섹션 13.5).
- SPRI 비드를 사용하여 청소하십시오(이전 청소에서와 같이 에탄올을 사용하지 마십시오).
- 샘플에 0.4x 부피의 SPRI 비드(RT)를 추가합니다. 상온에서 10분 동안 배양하고 혼합을 돕기 위해 간헐적으로 부드럽게 튕깁니다.
- 비드와 용액이 완전히 분리될 때까지 자석 위에 놓습니다(~5분). 상층액을 제거하고 버리십시오. 비드 펠릿을 방해하지 않도록 주의하십시오.
- 125μL의 SFB로 두 번 세척합니다.
- 피펫과 혼합하여 125μL의 SFB로 펠릿을 완전히 재현탁시킵니다. 30초 동안 배양합니다.
- 펄스 원심분리기를 사용하여 튜브 베이스에 액체를 모으고 자석에 놓습니다. 상층액을 제거하고 폐기하십시오.
- 13.4.4-13.4.5단계를 반복하여 펠릿을 두 번째로 세척합니다.
- 펄스 원심분리기를 작동시키고 과도한 SFB를 제거합니다.
- 15μL의 EB에 재현탁하고 RT에서 10분 동안 배양합니다.
- ~2분 동안 자석으로 돌아간 다음 용액을 깨끗한 1.5mL 미세 원심분리기 튜브로 조심스럽게 옮깁니다.
- 앞서 10.13단계에서 설명한 대로 용리된 라이브러리의 1μL를 정량합니다.
알림: 최상의 결과를 얻으려면 MinION 시퀀싱으로 직접 진행하십시오. 그러나 최종 라이브러리는 필요한 경우 최대 1주일 동안 4°C의 EB에 보관할 수 있습니다.
- 플로우 셀 품질 검사를 실행합니다.
- 시퀀싱 장치를 랩톱에 연결하고 시퀀싱 소프트웨어를 엽니다.
- 플로우 셀 유형을 선택한 다음 Flow Cell 확인 및 테스트 시작 (Check Flow Cell and Start Test)을 클릭합니다.
- 완료되면 활성(즉, 생존 가능한) 기공의 총 수가 표시됩니다. 새로운 플로우 셀에는 >800개의 활성 기공이 있어야 합니다. 그렇지 않은 경우 제조업체에 교체를 문의하십시오.
- 플로우 셀 프라이밍 및 로딩(20분)
- 다음 시약을 RT에서 해동한 다음 시퀀싱 완충액, 플러시 테더, 플러시 완충액 및 로딩 비드를 얼음 위에 놓습니다.
- 염기서열분석 완충액을 소용돌이치게 하고 완충액을 세척하고 펄스 원심분리기를 얼음 위에 놓습니다.
- 플러시 테더를 펄스 원심분리하고 피펫팅으로 혼합합니다. 그런 다음 얼음 위에 놓습니다.
- 플로우 셀 프라이밍 키트에서 플러시 버퍼 튜브에 직접 30μL의 플러시 테더를 추가하여 플로우 셀 프라이밍 믹스를 준비하고 피펫팅으로 혼합합니다.
- 로딩 비드는 빠르게 가라앉기 때문에 사용 직전에 피펫팅하여 혼합하십시오.
- 새 튜브에서 표 5에 언급된 대로 염기서열분석을 위한 최종 라이브러리 희석액을 준비합니다.
참고: 보충 파일 3 워크시트의 계산을 사용하여 50fmol에 해당하는 DNA 라이브러리의 부피를 얻습니다. 12μL 미만을 계산하는 경우 EB를 추가하여 최대 12μL를 만듭니다. - 시퀀싱 장치 덮개를 뒤로 젖히고 프라이밍 포트가 보이도록 프라이밍 포트 덮개를 시계 방향으로 밉니다(그림 3)
- P1000 피펫을 200 μL로 설정하여 기포를 조심스럽게 제거하고, 팁을 프라이밍 포트에 삽입한 다음, 피펫 팁에 들어가는 작은 부피가 보일 때까지(최대 230 μL로 회전) 휠을 돌립니다.
- 800μL의 플로우 셀 프라이밍 혼합물을 프라이밍 포트를 통해 플로우 셀에 로드하고 기포가 발생하지 않도록 주의합니다.
- 5분 동안 그대로 두십시오.
- 샘플 포트 커버를 부드럽게 들어 올리고 P1000 피펫을 사용하여 프라이밍 포트를 통해 200μL의 프라이밍 혼합물을 플로우 셀에 로드합니다.
- 로딩 전에 라이브러리 믹스를 위아래로 피펫팅하여 로딩 전에 마스터 믹스의 로딩 비드가 재현탁되도록 합니다.
- 75μL의 library mix를 샘플 포트를 통해 플로우 셀에 적하 방식으로 로드합니다. 다음 드립을 추가하기 전에 각 드립이 포트로 흐르는지 확인하십시오.
- 샘플 포트 커버를 부드럽게 교체하여 마개가 샘플 포트에 들어가는지 확인합니다.
- 프라이밍 포트를 닫고 시퀀싱 장치 덮개를 교체합니다.
- 시퀀싱 실행(최대 48시간)
- 시퀀싱 장치를 노트북에 연결하고 시퀀싱 소프트웨어를 엽니다.
- start(시작)를 클릭한 다음 Start Sequencing(시퀀싱 시작)을 클릭합니다.
- New Experiment(새 실험)를 클릭하고 시퀀싱 소프트웨어 GUI(그래픽 사용자 인터페이스) 워크플로에 따라 실행에 대한 매개 변수를 설정합니다.
- 실험 이름과 샘플 ID(예: rabv_run1)를 입력하고 드롭다운 메뉴에서 Flow Cell Type 을 선택합니다.
- 키트 선택을 계속하고 관련 ligation sequencing kit 및 사용된 native barcoding kit를 선택합니다.
- 실행 옵션을 계속합니다. 특정 시간 후에 실행이 자동으로 중지되도록 원하지 않는 한 기본값을 유지합니다(실행은 언제든지 수동으로 중지할 수 있음).
- 베이스콜링을 계속합니다. 컴퓨팅 리소스에 따라 Basecalling을 켜거나 끄도록 선택합니다(컴퓨터 설정 참조). 바코드에서 Edit Options(편집 옵션)를 선택하고 Barcode Both Ends(바코드 양끝)가 켜져 있는지 확인합니다. 저장하고 출력 섹션으로 계속 진행합니다.
- 기본값을 수락하고 최종 검토를 계속하고, 설정을 확인하고, 워크시트(보충 파일 3)에 세부 정보를 기록합니다. 시작을 클릭합니다.
알림: 플로우 셀을 재사용하는 경우 시작 볼륨을 조정하십시오.tage(실행 옵션의 고급 섹션에서), 보충 파일 3의 구성표에 표시된 대로. - 초기 활성 채널 기록 - 이 값이 품질 관리(QC) 검사보다 현저히 낮으면 시퀀싱 소프트웨어를 다시 시작합니다. 여전히 낮으면 컴퓨터를 재부팅하십시오.
- 스트랜드 대 단일 기공의 초기 채널을 기록하여 대략적인 기공 점유를 제공합니다. 이 숫자는 변동되므로 근사치를 제공하십시오.
- 실행이 진행됨에 따라 모니터링합니다.
14. 라이브 및 오프라인 베이스콜링
참고: 이 지침에서는 artic-rabv 리포지토리에 제공된 기존 디렉토리 구조와 프로토콜의 사전 요구 사항 섹션 1 및 3을 따랐다고 가정합니다.
- 로컬 파일 시스템에서 analysis라는 새 디렉터리를 만들어 모든 분석 출력을 저장합니다. 더 구성하려면 : MinKNOW에 제공된 샘플 ID를 run_name로 사용하여 프로젝트 이름과 그 안에 실행을위한 새 디렉토리를 만드는 하위 디렉토리를 만듭니다. 다음과 같이 하나의 명령으로 이 작업을 수행합니다.
mkdir -p
분석/project_name/run_name
그런 다음 해당 위치로 이동합니다.
시디
경로/분석/project_name/run_name - 라이브 베이스콜링
참고: 나노포어 베이스콜링을 실시간으로 수행하려면 노트북에 NVIDIA CUDA 호환 그래픽 처리 장치(GPU)가 필요합니다. GPU 베이스콜링 설정에 대한 지침이 guppy 프로토콜(56)을 사용하여 수행되었는지 확인하십시오.- 실행 설정 중에 라이브 베이스콜링을 켭니다.
- RAMPART를 사용하여 아래 지침에 따라 시퀀싱 커버리지를 실시간으로 모니터링하십시오.
- 컴퓨터 터미널에서 artic-rabv conda 환경을 활성화합니다.
conda 활성화 artic-rabv - run_name 디렉토리 내에 rampart 출력에 대한 새 디렉토리를 만들고 탐색합니다.
cd /경로/분석/project_name/run_name
mkdir rampart_output
CD rampart_output - 바코드와 샘플 이름을 페어링하는 바코드 .csv 파일을 만듭니다. 바코드당 한 줄이 있어야 하며 "바코드" 및 "샘플"이라는 제목과 함께 라이브러리에 있는 바코드만 지정해야 합니다. artic-rabv 디렉토리의 예를 따르십시오.
분석/example_project/example_run/rampart_output/바코드.csv - 실행에 대한 MinKNOW 출력의 fastq_pass 폴더에 대한 관련 프로토콜 폴더 및 경로를 제공하여 RAMPART를 시작합니다.
rampart --protocol /path/rampart/scheme_name_V1_protocol - basecalledPath - 브라우저 창을 열고 URL 상자에서 localhost:3000으로 이동합니다. 결과가 화면에 나타나기 전에 충분한 데이터가 기본 호출될 때까지 기다립니다.
- 오프라인 베이스콜링(실행 후 수행)
- 라이브 베이스콜링이 설정되지 않은 경우 MinKNOW의 출력은 원시 신호 데이터(fast5 files). 실행 중에는 RAMPART를 사용할 수 없습니다. guppy를 사용하여 실행 후 fast5 파일을 기본 데이터(fastq 파일)로 변환합니다(필수 구성 요소 1.1.1단계의 설정 참조). basecalled 데이터에서 RAMPART 사후 실행을 실행합니다.
- guppy basecaller를 실행합니다.
guppy_basecaller -c dna_r9.4.1_450bps_fast.cfg -i /path/to/reads/fast5_* -s /path/analysis/project_name/run_name -x 자동 -r
-c는 베이스콜링 모델을 지정하는 구성 파일이고, -i는 입력 경로이고, -s는 저장 경로이고, -x는 GPU 장치에 의한 베이스콜링을 지정하고(컴퓨터 버전의 guppy를 사용하는 경우 제외), -r은 입력 파일을 재귀적으로 검색하도록 지정합니다.
참고: 구성 파일(.cfg)은 _fast를 _hac로 대체하여 정확도가 높은 기본 호출자로 변경할 수 있지만 시간이 훨씬 더 오래 걸립니다.
15. 플로우 셀 세척
- 플로우 셀은 세척하여 공극이 여전히 생존 가능한 경우 새로운 라이브러리의 염기서열을 분석하는 데 재사용할 수 있습니다. ONT 플로우 셀 세척 프로토콜57에서 세척 지침을 참조하십시오.
16. 분석 및 해석
- ARTIC 생물정보학 파이프라인을 통한 합의 염기서열 생성
- rabv_protocols 폴더의 artic-rabv GitHub 저장소40 에 자세히 설명된 지침에 따라 원시 fast5 또는 basecalled fastq 파일에서 합의 시퀀스를 생성합니다.
참고: 자세한 내용은 Artic 파이프라인 - 코어 파이프라인58 을 참조하세요.
- rabv_protocols 폴더의 artic-rabv GitHub 저장소40 에 자세히 설명된 지침에 따라 원시 fast5 또는 basecalled fastq 파일에서 합의 시퀀스를 생성합니다.
- 선택 사항: 앰플리콘당 평균 판독 깊이를 분석합니다.
- 보충 파일 1을 참조하여 artic-rabv 저장소에서 사용할 수 있는 스크립트를 조정합니다. 간단히 말해서, SAMtools59 및 R에 표시된 뉴클레오티드당 커버리지를 사용하여 심층 통계가 생성됩니다.
- GLUE를 이용한 계통발생 분석
- 42 RABV_GLUE부터 분석 > 유전형 분석 및 해석 탭 > 파일 추가를 선택하고 합의 시퀀스의 fasta 파일을 선택합니다.
- 제출을 클릭하고 기다립니다. 분석이 완료되면 분석 표시 버튼을 클릭하여 clade 및 subclade 할당, 유전자당 커버리지, 참조 염기서열의 변이 및 가장 가까운 친척을 표시할 수 있습니다.
- 관련 컨텍스트 시퀀스는 Sequence Data > NCBI Sequences by Clade 섹션에서도 식별할 수 있습니다.
- 식별된 clade를 선택하거나 Rabies Virus (RABV) 를 클릭하여 사용 가능한 모든 염기서열을 확인합니다.
- 관련 염기서열(예: 원산지)을 필터링합니다.
- 분석 및 비교를 위해 이러한 시퀀스와 해당 메타데이터를 다운로드하십시오.
- MADDOG41을 사용한 계보 할당
- GitHub에서 MADDOG 리포지토리를 가져와 최신 버전으로 작업하고 있는지 확인합니다.
- 로컬 MADDOG 리포지토리(이전에 필수 구성 요소 섹션에서 생성됨) 내에 실행 이름이라는 할당 폴더를 만듭니다.
- 폴더 안에 합의 시퀀스가 포함된 fasta 파일을 추가합니다.
- 폴더에 메타데이터 파일을 추가합니다.
참고: 이 파일은 'ID', '국가', '연도' 및 '할당'이라는 4개의 열이 있는 csv여야 하며 시퀀스 ID, 샘플링 국가 및 샘플 수집 연도를 자세히 설명해야 하며 '할당' 열은 비어 있어야 합니다. - 명령줄 인터페이스에서 conda 환경(conda activate MADDOG)을 활성화합니다.
- 명령줄 인터페이스에서 MADDOG 리포지토리 폴더로 이동합니다.
- 처음에는 염기서열에 계보 할당을 수행하여 잠재적인 이상을 확인하고 더 긴 계보 지정 단계를 실행하는 것이 적절한지 확인합니다. 이를 위해 명령 줄에 sh assignment.sh 를 입력하십시오.
- 메시지가 표시되면 Y를 입력하여 리포지토리를 가져왔고 최신 버전의 MADDOG로 작업 중임을 나타냅니다.
- 메시지가 표시되면 fasta 파일이 포함된 MADDOG 리포지토리 폴더 내의 폴더 이름을 입력합니다.
- 계보 할당이 완료되면 폴더에서 출력 파일을 확인합니다. 출력이 예상대로이고 동일한 계보에 할당된 시퀀스가 여러 개 있는 경우 계보 지정을 실행합니다.
- 계보 지정을 실행하는 경우 방금 만든 할당 출력 파일을 삭제합니다.
- 터미널의 MADDOG 저장소 폴더 내에서 sh designation.sh 명령을 실행합니다.
- 메시지가 표시되면 Y를 입력하여 저장소를 가져왔고 최신 버전의 MADDOG로 작업 중임을 나타냅니다.
- 메시지가 표시되면 fasta 파일 및 메타데이터가 포함된 MADDOG 리포지토리 폴더 내에 폴더 이름을 입력합니다. 이것은 각 염기서열에 대한 계보 정보, 새롭고 관련된 이전 염기서열(16.3.6에서)의 계통 발생, 계보에 대한 계층적 정보, 잠재적으로 새로운 계보 및 과소 샘플링 영역에 대한 세부 정보를 출력합니다.
참고: 프로토콜, 사용법 및 출력에 대한 자세한 내용은 Campbell et al.60에서 확인할 수 있습니다. - 초기 분석이 완료되면 새로 출현하는 계보와 과소 샘플링된 계보도 테스트하라는 메시지가 표시되면 필요한 경우 Y를 입력합니다. 그렇지 않으면 N을 입력합니다.
- 새로 발견된 계보를 확인하라는 메시지가 표시되면 Y를 입력하고 결과 NEXT_STEPS.eml 파일의 지침을 따릅니다. 그렇지 않으면 N을 입력합니다.
대표적 결과
이 프로토콜에 설명된 RABV의 샘플-서열-해석 워크플로우는 탄자니아, 케냐, 나이지리아 및 필리핀과 같은 풍토병 국가의 다양한 실험실 조건에서 성공적으로 사용되었습니다(그림 4). 이 프로토콜은 신선 및 냉동 뇌 조직, 장기간 콜드 체인으로 운송된 뇌 조직의 cDNA 및 RNA 추출물, 뇌 조직 도말이 있는 FTA 카드 등 다양한 샘플 유형 및 조건에 사용되었습니다(표 6).
RAMPART를 사용한 실시간 베이스콜링(그림 5)은 거의 실시간으로 판독 생성과 샘플당 커버리지 비율을 보여줍니다. 이는 실행을 중지하고 재사용을 위해 플로우 셀을 저장할 시기를 결정할 때 특히 유용합니다. 실행 시간의 변화가 관찰되었으며, 일부는 2시간 내에 완료되는 반면 다른 일부는 적절한 적용 범위(x100)에 도달하는 데 12시간 이상 걸릴 수 있습니다. 증폭이 불량한 영역도 볼 수 있습니다. 예를 들어, 그림 6 은 커버리지 프로파일이 증폭이 매우 낮은 일부 앰플리콘을 보여주는 한 염기서열분석 실행의 스냅샷을 보여주며, 이는 잠재적으로 문제가 있는 프라이머를 나타냅니다. 이러한 증폭 불량 영역을 보다 철저하게 조사함으로써 프라이머 불일치를 식별할 수 있었으며, 이를 통해 개별 프라이머를 재설계하고 개선할 수 있었습니다. 일부 프라이머 체계는 다른 체계보다 더 많은 불일치를 보였습니다. 이는 필리핀과 비교하여 동아프리카 프라이머 계획에서 관찰되며, 동아프리카 계획은 훨씬 더 광범위한 다양성을 포착하는 것을 목표로 하기 때문에 목표 다양성에 부합합니다.
RABV 게놈 데이터 관리를 위한 범용 리소스인 RABV-GLUE42와 계통 분류 및 명명법 시스템인 MADDOG60을 사용하여 결과 RABV 염기서열을 컴파일하고 해석했습니다. 표 7 은 RABV-GLUE를 사용하여 할당된 각 국가에서 유통되는 주요 및 보조 분류를 보여줍니다. 또한 MADDOG 할당에 따른 로컬 계보의 고해상도 분류도 표시됩니다.
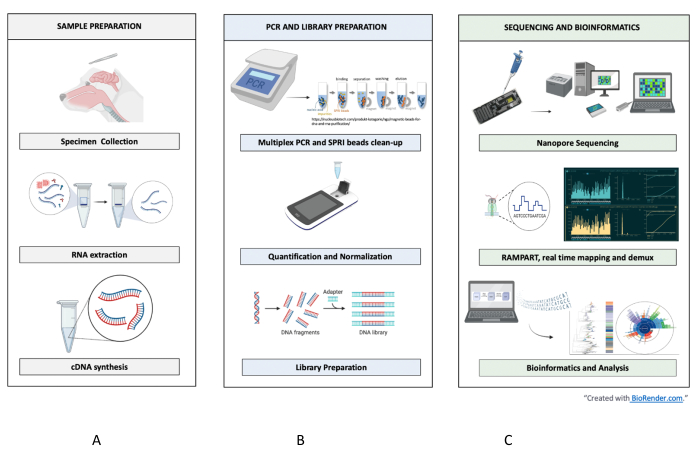
그림 1: RABV의 샘플-시퀀싱-해석 워크플로우. (A) 시료 전처리, (B) PCR 및 라이브러리 전처리, (C) 염기서열분석 및 생물정보학에서 분석 및 해석에 이르는 단계를 요약하여 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
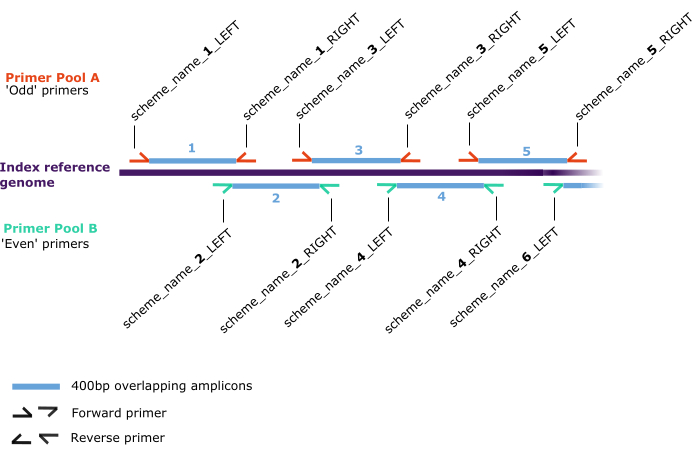
그림 2: 프라이머 체계 개략도. A (빨간색 )와 B (녹색)의 두 개의 개별 풀에 할당되는 정방향 및 역방향 프라이머(반쪽 화살표) 쌍에 대한 '인덱스 참조 게놈'(짙은 보라색)을 따라 어닐링 위치. 프라이머 쌍은 인덱스 참조 게놈을 따라 scheme_name_X_DIRECTION순차적으로 번호가 매겨진 400bp 중첩 앰플리콘(파란색)을 생성하며, 여기서 'X'는 프라이머에 의해 생성된 앰플리콘을 나타내는 숫자이고 'DIRECTION'은 각각 정방향 또는 역방향을 설명하는 'LEFT' 또는 'RIGHT'입니다. 'X'의 홀수 또는 짝수 값은 풀(각각 A 또는 B)을 결정합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
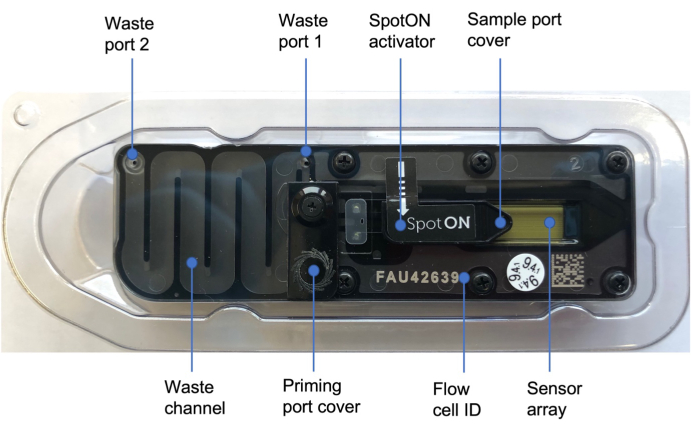
그림 3: 나노포어 플로우 셀48. 파란색 라벨은 프라이밍 용액이 추가되는 프라이밍 포트를 덮는 프라이밍 포트 커버, 샘플이 적하 방식으로 추가되는 샘플 포트를 덮는 SpotON 샘플 포트 커버, 폐기물 포트 1 및 2, 플로우 셀 ID를 포함하여 플로우 셀의 다양한 부분을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
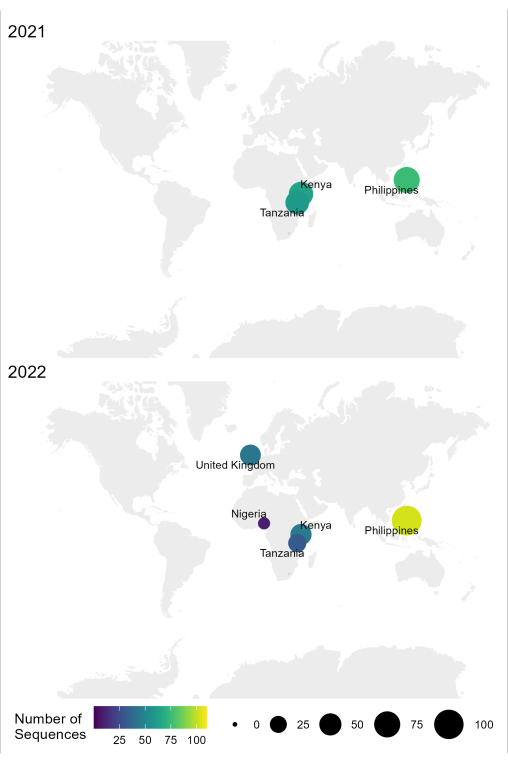
그림 4: 2021년과 2022년에 최적화된 워크플로우를 사용하여 RABV 염기서열분석이 수행된 위치를 보여주는 지도. 거품 크기와 색상은 위치당 시퀀스 수에 해당하며, 작고 어두울수록 적고 크고 밝을수록 많습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
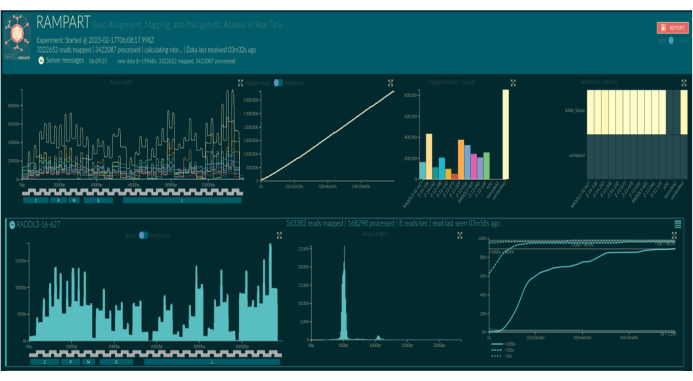
그림 5: 웹 브라우저의 RAMPART 시각화 스크린샷. 바코드 이름은 생물정보학 설정에 따라 샘플 이름으로 대체됩니다. 상단 3개의 패널은 인덱스 참조 게놈의 뉴클레오티드 위치당 각 바코드에 대한 매핑된 판독의 적용 범위(왼쪽 상단, 바코드로 색상), 시간 경과에 따른 모든 바코드의 매핑된 판독값 합계(상단 중앙) 및 바코드당 매핑된 판독(오른쪽 상단, 바코드로 색상)과 같은 전체 실행에 대한 요약 플롯을 보여줍니다. 아래쪽 패널에는 바코드당 플롯 행이 표시됩니다. 왼쪽에서 오른쪽으로: 인덱스 참조 게놈의 뉴클레오티드 위치당 매핑된 판독의 커버리지 깊이(왼쪽), 매핑된 판독의 길이 분포(가운데), 시간 경과에 따라 매핑된 판독의 10배, 100배 및 1,000배 커버리지를 얻은 인덱스 참조 게놈의 뉴클레오티드 위치 비율(오른쪽). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
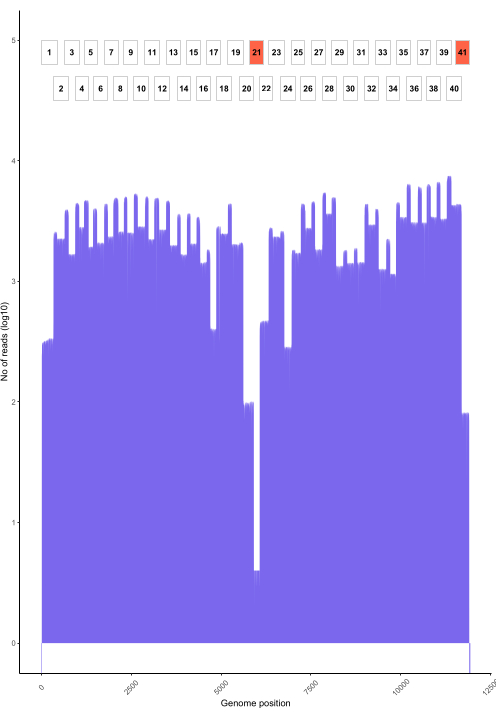
그림 6: 프로토콜을 사용하여 염기서열을 분석한 필리핀의 광견병 바이러스 샘플에 대한 게놈 전반의 판독 범위. 게놈의 각 뉴클레오티드 위치에서의 판독 범위는 라이브러리를 생성하는 데 사용된 중첩 앰플리콘(1-41)의 위치와 함께 표시됩니다. 커버리지 깊이의 스파이크는 앰플리콘 중첩 영역에 해당합니다. 커버리지 깊이가 낮은 앰플리콘은 앰플리콘 중첩 영역에 해당합니다. 커버리지 깊이가 낮은 앰플리콘은 빨간색으로 강조 표시되어 최적화가 필요할 수 있는 문제가 있는 영역을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 1: cDNA 준비를 위한 마스터 믹스 및 열 순환기 조건. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 2: 멀티플렉스 PCR을 위한 마스터 믹스 및 열 순환기 조건. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 3: 최종 분취 반응을 위한 마스터 믹스 및 열 순환기 조건. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 4: 바코드를 위한 마스터 믹스 및 열 순환기 조건. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 5: 염기서열분석을 위한 어댑터 결찰 마스터 믹스 및 최종 라이브러리 희석. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 6: 생성된 광견병 바이러스 전체 게놈 염기서열의 수와 샘플-염기서열-해석 워크플로우를 사용하는 여러 국가에서 사용된 샘플 유형. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 7: 워크플로우를 사용하여 생성된 염기서열에 대한 RABV-GLUE의 주요 및 보조 클래드 할당과 MADDOG의 계보 할당. 이 표를 다운로드하려면 여기를 클릭하십시오.
보충 파일 1: Primer scheme design and optimization, and amplicon read depth analysis. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 2: 계산 설정 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 3: RABV WGS 프로토콜 워크시트 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
접근 가능한 RABV, 나노포어 기반, 전체 게놈 시퀀싱 워크플로우는 Brunker et al.61에 의해 ARTIC 네트워크(46)의 자원을 사용하여 개발되었습니다. 여기에서는 샘플에서 시퀀스에서 해석까지의 전체 단계와 함께 업데이트된 워크플로우를 제시합니다. 워크플로우는 전체 게놈 염기서열 분석을 위한 뇌 조직 샘플 준비에 대해 자세히 설명하고, 판독을 처리하고 합의 염기서열을 생성하는 생물정보학 파이프라인을 제시하며, 계통 할당을 자동화하고 계통발생학적 맥락을 결정하기 위한 두 가지 광견병 관련 도구를 강조합니다. 업데이트된 워크플로우는 또한 적절한 계산 및 실험실 작업 공간 설정에 대한 포괄적인 지침을 제공하며, 다양한 상황(리소스 부족 설정 포함)에서의 구현에 대한 고려 사항을 제공합니다. 우리는 게놈 감시 능력이 없거나 제한된 4개의 RABV 풍토병 LMIC에서 학술 및 연구 기관 환경 모두에서 워크플로우를 성공적으로 구현했음을 입증했습니다. 워크플로는 다양한 설정에서 애플리케이션에 대한 복원력이 입증되었으며 다양한 전문 지식을 가진 사용자가 이해할 수 있습니다.
RABV 염기서열분석을 위한 이 워크플로우는 공개적으로 사용 가능한 가장 포괄적인 프로토콜(샘플-시퀀스-해석 단계 포함)이며 시작 및 운영 비용을 모두 절감하기 위해 특별히 조정되었습니다. 나노포어 기술을 사용한 라이브러리 준비 및 염기서열분석에 필요한 시간과 비용은 Illumina61과 같은 다른 플랫폼에 비해 크게 감소하며, 지속적인 기술 개발로 염기서열 품질과 정확도가 Illumina62와 비교할 수 있도록 개선되고 있습니다.
이 프로토콜은 다양한 저리소스 컨텍스트에서 복원력을 갖도록 설계되었습니다. 핵심 프로토콜과 함께 제공되는 문제 해결 및 수정 지침을 참조하여 사용자가 필요에 맞게 워크플로를 조정할 수 있습니다. 워크플로우에 사용자 친화적인 생물정보학 도구를 추가하는 것은 기존 프로토콜에 대한 주요 개발을 구성하며, 최소한의 사전 생물정보학 경험을 가진 사용자가 로컬 컨텍스트에서 염기서열 데이터를 해석하기 위해 적용할 수 있는 빠르고 표준화된 방법을 제공합니다. 현장에서 이를 수행할 수 있는 능력은 집중적이고 장기적인 기술 교육 투자가 필요한 특정 프로그래밍 및 계통발생 기술의 필요성에 의해 제한되는 경우가 많습니다. 이 기술은 염기서열 데이터를 철저하게 해석하는 데 중요하지만, 핵심 전문 지식이 습식 실험실 기반일 수 있는 현지 "염기서열 분석 챔피언"을 수용하여 데이터를 해석하고 소유권을 가질 수 있도록 하기 위해서는 기본적이고 접근 가능한 해석 도구도 똑같이 바람직합니다.
이 프로토콜은 여러 국가에서 수년 동안 수행되어 왔기 때문에 이제 커버리지를 개선하고 누적된 다양성을 처리하기 위해 멀티플렉스 프라이머 체계를 최적화하는 방법에 대한 지침을 제공할 수 있습니다. 또한 사용자가 비용 효율성을 개선하거나 특정 지역에서 조달을 용이하게 할 수 있도록 하기 위한 노력이 이루어졌는데, 이는 일반적으로 분자 접근법(63)의 지속 가능성에 대한 도전이다. 예를 들어, 아프리카(탄자니아, 케냐 및 나이지리아)에서는 어댑터 연결 단계에서 현지 공급업체에서 더 쉽게 구할 수 있고 다른 연결 시약에 비해 저렴한 대안인 blunt/TA ligase master mix를 선택했습니다.
경험에 비추어 볼 때, 샘플당 및 실행당 비용을 줄이는 방법에는 여러 가지가 있습니다. 실행당 샘플 수를 줄이면(예: 24개 샘플에서 12개 샘플로) 여러 번 실행에 걸쳐 플로우 셀의 수명을 연장할 수 있는 반면, 실행당 샘플 수를 늘리면 시간과 시약이 극대화됩니다. 우리는 염기서열분석 실행 3회 중 1회에 대해 플로우 셀을 세척하고 재사용할 수 있었으며, 이를 통해 추가로 55개의 시료를 염기서열분석할 수 있었습니다. 사용 직후 플로우 셀을 세척하거나, 가능하지 않은 경우 매 실행 후 폐기물 채널에서 폐액을 제거하면 두 번째 실행에 사용할 수 있는 기공 수를 보존하는 것 같았습니다. 플로우 셀에서 사용할 수 있는 초기 공극 수를 고려하여, 한 번의 실행을 최적화하여 특정 플로우 셀에서 실행할 샘플 수를 계획할 수도 있습니다.
워크플로는 가능한 한 포괄적이도록 하는 것을 목표로 하지만 자세한 지침과 표지판이 있는 리소스가 추가되어 절차가 여전히 복잡하고 신규 사용자에게 어려울 수 있습니다. 사용자는 대면 교육 및 지원을 받는 것이 좋으며, 이상적으로는 로컬에서 또는 외부 공동 작업자를 통해 찾을 수 있습니다. 예를 들어, 필리핀에서는 ONT를 사용한 SARS-CoV-2 게놈 감시를 위한 지역 실험실 내 역량 구축 프로젝트를 통해 RABV 염기서열 분석으로 쉽게 이전할 수 있는 의료 진단 전문가들의 핵심 역량을 개발했습니다. SPRI 비드 클린업과 같은 중요한 단계는 실습 교육 없이는 마스터하기 어려울 수 있으며, 비효율적인 클린업은 플로우 셀을 손상시키고 실행을 손상시킬 수 있습니다. 샘플 오염은 실험실에서 앰플리콘을 처리할 때 항상 주요 관심사이며 제거하기 어려울 수 있습니다. 특히, 시료 간의 교차 오염은 실행 후 생물정보학 중에 검출하기가 매우 어렵습니다. 깨끗한 작업 표면 유지, PCR 전후 영역 분리, 음성 대조군 통합과 같은 우수한 실험실 기술 및 관행은 품질 관리를 보장하는 데 필수적입니다. 나노포어 시퀀싱 개발의 빠른 속도는 일상적인 RABV 게놈 감시의 장점과 단점 모두입니다. nanopore의 정확도, 접근성 및 의정서 레퍼토리에 계속되는 개선은 그것의 신청을 위한 범위를 넓히고 개량합니다. 그러나 동일한 개발로 인해 표준 운영 절차와 생물정보학 파이프라인을 유지하는 것이 어려워집니다. 이 프로토콜에서는 더 오래된에서 현재 나노포어 라이브러리 준비 키트(재료 테이블)로의 전환을 지원하는 문서를 제공합니다.
LMIC에서 염기서열분석의 일반적인 장애물은 비용뿐만 아니라 적시에 소모품을 조달할 수 있는 능력(특히 조달 팀과 공급업체에 비교적 새로운 염기서열분석 시약) 및 계산 리소스, 안정적인 전력과 인터넷에 대한 액세스를 포함한 접근성입니다. 이 워크플로우의 기초로 휴대용 나노포어 시퀀싱 기술을 사용하면 이러한 접근성 문제의 많은 부분을 해결할 수 있으며, 당사는 다양한 설정에서 프로토콜의 사용을 시연하여 국내에서 전체 프로토콜 및 분석을 수행했습니다. 물론 적시에 장비를 조달하고 소모품을 시퀀싱하는 것은 여전히 어려운 과제이며, 많은 경우 영국에서 시약을 운반하거나 배송해야 했습니다. 그러나 일부 지역에서는 조달 프로세스를 간소화하고 병원체 유전체학의 적용을 정상화하기 시작한 SARS-CoV-2 염기서열 분석(예: 필리핀)에 대한 투자의 혜택을 받아 시약을 현지 공급 경로에 전적으로 의존할 수 있었습니다.
안정적인 인터넷 연결의 필요성은 일회성 설치로 최소화됩니다. 예를 들어, GitHub 리포지토리, 소프트웨어 다운로드 및 나노포어 시퀀싱 자체는 실행을 시작하기 위해 인터넷 액세스만 필요하거나(전체 액세스가 아님) 회사의 동의하에 완전히 오프라인으로 수행할 수 있습니다. 모바일 데이터를 사용할 수 있는 경우 휴대폰을 랩톱의 핫스팟으로 사용하여 실행 기간 동안 연결을 끊기 전에 시퀀싱 실행을 시작할 수 있습니다. 샘플을 일상적으로 처리할 때 데이터 스토리지 요구 사항이 빠르게 증가할 수 있으며 이상적으로는 데이터가 서버에 저장됩니다. 그렇지 않으면 솔리드 스테이트 드라이브(SSD) 하드 드라이브를 소싱하는 데 상대적으로 저렴합니다.
LMIC에서 유전체 감시에 여전히 장벽이 있다는 것을 알고 있지만, 유전체학 접근성 및 전문성 구축에 대한 투자가 증가함에 따라(예: Africa Pathogen Genomics Initiative [Africa PGI])64 이러한 상황이 개선될 것으로 예상됩니다. 게놈 감시는 팬데믹 대비에 매우 중요하며6 RABV와 같은 풍토성 병원체의 게놈 감시를 루틴화함으로써 역량을 구축할 수 있다. SARS-CoV-2 팬데믹 기간 동안 강조된 염기서열 분석 능력의 글로벌 격차는 이러한 구조적 불평등을 해결하기 위한 촉매 변화의 동인이 되어야 합니다.
접근 가능한 생물정보학 도구를 포함하여 RABV에 대한 이 샘플-서열-해석 워크플로우는 2030년까지 개 매개 광견병으로 인한 인간 사망 제로를 목표로 하는 통제 조치를 안내하고 궁극적으로 RABV 변종을 제거하는 데 사용할 수 있는 잠재력을 가지고 있습니다. 관련 메타데이터와 결합하여, 이 프로토콜로부터 생성된 게놈 데이터는 발병 조사 중 그리고 국가 또는 지역에서 순환하는 계통의 식별에 있어서 신속한 RABV 특성화를 용이하게 한다60,61,65. 우리는 주로 개 매개 광견병의 예를 사용하여 파이프라인을 설명합니다. 그러나 이 워크플로는 야생 동물 광견병에 직접 적용할 수 있습니다. 이러한 전이성과 저렴한 비용은 질병 관리 및 통제를 개선하기 위해 광견병뿐만 아니라 다른 병원체에 대해서도 일상적인 염기서열 분석을 쉽게 사용할 수 있도록 하는 문제를 최소화합니다46,66,67.
공개
저자는 공개할 것이 없습니다.
감사의 말
이 연구는 Wellcome [207569/Z/17/Z, 224670/Z/21/Z], 의학 연구 위원회[MR/R025649/1] 및 필리핀 과학기술부(DOST)의 Newton 자금 지원, COVID-19에 대한 UK Research and Innovation Global 노력 [MR/V035444/1], University of Glasgow Institutional Strategic Support Fund [204820], Medical Research Council New Investigator Award (KB) [MR/X002047/1], 및 국제 파트너십 개발 기금, DOST British Council-Philippines 학생(CB), National Institute for Health Research[17/63/82] GemVi 장학금(GJ) 및 MVLS DTP(KC) [125638-06], EPSRC DTP(RD) [EP/T517896/1] 및 Wellcome IIB DTP(HF) [218518/Z/19/Z). Daniel Streicker, Alice Broos, Elizabeth Miranda, DVM, Daria Manalo, DVM, Thumbi Mwangi, Kennedy Lushasi, Charles Kayuki, Jude Karlo Bolivar, Jeromir Bondoc, Esteven Balbin, Ronnel Tongohan, Agatha Ukande, Davis Kuchaka, Mumbua Mutunga, Lwitiko Sikana, Anna Czupryna 등 이 작업을 지원해 주신 동료 및 협력자에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| Brand name | |||
| Software | |||
| Sequencing software (MinKnow) | Oxford Nanopore Technologies | https://community.nanoporetech.com/downloads | |
| Bioinformatics tool kit (Guppy) | Oxford Nanopore Technologies | https://community.nanoporetech.com/docs/prepare/library_prep_protocols/Guppy-protocol/v/gpb_2003_v1_revao_14dec2018 | |
| Equipment | |||
| Thermal cycler (miniPCR™ mini16 thermal cycler) | Cambio | MP-QP-1016-01 | |
| Homogenizer (Precellys Evolution Touch Homogenizer) | Bertin Instruments | EQ02520-300 | |
| Cold Racks (0.2-0.5mL) (PCR Mini-cooler with transparent lid) | BRAND | 781260 | |
| Pipettor | |||
| (Pipetman L Fixed F1000L, 1000 uL) | Gilson | SKU: FA10030 | |
| (Pipetman L Fixed F100L, 100 uL) | Gilson | SKU: FA10024 | |
| (Pipetman L Fixed F10L, 10 uL) | Gilson | SKU: FA10020 | |
| (Pipetman L Fixed F1L, 1 uL) | Gilson | SKU: FA10025 | |
| (Pipetman L Fixed F20L, 20 uL) | Gilson | SKU: FA10021 | |
| (Pipetman L Fixed F250L, 250 uL) | Gilson | SKU: FA10026 | |
| Fluorometer (Qubit 4 Fluorometer) | Thermofisher scientific/Fisher scientific | Q33238 | |
| Laptop (Any brand with ~2 GB of drive space, minimum of 512 GB storage space, msi installer [GPU]) | |||
| Microcentrifuge (Refrigerated centrifuge) | Thermofisher scientific/Fisher scientific | 75004081 | |
| Vortex mixer (Basic vortex mixer) | Thermofisher scientific/Fisher scientific | 88882011 | |
| Magnetic rack (DynaMag -2 Magnet) | Thermofisher scientific/Fisher scientific | 12321D | |
| Sequencing device (MinION) | Oxford Nanopore Technologies | MinION Mk1B | |
| RNA Extraction | |||
| RNA extraction kit (Qiagen RNEasy Mini Kit 250) | Qiagen | 74106 | |
| RNA stabilizing reagents | |||
| (RNA later) | Invitrogen | AM7020 | |
| (DNA/RNA Shield) | Zymo Research | R1100-50 | |
| PCR | |||
| Nuclease-free Water (Nuclease-free Water [not DEPC-treated]) | Thermofisher scientific/Fisher scientific | AM9937 | |
| Master mix for first strand cDNA synthesis (LunaScript RT SuperMix Kit) | New England Biolabs | E3010S | |
| DNA amplification master mix (Q5® Hot Start High-Fidelity 2X Master Mix [NEB]) | New England Biolabs | M0494L | |
| Primer (Scheme) (Custom DNA oligos) | Invitrogen | ||
| SPRI Bead Clean-up | |||
| SPRI beads (Aline Biosciences PCR Clean DX ) | Cambio | AL-AC1003-50 | |
| Ethanol, Pure Absolute, >99.8% (GC) [Riedel-De Haen] | Merck | 818760 | |
| Short Fragment buffer (SFB expansion pack) | Oxford Nanopore Technologies | EXP-SFB001 | |
| DNA Quantification | |||
| DNA quantification kit (Qubit® dsDNA HS Assay Kit) | Thermofisher scientific/Fisher scientific | Q32854 | |
| DNA quantification assay tubes (Qubit™ Assay Tubes) | Thermofisher scientific/Fisher scientific | Q32856 | |
| End Prep and barcoding (Qubit™ Assay Tubes) | |||
| End Prep master mix (NEBNext Ultra End Repair/dA-Tailing Module) | New England Biolabs | E7546L | |
| Barcoding kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 | |
| (Native Barcoding Expansion 1-12) | EXP-NBD104 | ||
| (Native Barcoding Expansion 13-24) | EXP-NBD114 | ||
| (Native Barcoding Expansion 96) | EXP-NBD196 | ||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 | |
| (not compatible) | (not compatible) | ||
| (Native Barcoding Kit 24 V14) | SQK-NBD114.24 | ||
| (Native Barcoding Kit 96 V14) | SQK-NBD114.96 | ||
| Ligation mastermix (Blunt/TA Ligase Master Mix) | New England Biolabs | M0367S | |
| Adapter Ligation | |||
| Adapter ligation master mix | |||
| (NEBNext Quick Ligation Module) | New England Biolabs | E6056S | |
| (NEBNext Ultra II Ligation Module) | New England Biolabs | E7595S | |
| (Blunt/TA Ligase Master Mix) | New England Biolabs | M0367S | |
| Adapter mix | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 EXP-AMII001 | |
| (Adapter Mix II [AMII]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 EXP-NBA114 | |
| (Native adapter [NA]) | |||
| Sequencing | |||
| Flowcell priming kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 EXP-FLP002 | |
| (Flush Buffer [FB]) | |||
| (Flush Tether [FT]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 EXP-FLP004 | |
| (Flow Cell Flush [FCF]) | |||
| (Flow Cell Tether [FCT]) | |||
| Ligation Sequencing Kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 SQK-LSK109 | |
| Adapter Mix (Adapter Mix [AMX]) | |||
| Ligation Buffer (Ligation buffer [LNB]) | |||
| Short Fragment Buffer (Short Fragment buffer [SFB]) | |||
| Sequencing Buffer (Sequencing Buffer [SQB]) | |||
| Elution Buffer (Elution buffer [EB]) | |||
| Loading Beads (Loading Beads [LB]) | |||
| Sequencing Tether (Sequencing Tether [SQT]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 SQK-LSK114 | |
| Adapter Mix (Ligation Adapter [LA]) | |||
| Ligation Buffer (Ligation buffer [LNB]) | |||
| Short Fragment Buffer (Short Fragment buffer [SFB]) | |||
| Sequencing Buffer (Sequencing Buffer [SB]) | |||
| Elution Buffer (Elution buffer [EB]) | |||
| Loading Beads (Loading Beads [LIB]) | |||
| Sequencing Tether (Flow Cell Tether [FCT]) | |||
| Library solution (Library solution [LIS]) | |||
| Flush buffer (Flow Cell Flush [FCF]) | |||
| Flow Cell | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 FLO-MIN106D | |
| (Flow Cell [R9.4.1]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 FLO-MIN114 | |
| (Flow Cell [R10.4.1]) | |||
| Flow Cell wash | |||
| Flowcell wash kit (Flow cell wash kit) | Oxford Nanopore Technologies | EXP-WSH004 | |
| Consummables | |||
| Surface decontaminant | |||
| (DNA Away Surface Decontaminant, Squeeze Bottle [Molecular Bio]) | Thermofisher scientific/Fisher scientific | 7010PK | |
| (RNase Away Surface Decontaminant, Bottle [Molecular Bio]) | Thermofisher scientific/Fisher scientific | 7002PK | |
| PCR 8-Tube Strip 0.2ml, individual cap (PCR 8-Tube Strip 0.2ml, with Individual attached Flat Caps, Sterile, DNAse/RNAse, Pyrogen Free,Natural [Greiner]) | Greiner | 608281 | |
| PCR Tube 0.2ml (PCR Tube 0.2ml, Natural [Domed Cap] Bagged in 500s, Non-Sterile [Greiner]) | Greiner | 671201 | |
| 1000µL Filter Tips (500) (Stacked 1000µL Filter Tips [500]) | Thermofisher scientific/Fisher scientific | 11977724 | |
| 100µL Filter Tips (1000) | Thermofisher scientific/Fisher scientific | 11947724 | |
| 10µL Filter Tips (1000) (Stacked 100µL Filter Tips [1000]) | Thermofisher scientific/Fisher scientific | 11907724 | |
| Reinforced tubes tubes (2ml) with screw caps and o-rings (Fisherbrand™ Bulk tubes) | Thermofisher scientific/Fisher scientific | 15545809 | |
| Microcentrifuge tube (1.5ml) (1.5 ml Eppendorf Tubes [500]) | Eppendorf | 1229888 | |
| DNA LoBind Tubes (1.5ml) (DNA LoBind Tubes) | Thermofisher scientific/Fisher scientific | 10051232 | |
| Cryobabies labels | |||
| Gloves (S/M/L) | |||
| Paper towel |
참고문헌
- Rupprecht, C. E. Rhabdoviruses: rabies virus. Medical Microbiology. , (1996).
- Rabies. World Health Organization Available from: https://www.who.int/news-room/fact-sheets/detail/rabies (2023)
- WHO Expert Consultation on Rabies: WHO TRS N°1012. World Health Organization Available from: https://www.who.int/publications-detail-redirect/WHO-TRS-1012 (2018)
- Benavides, J. A., et al. Defining new pathways to manage the ongoing emergence of bat rabies in Latin America. Viruses. 12 (9), 1002 (2020).
- Hampson, K. Estimating the global burden of endemic canine rabies. PLoS Neglected Tropical Diseases. 9 (4), e0003709 (2015).
- Global genomic surveillance strategy for pathogens with pandemic and epidemic potential, 2022-2032. World Health Organization Available from: https://www.who.int/publications-detail-redirect/978924004679 (2022)
- Tsai, K. J., et al. Emergence of a sylvatic enzootic formosan ferret badger-associated rabies in Taiwan and the geographical separation of two phylogenetic groups of rabies viruses. Veterinary Microbiology. 182, 28-34 (2016).
- Chiou, H. -. Y., et al. Molecular characterization of cryptically circulating rabies virus from ferret badgers, Taiwan. Emerging Infectious Diseases. 20 (5), 790-798 (2014).
- Sabeta, C. T., Mansfield, K. L., McElhinney, L. M., Fooks, A. R., Nel, L. H. Molecular epidemiology of rabies in bat-eared foxes (Otocyon megalotis) in South Africa. Virus Research. 129 (1), 1-10 (2007).
- Scott, T. P. Complete genome and molecular epidemiological data infer the maintenance of rabies among kudu (Tragelaphus strepsiceros) in Namibia. PLoS One. 8 (3), e58739 (2013).
- Lembo, T., et al. Exploring reservoir dynamics: a case study of rabies in the Serengeti ecosystem. The Journal of Applied Ecology. 45 (4), 1246-1257 (2008).
- Coetzee, P., Nel, L. H. Emerging epidemic dog rabies in coastal South Africa: a molecular epidemiological analysis. Virus Research. 126 (1-2), 186-195 (2007).
- Ou de Munnink, B. B. First molecular analysis of rabies virus in Qatar and clinical cases imported into Qatar, a case report. International Journal of Infectious Diseases. 96, 323-326 (2020).
- Smith, J., et al. Case report: Rapid ante-mortem diagnosis of a human case of rabies imported into the UK from the Philippines. Journal of Medical Virology. 69, 150-155 (2003).
- Mahardika, G. N. K., et al. Phylogenetic analysis and victim contact tracing of rabies virus from humans and dogs in Bali, Indonesia. Epidemiology and Infection. 142 (6), 1146-1154 (2014).
- Tohma, K., et al. Molecular and mathematical modeling analyses of inter-island transmission of rabies into a previously rabies-free island in the Philippines. Infection, Genetics and Evolution. 38, 22-28 (2016).
- Tohma, K., et al. Phylogeographic analysis of rabies viruses in the Philippines. Infection, Genetics and Evolution. 23, 86-94 (2014).
- Saito, M., et al. Genetic diversity and geographic distribution of genetically distinct rabies viruses in the Philippines. PLoS Neglected Tropical Diseases. 7 (4), e2144 (2013).
- Biek, R., Henderson, J. C., Waller, L. A., Rupprecht, C. E., Real, L. A. A high-resolution genetic signature of demographic and spatial expansion in epizootic rabies virus. Proceedings of the National Academy of Sciences. 104 (19), 7993-7998 (2007).
- Reddy, G. B., et al. Molecular characterization of Indian rabies virus isolates by partial sequencing of nucleoprotein (N) and phosphoprotein (P) genes. Virus Genes. 43, 13-17 (2011).
- David, D., Dveres, N., Yakobson, B. A., Davidson, I. Emergence of dog rabies in the northern region of Israel. Epidemiology and Infection. 137 (4), 544-548 (2009).
- Benjathummarak, S. Molecular genetic characterization of rabies virus glycoprotein gene sequences from rabid dogs in Bangkok and neighboring provinces in Thailand, 2013-2014. Archives of Virology. 161 (5), 1261-1271 (2016).
- Denduangboripant, J., et al. Transmission dynamics of rabies virus in Thailand: implications for disease control. BMC Infectious Diseases. 5, 52 (2005).
- Talbi, C., et al. Phylodynamics and human-mediated dispersal of a zoonotic virus. PLoS Pathogens. 6 (10), e1001166 (2010).
- Bourhy, H., et al. Revealing the micro-scale signature of endemic zoonotic disease transmission in an African urban setting. PLoS Pathogens. 12 (4), e1005525 (2016).
- Zinsstag, J., et al. Vaccination of dogs in an African city interrupts rabies transmission and reduces human exposure. Science Translational Medicine. 9 (421), (2017).
- Yakovleva, A., et al. Tracking SARS-COV-2 variants using Nanopore sequencing in Ukraine in 2021. Scientific Reports. 12, 15749 (2022).
- Mannsverk, S., et al. SARS-CoV-2 variants of concern and spike protein mutational dynamics in a Swedish cohort during 2021, studied by Nanopore sequencing. Virology Journal. 19, 164 (2022).
- Soufi, M., et al. Fast and easy nanopore sequencing workflow for rapid genetic testing of familial Hypercholesterolemia. Frontiers in Genetics. 13, 836231 (2022).
- Cabibbe, A. M. Application of targeted next-generation sequencing assay on a portable sequencing platform for culture-free detection of drug-resistant tuberculosis from clinical samples. Journal of Clinical Microbiology. 58 (10), 00632 (2020).
- Xu, Y., et al. Nanopore metagenomic sequencing of influenza virus directly from respiratory samples: diagnosis, drug resistance and nosocomial transmission, United Kingdom, 2018/19 influenza season. Euro Surveillance. 26 (27), 2000004 (2021).
- Stubbs, S. C. B., et al. Assessment of a multiplex PCR and Nanopore-based method for dengue virus sequencing in Indonesia. Virology Journal. 17, 24 (2020).
- Croville, G., et al. Rapid whole-genome based typing and surveillance of avipoxviruses using nanopore sequencing. Journal of Virological Methods. 261, 34-39 (2018).
- Theuns, S., et al. Nanopore sequencing as a revolutionary diagnostic tool for porcine viral enteric disease complexes identifies porcine kobuvirus as an important enteric virus. Scientific Reports. 8, 9830 (2018).
- O'Donnell, V. K., et al. Rapid sequence-based characterization of African swine fever virus by use of the Oxford Nanopore MinION sequence sensing device and a companion analysis software tool. Journal of Clinical Microbiology. 58, 01104-01119 (2019).
- Brito, A. F. Global disparities in SARS-CoV-2 genomic surveillance. Nature Communications. 13, 7003 (2022).
- ONT login/register. Oxford Nanopore Technology Available from: https://nanoporetech.com/login-register (2023)
- Software Downloads. Oxford Nanopore Technology Available from: https://community.nanoporetech.com/downloads (2023)
- . GitHub Available from: https://github.com/ (2023)
- . Artic-rabv Available from: https://github.com/kirstyn/artic-rabv (2022)
- . MADDOG: Method for Assignment, Definition and Designation of Global Lineages Available from: https://github.com/KathrynCampbell/MADDOG (2022)
- RABV-GLUE. Centre for Virus Research Available from: https://github.com/KathrynCampbell/MADDOG (2022)
- Itokawa, K., Sekizuka, T., Hashino, M., Tanaka, R., Kuroda, M. Disentangling primer interactions improves SARS-CoV-2 genome sequencing by multiplex tiling PCR. PLoS ONE. 15 (9), e0239403 (2020).
- Davis, M. W., Jorgensen, E. M. ApE, A plasmid editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 818619 (2022).
- Döring, M., Pfeifer, N. . openPrimeR: Multiplex PCR primer design and analysis. , (2023).
- Quick, J. Multiplex PCR method for MiniON and Illumina sequencing of Zika and other virus genomes directly from clinical samples. Nature Protocols. 12 (6), 1261-1276 (2017).
- Laboratory Techniques in Rabies. World Health Organization Available from: https://apps.who.int/iris/bitstream/handle/10665/310836/9789241515153-eng.pdf (2018)
- Lembo, T. Partners for Rabies Prevention. The blueprint for rabies prevention and control: a novel operational toolkit for rabies elimination. PLoS Neglected Tropical Diseases. 6 (2), e1388 (2012).
- Terrestrial Manual Online Access. World Organization for Animal Health Available from: https://www.woah.org/en/what-we-do/standards/codes-and-manuals/terrestrial-manual-online-access/ (2023)
- Mauti, S. Field postmortem rabies rapid immunochromatographic diagnostic test for resource-limited settings with further molecular applications. Journal of Visualized Experiments. (160), e60008 (2020).
- Patrick, E., et al. Enhanced rabies surveillance using a direct rapid immunohistochemical test. Journal of Visualized Experiments. (146), e59416 (2019).
- Lembo, T., et al. Evaluation of a direct, rapid immunohistochemical test for rabies diagnosis. Emerging Infectious Diseases. 12 (2), 310-313 (2006).
- Marston, D. A., et al. Pan-lyssavirus real time RT-PCR for rabies diagnosis. Journal of Visualized Experiments. (149), e59709 (2019).
- . DNA quantification using the Qubit fluorometer Available from: https://www.protocols.io/view/dna-quantification-using-the-qubit-fluorometer-bc6vize6 (2020)
- . DNA quantification using the Quantus fluorometer Available from: https://www.protocols.io/view/dna-quantification-using-the-quantus-fluorometer-7pzhmp6 (2020)
- Guppy protocol. Nanopore Community Available from: https://community.nanoporetech.com/protocols/Guppy-protocol/v/gpb_2003_v1_revaq_14dec2018 (2023)
- Flow Cell Wash Kit (EXP-WSH004). Nanopore Community Available from: https://community.nanoporetech.com/protocols/flow-cell-wash-kit-exp-wsh004/v/wfc_9120_v1_revh_08dec2020 (2023)
- . Core Pipeline - arctic pipeline Available from: https://artic.readthedocs.io/en/latest/minion/ (2023)
- . Samtools Available from: https://www.htslib.org (2023)
- Campbell, K., et al. Making genomic surveillance deliver: A lineage classification and nomenclature system to inform rabies elimination. PLoS Pathogens. 18 (5), e1010023 (2022).
- Brunker, K., et al. Rapid in-country sequencing of whole virus genomes to inform rabies elimination programmes. Wellcome Open Research. 5, 3 (2020).
- Bull, R. A., et al. Analytical validity of nanopore sequencing for rapid SARS-CoV-2 genome analysis. Nature Communications. 11, 6272 (2020).
- Okeke, I. N., Ihekweazu, C. The importance of molecular diagnostics for infectious diseases in low-resource settings. Nature Reviews. Microbiology. 19 (9), 547-548 (2021).
- Inzaule, S. C., Tessema, S. K., Kebede, Y., Ouma, A. E. O., Nkengasong, J. N. Genomic-informed pathogen surveillance in Africa: opportunities and challenges. The Lancet Infectious Diseases. 21 (9), 281-289 (2021).
- Kennedy, L. Integrating contact tracing and whole-genome sequencing to track the elimination of dog-mediated rabies: an observational and genomic study. eLife. , (2023).
- Pallerla, S. R. Diagnosis of pathogens causing bacterial meningitis using Nanopore sequencing in a resource-limited setting. Annals of Clinical Microbiology and Antimicrobials. 21, 39 (2022).
- Quick, J. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228-232 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유