Studying Muscle Transcriptional Dynamics at Single-molecule Scales in Drosophila (초파리의 단일 분자 스케일에서 근육 전사 역학 연구)
요약
초파리 는 근육 형성을 조절하는 주요 분자를 연구하기 위한 잘 정립된 모델입니다. 그러나 현재의 방법은 mRNA 전사 역학 및 세포융합 내 공간 분포를 결정하기에 충분하지 않습니다. 이러한 한계를 해결하기 위해 RNA 형광 in situ hybridization 방법을 최적화하여 단일 분자 규모에서 mRNA를 검출하고 정량화할 수 있습니다.
초록
골격근은 힘을 생성하고 신체 움직임을 가능하게 하는 많은 묶인 근섬유로 구성된 큰 세포융합체입니다. 초파리 는 근육 생물학을 연구하는 고전적인 모델입니다. 초파리 유전학과 고급 오믹스 접근법의 조합은 근육 형태 형성 및 재생을 조절하는 주요 보존 분자의 식별로 이어졌습니다. 그러나, 이들 분자의 전사 역학과 세포융합체 내 메신저 RNA의 공간적 분포는 기존의 방법으로 평가할 수 없습니다. 여기서는 기존의 단일 분자 RNA 형광 in situ hybridization(smFISH) 방법을 최적화하여 성인 비행 근육과 근육 줄기 세포 내에서 개별 mRNA 분자를 검출하고 정량화할 수 있도록 했습니다. 개념 증명(PoC)으로 두 가지 진화적 보존 전사 인자인 Mef2 및 Zfh1/Zeb의 mRNA 발현 및 분포를 분석했습니다. 우리는 이 방법이 근육 전구체 세포, 성인 근육 및 근육 줄기 세포의 두 전사체 모두에 대해 단일 mRNA 분자를 효율적으로 검출하고 정량화할 수 있음을 보여줍니다.
서문
성인 골격근은 수축 특성이 움직임을 생성하는 분화된 다핵 근섬유로 구성되어 있습니다. 근육 성장, 유지 및 재생은 배아 발달 중에 지정된 근육 전구 세포와 근육 줄기 세포(MuSC)에 의존합니다1. 근육 생성 프로그램은 일련의 핵심 근육 전사 인자(TF)(예: Pax3/7, MYOD, Mef2 및 ZEB)2,3,4에 의해 미세하게 제어됩니다. 근육 생물학을 조절하는 분자 메커니즘을 해독하는 것은 근본적인 근육학 관련 질문을 해명하고 근육 퇴행성 질환을 치료하는 데 사용할 수 있는 치료법을 찾는 데 중요합니다.
초파리는 근형성을 연구하기 위한 유전적 모델로서 오랜 역사를 가지고있다 5. 최근 근육 재생 6,7,8을 조사하는 새로운 모델로 등장했습니다. 근육 구조와 핵심 근육 생성 프로그램은 파리와 포유류 사이에서 매우 잘 보존되어 있습니다. 예를 들어, TF Mef2 및 Zfh1/ZEB는 근육 발달 및 재생을 조절하는 보존 기능을 가지고있습니다 3,9,10,11. 간접 비행 근육(Indirect Flight Muscles, IFM)과 같은 성체 초파리 근육(Adult Drosophila muscle)은 성체 근육 전구 근육(Adult Muscle Progenitors, AMP)으로 알려진 MuSC의 특정 집단으로 형성됩니다12. 이러한 AMP는 배아 발생 중에 지정되며 날개 및 다리 디스크와 같은 상피 구조와 관련이 있습니다. 배아 및 유충 단계에 걸쳐, AMP는 IFM을 형성하기 위해 분화 및 융합에 관여할 때 변태될 때까지 미분화 상태로 남아 있습니다13,14. TF Spalt major(spalt, salm)는 비행 근육 발달 중에 발현되며 구조적 정체성을 결정하는 데 필요하다15. Mef2는 성인 근육 형성에 필수적인 또 다른 중요한 근육 생성 TF입니다16,17. 이는 AMP에서 발현되고 성인 IFM(10,18)에서 유지됩니다. 대부분의 AMP는 기능적 근육으로 분화하지만, 부분집합은 분화를 벗어나 성인 MuSC를 형성한다11. 척추동물과 유사하게, TF Zfh1/ZEB는 성체 MuSC의 조기 분화를 방지하고 줄기 9,10을 유지하는 데 필요합니다.
유전자 발현 역동성은 각종 근육 생물학 과정19,20를 통제하기 위하여 입증되었다. 고처리량 단일 세포 및 단일 핵 RNA 시퀀싱 기술의 출현으로 이러한 전사 역학21,22에 대한 포괄적인 탐구가 가능해졌습니다. 이러한 접근법의 한 가지 주목할 만한 한계는 다핵 근육 섬유에서 mRNA 분자의 공간적 분포를 제공할 수 없다는 것입니다. 이러한 특징은 두 가지 유형의 mRNA 바디를 나타내는 단일 분자 형광 현장 교잡화(smFISH)로 조사할 수 있습니다: 1. 세포질에 퍼져 있고 성숙한 RNA를 나타내는 개별 mRNA 분자 및 2. 최대 2개의 밝은 핵 병소는 초기 전사체에 해당하며 전사 활성 대립유전자23을 나타냅니다. 따라서 smFISH는 개별 mRNA 분자 정량화를 위해 선택되는 방법으로, 공간 분포를 조사하고 유전자 전사 역학의 스냅샷을 제공합니다.
smFISH 분석법은 표적 전사체를 보완하도록 특별히 설계된 짧은 형광 올리고뉴클레오티드 프로브 세트에 의존합니다. 어닐링 시 공초점 현미경24을 사용하여 단일 분자 수준에서 mRNA를 검출할 수 있는 고강도 점 광원을 생성합니다. 이 방법은 포유류 근육 조직(19,20)을 포함한 다양한 세포 유형에 점점 더 많이 적용되고 있다. 그러나 다른 동물 모델과 마찬가지로 초파리에서 성인 근육 유전자 발현에 대한 대부분의 지식은 mRNA 분자의 공간적 위치에 대한 정량적 정보가 부족한 벌크 분자 분석에서 파생됩니다. 여기서 우리는 근육 전구체 세포와 성인 초파리 근육23,25에서 smFISH를 수행하는 방법을 최적화했습니다. 이 프로토콜에는 핵 분할과 mRNA 계수 및 국소화를 위한 완전 자동화된 분석 파이프라인이 포함되어 있습니다.
프로토콜
1. 날개 디스크 및 성인 근육 해부 및 준비
참고: RNAse 억제제 용액으로 해부 벤치와 모든 해부 기구를 청소하십시오(재료 표). 실험실 장갑은 전체 실험 절차 동안 착용해야 합니다.
- 윙 디스크
- L3 단계별 유충을 차가운 1x 인산염 완충 식염수(PBS)에 넣습니다(그림 1A).
- 1x PBS로 유충을 3번 헹구고 이 용액에 5분 동안 보관합니다.
- 두 쌍의 날카로운 집게를 사용하여 앞쪽에서 유충을 잡고 나머지 신체 조직(약 2/3)을 당겨 버립니다.
- 한 쌍의 집게로 유충의 처음 1/3을 잡고 다른 쌍으로 입 고리를 잡습니다. 완전히 뒤집힐 때까지 유충 내부의 입 고리를 집어넣습니다.
참고: 분석을 위한 적절한 샘플 크기를 보장하기 위해 실험당 약 30마리의 유충을 해부합니다. - 날개 디스크가 유충의 양쪽을 따라 달리는 기관의 두 가지 주요 가지와 밀접하게 관련되어 있음을 관찰하십시오.
- 다른 모든 유충 구성 요소(다리 디스크 및 뇌)를 제거하여 기관에 부착된 한 쌍의 날개 디스크가 있는 깨끗한 거꾸로 된 유충 사체를 얻습니다(그림 1B).
- 샘플을 1mL 원심분리 튜브에 넣고 교반 하에 실온에서 45분 동안 1x PBS에 희석한 4% 파라포름알데히드(PFA)에 고정합니다.
참고: 샘플 교반은 진동 교반기에 의해 이루어집니다(재료 표). - 샘플을 70% 에탄올(EtOH, RNAse가 없는 물에 희석)로 3 x 5분 동안 세척하고 4°C의 동일한 세척 용액에서 최소 2일에서 최대 1주일 동안 보관합니다.
- 간접 비행 근육
- CO2 플라이 패드에 성체 파리를 마취하고 머리, 복부, 다리 및 날개를 제거합니다.
- 절개된 흉부를 차가운 1x PBS에 넣고 모든 샘플이 절개될 때까지 계속 수행합니다(그림 1D).
- 교반 하에 실온에서 20분 동안 4% PFA(1% Triton)에 흉부를 접두사로 붙입니다.
- 샘플을 1x PBT 세척액(1x PBS에서 1% Triton)으로 3 x 5분 동안 헹굽니다.
- 유리 슬라이드의 양면 테이프(Table of Materials)에 흉부를 놓고 날카로운 마이크로톰 블레이드로 이등분하여 두 개의 반흉부를 생성합니다(그림 1E,F).
- 샘플을 4% PFA(1% Triton)에 넣고 교반 중인 실온에서 45분 동안 고정합니다.
- 교반 중인 실온에서 1x PBT 세척액(1% Triton, 1x PBS)으로 2 x 20분 동안 세척합니다.
- 반흉을 차가운 1x PBS에 놓고 겸자를 사용하여 IFM을 반흉에서 부드럽게 분리합니다(그림 1G).
알림: 이를 위해서는 큐티클을 최대한 철저히 제거하는 것이 중요합니다. - 시료를 투과시키려면 분리된 IFM을 70% EtOH 용액에 4°C에서 최소 2일, 최대 1주일 동안 보관합니다.
참고: 분석을 위한 적절한 샘플 크기를 보장하기 위해 실험당 약 10개의 흉부에서 IFM을 분리합니다.
2. 혼성화, 면역염색 및 장착
참고: 유사한 하이브리드화 절차가 윙 디스크와 IFM 샘플 모두에 적용됩니다. 교잡을 시작하기 전에 유충 사체에서 날개 디스크를 분리하고 200μL의 완충액 A에 넣는 것이 중요합니다.
- 400μL의 최종 원액에 대해 10μL의 TE 버퍼(12.5μL Tris-HCl, 1mM EDTA, pH 8.0)에 프로브를 재현탁합니다.
- 샘플을 Buffer A(Table of Materials)가 들어 있는 0.5mL 원심분리 튜브로 옮깁니다.
알림: 용기의 부피를 줄이면 샘플을 시각화하는 것이 더 쉬워지고 세척 과정에서 손실되는 것을 방지할 수 있습니다. - 샘플을 버퍼 A로 2 x 20분 동안 세척합니다.
- 흡충액 A를 흡입하고 400μL의 혼성화 완충액으로 교체하고(재료 표), 열 혼합기에서 37°C에서 30분 동안 샘플을 사전 교성화합니다(재료 표).
- 하이브리드화 완충액에서 프로브를 날개 디스크의 경우 125nM, 성인 근육의 경우 200nM의 최종 농도로 희석하고 1차 항체를 추가합니다.
참고: 항체 희석은 사용 중인 특정 항체에 따라 다릅니다(재료 표). 항체 추가는 선택 사항입니다. 사용자는 단백질 염색을 포기하고 대신 항체 추가를 포함하지 않는 유사한 smFISH 프로토콜을 사용할 수 있습니다. - 혼성화 완충액을 흡인하고 프로브와 항체를 포함하는 100μL의 혼성화 완충액으로 교체합니다.
- 37°C의 어두운 곳에서 열 혼합기에서 300rpm 교반으로 최소 16시간 동안 샘플을 배양합니다.
- 버퍼 A의 부분 표본을 37°C에서 예열합니다.
- 300rpm 교반에서 37°C의 열 혼합기에서 3 x 10분 동안 샘플을 세척합니다.
- 마지막 세척 중에 2차 항체(1/200)와 DAPI(1/10,000)를 버퍼 A에 희석하고 이 용액에서 37°C에서 최소 1시간 동안 샘플을 배양합니다.
- 샘플을 세척합니다.ampBuffer B(Table of Materials)를 사용하여 3 x 20분 동안 세척합니다.
- 현미경 슬라이드에서 샘플을 조심스럽게 옮기고 잔류 버퍼 B를 닦아내고 30μL의 장착 매체를 추가하고 18 x 18mm 커버슬립으로 덮습니다. 매니큐어를 사용하여 준비를 밀봉하십시오.
- 샘플을 4°C에서 최대 1주일 동안 보관합니다.
알림: 그러나 프로브 신호 저하를 방지하기 위해 가능한 한 빨리 이미징을 수행하는 것이 좋습니다.
3. 이미징
- 40x 및 63x 오일 이멀젼 대물렌즈가 장착된 컨포칼 현미경을 사용하여 xyz 획득 모드로 이미지를 획득합니다(Table of Materials). smFISH 신호는 광자 계수 모드를 통해 HyD 검출기에 의해 감지됩니다.
- DAPI 신호와 UV 램프를 사용하여 샘플을 찾습니다.
- 날개 디스크 관련 근육 전구 및 IFM(그림 2 및 그림 3)을 이미지화하려면 레이저 라인 및 방출 필터에 대해 다음 설정을 적용합니다. DAPI (여기(예) 405nm, 방출(em.) 선택) 450nm), Alexa Fluor 488 (예: 496nm, em. 519 nm) 및 smFISH 프로브용 Quasar 670 (예: 647 nm, em. 670 nm)입니다.
- 성인 MuSC를 이미지화하려면(그림 2D) 레이저 라인 및 방출 필터에 대해 다음 설정을 적용합니다. DAPI (예: 405nm, em. 450nm), Alexa Fluor 488 (예: 496nm, em. 519nm), Alexa Fluor 555 (예: 555nm, em. 565 nm) 및 smFISH 프로브용 Quasar 670 (예: 647 nm, em. 670 nm)입니다.
참고: 이미징의 경우, 2차 항체 및 프로브와 관련된 염료에 따라 파장을 조정할 수 있습니다. - 공초점 이미지를 로 저장합니다. TIFF 파일.
4. 사후 이미징 분석
- 시작하려면 ImageJ를 실행하고 플러그인 메뉴로 이동합니다. 거기에서 Macros(매크로) | 편집 하여 매크로 소스 코드를 엽니다.
참고: 이렇게 하면 필요에 따라 매개변수를 조정할 수 있으며, 필요한 경우 매크로의 소스 코드 내에서 각 채널에 대한 폴더 디렉토리를 수정할 수 있습니다. - 이 프로토콜을 따르고 유충의 Mef2 smFISH 반점을 정량화하려면 Log_radius = 3.0 및 Log_quality = 20.0 설정을 사용합니다. 흐림이 있는 Segement Mef2 양성 핵 = 2; nucleus_scale_parameter = 30; nucleus_threshold = -8.0; nucleus_size = 300.
- 근육 섬유에서는 다음 매개 변수를 사용하십시오 : Log_radius = 2.5; Log_quality = 60.0; 흐림 = 2; nucleus_scale_parameter = 100; nucleus_threshold = 0.0; nucleus_size = 300입니다.
- 매크로를 시작하려면 실행 명령을 실행하고 매크로가 지정된 폴더에서 모든 이미지를 자동으로 로드하고 순차적으로 정량화할 때까지 기다리기만 하면 됩니다.
참고: 이 프로세스의 소요 시간은 분석되는 데이터의 양에 따라 몇 초에서 몇 시간까지 다양할 수 있습니다. - 두 개의 새 .csv 파일인 file_FISH_results 및 file_nuclei_results에 표시된 결과를 찾습니다. 첫 번째는 전사 반점의 수와 평균 및 최대 강도를 보여줍니다. 두 번째는 핵의 총 수와 각 핵 내부의 반점 수를 모두 제공합니다.
대표적 결과
이 프로토콜에서는 Mef2 및 zfh1 mRNA를 표적으로 하는 상업적으로 생산된 프로브를 사용했습니다. 제조업체의 FISH 프로브 설계자와 함께 프로브를 설계했습니다(재료 표). Mef2 세트는 모든 Mef2 RNA 동형(엑손 3에서 엑손 10까지)에 공통적인 엑손을 표적으로 합니다. zfh1 세트는 zfh1-RB 및 zfh1-RE 동형(isoform 10) 모두에 공통적인 세 번째 엑손을 표적으로 합니다. 두 프로브 모두 Quasar 670 형광 염료에 접합됩니다.
ImageJ 소프트웨어와 호환되는 매크로를 사내에서 생성하여 원시 .tif 데이터를 자동으로 분석했습니다. 매크로는 피지 플러그인 BIOP를 사용하여 Cellpose(26 )에 의한 핵의 3D 분할과 Trackmate 플러그인(27)에 구현된 가우스의 라플라시안에 의한 전사 스폿 정량화를 허용합니다. MorpholibJ 플러그인28 은 후처리에 사용된다(보충 파일 1).
첫 번째 단계로, Mef2 및 zfh1이 AMP에서 발현되는 것으로 알려진 날개 가상 디스크에서 smFISH를 수행했습니다. 이러한 데이터는 두 전사체 모두 Mef2 또는 Zfh1 항체와 함께 염색된 AMP 집단에서 균일하게 검출됨을 보여줍니다(그림 2A,B). 활성 전사 부위(TS)는 개별 세포질 전사체 또는 성숙 핵 전사체보다 더 크고 더 강렬한 신호를 갖는 경향이 있기 때문에 smFISH에 의해 성숙한 mRNA와 구별되고 구별될 수 있습니다. 일관되게 AMP의 배율이 높을수록 TS 병소와 세포질에 흩어져 있는 성숙한 mRNA를 구별할 수 있으며(그림 2A',B'), 이 smFISH 프로토콜의 감도를 검증할 수 있습니다. 그러나 zfh1과 Mef2 모두에 대해 핵당 하나의 활성 TS를 관찰했다는 점을 언급해야 합니다(그림 2A',B'). 이러한 데이터는 AMP의 Mef2 및 zfh1 전사 패턴이 해당 단백질의 검출과 함께 국소화되기 때문에 프로브 설계의 정확성을 추가로 검증합니다(그림 2).
두 번째 단계에서는 분화된 성체 IFM 및 관련 줄기세포에서 Mef2 및 zfh1 mRNA의 전사 부위와 분포를 조사했습니다(그림 2C,D). 이 데이터는 세포질 전체의 세포융합 근육 핵과 Mef2 mRNA 분포의 Mef2 TS를 명확하게 보여줍니다(그림 2C,C'). Zfh1은 특히 MuSC 인구10,11을 표시합니다. 이 smFISH 프로토콜을 사용하여 Zfh1-Gal4 > GFP 발현으로 표시된 MuSC에서 zfh1 전사를 성공적으로 검출했습니다. 더 높은 배율은 zfh1 TS와 세포질 단일 mRNA의 검출을 모두 보여줍니다(그림 2D').
마지막으로, 사내에서 구축한 ImageJ 매크로를 사용하여 Mef2 전사 역학 및 공간 분포를 정량적으로 분석했습니다. 계산 파이프라인은 Mef2 스폿과 근육 핵을 효율적으로 검출하고 분할할 수 있습니다(그림 3A-C). 그러나 신호 대 잡음비 문제로 인해 경우에 따라 일부 지점이 감지되지 않을 수 있습니다(그림 3A',B'). 이 자동화된 방법을 사용하여 Mef2 mRNA 풍부도가 근육 전구체 세포(AMP)와 분화된 성체 IFM 간에 다른지 여부를 조사했습니다. 분석 결과, 성체 IFM과 AMP의 핵당 Mef2 mRNA 수는 크게 다르지 않습니다(그림 3D).
성인 IFM 근육에서 Mef2 전사체의 공간적 분포에 대한 통찰력을 얻기 위해 세포질 대 핵 Mef2 mRNA 반점의 비율을 정량화했습니다(그림 3E,F). 이 프로그램을 사용하여 총 Mef2 mRNA 스폿 수와 핵 염색(Mef2)과 겹치는 Mef2 mRNA 스폿 수를 모두 계산했습니다. 총 mRNA 수에서 핵 관련 mRNA를 빼면 세포질 mRNA 스폿 수가 제공됩니다. 우리의 발견은 Mef2 mRNA의 약 92%가 세포질에 존재하고 나머지 8%는 그림 3E,F와 같이 근육 핵과 관련이 있음을 보여줍니다.
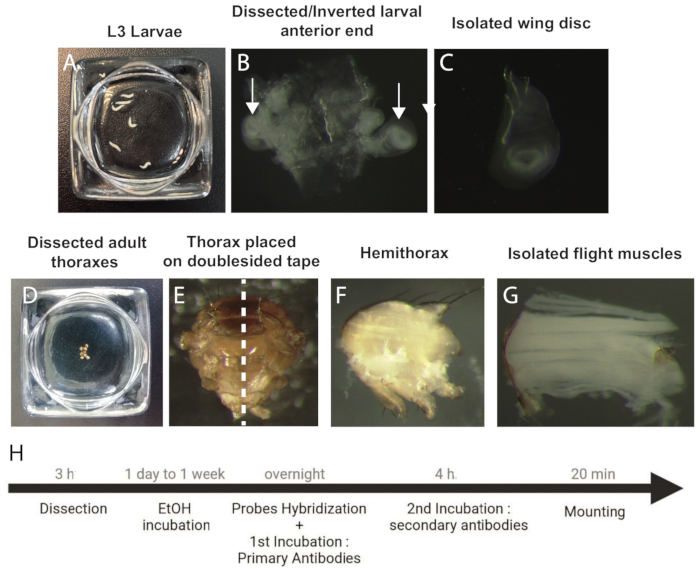
그림 1: smFISH에 대한 날개 디스크 및 IFM 샘플을 해부하고 준비하는 절차. (A) L3 단계의 유충. (B) 유충의 절개 및 거꾸로 된 앞쪽 끝. 화살표는 날개 디스크를 나타냅니다. (C) 고립된 날개 디스크. (D) 절개된 성인 흉부. (E) 성인 흉부는 이등분(점선) 전에 양면 테이프로 향하게 되어 있습니다. (F) 성인 반흉부. (G) 절연 IFM. (H) smFISH 프로토콜의 워크플로우. 약어: IFM = 간접 비행 근육; smFISH = single-molecule RNA fluorescence in situ hybridization; EtOH = 에탄올. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
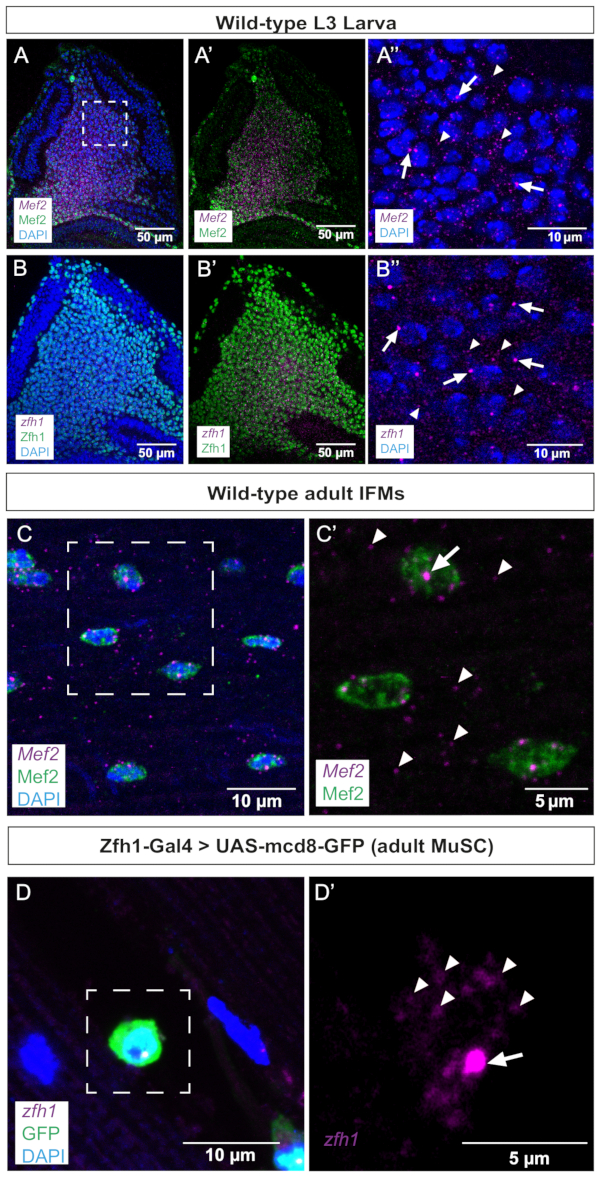
그림 2: 날개 디스크와 성체 IFM에서 Mef2 및 zfh1 smFISH의 대표 이미지. (A,B) Mef2 및 zfh1 mRNA(보라색)는 AMP에서 균일하게 검출되며 각각 (A,A') Mef2 및 (B,B') Zfh1 단백질(녹색)과 함께 국소화됩니다. (ᅡ',B'') AMP의 더 높은 배율. (C) Mef2 mRNA 및 Mef2 단백질은 성인 IFM에서 검출됩니다. (씨') C에서 박스형 영역의 더 높은 배율. (D) Zfh1-Gal4>UAS-mCD8GFP 발현(녹색)은 성체 MuSC를 라벨링합니다. zfh1 RNA는 성인 MuSC에서 검출됩니다. (디') D에서 박스형 영역의 더 높은 배율. Zfh1 전사 시작 부위와 단일 mRNA는 각각 화살표와 화살촉으로 표시됩니다. DAPI 염색(파란색). 스케일 바 = 50 μm (A,B,A',B'), 10 μm (A",B",C,D), 5 μm (C',D'). 약어: IFM = 간접 비행 근육; smFISH = single-molecule RNA fluorescence in situ hybridization; AMPs = 성인 근육 전구 세포; MuSCs = 근육 줄기 세포; DAPI = 4', 6- 디아 미디노 -2- 페닐 린돌; GFP = 녹색 형광 단백질. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
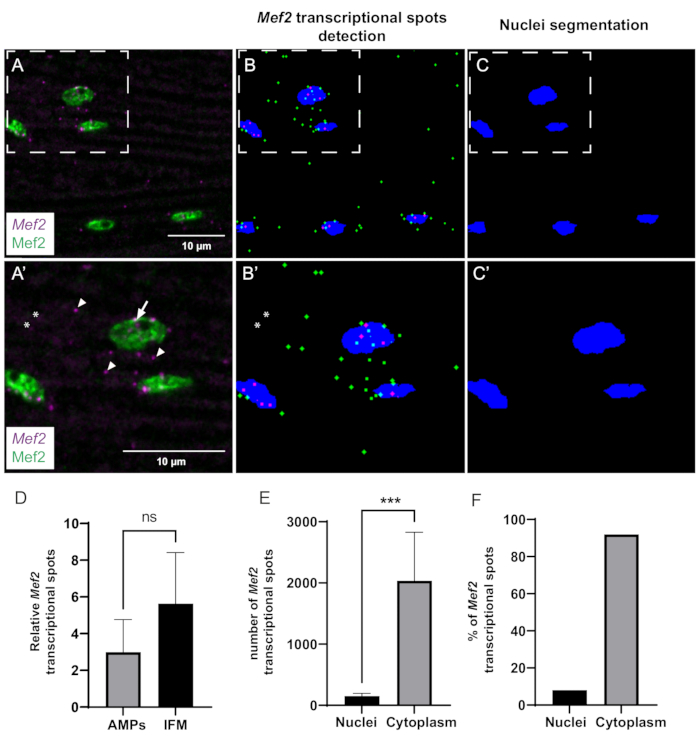
그림 3: smFISH에 의한 Mef2 전사 및 세포질 mRNA 축적의 정량화. (A) 야생형 성체 IFM에서 Mef2 전사체(보라색) 및 Mef2 단백질(녹색)의 분포. (ᅡ') A에서 박스형 영역의 더 높은 배율. 전사 시작 부위와 단일 mRNA는 각각 화살표와 화살촉으로 표시됩니다. (나,씨) 자동 Mef2 스폿 찾기 및 핵 분할. 세포질 및 핵 Mef2 mRNA는 각각 녹색과 자홍색으로 표시됩니다. (B',C')입니다. B 와 C의 박스 영역의 더 높은 배율. 별표는 매크로에서 계산되지 않는 지점을 나타냅니다. (D) 총 핵 수와 관련된 AMP 및 성인 IFM의 Mef2 스폿 정량화의 예. (p = 0,0785, 스튜던트 t-검정, 날개 디스크(n = 4), IFM(n = 11)). (E) 성인 IFM에서 Mef2 스폿 분포의 정량화 예. (*** p< 0.0007, 스튜던트 t-검정, n = 5). (F) 핵 내부 및 외부에서 검출된 Mef2 mRNA 반점의 백분율(n = 5). 스케일 바 = 10μm(A,A'). 약어: IFM = 간접 비행 근육; smFISH = single-molecule RNA fluorescence in situ hybridization; AMPs = 성인 근육 전구 세포. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 파일 1: 후처리에 사용되는 매크로입니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
smFISH 방법의 적용은 최근 인기를 얻었으며 다양한 세포 유형 및 모델 유기체로 사용이 확장되었습니다. 그러나 초파리의 경우 성인 근육 유전자 발현에 대한 대부분의 지식은 mRNA 분자의 정확한 공간 위치에 대한 정량적 정보를 제공하지 못하는 벌크 분자 분석에서 파생됩니다. 이 간극을 해결하기 위해 근육 전구체 세포와 성체 초파리 근육에서 smFISH를 수행하는 방법을 최적화했습니다. 이 접근법은 이전에 발표된 프로토콜23 에서 채택되었으며 초파리 근육 조직에 최적화되었습니다.
고품질 smFISH 이미지를 얻는 데 있어 가장 큰 장애물은 성인 근육의 두께로, 프로브의 최적 침투를 방해합니다. 따라서 성체 근육을 동물의 다른 조직과 분리하고 가벼운 교반으로 교잡 과정을 수행하는 것이 중요합니다. 이 특정 단계는 조직의 효과적인 투과를 보장합니다.
또 다른 중요한 측면은 신호 대 잡음비로 인해 계산 파이프라인이 특정 mRNA 스폿을 검출하지 못할 수 있다는 것입니다. 신호 대 잡음비를 높이는 것은 프로브의 최적 농도를 찾는 것으로 나타났습니다. 최적의 희석은 접합 염료 및 올리고뉴클레오티드 조성물을 포함한 각 프로브 세트의 조성에 따라 달라질 수 있습니다. 다른 희석을 시도하는 것이 좋습니다. 200nM의 최종 희석은 이 실험에서 성인 조직에 대한 최상의 신호 대 잡음비를 산출했습니다.
smFISH 방법을 사용하면 TS 초점에서 새로 합성된 RNA의 수를 정량화할 수 있습니다. 유전자가 활발하게 전사되면 TS에서 여러 초기 RNA가 동시에 생성됩니다. 결과적으로, TS의 강도는 성숙한 세포질 RNA의 강도를 능가할 것이며, 이 특징은 핵 국소화와 결합되어 TS를 개별 세포질 RNA와 구별할 수 있습니다. 근육 생물학의 맥락에서 TS 검출 및 정량화는 동일한 syncytium 내의 근육 핵 간 특정 유전자의 전사 동기화를 결정하는 데 특히 중요합니다. 그러나 이 계산 파이프라인은 세포질 mRNA와 TS 신호를 구별하도록 설계되지 않았습니다. 대안으로 이 smFISH 분석법을 BayFISH 또는 FISH-quant29,30과 같은 잘 정립된 다른 smFISH 분석 도구와 결합하는 것이 좋습니다. 이러한 도구는 RNA 응집체의 자동 분할 및 형광 강도 계산을 매우 정밀하게 용이하게 하는 것으로 입증되었습니다.
마지막으로, smFISH는 높은 공간 분해능으로 mRNA 분자를 검출하지만, 동시에 적은 수의 mRNA를 분석하는 데 한계가 있습니다. merFISH(multiplexed error-robust fluorescence in situ hybridization)와 같은 멀티스케일 분석법은 다수의 서로 다른 mRNA31을 동시에 분석할 수 있습니다. 올리고뉴클레오티드 프로브와 오류 수정 코드를 결합한 이 방법은 단일 세포에서 수백 종의 RNA 종을 쉽게 검출할 수 있습니다.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
원고를 비판적으로 읽어준 알랭 뱅상(Alain Vincent)에게 감사한다. Zfh1 항체를 제공해주신 Ruth Lehmann에게 감사드립니다. Stephanie Dutertre와 Xavier Pinson이 MRic 이미징 플랫폼의 컨포칼 현미경 검사에 도움을 준 것에 감사드립니다. EL은 ministère de l'enseignement supérieur et de la recherche scientifique의 박사 펠로우십의 지원을 받습니다. NA는 AFM-Telethon Ph.D. 펠로우십 23846의 지원을 받습니다. TP는 T.P.(2019-198009)에 대한 Chan Zuckerberg Initiative DAF 보조금으로 자금을 조달합니다. HB는 CNRS, Atip Avenir 프로그램 및 AFM-Telethon 트램펄린 보조금 23108의 지원을 받습니다.
자료
| Name | Company | Catalog Number | Comments |
| Confocal microscope SP8 | Leica | SP8 DMI 6000 | |
| DAPI | ThermoFisher | 62248 | |
| Double-sided tape | Tesa | 57910-00000 | |
| Dumont #55 Forceps | Fine Science Tools | 11255-20 | |
| Formaldehyde 16% | Euromedex | EM-15710 | |
| Mef2 antibody | DHSB | NA | |
| PBS 10x | Euromedex | ET330 | |
| RNaseZAP | Sigma-Aldrich | R2020 | |
| Stellaris probes | LGC | NA | |
| Stellaris RNA FISH Hybridization Buffer | LGC | SMF-HB1-10 | |
| Stellaris RNA FISH Wash Buffer A | LGC | SMF-WA1-60 | |
| Stellaris RNA FISH Wash Buffer B | LGC | SMF-WB1-20 | |
| Swann-Morton Blades | Fisher scientific | 0210 | |
| ThermoMixer | Eppendorf | 5382000015 | |
| Triton X-100 | Euromedex | 2000 | |
| UAS-mCD8::GFP line | Bloomington | RRID:BDSC_5137 | |
| Ultrapure water | Sigma-Aldrich | 95284 | |
| Watch Glass, Square, 1 5/8 in | Carolina | 742300 | |
| Zfh1 antibody | Gifted by Ruth Leahman | ||
| Zfh1-Gal4 | Bloomington | BDSC: 49924, FLYB: FBtp0059625 |
참고문헌
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nature Communications. 12 (1), 692 (2021).
- Chal, J., Pourquie, O. Making muscle: skeletal myogenesis in vivo and in vitro. Development. 144 (12), 2104-2122 (2017).
- Taylor, M. V., Hughes, S. M. Mef2 and the skeletal muscle differentiation program. Seminars in Cell and Developmental Biology. 72, 33-44 (2017).
- Siles, L., et al. ZEB1 imposes a temporary stage-dependent inhibition of muscle gene expression and differentiation via CtBP-mediated transcriptional repression. Molecular and Cellular Biology. 33 (7), 1368-1382 (2013).
- Weitkunat, M., Schnorrer, F. A guide to study Drosophila muscle biology. Methods. 68 (1), 2-14 (2014).
- Boukhatmi, H. Drosophila, an integrative model to study the features of musclestem cells in development and regeneration. Cells. 10 (8), 2112 (2021).
- Bothe, I., Baylies, M. K. Drosophila myogenesis. Current Biology. 26 (17), R786-R791 (2016).
- Catalani, E., et al. RACK1 is evolutionary conserved in satellite stem cell activation and adult skeletal muscle regeneration. Cell Death Discovery. 8 (1), 459 (2022).
- Siles, L., Ninfali, C., Cortes, M., Darling, D. S., Postigo, A. ZEB1 protects skeletal muscle from damage and is required for its regeneration. Nature Communications. 10 (1), 1364 (2019).
- Boukhatmi, H., Bray, S. A population of adult satellite-like cells in Drosophila is maintained through a switch in RNA-isoforms. Elife. 7, e35954 (2018).
- Chaturvedi, D., Reichert, H., Gunage, R. D., VijayRaghavan, K. Identification and functional characterization of muscle satellite cells in Drosophila. Elife. 6, e30107 (2017).
- Gunage, R. D., Reichert, H., VijayRaghavan, K. Identification of a new stem cell population that generates Drosophila flight muscles. Elife. 3, e03126 (2014).
- Figeac, N., Daczewska, M., Marcelle, C., Jagla, K. Muscle stem cells and model systems for their investigation. Developmental Dynamics. 236 (12), 3332-3342 (2007).
- Aradhya, R., Zmojdzian, M., Da Ponte, J. P., Jagla, K. Muscle niche-driven insulin-Notch-Myc cascade reactivates dormant adult muscle precursors in Drosophila. Elife. 4, e08497 (2015).
- Schonbauer, C., et al. Spalt mediates an evolutionarily conserved switch to fibrillar muscle fate in insects. Nature. 479 (7373), 406-409 (2011).
- Bryantsev, A. L., Baker, P. W., Lovato, T. L., Jaramillo, M. S., Cripps, R. M. Differential requirements for Myocyte Enhancer Factor-2 during adult myogenesis in Drosophila. Developmental Biology. 361 (2), 191-207 (2012).
- Soler, C., Han, J., Taylor, M. V. The conserved transcription factor Mef2 has multiple roles in adult Drosophila musculature formation. Development. 139 (7), 1270-1275 (2012).
- Soler, C., Taylor, M. V. The Him gene inhibits the development of Drosophila flight muscles during metamorphosis. Mechanisms of Development. 126 (7), 595-603 (2009).
- Pinheiro, H., et al. mRNA distribution in skeletal muscle is associated with mRNA size. Journal of Cell Science. 134 (14), jcs256388 (2021).
- Denes, L. T., Kelley, C. P., Wang, E. T. Microtubule-based transport is essential to distribute RNA and nascent protein in skeletal muscle. Nature Communications. 12 (1), 6079 (2021).
- Li, H., et al. Fly Cell Atlas: A single-nucleus transcriptomic atlas of the adult fruit fly. Science. 375 (6584), eabk2432 (2022).
- Santos, M. D., et al. Extraction and sequencing of single nuclei from murine skeletal muscles. STAR Protocols. 2 (3), 100694 (2021).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877-879 (2008).
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of single RNA transcripts in situ. Science. 280 (5363), 585-590 (1998).
- Raj, A., Tyagi, S. Detection of individual endogenous RNA transcripts in situ using multiple singly labeled probes. Methods in Enzymology. 472, 365-386 (2010).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: a generalist algorithm for cellular segmentation. Nature Methods. 18 (1), 100-106 (2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Legland, D., Arganda-Carreras, I., Andrey, P. MorphoLibJ: integrated library and plugins for mathematical morphology with ImageJ. Bioinformatics. 32 (22), 3532-3534 (2016).
- Mueller, F., et al. FISH-quant: automatic counting of transcripts in 3D FISH images. Nature Methods. 10 (4), 277-278 (2013).
- Gomez-Schiavon, M., Chen, L. F., West, A. E., Buchler, N. E. BayFish: Bayesian inference of transcription dynamics from population snapshots of single-molecule RNA FISH in single cells. Genome Biology. 18 (1), 164 (2017).
- Chen, K. H., Boettiger, A. N., Moffitt, J. R., Wang, S., Zhuang, X. RNA imaging. Spatially resolved, highly multiplexed RNA profiling in single cells. Science. 348 (6233), aaa6090 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유