3차원 피브린 하이드로겔에서 인간 흉선 오가노이드의 형성
* 이 저자들은 동등하게 기여했습니다
요약
여기에서는 흉선 상피 세포(TEC) 성숙 및 장기간의 유지 관리, 체외 흉선형성을 지원하는 것을 목표로 하는 3D 피브린 하이드로겔에서 성장한 인간 iPSC 유래 흉선 오가노이드 형성을 위한 프로토콜에 대해 설명합니다.
초록
기능적이고 자기 내성이 있는 T 세포 레퍼토리의 생성은 흉선 미세환경과 주로 세포외 기질(ECM)의 특성에 의존하는 복잡한 과정입니다. 흉선 상피 세포(TEC)는 흉선형성에 매우 중요하며, 자가 반응성 클론을 필터링하여 발달 중인 T 세포를 육성하고 선택합니다. TEC는 ECM에 의해 제공되는 물리적, 화학적 단서에 특히 민감한 것으로 경험적으로 입증되었으며, 기존의 단층 세포 배양은 죽을 때까지 빠르게 기능을 상실하게 됩니다. 이러한 섬세한 유지 관리와 상대적인 희소성으로 인해 체외에서 흉선 생물학을 모델링하는 데 큰 위험이 있음에도 불구하고 대규모로 그리고 시간이 지남에 따라 TEC 틈새 시장을 충실하게 모방할 수 있는 모델은 여전히 부족합니다. 여기에서는 인간 유도 만능 줄기 세포(iPSC)에서 TEC 구획을 유도하고 3차원(3D) 피브린 기반 하이드로겔에서 원발성 초기 흉선 세포 전구세포와 함께 재응집하는 다세포 인간 흉선 오가노이드 모델의 형성에 대해 설명합니다. 이 모델은 흉선 미세환경을 체외로 재현하고 기능성, 즉 T 세포를 생산하고 몇 주에 걸쳐 흉선 오가노이드 성장을 지원하는 능력을 보여주는 확장 가능한 배양 시스템에 대한 현재 요구 사항에 대한 답변입니다. 따라서 우리는 TEC 생물학 및 T 세포 생성 ex vivo 에 대한 연구에 도움이 될 iPSC 유래 오가노이드를 통한 흉선 기능성의 실용적인 in vitro 모델을 제안합니다.
서문
흉선은 유능하고 관대한 면역 체계 생성에 필수적인 역할을 하는 주요 림프 기관입니다 1,2,3. 초기 흉선 전구세포(ETPs)는 골수에서 흉선으로 이동하여 기능적 T 세포 1,2,4,5로 확장되고 분화합니다. 이러한 과정은 특화된 집단인 흉선 상피 세포(TEC)에 의해 매개됩니다2,6,7. TEC는 흉선 상피 전구 세포(TEPs)8,9에서 파생되며 T 세포 이동, 확장 및 성숙에 필요한 특수 3D 미세 환경을 생성하는 데 특정 역할을 하는 대뇌 피질 TEC(cTEC) 및 수질 TEC(mTEC)로 구성됩니다. TEC는 주로 성장 및 분화 인자(growth and differentiation factor) 1,10,11을 제공하고 자가 항원(self-antigen) 5,7,12의 제시를 통해 비기능성 및 비내성성 흉선세포를 음성으로 선택함으로써 T 세포 발달을 매개합니다. 발달 중인 T 세포와 TEC 사이의 복잡한 상호 작용은 흉선 누화(thymic crosstalk)로 알려진 과정에서 TEC 집단의 성숙 및 3D 조직화에 중심적인 역할을 합니다 1,11. 흉선의 세포 집단 간의 상호 작용은 세포외 기질(ECM)에 의해 형성된 특정 미세환경에 크게 의존합니다. 흉선 ECM은 흉선 세포 집단과 역동적인 호혜성 상태에 있으며, 유전자 조절에 영향을 미치고 효소 또는 매트릭스 단백질의 분비에 의해 지속적으로 재형성된다13. ECM은 성장 인자와 사이토카인의 생체이용률을 조절하고, 인테그린과 같은 막 결합 수용체를 통한 직접 신호전달을 통해 세포에 영향을 미치며, 물리적 힘을 통해 세포골격을 형성함으로써 세포에 영향을 미친다14. 콜라겐, 라미닌 등의 흉선 ECM 성분은 TEC 유지에 중요한 성장 인자인 TGFb와 FGF에 대한 친화성이 높은 것으로 나타났으며, 이는 복합체를 형성하여 이를 고정하는 데 중요한 역할을 합니다. 흉선 ECM 가소성, 탄성 계수 및 밀도는 또한 TEC의 운명을 지시하고 흉선의 기능에 필수적인 흉선의 구획화를 형성하는 데 중요한 역할을 합니다. 이러한 단서는 ECM과 그 3D 구조를 고려하여 생체 외 흉선을 모방하는 것의 중요성을 강조합니다. 이 점은 1차 TEC가 고전적 세포 배양 설정(15,16,17)에서 배양될 때 빠르게 역분화되고, 기능을 잃고, 결국 죽는다는 사실에 의해 뒷받침됩니다.
배양 모델은 ECM의 구조와 ECM에 제공하는 중요한 단서를 보존하기 위해 인간 흉선 외식에서 기능적 TEC 집단을 확장하기 위해 개발되었습니다 18,19,20. 이 배양 시스템은 in vitro에서 기능성 TEC의 집단을 성공적으로 확장하고 유지할 수 있었지만 배양 7-8일 후에는 유지할 수 없었다18. 따라서 흉선 미세환경과 그 기능을 체외에서 그리고 장기적으로 재현할 수 있는 접근 가능하고 실용적인 3D 배양 시스템의 개발은 이 분야에서 중요한 이해관계입니다. 최근 하이드로겔 기반 3D 배양 시스템의 개발로 인해 여러 인공 흉선 오가노이드 시스템이 등장하여 체외 흉선 모델링에 대한 주요 진전을 이루었습니다 15,16,21,22. 우리는 인간 1차 ETP와 유도만능줄기세포(iPSC)에서 유래한 인간 TEP를 스페로이드로 재집계하고 피브린 하이드로겔에 파종하여 인간 흉선 오가노이드(hTO) 공동 배양 시스템을 개발했습니다.
이 연구에서 재료 및 하이드로겔 설정의 선택은 흉선 ECM의 기본 구조를 재현하는 동시에 실용성과 실험을 위한 저렴하고 풍부한 재료 소스를 얻기 위해 프로세스를 확장할 수 있는 능력을 유지하는 것을 목표로 했습니다15. 이 hTO 시스템은 다중 계통 분화 잠재력을 보여주며 ETP23에서 생산적인 흉선형성을 지원할 수 있습니다. 이 오가노이드 시스템은 흉선 내 세포 상호 작용 연구와 정상 및 병리학적 인간 림프구 증 모델링을 위한 신뢰할 수 있는 도구입니다. iPS 세포의 사용은 또한 유전자 편집 기능을 모델에 도입합니다. iPSC를 기능성 흉선 조직으로 효과적으로 분화하는 것은 지난 15년 동안 이 분야의 오랜 목표였으며, 21,24,25,26,27 TEC 계통 운명 신호를 해독하는 데 상당한 진전이 있었습니다. 이러한 in vitro 3D 흉선 모델에 대한 요구에 부응하기 위해 이 기술 노트는 하이드로겔 스캐폴드 형성, 세포 마이크로매스 재응집 및 파종, 오가노이드 배양 및 수확에 중점을 두고 iPSC 유래 인간 흉선 오가노이드의 단계별 생성을 위한 방법 및 기술 세부 사항을 설명합니다.
프로토콜
hiPSC 라인 hiN.Fm.m.Lon71.019는 남성 성인 섬유아세포에서 생성되었으며 mRNA transfection을 통해 재프로그래밍되었습니다. hiPSC 라인 hiN.Fm.f.Lon80.002는 여성 성인 섬유아세포에서 생성되었으며 mRNA transfection을 통해 재프로그래밍되었습니다. hiPSC 라인 hiN.Fs.f.MIPS203.003은 여성 성인 섬유아세포에서 생성되었으며 재조합 센다이 바이러스 벡터 감염을 통해 재프로그래밍되었습니다. 모든 세포주는 Nantes iPSC 플랫폼에 의해 제공되었습니다. 환자는 자신의 세포가 연구 목적으로 사용되는 것에 대해 정보에 입각한 동의를 제공했습니다(익명 수집, Lonza, cat # CC-2511). 1차 ETP는 선언 DC-2017-2987에 따른 프랑스 CODECOH 규정에 따라 같은 날 낭트 병원(CHU Nantes)에서 소아 심장 수술을 받은 환자로부터 익명의 버려진 폐기물로 얻은 출생 후 인간 흉선 샘플의 해리에 의해 분리됩니다.
1. TEP 아이덴티티에 대한 iPSC의 차별화 지시
참고: Lai와 Jin이 발표한 첫 번째 연구가 흉선 상피 정체성에 대한 쥐 배아 줄기 세포(EScs)의 분화를 입증한 이래로28 여러 연구에서 인간 iPS 세포의 TEP 정체성 21,24,25,26,27,29에 대한 지시 분화를 설명하는 프로토콜을 개발하고 최적화했습니다. 이러한 연구는 FOXN1 및 PAX9 24,25,28,30과 같은 흉선 상피 식별 마커를 발현하는 TEP와 DLL4 및 AIRE26과 같은 기능성 마커를 발현하지만 TEC 성숙 마커24,25는 부족합니다. 두 가지 접근법이 분화된 TEP의 성숙을 성숙한 TEC 정체성으로 성숙시키는 것을 지원하는 것으로 나타났다: 마우스(29)와 같은 생체 내 모델로의 이식, 그리고 공기-액체 계면 설정(21)에서 배양된 3D 흉선 오가노이드 시스템으로의 재응집. 두 시스템 모두 in vivo 또는 in vitro T 림프포이에시스를 지원할 수 있는 기능적 TEC 집단의 성숙을 유지하고 지원하는 데 있어 3D 구조가 수행하는 중요한 역할을 입증했습니다 15,24,25,31.
- 이 연구에 사용된 흉선 오가노이드 시스템의 경우 Provin et al.23에서 개발되고 자세히 설명된 프로토콜에 따라 TEP 식별을 향한 iPS 세포의 분화를 수행합니다.
2. 소아 흉선 샘플에서 원발성 ETP의 분리
참고 : ETP는 T 세포 계통과 흉선 내의 수지상 세포를 발생시키고 CD3, CD4, CD8, CD14, CD19, CD56- CD45 + CD34 + CD7 + 32,33 표현형을 나타내는 골수 기원 전구 세포입니다.
- 공핍 비드의 준비
- 전날 자기 세포 분리 비드(재료 표)를 15mL 튜브에 옮기고 4mL의 분리 완충액(PBS + 0.1% BSA + 2mM EDTA)으로 세척합니다.
- 튜브를 마그네틱 스탠드에 놓고 상층액을 제거한 다음 2mL의 분리 완충액을 추가합니다.
- 마우스 항인간 CD3, CD4 및 CD8 항체를 비드에 추가하고 교반 하에 4°C에서 45분 동안 배양합니다. 튜브를 마그네틱 스탠드에 놓고 격리 버퍼에서 여러 번 세척한 다음 20mL의 격리 버퍼에 다시 현탁시킵니다.
- 흉선 샘플 해리
- 신선한 흉선 샘플을 RPMI1640로 채워진 페트리 접시에 옮깁니다(Table of Materials). 멸균 해부 가위와 펜치로 약1mm3 크기의 조각으로 자릅니다.
- 25mL 피펫을 사용하여 배지와 단편을 여러 번 세척한 다음(매체가 흐려져야 함) 단편이 침전되도록 하고 배지의 절반을 50mL 튜브에 수집합니다. 매체를 더 추가하고 매체가 깨끗해질 때까지 반복합니다.
- 배지를 필요한 만큼 50mL 튜브에 모으고 200 x g 에서 5분 동안 튜브를 회전시킵니다.
- 상층액을 제거하고 펠릿을 10mL의 적혈구 용액에 재현탁시킵니다(재료 표). 실온(RT)에서 5분 동안 배양하고 20mL의 세척 완충액(PBS + 0.5% BSA + 4mM EDTA + 1% 페니실린/스트렙토마이신)을 추가합니다.
- 200 x g에서 5분 동안 회전시키고 상층액을 제거합니다. 펠릿을 10mL의 세척 완충액에 재현탁시키고 70μm 메쉬 필터를 통해 걸러낸 다음 세포를 계수합니다.
- ETP 강화
- 세포를 계수한 후 세척 버퍼를 사용하여 부피를 튜브당 10mL로 조정합니다. 필요한 양의 세포 분리 비드를 추가하고(세포 2억 개당 20mL의 분리 완충액에 500μL의 비드 사용) 4°C에서 30분 동안 교반하면서 배양합니다.
- 튜브를 마그네틱 스탠드에 2분 동안 놓고 깨끗한 튜브에 상층액을 조심스럽게 모읍니다. 튜브를 제거하고 20mL의 분리 완충액으로 비드를 세척합니다. 튜브를 소용돌이치게 하고 마그네틱 스탠드에 다시 놓고 상층액을 수집합니다. 이 단계를 두 번 반복합니다.
- 상층액을 200 x g 에서 5분 동안 회전시킵니다. 펠릿을 2mL의 분리 완충액에 재현탁시키고 세포를 계수합니다.
- ETP 절연
- 농도를 mL당 2억 개의 세포로 조정하고 염색되지 않은 대조군으로 소량을 수집합니다.
- 마우스 항인간 계통(Lin)(CD3, CD4, CD8, CD14, CD19, CD56), CD7 및 CD34 항체(모든 Lin 마커에 동일한 형광 색소 사용)로 세포를 라벨링합니다. 4 °C에서 45 분 동안 배양합니다.
- 동일한 양의 세척 완충액으로 세포를 세척합니다. 200 x g 에서 5분 동안 세포를 회전시키고 펠릿을 1mL에 재현탁시킨 다음 세포를 계수합니다.
- 부피를 mL당 5,000만 개의 세포 농도로 조정하고 70μm 메쉬 필터를 통해 세포를 변형시킵니다.
- 선택한 생존도 마커를 추가하고 70μm 노즐을 사용하여 유세포 분석으로 살아있는 Lin- CD34+ CD7+ 세포를 분류합니다.
3. 3D 흉선 오가노이드 배양
- TEP 준비
- ETP 분리 직후, 13-15일에 TEP 배양의 품질을 제어합니다. 세포가 합류점에 도달하고 돌출부가 있는 조밀한 단층을 형성하는지 확인합니다(그림 1).
- 분화된 TEP를 수확하려면 DPBS-/-로 세포를 세척하고 제거한 다음 웰당 1mL의 TrypLE(Table of Materials)를 추가하고 37°C에서 5-7분 동안 배양합니다.
- 웰당 1mL의 XVIVO10(Table of Materials)를 추가하고, 여러 번 플러시하여 세포를 분리하고, 15mL 튜브로 옮기고, 200 x g 에서 5분 동안 회전시킵니다.
- 상층액을 제거하고 펠릿을 XVIVO10 1mL에 재현탁시키고 세포를 계수합니다.
참고: 분화의 효능을 미리 평가하려면 배양액을 별도로 사용하고 RT-qPCR에 의한 FOXN1 및 PAX9 의 발현과 유세포 분석에 의한 분화 수율(EPCAM+ CD205+ 세포의 분율로 계산, 50% 이상이어야 함)을 검증하십시오(그림 1). 이 분화 단계에서 거의 모든 EPCAM+ 세포는 CD205에 대해 양성이며, 이는 전구체 정체성11을 증명합니다.
- ETP 준비
- ETP 분리 직후 수집 튜브를 200 x g 에서 5분 동안 회전시킵니다. 펠릿을 XVIVO10 1mL에 재현탁시키고 세포를 계수합니다.
- 흉선 오가노이드 응집
- 적절한 부피를 피펫팅하고 mL당 2,00,000TEP 및 40,000ETP의 농도로 두 세포 현탁액을 합동하고 한 번 부드럽게 위아래로 피펫팅하여 균질화합니다.
- 표 1에 따라 적절한 보충제를 추가하고, 저결합 U 바닥 96-웰 플레이트에 웰당 100μL의 혼합 셀 현탁액을 플레이트한다. 생산량 증가를 위해 멀티채널 피펫을 사용하십시오. 그러나 기존의 단일 팁 피펫은 귀중한 세포의 부피 손실을 제한합니다. 플레이트를 37 ° C 및 5 % CO2 에서 밤새 배양합니다.
- 하이드로겔의 제조
참고: 하이드로겔 형성, 오가노이드 파종 및 배양 배지 분포에 사용되는 실험 설정은 그림 2에 나와 있습니다.- 다음 날(오가노이드 배양 단계 1일차)에 트롬빈(10U/mL), 아프로티닌(26,000U/mL) 및 피브리노겐(8mg/mL)의 부분 표본을 해동합니다(재료 표). 트롬빈과 아프로티닌을 얼음에 넣고 피브리노겐을 37°C 수조에서 해동합니다(침전되므로 얼음 위에 두지 마십시오). 소용돌이치지 말고 부분 표본을 후드 아래에 놓고 부드러운 피펫팅으로 균질화하십시오.
- 표 2에 제시된 비율에 따라 생산된 오가노이드의 수에 대해 필요한 만큼의 행잉 인서트를 준비합니다. 멸균 플라이어를 사용하여 삽입물을 배양 웰에 넣고 배양 플레이트에서 하나 이상의 열 또는 행을 비워 둡니다.
- 주조할 1.5mL 튜브를 겔만큼 준비합니다. 각 튜브에서 먼저 표 2에 자세히 설명된 대로 필요한 양의 피브리노겐과 아프로티닌을 피펫팅합니다.
- 두 번째로 필요한 트롬빈 부피를 단일 튜브에 추가하고 시약을 균질화하기 위해 기포를 생성하지 않고 빠르게 2회 세척한 다음 튜브의 전체 내용물을 끌어내고 혼합물을 행잉 인서트로 빠르게 세척합니다. 피펫을 인서트 중앙보다 수직으로 놓고 기포를 생성하지 않고 시약 혼합물을 부드럽게 세척합니다.
참고: 이 단계에서는 시약이 몇 초 안에 중합되므로 실행 속도가 매우 중요하며, 겔에 덩어리가 형성되거나 밀도가 고르지 않은 것을 방지하기 위해 올바르게 혼합하는 것이 중요합니다. 각 웰에 대해 다른 1.5mL 튜브를 사용하여 한 번에 하나의 웰을 진행합니다(튜브를 여러 웰에 재사용하는 경우 남아 있는 고체 겔 덩어리가 피펫의 팁을 막을 수 있음). 트롬빈의 높은 활성으로 인해 중합이 너무 빨리 일어나는 경우 1:2 희석을 사용하십시오. 주조 직후 겔은 투명하거나 약간 반투명해야 하며 플레이트를 수직으로 기울일 때 몇 초 후에도 여전히 흐릅니다. - 투명한 용액이 응고되어 불투명한 흰색으로 변하고 플레이트를 수직으로 기울일 때 겔이 제자리에 단단히 고정될 때까지 37°C에서 최소 1시간 동안 배양합니다(그림 2A).
- 오가노이드 시딩
- 집계 단계의 품질을 확인합니다. 마이크로매스가 구형 세포 덩어리를 형성하고 조밀한 코어가 ETP의 저밀도 후광으로 둘러싸여 있는지 확인합니다(그림 3).
- P200 콘의 끝을 자르고 접착 방지 용액으로 세척합니다(재료 표). 세포 덩어리를 채취하려면 플레이트를 거의 수직 위치로 기울이십시오: 미생물은 웰의 아래쪽 벽으로 가라앉고 흡인하는 동안 피펫 팁을 웰 바닥으로 점진적으로 밀어 쉽게 회수할 수 있습니다.
- 표 1(그림 2B)에 제시된 비율에 따라 피펫 팁으로 겔을 건드리지 않고 섬세하게 증착하여 하이드로겔 상단의 덩어리를 파종합니다. 이 단계에서 오가노이드가 자유롭게 떠다니는 것처럼 보일지라도, 배양액으로 부풀어 오르면 겔이 부드러워지고 오가노이드가 상층에 자리 잡게 됩니다. P96 웰에 오가노이드가 남아 있지 않은지 현미경으로 확인합니다.
- 표 1 및 표 2에 따라 필요한 부피의 배양 배지를 준비하고 각 웰에서 삽입 벽을 따라 피펫팅하여 하이드로겔 상단에 부피의 1/4을 천천히 추가하고 나머지 3/4은 행잉 인서트의 암 사이에 피펫을 배치하여 웰 바닥에 추가합니다(그림 2C).
- 플레이트의 습도를 유지하기 위해 빈 배양 웰에 PBS 1mL를 넣습니다. 37 °C 및 5 % CO 2 에서 배양합니다.
- 흉선 오가노이드 배양
- 2일차에 오가노이드가 잘 파종되었는지 확인합니다: 하이드로겔은 제자리에 있어야 하며 오가노이드는 인서트 바닥에 침전되지 않아야 합니다.
- 필요한 양의 배양액을 표 1 및 표 2에 따라 준비한다. 흡인 원뿔의 끝을 행잉 인서트의 암 사이로 향하게 하여 매체를 제거하고 젤을 만지지 않도록 합니다. 같은 방식으로 피펫을 배치하여 새 배지를 추가합니다.
- 2일마다 배지를 교체하고 2-4일 후(TEP 분화 시작 후 18일째) 2상 배지(표 1)로 전환합니다.
참고: 오가노이드 배치는 이러한 방식으로 최대 6주 동안 배양액에서 유지할 수 있습니다.
- 오가노이드 수확
- 웰당 1mL의 TrypLE가 있는 15mL 튜브를 준비하여 수확합니다.
- P1000 콘의 끝을 자르고 접착 방지 용액으로 코팅합니다. 피펫 팁을 인서트 중앙에 수직으로 놓고(멤브레인이 천공되지 않도록 주의) 겔을 부드럽게 피펫팅하고(멤브레인이 천공되지 않도록 주의) TrypLE 튜브로 옮깁니다. 인서트의 멤브레인을 TrypLE로 세척하고 튜브로 옮깁니다.
- 37 °C에서 15분 동안 배양하고 5분 간격으로 부드럽게 소용돌이칩니다. 겔과 오가노이드가 해리되는지 확인합니다.
- 15분 후 70μm 메쉬 필터를 걸러내고 200 x g 에서 5분 동안 회전시킵니다. 세척 버퍼에 펠릿을 재현탁시키고 선택한 분석 방법으로 진행합니다.
대표적 결과
프로토콜의 워크플로는 그림 4에 요약되어 있습니다. 이 3D 오가노이드 배양 모델을 위해 우리는 이전에 우리 팀이 제공한 물리적, 기계적 단서 덕분에 몇 일 동안 주요 마우스 mTEC를 유지하기 위해 사용했던 트롬빈과 피브리노겐 하이드로겔을 채택했습니다34. 중합 후 겔은 느슨한 스펀지 같은 메쉬 구조를 보여야 합니다(그림 5).
초기 파종 및 부착 단계 후, 오가노이드는 겔의 표면과 최상층 모두에서 점진적으로 성장하고 발달했습니다. 겔 특성, 파종 조건 및 겔에 파종된 오가노이드의 수에 따라 오가노이드는 구형에서 직사각형 구조로 형성되었으며(그림 6) 때때로 병합되어 더 큰 구조를 형성했습니다. 배양 첫 주 이후 오가노이드 내에서 조직의 두 가지 특정 하위 수준이 관찰되었습니다: 첫 번째는 오가노이드에서 방사선을 조사하고 하이드로겔을 모든 방향으로 식민지화하는 큰 세포에 의해 형성된 긴 세포 표면 돌출부와 같은 구조를 관찰했습니다(그림 6 및 그림 7). 둘째, 우리는 이러한 세포 돌출부 주위에 집중된 작은 세포에 의해 형성된 클러스터와 같은 구조를 관찰했습니다. 연구 가설을 확인하기 위해 두 세포 유형을 모두 분리할 수는 없었지만, 이 현상은 흉선 간호사 세포 복합체(thymic nurse cell complexes)로 알려진 다수의 훨씬 더 작은 발달 T 세포와 개별 cTEC의 상호 작용에 의해 형성된 흉선 피질 내에서 발견되는 3D 배열을 연상시킵니다11 (그림 8).
오가노이드 배양 단계의 여러 시점에서 유세포 분석을 통해 흉선 오가노이드의 세포 구성을 평가하고 TEC(EPCAM+ CD45-로 특징지어짐), 흉선세포(EPCAM-CD45+ CD3+)(그림 9) 및 흉선 조혈 비흉선 세포 subset으로 구성된 EPCAM-CD45+ CD3- 구획과 같은 몇 가지 주요 구획을 식별했습니다. 자세한 내용은 Provin et al.23에서 확인할 수 있습니다.
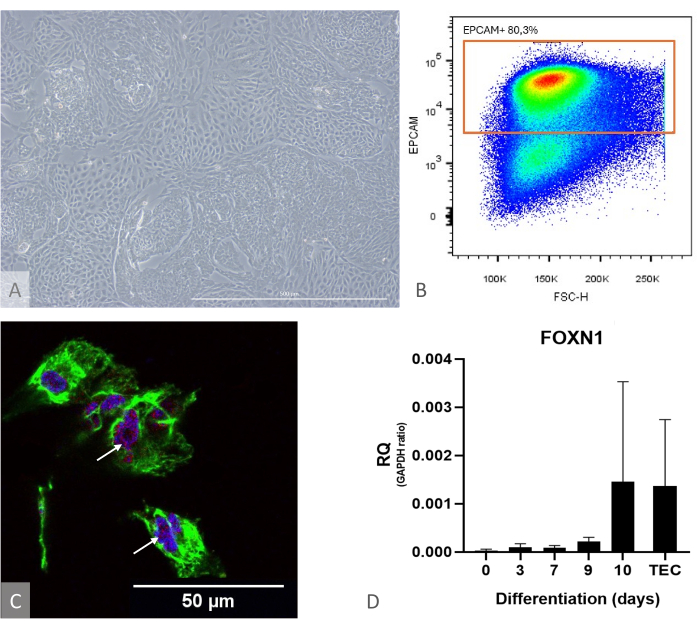
그림 1: iPSC에서 TEP로의 차별화 특성화. (A) D13에서 iPSC에서 TEP로의 분화의 예, 도립 위상차 현미경, 400x. 스케일 바: 500 μm. (B) 도트 플롯 예, 분화 14일째에 DAPI- 세포 중 EPCAM+ 세포의 비율, FlowJo 10.0.7의 이미지. (C) iPSC에서 TEP로 분화 16일째에 DAPI(파란색), PAX9(빨간색) 및 KRT8(녹색)에 대한 면역염색, 면역형광 및 컨포칼 이미징. 흰색 화살표는 anti-PAX9 염색의 예를 나타냅니다. 척도 막대: 50 μm (D) iPS에서 TEP로 분화하는 동안 FOXN1의 발현 수준(RQ에서 GAPDH까지). TEC: 양성 대조군 참조, 소아 흉선 샘플에서 분리한 1차 인간 TEC. Prism의 그래프(GraphPad 버전 8.0.1). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
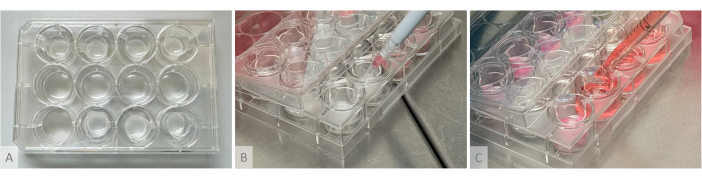
그림 2: 하이드로겔 형성, 오가노이드 파종 및 배양 배지 분포를 위한 실험 설정. (A) 하이드로겔이 주조된 배양 플레이트는 상단 및 하단 열에 배치된 행잉 인서트에 주조됩니다. (B) 오가노이드 시딩: 1개의 오가노이드가 포함된 절단된 피펫 콘을 하이드로겔 위에 건드리지 않고 놓고 오가노이드를 겔 표면에 부드럽게 파종합니다. (C) 배양 배지는 피펫의 팁을 행잉 인서트의 암 사이에 위치시켜 배양 웰에 증착됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
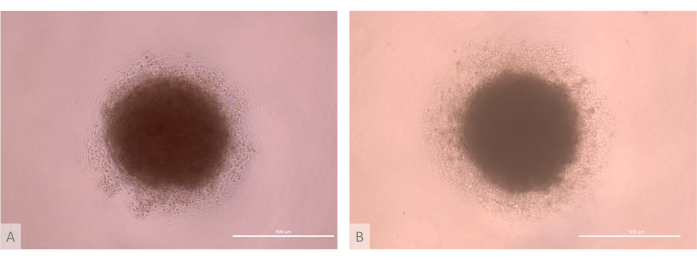
그림 3: 파종 전 흉선 오가노이드 배양의 D0(전체 프로토콜의 13-15일). (A) Lon71.019 iPS 라인에서 파생된 TEC로 생산된 오가노이드. (B) MIPS203.003 iPS 라인에서 파생된 TEC로 생산된 오가노이드. 도립 위상차 현미경, 1000×. 스케일 바: 500 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
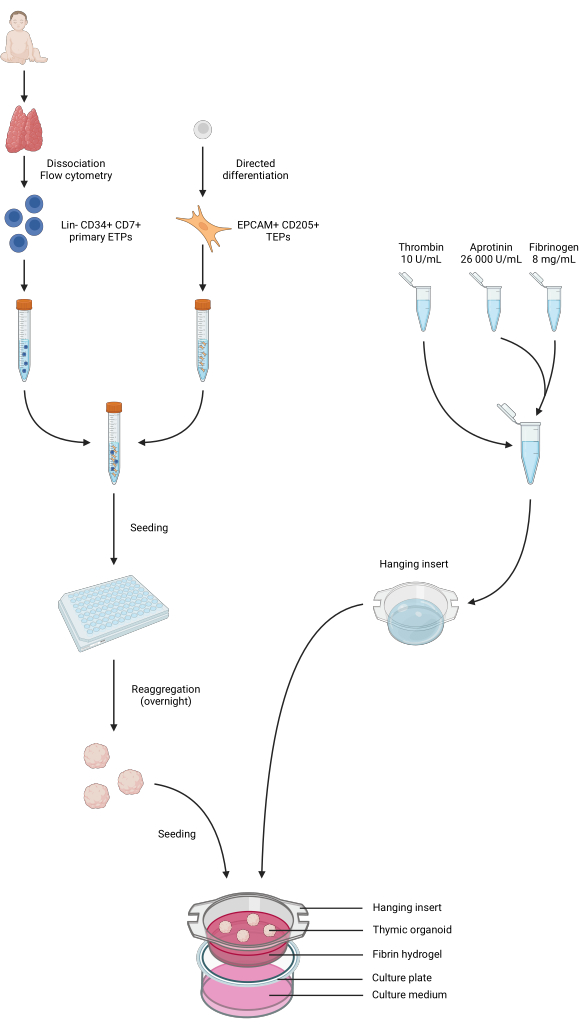
그림 4: 프로토콜의 모든 단계를 요약한 표현. 소아 흉선 샘플을 수집하고 해리하고, 1차 Lin-CD34+ CD7+ ETP를 유세포 분석으로 분류했습니다. iPS 세포의 분화는 TEP 정체성을 위해 수행되었습니다. ETP 및 iPS 유래 TEP를 저결합 96웰 플레이트에 모아서 파종하고 하룻밤 사이에 흉선 오가노이드로 응집했습니다. 피브린 하이드로겔은 아프로티닌, 피브리노겐 및 트롬빈으로 제조되어 교수형 인서트에 주조되었습니다. 중합 후, 오가노이드를 하이드로겔 위에 파종하고 1상 배양 배지를 웰에 첨가했습니다. 오가노이드는 최대 6주 동안 배양액에 보관되었습니다. BioRender에서 제작, 출판 라이선스 AG26EFCZOM. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
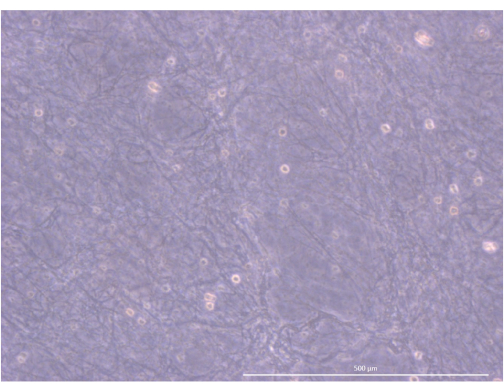
그림 5: 하이드로겔의 조직과 구조. 도립 위상차 현미경, 1000×. 스케일 바: 500 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
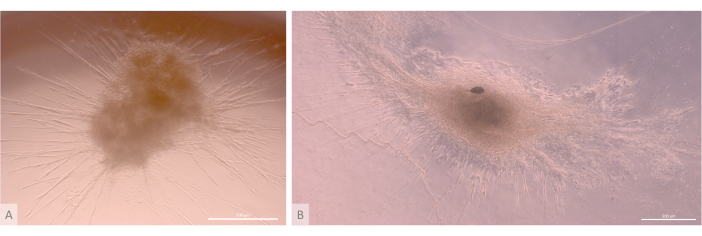
그림 6: 성숙한 오가노이드와 3차원 구조. (A) 3D 배양 24일째의 흉선 오가노이드, MIPS203.003 iPS 라인. (B) 3D 배양 32일째 흉선 오가노이드의 합성 이미지, Lon71.019 iPS 라인. 도립 위상차 현미경. 스케일 바: 500 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
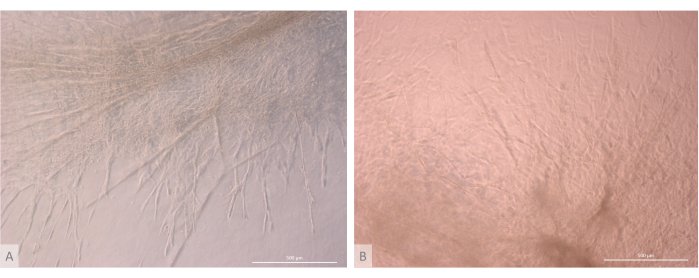
그림 7: 흉선 오가노이드의 구조 세부 정보. (A) 3D 배양 32일째의 흉선 오가노이드, L71.019 iPS 라인. (B) 3D 배양 27일째의 흉선 오가노이드, L80.002 iPS 라인. 도립 위상차 현미경, 400x. 스케일 바: 500 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
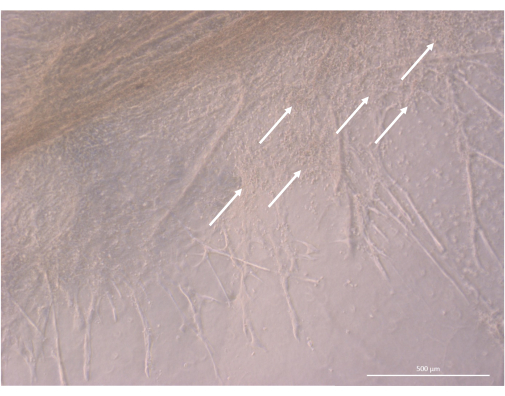
그림 8: 3D 배양 32일째 시점의 흉선 오가노이드의 구조 세부 정보. 흰색 화살표는 TEC 세포에 근접하여 증식하는 작은 흉선 세포 클러스터를 가리킵니다. 도립 위상차 현미경, 400x. 스케일 바: 500 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
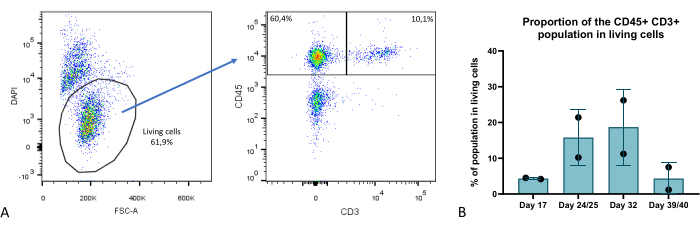
그림 9: 흉선 오가노이드 내 T 세포 구획의 비율. (A) 점도표 예, 3D 배양 35일째에 흉선 오가노이드의 살아있는(DAPI-) 세포 내 CD45+ CD3+ 세포의 비율, FlowJo 10.0.7의 이미지. CD45+ CD3- 분획은 조혈 비흉선 세포로 구성됩니다. (B) 3D 배양 17일, 24/25일, 32일, 39/40일에 흉선 오가노이드의 살아있는 세포 내 CD45+ CD3+ 세포의 비율, 기술적 중복 또는 삼중의 경우 n=2, Prism(GraphPad 버전 8.0.1) 그래프. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 단위 | 1단계 매체 14일차부터 18일차까지 | 2단계 중형 19일차 이후 | |
| 기지 | XVIVO10 | XVIVO10 | |
| BMP4 | ng/mL | 50 | |
| FGF8 | ng/mL | 10 | |
| FGF10 시리즈 | ng/mL | 10 | |
| IGF1 (이그에프1) | ng/mL | 10 | |
| EGF (영문) | ng/mL | 10 | |
| 랭크 L | ng/mL | 50 | 50 |
| 일리노이7 | ng/mL | 5 | 5 |
| FLT3 패 | ng/mL | 5 | 5 |
| 증권 시세 표시기 | ng/mL | 10 | 10 |
| 글루타맥스 | ng/mL | 1% | 1% |
표 1: 보충제 및 각각의 농도.
| 아프로티닌(μL) | 트롬빈(μL) | 피브리노겐 (μL) | 1단계 매체 | 오가노이드(단위) | |
| 24웰 플레이트 | 5 | 75 | 75 | 1 | 3 대 5 |
| 12웰 플레이트 | 9.2 | 138.2 | 138.2 | 1.8 | 5 |
| 6웰 플레이트 | 16 | 240.8 | 240.8 | 3.2 | 8에서 9 |
표 2: 6웰, 12웰 및 24웰 플레이트에서 하이드로겔을 준비하고 오가노이드를 파종하는 데 필요한 성분 비율.
토론
2D의 고전적인 단층 배양 또는 RTOC(재응집된 흉선 장기 배양)와 같은 훨씬 더 발전된 최첨단 3D 모델과 비교할 때 여기에서 설명하는 모델은 상당한 개선을 보여줍니다. 기술적 관점에서 이 모델은 TEC가 자가 재생 iPS 셀에서 파생되기 때문에 향상된 확장성과 재현성을 제공합니다. 또한 iPSC 단계에서 유전자 편집이 가능하여 TEC에서 더 쉽게 knock in 또는 knock out 연구를 수행할 수 있습니다. 이 연구에서 나타난 흉선 오가노이드의 생존율은 주목할 만하며, 최대 6주 동안 T 세포 생성이 입증되어 2D 또는 RTOC 배양에 비해 상당한 개선을 제공합니다(그림 9). 따라서 흉선 3D 구조 및 ECM 특성의 재구성은 흉선 오가노이드의 지속적인 흉선 기능, 즉 가장 성숙한 흉선 세포 구획에서 T 세포를 생성하는 능력, 즉 3D 배양 약 4주차에 최근 흉선 이주민을 생성하여 CD4+ 및 CD8+ T 세포를 모두 생성하는 능력으로 이어집니다23.
흉선 미세환경은 강렬한 확장 및 분화 활동을 지원하기 때문에 적절한 가스 교환은 모든 체외 흉선 모델에서 중요한 매개변수입니다. 실제로, 농축된 이산소 분위기 또는 공기-액체 계면(21,35)에서 유지되는 모델에서 개선된 결과가 관찰되었다. 우리의 관찰은 이 점을 뒷받침하며 공기 계면 바로 아래의 하이드로겔 상단에 올바른 오가노이드 파종의 중요성을 강조합니다. 점성이 액체 하이드로겔로 이어지는 중합 결함은 인서트 하단의 오가노이드 침몰을 유발하고 성장을 방해합니다. 내피 세포 온칩(endothelial cells on-chip)을 사용한 공동 배양은 혈관화(vascularization)를 추가하여 이 장벽을 허물 수 있는 유망한 대안입니다. 이 연구에서 생산된 흉선 오가노이드의 크기는 약 5mm로 제한되는데, 이는 핵심 영역에서 가스와 영양 교환이 부족하기 때문인 것으로 추정됩니다. 따라서 혈관화는 배양 스케일업을 가능하게 하고 공정 최적화와 결합하여 수백만 개의 TEC 및 T 세포를 포함하는 오가노이드의 생산을 가능하게 합니다. 하이드로겔의 밀도 또한 중요한 파라미터이며, 배치 전반에 걸친 재현성은 동결 및 해동 주기에 대한 효소의 민감성을 감안할 때 프로토콜의 주요 한계 중 하나입니다. 하이드로겔 주조 단계는 프로토콜에서 중요한 단계입니다. 시약 활성을 확인하기 위해 계획된 실험 1시간 전에 하이드로겔 1개를 주조하여 테스트를 수행하는 것이 좋습니다. 불충분한 효소 활성으로 인해 중합이 손상되고 iPSC 유래 TEP의 비용을 감안할 때, 새로운 시약 분취액으로 프로토콜을 다시 시작하는 것 외에 다른 문제 해결을 권장하지 않습니다. TEC는 ECM의 중요한 생산자입니다. 그러나 흉선 섬유아세포의 역할에 대한 이해의 최근 진전을 감안할 때 방사선 조사된 섬유아세포 집단을 오가노이드 모델에 추가하는 것은 흥미로울 수 있습니다. 이 집단은 TEC 및 T 세포 분화 및 유지에 긍정적인 영향을 미치는 흉선 환경 재생에 참여하는 성장 인자와 ECM을 분비할 수 있습니다. 이 흉선 오가노이드 모델의 또 다른 중요한 한계는 적절한 피질-수질 분리가 없다는 것입니다. 흉선의 캡슐 섬유아세포(capsular fibroblast)가 피질의 형성을 형성하는 것으로 나타났기 때문에 배양 모델에 추가하면 이러한 한계를 해결하는 데 도움이 될 수 있습니다. 따라서, 이 프로토콜은 흉선의 복잡한 시험관 내 모델의 기초를 소개합니다. 이는 iPSC 흉선 분화, 3D 하이드로겔 기반 배양 및 체외 림프구 형성 분야에서 이루어진 최근의 발전을 결합합니다. 이 모델은 예를 들어 중간엽 및 혈관 구획을 추가하여 확장성을 해결하고 복잡성을 증가시키기 위해 더욱 구체화될 수 있습니다. 따라서 면역에 대한 가치 있는 연구 플랫폼 또는 맞춤형 T 세포 기반 세포 치료의 응용 프로그램이 될 수 있습니다.
공개
저자는 이해 상충이 없다고 선언합니다.
감사의 말
로랑 데이비드(Laurent David)가 이끄는 프랑스 낭트의 iPSC 핵심 시설 직원들에게 감사의 말씀을 전하고 싶습니다. 이 연구는 JP-Rare Disease JTC2019 프로그램인 TARID 프로젝트(EJPRD19-208)의 지원을 받았으며, ANR(ANR-19-RAR40011-5)은 M.G.에게, RFI Bioregate 보조금(ThymIPS)은 la Région Pays de la Loire에서 M.G.로, ANR(ANR-22-CE15-0045)은 M.G.에게, "SATT Ouest Valorisation" 프로젝트인 OrgaTreg에서 M.G.N.P.는 "la fondation d'entreprise ProGreffe"의 지원을 받았습니다. M.d.A.는 "la Fondation pour la Recherche Médicale"의 지원을 받았습니다. IBiSA와 Biogenouest의 지원을 받는 낭트의 iPSC 핵심 시설의 자원과 기술 지원에 감사드립니다. 이 연구는 미래 프로그램 ANR-11-LABX-0016-01의 투자를 통해 National Research Agency가 지원하는 Labex IGO 프로그램에 의해 부분적으로 자금 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Aprotinin | Sigma Aldrich | 616370 | |
| BMP4 | Miltenyi | 130-111-165 | |
| CCR7 (CD197) | BD Biosciences | PE | Clone: 3D12; Dilution: 1: 200 |
| CD14 | BD Biosciences | FITC | Clone: M5E2; Dilution: 1: 200 |
| CD19 | BD Biosciences | PE | Clone: HIB19; Dilution: 1: 200 |
| CD205 | BioLegend | FITC | Clone: MG38; Dilution: 1: 200 |
| CD3 | BD Biosciences | PE | Clone: HIT3a; Dilution: 1: 200 |
| CD34 | BD Biosciences | FITC | Clone: 8G12; Dilution: 1: 100 |
| CD4 | BD Biosciences | PE | Clone: RPA-T4; Dilution: 1: 100 |
| CD4 | BD Biosciences | BV711 | Clone: L200; Dilution: 1: 200 |
| CD45 | BD Biosciences | PerCP | Clone: HI30; Dilution: 1: 200 |
| CD56 | BD Biosciences | PE | Clone: B159; Dilution: 1: 200 |
| CD62L | BD Biosciences | BV605 | Clone: DREG-56; Dilution: 1: 200 |
| CD69 | BD Biosciences | BV510 | Clone: FN50; Dilution: 1: 200 |
| CD7 | BD Biosciences | APC | Clone: M-T701; Dilution: 1: 200 |
| CD8 | BD Biosciences | PeCy7 | Clone: RPA-T8; Dilution: 1: 200 |
| CD8 | BD Biosciences | PE | Clone: HIT8a; Dilution: 1: 200 |
| Dynabeads Pan Mouse IgG | Invitrogen | 11041 | |
| EGF | Miltenyi | 130-097-751 | |
| EPCAM (CD326) | BD Biosciences | PE | Clone: HEA-125; Dilution: 1: 200 |
| EPCAM (CD326) | Miltenyi | BV711 | Clone: EBA-1; Dilution: 1: 200 |
| FGF10 | Miltenyi | 130-127-858 | |
| FGF8 | Biotechne R&D | 423-F8 | |
| Fibrinogen | Sigma Aldrich | 341578 | |
| FLT3 L | Peprotech | AF-300-19 | |
| Glutamax | Gibco | 35050-61 | |
| IGF1 | Miltenyi | 130-093-886 | |
| IL7 | Peprotech | AF-200-07 | |
| RANK L | Biotechne R&D | 6449-TEC | |
| Red blood cell lysis solution | Miltenyi | 130-094-183 | |
| RPMI1640 | Gibco | 11875093 | |
| SCF | Peprotech | AF-300-07 | |
| Thrombin | Sigma Aldrich | 605190 | |
| TrypLE | Gibco | 2605010 | |
| XVIVO10 | Lonza | LONBE04-380Q |
참고문헌
- Starr, T. K., Jameson, S. C., Hogquist, K. A. Positive and negative selection of T cells. Annu Rev Immunol. 21, 139-176 (2003).
- Carpenter, A. C., Bosselut, R. Decision checkpoints in the thymus. Nat Immunol. 11 (8), 666-673 (2010).
- Miller, J. F. A. P. The function of the thymus and its impact on modern medicine. Science. 369 (6503), (2020).
- Haddad, R., et al. Dynamics of thymus-colonizing cells during human development. Immunity. 24 (2), 217-230 (2006).
- Cumano, A., et al. New molecular insights into immune cell development. Annu Rev Immunol. 37, 497-519 (2019).
- Bautista, J. L., et al. Single-cell transcriptional profiling of human thymic stroma uncovers novel cellular heterogeneity in the thymic medulla. Nat Commun. 12 (1), 1096 (2021).
- Kadouri, N., Nevo, S., Goldfarb, Y., Abramson, J. Thymic epithelial cell heterogeneity: TEC by TEC. Nat Rev Immunol. 20 (4), 239-253 (2020).
- Alves, N. L., et al. Serial progression of cortical and medullary thymic epithelial microenvironments. Eur J Immunol. 44 (1), 16-22 (2014).
- Baik, S., Jenkinson, E. J., Lane, P. J. L., Anderson, G., Jenkinson, W. E. Generation of both cortical and Aire+ medullary thymic epithelial compartments from CD205+ progenitors. Eur J Immunol. 43 (3), 589-594 (2013).
- Tavian, M., Peault, B. Embryonic development of the human hematopoietic system. Int J Dev Biol. 49 (2-3), 243-250 (2005).
- Abramson, J., Anderson, G. Thymic epithelial cells. Annu Rev Immunol. 35 (1), 85-118 (2017).
- Anderson, M. S., et al. Projection of an immunological self shadow within the thymus by the aire protein. Science. 298 (5597), 1395-1401 (2002).
- Sharma, H., Moroni, L. Recent advancements in regenerative approaches for thymus rejuvenation. Adv Sci. 8 (14), 2100543 (2021).
- Alenghat, F. J., Ingber, D. E. Mechanotransduction: all signals point to cytoskeleton, matrix, and integrins. Sci STKE. 2002 (119), 6 (2002).
- Pinto, S., Schmidt, K., Egle, S., Stark, H. -. J., Boukamp, P., Kyewski, B. An organotypic coculture model supporting proliferation and differentiation of medullary thymic epithelial cells and promiscuous gene expression. J Immunol. 190 (3), 1085-1093 (2013).
- Hun, M., Barsanti, M., Wong, K., Ramshaw, J., Werkmeister, J., Chidgey, A. P. Native thymic extracellular matrix improves in vivo thymic organoid T cell output, and drives in vitro thymic epithelial cell differentiation. Biomaterials. 118, 1-15 (2017).
- Asnaghi, M. A., et al. Thymus extracellular matrix-derived scaffolds support graft-resident thymopoiesis and long-term in vitro culture of adult thymic epithelial cells. Adv Funct Mater. 31 (20), 2010747 (2021).
- Villegas, J. A., et al. Cultured human thymic-derived cells display medullary thymic epithelial cell phenotype and functionality. Front Immunol. 9, 1663 (2018).
- Hauri-Hohl, M., Zuklys, S., Holländer, G. A., Ziegler, S. F. A regulatory role for TGF-β signaling in the establishment and function of the thymic medulla. Nat Immunol. 15 (6), 554-561 (2014).
- Campinoti, S., et al. Reconstitution of a functional human thymus by postnatal stromal progenitor cells and natural whole-organ scaffolds. Nat Commun. 11 (1), 6372 (2020).
- Ramos, S. A., et al. Generation of functional thymic organoids from human pluripotent stem cells. Stem Cell Reports. 18 (4), 829-840 (2023).
- Fan, Y., et al. Bioengineering thymus organoids to restore thymic function and induce donor-specific immune tolerance to allografts. Mol Ther. 23 (7), 1262-1277 (2015).
- Provin, N., et al. Combinatory differentiation of human induced pluripotent stem cells generates thymic epithelium that supports thymic crosstalk and directs dendritic- and CD4/CD8 T-cell full development. bioRxiv. 2023, 572664 (2023).
- Parent, A. V., et al. Generation of functional thymic epithelium from human embryonic stem cells that supports host T cell development. Cell Stem Cell. 13 (2), 219-229 (2013).
- Sun, X., et al. Directed differentiation of human embryonic stem cells into thymic epithelial progenitor-like cells reconstitutes the thymic microenvironment in vivo. Cell Stem Cell. 13 (2), 230-236 (2013).
- Inami, Y., et al. Differentiation of induced pluripotent stem cells to thymic epithelial cells by phenotype. Immunol Cell Biol. 89 (2), 314-321 (2011).
- Gras-Pena, R., et al. Human stem cell-derived thymic epithelial cells enhance human T cell development in a xenogeneic thymus. J Allergy Clin Immunol. 149 (5), 1755-1771 (2022).
- Lai, L., Jin, J. Generation of thymic epithelial cell progenitors by mouse embryonic stem cells. Stem Cells. 27 (12), 3012-3020 (2009).
- Ramos, S. A., et al. Generation of functional human thymic cells from induced pluripotent stem cells. J Allergy Clin Immunol. 149 (2), 767-781 (2022).
- Provin, N., Giraud, M. Differentiation of pluripotent stem cells into thymic epithelial cells and generation of thymic organoids: Applications for therapeutic strategies against APECED. Front Immunol. 13, 930963 (2022).
- Montel-Hagen, A., et al. In vitro recapitulation of murine thymopoiesis from single hematopoietic stem cells. Cell Rep. 33 (4), 108320 (2020).
- Park, J. -. E., et al. A cell atlas of human thymic development defines T cell repertoire formation. Science. 367 (6480), 3224 (2020).
- Flippe, L., et al. Rapid and reproducible differentiation of hematopoietic and T cell progenitors from pluripotent stem cells. Front Cell Dev Biol. 8, 577464 (2020).
- Padonou, F., et al. Aire-dependent transcripts escape Raver2-induced splice-event inclusion in the thymic epithelium. EMBO Rep. 23 (3), e53576 (2022).
- Han, J., Zúñiga-Pflücker, J. C. High-oxygen submersion fetal thymus organ cultures enable FOXN1-dependent and -independent support of T lymphopoiesis. Front Immunol. 12, 652665 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유