장기 수준의 마이크로바이옴 분석을 위한 모기 난소, 중장 및 타액선 해부
요약
모기의 미생물 군집은 매개체 생물 방제 전략에 대한 큰 가능성을 가지고 있습니다. 대부분의 공생체는 배양할 수 없어 균유전체학 분석이 필요합니다. 암컷 모기를 해부하고 난소, 중장 및 타액선을 분리하여 교차 오염을 방지하고, 장기 수준에서 마이크로바이옴 연구를 촉진하고, 모기 생물학에서 미생물의 역할에 대한 이해를 높이는 방법을 설명합니다.
초록
말라리아, 뎅기열, 웨스트 나일, 지카, 우수투, 황열병을 포함한 모기 매개 질병의 부담이 전 세계적으로 계속 증가하여 심각한 공중 보건 위협이 되고 있습니다. 살충제 내성이 증가하고 효과적인 백신이 없어짐에 따라 모기의 미생물군에 초점을 맞춘 새로운 전략이 등장하고 있습니다. 그럼에도 불구하고 대다수의 공생체는 재배에 저항력이 있습니다. 따라서 모기 표본에서 박테리아 게놈의 다양성과 기능을 특성화하는 것은 균유전체학(metagenomics)과 그에 따른 조립 및 비닝 전략에 의존합니다. 분리된 장기에서 채취한 메타게놈 조립 게놈(MAG)의 관찰 및 분석은 특히 난소(생식 기관), 중장(음식물 소화 및 면역의 핵심) 또는 타액선(병원체가 타액으로 들어가 혈액 식사 중에 혈류에 도달하기 위해 집락을 형성해야 하므로 매개체 매개 질병의 전염에 필수적)에서 모기 관련 미생물의 특정 역할에 대한 주요 정보를 제공할 수 있습니다. 이렇게 새롭게 재구성된 게놈은 새로운 벡터 생물 제어 전략 개발을 위한 길을 열 수 있습니다. 이를 위해서는 모기 기관을 격리하면서 모기 기관 간 또는 다른 모기 기관에 존재하는 미생물과의 교차 오염을 피해야 합니다. 여기에서는 장기 수준에서 모기 마이크로바이옴을 연구하기 위한 최적화되고 오염되지 않은 해부 프로토콜에 대해 설명합니다.
서문
모기는 질병을 일으키는 광범위한 병원체를 퍼뜨리며 공중 보건에 심각한 위협이 됩니다. 모기 개체군에서 살충제 내성의 유병률이 증가하고 이러한 병원체에 대한 효과적인 백신이 없기 때문에 모기 마이크로바이옴에 초점을 맞춘 새로운 생물 방제 방법이 등장하고 있습니다. 특히 병원체 전달을 방해하고 숙주 생식을 조작할 수 있는 세포내 세균 Wolbachia가 눈에 띕니다 1,2,3. 또한, 다른 모기 공생체는 병원체의 감염 및 전파뿐만 아니라 숙주의 생존, 발달 또는 면역 체계의 중심이며, 벡터 매개 질병과 싸우기 위해 그들의 착취에 대한 큰 가능성을 보여줍니다 4,5,6,7,8.
모기와 관련된 미생물은 미생물 생명체의 모든 영역(박테리아, 진핵생물 및 곰팡이 포함)에 걸쳐 있으며, 이들은 숙주와 밀접하게 상호 작용할 뿐만 아니라 서로 다른 신체 구획에서 서로 상호 작용합니다(9,10). 따라서, 미생물총(microbiota), 잠재적인 계절적 변이(seasonal variation)11 및 그 구성원들이 별개의 모기 조직에서 직접 상호 작용하는 메커니즘에 대한 더 나은 이해는 새로운 표적 생물 방제 방법을 개발하거나 기존 방법을 개선하는 데 도움이 될 수 있다. 그러나 대다수의 공생체는 재배에 저항력을 가지고 있어 특성화가 불가능합니다.
2세대 및 3세대 염기서열분석 방법의 출현과 최첨단 조립 및 비닝 접근법은 미생물 게놈을 재구성하고 배양할 수 없는 미생물의 다양성과 기능적 잠재력에 접근할 수 있게 했습니다. 여기에서는 암컷 모기의 난소, 중장, 타액선을 절개하면서 교차 오염을 방지하는 방법을 제시합니다. 이 프로토콜은 게놈 DNA 추출과 후속 메타바코딩 또는 샷건 메타게놈 시퀀싱을 통해 장기 수준에서 모기 미생물군의 다양성과 기능을 탐구할 수 있습니다. 우리는 Culex spp. 표본에 대한 모기 해부 및 마이크로바이옴 데이터의 예를 제공하지만, 이 프로토콜은 Anopheles 또는 Aedes와 같은 다른 속의 벡터로 확장될 수 있습니다.
프로토콜
참고: 그림 1 은 프로토콜의 여러 단계를 요약한 방법의 개략도를 보여줍니다.
1. 재료 준비
- 실험을 시작하기 전에 0.5% 활성 염소(세계보건기구에서 권장)가 포함된 표백제 용액을 준비하여 장비와 작업대를 소독합니다.
- 900mL의 물에 각각 1.5g의 활성 염소를 함유한 3개의 수용성 정제를 녹입니다. 정제가 완전히 녹을 때까지 15분 동안 기다리면 0.5% 표백제 용액이 됩니다.
- 0.5% 표백제 용액을 스프레이 병에 넣습니다. 그런 다음 미세한 슬라이드와 작업대를 포함한 모든 해부 장비에 용액을 분무합니다.
알림: 장비와 작업대는 사용하기 전에 ~10분 동안 건조시켜야 합니다.
- 샘플이 외부 박테리아로 오염되는 것을 방지하기 위해 각 해부 전에 모기를 헹구려면 4개의 1.5mL 새로 멸균된 튜브(96% 에탄올이 포함된 튜브 2개)와 1x PBS 완충액(인산염 완충 식염수)이 포함된 다른 튜브 2개를 준비합니다.
- 각 모기를 해부한 후 샘플을 보관할 튜브를 준비합니다: 각각 100μL의 보존 완충액이 들어 있는 4개의 1.5mL 멸균 튜브.
참고: 타액선용 튜브 하나, 난소용 튜브 하나, 중창자용 튜브 하나, 모기 사체용 튜브를 준비하십시오.
2. 모기 채집
- 성인 여성 개체를 수집하여 표면 멸균 할 때까지 (현장 또는 실험실에서, 4-7 일) 생명을 유지하십시오.
3. 모기의 표면 살균
알림: 겸자와 바늘을 청소하고 각 장기와 개인 사이에 멸균된 새 튜브를 사용하십시오. 표백제를 사용할 수 없는 경우 생물학적 물질의 교차 오염을 방지하기 위해 각 개인과 장기의 절개 사이에 있는 절개 겸자와 바늘을 철저히 화염 멸균해야 합니다. 해부 기구는 다음 사용 전에 표백제/에탄올 잔류물 없이 건조해야 합니다.
- 암컷이 알코올에 노출되었을 때 장 내용물을 비우지 않도록 얼음이나 -20°C의 냉동고에서 냉 마취하십시오(조직과 장기가 얼지 않도록 4분 동안).
- 개별 모기를 96% 에탄올 1mL가 든 새로운 멸균된 1.5mL 튜브에 옮깁니다. 30초 동안 소용돌이를 부드럽게(수동) 합니다(곤충 표면의 비늘과 잠재적인 박테리아 오염 물질을 분리하기 위해 튜브에서 모기를 잘 흔듭니다). 에탄올이 들어있는 튜브를 사용하여 두 번 헹굽니다.
- 모기를 1.5mL의 멸균된 1x PBS 용액이 있는 새로운 멸균된 1mL 튜브로 옮기고 30초 동안 부드럽게(수동으로) 소용돌이칩니다(에탄올로 DNA 침전을 피하기 위해). 멸균 1x PBS가 들어 있는 튜브를 사용하여 두 번 헹굽니다.
4. 모기 해부
- 멸균 1x PBS의 작은 방울 몇 방울을 슬라이드에 올려 모기를 쉽게 배치하고 절개된 장기를 헹구는 데 도움이 됩니다.
알림: 멸균 PBS 방울은 모기 장기를 수용할 수 있을 만큼 충분히 커야 합니다. - 청소한 모기를 현미경 슬라이드의 멸균 1x PBS 한 방울에 놓습니다.
- 아래 설명에 따라 멸균된 겸자와 바늘로 타액선을 절개합니다.
- 모기가 움직이지 않도록 핀셋으로 모기의 흉부를 잡습니다. 그런 다음 머리 아래의 목에 바늘을 놓고 모기의 머리를 부드럽게 잡아당겨 흉부에서 분리합니다.
참고: 타액선은 위쪽 흉부에 위치하고 있으며 타액관으로 머리와 연결되어 있습니다. 절개 중에는 흉부에서 타액선을 뽑을 때 머리와 함께 오지 않으므로 이 관을 끊지 않는 것이 중요합니다. 이것이 얼린 모기가 아닌 신선한 모기를 갖는 것이 중요한 이유입니다. - 머리가 흉부에서 분리될 때 각각 세 개의 엽으로 구성된 두 개의 침샘이 나오는 것을 관찰하십시오. 타액관 수준에서 타액선을 자르고 바늘로 채취한 다음 멸균 1x PBS의 새 방울에 넣어 헹구고 장기 간 오염을 방지합니다.
- 바늘을 사용하여 타액선을 채취하고 100μL의 보존 완충액이 들어 있는 새로운 멸균 튜브에 넣습니다.
- 모기가 움직이지 않도록 핀셋으로 모기의 흉부를 잡습니다. 그런 다음 머리 아래의 목에 바늘을 놓고 모기의 머리를 부드럽게 잡아당겨 흉부에서 분리합니다.
- 다음과 같이 멸균된 겸자와 바늘로 난소를 절개합니다.
- 모기를 등에 눕히십시오. 해개 중 움직임을 방지하기 위해 집게를 사용하여 모기를 흉부로 잡고 바늘로 세 번째 복부 부분(바닥에서 시작)에 구멍을 뚫습니다.
알림: 선택적으로 모기의 다리와 날개를 제거하여 해부를 용이하게 합니다. - 세 번째 복부 부분을 뚫으면서 복부를 아래로 당겨 열고 내부 장기를 드러냅니다. 장기의 방출을 용이하게 하기 위해 남아 있는 모기 외골격 조각을 제거합니다.
참고: 장, Malpighian 세뇨관 및 난소와 같은 복부의 장기가 보입니다. - 멸균된 겸자로 양쪽 난소를 채취하고 현미경 슬라이드의 멸균 1x PBS의 새 방울에 넣어 헹굽니다. 난소를 잡고 100μL의 보존 완충액이 있는 1.5mL 튜브에 넣습니다.
- 모기를 등에 눕히십시오. 해개 중 움직임을 방지하기 위해 집게를 사용하여 모기를 흉부로 잡고 바늘로 세 번째 복부 부분(바닥에서 시작)에 구멍을 뚫습니다.
- 이제 복부가 열리고 장기가 노출되었으므로 멸균된 새 겸자 및/또는 바늘로 중장을 채취합니다.
- 앞창자와 뒷창자에서 중장을 분리합니다. 그런 다음 중장에서 Malpighian 세뇨관을 제거합니다. 멸균 바늘을 사용하여 중장을 잡고 새로운 멸균 1x PBS 방울에 올려 헹구고 장기 간 오염을 방지합니다.
- 새로운 멸균 1x PBS 드롭으로 옮깁니다. 겸자로 중앙장을 열어 보충 파일 1에 설명된 대로 박테리아를 제거하고 농축합니다. 겸자로 중장을 잡고 박테리아가 포함된 모든 PBS를 피펫팅하여 전체 물질(액체 및 조직)을 100μL의 보존 완충액이 있는 새로운 멸균된 1.5mL 튜브에 저장합니다.
- 현미경 슬라이드에서 이전에 사용한 위치에 5μL의 1x PBS를 다시 추가하고 PBS를 동일한 1.5mL 튜브에 직접 피펫팅하여 현미경 슬라이드에 달라붙었을 수 있는 박테리아를 회수합니다.
- 멸균 겸자를 사용하여 사체(남은 시체)를 잡고 100μL의 보존 완충액이 있는 새로운 멸균된 1.5mL 튜브에 보관합니다.
- 추가 처리까지 재료 보관
- 튜브에 'Species, Organ, Date, Researcher's name'이라고 라벨을 붙입니다.
- 절개된 난소, 중장, 타액선 및 사체는 처리할 때까지 -20°C에서 보관합니다.
- 종(또는 속 수준만 해당), 수집 날짜, 국가, 지방, 위도, 경도, 해부를 한 사람의 이름, 임신 여부, 혈액 식사 여부를 포함하여 샘플에 대한 모든 관련 메타데이터를 스프레드시트에서 추적합니다.
결과
x'Mosquito 해부
프로토콜에 따라 실험실 군락에서 두 개의 Culex pipiens molestus 표본(임신 암컷 포함)의 난소, 중창자, 타액선 및 사체를 수집하고 분리했습니다. 우리는 쌍안경 아래에 남아 있는 파편이 없는 전체(즉, 끊어지지 않은) 잘 격리된 장기를 관찰한 후 깨끗한 해부를 확인했습니다. Culex pipiens molestus 표본의 전신, 머리 및 흉부, 절개된 타액선, 난소 및 중장이 그림 2에 나와 있습니다. 예상했던 대로, 중창자와 침샘은 모기의 난소보다 훨씬 작았다. gravid Culex 표본의 난자는 midgut 및 Malpighian tubules와 함께 그림 3에 제시되어 있습니다. 주목할 점은 재료가 신선하지 않고 조직이 깨질 가능성이 있는 경우 해부가 실패하기 쉽다는 것입니다(그림 4B,C). 따라서 조직이 여전히 탄력적이므로 가능한 경우 채취 직후(12시간 이내) 재료를 해부하는 것이 좋습니다(그림 4A). 마찬가지로, 동결된 물질을 절개할 수 있지만 깨지기 쉬운 조직으로 인해 장기 간 교차 오염 및 실패의 위험이 훨씬 더 높습니다(그림 4).
마이크로바이옴 데이터
또한, 동일한 절차에 따라 뉴칼레도니아 누메아(Noumea)에서 채취한 3개의 Culex quinquefasciatus 표본에서 중창자와 난소를 채취하여 분리했다. 각 장기에서 DNA를 추출하고, 전체 게놈 염기서열 분석을 위한 샘플을 준비하고, 보충 파일 1에 자세히 설명된 대로 마이크로바이옴 데이터 분석을 수행했습니다. PhyloFlash12 를 사용하여 조립되지 않은 품질 필터링된 짧은 판독에 대한 박테리아 분류학적 다양성 분석은 난소에 비해 중장에서 뚜렷한 우성 분류군을 보여주었습니다(그림 5A). 특히, 난소 세균 군집은 Wolbachia에 의해 지배되었으며, 개별 3의 난소에 Spirochaetaceae 계통형이 추가로 존재했으며, midgut 군집은 Gammaproteobacteria, Spirochaetaceae 및 Firmicutes를 포함한 더 넓은 다양성을 나타냈습니다. 동일한 염기서열 분석 데이터에서 완료율이 80%<> 중복성이 5%인 4개의 메타게놈 조립 게놈(MAG)을 재구성했으며, 이는 Spirochaetia, Alpha- 및 Gammaproteobacteria 클래스에 속합니다(표 1).
예상한 바와 같이, 본원에서 재구성된 MAG는 메타게놈 짧은-판독을 이용한 게놈 재구성의 특정 단점으로 인해 PhyloFlash 결과에 의해 예측된 완전한 분류학적 다양성을 커버하지 못했다. Wolbachia에 할당된 MAG(표 1의 MAG 3 참조)는 모든 난소와 2개의 중장에서 검출되었으며 난소에서 더 높은 적용 범위를 가졌습니다(그림 5B). 또한 해당 난소에서 검출되지 않은 Culex 개체 2와 3에서 얻은 중창자에서 Pantoea 속(표 1 및 2 참조)을 포함하여 Enterobacteriaceae 계열에 속하는 두 개의 MAG를 재구성했습니다(그림 5B). 마지막으로, Graña-Miraglia와 동료13에 의해 Culex pipiens midguts에서 성공적으로 분리된 BR149 속에 할당된 Spirochaetaceae 박테리아 게놈 1개를 재구성했습니다(표 1). 흥미롭게도, 이 MAG는 개인 1과 3의 중창자뿐만 아니라 개인 3의 난소에서도 검출되었습니다(그림 5B).
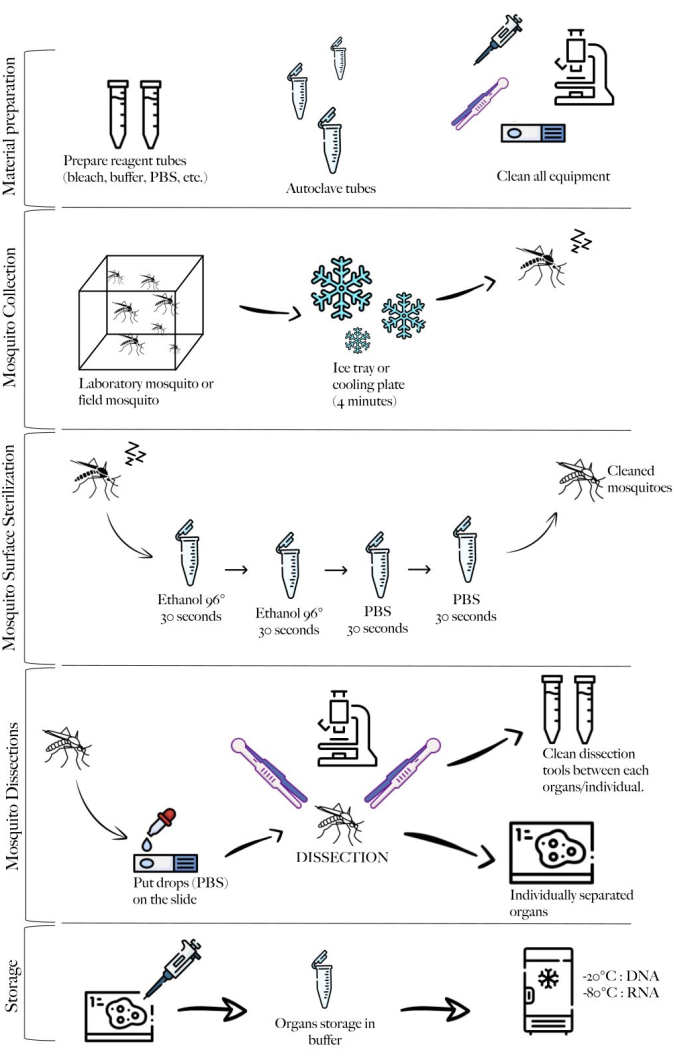
그림 1: 여러 단계를 요약한 방법의 개략도. 재료 준비, 모기 수집, 모기 청소, 모기 해부 및 보관. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
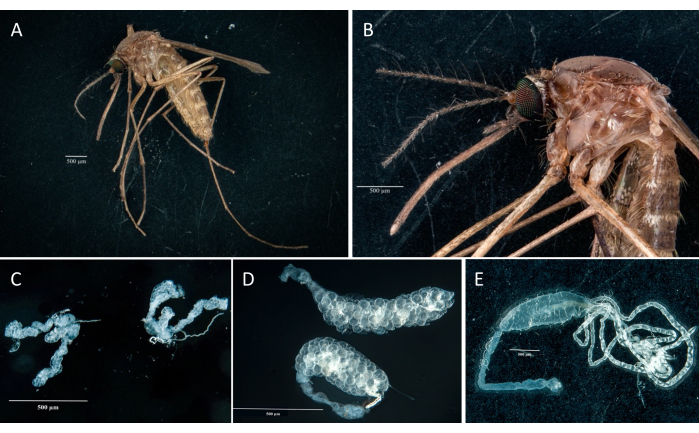
그림 2 : Culex pipiens molestus 암컷. (A) 전신. (B) 머리와 흉부. (C) 절제된 타액선. (D) 박리된 난소. (E) Malpighi 세뇨관으로 절개된 장. 스케일 바 = 500 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
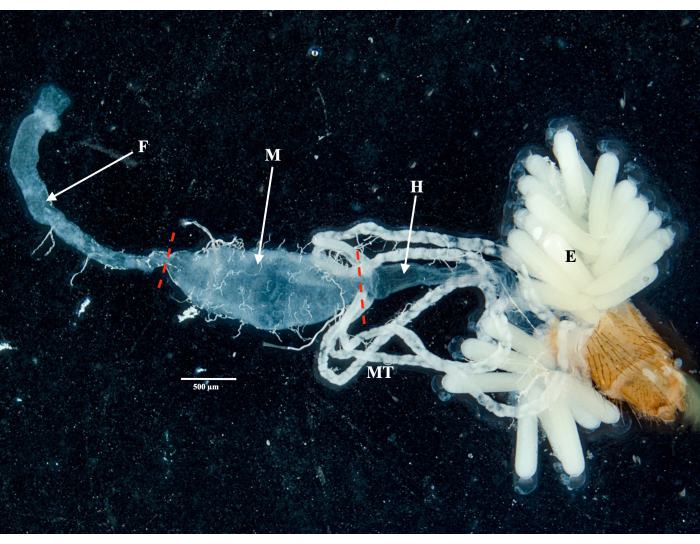
그림 3: gravid Culex pipiens molestus 암컷의 절개된 복부. 표본의 앞창자, 중창자, 뒷창자, Malpighian 세뇨관 및 난자가 표시됩니다. 빨간색 점선은 앞창자와 뒷창자에서 중장을 분리하기 위해 절단할 위치를 나타냅니다. 눈금 막대 = 500 μm. 약어: F = foregut; M = 중장; H = 뒷창자; E = 달걀; MT = Malpighian 세뇨관. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
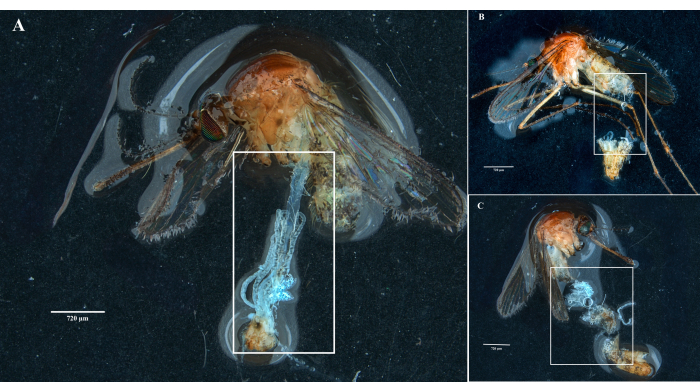
그림 4: 신선 및 냉동 Culex 모기 표본의 해부. 흰색 사각형은 (A) 갓 절개된 표본의 온전한 장기가 있는 모기 조직과 (B,C) 해부 전에 냉동된 표본의 깨진 생물학적 물질을 나타냅니다. 스케일 바 = 720 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
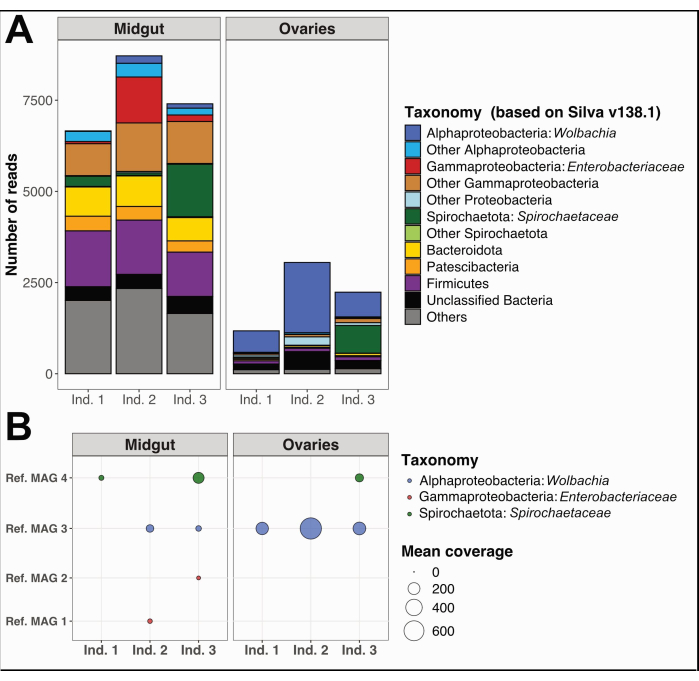
그림 5: 3명의 Culex quinquefasciatus 개체에 대한 마이크로바이옴 분석의 예. (A) SSU rRNA 추출을 통해 추정된 박테리아 다양성의 시각화는 3개 검체의 중창자와 난소에서 PhyloFlash12 로 판독합니다. (B) 샘플에 대한 재구성된 MAG의 평균 적용 범위. 약어: MAG = Metagenome-Assembled Genome; Ind = 개인. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 잡지 | 참조 MAG 4 | 참조 MAG 2 | 참조 MAG 1 | 참조 MAG 3 |
| 길이(bp) | 1,287,790 | 4,910,866 | 4,751,276 | 1,298,266 |
| contigs의 수 | 9 | 162 | 149 | 123 |
| GC % | 34.05 | 55.45 | 54.05 | 34.15 |
| 완료(%) | 84.5 | 97.18 | 98.59 | 91.55 |
| 이중화(%) | 0 | 2.82 | 4.22 | 0 |
| 도메인 | 박테리아 | 박테리아 | 박테리아 | 박테리아 |
| 문 | 스피로차에토타 | 프로테오박테리아 | 프로테오박테리아 | 프로테오박테리아 |
| 수업 | 스피로채티아 | 감마프로테오박테리아 | 감마프로테오박테리아 | 알파프로테오박테리아 |
| 주문 | WRBN01 시리즈 | 엔테로박테랄레스 | 엔테로박테랄레스 | 리케치알레스 |
| 가족 | WRBN01 시리즈 | 장내세균(Enterobacteriaceae) | 장내세균(Enterobacteriaceae) | 아나플라스마타과(Anaplasmataceae) |
| 속 | 브롬149 | 판토에아 | - | 볼바키아 |
표 1: 3개의 Culex quinquefasciatus 개체에서 재구성한 MAG. Anvi'o17 에서 사용할 수 있는 단일 복제 핵심 유전자 수집 및 GTDB18을 사용하여 얻은 분류학을 기반으로 한 게놈 크기, 콘티그 수, GC 비율, 완료 추정치 및 중복성.
보충 파일 1: 시료 채취, DNA 추출 및 전체 게놈 염기서열 분석부터 시작하여 게놈 재구성을 위한 생물정보학 워크플로우 및 원핵생물 및 진핵생물 판독 비율 추정을 위한 마이크로바이옴 데이터 분석을 위한 자세한 예시 절차 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
침샘부터 시작하여 장기 절개 순서에 특히 주의를 기울이는 것이 좋습니다. 실제로, 우리는 모기의 무결성이 보존되면 Culex 표본의 흉부에서 더 쉽게 추출되는 것을 관찰했습니다. 복부나 흉부가 손상되면 모기 몸의 압력이 감소하여 시술을 방해할 수 있습니다. 그러나 흉부와 복부 사이를 절개한 다음 머리와 흉부에서 침샘을 뽑는 것도 가능합니다(A. B. Failloux, personal communication). 또한 능숙한 해부 기술을 습득하는 것이 어려울 수 있으므로 실험 전에 적절한 수의 표본에 대해 연습하는 것이 좋습니다.
체계적인 방식으로 교차 오염을 피하면서 모기 장기를 격리하는 것은 광범위한 다운스트림 모기 미생물군 분석에 매우 중요합니다. 프랑스 남부의 Culex pipiens 표본에서 단일 난소를 절개한 후 게놈 분해 메타게놈 연구를 통해 Culex pipiens에서 Wolbachia의 첫 번째 플라스미드를 발견할 수 있었습니다(pWCP14). 유사한 접근 방식을 사용하여 다양한 환경 및 실험실 조건에 걸쳐 전 세계 대륙 및 섬 지역의 Culex pipiens 및 Culex quinquefasciatus 표본에서 pWCP의 분포와 빈도를 조사했습니다. 전반적으로, 이 데이터는 Culex 모기에서 눈에 띄게 보존된 Wolbachia 플라스미드 요소를 보여주었으며, 이는 내공생 생물학15에서 이 모바일 요소가 중요한 역할을 한다는 것을 시사하며, 추가 분석이 필요합니다.
여기에서는 이 체계적인 절차를 사용하여 얻은 중장 및 난소 샘플에 대한 모기 마이크로바이옴 분석의 추가 예를 제공합니다. 우리는 조직 간의 미생물군에서 명확한 차이를 관찰했으며(그림 5), 공유 분류군과 장기 특이적 박테리아 분류군 모두를 관찰했습니다. 예상했던 대로, Wolbachia 의 존재는 두 기관 모두에서 감지되었으며, midguts에 비해 모기 난소의 MAGs의 상대적 풍부도(조립되지 않은 짧은 판독 기준)와 평균 적용 범위가 더 높았으며, 이 내공생체가 난소를 통해 전달된 후 체세포 조직으로 퍼진다는 관찰과 일치했습니다. 이 연구는 뉴칼레도니아의 샘플로 제한되었지만, 이 프로토콜은 전 세계적으로 Wolbachia의 게놈 변동성에 대한 조사뿐만 아니라 자체의 밀도 조절 및 바이러스 보호를 포함한 다양한 표현형에서의 역할을 용이하게 할 수 있습니다. 더욱이, 이 연구는 여기에 제시된 해부 절차가 어떻게 중장 샘플 내에서 모기 공생체의 분류학적 다양성과 잠재적인 기능적 능력을 검사할 수 있게 하는지 보여줍니다.
우리는 두 개의 중장 샘플에만 존재하는 두 개의 장내 세균 초안 게놈을 획득하여 이 두 샘플에 대한 장기 간의 오염이 없음을 확인했습니다. 개체 3의 중장과 난소에서 모두 검출된 스피로헤테스와 관련하여, Juma와 동료들은 2020년에 알 뗏목의 표면에서 이러한 박테리아의 존재를 관찰했습니다. 저자들은 달걀 뗏목에서 발견되는 박테리아 군집은 달걀 뗏목이 탈이온화되고 박테리아가 없는 물에 보관되었다는 점을 감안할 때 주로 난소에서 모계로 유전될 수 있다고 제안했습니다. 그러나 그들은 산란 직후 세균 집락화가 일어날 가능성을 배제할 수 없었고, 난소 마이크로바이옴16에 대한 추가 연구를 권장했다.
처음에는 Culex pipiens 종 복합체의 표본을 위해 설계되었지만, 우리는 이 프로토콜이 Anopheles 또는 Aedes와 같은 다른 벡터를 연구하는 더 광범위한 의료 곤충학자 커뮤니티에 적용될 수 있을 것으로 예상합니다. 이 방법은 개별 장기 수준에서 작동함으로써 개체 내 및 개체 간 게놈 비교를 모두 가능하게 할 수 있으며, 벡터 제어 전략을 발전시킬 수 있는 잠재력과 함께 미세한 규모에서 공생체 게놈 변동성에 대한 통찰력을 제공할 수 있습니다. 침샘, 난소 및 중장을 절개하고 분리하여 미생물 교차 오염을 방지하는 이 방법은 이 세 기관 내 바이러스 감염 역학 연구에도 유용한 프로토콜이 될 수 있습니다.
공개
저자는 이해 상충이 없다고 선언합니다.
감사의 말
조던 투타가타(Jordan Tutagata)에게 모기의 침샘을 해부하는 방법을 가르쳐 준 길버트 레고프(Gilbert Legoff)와 모기 기관 사진을 촬영하는 데 도움을 준 줄리아노 무치(Giuliano Mucci)에게 감사드립니다. 프로토콜에 대한 유용한 토론을 해준 Anna-Bella Failloux와 Nonito Pages에게 감사드립니다. 이 작업은 ERC RosaLind Starting Grant "948135"의 지원을 받아 JR에 이루어졌습니다. Vectopole 플랫폼(IRD, Montpellier)이 기술 지원을 제공하고 모기 개체군의 사육 및 유지에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| Alcohol 96° | Fisher scientific | 10332562 | |
| Binocular magnifier | |||
| Bleach | RAJA | 145517 | 150 tablets of 1.5 g |
| DNA prep kit | Illumina | Provided by MGX sequencing platform | Previously known as Nextera DNA Flex |
| DNA-RNA Shield (50 mL) | Zymo research | ZR1100-50 | Preservation buffer |
| DNeasy Blood and Tissue Kit | Qiagen | 69504 | DNA extraction kit |
| Filter tips 20 µL | Starlab | S1120-3810 | |
| Filter tips 200 µL | Starlab | S1120-8710 | |
| Filter tips 1000 µL | Starlab | S1122-1730-C | |
| Forceps | FST (Fine Science Tools) | 11252-20 | Dumont Forceps #5 |
| Library quantification kit | Roche | Provided by MGX sequencing platform | KAPA Library Quanitification Kits |
| Micropipettes 2-20 µL | Eppendorf | 6.291704 | |
| Micropipettes 20-200 µL | Eppendorf | 6.291703 | |
| Micropipettes 100-1000 µL | Eppendorf | 7.648488 | |
| Microscope slides | Epredia | J1800BMNZ | dimension : 75 mm x 50 mm |
| Needles | Terumo | AN*2719R1 | |
| NGS kit | Agilent | Provided by MGX sequencing platform | Fragment Analyzer Systems HS Genomic DNA 50kb Kit |
| NovaSeq 6000 | Illumina | Provided by MGX sequencing platform | Sequencer |
| PBS Phosphate Buffered Saline (Sterile) | Fisher scientific | 10212990 | |
| Permanent black marker | |||
| Sterile Eppendorf | Dutscher | 33871 | 1.5 mL |
| Support for needles | FST (Fine Science Tools) | 26016-12 | Moria MC1 Pin Holder 12 cm |
참고문헌
- Achee, N. L., et al. Alternative strategies for mosquito-borne arbovirus control. PLOS Negl Trop Dis. 13 (1), e0006822 (2019).
- Hoffmann, A. A., et al. Successful establishment of Wolbachia in Aedes populations to suppress dengue transmission. Nature. 476 (7361), 454-457 (2011).
- Porter, J., Sullivan, W. The cellular lives of Wolbachia. Nat Rev Microbiol. 21 (11), 750-766 (2023).
- Coon, K. L., Brown, M. R., Strand, M. R. Mosquitoes host communities of bacteria that are essential for development but vary greatly between local habitats. Mol Ecol. 25 (22), 5806-5826 (2016).
- Didion, E. M., et al. Microbiome reduction prevents lipid accumulation during early diapause in the northern house mosquito, Culex pipiens pipiens. J Insect Physiol. 134, 104295 (2021).
- Gabrieli, P., et al. Mosquito trilogy: Microbiota, immunity and pathogens, and their implications for the control of disease transmission. Front Microbiol. 12, 630438 (2021).
- Garrigós, M., Garrido, M., Panisse, G., Veiga, J., Martínez-de La Puente, J. Interactions between West Nile Virus and the microbiota of Culex pipiens vectors: A literature review. Pathogens. 12 (11), 1287 (2023).
- Zheng, R., et al. Holobiont perspectives on tripartite interactions among microbiota, mosquitoes, and pathogens. ISME J. 17 (8), 1143-1152 (2023).
- Flores, G. A. M., et al. Wolbachia dominance influences the Culex quinquefasciatus microbiota. Sci Rep. 13 (1), 18980 (2023).
- Hegde, S., et al. Interkingdom interactions shape the fungal microbiome of mosquitoes. Anim Microbiome. 6 (1), 11 (2024).
- Suo, P., et al. Seasonal variation of midgut bacterial diversity in Culex quinquefasciatus populations in Haikou city, Hainan province, China. Biology. 11 (8), 1166 (2022).
- Gruber-Vodicka, H. R., Seah, B. K. B., Pruesse, E. phyloFlash: Rapid small-subunit rRNA profiling and targeted assembly from metagenomes. mSystems. 5 (5), e00920-e00920 (2020).
- Graña-Miraglia, L., et al. Spirochetes isolated from arthropods constitute a novel genus Entomospira genus novum within the order Spirochaetales. Sci Rep. 10 (1), 17053 (2020).
- Reveillaud, J., et al. The Wolbachia mobilome in Culex pipiens includes a putative plasmid. Nat Commun. 10 (1), 1051 (2019).
- Ghousein, A., et al. pWCP is a widely distributed and highly conserved Wolbachia plasmid in Culex pipiens and Culex quinquefasciatus mosquitoes worldwide. ISME Commun. 3 (1), 40 (2023).
- Juma, E. O., Kim, C. -. H., Dunlap, C., Allan, B. F., Stone, C. M. Culex pipiens and Culex restuans egg rafts harbor diverse bacterial communities compared to their midgut tissues. Parasit Vectors. 13 (1), 532 (2020).
- Lee, M. D. GToTree: a user-friendly workflow for phylogenomics. Bioinformatics. 35 (20), 4162-4164 (2019).
- Parks, D. H., et al. A complete domain-to-species taxonomy for bacteria and Archaea. Nat Biotechnology. 38 (9), 1079-1086 (2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유