Method Article
초저온 전자 단층촬영(cryo-electron tomography) 데이터의 몰입형 세분화 및 분석을 위한 가상 현실 활용
요약
초저온 전자 단층촬영(cryo-ET)을 사용하면 나노미터 분해능에서 세포 초미세 구조를 3D로 시각화할 수 있지만 수동 분할은 여전히 시간이 많이 걸리고 복잡합니다. 우리는 cryo-ET 단층촬영을 위한 고급 가상 현실 소프트웨어를 통합한 새로운 워크플로우를 제시하고, 포유류 세포의 미토콘드리아를 분할하여 그 효과를 보여줍니다.
초록
초저온 전자 단층촬영(Cryo-ET)은 나노미터 분해능에서 세포의 초미세 구조를 3차원(3D)으로 시각화하는 강력한 기술입니다. 그러나 cryo-ET 데이터에서 세포 구성 요소의 수동 분할은 복잡성과 시간 소모적인 특성으로 인해 상당한 병목 현상으로 남아 있습니다. 이 작업에서는 고급 가상 현실(VR) 소프트웨어를 통합하여 cryo-ET 데이터 세트 분할의 효율성과 정확성을 모두 향상시키는 새로운 세분화 워크플로우를 제시합니다. 이 워크플로우는 직관적인 3D 상호 작용이 가능한 몰입형 VR 도구를 활용하여 사용자가 보다 자연스럽고 인터랙티브한 환경에서 복잡한 세포 구조를 탐색하고 주석을 달 수 있도록 합니다. 워크플로우의 효과를 평가하기 위해 망막 색소 상피(RPE1) 세포의 미토콘드리아를 분할하는 데 적용했습니다. 세포 에너지 생산 및 신호 전달에 필수적인 미토콘드리아는 역동적인 형태학적 변화를 나타내므로 이상적인 테스트 샘플입니다. VR 소프트웨어는 미토콘드리아 막과 내부 구조를 정밀하게 묘사할 수 있도록 하여 분절된 막 구조의 다운스트림 분석을 가능하게 했습니다. 이를 통해 이 VR 기반 세분화 워크플로우가 사용자 경험을 크게 개선하는 동시에 cryo-ET 데이터에서 복잡한 세포 구조의 정확한 세분화를 유지한다는 것을 입증했습니다. 이 접근 방식은 구조 세포 생물학 및 과학 교육에 광범위하게 적용될 수 있을 것으로 기대되며, 상세한 세포 분석에 종사하는 연구자에게 혁신적인 도구를 제공합니다.
서문
초저온 전자 단층촬영(cryo-ET)은 고해상도 1,2에서 거의 네이티브 상태의 세포 구성 요소를 시각화하는 능력에 혁명을 일으켰습니다. 이 강력한 기술을 통해 연구원들은 세포 미세 구조를 특성화하여 세포 구조 및 기능에 대한 전례 없는 통찰력을 제공할 수 있습니다. 그러나 Cryo-ET에 한계가 없는 것은 아니며, 그 중 가장 중요한 것은 표준 초저온 투과 전자 현미경에서 이미징하기 위해 전자 투명(일반적으로 <0.5μm)할 수 있을 만큼 샘플이 얇아야 한다는 요구 사항입니다. 최근 극저온 집속 이온 빔 밀링의 발전으로 cryo-ET 분석을 위해 두꺼운 시료를 얇게 만들 수 있게 되었습니다 3,4.
일반적인 cryo-ET 워크플로우는 +60°에서 -60°에 이르는 다양한 각도에서 시료를 이미지화하는 틸트 시리즈 컬렉션으로 시작됩니다. 그런 다음 이러한 이미지를 계산적으로 정렬하고 역투영하여 3차원(3D) 볼륨 또는 단층촬영도 5,6,7을 생성합니다. 이 단층촬영은 세포 지형에 대한 상세한 3D 지도 역할을 하며, 세포 구조에 대한 공간적, 시간적 정보를 모두 제공합니다. 동일한 구조의 여러 복사본이 정렬되고 평균화되는 서브토모그램 평균화를 통한 추가 개선은 분해능 한계를 더욱 높일 수 있으며 때로는 나노미터 미만의 분해능 7,8,9,10을 달성할 수 있습니다.
이러한 단층촬영에서 의미 있는 생물학적 정보를 추출하는 데 있어 중요한 단계는 세분화(segmentation)입니다. 이 프로세스에는 3D 볼륨 내에서 멤브레인과 같은 특정 세포 구조에 주석을 추가하는 작업이 포함됩니다. 세분화를 통해 막간 거리 및 멤브레인 곡률 계산을 포함한 고급 분석을 가능하게 하여 세포 과정에 대한 귀중한 통찰력을 제공합니다11,12. 이 작업에 Dragonfly, Amira, MemBrain, EMAN2 및 tomomemsegtv13,14,15,16,17을 포함한 여러 소프트웨어 패키지를 사용할 수 있지만 세분화 프로세스는 cryo-ET 데이터 분석에서 여전히 심각한 병목 현상으로 남아 있습니다. 이는 종종 노동 집약적이고 시간이 많이 소요되는 수동 프로세스이며 완료하는 데 몇 주에서 몇 달이 걸릴 수 있습니다. 이러한 패키지 중 다수는 자동 세분화 기능을 제공하지만, 오탐(false positive)을 제거하기 위해 광범위한 수동 수정이 필요한 경우가 많으며, 이는 슬라이스 단위 또는 3D로 수행할 때 힘들고 직관적이지 않을 수 있는 프로세스입니다.
우리는 이러한 문제를 해결하기 위해 가상 현실(VR) 기술을 활용하여 대안적인 접근 방식을 제안합니다. VR은 데이터 시각화를 위한 몰입형 대화형 방법을 제공하여 사용자가 마치 세포 환경 내부에 있는 것처럼 단층 촬영 볼륨을 탐색할 수 있도록 합니다. 또한 이 접근 방식은 몰입감 있고 독특하고 매력적인 경험을 제공함으로써 과학 교육과 현장 Cryo-ET 데이터의 과학적 탐사 및 발견을 위한 귀중한 플랫폼을 제공합니다. 이 작업에서는 과학적 시각화를 위해 설계된 VR 소프트웨어인 syGlass18을 사용하여 cryo-ET 데이터 분할을 위한 프로토콜을 제시합니다. 이 소프트웨어는 수동 분할, 자동 생성된 세분화 개선, 단층촬영 내 입자 선별을 포함한 cryo-ET 데이터 분석을 위한 포괄적인 툴킷을 제공합니다. 우리의 연구는 VR이 cryo-ET 데이터 분석에서 세척 분할, 입자 선별 및 수동 분할을 위한 강력한 도구로서 실행 가능성을 보여줍니다.
cryo-ET 분할을 위한 VR 소프트웨어의 유용성을 설명하기 위해 망막 색소 상피(RPE1) 세포의 미토콘드리아 형태 분석에 중점을 둡니다. 미토콘드리아는 복잡한 구조와 외부 미토콘드리아 막과 내부 미토콘드리아 막 사이의 거리와 같이 쉽게 정량화할 수 있는 특징의 존재로 인해 세분화를 위한 훌륭한 테스트 케이스 역할을 합니다. 이러한 특징은 표면 형태 측정 분석 도구(12)를 사용하여 정확하게 측정할 수 있으며, 이는 세분화 품질을 평가하기 위한 강력한 메트릭을 제공합니다. 이 프로토콜은 syGlass를 사용하여 cryo-ET 데이터를 분할하기 위한 단계별 지침을 제공하여 cryo-ET 분할 파이프라인 내에서 그 유용성을 입증합니다. VR 기반 분할을 cryo-ET 워크플로우에 통합함으로써 세포 생물학에서 수동 구조 분석의 효율성과 정확성을 모두 향상시키는 것을 목표로 합니다.
프로토콜
1. 세분화를 위한 cryo-ET 데이터 준비
- 원시 Cryo-ET 단층촬영을 TIFF 스택과 같은 syGlass와 호환되는 데이터 형식으로 변환할 수 있습니다. 입자가 검은색 바탕에 흰색이 되도록 신호를 설정하고 ImageJ19로 히스토그램 평활화를 수행합니다.
참고: 신호 대 잡음비를 향상시키려면 소프트웨어로 가져오기 전에 Warp20, Topaz Denoise21, cryoCARE22,23 또는 IsoNet24와 같은 소프트웨어로 단층 촬영의 노이즈를 제거하는 것이 좋습니다. - 컴퓨터에서 VR 소프트웨어를 실행합니다. 파일 메뉴로 이동하여 프로젝트 만들기를 선택합니다.
- Create New Project(새 프로젝트 만들기) | 파일 추가를 클릭합니다. TIFF 파일이 저장된 위치로 이동하여 소프트웨어로 가져옵니다.
- 파일이 시계열인지 확인하라는 메시지가 표시되면 No(아니요)를 클릭합니다.
- 프로젝트 이름을 지정한 다음 저장을 클릭하여 프로젝트 목록 아래에 새 프로젝트를 만듭니다.
- 프로젝트를 두 번 클릭하여 단층 촬영을 열고 소프트웨어 대화형 가상 현실 환경에 로드합니다.
2. 가상 현실(VR) 설정
- VR 헤드셋과 핸드 컨트롤러를 컴퓨터에 연결합니다.
- 화면의 지시에 따라 VR 환경을 보정합니다.
참고: 보정은 세분화가 수행되는 3차원 공간을 나타냅니다. 작업 공간의 매개변수(길이, 너비 및 높이)는 VR 투영을 표시하기 전에 정의해야 합니다. VR 프로젝션은 이 매개변수에서만 볼 수 있습니다. - 세분화는 정의된 작업 3차원 공간 내에서만 수행됩니다. 시야각에 VR 환경에서 작업하고자 하는 영역이 포함되어 있는지 확인합니다.
3. 3D 시각화 최적화
- 소프트웨어 인터페이스에서 시각화 버튼을 클릭합니다. 대비, 윈도우, 밝기 및 임계값 슬라이더와 같은 다양한 시각화 옵션을 조정하여 신호를 향상시키고 노이즈를 최소화합니다.
- 핸드 컨트롤러를 사용하여 단층 촬영을 더 가까이 당기거나 필요에 따라 밀어냅니다.
- 왼쪽 컨트롤러를 사용하여 Cut 도구를 활성화하여 단층 촬영 내의 다양한 슬라이스를 시각적으로 검사합니다.
4. 세분화 프로세스
- 단층촬영을 통해 분할이 시작될 원하는 슬라이스로 이동합니다.
- 핸드 컨트롤러를 사용하여 주석 메뉴에서 Region of Interest (ROI) 옵션을 활성화합니다. 단층촬영(tomogram) 내에 녹색 상자가 나타납니다.
- 녹색 상자의 크기와 위치를 조정하여 세그먼트화할 영역으로 이동합니다.
- 왼쪽 컨트롤러를 사용하여 ROI를 잠그고 세분화를 시작합니다. ROI를 잠근 후 도구는 체적 데이터를 분할할 수 있는 페인트 모드로 자동 전환됩니다.
- 정확한 분할을 위해 단층촬영을 확대 또는 축소할 수 있습니다.
- 최적의 제어를 위해 시계 방향 또는 시계 반대 방향으로 회전하여 페인트 브러시 크기를 조정합니다.
- 선택한 3D 영역 내에서 ROI(예: 미토콘드리아 막)를 신중하게 분할합니다. 세그멘테이션 중에 오류가 발생하면 보조 컨트롤러 트리거를 길게 눌러 지우기 모드를 시작한 다음 세그멘테이션과 동일한 동작을 사용하여 오류를 지웁니다.
- 전체 단층촬영이 분할될 때까지 이 과정을 영역별로 반복합니다.
5. 세분화된 데이터 및 분석 내보내기
- 세분화를 완료한 후 프로젝트를 클릭하여 선택하여 강조 표시되어 있는지 확인합니다.
- 왼쪽 상단 모서리에 있는 Projects 탭을 클릭한 다음 ROIs를 선택합니다.
- 전체 볼륨 또는 특정 ROI를 내보내도록 선택합니다. 세그먼트화된 데이터의 내보내기 위치를 지정합니다.
- 선택한 소프트웨어를 사용하여 분할된 데이터를 로드 및 분석하고 출판을 위한 그림을 생성합니다.
6. 정리를 위해 바이너리 마스크를 소프트웨어로 가져오기
- 1.1-1.6단계를 수행합니다.
- 프로젝트를 마우스 오른쪽 버튼으로 클릭하고 마스크 데이터 추가를 클릭합니다. 그런 다음 초기 세분화가 저장된 위치로 이동하여 동일한 프로젝트 아래로 가져옵니다.
- ROI 주석을 사용하여 초기 세분화를 편집합니다.
- 세그멘테이션을 추가/지우기 위해 초기 세그멘테이션을 정리합니다.
- 마스크 탭의 ROI 옵션에서 마스크 ROI를 조정합니다. 마스크 ROI를 한 항목에서 다른 값으로 변경하려면 마스크 덮어쓰기 옵션만 켜고 핸드 컨트롤러로 페인트 모드로 이동합니다. 이 모드는 보이는 마스크 데이터만 덮어씁니다.
- 5.1-5.3단계에 설명된 단계를 사용하여 조정된 마스크 ROI를 내보냅니다.
7. VR 소프트웨어를 사용하여 입자 좌표 따기
- 섹션 1에 설명된 단계를 사용하여 단층촬영(TIFF 스택)을 사용하여 프로젝트를 만듭니다.
- Annotation (주석)을 클릭하십시오. Count: 수동 또는 자동 옵션이 있는 창을 엽니다.
- 수동 옵션의 경우 카운트의 색상을 선택하고 좌표 표시를 시작합니다.
- automated 옵션의 경우 셀 크기를 선택하고 해상도를 입력합니다. 자동 계산 실행을 클릭하고, 개수를 편집하고(좋은 선택은 유지하고 잘못된 선택은 폐기), 진행 상황을 저장합니다.
- Export the count coordinates by click on the project tab | annotations(주석)를 클릭합니다.
- 주석 원점의 경우 왼쪽 하단 모서리 를 선택하고 주석 단위의 경우 Voxels를 선택합니다.
- 계산 포인트 셀을 클릭하여 셀을 강조 표시합니다.
- Export selected를 클릭하여 선택 항목의 좌표를 .csv 파일에 저장합니다.
참고: 이 파일은 서브토모그램 평균화를 위해 단층촬영에서 서브토모그램을 추출하는 데 사용할 수 있습니다
결과
이 연구에서는 syGlass를 사용하여 미토콘드리아와 추가 막성 소기관(예: 소포, 소포체)을 포함하는 단층촬영을 분할했습니다. 단층 도선은 처음에 16.00 Å/pixel에서 가중 투영을 사용하여 Warp에서 재구성되었으며 소프트웨어 IsoNet을 사용하여 누락된 쐐기 보정 및 잡음 제거를 거쳤습니다. 다음 단층 촬영은 그림 1A 와 같이 가져오기를 위한 추가 처리를 거쳤습니다( 또한 보충 비디오 S1, 보충 비디오 S2, 보충 비디오 S3 및 보충 비디오 S4 참조).
전처리 후 MRC 형식의 단층촬영은 ImageJ를 사용하여 TIFF 스택으로 변환되었으며, 대비 반전이 적용되어 멤브레인이 검은색 배경에 흰색으로 보이도록 했습니다. 그런 다음 대비를 더욱 향상시키기 위해 히스토그램 이퀄라이제이션을 수행했습니다. 이를 통해 보다 효과적인 임계값을 설정할 수도 있습니다. TIFF 스택은 몰입형 VR 환경 내에서 3D로 가져와 시각화하여 그림 1B에 표시된 소프트웨어의 절단 도구를 사용하여 막질 구조를 자세히 검사할 수 있습니다.
소프트웨어에서 수동 세분화가 수행되었으며, 윈도잉은 자동으로 설정되고 밝기/임계값 조정이 이루어졌으며 셀룰러 기능 가시성을 최적화했습니다. 미토콘드리아와 다른 구조에 대한 ROI는 VR 컨트롤러를 사용하여 정의되었습니다. 세분화 도구를 사용하면 멤브레인 경계를 정확하게 묘사할 수 있었고 erase 기능을 사용하여 오류를 수정할 수 있었습니다. ROI 도구를 사용하여 단층촬영 전체 영역을 boxing하기 위한 슬라이스 별 탐색 또는 3D는 조정 가능한 페인트 브러시 도구와 결합되어 그림 1C와 같이 미토콘드리아 막 및 기타 소기관의 정확한 분할을 보장했습니다.
분할된 데이터는 ROI 도구의 표면 옵션을 사용하여 메시를 생성하여 시각화했으며, 스무딩 반복은 12로, 해상도 수준은 3으로 설정되었습니다. 최종 3D 렌더링은 그림 1D와 같이 외막과 내막, 크리스태 및 인산칼슘 침전물을 포함한 미토콘드리아 구조를 명확하게 보여줍니다(보충 동영상 S5, 보충 동영상 S6, 보충 동영상 S7 및 보충 동영상 S8 참조).
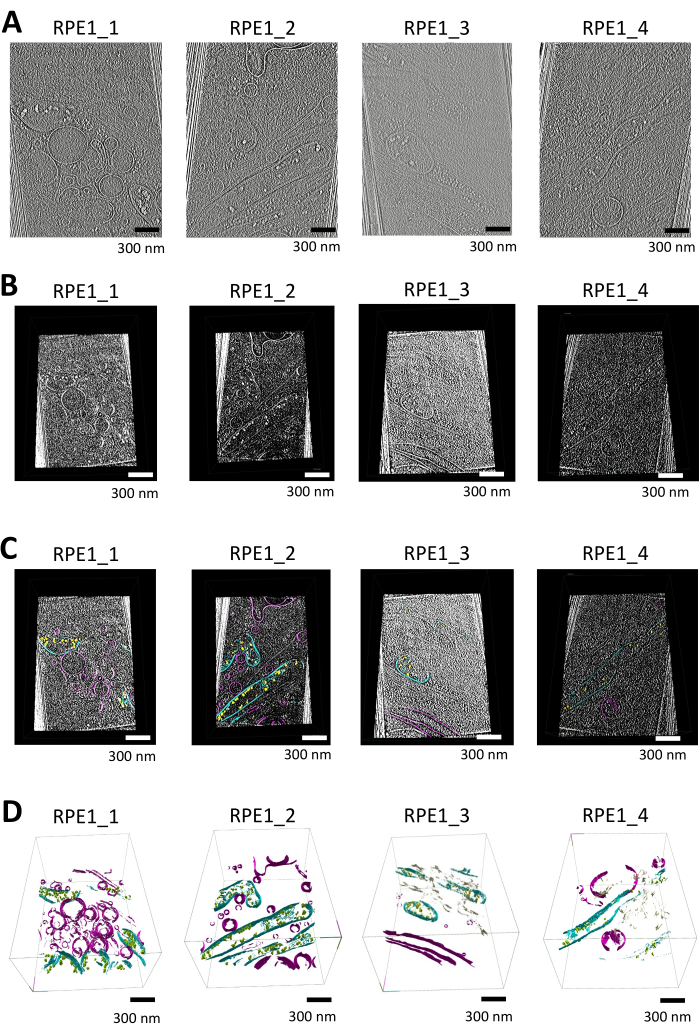
그림 1: syglass를 사용한 단층 촬영 시각화, 세분화 및 3D 렌더링의 작업 흐름. (A) 얇은 가장자리 RPE1 세포의 단층 촬영 절편. Warp에서 재구성하고 IMOD25로 시각화한 syGlass를 사용한 분할에 사용되는 IsoNet 보정 단층 촬영. 단층 촬영은 K3 검출기가 장착된 300keV Titan-Krios 현미경에서 수집되었으며 망막 상피 세포의 얇은 가장자리에 초점을 맞췄습니다. 각 단층촬영에는 다양한 다른 막 결합 소기관과 함께 적어도 하나의 미토콘드리아가 포함되어 있습니다. 이러한 이미지는 추가 비디오 S1, 추가 비디오 S2, 추가 비디오 S3 및 추가 비디오 S4에 표시된 비디오의 스틸 이미지입니다. (B) 소프트웨어에서 시각화된 해당 단층 촬영. VR 소프트웨어의 절단 도구를 사용하여 임계값, 밝기 및 윈도잉을 최적화한 후 생성된 단층 촬영 슬라이스는 뚜렷한 세포 구조를 드러냅니다. (C) 분절된 멤브레인이 있는 소프트웨어에서 시각화된 단층 촬영. VR 소프트웨어의 자르기 도구로 만든 단층 촬영 슬라이스는 해당 세분화로 오버레이됩니다. 인산칼슘 침전물은 노란색, 미토콘드리아 막은 청록색, 소포 및 원형질막과 같은 다른 막은 보라색으로 표시됩니다. (D) syGlass에서 분절된 멤브레인의 3D 렌더링. 크리스테(cristae), 바깥쪽 및 안쪽 미토콘드리아 막(outer and inner mitochondrial membranes)을 포함한 미토콘드리아 막(mitochondrial membranes)은 시안(cyan)으로 표시됩니다. 인산칼슘 침전물은 노란색 또는 녹색이고, 소포와 원형질막은 보라색이며, 소포체는 황갈색입니다. 이러한 이미지는 보충 동영상 S5, 추가 동영상 S6, 추가 동영상 S7 및 추가 동영상 S8에 표시된 동영상의 스틸 이미지입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 비디오 S1: 이 연구에서 분할에 사용된 샘플 RPE-1_1의 단층 촬영. 이 단층 촬영은 Warp를 사용하여 16 Å/pixel로 재구성한 다음 IsoNet을 사용하여 누락된 쐐기를 제거하고 수정합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 비디오 S2: 이 연구에서 분할에 사용된 샘플 RPE-1_2의 단층 촬영. 이 단층 촬영은 Warp를 사용하여 16 Å/pixel로 재구성한 다음 IsoNet을 사용하여 누락된 쐐기를 제거하고 수정합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 비디오 S3: 이 연구에서 분할에 사용된 샘플 RPE-1_3의 단층 촬영. 이 단층 촬영은 Warp를 사용하여 16 Å/pixel로 재구성한 다음 IsoNet을 사용하여 누락된 쐐기를 제거하고 수정합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 동영상 S4: 이 연구에서 분할에 사용된 샘플 RPE-1_4의 단층 촬영. 이 단층 촬영은 Warp를 사용하여 16 Å/pixel로 재구성한 다음 IsoNet을 사용하여 누락된 쐐기를 제거하고 수정합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 비디오 S5: 표면 생성 후 샘플 RPE-1_1의 결과 세분화. 미토콘드리아 막은 청록색으로 표시됩니다. 미토콘드리아 내의 인산칼슘은 노란색으로 표시됩니다. 소포체는 황갈색으로 표시됩니다. 다른 멤브레인은 자주색으로 표시됩니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 동영상 S6: 표면 생성 후 샘플 RPE-1_2의 결과 세분화. 미토콘드리아 막은 청록색으로 표시됩니다. 미토콘드리아 내의 인산칼슘은 노란색으로 표시됩니다. 소포체는 황갈색으로 표시됩니다. 다른 멤브레인은 자주색으로 표시됩니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 비디오 S7: 표면 생성 후 샘플 RPE-1_3의 결과 분할. 미토콘드리아 막은 청록색으로 표시됩니다. 미토콘드리아 내의 인산칼슘은 노란색으로 표시됩니다. 소포체는 황갈색으로 표시됩니다. 다른 멤브레인은 자주색으로 표시됩니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 비디오 S8: 표면 생성 후 샘플 RPE-1_4의 결과 세분화. 미토콘드리아 막은 청록색으로 표시됩니다. 미토콘드리아 내의 인산칼슘은 노란색으로 표시됩니다. 소포체는 황갈색으로 표시됩니다. 다른 멤브레인은 자주색으로 표시됩니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
이 작업에서는 특히 syGlass를 사용하는 VR을 cryo-ET 파이프라인에 통합하여 세포 구조를 효과적으로 분할하는 방법을 시연했습니다. 우리의 초점은 주로 멤브레인에 있었지만, 브러시 모양과 크기를 조정할 수 있고 원하는 세포 개체에 해당하는 복셀만 표시되도록 임계값을 조정할 수 있기 때문에 필라멘트 또는 리보솜과 같은 다른 세포 구조의 분할을 방해하는 고유한 제한은 없습니다. 세분화에서 VR의 주요 장점 중 하나는 데이터와의 직관적이고 직접적인 상호 작용으로, 이를 통해 사용자는 마치 물리적으로 그 안에 있는 것처럼 볼륨을 시각화하고 조작할 수 있습니다. 수동 세그멘테이션 또는 초기 세그멘테이션 마스크 정리의 전통적인 방법은 일반적으로 컴퓨터에서 직접 작업하는 것인데, 이는 몰입도가 떨어지고 주석 달기가 느릴 수 있습니다.
VR을 cryo-ET 워크플로우에 통합함으로써 사용자는 다른 소프트웨어에서 생성된 분할 마스크와 빠르게 상호 작용할 수 있을 뿐만 아니라 VR을 사용하여 부분적으로 분할된 구조의 분할을 안내하고 거짓 양성을 효율적으로 정리할 수 있습니다. 현재 대부분의 주석 사용 사례에는 여전히 수동 세분화가 필요하며, 제시된 워크플로를 통해 사용자는 향상된 용이성과 속도로 다운스트림 분석에 적합한 세분화를 생성할 수 있습니다. 이 연구를 위해 HTC Vive VR 헤드셋을 사용했지만 소프트웨어는 SteamVR 또는 OculusVR을 지원하는 장치와 호환됩니다.
이 프로토콜의 최적 적용을 위해 cryo-ET 데이터는 높은 신호 대 잡음비(SNR)의 고품질 단층촬영과 같은 특정 기준을 충족해야 합니다. SNR은 구조적 특징의 명확성이 VR에서 수동 세그멘테이션의 효율성에 직접적인 영향을 미치기 때문에 필수적인 매개변수입니다. 누락된 쐐기 보정 및 노이즈 제거와 같은 전처리 단계는 워크플로우의 핵심 부분입니다. 이 프로토콜에서는 이러한 목적으로 IsoNet을 활용했습니다. 단층촬영은 VR 시각화를 위해 관리 가능한 데이터 크기를 더욱 유지하면서 구조 간의 효과적인 분할을 가능하게 하기 위해 충분한 구조적 세부 사항과 세포 구조 간의 충분한 분리를 제공하는 복셀 크기를 사용하여 적절한 해상도로 재구성해야 합니다. 또한 검은색 배경에 흰색으로 나타나는 세포 구조와의 반전 대비와 히스토그램 이퀄라이제이션을 적용하여 syGlass 환경 내에서 멤브레인 및 기타 구조의 가시성을 향상시켜야 합니다.
이 프로토콜의 성공을 보장하기 위해 몇 가지 중요한 사항을 고려해야 합니다. 첫째, 데이터 전처리 중에 대비 반전 및 히스토그램 이퀄라이제이션의 정확한 적용이 중요합니다. 부적절한 조정은 VR에서 최적화되지 않은 시각화를 초래하여 세분화를 어렵게 만들 수 있습니다. 둘째, VR 소프트웨어 내에서 윈도잉, 밝기 및 임계값 설정을 적절하게 조정하는 것은 구조물의 최적 시각화를 위해 필수적입니다. 사용자는 특정 데이터 세트 및 VR 설정에 대해 최상의 결과를 얻으려면 이러한 설정을 실험해야 합니다.
문제 해결과 관련하여 사용자는 특히 대규모 단층 촬영 데이터 세트를 처리할 때 VR 시스템 성능 제한과 같은 문제에 직면할 수 있습니다. VR 환경이 느려지거나 반응이 없는 경우, syGlass의 ROI 툴에서 바운딩 박스를 조정하고 세그멘테이션이 수행될 때 토모그램을 따라 이동하여 데이터를 다운샘플링하거나 단층촬영을 더 작은 섹션으로 분할하는 것을 고려해 보십시오. 시각화 메뉴의 성능 슬라이더를 조정하여 세분화 프로세스 중에 지연을 줄일 수도 있습니다. 또한 멀미를 완화하기 위해 사용자는 VR 설정을 조정하여 모션 효과를 줄이거나 세분화 세션 중에 정기적으로 휴식을 취할 수 있습니다.
당사의 VR 기반 분할 프로토콜은 현재의 cryo-ET 분할 워크플로우에 상당한 이점을 제공합니다. 기존의 수동 분할 방법에는 종종 개별 슬라이스에 대한 2D 주석이 포함되는데, 이는 시간이 많이 소요될 수 있고 3차원 연속성을 완전히 포착하지 못할 수 있습니다13. 또한 VR 기반 세분화는 세포 구조에 대한 훨씬 더 몰입감 있는 보기를 제공하여 시각화를 지원합니다. 자동화된 머신 러닝 세분화는 세포 단층촬영 14,16,17에서 세분화를 얻는 강력한 방법으로 부상하고 있지만, cryo-ET 데이터에 존재하는 높은 노이즈 수준과 복잡한 구조로 인해 수동 개입이 필요한 갭과 거짓 긍정이 발생합니다. 이 프로토콜은 초저온 전자 단층촬영을 수동으로 분할하거나, 다른 신경망 소프트웨어의 훈련 데이터로 사용될 수 있는 초기 분할을 생성하거나, 다른 자동화된 접근 방식에서 생성된 초기 분할을 정리하는 대체 접근 방식을 제공합니다.
결론적으로, 이 연구는 VR 기반 세분화를 cryo-ET 데이터 분석 및 교육을 위한 유망한 도구로 강조하여 향상된 효율성과 보다 몰입감 있는 사용자 경험을 제공합니다. VR 기술은 향후 개발이 이루어짐에 따라 cryo-ET 데이터 세트에서 복잡한 세포 구조에 대한 과학적 발견을 해석하고 전파하는 방식에 혁명을 일으킬 수 있는 잠재력을 가지고 있으며, 기존의 세분화 및 교육 방법에 대한 가치 있는 대안을 제공합니다.
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
이 작업은 NIH(Common Fund U24GM129539, U24GM139171 및 NIGMS R24GM154192), Simons Foundation(SF349247) 및 NY State Assembly의 지원을 받아 New York Structural Biology Center에 위치한 NCCAT(National Center for CryoEM Access and Training)와 Simons Electron Microscopy Center에서 수행되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| CryoET Data | Format:TIFF-stack, TIFF | ||
| HTC VIVE Cosmos | HTC | 99HARL000-00 | https://www.vive.com/sea/product/vive-cosmos/features/ |
| Intel(R) Core(TM) i7-10870H CPU @ 2.20 GHz 2.21 GHz | Intel | https://ark.intel.com/content/www/us/en/ark/products/208018/intel-core-i7-10870h-processor-16m-cache-up-to-5-00-ghz.html | |
| NVIDIA GeForce RTX 3070 Laptop GPU | NVIDIA | https://www.nvidia.com/en-us/geforce/laptops/compare/30-series/ | |
| syGlass Software | syGlass | syGlass Software installed on a compatible Windows PC | |
| VIVE Cosmos Hand Controllers | HTC | 99HAFR001-00 | https://www.vive.com/us/accessory/cosmos-controller-right/ |
| Windows 11 Home | Microsoft | Microsoft Windows 11 Home |
참고문헌
- Plitzko, J. M., Schuler, B., Selenko, P. Structural biology outside the box-inside the cell. Curr Opin Struct Biol. 46, 110-121 (2017).
- Beck, M., Baumeister, W. Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail. Trends Cell Biol. 26 (11), 825-837 (2016).
- Lam, V., Villa, E. Practical approaches for cryo-FIB milling and applications for cellular cryo-electron tomography. Methods Mol Biol. 2215, 49-82 (2021).
- Berger, C., et al. Cryo-electron tomography on focused ion beam lamellae transforms structural cell biology. Nat Methods. 20 (4), 499-511 (2023).
- Watson, A. J. I., Bartesaghi, A. Advances in cryo-ET data processing: meeting the demands of visual proteomics. Curr Opin Struct Biol. 87, 102861 (2024).
- Pyle, E., Zanetti, G. Current data processing strategies for cryo-electron tomography and subtomogram averaging. Biochem J. 478 (10), 1827-1845 (2021).
- Wan, W., Briggs, J. A. G. Cryo-electron tomography and subtomogram averaging. Methods Enzymol. 579, 329-367 (2016).
- Ni, T., Liu, K., Zhang, J., Atanasov, I., Mladenov, M. G., Jiang, W. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nat Protoc. 17 (2), 421-444 (2022).
- Obr, M., et al. Exploring high-resolution cryo-ET and subtomogram averaging capabilities of contemporary DEDs. J Struct Biol. 214 (2), 107852 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nat Methods. 18 (2), 186-193 (2021).
- Salfer, M., Collado, J. F., Baumeister, W., Fernández-Busnadiego, R., Martínez-Sánchez, A. Reliable estimation of membrane curvature for cryo-electron tomography. PLoS Comput Biol. 16 (8), e1007962 (2020).
- Barad, B. A., et al. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. J Cell Biol. 222 (2), e202204093 (2023).
- Martinez-Sanchez, A., Garcia, I., Asano, S., Lucic, V., Fernandez, J. J. Robust membrane detection based on tensor voting for electron tomography. J Struct Biol. 186 (1), 49-61 (2014).
- Heebner, J. E., et al. Deep learning-based segmentation of cryo-electron tomograms. J Vis Exp. (189), e64435 (2022).
- Garza-López, E., Robles-Flores, M. Protocols for generating surfaces and measuring 3D organelle morphology using Amira. Cells. 11 (1), 65 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nat Methods. 14 (10), 983-985 (2017).
- Lamm, L., et al. MemBrain v2: an end-to-end tool for the analysis of membranes in cryo-electron tomography. bioRxiv. , (2024).
- Pidhorskyi, S., Morehead, M., Jones, Q., Spirou, G., Doretto, G. syGlass: interactive exploration of multidimensional images using virtual reality head-mounted displays. arXiv. , (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Tegunov, D., Cramer, P. Real-time cryo-electron microscopy data preprocessing with Warp. Nat Methods. 16 (11), 1146-1152 (2019).
- Bepler, T., Kelley, K., Noble, A. J., Berger, B. Topaz-Denoise: general deep denoising models for cryoEM and cryoET. Nat Commun. 11, 5208 (2020).
- Buchholz, T. -. O., Berninger, L., Maurer, M., Jug, F. Cryo-CARE: content-aware image restoration for cryo-transmission electron microscopy data. Proc IEEE Int Symp Biomed Imaging. 16, 502-506 (2019).
- Buchholz, T. -. O., Jordan, M., Pigino, G., Jug, F. Content-aware image restoration for electron microscopy. Methods Cell Biol. 152, 277-289 (2019).
- Liu, Y. -. T., et al. Isotropic reconstruction for electron tomography with deep learning. Nat Commun. 13, 6482 (2022).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J Struct Biol. 116 (1), 71-76 (1996).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유