Um Protocolo Simples, Rápido e Parcialmente Automatizado para o Isolamento de Núcleos Únicos de Tecidos Congelados de Mamíferos para Sequenciamento de Núcleo Único
Neste Artigo
Resumo
O estudo descreve um protocolo simples, rápido e parcialmente automatizado para isolar núcleos de alta qualidade de tecidos congelados de mamíferos para sequenciamento de RNA de núcleo único a jusante.
Resumo
O sequenciamento de RNA de célula única e de núcleo único tornou-se aplicações laboratoriais comuns devido à riqueza de informações transcriptômicas que fornecem. O sequenciamento de RNA de núcleo único, particularmente, é útil para investigar a expressão gênica em tecidos de difícil dissociação. Além disso, essa abordagem também é compatível com material congelado (arquivamento). Aqui, descrevemos um protocolo para isolar núcleos únicos de alta qualidade de tecidos congelados de mamíferos para sequenciamento de RNA de núcleo único a jusante de forma parcialmente automatizada usando instrumentos e reagentes disponíveis comercialmente. Especificamente, um dissociador robótico é usado para automatizar e padronizar a homogeneização do tecido, seguido por um gradiente químico otimizado para filtrar os núcleos. Por fim, contamos os núcleos de forma precisa e automática usando um contador automatizado de células fluorescentes. O desempenho deste protocolo é demonstrado no cérebro de camundongos, rins de ratos e tecido hepático e baço de cynomolgus. Este protocolo é simples, rápido e facilmente adaptável a vários tecidos de mamíferos sem a necessidade de otimização extensiva e fornece núcleos de boa qualidade para sequenciamento de RNA de núcleos únicos a jusante.
Introdução
O sequenciamento de RNA de célula única (sc) e núcleo único (sn) tornou-se um protocolo comumente utilizado em biologia molecular e celular devido à maior resolução da expressão gênica em comparação com o sequenciamento de RNA em massa. No entanto, o isolamento de preparações de células únicas e núcleos únicos de boa qualidade a partir de tecidos sólidos permanece um desafio e é frequentemente a etapa limitante da taxa em experimentos de sc/sn-RNAseq. De fato, uma infinidade de protocolos tem sido desenvolvida utilizando diversos procedimentos químicos e mecânicos para a obtenção de suspensões células/núcleos1,2,3,4,5,6,7,8,9,10,11,12,13,14,15 . Além disso, as estratégias para limpar essas preparações de detritos/aglomerados, etc., vão desde a classificação do fluxo até a filtragem e lavagem. Tais protocolos são frequentemente manuais (levando à variabilidade relacionada ao usuário), podem ser demorados (levando à redução da viabilidade celular/núcleo) e/ou podem exigir acesso a um citômetro de fluxo para a classificação célula/núcleo. Este estudo concentrou-se no desenvolvimento de um protocolo simples, rápido e parcialmente automatizado de isolamento de núcleos únicos a partir de tecidos congelados de mamíferos para aplicações de sequenciamento de RNA a jusante. Nós nos concentramos especificamente no isolamento de núcleos em oposição ao isolamento celular, pois é compatível com o uso de tecidos congelados, tornando a coleta/processamento de amostras mais prática e permitindo o lote imparcial de amostras, especialmente em experimentos de curso de tempo. Além disso, embora o transcriptoma nuclear não reflita completamente o transcriptoma celular, vários estudos já mostraram que os dados de sequenciamento de RNA de núcleo único são comparáveis aos dados de sequenciamento de RNA de célula única para identificação de tipo celular, embora as proporções de tipos celulares possam variar6,16,17,18,19.
O isolamento dos núcleos consiste em várias etapas: 1) ruptura mecânica ou química do tecido para liberar os núcleos, 2) limpeza de detritos e aglomerados e 3) contagem precisa dos núcleos para preparação para aplicações a jusante. Em vários protocolos, o passo 1 frequentemente envolve o uso de homogeneizador Dounce para romper o tecido 3,20. Alternativamente, métodos químicos podem ser utilizados, embora estes frequentemente precisem ser otimizados para diferentes tecidos 2,5,6. Experimentamos que um procedimento manual de ruptura tecidual é propenso à variabilidade associada ao operador, levando a qualidade e rendimento variáveis dos núcleos. Com o objetivo de minimizar a variabilidade técnica e ter um protocolo mais consistente e reprodutível que funcione entre os tecidos, foi desenvolvido um protocolo que utiliza um dissociador tecidual robótico disponível comercialmente21. Para a etapa 2, embora a troca tampão seja geralmente o meio mais simples para a lavagem dos núcleos, adotamos o uso de uma etapa relativamente curta de centrifugação por gradiente de sacarose para uma remoção mais completa dos debris. Para o tecido cerebral especificamente, usamos um gradiente coloide de sílica em vez de um gradiente de sacarose para uma remoção mais eficaz da mielina. Finalmente, para a contagem, o uso de um hemocitômetro é o padrão-ouro para contagem e inspeção visual dos núcleos. Em nosso protocolo, essa etapa pode ser automatizada de forma confiável usando um contador automatizado de células fluorescentes disponível comercialmente22. Este protocolo foi testado e é compatível com vários tecidos congelados de mamíferos, incluindo cérebro, rim, baço e fígado, de diferentes espécies de mamíferos (rato, camundongo e primata não humano) e fornece núcleos de boa qualidade para sequenciamento de RNA de núcleos únicos a jusante com uma plataforma comercial baseada em gotículas. O protocolo leva aproximadamente 75 min desde a preparação do tecido até o início do fluxo de sequenciamento de RNA de núcleos únicos.
Protocolo
Todos os estudos em animais foram realizados com a aprovação da autoridade veterinária cantonal de Basel-Stadt em estrita conformidade com os regulamentos federais suíços sobre proteção animal ou com a aprovação do Comitê Institucional de Cuidados e Uso de Animais em conformidade com a Lei Alemã de Bem-Estar Animal.
1. Preparação de tecidos e reagentes/instrumentos
- Limpeza e preparação de instrumentos
- Limpe bancadas e pinças com etanol 70% e solução de descontaminação da RNase. Pré-resfriar as centrífugas a 4 °C.
- Pré-resfriar os cartuchos de isolamento de núcleos na geladeira a 4 °C por pelo menos 30 min.
- Inicie o dissociador robótico e ligue o resfriamento definindo o controle deslizante no canto superior direito da tela para esfriar e clicando nele para começar a esfriar para que o controle deslizante apareça laranja. Verifique se o frasco de reagente de armazenamento de núcleos (NSR) anexado e o frasco de tampão de isolamento de núcleos (NIB) têm líquido suficiente e estão devidamente resfriados.
- Prepare uma caixa de espuma de poliestireno cheia de gelo seco e pré-resfrie placas de Petri e lâminas de bisturi sobre gelo seco.
- Preparação do tampão
- Preparar a solução de almofada de sacarose 1,5 M (SCS) conforme mostrado na Tabela 1. Distribuir o SCS em alíquotas de 500 μL em tubos de DNase/RNase de 2 mL para obter quatro alíquotas de SCS de 500 μL por amostra. Mantenha as alíquotas no gelo até o uso posterior.
- Em caso de processamento de tecido cerebral, preparar uma solução de coloide de sílica a 18%, conforme descrito na Tabela 2, diluindo a solução estoque de coloide de sílica em NSR e adicionando inibidor de RNase. Preparar 3 mL de solução de sílica coloide a 18% por amostra e mantê-la congelada.
2. Homogeneização tecidual e isolamento de núcleos
- Retire a amostra do congelador a -80 °C e coloque-a imediatamente sobre gelo seco.
- Corte a amostra numa placa de Petri pré-arrefecida ou placa de metal sobre gelo seco com um bisturi pré-arrefecido numa peça de 15-50 mg (se ainda não tiver o tamanho certo). Certifique-se de cortar a amostra na direção correta para que a amostra ainda seja representativa das estruturas do órgão de interesse.
NOTA: Com este protocolo, 15-50 mg é o tamanho ideal da amostra para extração nuclear. Para obter um bom rendimento após a limpeza, recomenda-se um tamanho de amostra de pelo menos 25 mg. Para amostras menores, estão disponíveis cartuchos especiais otimizados para processar pequenas entradas com o dissociador robótico. Cartuchos de isolamento de núcleos de entrada pequenos foram empregados para dissociar amostras de tecido pesando até 4 mg com rendimento suficiente para sequenciamento de RNA de núcleo único a jusante. Um exemplo de rendimento de núcleos usando o cartucho de baixa entrada do tecido hepático de rato é mostrado na Tabela 3. - Retire o cartucho de isolamento de núcleos da geladeira, desembale-o, remova o moedor e pipete 15 μL de inibidor de RNase (40 U/μL) para o fundo do cartucho.
NOTA: Durante a extração dos núcleos, o dissociador robótico adicionará NIB e NSR ao cartucho para um volume total de 3 mL (com o protocolo de extração de núcleos de baixo volume). Ao adicionar 15 μL de inibidor de RNase (40 U/μL) ao cartucho antes da extração, a suspensão terá a concentração desejada de inibidor de RNAse de 0,2 U/μL. - Coloque a amostra de tecido no fundo do cartucho usando uma pinça. Não coloque a amostra exatamente no centro do cartucho para ter a eficiência ideal de ruptura.
- Selecione Executar um protocolo no instrumento e clique na opção Núcleos no canto superior esquerdo.
- No menu, selecione o protocolo de isolamento de núcleos de baixo volume e verifique se a velocidade de interrupção está definida como rápida clicando em Modificar. Carregue o cartucho no instrumento abrindo a porta e deslizando para fora do palco levantando o botão vermelho.
- Insira o cartucho no local designado, gire a trava do cartucho e deslize no palco até que o botão vermelho se encaixe. Feche a porta e inicie a extração de núcleos no instrumento clicando em Avançar. A corrida dura aproximadamente 7 min.
- Quando a corrida terminar, remova o cartucho do instrumento levantando o botão vermelho e puxando o palco. Coloque imediatamente o cartucho no gelo.
- Para todos os tecidos, exceto o cérebro, avance para o passo 3.1. Para amostras de cérebro, prossiga diretamente para a etapa 3.2.
3. Limpeza dos núcleos
- Limpeza do gradiente de sacarose
NOTA: Para o tecido cerebral, ignore esta etapa e vá diretamente para a etapa 3.2. Todas as etapas de limpeza são realizadas no gelo, a fim de minimizar a degradação do RNA. Os tampões e tubos, bem como as centrífugas, precisam ser pré-resfriados. Todas as etapas de ressuspensão e mistura são realizadas apenas por pipetagem cuidadosa, pois o vórtice pode comprometer a qualidade e integridade dos núcleos.- Perfure cuidadosamente a folha redonda no cartucho do dissociador com uma ponta de pipeta.
NOTA: Após a dissociação, a suspensão de núcleos resultante tem um volume aproximado de 2 mL. Para facilitar a limpeza do gradiente de sacarose, divida a suspensão dos núcleos em duas alíquotas de 900 μL, resultando em um volume total de 1,8 mL de suspensão de núcleos sendo usado durante a limpeza. - Remova a primeira alíquota de 900 μL da suspensão de núcleos do cartucho e adicione-a a uma alíquota de 500 μL SCS previamente preparada em um tubo de 2 mL. Misture bem pipetando até que a mistura fique homogênea.
- Remova a mistura de 1400 μL de suspensão de núcleos - SCS e coloque-a cuidadosamente em uma nova alíquota SCS de 500 μL, segurando o tubo em um ângulo e adicionando a mistura em gotas, criando uma separação de fase claramente visível (ver Figura 1A).
- Feche cuidadosamente o tubo e coloque-o novamente no gelo sem perturbar a separação de fases.
- Repetir os passos 3.1.2-3.1.4 com a segunda alíquota de suspensão de 900 μL e uma nova alíquota SCS para ter dois tubos de 2 ml por amostra com uma separação de fases claramente visível para centrifugação por gradiente.
- Adicione cuidadosamente os tubos a uma centrífuga pré-resfriada e gire a 13.000 x g por 15 min a 4 °C.
- Enquanto isso, prepare o NSR descrito na Tabela 4, adicionando o inibidor de RNase a uma alíquota de NSR. Preparar 1 mL de NSR por amostra.
NOTA: Neste ponto, as esferas de gel reagente de expressão gênica de célula única podem ser removidas do freezer de -80 °C, permitindo que elas se equilibrem à temperatura ambiente (RT), e o oligo de interruptor de molde pode ser ressuspendido em buffer TE baixo. - Após a centrifugação, remova o sobrenadante de ambos os tubos sem perturbar o pellet e ressuspenda cuidadosamente o pellet em 50 μL de NSR gelado, de acordo com a recomendação do fabricante. Agrupar os dois pellets da mesma amostra em um novo tubo de 1,5 mL e adicionar 900 μL de NSR gelado a um volume total de 1 mL. Misture bem por pipetagem.
- Centrifugar a amostra a 500 x g durante 5 min a 4 °C com uma centrífuga de rotor de balde oscilante.
NOTA: É altamente recomendável usar um rotor de balde oscilante para minimizar a perda de núcleos, especialmente quando se espera que o rendimento dos núcleos seja baixo ou quando se inicia com pequenas quantidades de tecido. - Enquanto isso, preparar 500 μL por amostra de 1x PBS (sem Ca2+ e Mg2+) com 0,04% de albumina de soro bovino (BSA) e 0,2 U/μL inibidor de RNase, conforme descrito na Tabela 5.
- Remover cuidadosamente o sobrenadante sem descartar o pellet e ressuspendê-lo em 100 μL de solução de PBS conforme preparado acima (1x PBS + 0,04% BSA + inibidor de RNase a 0,2 U/μL).
NOTA: Para amostras de tecido pequenas, a concentração de núcleos pode ser baixa e, portanto, recomenda-se ressuspender o pellet em apenas 50 μL de solução de PBS para garantir concentrações suficientemente altas para o sequenciamento de RNA de núcleo único. - Prossiga diretamente para a etapa 4.
- Perfure cuidadosamente a folha redonda no cartucho do dissociador com uma ponta de pipeta.
- Limpeza do gradiente coloide de sílica
NOTA: Para o tecido cerebral, um gradiente de sílica coloide é mais adequado do que um gradiente de sacarose para remover mielina e detritos da suspensão dos núcleos. Todas as etapas de limpeza são realizadas no gelo para minimizar a degradação do RNA. Os tampões e tubos, bem como as centrífugas, precisam ser pré-resfriados. Todas as etapas de ressuspensão e mistura são realizadas apenas por pipetagem cuidadosa, pois o vórtice pode comprometer a qualidade e integridade dos núcleos.- Perfure cuidadosamente a folha redonda no cartucho do dissociador com uma ponta de pipeta.
- Retire a suspensão de núcleos do cartucho e adicione-a a um tubo de 5 mL.
- Centrifugar a 500 x g durante 5 min a 4 °C numa centrífuga pré-arrefecida.
- Remover cuidadosamente o sobrenadante sem perturbar o pellet e ressuspender o pellet em 1 mL de solução gelada de sílica coloide a 18%.
- Adicionar mais 2 mL de solução coloide de sílica a 18% para obter um volume total de 3 mL e misturar bem por pipetagem.
- Centrifugar a amostra a 700 x g durante 5 min a 4 °C num rotor de caçamba oscilante com o travão acionado.
- Enquanto isso, prepare o NSR descrito na Tabela 4 adicionando o inibidor de RNase a uma alíquota de NSR. Preparar 1 mL de NSR por amostra.
NOTA: Neste ponto, as esferas de gel reagente de expressão gênica de célula única podem ser removidas do freezer de -80 °C, permitindo que ele se equilibre para RT e o oligo do interruptor do molde pode ser ressuspendido em buffer TE baixo. - Retire cuidadosamente a amostra da centrífuga sem perturbar a camada de mielina que flutua no topo.
- Primeiro, remova a camada de mielina do topo e descarte; Em seguida, remova cuidadosamente todo o sobrenadante sem perturbar o pellet.
NOTA: A camada de mielina pode ser facilmente removida envolvendo um lenço estéril sem fiapos em torno de uma ponta de pipeta de 1 mL para aspirar a camada de mielina juntamente com 1-2 mL do sobrenadante. - Ressuspender o pellet em 1 mL de NSR gelado conforme recomendação do fabricante.
- Centrifugar a amostra a 500 x g durante 5 min a 4 °C numa centrífuga com rotor de balde oscilante.
NOTA: É altamente recomendável usar um rotor de balde oscilante para minimizar a perda de núcleos, especialmente quando se espera que o rendimento dos núcleos seja baixo ou quando se inicia com pequenas quantidades de tecido. - Enquanto isso, preparar 500 μL por amostra de 1x PBS (sem Ca2+ e Mg2+) com 0,04% de albumina de soro bovino (BSA) e 0,2 U/μL inibidor de RNase, conforme descrito na Tabela 5.
- Remover cuidadosamente o sobrenadante sem perturbar o pellet e ressuspender a amostra em 100 μL de solução de PBS conforme preparado acima (1x PBS + 0,04% BSA + inibidor de RNase a 0,2 U/μL).
NOTA: Para amostras de tecido pequenas, a concentração de núcleos pode ser baixa e, portanto, recomenda-se ressuspender o pellet em apenas 50 μL de solução de PBS para garantir concentrações suficientemente altas para o sequenciamento de RNA de núcleo único.
4. Contagem
- Para cada amostra a contar, diluir 10 μL de suspensão de núcleos em 20 μL de solução de PBS para obter uma diluição de 1:3.
- Para a contagem, adicionar 25 μL de solução corante de iodeto de propídio (PI) a um poço de mistura da placa de contagem de contadores fluorescentes. Adicionar 25 μL da suspensão de núcleos diluídos e misturar bem por pipetagem. Transfira a amostra corada de 50 μL do poço de mistura para o poço de carregamento.
- Carregue a placa de contagem no contador de células e inicie a contagem.
NOTA: A contagem de núcleos é retirada do canal fluorescente vermelho com um tempo de exposição de 700 ms. Este canal foi otimizado para obter uma contagem precisa de núcleos por comparação cruzada com contagem manual com uma câmara de Neubauer e coloração de azul de Trypan sob o microscópio. Em altas concentrações, os núcleos estão muito próximos, dificultando a separação entre os softwares. Neste caso, recomenda-se a recontagem da amostra numa diluição adequada. A integridade dos núcleos, bem como a limpeza, pode ser avaliada a partir da imagem de campo brilhante ou sob um microscópio. - Diluir as amostras com PBS (1x PBS + 0,04% BSA + inibidor de RNase a 0,2 U/μL) até a concentração desejada para sequenciamento de RNA de núcleo único.
NOTA: Concentrações entre 700-1.200 núcleos/μL são consideradas ideais para o sequenciamento de RNA de núcleo único. Concentrações celulares mais baixas, como 700 núcleos/μL, podem resultar em contaminação de fundo reduzida do RNA ambiente.
5. Preparação da biblioteca
- Realizar o sequenciamento de RNA de núcleo único com os reagentes de expressão gênica de célula única usando o protocolo do fabricante visando uma recuperação de núcleos de 8000-10.000 núcleos por amostra.
6. Sequenciamento
- Sequencie as bibliotecas com a profundidade de sequenciamento desejada com indexação dupla emparelhada, e as seguintes leituras de sequenciamento: Leia 1: 28 ciclos, i7 Índice: 10 ciclos, i5 Índice: 10 ciclos e Leia 2: 90 ciclos.
Resultados Representativos
O desempenho e a versatilidade deste protocolo são demonstrados pela realização de sequenciamento de RNA de núcleo único em tecido fresco congelado do córtex occipital cerebral de três camundongos B6, tecido renal fresco congelado transversalmente cortado de três ratos Wistar, fígado de arquivo (11 anos de idade) e tecido esplênico de três macacos Cynomolgus Maurícios. Todos os animais não foram perfundidos.
Como mostrado nas Figuras 1B,C, foram obtidos núcleos de boa qualidade e livres de sinais de blebbing, debris e aglomeração. A filtração baseada em gradiente de sacarose foi otimizada para remover a maioria dos detritos, testando diferentes densidades, velocidades de rotação e tempos, e avaliando a pureza/integridade nuclear em um microscópio, bem como avaliando a distribuição do tamanho dos núcleos e o rendimento (Figura 1D). Isso nos permitiu escolher uma densidade de gradiente de sacarose de 1,5 M e usar um curto tempo de rotação de 15 min. Em seguida, para avaliar melhor a qualidade dos núcleos, os dados foram pré-processados usando 10X Cell Ranger, e uma análise posterior dos dados a jusante foi realizada usando Besca23. Núcleos com >5% de conteúdo mitocondrial (pois estes tendem a ser núcleos danificados/estressados) foram filtrados, e núcleos com 500-7.000 genes (para minimizar gotículas e multipletos vazios) foram retidos. Foram incluídos apenas genes presentes em pelo menos 30 núcleos. Visamos 8.000 núcleos por amostra de córtex cerebral e 10.000 núcleos por amostra de rim, fígado e baço. Após filtração, 10.644 núcleos de alta qualidade das três amostras de cérebro, 14.960 núcleos de alta qualidade das três amostras de rim, 18.795 núcleos de alta qualidade das três amostras de fígado e 13.882 núcleos de alta qualidade das três amostras de baço foram obtidos. A Figura 2A,D,G,J mostra gráficos de violino representando a distribuição das contagens de UMI, contagens gênicas e conteúdo mitocondrial em cada amostra. O número mediano de contagens em todas as amostras de cérebro foi de 7.563 UMI/núcleo e 3.208 genes/núcleo. O número mediano de contagens em todas as amostras de rim foi de 3.841 UMI/núcleo e 1.915 genes/núcleo. O número mediano de contagens em todas as amostras de fígado foi de 2.649 UMI/núcleos e 1.676 genes/núcleos. O número mediano de contagens em todas as amostras de baço foi de 1.609 UMI/núcleos e 1.138 genes/núcleos. Em seguida, geramos clusters usando genes altamente variáveis e os anotamos usando genes marcadores conhecidos 17,24,25,26. Como visto na Figura 2B,E,H,K, conseguimos identificar os tipos celulares esperados de cada tecido. Além disso, como visto na Figura 2B,E,H,K, todos os animais contribuíram para todos os agrupamentos, indicando baixa variabilidade técnica geral introduzida pelo protocolo. Além disso, as proporções celulares foram comparáveis nas três amostras por tipo de tecido, assim como a UMI e a contagem gênica (Figura 2A,C,D,F,G,I,J,L). Uma exceção notável é o fígado, onde as populações de hepatócitos entre as três amostras de fígado foram diferentes em proporções e perfil. Isto é provavelmente devido a diferenças biológicas entre os animais (sexo, idade, estado metabólico).
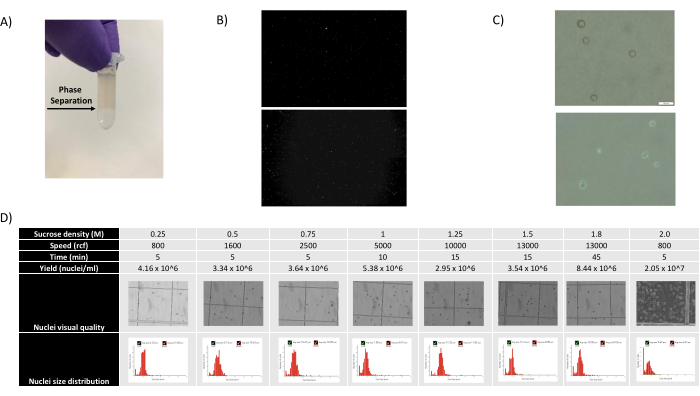
Figura 1: Avaliação da qualidade dos núcleos e otimização do gradiente de sacarose . (A) A separação de fase esperada durante a centrifugação do gradiente de sacarose é mostrada com uma seta. (B) Imagens fluorescentes representativas dos núcleos do rim de rato corados com iodeto de propídio (parte superior) e do baço de cynomolgus (abaixo) obtidas com o protocolo. (C) Imagens representativas de microscopia de campo claro de núcleos isolados de fígado de camundongo (superior) e cérebro de camundongo (inferior), barra de escala de 500 μm. Observe a superfície lisa regular dos núcleos indicando boa qualidade nuclear. (D) Otimização do gradiente de sacarose. Várias densidades de sacarose, velocidades de rotação e tempos de rotação foram testados. Imagens de microscopia de campo claro dos núcleos, distribuição do tamanho dos núcleos e rendimento dos núcleos são mostradas para cada condição. Clique aqui para ver uma versão maior desta figura.
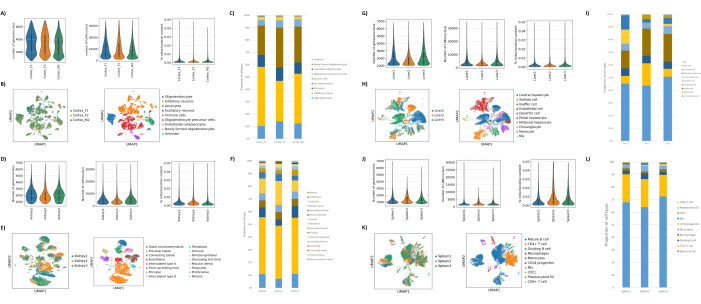
Figura 2: Dados representativos do snRNAseq no córtex occipital do cérebro de camundongos, rim de rato (córtex e medula) e fígado e baço de macaco cynomolgus. (A) Gráficos de violino mostrando a distribuição de genes/núcleo, UMIs/núcleo e porcentagem de conteúdo mitocondrial por amostra de cérebro. (B) Painel esquerdo: gráfico UMAP mostrando a contribuição de cada amostra para os conglomerados identificados no cérebro. Painel direito: UMAP mostrando as identidades dos agrupamentos anotados com base em genes marcadores no tecido cerebral. (C) Proporções celulares observadas nas 3 amostras de cérebro. (D) Gráficos de violino mostrando a distribuição de genes/núcleo, UMIs/núcleo e porcentagem de conteúdo mitocondrial por amostra de rim. (E) Painel esquerdo: gráfico UMAP mostrando a contribuição de cada amostra para os conglomerados identificados no rim. Painel direito: UMAP mostrando as identidades dos agrupamentos anotados com base em genes marcadores no tecido renal. (F) Proporções celulares observadas nas 3 amostras de rim. (G) Gráficos de violino mostrando a distribuição de genes/núcleo, UMIs/núcleo e porcentagem de conteúdo mitocondrial por amostra de fígado. (H) Painel esquerdo: gráfico UMAP mostrando a contribuição de cada amostra para os conglomerados identificados no fígado. Painel direito: UMAP mostrando as identidades dos agrupamentos anotados com base em genes marcadores no tecido hepático. (I) Proporções celulares observadas nas 3 amostras de fígado. (J) Gráficos de violino mostrando a distribuição de genes/núcleo, UMIs/núcleo e porcentagem de conteúdo mitocondrial por amostra de baço. (K) Painel esquerdo: gráfico UMAP mostrando a contribuição de cada amostra para os conglomerados identificados no baço. Painel direito: UMAP mostrando as identidades dos clusters anotados com base em genes marcadores no tecido do baço. (L) Proporções celulares observadas nas 3 amostras de baço. Clique aqui para ver uma versão maior desta figura.
| Componentes | Concentração de estoques | Volume por amostra | Concentração Final |
| Solução de almofada de sacarose | 2 milh | 1500 μL | 1.5 milh |
| Tampão de almofada de sacarose | - | 500 μL | - |
| Ditiotreitol (TDT) | 1 milh | 2 μL | 1 mM |
| Inibidor de RNAse | 40 U/μL | 10 μL | 0,2 U/μL |
Tabela 1: Preparação de solução de almofada de sacarose 1,5 M (SCS). Esta solução é utilizada para a centrifugação do gradiente de sacarose durante a limpeza na etapa 3.1 e deve ser preparada na hora de cada vez antes de iniciar o protocolo. Mantenha sempre o SCS no gelo durante o protocolo. As soluções mencionadas nesta tabela são referenciadas na Tabela de Materiais.
| Componentes | Concentração de estoques | Volume por amostra | Concentração Final |
| Solução-mãe de coloide de sílica | 90% | 600 μL | 18% |
| Reagente de Armazenamento de Núcleos (Genômica S2) | - | 2400 μL | - |
| Inibidor de RNAse | 40 U/μL | 15 μL | 0,2 U/μL |
Tabela 2: Preparação da solução coloide de sílica a 18%. Esta solução é utilizada para a centrifugação do gradiente coloide de sílica durante a limpeza na etapa 3.2 e deve ser preparada na hora de cada vez antes de iniciar o protocolo. Manter sempre a solução coloide de sílica a 18% congelada durante o protocolo.
| Tecido | Peso da amostra | Cartucho | Rendimento |
| Fígado de rato | magnésio 25 | Cartucho de isolamento de núcleos | 65.000 núcleos por mg de tecido |
| Fígado de rato | magnésio 4 | Cartucho de isolamento de núcleos de entrada pequeno | 32.000 núcleos por mg de tecido |
Tabela 3: Rendimento de núcleos do cartucho de isolamento de núcleos de baixa entrada versus o cartucho de isolamento de núcleos após a limpeza do gradiente de sacarose.
| Componentes | Concentração de estoques | Volume por amostra | Concentração Final |
| Reagente de Armazenamento de Núcleos | - | 1000 μL | - |
| Inibidor de RNAse | 40 U/μL | 5 μL | 0,2 U/μL |
Tabela 4: Preparação do reagente de armazenamento de núcleos (NSR). Esta solução é utilizada durante o isolamento de núcleos nos passos 3-5, bem como durante a limpeza no passo 3.1.8. Pode ser armazenado a 4 °C por até 4 meses. Prepare uma alíquota fresca com inibidor de RNase durante a etapa de centrifugação na etapa de limpeza 6. As soluções mencionadas nesta tabela são referenciadas na Tabela de Materiais.
| 1x PBS + 0,04% BSA solução de estoque | |||
| Componentes | Concentração de Estoques | Volume para estoque | Concentração Final |
| PBS (sem Ca 2+, sem Mg2+) | 1x | 30 mL | - |
| Albumina de Soro Bovino (BSA) | 30% | 40 μL | 0.04% |
| 1x PBS + 0,04% BSA + 0,2 U/μL inibidor de RNAse | |||
| Componentes | Concentração de Estoques | Volume por amostra | Concentração Final |
| 1x PBS + 0,04% BSA Solução de Estoque | - | 500 μL | - |
| Inibidor de RNAse | 40 U/μL | 2,5 μL | 0,2 U/μL |
Tabela 5: Preparo do PBS + BSA 0,04%. Esta solução é utilizada no final da limpeza na etapa 3.1.10 e após contagem para diluir a suspensão dos núcleos até a concentração desejada para a sequenciação de RNA de núcleo único 10X (etapa de contagem 4.4). A solução-mãe pode ser armazenada a 4 °C por até 1 mês. Prepare uma alíquota fresca com inibidor de RNase durante a etapa de centrifugação na etapa de limpeza 6.
Discussão
Desenvolvemos um protocolo versátil e parcialmente automatizado para obter núcleos únicos de alta qualidade a partir de tecidos congelados de mamíferos e demonstramos o protocolo em cérebro de camundongo, rim de rato e tecido hepático e baço de cynomolgus.
Ao compararmos o desempenho desse protocolo com o de outros protocolos publicados para sequenciamento de RNA de núcleo único em tecido cerebral, renal, baço e hepático 6,7,20,24,25,26, observamos que conseguimos detectar um número semelhante de genes e contagens de UMI por núcleo e somos capazes de recuperar os tipos celulares esperados. Comparado aos métodos existentes, existem várias vantagens para este protocolo. Primeiro, o protocolo deste estudo automatiza a homogeneização tecidual e o isolamento de núcleos únicos. Isso é conseguido com o uso de um disruptor de tecido robótico21. Na maioria dos protocolos, o tecido é homogeneizado com homogeneizador Dounce para liberar núcleos únicos 3,20. Entretanto, observamos que essa etapa manual pode levar a uma variabilidade experimental no rendimento e integridade dos núcleos dependendo da quantidade de força exercida durante a homogeneização, comprometendo a reprodutibilidade dos experimentos. Aqui, utilizando-se um moedor de tecidos automatizado com ajustes fixos, obteve-se boa qualidade dos núcleos e rendimento com maior consistência ao longo dos experimentos. Além disso, automatizar essa etapa também reduz o tempo prático do protocolo (a etapa de ruptura do tecido leva aproximadamente 7 min), permitindo que o usuário se prepare para as etapas subsequentes. Em segundo lugar, o protocolo descrito neste estudo é versátil, ou seja, é compatível com diferentes tecidos de diferentes espécies. Isso nos permite evitar a otimização de protocolos demorados, por exemplo, para identificar tampões/detergentes de homogeneização para diferentes tecidos 2,5,6. Em terceiro lugar, esse protocolo não depende do acesso a um classificador de fluxo para obter núcleos limpos, tornando-o mais acessível para laboratórios que não possuem os equipamentos/conhecimentos necessários para a triagem de fluxo. Em vez disso, otimizamos a filtração baseada em gradiente de sacarose para remover a maioria dos detritos. No entanto, para o tecido cerebral em particular, o uso de um gradiente coloide de sílica em vez de um gradiente de sacarose é recomendado para uma remoção mais eficiente da mielina. Verificamos também que o uso de um rotor de caçamba oscilante no final da etapa de centrifugação do gradiente sacarose/sílica coloide minimiza a perda de núcleos. Por isso, o uso de tal rotor é altamente recomendado. Quarto, após testar múltiplos métodos de contagem de núcleos (contagem manual ao microscópio, uso de vários contadores automatizados), recomenda-se o uso de um contador automático de células fluorescentes22. O uso de um corante intercalante de DNA, como o iodeto de propídio, aumenta a precisão da contagem de núcleos. Em quinto lugar, este protocolo leva cerca de 75 minutos desde o início até o carregamento do chip microfluídico. Isso ajuda a garantir que a integridade dos núcleos permaneça alta ao processar várias amostras. Finalmente, descobrimos que o protocolo também é compatível com o tecido composto de temperatura de corte ideal (OCT). Se usar esse material, o tecido pode ser removido do bloco OCT usando um bisturi antes da homogeneização.
Um desafio frequente em conjuntos de dados de sequenciamento de RNA de núcleo único é a presença de RNA ambiente, que pode ser não nuclear (por exemplo, mitocondrial), bem como derivado nuclear27,28. Em nosso protocolo, o RNA mitocondrial (um proxy para o RNA ambiente não nuclear) é baixo mesmo antes da filtragem (0,1-1,6% para os tecidos mostrados). No entanto, à semelhança de outros protocolos e conjuntos de dados, a contaminação por RNA ambiente a partir de genes altamente expressos nos núcleos de tipos celulares abundantes (como hepatócitos no fígado, neurônios no cérebro, etc.) ainda está presente27. Existem várias ferramentas de bioinformática, como CellBender, SoupX, etc., que podem remover essa contaminação do RNA ambiente antes da anotação dosnúcleos 29,30,31. Outra limitação desse protocolo é que, embora as etapas de ruptura tecidual e isolamento de núcleos sejam automatizadas, o rendimento dessa etapa ainda é limitado, pois apenas uma amostra pode ser processada por vez. No entanto, como essa etapa leva apenas aproximadamente 7 min por pedaço de tecido, várias amostras ainda podem ser processadas em um lote. Normalmente, processamos quatro amostras por lote, mas fizemos até seis amostras por lote com bons resultados. Melhorias recentes no dissociador robótico para permitir o processamento paralelo de duas amostras simultaneamente permitirão o processamento de 8-12 amostras por lote, o que é compatível com o rendimento do chip microfluídico que é usado para encapsulamento de núcleos únicos.
Embora não tenhamos utilizado os núcleos isolados por este protocolo para outras aplicações a jusante como ATAC-seq ou snRNAseq utilizando outras plataformas, com base na qualidade dos dados obtidos com os reagentes de expressão gênica aqui utilizados, acreditamos que nosso protocolo deva ser compatível com aplicações adicionais a jusante. No entanto, trabalhos futuros envolverão o teste desse protocolo com outras aplicações downstream, como ATAC-seq.
Em conclusão, desenvolvemos um protocolo de isolamento de núcleos rápido, simples e parcialmente automatizado para sequenciamento de RNA de núcleo único a jusante que demonstrou ser compatível com diferentes tipos de tecidos congelados de mamíferos.
Divulgações
Todos os autores são/foram funcionários da F. Hoffmann-La Roche durante a realização do estudo.
Agradecimentos
Os autores gostariam de agradecer a Filip Bochner, Marion Richardson, Petra Staeuble e Matthias Selhausen pelo fornecimento dos tecidos animais que foram analisados neste manuscrito. Também gostaríamos de agradecer a Petra Schwalie, Klas Hatje, Roland Schmucki e Martin Ebeling por seu apoio em bioinformática.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 M DTT | Thermo Fisher Scientific | P2325 | |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 10x Magnetic Separator | 10x genomics | PN-120250 | |
| 10x Vortex Adapter | 10x genomics | PN-120251 | |
| 1x DPBS (10x), no calcium, no magnesium | Thermo Fisher Scientific | 14190144 | stored at 4°C |
| 30% Bovine Serum Albumin | Sigma-Aldrich | A9576_50ML | |
| 400 mM Tris-HCl, pH 8.0 | Thermo Fisher Scientific | 15568025 | |
| 40U/μl RNaseOUT Recombinant Ribonuclease Inhibitor | Thermo Fisher Scientific | 10777019 | Stored at -20 °C |
| Agilent High Sensitivity DNA Kit | Agilent | 5067-4626 | |
| Cellaca MX High-throughput Automated Cell Counter | Nexcelom Bioscience | CELMXSYSF2 | Automated fluorescent cell counter |
| Chromium Next GEM Chip G Single Cell Kit, 16 rxns | 10x genomics | PN-1000127 | Single cell gene expression reagent, stored at room temperature |
| Chromium Next GEM Secondary Holder | 10x genomics | PN-1000195 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1, 4 rxns | 10x genomics | PN-1000129 | Single cell gene expression reagent, stored at -80 °C |
| Chromium Next GEM Single Cell 3' GEM, Library & Gel Bead Kit v3.1, 4 rxns | 10x genomics | PN-1000128 | Single cell gene expression reagent |
| Chromium Next GEM Single Cell 3' Library Kit v3.1 4 rxns | 10x genomics | PN-1000158 | Single cell gene expression reagent, stored at -20 °C |
| Chromium Next GEM Single Cell 3'GEM Kit v3.1 4 rxns | 10x genomics | PN-1000130 | Single cell gene expression reagent, stored at -20 °C |
| Divided Polystyrene Reservoirs | VWR | 41428-958 | |
| DNA LoBind Tubes 1.5ml Eppendorf | Sigma-Aldrich | EP0030108051 | |
| DNA LoBind Tubes 2ml Eppendorf | Sigma-Aldrich | EP0030108078 | |
| Dry ice | - | - | |
| Dynabeads MyOne SILANE | 10x genomics | PN-2000048 | Single cell gene expression reagent, stored at 4 °C |
| Ethanol Pure | Sigma-Aldrich | E7023 | |
| Glycerin (Glycerol), 50% (v/v) | Ricca Chemical Company | 3290-16 | |
| Heatblock | |||
| High-Throughput Nexcelom Counting Plates | Nexcelom Bioscience | CHM24-A100-001 | Cell counter counting plate |
| Low TE Buffer (10 mM Tris-HCl pH 8.0, 0.1 mM EDTA) | Thermo Fisher Scientific | 12090015 | |
| Mini Centrifuge | - | - | |
| NovaSeq 6000 SP Reagent Kit v1.5 (100 cycles) | Illumina | 2002840 | |
| Nuclei Isolation Buffer | S2 Genomics | 100-063-396 | Stored at 4 °C |
| Nuclei Isolation Cartridge | S2 Genomics | 100-063-287 | Precooled at 4 °C before use |
| Nuclei PURE 2 M Sucrose Cushion Solution | Sigma-Aldrich | NUC201-1KT | Sucrose cushion solution |
| Nuclei PURE Sucrose Cushion Buffer | Sigma-Aldrich | NUC201-1KT | |
| Nuclei Storage Reagent | S2 Genomics | 100-063-405 | Stored at 4 °C |
| PCR Tubes 0.2 ml 8-tube strips | Eppendorf | 30124359 | |
| Percoll | GE Healthcare | 17-0891-02 | Silica colloid solution |
| PhiX Control v3 | Illumina | FC-110-3001 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific | Q32854 | |
| Refrigerated Centrifuge (Eppendorf 5804R) | Eppendorf | 5805000010 | |
| Refrigerated Centrifuge with Swinging-Bucket Rotor (Eppendorf 5810R) | Eppendorf | 5811000015 | |
| RNAseZap | Ambion | AM9780 | RNAse decontamination solution |
| Round cell culture petri dish | SPL | 330005 | |
| Scalpel disposable | Aesculap AG | BA210 | pre-cooled on dry ice before use |
| Single Index Kit T Set A, 96 rxns | 10x genomics | PN-1000213 | Single cell gene expression reagent, stored at -20 °C |
| Singulator 100 System | S2 Genomics | - | Commercially available robotic tissue dissociator |
| Sodium Hydroxide 1M | Sigma-Aldrich | 72068 | |
| SPRIselect Reagent Kit | Beckman Coulter | b23318 | |
| Sterile tweezers | - | - | |
| UltraPure DNase/RNase-Free Distilled Water | Thermo Fisher Scientific | 10977049 | |
| ViaStain PI Staining Solution | Nexcelom Bioscience | CS1-0109-5mL | Propidium iodide staining solution |
| Vortex Mixer+A2:D44 | VWR | - |
Referências
- Burja, B., et al. An Optimized Tissue Dissociation Protocol for Single-Cell RNA Sequencing Analysis of Fresh and Cultured Human Skin Biopsies. Front Cell Dev Biol. 10, 872688 (2022).
- Kimbley, L. M., et al. Comparison of optimized methodologies for isolating nuclei from esophageal tissue. Biotechniques. 72 (3), 104-109 (2022).
- Maitra, M., et al. Extraction of nuclei from archived postmortem tissues for single-nucleus sequencing applications. Nature Protocols. 16 (6), 2788-2801 (2021).
- Nadelmann, E. R., et al. Isolation of nuclei from mammalian cells and tissues for single-nucleus molecular profiling. Current Protocols. 1 (5), e132 (2021).
- Rousselle, T. V., et al. An optimized protocol for single nuclei isolation from clinical biopsies for RNA-seq. Scientific Reports. 12, 9851 (2022).
- Slyper, M., et al. A single-cell and single-nucleus RNA-Seq toolbox for fresh and frozen human tumors. Nature Medicine. 26 (5), 792-802 (2020).
- Leiz, J., et al. Nuclei isolation from adult mouse kidney for single-nucleus RNA-sequencing. Journal of Visualized Experiments: JoVE. (175), 62901 (2021).
- Alvarez, M., et al. Isolation of nuclei from human snap-frozen liver tissue for single-nucleus RNA sequencing. Bio-Protocol. 13 (3), e4601 (2023).
- Ayhan, F., Douglas, C., Lega, B. C., Konopka, G. Nuclei isolation from surgically resected human hippocampus. STAR Protocols. 2 (4), 100844 (2021).
- Joshi, N., Misharin, A. Single-nucleus isolation from frozen human lung tissue for single-nucleus RNA-seq. Protocols.io. , (2019).
- Martelotto, L. G., Luciano Martelotto, L. 'Frankenstein' protocol for nuclei isolation from fresh and frozen tissue for snRNAseq. Protocols.io. , (2020).
- Masilionis, I., Chaudhary, O., Chaligne, R., Mazutis, L. Nuclei extraction for single-cell RNAseq from frozen tissue using Singulator™ 100. Protocols.io. , (2022).
- Matson, K. J. E., et al. Isolation of adult spinal cord nuclei for massively parallel single-nucleus RNA sequencing. Journal of Visualized Experiments: JoVE. (140), 58413 (2018).
- Mendelev, N., et al. Multi-omics profiling of single nuclei from frozen archived postmortem human pituitary tissue. STAR Protocols. 3 (2), 101446 (2022).
- Soule, T. G., et al. A protocol for single nucleus RNA-seq from frozen skeletal muscle. Life Science Alliance. 6 (5), e202201806 (2023).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648 (2018).
- Ding, J., et al. Systematic comparison of single-cell and single-nucleus RNA-sequencing methods. Nature Biotechnology. 38, 737-746 (2020).
- Hu, P., et al. Single-nucleus transcriptomic survey of cell diversity and functional maturation in postnatal mammalian hearts. Genes & Development. 32 (19-20), 1344-1357 (2018).
- Lake, B. B., et al. A comparative strategy for single-nucleus and single-cell transcriptomes confirms accuracy in predicted cell-type expression from nuclear RNA. Scientific Reports. 7, 6031 (2017).
- Narayanan, A., et al. Nuclei Isolation from Fresh Frozen Brain Tumors for Single-Nucleus RNA-seq and ATAC-seq. Journal of Visualized Experiments: JoVE. (162), 61542 (2020).
- Jovanovich, S., et al. . Automated processing of solid tissues into single cells or nuclei for genomics and cell biology applications with the Singulator™ 100 and 200 systems. , (2022).
- Bell, J., et al. Characterization of a novel high-throughput, high-speed and high-precision plate-based image cytometric cell counting method. Cell & Gene Therapy Insights. 7 (4), 427-447 (2021).
- Madler, S. C., et al. Besca, a single-cell transcriptomics analysis toolkit to accelerate translational research. NAR Genomics and Bioinformatics. 3 (4), lqab102 (2021).
- Wu, H., et al. Mapping the single-cell transcriptomic response of murine diabetic kidney disease to therapies. Cell Metabolism. 34 (7), 1064-1078 (2022).
- Han, L., et al. Cell transcriptomic atlas of the non-human primate Macaca fascicularis. Nature. 604 (7907), 723-731 (2022).
- Madissoon, E., et al. scRNA-seq assessment of the human lung, spleen, and esophagus tissue stability after cold preservation. Genome Biology. 21 (1), 1 (2019).
- Caglayan, E., Liu, Y., Konopka, G. Neuronal ambient RNA contamination causes misinterpreted and masked cell types in brain single-nuclei datasets. Neuron. 110 (24), 4043-4056 (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Molecular Systems Biology. 15 (6), e8746 (2019).
- Fleming, S. J., et al. Unsupervised removal of systematic background noise from droplet-based single-cell experiments using CellBender. bioRxiv. , (2022).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biology. 21 (1), 57 (2020).
- Young, M. D., Behjati, S. SoupX removes ambient RNA contamination from droplet-based single-cell RNA sequencing data. Gigascience. 9 (12), giaa151 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados