Imagem de microdomínios iniciais de Ca2+ em células T primárias
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, fornecemos um protocolo abrangente para resolver sinais locais iniciais de Ca2+ , conhecidos como microdomínios de Ca2+ , em células T murinas e humanas primárias usando microscopia de fluorescência. Este protocolo serve como um recurso valioso para pesquisadores que examinam as vias de sinalização de Ca2+ dentro das células imunes e para desvendar ainda mais sua função.
Resumo
Sinais locais de Ca2+ em subsegundos, denominados microdomínios de Ca2+ , são sinais de Ca2+ altamente dinâmicos e de curta duração, que resultam em uma elevação global de [Ca2+]i e já podem determinar o destino de uma célula T. Após a ativação do receptor de células T, o NAADP é formado rapidamente, ligando-se às proteínas de ligação ao NAADP (HN1L / JPT2, LSM12) e seus respectivos receptores (RyR1, TPC2) localizados nos estoques intracelulares de Ca2+ , como o ER e os lisossomos, e levando à liberação e elevação subsequentes de [Ca2+]i. Para capturar esses sinais de Ca2+ rápidos e dinâmicos, desenvolvemos uma técnica de imagem de alta resolução usando uma combinação de dois indicadores de Ca2+ , Fluo-4 AM e FuraRed AM. Para o pós-processamento, uma abordagem de detecção de microdomínio Ca2+ semiautomatizada e de código aberto foi desenvolvida com base na linguagem de programação Python. Usando esse fluxo de trabalho, somos capazes de detectar de forma confiável microdomínios Ca2+ em nível subcelular em células T murinas e humanas primárias em vídeos de fluorescência de alta resolução temporal e espacial. Este método também pode ser aplicado a outros tipos de células, como células NK e linhagens celulares neuronais murinas.
Introdução
A técnica de microscopia de fluorescência apresentada permite a visualização de sinais iniciais locais e temporais de cálcio (Ca2+) em células T primárias de camundongos, denominadas microdomínios de Ca2+ . Os microdomínios de Ca2+ representam eventos de sinalização de Ca2+ altamente dinâmicos e de curta duração, apresentando desafios para imagens e análises eficazes de células vivas1.
As células T são desafiadoras para imagens de células vivas devido às diferenças relativas na intensidade de fluorescência central e periférica, que podem ser atribuídas à sua forma esférica e seu pequeno diâmetro de ~ 6-8 μm. Após a estimulação e formação de sinapses imunes, as células T sofrem alterações morfológicas, complicando ainda mais a imagem das células T1. Portanto, empregar uma análise raciométrica torna-se imperativo, obtido registrando duas imagens representando propriedades diferentes de um corante Ca2+ ou utilizando uma combinação de dois corantes Ca2+ . As características exigentes dos microdomínios de Ca2+ incluem sua natureza rápida, temporal e espacialmente limitada. Para capturar isso, os corantes de Ca2+ usados devem possuir um alto brilho basal e uma alta relação sinal-ruído (SNR) para obter a maior resolução temporal e espacial possível. Os melhores resultados foram alcançados usando uma combinação do corante de comprimento de onda duplo Fura Red e do corante de comprimento de onda único Fluo-4. O co-carregamento de células com Fluo-4 e Fura Red mitiga os desafios impostos pelo forte fotobranqueamento de corantes de comprimento de onda de dupla emissão e o atraso temporal associado aos corantes de dupla excitação, garantindo adequação para aquisição rápida de imagens. Essa abordagem facilita ainda mais a visualização de mudanças na forma e movimentos sutis. Exigências especiais também são colocadas no sistema de imagem em termos de resolução espacial para permitir a visualização de sinais de Ca2+ provenientes da abertura de pequenos aglomerados de canais ou mesmo canais únicos1.
A sinalização de Ca2+ desempenha um papel fundamental na ativação de funções imunes dentro das células T, incluindo a formação de sinapses e a produção e liberação de citocinas 2,3. O destino específico da célula é regulado através dos sinais de Ca2+ pronunciados de forma diferente e distribuídos localmente, os microdomínios de Ca2+ 3. Notavelmente, esses sinais locais de Ca2+ precedem um aumento generalizado nos níveis intracelulares de Ca2+ nas células T e a formação de microdomínios de Ca2+ depende da entradae liberação de Ca2+ 1,4,5. Após a estimulação do receptor de células T (TCR)/CD3, a formação de segundos mensageiros liberadores de Ca2+, como o ácido nicotínico adenina dinucleotídeo fosfato (NAADP), D-mio-inositol 1,4,5-trifosfato (IP3) e ADP ribose cíclica (cADPR) é desencadeada, levando ao aumento dos níveis intracelulares de Ca2+ até 1 μM 6,7. Os eventos iniciais de sinalização de Ca2+ estão ligados à liberação de Ca2+ dos estoques intracelulares de Ca2+, como o retículo endoplasmático (RE), sendo canais como o receptor de rianodina 1 (RyR1) e o receptor IP3 (IP3R) predominantemente responsáveis por essa sinalização. Posteriormente, isso desencadeia o influxo extracelular de Ca2+ e resulta em um sinal global de Ca2+ por meio da entrada de Ca2+ operada por armazenamento (SOCE)8. Além disso, existem outros canais envolvidos na sinalização de Ca2+ durante a ativação das células T9, por exemplo, os canais P2X4 e P2X7 garantem o influxo de cátions dependente de adenosina trifosfato (ATP), contribuindo para o aumento do Ca2+ intracelular. Notavelmente, os microdomínios iniciais de Ca2+ dependentes de adesão (ADCMs) já são formados antes da estimulação do TCR, mas com amplitudes e frequências mais baixas de Ca2+. Esses sinais iniciais de Ca2+ independentes de TCR provavelmente servem para a migração da célula T para o local da inflamação e preparam as células T para reestimulação no local da infecção10,11.
Ao desenvolver o método descrito para imagens locais de Ca2+ , ganhamos uma ferramenta adicional para explorar a origem e o significado dos primeiros sinais de Ca2+ na ativação de células T. Este método permite que o usuário detecte sinais de Ca2+ menores, de curta duração e de ocorrência mais rápida do que era possível anteriormente. Além disso, o DARTS (Deconvolution, Analysis, Registration, Tracking, and Shape Normalization), o pipeline de análise baseado em Python, permite compartilhar as ferramentas de análise com um público mais amplo12.
Protocolo
Todos os experimentos com animais foram aprovados e realizados de acordo com as diretrizes de bem-estar animal do Comitê Institucional de Cuidados e Uso de Animais do Centro Médico Universitário de Hamburgo-Eppendorf.
1. Isolamento de células T primárias de camundongos de linfonodos e baços
- Colha os baços/gânglios linfáticos em condições estéreis de acordo com as diretrizes éticas e coloque-os em um tubo com meio Clicks gelado (10% de soro fetal de bezerro (FCS), 2 mM de L-glutamina, penicilina/estreptomicina (P/S) 100 U/mL, 50 nM β-mercaptoetanol).
NOTA: As células T do tipo selvagem e do camundongo KO são isoladas da mesma maneira. - Arrefecer a centrífuga a 4 °C.
- Colocar o baço e os gânglios linfáticos num coador de células de 70 μm numa placa de Petri estéril e adicionar meio de isolamento do baço (meio RPMI + 7,5 % NCS + 1 % P/S) até um volume total de 20 ml.
- Rompa o baço completamente, mas suavemente, usando uma argamassa plástica. Transfira as células depois para um tubo de centrífuga de 50 mL.
NOTA: Um volume total de 20 mL deve estar agora no tubo de centrífuga de 50 mL. O baço e os gânglios linfáticos devem ser completamente interrompidos. Trabalhe no gelo a partir deste ponto. Manter as células no gelo retarda seu metabolismo e evita a morte celular, garantindo que permaneçam viáveis e funcionalmente intactas. Isso ajuda a preservar seu estado fisiológico por um longo período de tempo, o que é especialmente importante para experimentos precisos a jusante. - Centrifugar a suspensão da célula a 300 x g durante 5 min a 4 °C.
- Descarte o sobrenadante e garanta a ressuspensão do pellet resultante em 2 mL de solução salina tamponada com fosfato de Dulbecco (DPBS) (sem Ca2+ e Mg2+). Transfira para um novo tubo de centrífuga de 12 mL.
2. Seleção negativa de células T CD4+
NOTA: Para a seleção negativa de células T CD4+ , é usado um kit de isolamento de células T contendo um bloqueador FcR, anticorpos biotinilados contra células T não CD4+ e partículas magnéticas revestidas com estreptavidina.
- Adicione 20 μL / mL de bloqueador FcR de camundongo do kit de isolamento de células T à suspensão celular.
- Além disso, adicione 100 μL de coquetéis de isolamento de células T CD4 + de camundongo a uma concentração de 50 μL / mL, misture bem e incube por 10 min em temperatura ambiente (RT).
- Partículas magnéticas revestidas com estreptavidina em vórtice por 30 s, adicione 150 μL à suspensão celular, misture bem e incube por 2 min 30 s em RT.
- Adicione DPBS à suspensão celular, preenchendo-a até um volume total de 7 mL. Certifique-se de pipetar suavemente para cima e para baixo e, em seguida, incubar no ímã por 2 min 30 s em RT.
- Em seguida, colete suavemente as células T enriquecidas no sobrenadante e transfira-as para um novo tubo de centrífuga de 12 mL.
- Centrifugue as células a 300 x g em RT por 5 min.
- Descarte o sobrenadante.
- Ressuspenda as células T CD4+ isoladas e enriquecidas em 1 mL de DPBS, pipetando completamente para cima e para baixo. Incube a mistura por 2 min 30 s no ímã em RT para garantir que não haja contaminação do grânulo.
- Colete o sobrenadante, transfira-o para um novo tubo de centrífuga e conte as células T enriquecidas.
- Para contar as células, core-as com azul de tripano e conte usando um contador de células automatizado.
NOTA: É melhor carregar diretamente as células T CD4+ isoladas e obter imagens delas no mesmo dia do isolamento. No entanto, as células T murinas primárias podem ser cultivadas sem qualquer estimulação durante a noite em uma incubadora a 37 ° C com 5% de CO2 em um meio de isolamento (RPMI 1640 + 10% FCS + P / S).
3. Carregamento de células T CD4+ primárias de camundongo
NOTA: Para medir as concentrações citosólicas livres de Ca2+ , corantes fluorescentes e permeáveis à membrana são empregados nos experimentos de imagem de Ca2+ . Uma combinação de éster Fluo-4-acetoximetílico (AM) e o corante raciométrico Fura Red-AM serve como um indicador para a detecção rápida de sinais locais de Ca2+ . Certifique-se de trabalhar no escuro enquanto usa corantes fluorescentes.
- Centrifugue 2-5 x 106 células por 5 min, 300 x g, descarte o sobrenadante e ressuspenda em 480 μL de meio de células T (RPMI 1640, 10% FCS) com 10 μM Fluo-4-AM (Estoque: [1 mM]) e 20 μM Fura Red-AM (Estoque: [4 mM]).
- Incube inicialmente por 20 min em RT sob a bancada no escuro, cobrindo o falcão com papel alumínio.
NOTA: Não exceda o primeiro tempo de incubação, pois os indicadores de Ca2+ são diluídos em DMSO, o que pode ser prejudicial às células em um tempo de exposição mais longo no baixo volume total. Mantenha as células protegidas da luz após o carregamento devido à sensibilidade à luz dos corantes. - No final da incubação inicial de 20 minutos, adicione 2 mL de meio de células T às células e continue a incubação por mais 30 minutos no escuro em RT.
- Centrifugue a suspensão da célula a 300 x g por 5 min em RT.
- Descarte o sobrenadante e limpe o pellet lavando-o com 2 mL de tampão de medição de Ca2+ (140 mM NaCl, 5 mM KCl, 1 mM MgSO4, 1 mM CaCl2, 20 mM HEPES, 1 mM NaH2PO4, 5 mM de glicose, pH 7,4) seguido de centrifugação a 300 x g por 5 min em RT.
- Por fim, rejeitar o sobrenadante e ressuspendê-lo com o tampão de medição de Ca2+ . Ajuste o número da célula para ~ 100.000 células por 10 μL.
NOTA: Um volume de 10 μL é usado para uma medição. Evite diluir demais as células, pois pode ser necessário ajustar ainda mais a contagem de células ao microscópio para atingir um ideal para as condições de medição. - Deixe as células incubarem por ~ 20 min para garantir a infiltração celular completa e a desesterificação dos corantes. Armazene as células no gelo e no escuro até a medição.
NOTA: As células T murinas primárias carregadas podem ser usadas até ~ 4 h após o carregamento.
4. Imagem local de Ca2+
- Preparação de slides
- Revestir lamínulas de microscópio (24 mm x 46 mm) com albumina de soro bovino (BSA, 5 mg/mL) e poli-L-lisina (PLL, 0,1 mg/mL) (Figura 1A). Deixe o BSA descansar por ~ 20 min antes de aplicar o PLL.
NOTA: O revestimento facilita a adesão celular. Para uma adesão celular adequada, espalhe com precisão o BSA e o PLL até que não haja mais estrias visíveis. - Para criar câmaras de reação, cole anéis de vedação de borracha reutilizáveis nas lâminas usando uma pasta de silicone (Figura 1B, C).
NOTA: Outras lâminas/câmaras ou placas, como lâminas de 35 mm, câmaras de metal reutilizáveis ou placas de 8, 24 ou 48 poços, podem ser usadas para aquisição de imagens.
- Revestir lamínulas de microscópio (24 mm x 46 mm) com albumina de soro bovino (BSA, 5 mg/mL) e poli-L-lisina (PLL, 0,1 mg/mL) (Figura 1A). Deixe o BSA descansar por ~ 20 min antes de aplicar o PLL.
- Revestimento de esferas magnéticas de proteína G (10 μm de diâmetro) com anticorpos
- Misture suavemente a suspensão de esferas magnéticas de proteína e transfira 12,5 μL para um novo tubo.
- Coloque o tubo em um suporte magnético para remover o buffer de armazenamento e deixe as contas migrarem para o ímã. Remova cuidadosamente o buffer de armazenamento pipetando-o no lado oposto do ímã.
- Para remover qualquer tampão de armazenamento restante, lave os grânulos adicionando 500 μL de PBS-T (PBS, 0,1% Tween) e vórtice por 10 s. Coloque o tubo no suporte magnético novamente e remova o tampão.
- Para revestir as esferas com anticorpos, ressuspenda-as em 7,5 μL de PBS-T e adicione 5 μL de anti-CD3 (0,5 mg/mL) e anti-CD28 (0,5 mg/mL), respectivamente. Incubar por 30-60 min com mistura contínua em RT.
- Lave os grânulos revestidos três vezes com 500 μL de PBS-T, seguido de lavagem uma vez com 500 μL de tampão de medição de Ca2+ usando o suporte magnético.
- Ressuspenda os grânulos em 200-400 μL de tampão de medição de Ca2+.
NOTA: Verifique a densidade do cordão durante as medições, dilua ainda mais se necessário.
- Microscopia de imagem local de Ca2+
NOTA: A imagem é realizada usando um microscópio de luz de campo claro com ampliação de 100 vezes, equipado com uma lâmpada de arco de xenônio como fonte de luz. Os quadros são capturados no modo de 14 bits com binning duplo usando uma câmera acoplada de carga multiplicadora de elétrons. Para registrar e dividir os comprimentos de onda de emissão de ambos os corantes, um módulo de visão dupla é empregado, apresentando os seguintes filtros em nm (ex, 480/40; bs, 495; em1, 542/50; em2, 650/57). A configuração de imagem inclui um hub de aquisição e software de imagem para aquisição de imagem.- Coloque 10 μL das células carregadas na lâmina preparada e deixe-as presas à lâmina por 3-5 min.
- Adicione suavemente 80 μL de tampão de medição de Ca2+ à lâmina.
- Selecione a lente de imersão em óleo 100x e aplique uma pequena gota de óleo de imersão. Coloque a lâmina na mesa do microscópio.
- Ajuste o foco no modo de campo claro, selecione cuidadosamente um campo de visão com até 10 células que não estejam em contato umas com as outras e adquira uma imagem.
NOTA: Células sobrepostas e tocando serão difíceis de analisar posteriormente.- Ligue a lâmpada, verifique a fluorescência e o carregamento das células T e compare as células em ambos os canais para verificar se elas não estão pré-ativadas.
- Certifique-se de tirar fotos do campo de view para canais de campo claro e fluorescência antes e depois da medição para verificar o movimento e o carregamento da célula.
- Inicie a medição e capture a atividade basal por 1 min com uma taxa de aquisição de 1 quadro por 5 s.
- Adicione 10 μL de composto/estimulante (grânulo ou estimulador/inibidor) após 1 min e meça para um total de 3 min usando 40 quadros por s ou a taxa de quadros máxima possível.
NOTA: A adição de contas é a etapa crucial; Certifique-se de que as contas sejam adicionadas perto da luz de excitação na corrediça sem mover a corrediça. Exemplos representativos de microdomínios Ca2+ em células T CD4+ primárias após contato com esferas de camundongos WT e P2x4-/- e P2x7-/- são mostrados na Figura 2.
5. Pós-processamento/análise de dados
NOTA: Para processamento de imagem e análise de dados, o DARTS de pipeline de software livre baseado em Python é usado. Foi desenvolvido por Woelk et al.12 com base no trabalho de Diercks et al.13.
- Instalação do pipeline DARTS
- Instale o Python 3.10.014, o anaconda15 e o git16 e clone o repositório GitHub usando o comando de terminal git clone: https://github.com/IPMI-ICNS-UKE/DARTS.git
- Crie um ambiente conda com conda create --name DARTS e instale todos os pacotes Python necessários com pip install <package>.
NOTA: Siga as instruções de instalação no repositório DARTS GitHub17,18 para obter informações mais detalhadas. - Antes de usar Bioformats, certifique-se de que um Java Runtime Environment esteja instalado corretamente19.
- Uso do pipeline DARTS
NOTA: Uma vez instalado, o DARTS pode ser iniciado na janela do terminal.- Navegue até a cópia local do repositório DARTS (cd path/to/DARTS) que contém o arquivo main.py. Certifique-se de que o ambiente conda esteja ativado (conda ative DARTS).
- Execute o DARTS digitando python main.py na janela do terminal e pressionando Enter.
- Na interface gráfica do usuário (GUI), especifique o diretório de origem e resultados, bem como o formato da imagem na seção superior esquerda (consulte a Figura 3).
- Defina as propriedades da medição, como a escala (pixels por micrômetro), taxa de quadros, tipo de célula, etc. O pipeline de processamento pode ser montado com base nos dados de imagem específicos e na questão de pesquisa.
NOTA: Leia mais sobre os parâmetros de deconvolução, por exemplo, na documentação correspondente20. - Salve as configurações no computador antes de clicar em Iniciar. Uma descrição mais detalhada da GUI pode ser encontrada na documentação do DARTS.
- Imagem de microdomínio de Ca2+ e contatos de esferas
NOTA: Para imagens locais e estimulação com esferas e análise de imagem subsequente, os contatos de esferas específicos da célula precisam ser definidos pelo usuário para identificar o ponto de partida (t = 0) do período de medição de interesse (consulte a Figura 4). Um contato com o cordão é definido como o local de contato de um cordão (estimulante) com uma célula de interesse. Consiste na especificação de (1) o tempo de contato do cordão, por exemplo, quadro 300, e (2) a posição de contato do cordão em relação ao centro da célula, por exemplo, 1 hora ou 12 horas. Com a versão atual do DARTS, o usuário precisa definir e inserir manualmente as informações de cada contato do cordão. Use o procedimento a seguir para definir os contatos do cordão:- Use o controle deslizante para identificar o tempo de contato entre uma conta e uma célula de interesse.
- No menu de opções do lado direito, selecione o contato do cordão: x, y, t.
- Selecione o ponto no lado esquerdo da imagem onde a célula e a conta fazem contato.
- Selecione Escolher célula clicando em um ponto dentro. Clique na célula estimulada por esta conta, de preferência no centro.
- Clique em ADICIONAR contato do cordão.
- Repita as etapas 5.3.1 a 5.3.5 para cada contato adicional do cordão neste arquivo. Depois que todos os contatos do cordão estiverem definidos, clique no botão Continuar .
- Prossiga com arquivos adicionais. Depois que o último arquivo for alcançado, o script iniciará automaticamente a análise de todos os arquivos.
- Análise de dados
- Encontre as imagens de proporção processadas, dados de microdomínio para cada célula ao longo do tempo (incluindo localização, amplitude, tamanho), dados de origem para a projeção do alvo de dardos e outros recursos na pasta de resultados.
- Para criar alvos de dardos, navegue até a pasta /src/analysis/ dentro da pasta DARTS (padrão: cd path/to/DARTS/src/analysis) que contém o script DartboardPlotGUI.py. Em seguida, digite python DartboardPlotGUI.py e pressione Enter.
- Certifique-se de que as informações necessárias sejam fornecidas e que os arquivos de planilha da pasta de resultados sejam selecionados com precisão como os arquivos de origem para a geração do alvo de dardos.
Resultados Representativos
Neste protocolo, delineamos um método atualizado para obter imagens e analisar microdomínios iniciais de Ca2+ em células T primárias de camundongos com base em trabalhos anteriores de nosso grupo 1,13. Essa abordagem foi fundamental para desvendar o envolvimento de canais CRAC, como ORAI1, STIM1 e STIM2, bem como canais de liberação intracelular de Ca2+, como RyR1, nos primeiros eventos de sinalização de Ca2+ 4.
Para fazer isso, investigamos a formação espontânea do microdomínio Ca2+ por imagem de camundongos primários não estimulados Orai1-/-, Stim1-/-, Stim2-/- e Ryr1-/- e os comparamos com células T primárias de camundongo WT. A análise da formação do microdomínio de Ca2+ abrangeu a velocidade de início do sinal, a amplitude de Ca2+ e o número de sinais por plano confocal. Notavelmente, exceto para as células T Stim2-/-, todas as células T KO exibiram uma diminuição significativa nos sinais locais de Ca2+ e uma concentração reduzida de Ca 2+ citosólico livre basal em comparação com as células WT. Isso nos levou a concluir que a formação de microdomínios de Ca2+ está intrinsecamente ligada à interação de ORAI1, STIM1 e RyR14. Além disso, identificamos e caracterizamos com sucesso os microdomínios espontâneos de Ca2+ na membrana plasmática. Esses microdomínios de Ca2+ foram caracterizados por uma amplitude de Ca2+ de 290 nm ± 12 nm. A utilização de uma abordagem codificada por cores para sinais de Ca2+ permitiu a visualização de microdomínios de Ca2+ em toda a célula. Os resultados destacaram ainda o rápido início dos microdomínios de Ca2+, visíveis em milissegundos, e a capacidade desse método de detectar sinais de Ca2+ com uma longevidade de alguns milissegundos4. Esses microdomínios espontâneos de Ca2+ foram posteriormente identificados como microdomínios de Ca2+ dependentes de adesão (ADCM), dependentes não apenas de SOCE, mas também atuando por meio da cascata de sinalização FAK/PLC-γ/IP3 10 e envolvimento de P2X49. Além disso, esta técnica foi fundamental na confirmação das oxidases duplas 1 e 2 (DUOX1/2) como enzimas produtoras de NAADP21 e HN1L/JPT222 como uma das proteínas de ligação NAADP recém-descobertas23.
A Figura 2 mostra exemplos representativos de microdomínios de Ca2+ em células T CD4+ primárias após contato com o grânulo de WT, bem como de camundongos P2x4-/- e P2x7-/-. As células foram carregadas com os corantes Ca2+ Fluo-4 AM e Fura Red AM e fotografadas a uma taxa de aquisição de 25 ms (40 quadros / s). Para imitar a formação de sinapses de células T, as células foram estimuladas com esferas revestidas anti-CD3 / anti-CD28. A formação inicial do microdomínio Ca2+ foi analisada 1 s antes e até 15 s após o contato do cordão usando o pipeline DARTS. Após o contato com o grânulo, a célula WT mostrou rápida formação de microdomínios de Ca2+ no primeiro segundo após a estimulação no local de contato do grânulo (Figura 2A). Esses microdomínios de Ca2+ se expandiram ainda mais por toda a célula nos 15 s seguintes após o contato com o grânulo. Ao contrário da célula WT, as células P2x4-/- e P2x7-/- (Figura 2B) mostraram diminuição da formação do microdomínio Ca2+ após a estimulação do bead, bem como para o nível basal inferior de P2x4-/-a antes do contato com o bead. Esses achados representativos estão de acordo com os resultados publicados anteriormente por Brock et al.9, indicando a formação do microdomínio Ca2+ em células WT T diretamente após o contato com o bead por mais de 15 s e sinais mais baixos por quadro em células P2x4-/- e P2x7-/- . Além disso, a amplitude nas células P2x4-/-foi significativamente reduzida, estabelecendo ainda mais o papel da sinalização purinérgica nos microdomínios Ca2+ dependentes de adesão.
Além disso, este método também pode ser usado para visualizar microdomínios iniciais de Ca2+ em células T CD4+ humanas primárias (Figura 3). De acordo com as células T murinas primárias, os microdomínios iniciais de Ca2+ são evocados no local de contato do grânulo. No entanto, a resposta geral de Ca2+ parece ocorrer em uma escala de tempo diferente em comparação com as células T CD4+ murinas.
A análise dos sinais locais de Ca2+ de maneira manual não é viável, pois é bastante trabalhosa e subjetiva para o investigador individual. Portanto, desenvolvemos anteriormente um algoritmo no MATLAB Simulink usando suas caixas de ferramentas de processamento e otimização de imagens para pós-processamento13 para análise de microdomínios locais de Ca2+ .
Recentemente, desenvolvemos um novo pipeline de pós-processamento de código aberto chamado DARTS para análise de microdomínio de Ca2+ em imagens de células vivas de alta resolução usando a plataforma de software Python12. Aqui, diferentes algoritmos de deconvolução podem ser selecionados, dependendo da preferência do usuário, uma normalização da forma da célula realizada para compensar as mudanças morfológicas da forma da célula e parâmetros específicos do microscópio e da medição definidos (por exemplo, escala, taxa de quadros, tempo medido) (Figura 4).
Depois de selecionar os parâmetros para a análise do microdomínio de Ca2+ , uma segunda janela pop-up é aberta para cada medição individual para definir o contato do cordão (Figura 5). Para definir o contato do cordão, o usuário pode rolar manualmente pelo arquivo tiff usando o controle deslizante e selecionar a moldura de contato do cordão individualmente. O contato do cordão é selecionado clicando no local de contato do cordão (Figura 5, cordão e contato do cordão indicados por anel e seta amarelos), bem como na seleção da célula. Esta etapa deve ser repetida para cada célula de interesse. Por fim, o pós-processamento automatizado de imagens é aplicado e os dados dos resultados são resumidos e salvos em uma planilha.
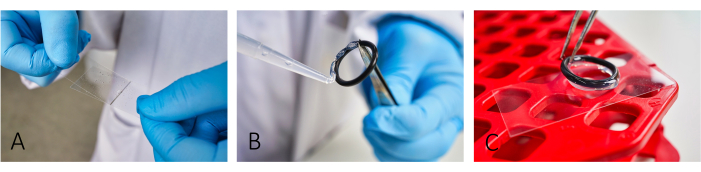
Figura 1: Fluxo de trabalho de preparação de lâminas para imagens. (A) Adicione e espalhe BSA e PLL na lâmina usando uma segunda lamínula de vidro. (B, C) Para construir uma câmara, cole os anéis de vedação de borracha usando graxa de silicone na lâmina. Certifique-se de que todo o anel esteja coberto com uma fina camada de graxa para ter o isolamento adequado da câmara. Clique aqui para ver uma versão maior desta figura.
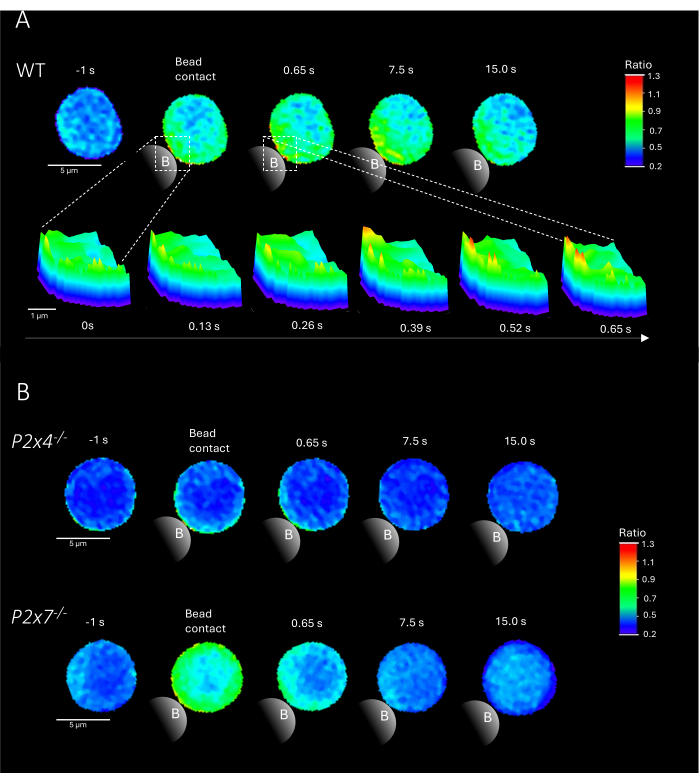
Figura 2: Células representativas dos microdomínios Ca2+ dependentes do receptor de células T em uma célula T CD4+ do tipo selvagem murino primário (WT) (A), P2x4-/- ou P2x7-/-(B). As células T CD4+ foram isoladas negativamente e carregadas com Fluo-4 AM e Fura Red, conforme descrito acima. As células T foram analisadas usando o pipeline DARTS, resultando em imagens celulares comparáveis aos resultados publicados anteriormente9. (A) Célula T primária WT 1 s antes da estimulação com esferas revestidas anti-CD3 / anti-CD28 e até 15 s após a estimulação (barra de escala de 5 μm), bem como gráfico de superfície 3D de um zoom de 0 s a 0,65 s na região de contato do cordão (barra de escala de 1 μm). (B) Faixa superior: célula T primária P2x4-/- representativa 1 s antes e até 15 s após a estimulação do grânulo. Faixa inferior: célula T primária P2x7-/- representativa 1 s antes e até 15 s após a estimulação do grânulo. Clique aqui para ver uma versão maior desta figura.
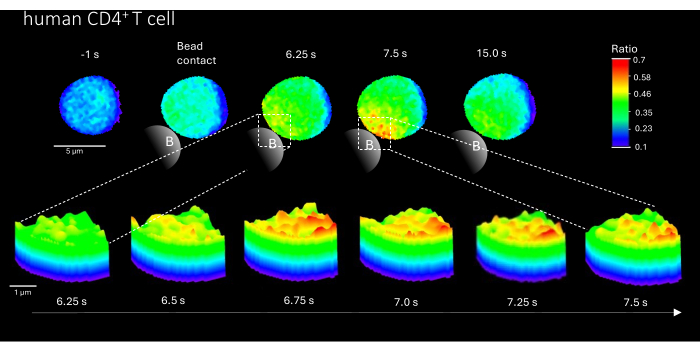
Figura 3: Microdomínios de Ca2+ em uma célula T humana primária representativa após estimulação com TCR. As células T CD4+ humanas primárias foram isoladas de células mononucleares do sangue periférico (PBMCs) por classificação de células ativadas por fluorescência (FACS) de revestimentos leucocitários e carregadas com Fluo-4 AM e Fura Red, conforme descrito acima. A figura mostra uma célula T humana primária 1 s antes da estimulação com esferas revestidas com anti-CD3 e até 15 s após a estimulação (barra de escala de 5 μm), bem como um gráfico de superfície 3D de um zoom de 6,25 s a 7,5 s na região de contato do grânulo (barra de escala de 1 μm). Clique aqui para ver uma versão maior desta figura.
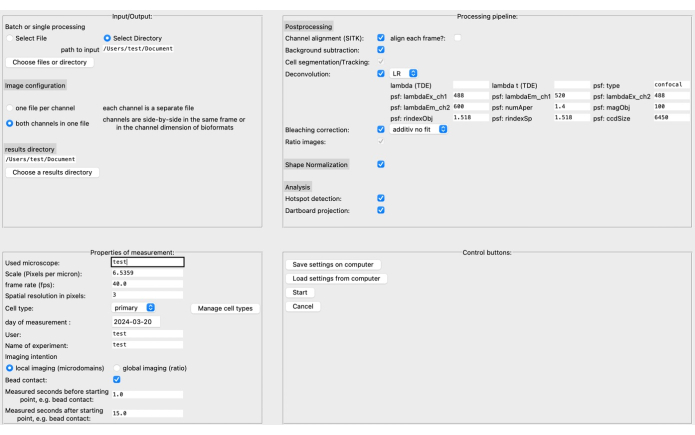
Figura 4: A interface gráfica do usuário (GUI) do DARTS. A GUI é dividida em quatro áreas. Na área Entrada/Saída, você precisa fornecer informações sobre os dados brutos, incluindo o diretório de origem e a configuração da imagem (dois canais por arquivo ou canais separados), bem como o diretório de resultados. Na área Propriedades de Medição, o experimento precisa ser descrito com todas as suas informações relevantes, como escala (mícrons por pixel), taxa de quadros e intervalo de medição em relação ao ponto de partida determinado posteriormente. Em seguida, um pipeline de processamento que consiste em etapas de pós-processamento, normalização de forma e análise real (detecção de microdomínio e acúmulo de dados do alvo de dardos) pode ser montado. Por fim, as configurações podem ser salvas ou carregadas do computador. Uma vez configurada a análise, clique em Iniciar para prosseguir. Para ler mais sobre a configuração, visite https://ipmi-icns-uke.github.io/DARTS/General/Usage.html. Clique aqui para ver uma versão maior desta figura.
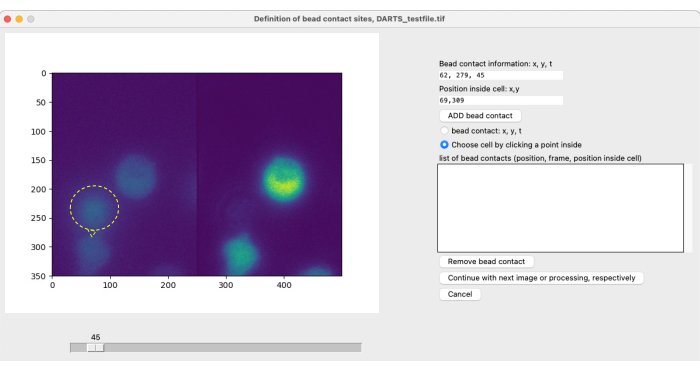
Figura 5: Definição manual dos contatos do cordão. Se contas forem adicionadas às células durante o experimento, o tempo de contato inicial da conta com uma célula de interesse e o local de contato devem ser definidos manualmente. Isso é feito rolando pelos quadros com o controle deslizante e encontrando uma posição (x,y) em um ponto de tempo t. Para preencher automaticamente o campo de informações de contato do cordão, o usuário clica na metade esquerda da imagem do microscópio na posição de contato do cordão. Em seguida, para associar uma célula ao contato do cordão, o usuário clica em uma posição dentro da célula que tenha um contato do cordão. A informação tem de ser confirmada selecionando ADD bead. Clique aqui para ver uma versão maior desta figura.
Discussão
Descrevemos um extenso protocolo para imagens de células vivas de alta resolução de microdomínios locais de Ca2+ em células T murinas e humanas primárias desencadeadas pela estimulação de TCR/CD3 através de esferas revestidas com anticorpos. Além disso, implementamos um algoritmo baseado em Python de código aberto e fácil de usar para identificar e analisar sinais locais de Ca2+. Notavelmente, o protocolo não se limita à detecção de microdomínios de Ca2+ no contexto da estimulação de TCR/CD3, mas é adaptável a outros tipos de células (imunes), como linhagens de células NK (KHYG-1)12 ou microdomínios de Ca2+ independentes de TCR10,11.
Uma etapa crítica dentro do protocolo é o tamanho e o número das esferas estimulantes. Para imitar uma sinapse imunológica, as contas devem ser semelhantes em tamanho às células. Portanto, para células T murinas e humanas primárias, bem como linhas celulares (Jurkat e KHYG1), usamos esferas magnéticas com um diâmetro de 10 μm. Além disso, cada célula deve ser estimulada apenas por uma única conta. Portanto, o número de contas adicionadas a cada slide deve ser suficiente, por um lado, mas se houver muitas contas no campo de visão, o fundo aumenta e não é possível detectar um único ponto de tempo de ativação e lado de contato.
O protocolo utiliza os corantes fluorescentes Ca2+ Fluo-4 AM e FuraRed AM de forma raciométrica, permitindo a calibração dos dados13. Além disso, o protocolo pode ser adaptado a outros pares de indicadores de Ca2+ , mas deve-se ter cuidado no processo de seleção em termos de cinética de ligação de Ca2+ , distribuição subcelular e fotobranqueamento1. Além disso, as condições de carga devem ser desenvolvidas e otimizadas para cada tipo de célula individual, mas as concentrações indicadas aqui são um bom ponto de partida. Para visualizar os microdomínios de Ca2+ , o Kd dos corantes de Ca2+ deve estar na faixa de 300-1200 nM e o tempo de aquisição por quadro deve ser de ≤60 ms. Se a intensidade da fluorescência for muito baixa, o conjunto de filtros deve ser verificado, mas também é possível carregar uma quantidade dupla de corante Ca2+ nas células T. No entanto, o corante Ca2+ pode se deslocar incorretamente para outras organelas ou sequestrar para vesículas, mas também pode atuar como um tampão Ca2+ e afetar as respostas de Ca2+ .
Uma limitação do algoritmo de análise é que uma forma esférica da célula é assumida; portanto, tipos de células com morfologias diferentes podem precisar de adaptação da caixa de ferramentas de análise. O algoritmo tem sido usado para analisar microdomínios locais de Ca2+ em células T murinas primárias, bem como células T Jurkat e uma linhagem celular NK (KHYG-1)12 e foi bem-sucedido na análise de microdomínios de Ca2+ para uma linhagem celular neuronal murina (N2a, dados não publicados). Em princípio, o protocolo e a caixa de ferramentas de análise podem ser usados para analisar tipos de células não esféricas, como células HEK293 ou HeLa, mas para esses tipos de células, a projeção do alvo não pode ser adaptada porque é baseada em uma estrutura redonda e normalização de forma das células. Além disso, o protocolo para detectar microdomínios iniciais localizados de Ca2+ após a estimulação do grânulo pode ser adaptado para analisar sinais locais de Ca2+ derivados de outros estímulos, como compostos ativadores ou inibidores solúveis, bem como microdomínios de Ca2+ dependentes de adesão e independentes de TCR/CD310,11. É importante notar que é mais fácil definir um único contato de cordão em termos de tempo e localização do que determinar o ponto de partida de ativação após compostos solúveis.
Uma limitação geral para a detecção da formação do microdomínio Ca2+ reside na alta resolução temporal-espacial necessária e na alta relação sinal-ruído (SNR) necessária. Atualmente, a resolução derivada de nossa configuração atinge uma resolução espacial calculada de ~0,368 μm e resolução temporal de ~40 quadros por segundo (fps)1. Avanços recentes no desenvolvimento de câmeras e detectores, bem como a melhoria de corantes fluorescentes, podem levar à possibilidade de alcançar gravações ópticas de canal único, como foram descritas para construções ORAI-GECI (indicadores de Ca2+ geneticamente expressos)24 para imagens de células vivas usando indicadores de Ca2+ com maior resolução temporal e espacial no futuro.
Em conjunto, o protocolo e a ferramenta de análise para imagens de microdomínio de Ca2+ de alta resolução descritos aqui podem ser usados não apenas para analisar os sinais locais iniciais de Ca2+ em células T, mas também podem ser adaptados a outros tipos de células para decifrar a importância da sinalização local de Ca2+ nestas.
Divulgações
Os autores declaram que a pesquisa foi realizada na ausência de qualquer relação comercial ou financeira que pudesse ser considerada um potencial conflito de interesses.
Agradecimentos
Este trabalho foi apoiado pela Deutsche Forschungsgemeinschaft (DFG) (projeto número 335447717; SFB1328, A02 para B-PD e RW; A14 para ET; número do projeto 516286863 ao B-PD). Os autores agradecem aos doadores de sangue e ao Departamento de Medicina Transfusional do UKE por sua cooperação.
Materiais
| Name | Company | Catalog Number | Comments |
| α-CD3 | BD Pharmingen | 553058 | |
| α-CD28 | BD Pharmingen | 553295 | |
| Anaconda | Anaconda | https://docs.anaconda.com/free/anaconda/install/index.html | |
| Bovine serum albumin | Sigma-Aldrich | A2058 | |
| Cell strainer | Biofil | CSS-013-070 | 70 µm diameter |
| Countess Automated Cell Counter | Invitrogen | A49865 | |
| Cover slips | Paul Marienfeld GmbH | 101202 | |
| DARTS | GitHub | https://github.com/IPMI-ICNS-UKE/DARTS | |
| Dulbecco’s Phosphate buffered saline | Gibco, Thermo Fisher | 14190144 | |
| EasySep CD4+ T cell Isolation Kit | Stemcell | 19852 | |
| EasySep Magnetic Multistand | Stemcell | 18010 | |
| Fluo 4-AM | Invitrogen | F14201 | |
| Fura Red-AM | Invitrogen | F3020 | |
| Gibco RPMI 1640 medium | Gibco, Thermo Fisher | 11875093 | |
| Git | GitHub | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git | |
| immersion oil | Leica | 11513859 | |
| Newborn calf serum | Sigma-Aldrich | N4637 | |
| Oracle | Oracle | https://www.oracle.com/de/java/technologies/downloads/ | |
| Penicillin/Streptamycin | Gibco, Thermo Fisher | 15240062 | |
| Poly-L-lysine | Sigma-Aldrich | P4707 | |
| Protein G magnetic beads | Merck | LSKMAGG02 | 10 µM diameter |
| Python | Python | https://www.python.org/downloads/ | |
| Silicon grease (basylone) | Bayer | 291-1220 | |
| Time-Dependent Entropy Deconvolution | GitHub | https://ipmi-icns-uke.github.io/TDEntropyDeconvolution/General/2-usage.html#input-parameters | |
| Tween | q-biogene | TWEEN201 |
Referências
- Wolf, I. M. A., et al. Frontrunners of T cell activation: Initial, localized Ca2+ signals mediated by NAADP and the type 1 ryanodine receptor. Sci Signal. 8 (398), ra102 (2015).
- Taguchi, T., Mukai, K. Innate immunity signalling and membrane trafficking. Curr Opin Cell Biol. 59, 1-7 (2019).
- Trebak, M., Kinet, J. -. P. Calcium signalling in T cells. Nat Rev Immunol. 19 (3), 154-169 (2019).
- Diercks, B. -. P., et al. ORAI1, STIM1/2, and RYR1 shape subsecond Ca2+ microdomains upon T cell activation. Sci Signal. 11, eaat0358 (2018).
- Cheng, H., Lederer, W. J., Cannell, M. B. Calcium sparks: Elementary events underlying excitation-contraction coupling in heart muscle. Science. 262 (5134), 740-744 (1993).
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Berridge, M. J., Lipp, P., Bootman, M. D. The versatility and universality of calcium signalling. Nat Rev Mol Cell Biol. 1 (1), 11-21 (2000).
- Kummerow, C., Junker, C., Kruse, K., Rieger, H., Quintana, A., Hoth, M. The immunological synapse controls local and global calcium signals in T lymphocytes. Immunol Rev. 231 (1), 132-147 (2009).
- Brock, V. J., et al. P2X4 and P2X7 are essential players in basal T cell activity and Ca2+ signaling milliseconds after T cell activation. Sci Adv. 8 (5), eabl9770 (2022).
- Weiß, M., et al. Adhesion to laminin-1 and collagen IV induces the formation of Ca2+ microdomains that sensitize mouse T cells for activation. Sci Signal. 16 (790), eabn9405 (2023).
- Diercks, B. -. P. The importance of Ca2+ microdomains for the adaptive immune response. Biochim Biophys Acta Mol Cell Res. 1871 (5), 119710 (2024).
- Woelk, L. -. M., et al. DARTS: an open-source Python pipeline for Ca2+ microdomain analysis in live cell imaging data. Front Immunol. 14, 1299435 (2024).
- Diercks, B. -. P., Werner, R., Schetelig, D., Wolf, I. M. A., Guse, A. H. High-resolution calcium imaging method for local calcium signaling. Methods Mol Biol. 1929, 27-39 (1929).
- . Python Available from: https://www.python.org/downloads/ (2024)
- . Anaconda Available from: https://docs.anaconda.com/free/anaconda/install/index.html (2024)
- . git Available from: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git (2024)
- . DARTS Available from: https://github.com/IPMI-ICNS-UKE/DARTS (2024)
- . DARTS GitHub Repository Available from: https://ipmi-icns-uke.github.io/DARTS/ (2024)
- . Oracle Available from: https://www.oracle.com/de/java/technologies/downloads/ (2024)
- . Time-Dependent Entropy Deconvolution Available from: https://ipmi-icns-uke.github.io/TDEntropyDeconvolution/General/2-usage.html#input-parameters (2024)
- Gu, F., et al. Dual NADPH oxidases DUOX1 and DUOX2 synthesize NAADP and are necessary for Ca2+ signaling during T cell activation. Sci Signal. 14 (709), eabe3800 (2021).
- Roggenkamp, H. G., et al. HN1L/JPT2: A signaling protein that connects NAADP generation to Ca2+ microdomain formation. Sci Signal. 14 (675), eabd5647 (2021).
- Heßling, L. D., Troost-Kind, B., Weiß, M. NAADP-binding proteins - Linking NAADP signaling to cancer and immunity. Biochim Biophy. Acta Mol Cell Res. 1870 (7), 119531 (2023).
- Dynes, J. L., Amcheslavsky, A., Cahalan, M. D. Genetically targeted single-channel optical recording reveals multiple Orai1 gating states and oscillations in calcium influx. Proc Natl Acad Sci U S A. 113 (2), 440-445 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados