Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Выделение и идентификация бактерий, устойчивых к антибиотикам, передающихся через воду, и молекулярная характеристика их генов устойчивости к антибиотикам
В этой статье
Резюме
Здесь мы представляем подробный протокол выделения и идентификации устойчивых к антибиотикам бактерий из воды и молекулярную характеристику их генов устойчивости к антибиотикам (ARG). Использование методов, основанных на культурах и не основанных на культурах (метагеномный анализ), дает полную информацию об общем бактериальном разнообразии и общем пуле различных ARG, присутствующих в пресных водах из Мумбаи, Индия.
Аннотация
Развитие и распространение устойчивости к антибиотикам (АР) через микробиоту, связанную с пресноводными водоемами, является серьезной глобальной проблемой здравоохранения. В настоящем исследовании образцы пресной воды были собраны и проанализированы в отношении общего бактериального разнообразия и генов AR (ARG) с использованием как традиционных методов, основанных на культурах, так и метагеномного подхода, независимого от культуры. В данной работе представлен систематический протокол перечисления общих и устойчивых к антибиотикам культивируемых бактерий из образцов пресной воды и определения фенотипической и генотипической резистентности в культивируемых изолятах. Кроме того, мы сообщаем об использовании полного метагеномного анализа общей метагеномной ДНК, извлеченной из образца пресной воды, для идентификации общего бактериального разнообразия, включая некультивируемые бактерии, и идентификации общего пула различных ARG (резистома) в водном объекте. Следуя этим подробным протоколам, мы наблюдали высокую устойчивую к антибиотикам бактериальную нагрузку в диапазоне 9,6 × 10 5-1,2 × 109 КОЕ/мл. Большинство изолятов были устойчивы к множественным протестированным антибиотикам, включая цефотаксим, ампициллин, левофлоксацин, хлорамфеникол, цефтриаксон, гентамицин, неомицин, триметоприм и ципрофлоксацин, с индексами множественной устойчивости к антибиотикам (MAR) ≥0,2, что указывает на высокие уровни резистентности в изолятах. Секвенирование 16S рРНК выявило потенциальные патогены человека, такие как Klebsiella pneumoniae, и оппортунистические бактерии, такие как Comamonas spp., Micrococcus spp., Arthrobacter spp. и Aeromonas spp. Молекулярная характеристика изолятов показала наличие различных ARG, таких как blaTEM, blaCTX-M (β-лактамы), aadA, aac (6')-Ib (аминогликозиды) и dfr1 (триметопримы), что также было подтверждено всем метагеномным анализом ДНК. В метагеномной ДНК также была обнаружена высокая распространенность других АРГ, кодирующих для насосов оттока антибиотиков - mtrA, macB, mdtA, acrD, β-lactamases-SMB-1, VIM-20, ccrA, ampC, blaZ, гена хлорамфениколацетилтрансферазы catB10 и гена устойчивости к рифампицину rphB-. С помощью протоколов, обсуждаемых в этом исследовании, мы подтвердили наличие передаваемых через воду бактерий MAR с различными фенотипическими и генотипическими признаками AR. Таким образом, весь метагеномный анализ ДНК может быть использован в качестве дополнительного метода к традиционным методам, основанным на культурах, для определения общего статуса AR водного объекта.
Введение
Устойчивость к противомикробным препаратам (УПП) была определена в качестве одной из наиболее актуальных глобальных проблем. Быстрая эволюция УПП и его распространение во всем мире являются одной из самых больших угроз здоровью человека и мировой экономике с точки зрения связанных с ним расходов наздравоохранение1. Чрезмерное и неправильное использование антибиотиков привело к увеличению AR. Это было подчеркнуто пандемией COVID-19, во время которой лечение ассоциированных вторичных инфекций во многих случаях было чрезвычайно скомпрометировано из-за УПП у пострадавших пациентов2. Помимо прямого использования/неправильного использования антибиотиков людьми, серьезнойпроблемой является чрезмерное и неправильное использование антибиотиков в сельском хозяйстве и животноводстве и их ненадлежащий сброс в окружающую среду, включая водные объекты3. Появление новых признаков резистентности и множественной лекарственной устойчивости у бактерий настоятельно подчеркивает необходимость лучшего понимания факторов, ведущих к развитию АР и его распространению. Множественные устойчивые к антибиотикам бактерии, которые часто несут несколько генов AR (ARG) на мобильных генетических элементах, таких как плазмиды, могут передавать эти гены устойчивости нерезистентным микроорганизмам, включая потенциальные патогены человека, что приводит к появлению супербактерий, которые не поддаются лечению даже антибиотиками последней инстанции4. Эти множественные устойчивые к антибиотикам бактерии, если они присутствуют в водных экосистемах, могут непосредственно проникать в кишечник человека через потребление загрязненных продуктов на водной основе, таких как рыба, крабы и моллюски. Предыдущие исследования показали, что распространение бактерий AR в естественных водных системах может также достигать других источников воды, включая питьевую воду, и, таким образом, может попасть в пищевую цепь человека 5,6,7.
Целью настоящего исследования является предоставление всеобъемлющего протокола с использованием комбинации методов, основанных на культурах и не основанных на культурах (весь метагеномный анализ), для получения полной информации об общем бактериальном разнообразии и общем пуле различных ARG, присутствующих в водном объекте в Мумбаи, Индия. Традиционно для изучения бактериального разнообразия в водоемах используются методы, основанные на культурах. Поскольку культивируемые микроорганизмы составляют лишь небольшой процент от общей микробиоты в любой нише, чтобы лучше понять общее состояние бактериального разнообразия и различные устойчивые признаки, преобладающие в любом образце, необходимо использовать различные методы, основанные на культуре и независимые от культуры методы. Одним из таких надежных и надежных методов, не зависящих от культуры, является анализ всей метагеномной ДНК. Этот высокопроизводительный метод был успешно использован в различных исследованиях бактериального разнообразия или функциональных аннотаций различных ARG 8,9. Этот метод использует метагеном (общий генетический материал в образце) в качестве исходного материала для различных анализов и, следовательно, не зависит от культуры. Протоколы в настоящем исследовании могут быть использованы для анализа всей метагеномной ДНК для получения информации об общем бактериальном разнообразии и различных ARG (резистоме) в образцах воды.
протокол
1. Сбор и обработка образцов
- Сбор образцов
- Соберите соответствующий объем пробы воды в стерильный контейнер (контейнеры) для проб, обеспечив заполнение не более 3/4 контейнера.
- Транспортируйте образцы в лабораторию в асептических условиях как можно скорее после сбора и немедленно обрабатывайте их.
- Обработка образцов
- Асептически фильтруйте образец воды через стерильную муслиновую ткань, чтобы удалить любые твердые частицы.
- Проводят соответствующие последовательные разбавления отфильтрованной воды для дальнейшего анализа.
2. Оценка общей бактериальной нагрузки и количества устойчивых к антибиотикам бактерий
- Определение общей бактериальной нагрузки
- Суспендировать 18,12 г агара R2A, модифицированный порошок в 1000 мл двойной дистиллированной воды, и растворить смесь путем нагревания. Автоклав растворенной смеси при 121 °C, 15 psi в течение 20 мин. Готовят R2A Agar, модифицированные пластины, заливая соответствующее количество автоклавной смеси в стерильные пластины Петри (например, добавляют приблизительно 20 мл автоклавной стерильной среды к 90 мм стерильной пластине Петри).
- Равномерно распределите 100 мкл соответствующих разбавлений образца отфильтрованной воды на Агар R2A, модифицированную пластину, как только среда затвердеет. Выполните эксперимент в двух экземплярах.
- Инкубировать все вышеперечисленные пластины при 35-37 °C в течение 48 ч (изменять температуру и время инкубации в зависимости от среды, используемой для изоляции).
- Выражайте общую бактериальную нагрузку в единицах колониеобразования на миллилитр (КОЕ/мл) с помощью уравнения (1):
 (1)
(1)
- Определение количества бактерий AR
- Выполните шаги 2.1.1-2.1.4. Однако вместо R2A Agar, модифицированных пластин, используйте R2A Agar, модифицированные пластины, дополненные индивидуально пятью различными антибиотиками, а именно цефотаксимом (3 мкг / мл), ципрофлоксацином (0,5 мкг / мл), эритромицином (20 мкг / мл), канамицином (15 мкг / мл) и ванкомицином (3 мкг / мл).
- Добавляют антибиотики отдельно в пробирки, содержащие 20 мл стерильного расплавленного R2A-агара, модифицированного (с температурой расплавленного R2A-агара, модифицированного при ≤40 °C) для достижения конечной концентрации антибиотика, как указано на этапе 2.2.1.
- Закрутите для равномерного перемешивания и вылейте на стерильные тарелки Петри до того, как агар затвердеет. Выполните эксперимент в двух экземплярах.
- Инкубировать все вышеперечисленные пластины при 35-37 °С в течение 48 ч (при использовании другой среды температура и время инкубации могут варьироваться).
- Для контроля качества и проверки эффективности антибиотиков наносите 100 мкл бактериальных суспензий Escherichia coli ATCC 25922 и Staphylococcus aureus ATCC 29213 на их соответствующие антибиотикосодержащие R2A agar, модифицированные пластины (убедитесь, что плотность свежей культуры, используемой для посева, составляет OD = 0,5 при 600 нм).
- Определите количество устойчивых к антибиотикам бактерий в пересчете на КОЕ/мл, как описано в шаге 2.1.4.
- Глицериновые запасы изолятов
- Выберите морфологически различные колонии AR.
- Суспендировать одну изолированную колонию в 2 мл стерильного бульона Луриа-Бертани, содержащего соответствующий антибиотик (например, если колонию отобрали из пластины, содержащей цефотаксим, инокулируют колонию со стадии 2.3.1 в стерильный бульон Лурия-Бертани, содержащий цефотаксим в соответствующей концентрации).
- Инкубируйте инокулированные трубки при 37 °C при 80 об/мин до тех пор, пока OD600 не достигнет 0,5.
- Получают глицериновые запасы изолятов путем смешивания 750 мкл суспензии культуры со стадии 2.3.3 в 250 мкл стерильного 100% глицерина в асептических условиях.
- Хранить запасы глицерина при температуре −80 °C до дальнейшего анализа.
ПРИМЕЧАНИЕ: Для возрождения культур из запасов глицерина разморозьте запасы глицерина при 4 °C. Инокулируют петлю этого бульона в 2 мл стерильного бульона Лурия-Бертани, содержащего соответствующий антибиотик, и дают расти.
3. Идентификация культивируемых бактерий путем секвенирования гена 16S рРНК
- Подготовка шаблона ДНК из изолятов для ПЦР
ПРИМЕЧАНИЕ: Протокол, описанный для подготовки шаблона ДНК для ПЦР для выделения сырой ДНК из бактерий, приведен Carlson et al.10.- Используя стерильную зубочистку, возьмите единичную, изолированную, чистую колонию изолята, растущую на пластине Петри. Суспендировать бактериальную колонию в 100 мкл стерильной двойной дистиллированной воды в стерильной микроцентрифужной трубке и кипятить в течение 10 мин.
- Центрифугируйте суспензию при 10 000 × г в течение 2 мин, чтобы гранулировать мусор, и перенесите супернатант в свежую стерильную микроцентрифужную трубку для использования в качестве шаблона сырой ДНК.
- Таргетная амплификация ПЦР области V3 гена 16S рРНК и секвенирование
- Готовят 40 мкл реакционной смеси в ПЦР-трубке для амплификации ПЦР, как указано в таблице 1.
ПРИМЕЧАНИЕ: Подготовка ДНК должна проводиться на ледяной глыбе, минимизируя вероятность загрязнения (надевайте перчатки при работе с реагентами и тщательно очищайте рабочую поверхность 70% этанолом). - Поместите трубку в термоблок и запустите соответствующую программу в термоциклере ПЦР. См. таблицу 2 для стандартизированных условий цикла ПЦР и информацию праймера для амплификации областей V3 генов 16S рРНК.
- Для разрешения ампликонов и визуализации проводят электрофорез агарозного геля (AGE). Смешайте 10 мкл амплифицированного продукта ПЦР и 2 мкл 6-кратного гелевого нагрузочного буфера (таблица 3) и загрузите эту смесь в лунки на 1,5% агарозном геле (растворите 1,5 г порошка агарозы в 100 мл 1x буфера TAE [Таблица 3]), содержащего 5 мкл 10 мг/мл бромида этидия (EtBr) до конечной концентрации 0,5 мкг/мл EtBr в 100 мл агарозного геля.
ВНИМАНИЕ: EtBr является мощным канцерогеном. Перчатки следует носить постоянно при обращении с EtBr и гелями, содержащими EtBr. - Добавьте лестницу ДНК для оценки размера ампликонов.
- Проводят электрофорез геля в буфере резервуара TAE при 80-100 В.
- Как только следящий краситель пройдет 3/4 геля, остановите электрофорез и визуализируйте ампликонные полосы под УФ-трансиллюминатором.
- Используйте продукт ПЦР (ампликон) для секвенирования гена 16S рРНК для идентификации изолята.
- Количественно оцените ампликон, подвергнув его спектрофотометрическому анализу с помощью уравнения (2).
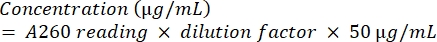 (2)
(2) - Чтобы проверить чистоту ДНК, рассчитайте соотношение А260/А280.
ПРИМЕЧАНИЕ: В идеале это число должно быть выше 1,5 и, предпочтительно, между 1,8 и 2,0. - Чтобы определить изоляты, сравните последовательности, полученные с базами данных последовательностей, используя соответствующее средство поиска выравнивания.
- Готовят 40 мкл реакционной смеси в ПЦР-трубке для амплификации ПЦР, как указано в таблице 1.
4. Выявление устойчивости к антибиотикам в изолятах с помощью тестирования чувствительности к антибиотикам
ПРИМЕЧАНИЕ: Этот протокол описывает метод тестирования чувствительности к антибиотикам (АСТ) путем диффузии диска. Использовались следующие диски антибиотиков: цефотаксим (5 мкг), ампициллин (10 мкг), левофлоксацин (5 мкг), левофлоксацин (5 мкг), хлорамфеникол (30 мкг), тигециклин (15 мкг), цефтриаксон (30 мкг), имипенем (10 мкг), гентамицин (10 мкг), неомицин (10 мкг), триметоприм (5 мкг) и ципрофлоксацин (5 мкг).
- Подготовка инокулята к АСТ
- Асептически инокулируют одну, изолированную, очищенную колонию AR, используя стерильную петлю в 2 мл стерильной неселективной среды, такой как бульон Лурии-Бертани (без какого-либо антибиотика), и инкубируют при 37 °C при 80 об/мин в течение ночи.
- Повторно суспендировать путем взятия 100-150 мкл выращенной на ночь культуры (приблизительно, OD600 = 1,8-2,0) в 2 мл свежей неселективной среды бульона Лурия-Бертани и инкубации в течение 2-4 ч (до тех пор, пока ОД600 не достигнет 0,4-0,5).
- Разбавляют эту свежевыращенную культуральную суспензию стерильным 0,85% физиологическим раствором таким образом, чтобы плотность культуры была равна 0,5 мкфарландской стандартной (приблизительно, OD600 = 0,1), что примерно соответствует 1-2 × 108 клеток/мл.
- Осторожно перемешайте бактериальную суспензию для равномерного распределения клеток.
- Используйте вышеуказанную суспензию в течение 15 мин после разведения.
- Инокуляция агаровых пластин
- Подготовьте пластины Мюллера-Хинтона Агара (MHA) для выполнения АСТ путем смешивания 38 г MHA в 1000 мл двойной дистиллированной воды и растворите смесь путем нагревания. Автоклав растворенной смеси при 121 °C, 15 psi в течение 15 мин.
- Убедитесь, что глубина MHA в пластинах составляет 4 мм (25 мл среды на пластину).
- Одновременно извлеките диски антибиотиков из морозильной камеры и нагрейте их до комнатной температуры.
ПРИМЕЧАНИЕ: Диски антибиотиков следует постепенно размораживать путем первоначального размораживания дисков при 4 °C, а затем при комнатной температуре, чтобы уменьшить любую потенциальную опасность конденсации на дисках, которая впоследствии может повлиять на зону ингибирования (ZOI). - В асептических условиях окуните стерильный ватный тампон в инокулят, приготовленный на этапе 4.1, и удалите лишнюю суспензию, чтобы избежать чрезмерной прививки пластин.
- Равномерно распределите культуру по тарелкам, начиная с верхней части пластины MHA и переходя взад и вперед от края к краю. Поверните пластину на 60° во время смазывания.
- Применение антибиотических дисков
- С помощью пламенно-стерилизованных щипцов асептически перенесите диски антибиотиков на инокулированные пластины MHA и осторожно надавите на диски, чтобы обеспечить полный контакт уровня с агаром.
ПРИМЕЧАНИЕ: Эта процедура должна быть выполнена в течение 15 минут после посева культуры на пластинах. - Поместите соответствующее количество антибиотических дисков на агаровую пластину с учетом организма, используемого антибиотика и размера пластины, чтобы избежать перекрытия зон ингибирования.
ПРИМЕЧАНИЕ: Четыре-пять дисков могут быть размещены на круглой пластине диаметром 90 мм.
- С помощью пламенно-стерилизованных щипцов асептически перенесите диски антибиотиков на инокулированные пластины MHA и осторожно надавите на диски, чтобы обеспечить полный контакт уровня с агаром.
- Инкубация плит
- В течение 15 мин после применения антибиотических дисков инвертируют пластины и инкубируют при 37 °C в течение ночи.
- Интерпретация результатов
- Измерьте диаметр ZOI в миллиметрах (мм) и интерпретируйте в соответствии со значениями точек останова, заданными EUCAST11. Смотрите два примера, приведенных ниже.
- Точка разрыва диаметра зоны (мм) для диска антибиотика ципрофлоксацина (5 мкг) для Enterobacterales составляет S ≥ 25 и R < 22, что означает, что он считается чувствительным (S), если ZOI ≥ 25 мм, в то время как он устойчив (R), если ZOI < 22 мм. Если диаметр ZOI падает между 22 и 25, изолят считается промежуточным (I).
- Точка разрыва диаметра зоны (мм) для диска антибиотика левомицетина (30 мкг) для Staphylococcus spp. составляет S ≥ 18 и R < 18, что означает, что он считается чувствительным, если ZOI ≥ 18 мм, в то время как он устойчив, если ZOI < 18 мм.
- Определите индекс множественной устойчивости к антибиотикам (MAR), найдя отношение количества антибиотиков, к которым изолят устойчив, к общему количеству антибиотиков, которым подвергается изолят.
ПРИМЕЧАНИЕ: Для контроля качества , E. coli ATCC 25922 и S. Стафилококк ATCC 29213 используются в качестве эталонных штаммов в соответствии с протоколом, как на шаге 4.
- Измерьте диаметр ZOI в миллиметрах (мм) и интерпретируйте в соответствии со значениями точек останова, заданными EUCAST11. Смотрите два примера, приведенных ниже.
5. Обнаружение генов устойчивости к антибиотикам в изолятах на основе ПЦР
- Используйте стандартный протокол ПЦР для идентификации ARG в изолятах. Подготовьте шаблон ДНК, используя протокол, приведенный в шаге 3.1.
ПРИМЕЧАНИЕ: Условия цикла ПЦР, использованные в этом исследовании, составляли 94 °C в течение 10 мин, за которыми следовали 35 циклов 94 °C в течение 30 с, отжиг в течение 30 с при соответствующей температуре (как стандартизовано для каждого набора грунтовки), удлинение при 72 °C в течение 40 с и окончательное расширение при 72 °C в течение 5 мин. Реакционная смесь описана в таблице 4. Список АРГ, грунтовок и температур отжига приведен в таблице 5. - Чтобы разрешить, визуализировать и проверить чистоту ампликонов, выполните шаги 3.2.3-3.2.10.
6. Полный метагеномный анализ ДНК для идентификации общего бактериального разнообразия и обнаружения ARG в метагеноме
- Извлечение общей ДНК (метагенома) из образца воды
- Извлеките метагеномную ДНК из образцов отфильтрованной воды.
ПРИМЕЧАНИЕ: В текущем исследовании метагеномная ДНК (общая ДНК) была извлечена из образцов отфильтрованной воды с использованием ссылочного набора изоляции ДНК в соответствии с протоколом производителя (см. Таблицу материалов). - Проверьте качество ДНК, загрузив 3 мкл экстрагированной метагеномной ДНК на 0,8% агарозный гель, и запустите гель при 80-110 В в течение примерно 30 минут.
- Проверьте наличие одной неповрежденной полосы.
- Проверьте концентрацию ДНК с помощью флуорометра.
- Извлеките метагеномную ДНК из образцов отфильтрованной воды.
- Определение бактериального разнообразия и обнаружение ARG с помощью секвенирования всей метагеномной ДНК
- Подготовка библиотеки и ПЦР-амплификация:
- Подготовьте библиотеку парного секвенирования с помощью набора для подготовки библиотеки ДНК, на который имеются ссылки (см. Таблицу материалов).
- Подготовьте ДНК к перевязке адаптера, взяв 200 нг ДНК и механически разрезав ее на более мелкие фрагменты, после чего следует непрерывный этап конечного восстановления, в котором «А» добавляется к 3'-концам.
- В зависимости от платформы, используемой для секвенирования, разложите конкретные адаптеры на обоих концах фрагментов ДНК.
ПРИМЕЧАНИЕ: Последовательности, имеющие решающее значение для привязки библиотек с двойным штрих-кодом к проточной ячейке для секвенирования, присутствуют в этих адаптерах. Это позволяет проводить ПЦР-амплификацию лигированных адаптером фрагментов и связывание стандартных праймеров секвенирования. - Чтобы проверить качество и количество, проанализируйте амплифицированную библиотеку с помощью высокочувствительного днк-чипа в соответствии с инструкциями производителя.
- Генерация и секвенирование кластеров:
- Загрузите усиленную библиотеку на соответствующую платформу виртуализации для создания кластера и последующего секвенирования.
ПРИМЕЧАНИЕ: Молекулы библиотеки связываются с комплементарным адаптером олигоса на парной концевой проточной ячейке. Во время секвенирования передние нити избирательно расщепляются после ресинтеза обратной нити. Эта скопированная обратная нить затем секвенируется с противоположного конца фрагмента.
- Загрузите усиленную библиотеку на соответствующую платформу виртуализации для создания кластера и последующего секвенирования.
- Биоинформационный анализ:
- Генерируйте каркасы из высококачественных данных с помощью соответствующей платформы.
- Подвергнуть эти каркасы биоинформаматическому анализу для таксономической классификации и идентификации ARG.
ПРИМЕЧАНИЕ: Рабочий процесс для всего метагеномного анализа ДНК для идентификации общего бактериального разнообразия и обнаружения ARG в метагеноме приведен на рисунке 1. Технологическая схема полной методологии, описанной в рукописи, приведена на рисунке 2.
- Подготовка библиотеки и ПЦР-амплификация:
Результаты
Общая бактериальная нагрузка и количество устойчивых к антибиотикам (AR) бактерий
Перечисление общей бактериальной нагрузки проводилось путем распределения 10−4-10−6-кратных разбавлений проб воды на R2A Agar, модифицированной среде. Для перечисления количества бакте...
Обсуждение
Сбор и обработка образцов играют важную роль и могут повлиять на результаты и интерпретацию исследования. Следовательно, чтобы исключить изменчивость проб, важно проводить отбор проб в нескольких местах исследуемого пресноводного водоема. Поддержание надлежащих асептических услови?...
Раскрытие информации
У авторов нет конфликтующих интересов для раскрытия.
Благодарности
Эта работа была частично поддержана финансовыми грантами Департамента науки и техники - Содействие университетским исследованиям и научному совершенству (DST-PURSE) Схемы Университета Мумбаи. Девика Гадигаонкар работала в качестве стипендиата проекта по этой схеме. Техническая помощь, предоставленная Харшали Шинде, старшим научным сотрудником Департамента науки и техники, науки и инженерных исследований (DST-SERB) Проект No: CRG/2018/003624, приветствуется.
Материалы
| Name | Company | Catalog Number | Comments |
| 100 bp DNA ladder | Himedia | MBT049-50LN | For estimation of size of the amplicons |
| 2x PCR Taq mastermix | HiMedia | MBT061-50R | For making PCR reaction mixture |
| 37 °C Incubator | GS-192, Gayatri Scientific | NA | For incubation of bacteria |
| 6x Gel Loading Buffer | HiMedia | ML015-1ML | Loading and Tracking dye which helps to weigh down the DNA sample and track the progress of electrophoresis |
| Agarose powder | Himedia | MB229-50G | For resolving amplicons during Agarose Gel Electrophoresis (AGE) |
| Ampicillin antibiotic disc | HiMedia | SD002 | For performing AST |
| Autoclave | Equitron | NA | Required for sterilization of media, glass plates, test tubes, etc |
| Bioanalyzer 2100 | Agilent Technologies | NA | To check the quality and quantity of the amplified library |
| Bisafety B2 Cabinet | IMSET | IMSET BSC-Class II Type B2 | Used for microbiological work like bacterial culturing, AST etc. |
| Cefotaxime antibiotic disc | HiMedia | SD295E-5VL | For performing AST |
| Cefotaxime antibiotic powder | HiMedia | TC352-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Ceftriaxone antibiotic disc | HiMedia | SD065 | For performing AST |
| Centrifuge Minispin | Eppendorf | Minispin Plus-5453 | Used to pellet the debris during crude DNA preparation |
| Chloramphenicol antibiotic disc | HiMedia | SD006-5x50DS | For performing AST |
| Ciprofloxacin antibiotic disc | HiMedia | SD060-5x50DS | For performing AST |
| Ciprofloxacin antibiotic powder | HiMedia | TC447-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Colorimeter | Quest | NA | For checking the OD of culture suspensions |
| Comprehensive Antibiotic Resistance Database (CARD) database | functional annotation of ARGs; https://card.mcmaster.ca/ | ||
| Cooling Shaker Incubator | BTL41 Allied Scientific | NA | For incubation of media plates for culturing bacteria |
| Deep Freezer (-40 °C) | Haier | DW40L, Haier Biomedicals | For storage of glycerol stocks |
| DNA Library Prep Kit | NEB Next Ultra DNA Library Prep Kit for Illumina | NA | Paired-end sequencing library preparation |
| EDTA | HiMedia | GRM1195-100G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Electrophoresis Apparatus | TechResource | 15 cm gel casting tray | For making the agarose gel and carrying out electrophoresis |
| Electrophoresis Power pack with electrodes | Genei | NA | For running the AGE |
| Erythromycin antibiotic disc | HiMedia | SD222-5VL | For performing AST |
| Erythromycin antibiotic powder | HiMedia | CMS528-1G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Erythromycin antibiotic powder | HiMedia | TC024-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Escherichia coli ATCC 25922 | HiMedia | 0335X-1 | Used as a control while performing AST |
| Ethidium Bromide | HiMedia | MB071-1G | Intercalating agent and visualizaion of DNA after electrophoresis under Gel Documentation System |
| Fluorometer | Qubit 2.0 | NA | For determining concentration of extracted metagenomic DNA |
| Gel Documentation System | BioRad | Used for visualizing PCR amplicons after electrophoresis | |
| Gentamicin antibiotic disc | HiMedia | SD170-5x50DS | For performing AST |
| Glacial Acetic Acid | HiMedia | AS119-500ML | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Glycerol | HiMedia | GRM1027-500ML | For making glycerol stocks |
| Imipenem antibiotic disc | HiMedia | SD073 | For performing AST |
| Kaiju Database | NA | NA | For taxonomical classification of reads; https://kaiju.binf.ku.dk/ |
| Kanamycin antibiotic disc | HiMedia | SD017-5x50DS | For performing AST |
| Kanamycin antibiotic powder | HiMedia | MB105-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Levofloxacin antibiotic disc | HiMedia | SD216-5VL | For performing AST |
| Luria Bertani broth | Himedia | M1245-500G | For enrichment of cultures |
| McFarland Standards | Himedia | R092-1No | To compare density of culture suspension |
| Molecular Biology water | HiMedia | TCL018-500ML | For making PCR reaction mixture |
| Mueller-Hinton Agar (MHA) | HiMedia | M173-500G | For performing Antibiotc Susceptibility Testing (AST) |
| Neomycin antibiotic disc | HiMedia | SD731-5x50DS | For performing AST |
| PCR Gradient Thermal Cycler | Eppendorf | Mastercycler Nexus Gradient 230V/50-60 Hz | Used for performing PCR for amplification of 16S rRNA region and various Antibiotic Resistance genes |
| Primers | Xcelris | NA | For PCR amplication |
| R2A Agar, Modified | HiMedia | M1743 | For preparation of media plates for isolation of total and antibiotic resistant (AR) bacterial load |
| Scaffold generation | CLC Genomics Workbench 6.0 | NA | For generation of scaffolds |
| Sequencer | Illumina platform (2 x 150 bp chemistry) | NA | Sequencing of amplified library |
| Sodium Chloride | HiMedia | TC046-500G | For preparation of 0.85% saline for serially diluting the water sample |
| Soil DNA isolation Kit | Xcelgen | NA | For extraction of whole metagenomic DNA from the filtered water sample |
| Staphylococcus aureus subsp. aureus ATCC 29213 | HiMedia | 0365P | Used as a control while performing AST |
| Taxonomical Classification | Kaiju ioinformatics tool | NA | For classification of reads into different taxonomic groups from phylum to genus level |
| The Comprehensive Antibiotic Resistance Database (CARD) | NA | NA | For functional annotation of ARGs |
| Tigecycline antibiotic disc | HiMedia | SD278 | For performing AST |
| Trimethoprim antibiotic disc | HiMedia | SD039-5x50DS | For performing AST |
| Tris base | HiMedia | TC072-500G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Vancomycin antibiotic powder | HiMedia | CMS217 | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Weighing Balance | Mettler Toledo | ME204 Mettler Toledo | Used for weighing media powders, reagent powders etc. |
| NA - Not Applicable |
Ссылки
- Prestinaci, F., Pezzotti, P., Pantosti, A. Antimicrobial resistance: A global multifaceted phenomenon. Pathogens and Global Health. 109 (7), 309-318 (2015).
- Knight, G., et al. Antimicrobial resistance and COVID-19: Intersections and implications. Elife. 10, 64139 (2021).
- Ventola, C. L. The antibiotic resistance crisis: Part 1: Causes and threats. Pharmacy and Therapeutics. 40 (4), 277-283 (2015).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J. R., Rath, A. Metagenomic analysis of total microbial diversity and antibiotic resistance of culturable microorganisms in raw chicken meat and mung sprouts (Phaseolus aureus) sold in retail markets of Mumbai. India. Current Science. 113 (1), 71-79 (2017).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J., Rath, A. Characterization of multiple antibiotic resistance of culturable microorganisms and metagenomic analysis of total microbial diversity of marine fish sold in retail shops in Mumbai, India. Environmental Science and Pollution Research. 25 (7), 6228-6239 (2018).
- Czekalski, N., GascónDíez, E., Bürgmann, H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake. The ISME Journal. 8 (7), 1381-1390 (2014).
- Kraemer, S., Ramachandran, A., Perron, G. Antibiotic pollution in the environment: From microbial ecology to public policy. Microorganisms. 7 (6), 180 (2019).
- Edmonds-Wilson, S., Nurinova, N., Zapka, C., Fierer, N., Wilson, M. Review of human hand microbiome research. Journal of Dermatological Science. 80 (1), 3-12 (2015).
- de Abreu, V., Perdigão, J., Almeida, S. Metagenomic approaches to analyze antimicrobial resistance: An overview. Frontiers in Genetics. 11, 575592 (2021).
- Carlson, S., et al. Detection of multiresistant Salmonella typhimurium DT104 using multiplex and fluorogenic PCR. Molecular and Cellular Probes. 13 (3), 213-222 (1999).
- Breakpoint tables for interpretation of MICs and zone diameters, Version 12.0. European Committee on Antimicrobial Susceptibility Testing Available from: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_12.0_Breakpoint_Tables.pdf (2022)
- Bharti, R., Grimm, D. Current challenges and best-practice protocols for microbiome analysis. Briefings in Bioinformatics. 22 (1), 178-193 (2019).
- Choo, J., Leong, L., Rogers, G. Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports. 5, 16350 (2015).
- Clinical and Laboratory Standards Institute. . Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria that Grow Aerobically, 11th edition. , (2015).
- Bayot, M., Bragg, B. Antimicrobial Susceptibility Testing. StatPearls. , (2021).
- Joseph, A. A., Odimayo, M. S., Olokoba, L. B., Olokoba, A. B., Popoola, G. O. Multiple antibiotic resistance index of Escherichia coli isolates in a tertiary hospital in South-West Nigeria. Medical Journal of Zambia. 44 (4), 225-232 (2017).
- Lorenz, T. Polymerase chain reaction: Basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Rolin, J. Food and human gut as reservoirs of transferable antibiotic resistance encoding genes. Frontiers in Microbiology. 4, 173 (2013).
- Racewicz, P., et al. Prevalence and characterisation of antimicrobial resistance genes and class 1 and 2 integrons in multiresistant Escherichia coli isolated from poultry production. Scientific Reports. 12, 6062 (2022).
- Gebreyes, W., Thakur, S. Multidrug-resistant Salmonella enterica serovar Muenchen from pigs and humans and potential interserovar transfer of antimicrobial resistance. Antimicrobial Agents and Chemotherapy. 49 (2), 503-511 (2005).
- Li, L., et al. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui Province, China. PLoS One. 9 (8), 104356 (2014).
- Akers, K., et al. Aminoglycoside resistance and susceptibility testing errors in Acinetobacter baumannii-calcoaceticus complex. Journal Of Clinical Microbiology. 48 (4), 1132-1138 (2010).
- Ciesielczuk, H. . Extra-intestinal pathogenic Escherichia coli in the UK: The importance in bacteraemia versus urinary tract infection, colonisation of widespread clones and specific virulence factors. , (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены