BioID 细胞环境中特异蛋白复合物的分离-蛋白质组学分析
In This Article
Summary
我们提供了一步一步的协议为分裂-BioID, 一种蛋白质片段互补试验的基础上的接近标记技术 BioID。激活在两个给定的蛋白质的相互作用, 它允许蛋白质组学分析的上下文相关的蛋白质复合物在他们的本机细胞环境。该方法简单, 成本效益高, 只需要标准的实验室设备。
Abstract
为了补充现有的亲和纯化 (AP) 方法, 以确定蛋白质-蛋白质相互作用 (PPI), 已经引入酶, 允许接近相关的标签蛋白质在活细胞。一种这种酶, 比拉 * (用于 BioID 方法), 介导蛋白质的 biotinylation 在大约10毫微米范围内。因此, 当融合到一个感兴趣的蛋白质, 并表达在细胞, 它允许标签的近端蛋白在其本土环境。与依赖于组装蛋白复合物纯化的 AP 相反, BioID 检测在细胞中被标记的蛋白质, 无论它们是否仍然与它们在分离时的兴趣蛋白相互作用。由于它 biotinylates 近端蛋白质, 你可以而且利用链亲和素的特殊亲和性, 以非常有效地分离它们。虽然 BioID 在识别瞬态或弱相互作用方面表现优于 ap, 但 ap 和 BioID 质谱方法都提供了一种已知蛋白质可能的相互作用的概述。但是, 它们不提供有关每个已确定 PPI 的上下文的信息。事实上, 大多数蛋白质通常是几个复合体的一部分, 对应于不同的成熟步骤或不同的功能单元。为了解决这两种方法的共同局限性, 我们设计了一种基于比拉酶的蛋白质片段互补试验。在这项试验中, 两个非活性片段的比拉 * 可以重组成一个活跃的酶, 当接近接近由两个相互作用的蛋白质, 他们被融合。由此产生的分裂-BioID 的检测, 从而允许标签的蛋白质, 聚集在一对相互作用的蛋白质。如果这两个仅在给定的上下文中进行交互, 则 BioID 将允许对其本地蜂窝环境中特定的上下文相关功能单元进行分析。在这里, 我们提供了一步一步的协议, 以测试和应用分裂 BioID 的一对相互作用的蛋白质。
Introduction
由于大多数细胞功能是由动态组装大分子络合物的蛋白质进行的, 蛋白质相互作用的鉴定是生物医学研究中的一项重要努力。事实上, PPI 在疾病中经常被解除管制, 并代表治疗1的潜在目标。PPI 鉴别的最广泛使用的方法是亲和纯化 (AP) 方法, 在细胞裂解后, 一种感兴趣的蛋白质被专门纯化在基质上, 相关蛋白随后通过质谱鉴定(MS)。虽然 AP MS 是一个强有力的方法, 它通常不会表现良好的可溶性蛋白复合物, 非常短暂的相互作用或 PPI, 需要一个完整的亚细胞结构。而且, 由于 PPI 网络的动态特性, 对数据的解释可能会很复杂, 因为单个蛋白质通常是几个不同蛋白质复合体的一部分。
最近开发了接近标记技术, 如 BioID2或 APEX23、4 , 以解决 AP MS 方法的一些局限性。在 BioID, 酶比拉 * (对应于野生类型的大肠杆菌酶的 G115R 变种) 催化形成的不稳定的 biotinyl 安培 (生物放大器), 可以与初级胺反应。相对于野生型酶, 它保留生物安培在其活跃的中心, 比拉 * 释放生物放大器允许其扩散到其邻近的环境。因此, 当融合到一个感兴趣的蛋白质, 并表达在细胞, 近端蛋白可以生物素化在估计范围内 10毫微米5。这些标记的近端蛋白质然后被链亲和素下拉和由 MS 辨认。与 AP MS 相反, BioID 需要融合蛋白的表达。因此, 它只能适用于功能不受标签限制的蛋白质。此外, 标签速度很慢, 通常 6–24 h2,6, 使得检测短寿命蛋白质具有挑战性。然而, 与 AP ms 相比, BioID 提供了几个关键优势: 首先, 它捕捉到了其本地细胞环境中的相互作用;第二, 细胞裂解后分离出标记蛋白而非组装复合物;第三, 链亲和素 pulldowns 允许使用变性缓冲器和苛刻的洗涤条件。因此, 该方法对于检测瞬态或弱交互7或发生在特定和难以隔离亚细胞结构8上的交互更敏感。
然而, 大多数蛋白质通常是较大的复合体的一部分, 可以根据细胞的暗示或需要执行的功能进行改造。因此, 单个蛋白质通常是几个配合物的一部分, 对应于不同的功能单位, 涉及不同的和/或重叠的 PPI。这两种方法都概述了给定蛋白质可能具有的所有关联, 但它们未能解决单个 PPI 的上下文。为了增加后者的分辨率, 我们设计了一种蛋白质片段互补试验 (PCA), 其中两个不活跃的比拉 * (NBirA *, 包含催化领域, CBirA *, 可以被视为重新激活域) 可以当两种相互作用的蛋白质在接近的接近度时, 重新组装成活性酶9。由此产生的分裂-BioID 检测聚焦于接近依赖的 biotinylation 蛋白质, 聚集在一对相互作用的蛋白质, 从而允许识别上下文相关的蛋白质组件。最近, 我们通过解决 miRNA 介导的基因沉默通路9中的两个不同的蛋白质复合体, 证明了 BioID 的杰出分辨力。
总之, 在一个单一和简单的检测, 分裂-BioID 允许发现和具体分配 PPI 到定义的功能单位, 其中一个特定的蛋白质, 提供一个额外的互动蛋白的相应的蛋白质复合体已知。
Protocol
注意: 方法的概述显示在图 1上。
1. 克隆战略的规划
- 选择两个认定相互作用的蛋白质进行测试。
注意: 这两种蛋白质中的每一个都将被融合成一个分裂 BioID 片段: NBirA * 或 CBirA *。作为一个负控制, CBirA * 融合蛋白将测试与 NBirA * 融合到绿色荧光蛋白 (NBirA * GFP)。作为一个额外的控制, NBirA * 融合可以单独测试, 没有同源 CBirA * 融合或结合 CBirA * 融合到一个无关的蛋白质。最好不要使用 CBirA * GFP 作为负控制, 因为它被证明始终如一地导致主要背景, 当结合到任何 NBirA * 融合蛋白9。这种观察的原因可能是 CBirA 的表达水平, 远远高于任何其他 CBirA * 融合蛋白我们已经使用了, 这可能导致显著的 reassociation 与 NBirA * 片段。 -
一旦选择了两种蛋白质, 检查文献以发现两者是否已经成功地标记在功能研究中 (例如, 作为 GFP 标记的融合蛋白)。
- 如果存在此类研究, 请记下标记的位置 (在 N 或 C 终点), 并使用相同的方向标记与 BioID 碎片有关的感兴趣的蛋白质。
- 如果不存在这样的研究, 计划构建在 N 和 C 终点标记的两种蛋白质的编码, 并计划一项测试, 以检测融合蛋白的功能 (例如, 在细胞线上的抢救实验, 耗尽的蛋白质)。
注: 当配对两种融合蛋白时, 使用图 2中描述的质粒, 编码长柔连接器, 融合蛋白 (比拉) 的方向 (在感兴趣的蛋白质的上游或下游融合) 一般无关紧要。.事实上, 使用 FRB 和 FKBP 作为模型蛋白, 它表明, 所有四可能的迭代 (两个碎片在 n 总站, 无论是在 c 总站, 一个在 n 总站和其他 C 总站, 反之亦然) 产量可比 biotinylation9。
- 设计底漆, 以放大码的两个感兴趣的蛋白质克隆成分裂-BioID 质粒。注意翻译框架与比拉的片段相同。如果使用图 2中描述的质粒, 则使用限制酶 PmeI 和 PacI 构造 CBirA * 融合, 以及 MluI * 融合的酶快和 NBirA。
注:图 2中描述的质粒在 F3/FRT 重组点两侧带有双向响应的应答元素。这允许被调控的共同表达在大致相同水平的两种融合蛋白从同一质粒10, 并有可能通过重组介导的盒式交换 (RMCE) 快速构造稳定的细胞系。在这些质粒中, NBirA * 有一个 c-myc 标记, 而 CBirA * 有一个标志标记。当然, 码也可以用本构促进剂在单个质粒上克隆。
关键步骤:在测试两种相互作用的蛋白质时, 建议将 NBirA * 融合到蛋白质1结合 CBirA * 融合到蛋白质 2, NBirA * 融合到蛋白质2结合 CBirA * 融合到蛋白质1。的确, 经常会发现一个迭代比另一种更有效。
2. 将感兴趣基因的码克隆成分裂-BioID 质粒
注: 在这个例子中, 两个可以被标记在 N 终点的蛋白质被考虑。将测试四条件, 并将其与未转染的单元格 (表 1) 进行比较。
- 使用聚合酶链反应 (PCR) 放大码的两种蛋白质, 以适当的引物测试。设计引物, 介绍了 NBirA * 融合蛋白的快和 MluI 限制点, 以及 PmeI * 融合蛋白的 PacI 和 CBirA 点。
注: PCR 可以进行任何商业, 优先校对, DNA 聚合酶。按照标准的协议, 从手册和适应它的熔化温度的底漆和长度的 ORF 将被放大根据制造商的指导方针。 -
亚克隆第一个 ORF
- 消化两种质粒 (约2微克) 和 PCR 扩增 ORF1 (与 NBirA *) 快和 MluI。使用每种酶的 1 ul 进行消化反应, 在1.5 毫升管中与适当数量的 DNA 和反应缓冲剂混合, 在 20 ul 中的总容积为1小时, 37 摄氏度。
- 在1% 琼脂糖泰 DNA 凝胶上运行两种消化样品。用干净的手术刀将与所消化的质粒和 ORF1 相对应的带转移到1.5 毫升管。
- 使用标准的 DNA 提取试剂盒净化两个波段。
- 用标准试剂结扎消化质粒和 ORF1。
- 如果使用材料表中所示的结扎套件, 结扎100的 DNA 含有三至五次摩尔过量插入质粒, 总体积为 4.5 ul。添加 5 ul 2x T4 连接酶缓冲器和 0.5 ul 的 T4 DNA 连接酶从结扎试剂盒。在室温下 (RT) 在1.5 毫升管中进行结扎反应10分钟。
- 转换成标准 DH-5α大肠杆菌主管细胞 (根据井下的方法11编写)。
- 混合 3 ul 的结扎反应与 50 ul 的主管细胞在1.5 毫升管冰和孵化30分钟. 将单元格转换为30-45 秒的42摄氏度, 然后在冰上孵化2分钟. 添加 250 ul 预预热 (37 °c)溶源性汤 (磅) 培养基和板 100 ul 的细胞在预热 (37 °c) 氨苄西林含 LB 琼脂板。在37摄氏度的夜间孵化细胞。
- 第二天, 选择四到六个殖民地和孵化他们在37°c, 180 rpm, 隔夜在3毫升的 LB 培养基含有100μg⋅mL-1氨苄西林在15毫升管。
- 使用标准 DNA MiniPrep 试剂盒将所选菌落中的质粒分离出来。
- 使用卡带2反向底漆 (表 2) 验证该质粒的正确性。
- 一旦发现含有第一个 ORF 的质粒, 亚克隆在该质粒中的第二个 orf 与步骤2.2 相同, 但使用 PmeI 和 PacI 酶。
- 使用卡带1反向底漆 (表 2) 序列生成的质粒。
3. 融合蛋白的检测
注: 以下说明是为双诱导表达质粒 (图 2) 和 HeLa-11ht 细胞, subclonal HeLa-CCL2 细胞系, 稳定地表达反向四环素控制转录激活剂 rtTA-M2 和含有RMCE12的轨迹。这些细胞的生长培养基是 Dulbecco 的改良鹰培养基 (DMEM), 含有10% 无四环素的胎牛血清 (血清)。当使用另一种细胞类型, 确切的播种条件和生长培养基将需要适应。
-
瞬变转染
- 种子细胞在浓度为 1 x 105细胞在2毫升每井六个井板材在转染和孵化细胞隔夜在37°c, 5% CO2在细胞培养孵化器的前一天。
- 在转染日, 除去每一个井的培养基, 并用2毫升的新鲜培养基替换。
- 根据表 1准备四转染反应。为每个井染, 添加6µg 聚乙烯亚胺到3µg 的质粒 DNA 在1.5 毫升无菌管和填充到500µL 与 DMEM 培养基没有血清。
- 在室温下将每个转染的混合体孵化至少5分钟, 然后再加滴明智的每一个井。在37摄氏度、5% CO2上过夜孵化细胞。
-
感应和接近标记
- 在转染后的一天, 去除培养基, 并替换为培养基补充与生物素在50µM, 以刺激 biotinylation 和强力霉肽在 200 ng⋅mL−1 , 以诱导融合蛋白的表达。在37°c, 5% CO2上孵化细胞至少20小时。
- 为了使生物素的库存解决方案, 将生物素分解为 50 mg⋅mL-1 (对应于ca200 毫米) 在2米的氢氧化铵中。一旦它被完全溶解, 稀释它到50毫米在500毫米 Hepes, ph 7.4, 然后调整 ph 值到7.4 与 HCl. 整除并且存放结果1,000x 存货解答在-20 °c。溶解强力霉素在 10 mg⋅mL-1在70% 乙醇和存储在螺钉帽微细在-20 °c 在黑暗中。
- 在转染后的一天, 去除培养基, 并替换为培养基补充与生物素在50µM, 以刺激 biotinylation 和强力霉肽在 200 ng⋅mL−1 , 以诱导融合蛋白的表达。在37°c, 5% CO2上孵化细胞至少20小时。
-
细胞裂解制剂
- 用1毫升的冷 (4 °c) 磷酸盐缓冲生理盐水 (PBS) 清洗细胞。
- 对每个井, 增加100µL 溶解缓冲 (50 毫米三 pH 值 7.4, 150 毫米氯化钠, 2 毫米 EDTA, 0.5% NP-40, 0.5 毫米和蛋白酶抑制剂)。
- 用细胞刮刀和转移到1.5 毫升管来收割细胞。
- 离心样品在 1.4万 x g 10 分钟在4°c 去除细胞碎片。
- 把上清液转移到新鲜的管子上, 放在冰上。
- 用布拉德福德测定蛋白质量。
-
聚丙烯酰胺凝胶电泳 (页) 与西方印迹
- 准备一个 SDS-聚丙烯酰胺凝胶。
- 对于每一个清除的裂解液, 准备一页样本30µg (至少15µg) 蛋白质总共30µL 的 SDS 加载缓冲区。然后在 SDS-聚丙烯酰胺凝胶上加载等量的每个样品 (20 µL/井), 然后进行电泳。
- 电泳后, 使用任何标准协议将分馏蛋白转化为低荧光 PVDF 印迹膜。
注: 为分析蛋白质 biotinylation, 10 分钟的传输时间与 "高兆瓦" 程序的快速转移半干西方印迹装置通常工作良好。 - 转移后, 将5% 块干牛奶中的膜阻挡在 RT 中30分钟。
- 在 2% BSA 和 0.1% Tween-20 的 PBS 中, 用荧光链亲和素共轭稀释1:15,000 在 RT 中孵化30–60分钟的膜。
- 冲洗膜三次, 每10分钟, 与 pbs 含有 0.1% Tween-20, 然后再一次与 pbs。
- 在荧光扫描仪成像系统上扫描膜。
注意: 典型的西方印迹显示在图 3上。上述过程描述了一种基于荧光的检测方法, 但使用链亲和素耦合的光检测技术也同样有效。
4. 蛋白质组学研究的分 BioID
关键注意事项:对于最终的质谱分析, 所有的步骤都要在无角蛋白的条件下进行, 所有的材料和试剂应该尽可能的无角蛋白。
- 瞬变转染
- 在转染前的前一天, 种子三到四 10 cm 板材为每个条件在 8 x 105细胞的集中在10毫升每板, 并且孵化细胞在37°c, 5% CO2在细胞培养孵化器中过夜。
- 第二天, 为每个转染条件准备一个主转染组合: 每条件三个板块, 36 µg 聚乙烯亚胺, 18 µg 质粒 DNA 溶解于900µL 无血清 DMEM。在 RT 中孵化每个主混合至少5分钟。
- 同时, 用新鲜的培养基取代每道菜的培养基, 然后添加300µL 的转染混合物, 使每个盘子都更清爽。
- 在37摄氏度、5% CO2上过夜孵化细胞。
- 感应和接近标记
- 转染后的一天, 把细胞转移到15厘米的盘子里。每道菜, 取出培养基, 用7毫升 PBS 冲洗细胞, 加入1.5 毫升的胰蛋白酶-EDTA 溶液, 在 RT 中孵育5分钟. 添加3.5 毫升的生长培养基并用重悬细胞, 将细胞悬浮液转移到15厘米的盘子里, 填充20毫升的生长培养基。辅以生物素在50µM, 以刺激 biotinylation 和强力霉肽在 200 ng⋅mL−1 (最终浓度), 以诱导融合蛋白的表达。
- 在37°c, 5% CO2上孵化细胞至少20小时。
- 细胞的收获和存贮
- 用 pbs 冲洗两次细胞, 然后在每个盘子中加入1.5 毫升的 pbs, 用刮刀来收割细胞。
- 将所收获的细胞与一个条件相对应, 转移到15毫升管中, 并通过离心 1200 x g、5分钟、4°c 来收获它们。
- 除去上清液和快速冷冻的颗粒在液氮, 然后存储在80°c, 直到进一步处理。
注: 另外, 细胞也可以由 trypsinization, 收获在生长培养基, 转移到15毫升管和洗涤三时间与 PBS 在结冰之前。
- 细胞裂解物的制备
- 并用重悬1毫升的裂解缓冲液中的细胞颗粒 (50 毫米三 pH 7.4, 500 毫米氯化钠, 0.4% SDS, 5 毫米 EDTA, 1 毫米, 1x 完全蛋白酶抑制剂) RT. 通过五克针将细胞10–20乘以十到25笔画。
- 用发音设备油脂实验样品。
注: 在材料表中指示的发音设备中, 可以使用以下程序: 四周期在高强度, 三十年代每周期在冷 (4 °c) 水浴。任何其他发音设备都是合适的, 但参数可能需要相应调整。 - 添加海卫 X-100 到回收的微气泡裂解液达到最终浓度的 2% (通常, 添加 100 ul 20% 海卫 X-100 到 900 ul 微气泡细胞裂解), 然后2.3 毫升50毫米三, pH = 7.4 每1毫升的裂解液, 以调整氯化钠浓度到150毫米之前 binding 链亲和素耦合珠。
关键注意事项:盐浓度越高, 对珠子的粘结效果就越低。 - 将调整后的裂解物分布在1.5 毫升管 (ca 1.1 毫升三管) 中, 并将其离心在 1.6万 x g, 10 分钟, 4°c.
- 将上清液(ca 3.2 毫升) 传输到15毫升管, 并保持 50–100 ul 作为输入材料。
- 测量每个样品的浓度与布拉德福德化验, 并使用相当于3到3.5 毫克蛋白质含量的链亲和素下拉。
- 链亲和素下拉
注意: 下拉过程的概述显示在图 4上。它几乎完全相同的原始协议, 由和同事发布的2。第一次执行下拉时, 可以将流通过和清洗第1-4 的样本放在一旁进行西方污点分析, 以确保程序正常工作 (图 5)。- 对于每个条件, 转移 200 ul 的链亲和素耦合磁珠悬浮到1.5 毫升管。把管子放在磁力架上, 等到珠子粘到管子的侧面 (ca. 1 分钟) 并卸下存储缓冲区。
- 用1毫升平衡缓冲器 (50 毫米三 pH 7–4、150毫米氯化钠、0.05% 海卫 X-100 和1毫米) 轻轻地混合来洗涤珠子。
- 派遣平衡珠在必要数量的管子 (通常当开始与3–3.5 镁蛋白质含量, 裂解物从每个情况可以被派遣在二到四1.5 毫升管子) 并且安置回到磁性架子。
- 移除平衡缓冲器, 并并用重悬从步骤4.4.7 中的相应单元格裂解物相等数量的每组珠子。在4摄氏度的旋转车轮上孵化过夜。
- 在第二天, 把1.5 毫升管放在一个磁性架子上, 等到珠子粘在管子的侧面, 然后把上清液转移到一个15毫升的管子上, 把它标记为流过。
注: 从现在起, 所有步骤都是在室温下执行的, 除非另有说明。 - 并用重悬每管中的珠子, 200 ul 的洗涤缓冲器 1 (2% SDS 在水中), 并结合每组悬浮珠对应一个条件在1.5 毫升管。
- 用1毫升的洗涤缓冲器 1, 在旋转轮上洗两次珠子8分钟。
- 用1毫升洗涤缓冲器 2 (50 毫米 HEPES pH 7.4, 1 毫米 EDTA, 500 毫米氯化钠, 1% 海卫 X-100 和0.1% 钠脱氧) 在旋转轮上洗两次珠子, 8 分钟。
- 用1毫升的洗涤缓冲器 3 (10 毫米三 pH 8, 250 毫米 LiCl, 1 毫米 EDTA, 0.5% NP-40 和0.5% 脱氧) 在旋转轮上洗两次珠子, 8 分钟。
- 用1毫升的洗涤缓冲器 4 (50 毫米三 pH 值 7.4, 50 毫米氯化钠, 0.1% NP-40), 在旋转轮上洗两次珠子8分钟。
- 要确保清洗缓冲液在最后一次清洗步骤后完全移除, 取出大部分上清, 然后向下旋转样品。把它们放回磁力架上, 等到珠子粘到管子的侧面, 然后取出剩余的缓冲器。
- 添加 30 ul 的洗脱缓冲 (10 毫米三 pH 7.4, 2% SDS, 5% β巯基乙醇, 和2毫米生物素) 的珠子。孵育在98°c 15 分钟, 然后立即删除磁性机架上的珠子。
- 将洗脱样品转移到一个新鲜的管, 并储存在-20 °c, 直到进一步处理。
- SDS 页和西部印迹
注: 在质谱分析之前, 建议用 SDS 页和 biotinylation 的方法评估其成功与否。如果没有观察到 biotinylation 模式, 你可能会认为 dox 或生物素没有添加到介质中, 或者两种股票溶液中的一个被破坏。- 对于每个输入样本, 通过将等量的蛋白质样本与适当体积的 3x SDS 加载缓冲器混合在一起, 准备一个页面样本, 总共 28 ul。通过将每个洗脱样品的5µL 与2.5 µL 的 3x SDS 加载缓冲器混合, 准备页面样品。
- 在 SDS-聚丙烯酰胺凝胶上加载样品 (25 µL/井输入, 7 µL/井用于洗脱样品)。按照3.4 节的说明进行电泳和西印迹。
- 如果第一次执行下拉, 通过将每个样品的20µL (输入、流经、洗涤第1-4) 与10µL 3x SDS 加载缓冲器或5µL (洗脱) 与2.5 µL 3x S 混合, 为下拉 (输入、流经、洗涤第1-4、洗脱) 的每一步准备页面样本。DS 加载缓冲区并继续执行步骤 4.6.4 (图 5)。
- MS 分析页面
注: 为了尽量减少潜在的角蛋白污染, 可以使用预制凝胶和商用样品加载缓冲器。- 添加 6.25 ul 的4x 样本缓冲区到 18.75 ul 的每一个洗脱样品, 并运行样品在4–20% 预制 SDS 凝胶, 直到他们迁移到2–3厘米到凝胶。
- 在15厘米的培养皿中, 用胶体考马斯亮蓝色 G250 染色13着色凝胶。
- 使用干净的手术刀为每个样本扣除整个车道, 不包括链亲和素带 (运行在 ca 17 kDa ,图 6), 并将切除带转移到1.5 毫升管。
- 将这些样本发送到蛋白质组设备进行进一步分析。
注意: 典型的 MS 结果可以在参考 9 (图 3和图 7和辅助表) 中看到。整个数据集可在自豪感存储库 (加入编号 PXD005005) 中获得。
Representative Results
为了说明该方法的工作原理, 在分离 miRNA 质粒中克隆了蛋白质 Ago2、TNRC6C 和 Dicer (所有参与 BioID 介导的基因沉默通路) 的开放阅读框架 (码)。Ago2 在 miRNA 诱导的沉默情结 (miRISC) 中与 TNRC6C 交互, 压抑翻译和刺激目标基因14的衰减。在组装 miRISC 之前, Ago2 与 Dicer, 这种酶产生成熟的 miRNAs, 在一个复杂的, 它可能会加载 miRNA15。因此, 拆分 BioID 应用于 Ago2/Dicer 对或 Ago2/TNRC6C 对。对于每对被测试的蛋白质, Ago2 是融合到 NBirA * 或 CBirA * 使用我们的分裂 BioID 质粒 (图 2), Dicer 和 TNRC6C 到相应的同源比拉 * 片段。此外, 每个蛋白质被融合到 CBirA * 和配对的 NBirA * GFP 融合作为一个负控制。这将导致对每对测试的蛋白质 (表 1) 测试四次迭代。
为了测试对被测试的蛋白质的相互作用是否激活了分裂 BioID, 我们遵循了图 1中描述的方案。该质粒被瞬态转染在一个春节系统兼容的 HeLa 细胞线。以强力霉素 (dox) 诱导融合蛋白的表达, 并在生长培养基中加入过量的生物肽刺激 biotinylation。经过20小时的 dox 和生物素的孵化时间, 细胞被裂解和分析的西方印迹使用共轭链亲和素检测生物素化蛋白。在哺乳动物细胞中, untransfected 样品中的共轭链亲和素通常检测到两个主要波段(图 3, 恒星), 并对应于内在生物素化蛋白 (最可能是线粒体羧基酶)。这两个波段都存在于所有样品中, 可以方便地用作内部负荷控制, 因此, 检测一个管家蛋白来控制等量蛋白质含量的负荷是多余的。典型的 BioID/分裂-BioID 实验, 可以观察到的其他主要波段是融合蛋白, 得到自生物素化。即使没有其他生物素化蛋白, 检测 biotinylation 的融合蛋白在这个阶段已经表明, 两个测试的蛋白质相互作用的细胞。在图 3中描述的实验中, 很明显, 具有与 CBirA * 融合成 TNRC6C 或 Dicer 的 NBirA*-Ago2 融合蛋白比相反的组合更有效, 其中 CBirA*-Ago2 与另两个 NBirA 的融合相配对。蛋白质 (图 3, 上部板, 比较车道2-3 的强度到车道 6-7)。此外, 激活是具体的, 因为没有 CBirA * 融合可以激活 NBirA * GFP 控制融合蛋白到可观的水平 (图 3, 比较车道 1, 4-5 到车道8对应于 untransfected 细胞)。因为在我们的质粒, NBirA * 有一个 c-myc 标记和 CBirA * 有一个标志标记 (图 2), 每个融合蛋白的表达水平可以用抗体对这两个标记 (图 3, 底部面板) 进行分析。
当观察到相互作用诱发的 biotinylation 时, 可以放大实验, 并在协议 (图 4) 4 段所示的链亲和素耦合珠上隔离生物素化蛋白。第一次执行隔离时, 净化的所有步骤都可以通过西方印迹 (图 5) 来分析。通常, 绑定到珠子应该是几乎定量的, 几乎没有泄漏通过应该看到在洗涤。前处理质谱样品, 我们建议运行一个西方的印迹, 以确保诱导 biotinylation 工作的预期和融合蛋白表达。融合蛋白的表达缺乏是由于转染效率差或 dox 诱导错误所致。如果融合蛋白表达, 但没有观察到 biotinylation, 检查是否过量的生物素 (50 微米) 实际添加到培养基中, 并认为该股票生物素仍然活跃。当对考马斯染色的蛋白质凝胶 (图 6) 进行洗脱材料分析时, 通常要观察到的最强波段的运行时间约为 17 kDa, 对应单体链亲和素。也可以观察与内源性生物素化蛋白和融合蛋白相对应的带。我们通常将示例车道上的区域链亲和素到加载井(图 6)。该切除带可存储在1.5 毫升管, 并送往质谱设施。另外, 绑定蛋白也可以在链亲和素耦合的珠子上进行胰蛋白酶消化, 而洗脱的消化肽则形成柱状体。我们通常使用 MaxQuant 软件16 (使用大多数默认参数并添加赖氨酸 biotinylation 作为可能的平移后修改, 请参阅参考9以了解更多详细信息和典型 ms 结果), 以分析 ms 原始数据和英仙座套件17为随后的统计分析, 都是免费软件。样本通常在三生物复制中运行。使用无标签量化, 特别是丰富的蛋白质可以确定在控制条件。为了过滤内在生物素化蛋白和非特异标记的蛋白质比拉 * 酶, 我们只考虑蛋白质, 是显著丰富超过六数据集产生的六无关的蛋白质。此外, 我们只考虑在一个分裂 BioID 数据集丰富的命中, 其中 NBirA * 融合蛋白已被 NBirA * GFP 取代。另外, 还提出了一些数据分析策略, 特别是在细胞培养 (SILAC) 中使用氨基酸的稳定同位素标记, 用于定量蛋白质组学18。此外, 已经描述了各种战略, 以直接隔离生物素化肽使用一个链亲和素变弱亲和性的生物素 18, 特殊洗脱条件使用有机溶剂19或生物素特定抗体20,21。虽然不一定导致发现更多的蛋白质, biotinylation 站点的识别增加了对命中的特殊性的信心, 并且在处理交互的拓扑时是有用的。
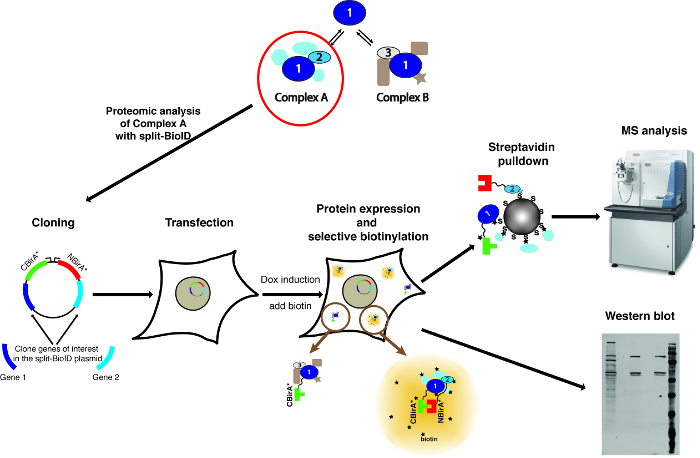
图 1: 拆分 BioID 过程的概述.蛋白质1与蛋白质2相互作用作为复杂 A 的一部分, 或以蛋白质3作为复杂 B 的一部分。为了特别探讨复杂 A 的组成, 分裂 BioID 可以应用于蛋白质1和2。质谱仪的相片是在创造性的共同性归属-分享同样 3.0 Unported 执照并且从 https://commons.wikimedia.org 下载了以 ThermoScientificOrbitrapElite 的文件名. JPG。请单击此处查看此图的较大版本.
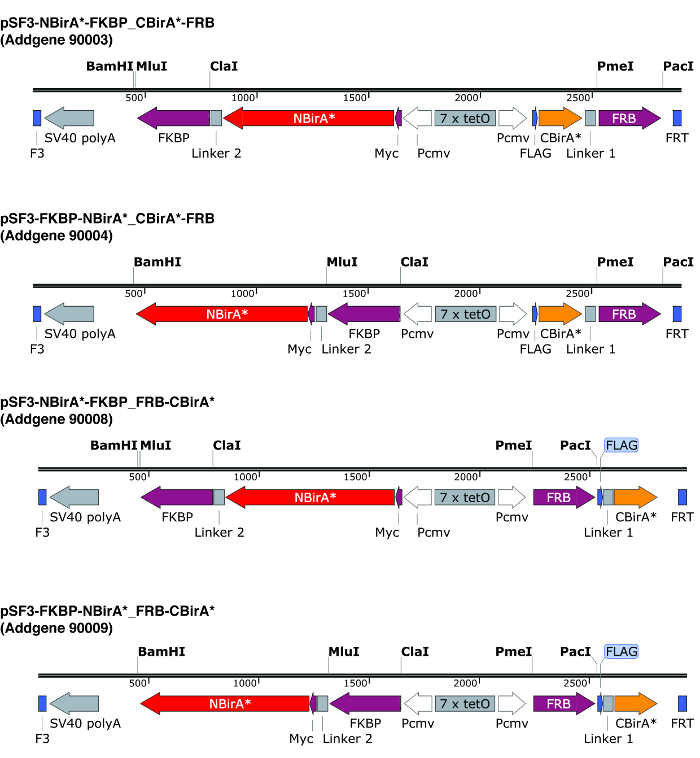
图 2: 分离 BioID 质粒的表达式磁带.我们提供四种质粒, 允许测试 NBirA * 和 CBirA * 融合蛋白的所有组合。质粒和完整的地图可在 addgene.org 下标明的数字。该质粒有一个可响应的元素 (7x tetO), 需要用在与春节表达系统兼容的细胞系中。还请注意, 在所有质粒中, FKBP 和 FRB 的码分别与 NBirA * 和 CBirA * 片段融合在一起。这两种蛋白质只在雷帕霉素存在的情况下相互作用, 因此在这种化学物质9存在或不存在的情况下, 质粒可用于快速测试系统。指定的限制站点是唯一的。请单击此处查看此图的较大版本.
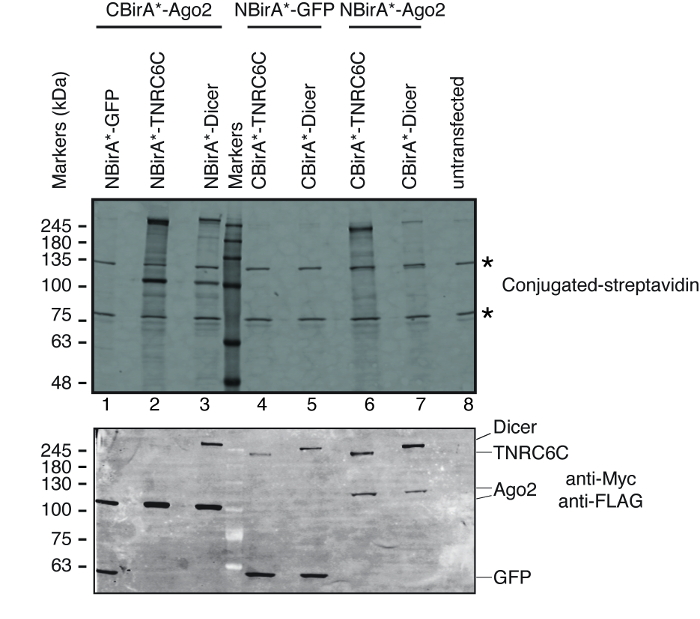
图 3: 用于分裂 BioID 实验的典型的西方印迹.上部面板: 检测生物素化蛋白与荧光标记链亲和素。下面板: 检测融合蛋白的抗 c-myc 和抗旗抗体。测试了两对蛋白质: Ago2/TNRC6C 和 Ago2/Dicer。在车道 2 & 3, Ago2 被追加了 CBirA * 片断。在车道 6 & 7, Ago2 被追加了 NBirA * 片断。当三种蛋白中的任何一个与 NBirA (1、4-5) 结合使用时, 没有发现明显的信号。星表示与内在生物素化蛋白质相对应的带, 可以作为内部负荷控制。此数字是从图 5B Schopp et . 进行调整的。9在创作共同性归属4.0 国际执照之下。请单击此处查看此图的较大版本.
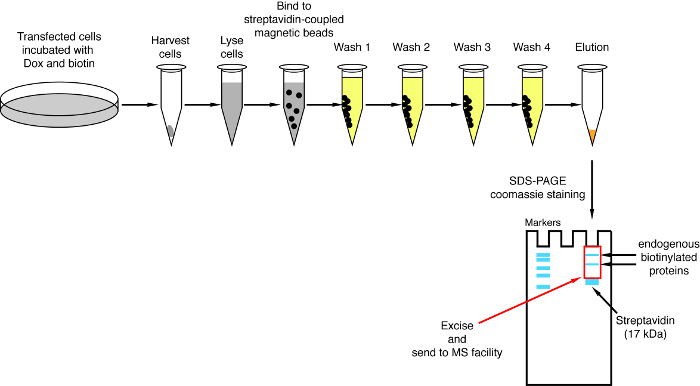
图 4: 链亲和素下拉过程的概述。对生物素化蛋白分离的主要步骤进行了质谱分析。请单击此处查看此图的较大版本.
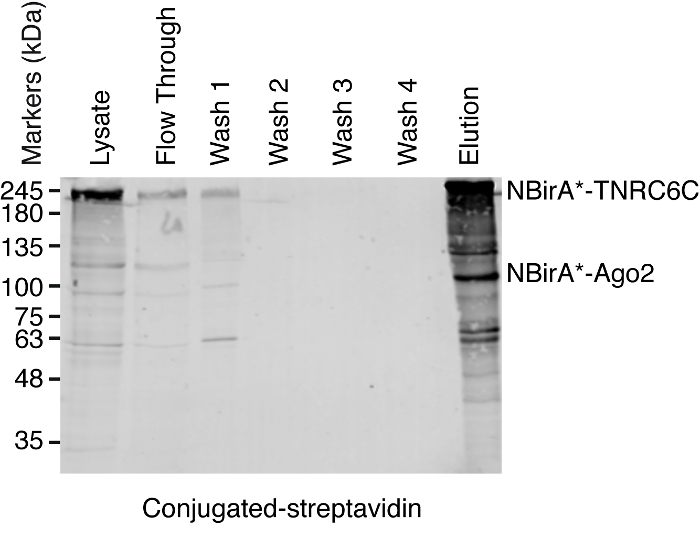
图 5: 链亲和素下拉试验的典型西方印迹。在 SDS-聚丙烯酰胺凝胶上载入每个所示样品的等量体积。随着西方印迹, 生物素化蛋白的检测与酶联链亲和素。表示与 NBirA*-TNRC6C 和 CBirA*-Ago2 对应的带。请单击此处查看此图的较大版本.
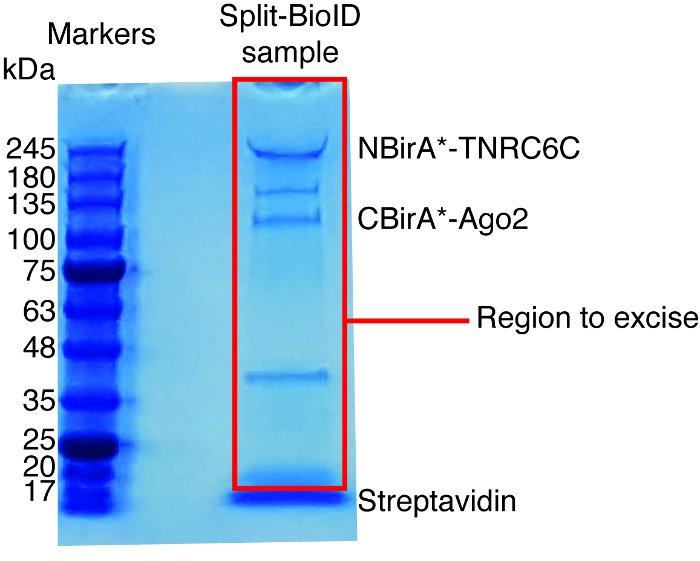
图 6: 用于质谱分析的典型考马斯染色蛋白凝胶.洗脱样品从链亲和素耦合珠被装载了预制蛋白质凝胶并且跑直到样品迁移 2-3 cm。在大约 17 kDa 看到的主要波段是链亲和素。直接在该波段上方的区域被切除并送往质谱设备。表示与 NBirA*-TNRC6C 和 CBirA*-Ago2 对应的带。请单击此处查看此图的较大版本.
| 转染样品 | 条件测试 | ||
| 1 | NBirA*-protein1/CBirA*-protein2 | ||
| 2 | CBirA*-protein1/NBirA*-protein2 | ||
| 3 | NBirA*-GFP/CBirA*-protein1 | ||
| 4 | NBirA*-GFP/CBirA*-protein2 | ||
| 5 | 无转染 | ||
表 1: 通常被测试的条件, 当应用分裂 BioID 到两种蛋白质。
| 测序底漆 | 序列 |
| 卡带1反向底漆 (CBirA * 融合) | TATACTTTCTAGAGAATAGGAAC |
| 卡带2反向底漆 (NBirA * 融合) | GTGGTTTGTCCAAACTCATC |
表 2: 分 BioID 质粒的测序底漆。
Discussion
概述了如何将感兴趣的基因克隆到分裂-BioID 质粒中, 如何检测相互作用诱发的 biotinylation, 以及如何分离生物素化蛋白进行质量谱分析。这里我们描述了一个基于瞬变转染的过程。虽然融合蛋白的表达可以通过添加到培养基中的 dox 量来调节, 但当与内源性相比, 瞬变转染可能导致与某些细胞严重过度表达 tshr 融合蛋白的非同源蛋白表达。同行。这可能会导致相应的 interactomes 和 PPI 的扭曲, 不忠实地反映涉及内源性蛋白质的相互作用。因此, 在瞬态系统建立 BioID 后, 一般都应该构造稳定的细胞系。该质粒与 Flp 介导的重组系统相容, 并将两个感兴趣的基因置于同一种应答元素的调节之下。如果需要, 并且当使用与兼容哺乳动物细胞, 他们允许容易地创造稳定的诱导细胞线。例如, 我们使用 HeLa-EM2-11 线表达 rtTA 四环素激活转录激活剂和一个独特的多弹头基因组位点, 其中四环素介导的基因表达可以严格调节12。使用这一细胞线和 Flp 介导的重组, 仅含有一份转基因的稳定细胞系可以在2/3 周内获得。或者, 你也可以使用目前的基因组编辑技术, 在基因的原生基因座中引入比拉片段。
在任何依赖于标记蛋白质的化验中, 人们都需要考虑所产生的融合蛋白是否起作用。有兴趣的蛋白质被标记的可利用的数据 (例如与 GFP 为成像研究) 和功能上被测试是有用的决定是否应该克隆比拉的片断在上游或下游基因感兴趣。如果没有此类数据, 则应在功能检测中测试 n-末期或 c-末期标记蛋白。例如, 融合蛋白的活性可以在细胞系中进行检测, 其中内源蛋白已被淘汰, 并与野生类型的情况进行比较。如果兴趣的蛋白质容忍 N 和 C 终端标记, 两个应该被测试。事实上, 在 BioID 实验中, 融合蛋白的定位会影响标记22的效率。此外, 我们观察到, 当对一对蛋白质应用分裂 BioID 时, 两种蛋白质中的哪一种被追加到 NBirA * 或 CBirA * 片段也会影响标记9的效率。在分裂-BioID 质粒, 16 氨基酸长甘氨酸/丝氨酸丰富的连接器耦合蛋白质的比拉 * 片段取自另一个 PCA 23, 并为我们的所有相互作用的蛋白质, 我们已经测试到目前为止.然而, 人们应该认为, 一些蛋白质对可能会更好地使用较短或更长的连接器。最后说明中, 博伦组24描述了另一种检测方法。在这种化验中, 比拉 * 是分裂在另一个地点 (E140/Q141) 比我们 (E256/G257)。我们同时测试了两种分离 BioID 的风味, 发现在本协议中描述的 E256/G257 在耦合到两个相互作用的蛋白质9时会导致更强的重新激活。
这种方法的一个一般缺点是标签速度慢。通常, 6 到24小时的孵化时间与生物素是必要的, 以获得可观的 biotinylation6, 排除使用此技术研究动态重塑的蛋白质复合物。虽然这一分析部分解决了这一告诫, 因为它只在两种蛋白质相互作用的情况下激活, 标签的缓慢速度排除了它用于研究对非常动态过程的反应或分析短寿命蛋白质。工程过氧化物酶 APEX2 是已知的促进有效标记近端蛋白在1分钟3。因此, 基于 APEX2 的 PCA 可以解决 BioID 的检测速度慢的局限性。一项原则证明研究描述了这种 split-APEX2 化验25。然而, 虽然 homodimerizing 蛋白是成功地生物素化, 是否也可以用于标签和识别蛋白质组装周围的一对相互作用的蛋白质仍有待证明。最近, 定向进化被用来创建 TurboID 和 miniTurbo, 两个变体的比拉 * 与增强的活动, 允许更短的标签时间窗口, 降低到10分钟26。将 BioID 与这些新变体相适应将进一步将此技术应用于更广泛的应用领域。
Disclosures
作者没有什么可透露的。
Acknowledgements
这项工作由德国研究理事会 (DFG) 通过德国卓越倡议 (CellNetworks DFG-激动 81) 资助, 并由合作研究中心 SFB638 提供部分资金。
Materials
| Name | Company | Catalog Number | Comments |
| Acetic acid (glacial) | VWR | 20104.298 | To make TAE buffer |
| Agarose | Sigma | A9539 | Take TAE-agarose gels for DNA analysis and extraction |
| Ammonia solution 25% NH3 | Bernd Kraft | 6012 | To dissolve biotin |
| Ampicillin | Sigma | A9518 | To select transformed bacteria |
| Bioruptor plus sonification device | Diagenode | B01020001 | Other sonification devices are also ok |
| Biotin | Sigma | B4639 | To be added to the growth medium to stimulate efficient biotinylation |
| Bovine serum albumin fraction V | Carl Roth | 8076 | Used in Western blot buffers and a protein standard in Bradford assays |
| Bradford Ultra reagent | Expedeon | BFU05L | Any other method/kit for protein determination is fine, this particular reagent is more tolerant to detergent than other Bradford reagents |
| Cell scrappers | TPP | 99002 | Any other model is also fine |
| ClaI | New England Biolabs | R0197 | Restriction enzyme for cloning into the split-BioID plasmids (NBirA* fusion) |
| DMEM medium | Sigma | D6046 | If using another cell line, use the corresponding optimal growth medium |
| DNA miniprep kit | Sigma | PLN350 | Any other kit is also fine |
| Doxycycline | Applichem | A2951 | Dox is light sensitive |
| DTT | Applichem | A2948 | Make 1M stock solution, store at -20 °C and always use fresh |
| DyLight 680-conjugated streptavidin | Thermo scientific | 21848 | to use with a LiCor Western blot scanning device |
| Dynabeads MyOne Streptavidin C1 | Invitrogen | 65002 | The C1 beads are not BSA coated which is preferable for downstream MS applications (no leakthrough of BSA in the final elution) |
| EDTA | Applichem | A5097 | Make a 500 mM stock, adjust pH to 8 while dissolving the EDTA powder |
| Ethanol | Sigma | 32205 | Make a 70% stock solution in which Doxycycline can be dissolved at 10 mg.mL-1 |
| Fastgene Gel/PCR DNA Extraction Kit | Nippon Genetics | FG-91302 | Any other kit is also fine |
| HCl 37% | Merck | 1.00317.1000 | To adjust pH of biotin stock solution |
| HEPES | Carl Roth | 6763 | Make a 500 mM stock solution, adjust the pH to 7.4 |
| Immobilon-FL PVDF membrane, 0.45 µm | Millipore | IPFL00010 | This membrane shows minimal autofluorescence when used with a LiCor Western blot scanning device |
| LiCl | Grüssing GmbH | 12083 | Make a 5M stock solution |
| Odyssey CLx imaging system | LI-COR | N/A | To scan Western blot membrane decorated with fluorophore-labeled antibody |
| Linear polyethylenimine (PEI) | Polysciences | 23966-2 | Any other transfection reagent is also fine |
| Milk powder | Carl Roth | T145 | To block Western blot membranes |
| MluI-HF | New England Biolabs | R3198 | Restriction enzyme for cloning into the split-BioID plasmids (NBirA* fusion) |
| Na-deoxycholate | Sigma | 30970 | Make a 10% (w/v) stock solution |
| NaCl | Sigma | 31434 | Make a 5M stock solution |
| NP-40 (Nonidet P40 substitute) | Sigma | 74385 | Make a 20% (v/v) stock solution |
| PacI | New England Biolabs | R0547 | Restriction enzyme for cloning into the split-BioID plasmids (CBirA* fusion) |
| Phosphate buffer saline (PBS) | Sigma | 806552 | To wash cells before scrapping |
| PmeI | New England Biolabs | R0560 | Restriction enzyme for cloning into the split-BioID plasmids (CBirA* fusion) |
| Protease inhibitor cocktail | Roche | 4693132001 | Added to the lysis buffer to prevent protein degradation |
| pSF3-Flag-CBir-FRB_Myc-NBir-FKBP | Addgene | 90003 | Split-BioID plasmid, mediates the co-expression of NBirA*-FKBP and CBirA*-FRB, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-CBir-FRB_FKBP-Myc-Nbir | Addgene | 90004 | Split-BioID plasmid, mediates the co-expression of FKBP-NBirA* and CBirA*-FRB, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-FRB-Cbir_Myc-NBir-FKBP | Addgene | 90008 | Split-BioID plasmid, mediates the co-expression of NBirA*-FKBP and FRB-CBirA*, FKBP and FRB can be replaced by two other ORFs |
| pSF3-Flag-FRB-Cbir_FKBP-Myc-Nbir | Addgene | 90009 | Split-BioID plasmid, mediates the co-expression of FKBP-NBirA* and FRB-CBirA*, FKBP and FRB can be replaced by two other ORFs |
| Q5 High-Fidelity PCR kit | New England Biolabs | E0555S | To amplify the ORF coding for the proteins to be tested. Any other thermostable DNA polymerase is fine. |
| Quick ligation kit | New England Biolabs | M2200S | To ligate DNA fragments into the split-BioID plasmids, any other DNA ligation system is fine. |
| RunBlue 4-20% SDS precast gels | Expedeon | BCG42012 | To use when running samples for MS analysis |
| RunBlue LDS Sample Buffer | Expedeon | NXB31010 | Running buffer for the RunBlue precast gels |
| SDS | Sigma | 5030 | Comes as a 20% stock solution |
| tet-free serum | Biowest | S181T | we use tet-free serum to minimize basal expression of the fusion proteins |
| Trans-Blot Turbo Transfer system | Bio-Rad | 1704150 | High speed Western blotting transfer system, any other transfer system is also fine |
| Tris | Carl Roth | 4855 | Make 1M stock solutions with adequate pH (7.4 and 8) |
| Triton X-100 | Applichem | A4975 | Make a 20% (v/v) stock solution |
| Tween-20 | Carl Roth | 9127 | Used in Western blot buffers, Tween 20 leads to high background fluorescence and should be omitted in the blocking and last wash step |
References
- Scott, D. E., Bayly, A. R., Abell, C., Skidmore, J. Small molecules, big targets: drug discovery faces the protein-protein interaction challenge. Nat Rev Drug Discov. 15 (8), 533-550 (2016).
- Roux, K. J., Kim, D. I., Raida, M., Burke, B. A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells. J Cell Biol. 196 (6), 801-810 (2012).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Rhee, H. W., et al. Proteomic mapping of mitochondria in living cells via spatially restricted enzymatic tagging. Science. 339 (6125), 1328-1331 (2013).
- Kim, D. I., et al. Probing nuclear pore complex architecture with proximity-dependent biotinylation. P Natl Acad Sci USA. 111 (24), E2453-E2461 (2014).
- Kim, D. I., et al. An improved smaller biotin ligase for BioID proximity labeling. Mol Biol Cell. 27 (8), 1188-1196 (2016).
- Lambert, J. P., Tucholska, M., Go, C., Knight, J. D., Gingras, A. C. Proximity biotinylation and affinity purification are complementary approaches for the interactome mapping of chromatin-associated protein complexes. J Proteomics. 118, 81-94 (2015).
- Morriswood, B., et al. Novel bilobe components in Trypanosoma brucei identified using proximity-dependent biotinylation. Eukaryot Cell. 12 (2), 356-367 (2013).
- Schopp, I. M., et al. Split-BioID a conditional proteomics approach to monitor the composition of spatiotemporally defined protein complexes. Nat Commun. 8, (2017).
- Béthune, J., Artus-Revel, C. G., Filipowicz, W. Kinetic analysis reveals successive steps leading to miRNA-mediated silencing in mammalian cells. EMBO Rep. 13 (8), 716-723 (2012).
- Inoue, H., Nojima, H., Okayama, H. High efficiency transformation of Escherichia coli with plasmids. Gene. 96 (1), 23-28 (1990).
- Weidenfeld, I., et al. Inducible expression of coding and inhibitory RNAs from retargetable genomic loci. Nucleic Acids Res. 37 (7), e50 (2009).
- Dyballa, N., Metzger, S. Fast and sensitive colloidal coomassie G-250 staining for proteins in polyacrylamide gels. J Vis Exp. (30), (2009).
- Jonas, S., Izaurralde, E. Towards a molecular understanding of microRNA-mediated gene silencing. Nat Rev Genet. 16 (7), 421-433 (2015).
- MacRae, I. J., Ma, E., Zhou, M., Robinson, C. V., Doudna, J. A. In vitro reconstitution of the human RISC-loading complex. P Natl Acad Sci USA. 105 (2), 512-517 (2008).
- Cox, J., Mann, M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol. 26 (12), 1367-1372 (2008).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nat Methods. 13 (9), 731-740 (2016).
- Opitz, N., et al. Capturing the Asc1p/Receptor for Activated C Kinase 1 (RACK1) Microenvironment at the Head Region of the 40S Ribosome with Quantitative BioID in Yeast. Mol Cell Proteomics. 16 (12), 2199-2218 (2017).
- Mackmull, M. T., et al. Landscape of nuclear transport receptor cargo specificity. Mol Syst Biol. 13 (12), 962 (2017).
- Kim, D. I., et al. BioSITe: A Method for Direct Detection and Quantitation of Site-Specific Biotinylation. J Proteome Res. , (2017).
- Udeshi, N. D., et al. Antibodies to biotin enable large-scale detection of biotinylation sites on proteins. Nat Methods. 14 (12), 1167-1170 (2017).
- Chapat, C., et al. Cap-binding protein 4EHP effects translation silencing by microRNAs. P Natl Acad Sci USA. 114 (21), 5425-5430 (2017).
- Luker, K. E., et al. Kinetics of regulated protein-protein interactions revealed with firefly luciferase complementation imaging in cells and living animals. P Natl Acad Sci USA. 101 (33), 12288-12293 (2004).
- De Munter, S., et al. Split-BioID: a proximity biotinylation assay for dimerization-dependent protein interactions. FEBS Lett. 591 (2), 415-424 (2017).
- Xue, M., et al. Optimizing the fragment complementation of APEX2 for detection of specific protein-protein interactions in live cells. Sci Rep. 7 (1), 12039 (2017).
- Branon, T. C., et al. Directed evolution of TurboID for efficient proximity labeling in living cells and organisms. bioRxiv. , (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved