Method Article
转化、基因组编辑和拟合氮的热带大麻树帕拉斯波尼亚安德森
摘要
寄生虫树是一种快速生长的热带树,属于大麻家族(大麻),可以形成固氮根结节与rhizobium。在这里,我们描述了基于农业细菌-肿瘤介导的稳定转化和CRISPR/Cas9基于基因组编辑的P.andersonii中反向遗传分析的详细方案。
摘要
帕拉斯波尼亚安德森是一种快速生长的热带树,属于大麻家族(大麻)。与另外4个物种一起,它形成了唯一已知的非豆类血统,能够建立氮固定结核共生与rhizobium。豆类和P.安德森尼之间的比较研究可以为根结核形成背后的遗传网络提供有价值的见解。为了便于比较研究,我们最近对P.andersonii基因组进行了测序,并建立了以肿瘤为介导的农业细菌,以及基于CRISPR/Cas9的基因组编辑。在这里,我们提供了一个详细的描述,为P.安德森ii开发的转换和基因组编辑程序。此外,我们描述了种子发芽和共生表型表型表征的过程。使用该协议,可在2-3个月内生成稳定的转基因突变线。T0转基因系的植物体外繁殖允许在A.tumefaciens共同培养后4个月开始出型化实验。因此,该协议只比P.andersonii可用的基于根基因的瞬态农业细菌根转化方法稍长,尽管提供了几个明显的优点。总之,这里描述的程序允许P.andersonii被用作研究模型,旨在了解共生结系以及这种热带树生物学的潜在其他方面。
引言
帕拉斯波尼亚安德森尼是属于大麻家族(大麻)的热带树,原产于巴布亚新几内亚和几个太平洋岛屿1,2,3。连同另外4个寄生虫物种,它代表了唯一能与根瘤建立固氮结核共生的非豆类血统。这种共生症在豆类(Fabaceae)模型中得到了很好的研究,即Medicago截断和莲花的黄斑,从而获得了关于结核形成和功能4的分子遗传特性的详细知识。此外,据证明,根结核共生在豆类是建立在更古老,和广泛的眼膜霉菌共生5。植物基因组比较表明,豆类、帕拉斯波尼亚以及寄生菌的固氮结核共生,以及所谓的含有二氮营养法兰卡细菌的actininhizal植物物种,具有共同的进化起源6,7,8.为了确定被确定为与豆类结核形成有关的基因是否是保守遗传基础的一部分,对非豆类物种的研究是必不可少的。为此,我们建议利用P.andersonii作为比较研究模型,与豆类一起,确定根结核形成和功能背后的核心遗传网络。
P. 安德森尼是一个先驱,可以在火山山的山坡上找到。它每月可以达到45厘米的生长速度,达到10米9的长度。P. 安德森尼树是风授,这是由形成单独的男性和女性花3,10。我们最近对P.andersonii的二倍体基因组(2n = 20;560 Mb/1C)进行了测序和批选,并收集了另外2个寄生虫物种的基因组序列草案;P. 硬地加和P.鲁戈萨6。这揭示了#35,000 P. 安德森尼基因模型,可以聚集在 >20,000 正交组与基因一起从M. 截管,大豆 (甘氨酸最大),阿拉伯拟丽多普西斯 thaliana,林地草莓 (弗拉加里亚维斯卡),特雷马东方,黑棉杨树 (波普斯三乔卡帕) 和紫杉树 (紫杉树大)6。此外,M.truncatula和P.Andersonii之间的转录组比较确定了一组290个假定矫形器,显示两个物种6的结节增强表达模式。这为比较研究提供了极好的资源。
为了研究P.安德森基根和结核的基因功能,建立了11个农业细菌根系介根转化方案。利用该协议,在较短的时间内生成具有转基因根的复合植物。该方法在豆类共生研究12、13、14中也得到了广泛的应用。然而,这种方法的缺点是,只有根被转换,并且每个转基因根代表一个独立的转化事件,导致大量变化。此外,变换是瞬态的,转基因线无法维持。这使得基于根系的根转化不太适合CRISPR/Cas9介导的基因组编辑。此外,Rhizo基因将其根诱导位点(rol)基因转移到植物基因组,该基因组曾经表示干扰激素平衡15。这使得研究植物激素在A.根系转化根中的作用具有挑战性。为了克服这些限制,我们最近开发了一种基于农业细菌的基于肿瘤的转化和P.andersonii10的CRISPR/Cas9介导诱变的突变的规程。
在这里,我们提供了一个详细的描述,基于A.tumefaciens的转换过程和反向遗传学管道开发的P.安德森。此外,我们还为转基因植物的下游处理提供方案,包括研究共生相互作用的测定。使用此处描述的协议,可在 2-3 个月内生成多条转基因线。结合CRISPR/Cas9介导的诱变,这允许高效生成敲除突变线。这些突变线可以在体外10、16、17中植物性繁殖,这允许在转化过程后4个月内生成足够的物质,以开始表型表征特征已启动10。总之,这套程序应该允许任何实验室采用P.andersonii作为研究模型,旨在了解根索比菌和霉菌的关联,以及这种热带树生物学的潜在其他方面。
研究方案
1. 在温室中种植P.安德森尼树
-
Germinate P. 安德森I WU1 种子18.
- 使用新鲜的帕拉斯波尼亚浆果或将干浆果浸入水中2小时,以补水。将浆果挤压在一张纸巾上,或擦在茶筛内部以去除种子。
- 使用商业漂白剂(+4%次氯酸盐)对种子进行消毒15-20分钟,然后用消毒水清洗种子6次。
- 将种子转移到无菌 200 μL PCR 管中。用消毒水填充管子,使种子完全浸没。在运行以下程序的热循环器中孵育管子 10 天:30 个循环(7 °C 4 小时,28 °C 4 小时)。不要使用加热盖,因为这可能会杀死种子。
- 准备 SH-0 板(见表 1)。将种子转移到 SH-0 板,并在 28°C,16 小时:8 小时:夜间孵育。用2层弹性密封箔封闭板,防止在28°C孵育过程中干燥。
- 幼苗发育出第一组真叶(在28°C孵育后约3-4周)后,将幼苗转移到充满商业盆栽土壤的盆中,用半透明的塑料杯覆盖幼苗,以防止干燥。在 28°C 的气候室或温室中放置盆栽,+85% RH,在 16 小时 8 小时:夜间下。
- 1周后,取出半透明的塑料杯。定期浇锅,当树木长得更大时,用肥料补充肥料来维持生长。
2. CRISPR/Cas9介导的P.安德森的构造的克隆
注:标准二进制变换向量可用于P.安德森ii的稳定变换。例如,下面是使用模块化克隆(例如金门)19生成 CRISPR/Cas9 介导诱变的构造的过程。
- 使用具有内置CRISPR设计工具的生物信息学软件,识别感兴趣的基因的引导RNA靶序列。选择位于目标基因编码序列5'端的引导RNA序列,以增加获得完全敲除的机会。一定要通过搜索P.安德森尼基因组6来检查脱靶效果。
注:每个靶基因使用2个sgRNA,最好分开200-300bp。这可能产生缺失,PCR和随后通过胶质电泳识别。 - 生成包含 sgRNA 序列的 1 级金门构造。
- 设计引物以放大每个单独的sgRNA,在以下引物序列中插入N(20)位置的20 bp导序列:5'-TGTGGTTATAtTGN(20) GTTTAGAGCAAAAAAG-3'。
注: 如果导引序列等于 GN(19),则在插入引物序列之前,请移除导引序列 5' 端的 G。 - PCR 扩增了 pICH86966::AtU6p::sgRNA_PDS20,使用步骤 2.2.1 中设计的正向引基和通用反向引基:5' -TGTGGTCTCAGCATGCCATAC-3'。使用高保热热稳定的DNA聚合酶和以下PCR条件:98°C30s;30 个周期(98 °C 表示 10 s;53 °C 表示 20 s;72°C 表示 10 s);72 °C 7分钟, 成功的PCR反应产生165 bp放大素。
- 使用商用PCR纯化试剂盒对PCR放大剂进行柱式纯化。随后,设置金门反应克隆sgRNA背后的阿拉伯拟南芥塔阿图6p小RNA启动子:10 ng的sgRNA PCR放大组,150纳克的pICSL01009::AtU6p 20,60ng适当的1级接受载体载体,2μL的T4加酶缓冲液,牛血清白蛋白(BSA)的0μL0.1%,BaI的0.5μL,T4连带酶的0.5μL,用超纯水填充至20μL。确保所有 sgRNA 都以相同的方向克隆,以防止发夹形成。
- 在运行以下程序的热循环器中孵育反应:37 °C,20 s;26 个循环(37 °C 3 分钟;16 °C 4 分钟);50°C 5分钟;80 °C 5分钟.在含有阿霉素(50mg/L)、X-Gal(200毫克/升)和IPTG(1 mM)的LB培养基21上改变金门对大肠杆菌和板的反应。
注:分别在超纯水和二甲基甲酰胺中制备IPTG和X-Gal的库存溶液。过滤灭菌素和IPTG库存溶液,并将所有库存储存在-20°C。处理二甲基甲酰胺时,请戴上手套。 - 使用商业质粒分离试剂盒选择白色菌落和分离质粒。序列验证分离质粒,然后继续金门2级组装。
- 设计引物以放大每个单独的sgRNA,在以下引物序列中插入N(20)位置的20 bp导序列:5'-TGTGGTTATAtTGN(20) GTTTAGAGCAAAAAAG-3'。
- 组装2级金门结构,实现稳定转型。
- 使用级别 1 AtU6p::sgRNA 构造(根据第 2.2 节生成)以及 pICH47802::NPTII, pICH47742::35Spro:_NLS-aCas9:35Ster,2 级接受器 pICSL4723 和相应的端链接器(见恩格勒等人22)。执行以下反应:使用每个供体载体的±100 fmol和受体载体的±20 fmol,加入2μL的T4连带缓冲液,2μL的0.1%BSA,0.5 μL的BpiI,0.5μL的T4连带,用超纯水填充至20μL。
注: 1 级质粒 pICH47802::NPTII, pICH47742::35Spro::_NLS-aCas9::35Ster需要首先克隆 (参见补充文件 1),如第 2.2 20、22 节所述的 sgRNA ,23. - 孵化反应,如步骤2.2.4,并转化为大肠杆菌。含有卡那霉素的LB介质上的板。第二天,选择白色菌落并分离质粒。通过限制消化分析确定正确的质粒组件。
- 使用级别 1 AtU6p::sgRNA 构造(根据第 2.2 节生成)以及 pICH47802::NPTII, pICH47742::35Spro:_NLS-aCas9:35Ster,2 级接受器 pICSL4723 和相应的端链接器(见恩格勒等人22)。执行以下反应:使用每个供体载体的±100 fmol和受体载体的±20 fmol,加入2μL的T4连带缓冲液,2μL的0.1%BSA,0.5 μL的BpiI,0.5μL的T4连带,用超纯水填充至20μL。
- 将2级结构转换为农业细菌菌株AGL124。
3. 稳定转换P. 安德森ii
- 接种2LB板含有适当的抗生素与A.tumefaciens应变AGL1改变与兴趣结构。在28°C下孵育板2天。
- 从温室种植的树上收获幼枝。每次变换使用约5根长度为5-8厘米的树枝。确保仅使用健康的未感染分支。去除叶子,这样,约1厘米2的叶组织留在每个小叶的末端。丢弃叶子。
- 使用1:1稀释商业漂白剂(稀释后+2%次氯酸盐)对组织进行15分钟的消毒,含有几滴聚糖酸盐20。然后,用高压灭水冲洗组织6次。
注:此步骤以及以下步骤需要在层压柜内执行,以保持组织无菌。 - 在25 mL的渗透介质(见表1)中,从1-2个板中重新悬浮A.tumefaciens细胞,其中含有醋酸酮(20mg/L)和非离子表面活性剂(0.001%v/v),达到+5的光学密度(OD 600)。
注:在70%乙醇中制备醋酸酮溶液,并储存在-20°C。非离子表面活性剂在添加到渗透介质之前需要过滤消毒。 - 将茎和小动物组织切成1厘米长的片断,在A.tumefaciens悬浮液内,从而在两侧产生新的伤口。将组织片留在A.tumefaciens悬浮液中10-30分钟。
- 准备生根介质(见表1),并在高压灭菌后加入醋酸酮(20mg/L)。将纸巾干块放在一块无菌的滤纸上,并将其放在介质上(+10 个外植/板)。在21°C的黑暗下孵育板2天。
注:在添加醋酸酮之前,允许介质冷却至+60°C。 - 2天后,检查板有无真菌或明显的细菌污染(A. tumefaciens以外的细菌)。受污染的板材需要丢弃。
- 准备液体SH-10介质(见表1)。高压灭菌后,添加聚山梨酸酯20(0.01%,v/v)。将组织片转移到含有多糖醇20的SH-10的10mL。在至少10分钟的期间,每2-3分钟轻轻搅拌一次,以洗涤组织。
- 用含有聚糖20的新鲜SH-10再洗两次。这些时候,每个洗涤步骤2-3分钟的孵育时间就足够了。
- 准备生根介质(见表 1)。高压灭菌后,加入西法塞米(300毫克/升)和卡那霉素(50毫克/升)和倒板。对于二次转化(转基因耐卡那霉素抗线转化),应用湿霉素(15mg/L)选择。
- 在无菌滤纸片上干燥组织片。之后,将组织片转移到步骤 3.9 中准备的板中。
- 在 28 °C,16 小时:8 小时:夜间孵育板 7 天。每2天检查板的真菌或细菌污染和过度生长的A.肿瘤。在污染的情况下,将未感染的部件转移到新鲜的盘子中。
- 7天后,将组织片转移到繁殖培养基(见表1),含有西法塞米(300毫克/升)和卡那霉素(50毫克/升)。孵育板在28°C, 16 h:8 h 白天:晚上.每周刷新一次板,直到转基因芽发展。确保只将未感染的组织片转移到新鲜板中。丢弃由A.tumefaciens过度生长的碎片。
- 一旦假定转基因芽的长度为 ±1 厘米,在含有西法酮(300 mg/L) 和卡那霉素 (50 mg/L) 的传播培养培养中独立切割芽和培养它们。为了确保芽代表独立的变换剂,只需从植物的两侧拍摄一次。
- 如步骤5.2所述,植物性地繁殖假定的转基因芽。
4. 假定转基因芽的基因分型
- 设计跨越sgRNA识别站点的引注。要允许PCR大分子测序,请选择引物150-250bp远离sgRNA识别位点。
- 从每个转基因芽切叶尖(±5毫米)进行基因型。此外,收获野生型控制样本。
- 使用步骤 4.1 中设计的引基器和用于直接放大植物样品中的 DNA 的商业试剂盒,执行 50 μL PCR 反应。或者,PCR反应可以使用高保真聚合酶在纯化DNA上进行。
- 在1.5-2%的甘蔗凝胶上分离PCR放大器。
- 分析凝胶电泳的结果。检查产生多个频段(超过 1 个等位)和 PCR 均位放大器的样本,其大小与野生类型不同,这表明存在中型环状体。
- 序列 PCR 增利剂,以识别确切的突变。对于生产单个PCR放大剂的样品,PCR产品可以直接排序。在凝胶电泳后产生1个以上带的样品,或直接测序PCR放大子后呈杂合体,需要首先克隆成钝端克隆载体。随后,对每个样本的多个克隆进行序列,以识别样品中存在的所有可能的等位基因。
- 使测序结果与感兴趣的基因对齐,并检查对齐方式,以检查sgRNA靶点附近的突变。随后,检查这些突变是否产生帧移位。丢弃具有 > 2 等位基因的线和包含帧内突变的行。
- 选择几行进行进一步分析。
- 按照步骤 5.2 中所述传播所选行。
- 当线条开发出若干新芽时,从 #3 叶尖取新样本,并重复步骤 4.3-4.7。确定来自同一行的每个样本和原始 PCR 样本中的突变是否相同。在所有样品中产生相同突变的线是均匀突变的,可用于进一步的实验。丢弃不产生与这些线相同的结果的行是嵌合的。
5. 准备根P.安德森尼植物进行实验
-
启动P.安德森尼的新组织培养线。
- 从健康树上收获开枝芽、幼苗或叶组织。或者,幼苗可用作起始材料。
- 使用1:1稀释商业漂白剂(稀释后+2%次氯酸盐)对组织进行消毒,其中含有几滴聚糖20,15分钟。 之后,使用高压灭菌水冲洗组织6次。
注:此步骤以及以下步骤需要在层压或层交叉流柜内执行,以保持组织无菌。 - 将组织转移到传播介质(见表1)。在 28°C,16 小时:8 小时:夜间用 2 层弹性密封箔和孵育板关闭板。
- 在前 2 周内每隔几天检查一次板,以确保组织没有真菌或细菌污染。
- 将 +10 芽放在新的繁殖介质板上,用 2 层弹性密封箔合上板,从而传播组织。孵育板在28°C, 16 h:8 h 白天:晚上.每 4 周重复此步骤。
- 当芽的长度为 >1 厘米时,在底部切割芽,并将其放置在根茎介质上(参见表 1)。约10芽可以放置在一个根板。将射击的基底尖端插入介质,使位置直立射击。根在28°C,16小时8小时白天:夜间在板孵育后的10-14天出现。
注:不要根植于所有芽,但保留部分用于组织培养传播(参见步骤5.2)。
6. 盆中P.安德森尼植物的点缀
-
准备气管接种。
- 从一个多聚体BOR26的单个菌落中接种10mL的液体YEM介质(见表2),并在28°C孵育2天。
注: M. 多聚物BOR2 是首选,因为它有效地对P. 安德森ii进行分增。然而,其他rhizobium菌株也可用于P.安德森尼的点缀(例如,叶尔巴多尼·埃尔卡尼WUR325,热带红豆杉CIAT89926,27或布雷迪氏菌sp. Kelud2A4)。 - 使用 10 mL 培养基为较大体积的液体 YEM 培养基接种。这种文化的数量取决于需要接种的罐子数量。
- 准备液体 EKM 介质(见表 3,表 4)。在3,500 x g下将细菌培养物离心10分钟,以收获细胞。随后,重新悬浮液体EKM中的细菌颗粒(使用与原始YEM培养物大致相同的体积),并确定光学密度(OD600)。
- 从一个多聚体BOR26的单个菌落中接种10mL的液体YEM介质(见表2),并在28°C孵育2天。
- 对于 +20 罐,准备 3 L 液体 EKM 介质,并在步骤 6.1.3 中制备根瘤悬浮液。达到 OD600 = 0.025。
- 混合 3 L 含有 rhizobia 的 EKM 与 1,250 克的珀尔特。随后,将210克这种混合物加入无菌半透明聚丙烯锅中。或者,使用沙作为结点测定的基板,而不是珍珠石。
- 在每个盆中种植1-3P.安德森尼植物。此外,准备几个装有P.安德森尼植物的罐子,用CRISPR控制结构转换(见补充表1)。称量几个锅,以便能够在实验期间确定水损失。盖住每个锅的底部,以保护根部免受光照射。
- 在气候生长室(28°C,16小时:8小时:晚上)孵育盆,持续4-6周。每周一次,称量几个锅,以确定水损失。如果水损失超过10 mL,补充超纯水以补偿损失。
- 4-6周后,从珍珠岩中清洁根部,并使用双筒望远镜确定结核数,以检查结点效率。
7. 板上的P. 安德森植物的点缀
-
制备玻璃纸膜28.
- 切割玻璃纸膜,以适合一个正方形 12 厘米 x 12 厘米培养皿。将顶部的膜缩短一点,为芽提供生长空间。
- 为了增加玻璃纸膜的渗透性,将EDTA溶液(1g/L)中的膜煮沸20分钟。 之后,用脱矿水冲洗至少6次,以去除EDTA。
注:由于干膜在与水接触时容易起皱,因此将干膜一个接一个地浸入溶液中。 - 将膜水平排列在圆形玻璃板中的一层薄薄的水中。通过高压灭菌两次对膜进行消毒。
- 将1个高压化玻璃纸膜放在一个正方形12 x 12厘米的培养皿上,含有琼脂凝固的EKM介质(见表3,表4)。将两个3周长的根植植物(见第5节)或4周老幼苗(见第1.1节)放在膜顶部。确保只采摘根部有白根尖的幼苗,表明这些根仍在生长。
- 用第二个玻璃纸膜轻轻覆盖根部,形成三明治层。用3层弹性密封箔密封板。用铝箔包裹板的下半部分,以覆盖光照射的根部。
- 在气候生长室(28°C,16小时:8小时:晚上)孵育板3-4周。标记根提示的位置,以跟踪根随时间的增长。
- 如果EKM板由于长期孵育而开始干燥,在细菌接种前几天将植物转移到新鲜的EKM板。
- 如步骤 6.1 所述,准备细菌接种。
- 取出顶部玻璃纸膜,在根部涂上 1 mL 的草本培养物(OD600 = 0.025)。随后,在接种根上放置新的玻璃纸膜。使用铝箔包裹板外,以覆盖光照射的根部。
- 4周后,使用双筒望远镜检查结核数以确定结点效率。
8. 在袋中点缀P. 安德森尼幼苗
- 如第 1.1 节所述,Germinate P. 安德森种子。在囊泡完全出现后(在28°C的SH-0板上约12天),将幼苗转移到袋中。
- 要准备袋,撕开纸芯的折叠部分,并添加 7 mL 的改性 EKM 介质(参见表 3,表 4)。
- 将根放在形成纸芯和袋前塑料片的两张纸之间,插入 1 或 2 个幼苗。
- 通过在袋周围折叠铝箔,保护根部免受光线照射。将袋装在塑料盒中,盖上半透明盖,以保持高湿度。将盒子放在一个气候生长室(28 °C,16 h:8 h 白天:晚上)。
- 通过添加无菌超纯水来补偿水蒸发,使纸芯保持湿润(避免在袋底部站立水)。第一周后,这通常需要每4天加入2-3毫升。
- 如步骤 6.1 所述,准备细菌接种。
- 幼苗在袋中生长10-12天后,用500 μL的根系接种(OD600 = 0.025)。
- 通过时间跟随结核形成。接种后四周,结节可以计数和收获,以确定结点效率。
9. 结核细胞结构分析
- 在含有固定剂的 2 mL 管中收集 10-15 结核(0.1 M 磷酸盐缓冲液中的 5% 谷醛,pH 7.2)。吸尘1/2-1小时,在4°C孵育过夜。在此期间,样品沉入管底。
注:固定溶液可储存在4°C前2-4周使用。使用组织固定剂时,请务必戴上手套。 - 用 0.1 M 磷酸盐缓冲液清洗结节 2x,pH 7.2。在每个洗涤步骤之间间隔 10 分钟。
- 随后,在30%、50%、70%和100%乙醇中孵育,使样品脱水。为确保从样品中取出所有水,请重复 100% 乙醇步骤 3x。在每个脱水步骤之间应用 10 分钟间隔。
- 通过将 1 包硬化剂 I 加入 2.5 mL 的 PEG400 与 100 mL 的 HEMA(2-羟基乙酰丙烯酸酯)基树脂溶液混合,制备聚合混合物 I (PM-I)。搅拌溶液约15分钟,完全溶解硬化剂I。随后,将 PM-I 存储在 -20°C。
- 从步骤 9.3 中取出乙醇。按以下顺序渗透样品:PM-I:100% 乙醇(1:3,v/v)、PM-I:100% 乙醇(1:1,v/v)和 PM-I:100% 乙醇(3:1,v/v)。在RT下孵育每个溶液中的样品1/2-1小时,或直到样品沉入底部。
- 在100%PM-I溶液中,在4°C下孵育样品过夜。
- 将PM-I和硬化剂II混合在15:1(v/v)的比例中制备聚合混合物II。用聚合溶液填充塑料模具,将样品水平定向在模具底部,并盖上一块弹性密封箔。避免形成气泡。
注:当溶液在暴露于RT时开始聚合,尝试在塑料支架中尽快定位样品。聚合在RT过夜孵育后完成,或在37°C下1小时。 - 从步骤 9.7 中取出弹性密封箔盖,并将支架放在聚合样品上。要将支架安装到样品上,请将10 mL的甲基丙烯酸酯基树脂粉末溶解在5 mL的甲基丙烯酸酯树脂溶液中。快速将溶液添加到支架顶部的孔中。
注:在烟机罩中执行聚合步骤(RT时约30分钟)。 - 微孔部分样品厚度为4-5 μm。将显微镜滑块放在58°C的热板上,并在每张幻灯片中加入一大滴水。把各部分放在水的顶部。一旦水蒸发,各部分将粘附在滑道上。
- 浸入 0.05 % (w/v) 托卢丁蓝色 2 分钟,染色滑动。随后,用超纯水冲洗幻灯片3倍。使用明亮的现场显微镜可以观察幻灯片。
10. P. 安德森尼植物的真菌化
-
准备Rhizhag-不规则孢子的接种
- 准备一叠具有以下尺寸(从上到下):210 μm、120 μm 和 36 μm 网格尺寸的聚酯编织过滤器。
- 将商业孢子悬浮液所需量移至聚酯过滤器堆栈上。用 100 mL 的脱矿水冲洗过滤器 3x。孢子保留在 36 μm 过滤器的表面。
注:在层交叉流柜中准备孢子悬浮液,以防止污染。 - 拆卸聚酯堆栈,仅保留 36 μm 过滤器。使用脱盐水重复洗涤步骤至少6次。
- 将过滤器放在培养皿上,将孢子重新悬浮在脱盐水中。使用与步骤 10.1.2 中使用的孢子悬浮液体积相等的水量。通过移液将孢子悬浮液转移到无菌管中。
- 将5滴20μL的孢子悬浮液放在玻璃幻灯片上,并使用明场显微镜计算孢子数量。将孢子计数转换为孢子/mL的比率,并稀释孢子悬浮液,直到达到250孢子/mL。将孢子悬浮液储存在4°C。
- 执行真菌测定。为此,加入800克的高压沙,辅以70 mL的1⁄2-霍格兰中度至无菌半透明聚丙烯锅(见表5-6)。 通过剧烈摇动,将沙和中沙直接混合在锅中。
- 将一个P. 安德森尼植物放在每个锅中,将孢子悬浮液器 1 mL 直接放在P. 安德森尼植物根上。确保包括几个装有P.安德森尼植物的罐子,这些植物是经过CRISPR控制结构转化的(见补充表1)。
- 在气候生长室(28°C,16小时:8小时:晚上)孵育盆,持续6周。
- 从盆中取出植物,用自来水清洗根部,以清除尽可能多的沙。
- 将根切成 1 厘米长的片断,在 90°C 下将根片煮成 10% KOH(w/v)20 分钟。随后,将煮沸的根放在100μm网粒大小的细胞过滤器上,用50 mL的水冲洗3x。
- 在水浴或加热块中,在90°C下,在乳糖醇(300 mL乳酸;300 ml甘油;400 mL脱矿水)中,将0.05%(w/v)试青成根在90°C下染色5分钟。随后,将根转移到30%甘油。根样本可以存储在 RT 中。
- 将 15-25 根碎片放在单个显微镜幻灯片上。加入30%甘油,盖上盖玻璃,然后按压,直到根片变平。使用明亮的场显微镜观察根片段,并评分霉菌殖民化。
注:根据Trouvelot等人29,描述了一种对霉菌进行评分的方法。此方法使用多个类(%F、%M 和 %A),这允许快速估计每个根片段的霉菌殖民化水平和丰度。
结果
P. andersonii trees 可在 28°C 和 +85% 相对湿度的空调温室中生长(图 1A)。在这些条件下,树木在种植后6-9个月开始开花。雌性P.安德森尼花产生浆果,每个都含有一个种子。在成熟过程中,浆果会变色;先从绿色到白色,然后从白色到棕色(图1B)。从成熟的棕色浆果中提取的种子,在10天的温度周期后发芽,在SH-0板上孵育7天(图1C)。发芽的种子继续发育成幼苗,可在+4周后用于实验(图1D)。
我们之前已经表明,小瓣和部分年轻的P.安德森尼茎可以有效地转换使用A.tumefaciens应变AGL1 10。在转化程序开始时,组织外植株与A.tumefaciens在21°C下共同培养2天(图2A)。长期共同栽培导致A.tumefaciens过度殖民化组织外植,因此应加以预防(图2B)。共栽培期后,组织外植被转移到选择性培养,促进转化组织的生长。两到三周后,沿原始伤口表面通常观察到小绿色微卡(图2C)。这些卡利应该继续生长和开发1个或更多假定的转化芽在6-8周后,转换程序已经启动(图2D)。在此阶段,转化效率通常从 ±10-30% 到 +10-30%, 使用从成熟和部分木质树枝取走的组织外枝开始的转化(表 7)。如果从尚未开花的枝头的幼小和快速生长的尖端中取下外植,就可以实现65-75%的转化效率(表7)。偶尔,在未与介质接触的外植的侧面形成白色卡利,因此,不体验卡那霉素选择。这些卡利通常不是转基因的,从这些卡利形成的任何芽一般会漂白和死亡后直接接触含卡那霉素的介质(图2E)。如果转化率低和/或起始材料不理想,组织片可能会变成棕色(图2F),并遭受A.tumefaciens的过度增殖(图2G)。为了防止A. tumefaciens传播和过度生长附近的外植植物,需要定期更新培养基,并且需要清除严重感染的外植。一旦将单个转基因芽置于繁殖介质中,A. tumefaciens的过度增殖一般不再发生(图2H)。转基因芽可以通过体外繁殖成倍增加,在一个月内产生数十次芽(图3A-B)。这些芽可以放置在根培养基上,在+2周后诱导根的形成(图3C-D)。根植植物随后可用于实验。
为了创建敲除突变线,我们使用CRISPR/Cas9介导的诱变。为此,我们使用含有卡那霉素抗基因NPTII的二元载体,由CaMV35S启动子驱动的Cas9编码序列和2个sgRNA每个靶基因,从AtU6p小RNA启动子20表示。图4A提供了用于CRISPR/Cas9介导的P.andersonii的诱变的构造的图形表示。使用这种方法,基因组编辑在+40%的假定转化芽10。为了识别突变线,假定转化的芽在sgRNA靶点使用跨越目标区域的引物进行基因型突变。图 4给出了预期结果的示例。从凝胶电泳后拍摄的照片中可以看出,几个样品会产生与野生类型大小相似的PCR放大子(图4B)。这些植物可能含有小的印贝,不能通过角胶凝胶电泳或保持未经Cas9酶编辑。此外,几个样本产生的条带大小与野生类型不同(例如,图 4B中的行 2、4、7 和 8)。在这些行中,1(行 4、7 和 8)或两者(第 2 行)等位线包含易于可视化的较大内德尔。在PCR增生测序后,揭示了目标位点突变的确切性质。如图4C所示,CRISPR/Cas9突变后,可以获得1-4 bp的小印数,以及较大的缺失。在图 4C中,行 1 的顺序与野生类型的序列相同,表示此行已转义编辑,因此应丢弃。在包含突变的线中,可以识别异体突变体、同源性突变体和双等位突变体(图4C)。然而,异体突变体一般很少10。同源形或双等位敲除突变体可植物性繁殖,以获得足够的材料进行型型分析。由于在 T0代中执行型板分析,因此检查突变线是否可能是嵌合体非常重要。为此,基因分型需要在从每个突变系中抽取的至少3个不同的样本上重复。如果基因分型结果彼此相同,并且原始基因分型样本(例如图 4D中的第 8 行),则该线是均匀突变的,可用于进一步分析。但是,如果基因分型结果在独立样本之间不同(例如,图 4D中的第 4 行),则突变线是嵌合的,需要丢弃。
用M.多尿体BOR2接种P.安德森尼,导致根结节的形成(图5)。如图5A所示,这些结核沿根系分布。P. andersonii的结节呈浅棕色,但很容易根据根组织的形状进行区分(图5B)。在盆中进行接种实验和随后生长4-6周通常会导致±10-30结核的形成(图6A)。接种后4周接种EKM板状P.安德森植物后形成类似数量的结节(图6A)。在袋中,P.安德森的幼苗通常在接种后 5 周形成 #5-15 结核 (图 5C-D, 6A)。为了分析结核细胞结构,可以使用明场显微镜对结核进行分割和观察。图6B显示了一个纵向截面穿过P.安德森尼结核中间的例子。本节显示P. Andersonii结核的中心血管束,其两侧是含有受感染细胞的结核叶(图6B)。
P. 安德森尼植物也可以被霉化。在6周的接种与R.不规则,霉菌的殖民化频率通常达到>80%(图6C)。此时,一般+30%的细胞含有阿布斯库(图6C)。图6D显示了一个P.安德森基根段的代表性图像,其中包含了abuscles。

图 1:A 的代表性图像P. 安德森尼树,种子和幼苗。(A) 在28°C的温室中生长在盆栽土壤中的六个月大的P.安德森尼树。(B) 描绘不同成熟阶段的P.安德森浆果的代表形象。年轻的P.安德森尼浆果(未成熟)将颜色从绿色变为白色,最后在成熟时变成棕色(成熟)。(C) 在 SH-0 介质上孵育 1 周的P. 安德森种子。黑色圆圈表示发芽的幼苗。(D) 在SH-0介质中生长的四周老P.安德森尼幼苗。刻度条等于 25 厘米 (A) 和 1 厘米 (B-D).请点击此处查看此图的较大版本。
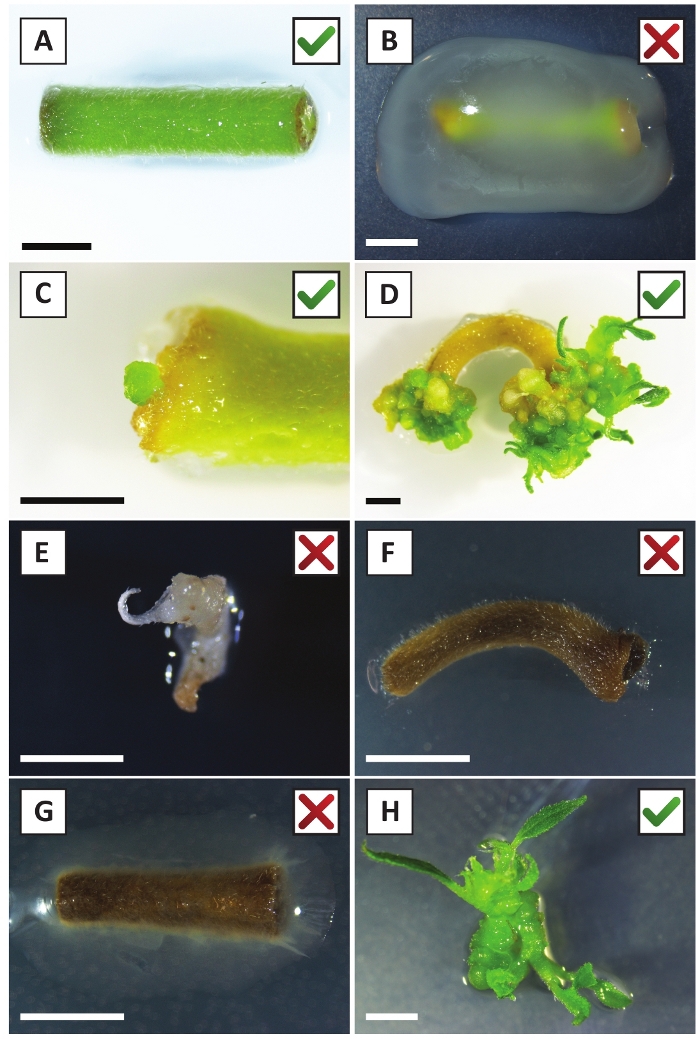
图2:在稳定转化过程的不同阶段,有代表性的外植图像。(A) 与A. tumefaciens共同种植的外植植物。(B) 在改造后的前2周内,由A.tumefaciens过度生长的植物。(C) 转基因微调用在联合栽培后2.5周在外植的伤口部位附近形成。(D) 在共同种植后 6 周内,一种出树的代表性图像,显示了来自(转基因)卡利的芽的出现。(E) 在直接接触含卡那霉素的介质时,会变成白发并最终死亡的拍摄代表图像。这种芽最有可能是非转基因和逃生的卡那霉素选择时,附加到外植。(F) 未成功转化的外植的代表性形象.(G) 代表形象,指由A. tumefaciens过度生长的未成功转化的植物。(H) 单转基因芽在与A.tumefaciens共同培养后8周内在繁殖培养基上生长。比例尺等于2.5毫米。包含绿色复选标记或红色十字的盒子分别表示外植的转化成功或不成功。请点击此处查看此图的较大版本。

图 3:代表图像体外传播。(A) 在传播媒介上生长的芽.这张照片是在板块刷新1周后拍摄的。(B) 在传播媒介上生长的芽。这张照片是在板块刷新4周后拍摄的。(C) 放在生根介质上的新鲜切芽。(D) 在生根培养基孵育2周。请注意根的存在。比例尺等于2.5厘米。请点击这里查看这个数字的较大版本。
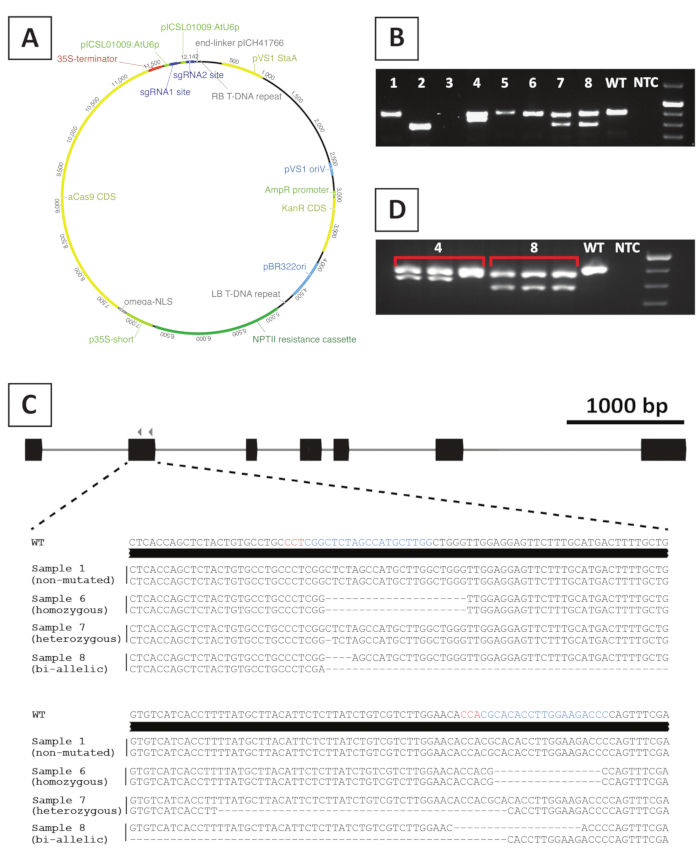
图4:P.安德森I T0转基因CRISPR/Cas9突变线基因分型后的代表性结果。(A) 用于 CRISPR/Cas9 介导的 P. andersonii的二进制向量的代表性地图。(B) 使用跨越sgRNA靶点基的引物对潜在的CRISPR/Cas9突变线进行基于PCR的基因分型后,具有代表性的结果。图中所示为安胶胶胶胶电泳后的图像。从单个转基因线采集的样本由数字表示。野生类型 (WT) 和无模板控件 (NTC) 分别指示包含正负控制的通道。(C) 在CRISPR/Cas9介导的基因编辑后获得的突变等位基因的架构表示。以蓝色和红色突出显示的分别是 sgRNA 目标位点和 PAM 序列。(D) 基于PCR的筛选潜在嵌合突变线后,具有代表性的结果。图中所示为从突变体4和8中采集的3个单独样品的胶质胶电泳后的图像。请注意,转基因突变线4是嵌合的。请点击此处查看此图的较大版本。
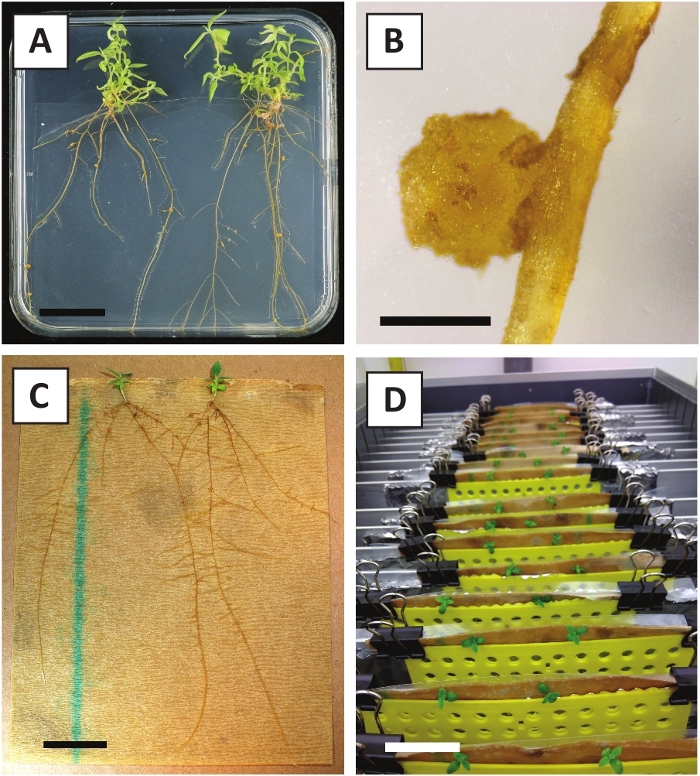
图5:板和袋中结点测定的代表性图像。(A) 在含有琼脂凝固EKM介质的板上点缀,用M.多聚物BOR2接种4周。(B)一个P.安德森根结核的代表形象。该图像是在接种后4周与M.多尿体BOR2。(C) 在含有液体EKM介质的袋中点缀。幼苗接种了5周的凯卢德2A4。(D) 用于袋装的完全设置的代表性图像。刻度条等于 2.5 厘米(A,C),1 毫米 (B) 和 5 厘米 (D).请点击此处查看此图的较大版本。
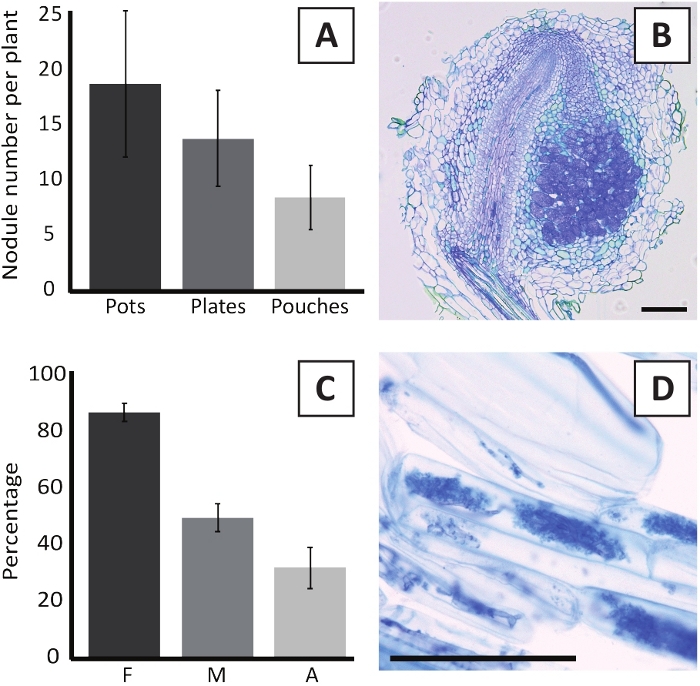
图6:结化和霉菌化测定的代表性结果。(A) 代表条形图显示每个植物在接种后 4 周内与罐中或板上的M. 多尿体BOR2 结合 4 周形成的结核数,并在接种后 5 周内用 Bradyrizobium sp. Kelud2A4 在袋中接种。 数据表示均值 = SD (n = 10)。(B) 在接种后4周与M.多尿体BOR2形成结核的纵向部分的代表性图像。该部分沾有托卢丁蓝色。(C) 代表条形图显示霉菌的定量。根据Trouvelot等人29量化的变量是F,分析的根片段被霉化的频率;M,感染强度;A、总根系中成熟亚数的丰度。在接种后6周用R.不规则(菌株DAOM197198)对肌分化进行量化。数据表示均值 = SD (n = 10)。(D) 在接种后6周,在P.安德森根皮质细胞中存在的成熟肌细胞的代表图像与R.不规则性。比例尺等于75μm。请点击这里查看此图的较大版本。
| 复合 | SH-0 | SH-10 | 传播介质 | 根茎介质 | 渗透介质 |
| SH 基盐介质 | 3.2 克 | 3.2 克 | 3.2 克 | 3.2 克 | 3.2 克 |
| SH-维生素混合物 | 1 克 | 1 克 | 1 克 | 1 克 | 1 克 |
| 蔗糖 | - | 10克 | 20克 | 10克 | 10克 |
| BAP (1 毫克/升) | - | - | 1 mL (4.44 μM) | - | - |
| IBA (1毫克/升) | - | - | 100 μL (0.49 μM) | 1 mL (4.92 μM) | - |
| NAA (1 毫克/升) | - | - | - | 100 μL (0.54 μM) | - |
| 1 M MES pH=5.8 | 3 mL | 3 mL | 3 mL | 3 mL | 3 mL |
| 1 M KOH | 将 pH 调整到 5.8 | 将 pH 调整到 5.8 | 将 pH 调整到 5.8 | 将 pH 调整到 5.8 | 将 pH 调整到 5.8 |
| 大信阿加 | 8 克 | - | 8 克 | 8 克 | - |
表1:基于申克-希尔德布兰特的30种介质的组成,用于生长P.安德森尼幼苗、稳定转化和体外繁殖。在加入液体储存之前,将固体化合物溶解到750 mL的超纯水中。之后,在0.1M KOH中填充完整的介质至1 L.准备BAP、IBA、NAA库存,并储存在-20oC。
| 高压灭菌前: | ||
| 复合 | 每升金额 | 最终浓度 |
| 甘露醇 | 5克 | 27.45 mM |
| 纳-格卢松酸酯 | 5克 | 22.92 mM |
| 酵母提取物 | 0.5 克 | - |
| MgSO4+7H2O | 0.2 克 | 0.81 mMM |
| Nacl | 0.1 克 | 1.71 mM |
| K2HPO4 | 0.5 克 | 2.87 mM |
| 高压灭菌后: | ||
| 复合 | 每升金额 | 最终浓度 |
| 1.5 M CaCl2 | 1 mL | 1.5 mM |
表2:用于生长红豆的酵母-甘醇(YEM)介质的组成。将 pH 调整到 7.0,将超纯水加注到 1 L。要制备琼脂凝固YEM介质,在高压灭菌前加入15克微阿加。
| 高压灭菌前: | |||
| 复合 | 库存集中 | 每升介质的量 | 最终浓度 |
| KH2PO4 | 0.44 米 | 添加 2 mL | 0.88 mM |
| K2HPO4 | 1.03 M | 添加 2 mL | 2.07 mM |
| 500x 微元件库存解决方案 | - | 添加 2 mL | - |
| MES pH=6.6 | 1米 | 添加 3 mL | 3 mM |
| 盐酸 | 1米 | 将 pH 调整到 6.6 | - |
| 超纯水 | - | 填充至 990 mL | - |
| 高压灭菌后: | |||
| 复合 | 库存集中 | 每升介质的量 | 最终浓度 |
| MgSO4+7H2O | 1.04 M | 2 mL | 2.08 mM |
| 娜2SO4 | 0.35米 | 2 mL | 0.70 mM |
| NH4否3 | 0.18 米 | 2 mL | 0.36 mM |
| CaCl2#2H2O | 0.75米 | 2 mL | 1.5 mM |
| 铁(III)-酸盐 | 27 mM | 2 mL | 54 μM |
表3:1 L改进EKM介质31的组成,用于P.安德森尼点测定。表4列出了500x微元件库存溶液的组成。要制备 2% 琼脂凝固 EKM 介质,在高压灭菌前加入 20 克 Daishin Agar。高压灭菌液 MgSO4#7H2O,Na2SO 4,CaCl2+2H2O,和 Fe(III)-酸盐库存进行灭菌。过滤消毒 NH4NO3库存溶液进行灭菌。
| 复合 | 每升金额 | 库存集中 |
| MnSO4 | 500毫克 | 3.31 mM |
| ZnSO4+7H2O | 125毫克 | 0.43 mM |
| CuSO4+5H2O | 125毫克 | 0.83 mM |
| H3BO3 | 125毫克 | 2.02 mM |
| Na2Moo4+2H2O | 50毫克 | 0.21 mM |
表4:用于制备改性EKM介质的500x微元件库存溶液的组成。将微元件库存溶液储存在 4°C。
| 化合物 | 库存集中 | 每升介质的量 | 最终浓度 |
| K2HPO4 | 20 mM | 1 mL | 0.2 mM |
| NH4否3 | 0.28 米 | 10 mL | 2.8 mM |
| MgSO4 | 40 mM | 10 mL | 0.4 mM |
| K2SO4 | 40 mM | 10 mL | 0.4 mM |
| Fe(II)-EDTA | 9 mM | 10 mL | 0.9 mM |
| CaCl2 | 80 mM | 10 mL | 0.8 mM |
| 50x 微元件库存解决方案 | - | 10 mL | - |
表5:用于真菌化测定的1/2-霍格兰32介质的组成。表6列出了50x微元件库存溶液的组成。通过组合 FeSO4+7H2O (9 mM) 和 Na2|EDTA (9 mM) 成 1 种库存溶液,并储存在 4°C。使用 1 M KOH 将介质的 pH 度调整为 6.1,并将超纯水填充到 1 L。
| 化合物 | 每升金额 | 库存集中 |
| H3BO3 | 71.1毫克 | 1.15 mM |
| MnCl2+4H2O | 44.5毫克 | 0.22 mM |
| CuSO4+5H2O | 3.7毫克 | 23.18 μM |
| ZnCl2 | 10.2毫克 | 74.84 μM |
| 纳2MoO4+2H2O | 1.2毫克 | 4.96 μM |
表6:用于制备1⁄2-霍格兰介质的50倍微元件库存溶液的组成。
| 植物外龄 | 转换效率 |
| 年轻 | 69.4 ± 6.2% (n = 2) |
| 成熟 | 18.3 ± 10.2% (n = 15) |
表7:P.安德森的转换效率。在这里,转化效率被定义为至少形成1个转基因的成骨或芽的植物的百分比。转换效率在改造后6周内评分,并描述为均值 = SD.n 表示确定转换效率的转换实验数。
补充文件 1:用于 CRISPR/Cas9 诱变的级别 1 和级别 2 构造的概述。请点击此处下载此文件。
讨论
豆类和远亲的大麻素属帕拉斯波尼亚是植物物种中仅有的两种,能够与固氮的根瘤建立内共生关系,形成根结节。两种包层之间的比较研究与提供对允许这种共生的核心遗传网络的见解高度相关。目前,基因研究主要在豆类中进行;特别是两个模型物种M.截管卡图拉和L.japonicus。为了提供一个额外的实验平台,并方便比较研究与点缀非豆类,我们在这里描述了一个详细的协议,在P.andersonii的稳定转化和反向遗传分析。提出的方案使用T0转基因P.安德森尼线的体外繁殖,允许在A.tumefaciens共同培养后4个月内开始体型分析。这比目前为稳定转化豆类33而建立的协议要快得多。这使得P.安德森ii成为一个有吸引力的研究模型。
此处描述的协议包含几个关键步骤。第一个问题涉及种子发芽。为了准备P.安德森子种子发芽,种子需要从浆果中分离出来。这是通过在一张纸巾上或茶筛内部摩擦浆果来完成的。需要轻轻地执行此步骤,以防止损坏种子涂层。如果种子涂层损坏,漂白剂可能在灭菌过程中进入种子,从而降低种子的生存能力。为了打破种子休眠状态,种子要经过10天的温度循环。然而,尽管这种治疗,发芽不完全同步。一般来说,第一批种子在7天后出现,但其他种子可能需要几天时间才能发芽。
改造过程中的关键点涉及起始材料的选择和共栽培步骤的持续时间。为了达到有效的转化,最好使用非无菌温室种植植物的健康和年轻的茎或小瓣作为起始材料。为了诱导幼枝的生长,建议每2-3个月修剪一次寄生虫树,每年更新一次树木。此外,共同培养步骤只需执行 2 天。长期共同栽培可促进A.tumefaciens对组织外植的过度殖民化,并普遍降低转化效率。为了防止A.tumefaciens的过度殖民化,定期刷新种植外植植物的盘子也很重要。如果确实发生过度殖民化,可以清洗组织外植(见第3.8节),以去除A.tumefaciens细胞。我们建议在用于洗涤的SH-10溶液中加入漂白剂(最终浓度:+2%次氯酸盐)。需要注意的是,这个额外的洗涤步骤可能不适用于严重感染的外植(图2B)。如果使用CRISPR/Cas9构造的转化只产生有限数量的假定转化芽,或者如果特定基因的突变会导致再生问题,则建议将空载体控制结构作为正控制。最后,必须确保选择的所有转基因线都是由独立的T-DNA整合事件产生的。因此,我们指示只从植物的两侧只采取一个单一的假定转基因射击。但是,我们意识到这减少了独立行的潜在数量。如果需要许多线,研究人员可以决定从原始外植分离假定变换的卡利,当这些卡里是+2毫米的大小和培养这些卡利独立。这样,可以从每个外植株中分离出多条线,从而增加潜在的转基因线数。
在目前的协议中,P.安德森的转基因线通过体外繁殖以植物方式繁殖。这样做的好处是,许多转基因植物可以在相对较短的时间段内产生。但是,此方法也有几个限制。首先,通过体外繁殖维持T0转基因线是劳动密集型的,可能导致不必要的遗传或表观遗传改变34,35。其次,T0线仍然包含T-DNA的副本,包括抗生素耐药性盒。这将限制可能的重新转换的数量,因为每次重新转换都需要不同的选择标记。目前,我们只测试了使用卡那霉素或湿霉素选择的转化(未显示数据)。此外,在T0转基因线中存在Cas9编码序列和sgRNA使补充研究复杂化。补充性检测是可能的,但需要sgRNA靶点突变,以便防止补体结构的基因编辑。第三,使用 T0线的缺点是 CRISPR/Cas9 突变体可能是嵌合体。为了防止对嵌合突变系进行型板分析,我们建议在至少3种不同的芽体外繁殖后重复基因分型分析。虽然,使用此处描述的协议获得的嵌合突变体的数量是有限的,它们偶尔被观察到10。为了克服使用T0线的局限性,P.安德森二突变线可以生成传播。P. 安德森尼的树木是有毒的和风授2。这意味着,每个转基因线需要纵,这样,男性和女性的花朵产生在一个个人,并随后种植,这样,交叉授粉不会发生。由于P.andersonii是一种快速生长的树,它需要大量的空间在热带温室(28°C,约85%的相对湿度)。因此,尽管在技术上是可能的,但P.安德森尼转基因线的生成传播在后勤上具有挑战性。
在协议部分,我们描述了P.安德森一的3种点缀方法。板和袋系统的优点是,根很容易获得,这可能允许细菌的点接种和随后结核的形成随着时间的推移。然而,板材系统相当劳动密集型,因此不太适合进行大规模的结点实验。袋系统的一个缺点是很难防止真菌污染。袋不是无菌的,因此真菌生长经常在袋的上半部分观察到。然而,这不会影响P.安德森生长,因此不会干扰结结测定。此外,袋系统仅适用于幼苗。尽管进行了几次尝试,我们还是无法种植通过体外繁殖在袋中获得的植物。
与现有的基于根系的根转化方法11相比,这里描述的P.andersonii反向遗传学管道提供了实质性的改进。使用所述程序,可以有效地生成稳定的转基因线,并通过体外繁殖保持。相反,根茎转化是短暂的,只会导致转基因根的形成。由于每个转基因根由独立转化产生,基于根系基因转化的A.根基因转化具有大量型型变异。这种变化在稳定线的情况下要小得多,尽管体外传播也会产生某种程度的变异。由于这种减少的变异和每个稳定线可以表型的多个植物,与A.根茎转化根相比,稳定线更适合定量测定。此外,稳定的转化不依赖于A.根诱导位点(罗尔)的引入,影响内源激素平衡15。因此,与A.根系转化根相比,稳定线更适合对参与激素平衡的基因进行反向遗传分析。P. andersonii作为研究模型的一个更普遍的优势是,它最近没有经历全基因组复制(WGD)。豆类帕皮利奥尼亚亚家族,其中包括模型豆类M.truncatula和L.japonicus,以及萨拉卡凯(订单马尔皮希亚莱斯),其中包括模型树波普卢斯三乔卡帕经验丰富的WGDs#65百万年前36,37。这些WGD产生的许多寄生基因拷贝被保存在M.truncatula,L.japonicus和P.trichocarpa37,38,39的基因组中,这创造了冗余,这可能会使反向遗传分析复杂化。由于P.安德森未经历最近的WGD,对P.安德森尼的反向基因分析可能较少受到寄生基因副本冗余功能的影响。
综合起来,我们提供了一个详细的方案,反向遗传分析在P.安德森ii。使用该协议,可以在2-3个月的10的时间内有效地生成单突变线。该协议可以扩展,通过同时针对不同基因的sgRNA的多路复用来产生更高阶的突变体,如其他植物物种40、41、42所示。此外,此处描述的稳定转换过程不仅限于 CRISPR/Cas9 基因靶向,但也可用于引入其他类型的构造(例如,用于启动器-报告器检测、异位表达或转-补充)。我们建立了P.andersonii作为一种比较研究模型,以研究与固氮性rhizobia或内霉菌的共生共生。然而,这里描述的协议也提供了工具,研究该热带树的生物学的其他方面,如木材形成,双性花的发展或卡纳巴塞特异性二次代谢物的生物合成。
披露声明
作者没有什么可透露的。
致谢
作者们感谢马克·尤尔斯、索菲恩·卡蒙和西尔维斯特·马里隆内特通过Addgene数据库提供金门克隆部件。此外,我们要感谢E.詹姆斯,P.哈多巴和T.J.希根斯为P.安德森尼种子。这项工作得到了荷兰科学研究组织(NWO-VICI赠款865.13.001;865.13.001;荷兰科学研究组织,第865.13.001年,第3001次,第3次,第3次,第300000名,荷兰科学研究组织,第310000名,NWO-开放竞赛补助金819.01.007)和印度尼西亚共和国研究、技术和高等教育部(RISET-PRO赠款8245-ID)。
材料
| Name | Company | Catalog Number | Comments |
| Sigma-Aldrich | N0640 | NAA | |
| Duchefa Biochemie | M1503.0250 | MES | |
| Sigma-Aldrich | D134406 | Acetosyringone | |
| Duchefa Biochemie | X1402.1000 | X-Gal | |
| Merck | 101236 | For nucleic acid electrophoresis gel | |
| - | - | Pouches box material, hangers | |
| Merck | 101188 | NH4NO3 | |
| Sigma-Aldrich | B3408-1G | BAP | |
| Merck | 100156 | H3BO3 | |
| Thermo-Fisher | ER1011 | Used as restriction enzyme in Golden Gate cloning assembly | |
| Thermo-Fisher | 15561020 | Used in Golden Gate cloning assembly | |
| Merck | 137101 | CaCl2·2H2O | |
| Duchefa Biochemie | C0111.0025 | C16H16N5O7S2Na | |
| Thermo-Fisher | K1231 | Used for cloning the blunt-ended PCR amplicons in genotyping procedure | |
| Agronutrition | AP2011 | Containing Rhizophagus irregularis DAOM 197198 (1,000 spores/mL), used for mychorrization assay | |
| Merck | 102790 | CuSO4·5H2O | |
| Duchefa Biochemie | D1004.1000 | Used for plant tissue culture agar-based medium | |
| Merck | 105101 | K2HPO4 | |
| VWR Chemicals | 20302.293 | Na2·EDTA | |
| Duchefa Biochemie | M0803.1000 | C6H14O6 | |
| Thermo-Fisher | ER0291 | Used as restriction enzyme in Golden Gate cloning assembly | |
| Merck | 100983 | C2H5OH | |
| VWR Chemicals | BDH9232-500G | EDTA | |
| Sigma-Aldrich | Z377600-1PAK | Cellophane membrane | |
| Biomatters, Ltd. | R9 or higher | Bioinformatics software for in silico cloning and designing of sgRNAs | |
| Mega International | - | Technical information at https://mega-international.com/tech-info/ | |
| Sigma-Aldrich | 65882 | Used for fixating nodule tissues | |
| VWR Chemicals | 24385.295 | - | |
| Vink | 219341 | Pouches box material, bottom part | |
| Leica Biosystems | 14702218311 | Used as a template for plastic embedding | |
| Merck | 100317 | HCl | |
| Sigma-Aldrich | I5386-1G | IBA | |
| Merck | 103862 | C6H5FeO7 | |
| Merck | 103965 | FeSO4O·7H2O | |
| Duchefa Biochemie | I1401.0005 | IPTG | |
| Duchefa Biochemie | K0126.0010 | ||
| Sigma-Aldrich | L2000 | ||
| Merck | 105886 | MgSO4O·7H2O | |
| Merck | 105934 | MnCl2·4H2O | |
| Merck | 102786 | MnSO4O | |
| Duchefa Biochemie | M1002.1000 | Used for bacterial culture agar-based medium | |
| Manutan | 92007687 | Pouches material | |
| Paraxisdienst | 130774 | Elastic sealing foil | |
| Pull Rhenen | Agra-Perlite No.3 | Used as growing substrate in pots for nodulation assay | |
| VWR Chemicals | 391-0581 | Used as container for cellophane membranes | |
| Thermo-Fisher | F130WH | For genotyping transgenic lines | |
| Addgene | 50337 | Level 0 terminator, 3'UTR, 35s (Cauliflower Mosaic Virus) | |
| Addgene | 48017 | End-link 2 for assembling 2 level one part into a level 2 acceptor | |
| Addgene | 48018 | End-link 3 for assembling 3 level one part into a level 2 acceptor | |
| Addgene | 48001 | Level 1 acceptor. Position 5. Forward orientation | |
| Addgene | 48007 | Level 1 Acceptor. Position 1. Reverse orientation | |
| Addgene | 50268 | Level 0 promoter (0.4 kb), 35s (Cauliflower Mosaic Virus) + 5'UTR, Ω (Tobacco Mosaic Virus) | |
| Addgene | 46966 | Used for designing CRISPR/Cas9 module | |
| Addgene | 46968 | Used for designing CRISPR/Cas9 module | |
| Addgene | 50334 | Level 0 Kanamycin/Neomycin/Paromomycin resistance cassette | |
| Topzeven | - | Used as filters for washing spore suspension | |
| Sigma-Aldrich | 8.17003 | PEG400 | |
| Duchefa Biochemie | E1674.0001 | Pots to grow Parasponia plantlets/seedlings | |
| Merck | 104871 | KH2PO4 | |
| Merck | 105033 | KOH | |
| Merck | 105153 | K2SO4O | |
| Van Leusden b.v. | - | Used as growing substrate for mychorrhization assay | |
| Duchefa Biochemie | S0225.0050 | SH-basal salt medium | |
| Duchefa Biochemie | S0411.0250 | SH-vitamin mixture | |
| Lehle Seeds | VIS-02 | Used as non-ionic surfactant in the washing step of stable transformation | |
| Merck | 137017 | NaCl | |
| VWR Chemicals | 89230-072 | C6H11NaO7 | |
| Merck | 106521 | Na2MoO4·2H2O | |
| Merck | 106574 | Na2HPO4·7H2O | |
| Merck | 567549 | NaH2PO4·H2O | |
| Sigma-Aldrich | 239313 | Na2SO4O | |
| Duchefa Biochemie | S0809.5000 | C12H22O11 | |
| Thermo-Fisher | B69 | Used in Golden Gate cloning assembly | |
| Thermo-Fisher | EL0013 | Used in Golden Gate cloning assembly | |
| Kulzer-Mitsui Chemicals Group | 64708806 | Methyl methacrylate-based resin powder | |
| Kulzer-Mitsui Chemicals Group | 64709003 | HEMA (2-hydroxyethyl methacrylate)-based resin solution | |
| Kulzer-Mitsui Chemicals Group | 66022678 | Methyl methacrylate-based resin solution | |
| Merck | 1159300025 | ||
| Acros | 189350250 | ||
| VWR Chemicals | 663684B | Polysorbate 20 | |
| Stout Perspex | - | pouches box material, lid | |
| Duchefa Biochemie | Y1333.1000 | ||
| Merck | 108816 | ZnCl2 | |
| Alfa Aesar | 33399 | ZnSO4O·7H2O |
参考文献
- Clason, E. W. THE VEGETATION OF THE UPPER-BADAK REGION OF MOUNT KELUT (EAST JAVA). Bulletin du Jardin Botanique de Buitenzorg. Serie III, 509-518 (1936).
- Soepadmo, E. Ulmaceae. Flora Malesiana-Series 1, Spermatophyta. 8, 31-76 (1974).
- Becking, J. H. The Rhizobium symbiosis of the nonlegume Parasponia. Biological Nitrogen Fixation. , 497-559 (1992).
- Oldroyd, G. E. D. Speak, friend, and enter: signalling systems that promote beneficial symbiotic associations in plants. Nature Reviews Microbiology. 11, 252-263 (2013).
- Gutjahr, C., Parniske, M. Cell and developmental biology of arbuscular mycorrhiza symbiosis. Annual Review of Cell and Developmental Biology. 29, 593-617 (2013).
- van Velzen, R., et al. Comparative genomics of the nonlegume Parasponia reveals insights into evolution of nitrogen-fixing rhizobium symbioses. Proceedings of the National Academy of Sciences of the United States of America. 115, E4700-E4709 (2018).
- van Velzen, R., Doyle, J. J., Geurts, R. A Resurrected Scenario: Single Gain and Massive Loss of Nitrogen-Fixing Nodulation. Trends in Plant Science. 24, 49-57 (2019).
- Griesmann, M., et al. Phylogenomics reveals multiple losses of the nitrogen-fixing root nodule symbiosis. Science. 1743, eaat1743 (2018).
- Becking, J. H. Root-Nodule Symbiosis Between Rhizobium And Parasponia (Ulmaceae). Plant and Soil. 51, 289-296 (1979).
- van Zeijl, A., et al. CRISPR/Cas9-Mediated Mutagenesis of Four Putative Symbiosis Genes of the Tropical Tree Parasponia andersonii Reveals Novel Phenotypes. Frontiers in Plant Science. 9, 284 (2018).
- Cao, Q., et al. Efficiency of Agrobacterium rhizogenes–mediated root transformation of Parasponia and Trema is temperature dependent. Plant Growth Regulation. 68, 459-465 (2012).
- Limpens, E., et al. RNA interference in Agrobacterium rhizogenes-transformed roots of Arabidopsis and Medicago truncatula. Journal of Experimental Botany. 55, 983-992 (2004).
- Boisson-Dernier, A., et al. Agrobacterium rhizogenes-Transformed Roots of Medicago truncatula for the Study of Nitrogen-Fixing and Endomycorrhizal Symbiotic Associations. Molecular Plant-Microbe Interactions. 14, 695-700 (2001).
- Kumagai, H., Kouchi, H. Gene Silencing by Expression of Hairpin RNA in Lotus japonicus Roots and Root Nodules. Molecular Plant-Microbe Interactions. 16, 663-668 (2003).
- Nilsson, O., Olsson, O. Getting to the root: The role of the Agrobacterium rhizogenes rol genes in the formation of hairy roots. Physiologia Plantarum. 100, 463-473 (1997).
- Davey, M. R., et al. Effective Nodulation of Micro-Propagated Shoots of the Non-Legume Parasponia andersonii by Bradyrhizobium. Journal of Experimental Botany. 44, 863-867 (1993).
- Webster, G., Poulton, P. R., Cocking, E. C., Davey, M. R. The nodulation of micro-propagated plants of Parasponia andersonii by tropical legume rhizobia. Journal of Experimental Botany. 46, 1131-1137 (1995).
- Op den Camp, R., et al. LysM-type mycorrhizal receptor recruited for rhizobium symbiosis in nonlegume Parasponia. Science. 331, 909-912 (2011).
- Weber, E., Engler, C., Gruetzner, R., Werner, S., Marillonnet, S. A Modular Cloning System for Standardized Assembly of Multigene Constructs. PLOS ONE. 6, e16765 (2011).
- Nekrasov, V., Staskawicz, B. J., Weigel, D., Jones, J. D. G., Kamoun, S. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease. Nature Biotechnology. 31, 691-693 (2013).
- Bertani, G. Studies On Lysogenesis. I. The Mode Of Phage Liberation By Lysogenic Escherichia Coli. Journal of Bacteriology. 62, 293-300 (1951).
- Engler, C., et al. A Golden Gate modular cloning toolbox for plants. ACS Synthetic Biology. 3, 839-843 (2014).
- Fauser, F., Schiml, S., Puchta, H. Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering in Arabidopsis thaliana. The Plant Journal. 79, 348-359 (2014).
- Lazo, G. R., Stein, P. A., Ludwig, R. A. A DNA Transformation-Competent Arabidopsis Genomic Library in Agrobacterium. Biotechnology. 9, 963-967 (1991).
- Op den Camp, R. H. M., et al. N Nonlegume Parasponia andersonii Deploys A Broad Rhizobium Host Range Strategy Resulting in Largely Variable Symbiotic Effectivenes. Molecular Plant-Microbe Interactions. 25, 954-963 (2012).
- Graham, P. H., Viteri, S. E., Mackie, F., Vargas, A. T., Palacios, A. Variation in acid soil tolerance among strains of Rhizobium phaseoli. Field Crops Research. 5, 121-128 (1982).
- Martinez-Romero, E., et al. Rhizobium tropici, A Novel Species Nodulating Phaseolus vulgaris L. Beans and Leucaena sp. Trees. International Journal of Systematic and Evolutionary Microbiology. 41, 417-421 (1991).
- Felten, J., et al. The Ectomycorrhizal Fungus Laccaria bicolor Stimulates Lateral Root Formation in Poplar and Arabidopsis through Auxin Transport and Signaling. Plant Physiology. 151, 1991-2005 (1991).
- Trouvelot, A., Kough, J. L., Gianinazzi-Pearson, V. Mesure du taux de mycorhization VA d’un systeme radiculaire. Recherche de methods d’estimation ayant une signification fonctionnelle. Aspects Physiologiques et Genetiques des Mycorhizes. , 217-221 (1986).
- Schenk, R. U., Hildebrandt, A. C. Medium and techniques for induction and growth of monocotyledonous and dicotyledonous plant cell cultures. Canadian Journal of Botany. 50, 199-204 (1972).
- Becking, J. H. The Parasponia parviflora - Rhizobium symbiosis. Host specificity, growth and nitrogen fixation under various conditions. Plant and Soil. 75, 309-342 (1983).
- Hoagland, D. R., Arnon Revised, D. I., Arnon, D. I. The Water-Culture Method for Growing Plants without Soil. Circular California Agricultural Experiment Station. 347, 1-32 (1950).
- Wang, K. . Methods in Molecular Biology: Agrobacterium Protocols. 1, (2015).
- Smulders, M. J. M., de Klerk, G. J. Epigenetics in plant tissue culture. Plant Growth Regulation. 63, 137-146 (2011).
- Larkin, P. J., Scowcroft, W. R. Somaclonal variation — a novel source of variability from cell cultures for plant improvement. Theoretical and Applied Genetics. 60, 197-214 (1981).
- Cannon, S. B., et al. Legume genome evolution viewed through the Medicago truncatula and Lotus japonicus genomes. Proceedings of the National Academy of Sciences of the United States of America. 103, 14959-14964 (2006).
- Tuskan, G. A., et al. The Genome of Black Cottonwood, Populus trichocarpa (Torr. & Gray). Science. 313, 1596-1604 (2006).
- Young, N. D., et al. The Medicago genome provides insight into the evolution of rhizobial symbioses. Nature. 480, 520 (2011).
- Sato, S., et al. Genome Structure of the Legume, Lotus japonicus. DNA Research. 15, 227-239 (2008).
- Xing, H. L., et al. A CRISPR/Cas9 toolkit for multiplex genome editing in plants. BMC Plant Biology. 14, 327 (2014).
- Lowder, L. G., et al. A CRISPR/Cas9 Toolbox for Multiplexed Plant Genome Editing and Transcriptional Regulation. Plant Physiology. 169, 971 (2015).
- van Zeijl, A. . Dissecting Hormonal Pathways in Nitrogen-Fixing Rhizobium Symbioses. , (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。