रूपांतरण, जीनोम संपादन और Phenotyping नाइट्रोजन फिक्सिंग उष्णकटिबंधीय कैनाबी ट्री Parasponia andersonii
In This Article
Summary
पैरास्पोनिया एंडरसनी एक तेजी से बढ़ते उष्णकटिबंधीय पेड़ है जो कैनबिस परिवार (कैनाबेसी) से संबंधित है और राइजोबियम के सहयोग से नाइट्रोजन-फिक्सिंग रूट नोड्यूल बना सकता है। यहाँ, हम पी एंडरसनी में रिवर्स आनुवंशिक विश्लेषण के लिए एक विस्तृत प्रोटोकॉल का वर्णन Agrobacterium tumefaciensपर आधारित -मध्यस्थ स्थिर परिवर्तन और CRISPR/Cas9 आधारित जीनोम संपादन.
Abstract
पैरास्पोनिया एंडरसनी एक तेजी से बढ़ते उष्णकटिबंधीय पेड़ है जो कैनबिस परिवार (कैनाबासी) से संबंधित है। 4 अतिरिक्त प्रजातियों के साथ मिलकर, यह राइजोबियम के साथ नाइट्रोजन-फिक्सिंग नोड्यूल सिम्बायोसिस स्थापित करने में सक्षम केवल ज्ञात गैर-लेगम वंश बनाता है। फलियां और पी एंडरसनी के बीच तुलनात्मक अध्ययन आनुवंशिक नेटवर्क अंतर्निहित रूट नोड्यूल गठन में मूल्यवान अंतर्दृष्टि प्रदान कर सकता है। तुलनात्मक अध्ययन की सुविधा के लिए, हमने हाल ही में पी एंडोर्नेई जीनोम का अनुक्रम किया और एग्रोबैक्टीरियम टयूफेसिएन्स-मध्यस्थ स्थिर परिवर्तन और CRISPR/Cas9-आधारित जीनोम संपादन की स्थापना की। यहाँ, हम परिवर्तन और जीनोम संपादन प्रक्रियाओं के बारे में एक विस्तृत विवरण प्रदान करते हैं जो P. andersoniiके लिए विकसित की गई है। इसके अतिरिक्त, हम बीज अंकुरण की प्रक्रियाओं और सहजीवी फीनोटाइप्स की विशेषता का वर्णन करते हैं। इस प्रोटोकॉल का उपयोग करना, स्थिर ट्रांसजेनिक उत्परिवर्ती लाइनों 2-3 महीने की अवधि में उत्पन्न किया जा सकता है. टी0 ट्रांसजेनिक लाइनों के इन विट्रो प्रचार में वनस्पति फेनोटाइपिंग प्रयोगों को ए tumefaciens सह-कृषि के बाद 4 महीने में शुरू करने की अनुमति देता है। इसलिए, इस प्रोटोकॉल केवल मामूली क्षणिक Agrobacterium राइजोजीन्स -आधारित रूट रूपांतरण विधि पी andersoniiके लिए उपलब्ध से अधिक समय लगता है, हालांकि कई स्पष्ट लाभ प्रदान करता है. साथ में, यहाँ वर्णित प्रक्रियाओं पी andersonii सहजीवी संघों के रूप में के रूप में अच्छी तरह से इस उष्णकटिबंधीय पेड़ के जीव विज्ञान के संभावित अन्य पहलुओं को समझने के उद्देश्य से अध्ययन के लिए एक अनुसंधान मॉडल के रूप में इस्तेमाल किया जा करने की अनुमति.
Introduction
पैरास्पोनिया एंडरसनी कैनबिस परिवार (कैनाबासी) से संबंधित एक उष्णकटिबंधीय वृक्ष है और पापुआ न्यू गिनी और कई प्रशांत द्वीप समूह1,2,3के मूल निवासी है . 4 अतिरिक्त पैरास्पोनिया प्रजातियों के साथ, यह केवल गैर-लेगम वंश का प्रतिनिधित्व करता है जो राइजोबिया के साथ नाइट्रोजन-फिक्सिंग नोड्यूल सिम्बायोसिस स्थापित कर सकता है। इस सहजीवन का अच्छी तरह से फली (फैबेसी) मॉडल मेडिकागो ट्रंकोटला और लोटस जैपोनिकसमें अध्ययन किया जाता है जिसके परिणामस्वरूप नोड्यूल गठन और कार्य4के आण्विक आनुवंशिक प्रकृति का विस्तृत ज्ञान प्राप्त होता है . इसके अतिरिक्त, यह प्रदर्शित किया गया था कि फलियां में जड़ नोड्यूल सहजीवन बहुत पुराने पर स्थापित किया गया है, और व्यापक arbuscular mycorrhizal सहजीवन5. Phylogenomic तुलना का सुझाव है कि फलियां, Parasponiaके नाइट्रोजन फिक्सिंग नोड्यूल symbioses, साथ ही, तथाकथित actinorhizal संयंत्र प्रजातियों है कि मेजबान diazotrophic Frankia बैक्टीरिया, एक साझा विकासवादी मूल है 6,7,8. यह निर्धारित करने के लिए कि क्या फली नोड्यूल गठन में शामिल होने के लिए पहचाने गए जीन संरक्षित आनुवंशिक आधार का हिस्सा हैं, गैर-लेगम प्रजातियों पर अध्ययन आवश्यक हैं। यह अंत करने के लिए, हम एक तुलनात्मक अनुसंधान मॉडल के रूप में पी andersonii का उपयोग करने का प्रस्ताव, फलियां के साथ, मूल आनुवंशिक नेटवर्क अंतर्निहित रूट नोड्यूल गठन और कामकाज की पहचान करने के लिए.
पी andersonii एक अग्रणी है कि ज्वालामुखी पहाड़ियों की ढलानों पर पाया जा सकता है. यह प्रति माह 45 सेमी की वृद्धि की गति को पूरा कर सकता है और 10 मीटर9तक की लंबाई तक पहुंच सकता है . पी एंड्रूनाई के पेड़ हवा-परागण होते हैं, जो अलग-अलग नर और मादा फूलों के गठन से सुविधाजनकहोते हैं 3,10. हमने हाल ही में पी एंडरसनीके द्विगुणित जीनोम (2n ] 20; 560 Mb/1C) का अनुक्रम और एनोटेट किया है, और 2 अतिरिक्त पैरास्पोनिया प्रजातियों के प्रारूप जीनोम अनुक्रमों को इकट्ठा किया है; पी. इससे पता चला कि 35,000 पी एंडरसनी जीन मॉडल जो एम ट्रंकटुला, सोयाबीन (ग्लिसिन मैक्स), अरबीडोप्सिस थालियाना, वुडलैंड स्ट्रॉबेरी ( फ्रेगेरिया वेस्का, ट्रेमा ओरिएन्टेलिस , ब्लैक कॉटन पॉपलर (पॉपुलस ट्राइकोकार्पा) और यूकेलिप्टस (यूकेलिप्टस ग्रैन्डिस)6. इसके अतिरिक्त, M. truncatula और P. andersonii के बीच ट्रांसक्रिप्टोम तुलना 290 putative orthologues का एक सेट है कि दोनों प्रजातियों में एक नोड्यूल बढ़ाया अभिव्यक्ति पैटर्न प्रदर्शित की पहचान6. यह तुलनात्मक अध्ययन के लिए एक उत्कृष्ट संसाधन प्रदान करता है.
पी एंडोर्नाली जड़ों और नोड्यूल में जीन प्रकार्य का अध्ययन करने के लिए एग्रोबैक्टीरियम राइजोजेनेस-मध्यस्थ मूल परिवर्तन के लिए एक प्रोटोकॉलस्थापितकिया गया है . इस प्रोटोकॉल का उपयोग करना, ट्रांसजेनिक जड़ों असर यौगिक पौधों एक अपेक्षाकृत कम समय सीमा में उत्पन्न किया जा सकता है. इस विधि को फली-सहजीवन अनुसंधान12,13,14में भी व्यापक रूप से लागू किया जाता है। हालांकि, इस विधि का नुकसान यह है कि केवल जड़ों बदल रहे हैं और प्रत्येक transgenic जड़ एक स्वतंत्र परिवर्तन घटना का प्रतिनिधित्व करता है, पर्याप्त भिन्नता में जिसके परिणामस्वरूप. इसके अलावा, परिवर्तन क्षणिक है और ट्रांसजेनिक लाइनों बनाए रखा नहीं जा सकता है. यह ए राइजोजीन्स-आधारित रूट ट्रांसफॉर्मेशन को CRISPR/Cas9-मध्यस्थ जीनोम संपादन के लिए कम अनुकूल बनाता है। इसके अतिरिक्त, ए राइजोजेने अपने रूट इनोवेन्चिंग टिड्डी (रोल ) जीनों को पौधे के जीनोम में स्थानांतरित कर देते हैं, जो एक बार हार्मोन होमोस्टेसिस15के साथ हस्तक्षेप व्यक्त करते थे। यह ए राइजोजेनेसमें पौधे हार्मोन की भूमिका का अध्ययन करना - रूपांतरित जड़ों को चुनौतीपूर्ण बनाता है। इन सीमाओं को पार करने के लिए, हमने हाल ही में एग्रोबैक्टीरियम tumefaciens-आधारितपरिवर्तन और CRISPR/Cas9-मध्यस्थमटजनन के लिए एक प्रोटोकॉल विकसित किया है।
यहाँ, हम ए tumefaciensआधारित परिवर्तन प्रक्रिया का एक विस्तृत विवरण प्रदान करते हैं और रिवर्स आनुवंशिकी पाइप लाइन पी andersoniiके लिए विकसित की है. इसके अतिरिक्त, हम ट्रांसजेनिक plantlets के बहाव से निपटने के लिए प्रोटोकॉल प्रदान करते हैं, symbiotic बातचीत का अध्ययन करने के लिए assays सहित. यहाँ वर्णित प्रोटोकॉल का उपयोग करके, एक 2-3 महीने की अवधि में एकाधिक ट्रांसजेनिक लाइनें उत्पन्न की जा सकती हैं। CRISPR/Cas9-मध्यस्थ उत्तजनन के साथ संयोजन में, यह नॉकआउट उत्परिवर्ती लाइनों के कुशल पीढ़ी की अनुमति देता है. इन उत्परिवर्ती रेखाओं को इन विट्रो10,16,17में वनस्पति रूप से प्रचारित किया जा सकता है , जो परिवर्तन प्रक्रिया के 4 महीनों में फेनोटाइपिक लक्षणीकरण शुरू करने के लिए पर्याप्त सामग्री उत्पन्न करने की अनुमति देता है 10शुरू किया गया है . साथ में, प्रक्रियाओं के इस सेट किसी भी प्रयोगशाला के अध्ययन के लिए एक अनुसंधान मॉडल के रूप में पी andersonii अपनाने के लिए rhizobial और mycorrhizal संघों, साथ ही संभावित रूप से इस उष्णकटिबंधीय पेड़ के जीव विज्ञान के अन्य पहलुओं को समझने के उद्देश्य से अनुमति देनी चाहिए.
Protocol
1. ग्रीन हाउस में पी एंडरसनी पेड़ बढ़ाएँ
-
अंकुर P. andersonii WU1 बीज18|
- ताजा परपोनिया जामुन का प्रयोग करें या सूखे जामुन को पानी में 2 ज के लिए फिर से हाइड्रेट करने के लिए भिगो दें। ऊतक कागज के एक टुकड़े पर स्क्वैश जामुन या बीज को हटाने के लिए एक चाय छलनी के अंदर के खिलाफ रगड़।
- 15-20 मिनट के लिए वाणिज्यिक ब्लीच ($4% हाइपोक्लोराइट) का उपयोग करके बीजों को संक्रमित करें और बाद में बाँझ पानी का उपयोग करके बीज 6 बार धोएं।
- बीज बाँझ 200 डिग्री एल पीसीआर ट्यूबों के लिए स्थानांतरण। नलियों को बाँझ पानी से भरें, ताकि बीज पूरी तरह से जलमग्न हो जाएं। निम्नलिखित कार्यक्रम चलाने वाले थर्मोसाइकिलर में 10 दिनों के लिए ट्यूबों को इनक्यूबेट करें: 30 चक्र (7 डिग्री सेल्सियस 4 एच के लिए, 4 एच के लिए 28 डिग्री सेल्सियस)। एक गर्म ढक्कन का उपयोग न करें, क्योंकि यह बीज को मार सकता है।
- एसएच-0 प्लेट्स तैयार करें (तालिका 1देखें )। एसएच-0 प्लेटों में बीज स्थानांतरित करें और 28 डिग्री सेल्सियस, 16 ज:8 एच दिन: रात में इनक्यूबेट करें। 28 डिग्री सेल्सियस पर ऊष्मायन के दौरान सुखाने को रोकने के लिए लोचदार सील पन्नी की 2 परतों के साथ बंद प्लेटें।
- अंकुरों के बाद सच पत्तियों का अपना पहला सेट विकसित किया है ($3-4 सप्ताह 28 डिग्री सेल्सियस पर ऊष्मायन के बाद), वाणिज्यिक potting मिट्टी से भरा बर्तन में अंकुर हस्तांतरण और एक पारदर्शी प्लास्टिक कप के साथ अंकुर को कवर करने के लिए शुष्कता को रोकने के. एक 28 डिग्री सेल्सियस जलवायु कमरे या ग्रीनहाउस में बर्तन प्लेस, $ 85% आरएच, एक 16 h:8 एच दिन के तहत: रात शासन.
- 1 सप्ताह के बाद, पारदर्शी प्लास्टिक कप को हटा दें। बर्तन नियमित रूप से पानी और जब पेड़ विकास को बनाए रखने के लिए उर्वरक के साथ बड़ा पूरक हो जाना.
2. पी एंडरसनी के CRISPR/Cas9-मध्यस्थ म्यूटजेनेसिस के लिए निर्माणों की क्लोनिंग
नोट: मानक द्विआधारी रूपांतरण सदिशों का उपयोग पी एंडरसनीके स्थिर रूपांतरण के लिए किया जा सकता है। यहाँ, एक उदाहरण के रूप में, मॉड्यूलर क्लोनिंग (जैसे, गोल्डन गेट)19का उपयोग कर CRISPR/Cas9-मध्यस्थ mutagenesis के लिए निर्माण उत्पन्न करने के लिए एक प्रक्रिया है।
- एक निर्मित CRISPR डिजाइन उपकरण की विशेषता bioinformatics सॉफ्टवेयर का उपयोग कर, ब्याज की जीन (ओं) के लिए गाइड आरएनए लक्ष्य दृश्यों की पहचान करें। पूर्ण नॉकआउट प्राप्त करने की संभावना को बढ़ाने के लिए लक्ष्य जीन की कोडिंग अनुक्रम के 5'अंत में स्थित गाइड आरएनए दृश्यों का चयन करें। P. andersonii जीनोम6के खिलाफ खोज करके ऑफ-लक्ष्य प्रभाव के लिए जाँच करने के लिए सुनिश्चित करें।
नोट: लक्ष्य जीन प्रति 2 sgRNAs का प्रयोग करें, अधिमानतः 200-300 बीपी के अलावा. यह हटाने कि पीसीआर द्वारा और बाद में agarose जेल electrophoresis द्वारा पहचाना जा सकता उत्पन्न कर सकते हैं. - स्तर उत्पन्न 1 गोल्डन गेट sgRNA दृश्यों युक्त निर्माण.
- डिजाइन प्राइमर निम्नलिखित प्राइमर अनुक्रम में एन(20) की स्थिति में 20 बीपी गाइड अनुक्रम डालने के द्वारा प्रत्येक व्यक्ति sgRNA बढ़ाना करने के लिए: 5'-TGTGGTCTCAATGएन(20) GTTTAGCTAGAATAG-3'।
नोट: यदि मार्गदर्शिका अनुक्रम GN(19)के बराबर होती है, तो प्राइमर अनुक्रम में सम्मिलित करने से पहले मार्गदर्शिका अनुक्रम के 5' अंत में G निकालें. - PCR amplify sgRNAs से pICH86966::AtU6p::SgRNA$PDS20 कदम 2.2.1 और सार्वभौमिक रिवर्स प्राइमर पर डिजाइन आगे प्राइमर का उपयोग कर: 5'-TGTCTCCGTAGTGTACGTGTTAC-3'. एक उच्च निष्ठा गर्मी-स्थिर डीएनए polymerase और निम्नलिखित पीसीआर शर्तों का उपयोग करें: 30 s के लिए 98 डिग्री सेल्सियस; 30 चक्र (98 डिग्री सेल्सियस के लिए 10 s; 20 s के लिए 53 डिग्री सेल्सियस; 10 s के लिए 72 डिग्री सेल्सियस); 72 डिग्री सेल्सियस के लिए 7 मिनट सफल पीसीआर प्रतिक्रियाओं एक 165 बीपी amplicon उपज.
- एक वाणिज्यिक पीसीआर शुद्धि किट का उपयोग कर पीसीआर amplicon स्तंभ शुद्ध। बाद में, Arabidopsis thaliana AtU6p छोटे आरएनए प्रमोटर के पीछे क्लोन sgRNAs के लिए गोल्डन गेट प्रतिक्रियाओं की स्थापना: sgRNA पीसीआर amplicon के 10 एनजी, 150 एनजी PICSL01009::AtU6p20, उचित स्तर 1 स्वीकारकर्ता वेक्टर के 60 एनजी, T4 के 2 $L लिगेज बफर, 2 डिग्री एल 0.1% गोजातीय सीरम एल्बुमिन (बीएसए), 0.5 डिग्री सेल्सियस, टी 4 लिगेज़ के 0.5 डिग्री एल, अल्ट्रा-शुद्ध पानी के साथ 20 डिग्री सेल्सियस को भरें। सुनिश्चित करें कि सभी sgRNAs एक ही अभिविन्यास में क्लोन कर रहे हैं hairpin गठन को रोकने के लिए.
- निम्नलिखित कार्यक्रम चलाने वाले थर्मोसाइकिलर में इनक्यूबेट प्रतिक्रियाएं: 20 s के लिए 37 डिग्री सेल्सियस; 26 चक्र (37 डिग्री सेल्सियस 3 मिनट के लिए; 4 मिनट के लिए 16 डिग्री सेल्सियस); 5 मिनट के लिए 50 डिग्री सेल्सियस; 5 मिनट के लिए 80 डिग्री सेल्सियस, एलबी मध्यम21 पर एस्चेरीचिया कोलाई और प्लेट के लिए गोल्डन गेट प्रतिक्रियाओं को बदलदें जिसमें एम्पीसिलिन (50 मिलीग्राम/एल), एक्स-गल (200 मिलीग्राम/एल) और आईपीटीजी (1 एम एम) शामिल हैं।
नोट: अल्ट्रा शुद्ध पानी और dimethylformamide में क्रमशः IPTG और एक्स-गल के शेयर समाधान तैयार करें। एम्पीसिलिन और IPTG स्टॉक समाधान को फ़िल्टर करें और सभी स्टॉक को -20 डिग्री सेल्सियस पर स्टोर करें। डाइमेथिलफॉर्मामीड से निपटने के दौरान दस्ताने पहनें। - सफेद कालोनियों का चयन करें और एक वाणिज्यिक प्लाज्मिड आइसोलेशन किट का उपयोग कर के लिए प्लाज्मिड को अलग करें। अनुक्रम गोल्डन गेट स्तर 2 विधानसभा के साथ जारी रखने से पहले अलग प्लाज्मिड सत्यापित करें।
- डिजाइन प्राइमर निम्नलिखित प्राइमर अनुक्रम में एन(20) की स्थिति में 20 बीपी गाइड अनुक्रम डालने के द्वारा प्रत्येक व्यक्ति sgRNA बढ़ाना करने के लिए: 5'-TGTGGTCTCAATGएन(20) GTTTAGCTAGAATAG-3'।
- इकट्ठा स्तर 2 गोल्डन गेट स्थिर परिवर्तन के लिए निर्माण।
- स्तर 1 AtU6p का उपयोग कर एक गोल्डन गेट प्रतिक्रिया प्रदर्शन::SgRNA निर्माण (धारा 2.2 के तहत उत्पन्न) के रूप में के रूपमें अच्छी तरह से pICH47802::NPTII, pICH4742::35S समर्थक::NLS-aCas9::35Ster, स्तर 2 स्वीकारकर्ता pICSL4723 और उपयुक्त अंत-लिंकर (Engler एट अल22देखें)। निम्नलिखित के रूप में प्रतिक्रियाओं का प्रदर्शन करें: प्रत्येक दाता वेक्टर के 100 फ्मोल का उपयोग करें और स्वीकारकर्ता वेक्टर के $20 fmol का उपयोग करें और टी 4 लिगे बफर के 2 डिग्री एल, 0.1% बीएसए के 2 $L, BpiI के 0.5 डिग्री एल, टी 4 लिगाज़ के 0.5 डिग्री एल, अल्ट्रा-शुद्ध पानी के साथ 20 डिग्री सेल्सियस को भरें।
नोट: स्तर 1 plasmids pICH47802::NPTII, pICH47742::35S समर्थक::[NLS-aCas9::35Ster क्लोन किया जा करने के लिए पहले की जरूरत है ( पूरक फ़ाइल 1देखें), के रूप में धारा 2.220,22 के तहत sgRNAs के लिए वर्णित है ,23. - इनक्यूबेट प्रतिक्रियाओं के रूप में के रूप में कदम 2.2.4 और ई. कोलाईमें बदलना . एलबी मध्यम पर प्लेट जिसमें कानामाइसिन होता है। अगले दिन, सफेद कालोनियों का चयन करें और प्लाज्मिड को अलग करें। प्रतिबंध-पाचन विश्लेषण द्वारा सही प्लाज़्मिड असेंबली का निर्धारण करें।
- स्तर 1 AtU6p का उपयोग कर एक गोल्डन गेट प्रतिक्रिया प्रदर्शन::SgRNA निर्माण (धारा 2.2 के तहत उत्पन्न) के रूप में के रूपमें अच्छी तरह से pICH47802::NPTII, pICH4742::35S समर्थक::NLS-aCas9::35Ster, स्तर 2 स्वीकारकर्ता pICSL4723 और उपयुक्त अंत-लिंकर (Engler एट अल22देखें)। निम्नलिखित के रूप में प्रतिक्रियाओं का प्रदर्शन करें: प्रत्येक दाता वेक्टर के 100 फ्मोल का उपयोग करें और स्वीकारकर्ता वेक्टर के $20 fmol का उपयोग करें और टी 4 लिगे बफर के 2 डिग्री एल, 0.1% बीएसए के 2 $L, BpiI के 0.5 डिग्री एल, टी 4 लिगाज़ के 0.5 डिग्री एल, अल्ट्रा-शुद्ध पानी के साथ 20 डिग्री सेल्सियस को भरें।
- रूपांतरण स्तर 2 एग्रोबैक्टीरियम tumefaciens तनाव एजीएल 124के लिए निर्माण |
3. पी एंडरसनी का स्थिर परिवर्तन
- ए tumefaciens तनाव AGL1 ब्याज के निर्माण के साथ बदल के साथ उपयुक्त एंटीबायोटिक दवाओं युक्त 2 LB प्लेटें टीका. 2 दिनों के लिए 28 डिग्री सेल्सियस पर इनक्यूबेट प्लेटें।
- ग्रीन हाउस विकसित पेड़ों से युवा शाखाओं फसल। प्रत्येक परिवर्तन के लिए लंबाई में 5-8 सेमी की लगभग 5 शाखाओं का प्रयोग करें। केवल स्वस्थ गैर-संक्रमित शाखाओं का उपयोग करना सुनिश्चित करें। पत्तियों को इस प्रकार काट कर निकालें कि प्रत्येक डंठल के अंत में पत्ती के ऊतकों के 1 सेमी2 को छोड़ दिया जाता है। पत्तियों को छोड़ दें।
- 15 मिनट के लिए ऊतक को संक्रमित 1:1-धुंधला वाणिज्यिक ब्लीच का उपयोग कर ($2% हाइपोक्लोराइट कमजोर पड़ने के बाद) जिसमें polysorbate 20 की कुछ बूँदें होती हैं। फिर, ऊतक 6 बार autoclaved पानी के साथ कुल्ला.
नोट:यह कदम है, साथ ही, निम्नलिखित चरणों के ऊतक बाँझ रखने के लिए एक laminar नीचे प्रवाह कैबिनेट के अंदर आयोजित करने की आवश्यकता है. - घुसपैठ माध्यम के 25 एमएल में 1-2 प्लेटों से ए tumefaciens कोशिकाओं को फिर से निलंबित (तालिका देखें 1) एसीटोसिरिंगोन युक्त (20 mg/L) और एक गैर-आयनिक सर्फैक्टेंट (0.001% v/v) $ 5 के ऑप्टिकल घनत्व (ओडी600)तक पहुंचने के लिए।
नोट:70% इथेनॉल में एसीटोसिरिंगोन स्टॉक समाधान तैयार करें और -20 डिग्री सेल्सियस पर स्टोर करें। घुसपैठ माध्यम में जोड़ने से पहले गैर-आयनिक सर्फैक्टेंट को फिल्टर-स्टरलाइज़ करने की आवश्यकता है। - ए tumefaciens निलंबन के अंदर लंबाई में $ 1 सेमी के टुकड़े में स्टेम और डंठल दोनों ऊतक कट, जिससे दोनों पक्षों में ताजा घाव पैदा. 10-30 मिनट के लिए ए tumefaciens निलंबन में ऊतक टुकड़े छोड़ दें।
- मूल माध्यम तैयार करें (तालिका 1देखें ) और ऑटोक्लेविंग के बाद एसीटोसीरिंगोन (20 मिलीग्राम/एल) जोड़ें। फिल्टर कागज के एक बाँझ टुकड़े पर सूखी ऊतक टुकड़े और यह मध्यम पर जगह ($ 10 explants / 2 दिनों के लिए 21 डिग्री सेल्सियस पर अंधेरे में इनक्यूबेट प्लेटें।
नोट:एसीटोसेरिंगोन जोड़ने से पहले माध्यम को $60 डिग्री सेल्सियस तक ठंडा करने की अनुमति दें। - 2 दिनों के बाद, फंगल या स्पष्ट जीवाणु संदूषण (ए tumefaciensके अलावा अन्य बैक्टीरिया) के लिए प्लेटों का निरीक्षण करें। दूषित प्लेटों को खारिज करने की आवश्यकता है।
- द्रव एसएच-10 माध्यम तैयार करें (तालिका 1देखें)। ऑटोक्लेविंग के बाद, पॉलीसोर्बेट 20 (0.01%, v/ एसएच-10 के 10 एमएल में ऊतक के टुकड़े स्थानांतरित करें जिसमें पॉलीसोर्बेट 20 होता है। कम से कम 10 मिनट की अवधि के दौरान, धीरे ऊतक धोने के लिए हर 2-3 मिनट आंदोलन.
- पॉलीसर्बेट 20 युक्त ताजा एसएच-10 के साथ दो अतिरिक्त बार धोएं। इन बार, धोने के कदम प्रति एक 2-3 मिनट ऊष्मायन समय पर्याप्त है।
- पक्ष माध्यम तैयार करें (तालिका 1देखें)। ऑटोक्लेविंग के बाद, सेफोटैक्सीम (300 मिलीग्राम/एल) और कानामीसिन (50 मिलीग्राम/एल) जोड़ें और प्लेटें डालें। द्वितीयक रूपांतरणों (ट्रांसजेनिक कानामी-प्रतिरोधी लाइनों के रूपांतरण) के लिए, हाइग्रोमाइसिन (15 मिलीग्राम/एल) चयन लागू करें।
- फिल्टर कागज के बाँझ टुकड़े पर सूखी ऊतक टुकड़े. बाद में, चरण 3.9 में तैयार प्लेटों में ऊतक के टुकड़े स्थानांतरित करें।
- 28 डिग्री सेल्सियस, 16 h:8 ज दिन:रात में 7 दिनों के लिए इनक्यूबेट प्लेटें। कवक या जीवाणु संदूषण और ए tumefaciensके अत्यधिक विकास के लिए हर 2 दिन की जांच प्लेटें . संदूषण के मामले में, गैर-संक्रमित टुकड़ों को एक ताजा प्लेट में स्थानांतरित करें।
- 7 दिनों के बाद, ऊतक के टुकड़ों को प्रचार माध्यम में स्थानांतरित करें (तालिका 1देखें ) जिसमें सेफोटैक्सीम (300 मिलीग्राम/एल) और कानामीसिन (50 मिलीग्राम/एल) शामिल हैं। 28 डिग्री सेल्सियस, 16 h:8 h दिन: रात में इनक्यूबेट प्लेटें। ट्रांसजेनिक शूटिंग विकसित होने तक सप्ताह में एक बार प्लेटें ताज़ा करें। केवल ताजा प्लेटों के लिए गैर संक्रमित ऊतक टुकड़े हस्तांतरण करने के लिए सुनिश्चित करें. ए tumefaciensद्वारा बढ़ रहे हैं कि टुकड़े को छोड़ दें ।
- एक बार putatively-transgenic शूटिंग लंबाई में 1 सेमी कर रहे हैं, कटौती शूटिंग और संस्कृति उन्हें स्वतंत्र रूप से प्रचार माध्यम में cefotaxime युक्त (300 mg/L) और kanamycin (50 mg/L). यह सुनिश्चित करने के लिए कि शूटिंग स्वतंत्र transformants का प्रतिनिधित्व करते हैं, एक explant के प्रत्येक पक्ष से केवल एक ही गोली मार ले.
- चरण 5-2 के अंतर्गत वर्णित के रूप में वनस्पति-परिवर्तनात्मक रूप से पादात्मक-ट्रांसजेनिक शूट का प्रचार करें।
4. Putatively-ट्रांसजेनिक शूट्स के जेनोटाइपिंग
- डिजाइन प्राइमर sgRNA मान्यता साइट (ओं) फैले. PCR amplicon अनुक्रमण की अनुमति देने के लिए, प्राइमर 150-250 बीपी sgRNA मान्यता साइट (ओं) से दूर का चयन करें।
- एक पत्ती टिप ($ 5 मिमी) प्रत्येक ट्रांसजेनिक गोली से काट genotyped किया जा करने के लिए। इसके अलावा, एक जंगली प्रकार नियंत्रण नमूना फसल।
- चरण 4.1 पर डिजाइन प्राइमर और एक वाणिज्यिक किट का उपयोग कर 50 डिग्री एल पीसीआर प्रतिक्रियाओं प्रदर्शन सीधे संयंत्र के नमूने से डीएनए बढ़ाना. वैकल्पिक रूप से, पीसीआर प्रतिक्रियाओं एक उच्च निष्ठा polymerase का उपयोग कर शुद्ध डीएनए पर किया जा सकता है।
- एक 1.5-2% agarose जेल पर अलग पीसीआर amplicons.
- जेल इलेक्ट्रोफोरोसिस से परिणामों का विश्लेषण करें। कई बैंड (1 से अधिक एलील) और जंगली प्रकार से अलग आकार के साथ पीसीआर amplicons उत्पादन नमूने के लिए जाँच करें, जो मध्यम आकार indels की उपस्थिति इंगित करता है.
- अनुक्रम PCR amplicons सटीक उत्परिवर्तनों की पहचान करने के लिए. एक एकल पीसीआर amplicon उत्पादन नमूने के लिए, पीसीआर उत्पादों सीधे अनुक्रम किया जा सकता है. नमूने है कि जेल electrophoresis के बाद अधिक से अधिक 1 बैंड का उत्पादन या कि पीसीआर amplicon के प्रत्यक्ष अनुक्रमण के बाद विषमयुग्मज होने के लिए दिखाई देते हैं, एक कुंद अंत क्लोनिंग वेक्टर में क्लोन किया जा करने की जरूरत है पहले. बाद में, नमूने में मौजूद सभी संभव alleles की पहचान करने के लिए प्रत्येक नमूने के लिए अनुक्रम कई क्लोन।
- ब्याज के जीन के लिए अनुक्रमण परिणाम संरेखित करें और sgRNA लक्ष्य साइट (ओं) के पास उत्परिवर्तनों के लिए जाँच करने के लिए संरेखण का निरीक्षण. इसके बाद, जाँच करें कि क्या ये उत्परिवर्तन frameshifts बनाते हैं। 2 alleles, और इन-फ्रेम उत्परिवर्तनों युक्त लाइनों के साथ लाइनों को छोड़ें।
- आगे के विश्लेषण के लिए कई पंक्तियों का चयन करें.
- चरण 5.2 के अंतर्गत वर्णित के रूप में प्रोपेगेट चयनित पंक्तियाँ.
- जब लाइनों कई नए शूटिंग विकसित की है, $ 3 पत्ती सुझावों से नए नमूने लेने के लिए और कदम दोहराने 4.3-4.7. निर्धारित करें कि क्या एक ही पंक्ति से निकलने वाले नमूनों में से प्रत्येक में मौजूद उत्परिवर्तन ों के साथ-साथ मूल PCR नमूने समान हैं। सभी नमूनों में एक ही उत्परिवर्तन उपज लाइनों सजातीय रूप से उत्परिवर्तित कर रहे हैं और आगे प्रयोग के लिए इस्तेमाल किया जा सकता है। इन पंक्तियों के रूप में एक ही परिणाम उपज नहीं है कि लाइनों को छोड़ें chimeric हैं.
5. प्रयोग के लिए जड़ें पी एंडरसनी प्लांटलेट की तैयारी
-
पी एंडरसनीकी एक नई ऊतक संस्कृति लाइन शुरू .
- फसल कक्षीय कलियों, युवा आकस्मिक शूटिंग या स्वस्थ पेड़ों से पत्ती ऊतक. वैकल्पिक रूप से, अंकुर एक प्रारंभिक सामग्री के रूप में इस्तेमाल किया जा सकता है.
- 1:1-diluted वाणिज्यिक ब्लीच का उपयोग कर ऊतक को संक्रमित करें ($2% हाइपोक्लोराइट कमजोर पड़ने के बाद) जिसमें पॉलीसोर्बेट की कुछ बूंदें 15 मिनट के लिए होती हैं। बाद में, ऑटोक्लेव्ड पानी का उपयोग करके ऊतक 6 बार कुल्ला करें।
नोट:यह कदम है, साथ ही, निम्नलिखित चरणों के ऊतक बाँझ रखने के लिए एक laminar downflow या laminar crossflow कैबिनेट के अंदर आयोजित किया जाना चाहिए. - ऊतक को संचरण माध्यम में स्थानांतरित करें (तालिका 1देखें )। 28 डिग्री सेल्सियस, 16 एच:8 एच दिन: रात में लोचदार सील पन्नी और इनक्यूबेट प्लेटों की 2 परतों के साथ बंद प्लेटें।
- यह सुनिश्चित करने के लिए कि ऊतक फंगल या जीवाणु संदूषण से मुक्त है, पहले 2 सप्ताह के दौरान हर कुछ दिनों में प्लेटों का निरीक्षण करें।
- प्रचार माध्यम की एक ताजा थाली पर $ 10 शूटिंग रखकर और लोचदार सील पन्नी के 2 परतों के साथ प्लेट बंद करके ऊतक का प्रचार करें। 28 डिग्री सेल्सियस, 16 h:8 h दिन: रात में इनक्यूबेट प्लेटें। इस चरण को हर 4 सप्ताह में दोहराएँ.
- जब शूटिंग लंबाई में होती है, तो उनके आधार पर शूटिंग काटकर उन्हें पक्ष माध्यम पर रखते हैं (सारणी 1देखें)। के बारे में 10 शूटिंग एक ही पक्ष प्लेट पर रखा जा सकता है. स्थिति मध्यम में गोली मार के बेसल टिप डालने से ईमानदार गोली मारता है. जड़ें 28 डिग्री सेल्सियस, 16 ज:8 एच दिन:रात में प्लेटों के ऊष्मायन के बाद 10-14 दिनों में दिखाई देते हैं।
नोट: सभी शूटिंग जड़ नहीं है, लेकिन ऊतक संस्कृति संचरण के लिए हिस्सा रखने के लिए (चरण 5.2 देखें).
6. पॉट्स में पी एंडरसनी प्लांटेट की नोडुलेशन
-
राइजोबियम इनोकुलम तैयार करें।
- तरल YEM माध्यम के 10 एमएल inoculate (तालिका 2देखें ) Mesorhizobium plurifarium BOR26 की एक कॉलोनी से और 2 दिनों के लिए 28 डिग्री सेल्सियस पर इनक्यूबेट.
नोट: एम plurifarium BOR2 पसंद किया जाता है के रूप में यह कुशलता से P. andersoniinodulates . हालांकि, अन्य राइजोबियम उपभेदों का उपयोग पी एंडरसनी (उदाहरण के लिए ब्रैड्रिजोबियम एलकानी WUR325, राइजोबियम उष्णकटिबंधीय CIAT89926,27 या ब्रैड्रिजोबियम) के नोडुलेशन के लिए भी किया जा सकता है। sp. Kelud2A4). - तरल YEM माध्यम की एक बड़ी मात्रा टीका करने के लिए 10 एमएल संस्कृति का प्रयोग करें। इस संस्कृति की मात्रा बर्तन है कि टीका लगाने की जरूरत की संख्या पर निर्भर है.
- द्रव EKM माध्यम तैयार करें (तालिका3, तालिका 4) देखिए। कोशिकाओं को काटने के लिए 3,500 x ग्राम पर 10 मिनट के लिए जीवाणु संस्कृति को सेंट्रीफ्यूज करें। बाद में, तरल EKM में जीवाणु गोली फिर से निलंबित (मूल YEM संस्कृति के रूप में लगभग एक ही मात्रा का उपयोग करें) और ऑप्टिकल घनत्व (ओडी600)का निर्धारण.
- तरल YEM माध्यम के 10 एमएल inoculate (तालिका 2देखें ) Mesorhizobium plurifarium BOR26 की एक कॉलोनी से और 2 दिनों के लिए 28 डिग्री सेल्सियस पर इनक्यूबेट.
- $ 20 बर्तन के लिए, तरल EKM मध्यम के 3 एल तैयार करने और चरण 6.1.3 पर तैयार राइजोबियल निलंबन के साथ टीका. OD तक पहुँचने के लिए600 $ 0.025.
- 1,250 ग्राम पर्लाइट के साथ राइजोबिया युक्त ईकेएम के 3 एल मिलाएं। इसके बाद, इस मिश्रण के 210 ग्राम बाँझ पारदर्शी polypropylene बर्तन में जोड़ें. वैकल्पिक रूप से, perlite के बजाय, nodulation assays के लिए एक सब्सट्रेट के रूप में रेत का उपयोग करें.
- प्रत्येक बर्तन में 1-3 पी एंडरसनी पौधे लगाएं। इसके अलावा, CRISPR नियंत्रण निर्माण के साथ बदल P. andersonii plantlets युक्त कई बर्तन तैयार (देखें अनुपूरक तालिका1). कई बर्तन वजन प्रयोग के दौरान पानी की हानि का निर्धारण करने में सक्षम होने के लिए. प्रत्येक बर्तन के नीचे कवर करने के लिए प्रकाश जोखिम से जड़ों ढाल.
- एक जलवायु विकास के कमरे में इनक्यूबेट बर्तन (28 डिग्री सेल्सियस, 16 एच:8 एच दिन: रात) 4-6 सप्ताह के लिए। सप्ताह में एक बार, पानी की हानि का निर्धारण करने के लिए कई बर्तन वजन। यदि पानी की हानि 10 एमएल से अधिक है, तो नुकसान की भरपाई करने के लिए अल्ट्रा-शुद्ध पानी के साथ पूरक करें।
- 4-6 सप्ताह के बाद, perlite से जड़ों को साफ और nodulation दक्षता की जांच करने के लिए एक दूरबीन का उपयोग कर नोड्यूल संख्या का निर्धारण।
7. प्लेट्स पर पी एंडरसनी प्लांटलेट की नोडुलेशन
-
सेलोफेन झिल्ली तैयार करें 28.
- एक वर्ग 12 सेमी x 12 सेमी पेट्री डिश में फिट करने के लिए सेलोफेन झिल्ली को काटें। शूटिंग बढ़ने के लिए स्थान की अनुमति देने के लिए शीर्ष पर थोड़ा छोटा झिल्ली काटें।
- सेलोफेन झिल्ली की पारगम्यता को बढ़ाने के लिए, 20 मिनट के लिए EDTA समाधान (1 ग्राम/एल) में झिल्ली को उबाललें, बाद में, EDTA को हटाने के लिए कम से कम 6x को डिमिनार्जिनाइज्ड पानी से धो लें।
नोट:के रूप में सूखी झिल्ली शिकन जब पानी के साथ संपर्क में करते हैं, सूखी झिल्ली एक के बाद एक समाधान में डूब. - झिल्ली को गोल काँच की प्लेट में पानी की पतली परत में क्षैतिज रूप से व्यवस्थित करें। झिल्ली को दो बार स्वावक्षण द्वारा बंध्याकरण करें।
- एक वर्ग 12 x 12 सेमी पेट्री डिश पर 1 ऑटोक्लेव्ड सेलोफेन झिल्ली रखें जिसमें आगर-ठोस ईकेएम माध्यम (तालिका 3, सारणी4) देखें। दो 3 सप्ताह पुराने जड़ें पी andersonii plantlets प्लेस (खंड 5 देखें) या 4 सप्ताह पुराने अंकुर (अनुभाग 1.1 देखें) झिल्ली के शीर्ष पर. केवल सफेद जड़ युक्तियाँ है कि जड़ों के साथ पौधों या अंकुर लेने के लिए सुनिश्चित करें, यह दर्शाता है कि इन जड़ों अभी भी बढ़ रहे हैं.
- धीरे से एक दूसरे सेलोफेन झिल्ली के साथ जड़ों को कवर, एक सैंडविच परत बनाने. लोचदार सील पन्नी के 3 परतों के साथ प्लेट सील। एल्यूमीनियम पन्नी के साथ प्लेटों के नीचे आधा लपेटें, प्रकाश जोखिम से जड़ों को कवर करने के लिए.
- एक जलवायु विकास कक्ष में प्लेटों इनक्यूबेट (28 डिग्री सेल्सियस, 16 h:8 h दिन: रात) 3-4 सप्ताह के लिए। समय के साथ रूट ग्रोथ का पालन करने के लिए रूट टिप्स की स्थिति को चिह्नित करें।
- यदि ईकेएम प्लेटलंबे ऊष्मायन के कारण सूखने लगती हैं, तो पौधों को जीवाणु संरोपण से कुछ दिन पहले ताजा ईकेएम प्लेटों में स्थानांतरित करें।
- चरण 6.1 पर वर्णित जीवाणु इनोकुलम तैयार करें।
- शीर्ष सेलोफेन झिल्ली निकालें और जड़ों के लिए राइजोबियम संस्कृति (ओडी600 डिग्री 0.025) के 1 एमएल लागू होते हैं। इसके बाद, टीकालगाए जड़ों पर एक नई सेलोफेन झिल्ली रखें। प्रकाश जोखिम से जड़ों को कवर करने के लिए एल्यूमीनियम पन्नी का उपयोग कर थाली के बाहर लपेटें।
- 4 सप्ताह के बाद, nodulation दक्षता निर्धारित करने के लिए एक दूरबीन का उपयोग कर नोड्यूल संख्या की जांच करें।
8. पाउच में पी एंडरसनी बीज की नोडेशन
- के रूप में अनुभाग 1.1 में वर्णित पी andersonii बीज अंकुरित. के बाद cotyledons पूरी तरह से उभरा है (एसएच-0 प्लेटों पर 28 डिग्री सेल्सियस पर 12 दिन), पाउच के लिए अंकुर स्थानांतरण.
- पाउच तैयार करने के लिए, कागज की बाती के मुड़े हुए भाग को फाड़ें और संशोधित ईकेएम माध्यम के 7 एमएल जोड़ें (तालिका 3, तालिका 4देखें)।
- कागज की दोनों शीटों के बीच जड़ों को रखकर 1 या 2 अंकुर डालें जो कागज की बाती और थैली के सामने प्लास्टिक शीट बनाते हैं।
- थैली के चारों ओर एल्यूमीनियम पन्नी तह द्वारा, प्रकाश जोखिम से जड़ों ढाल. उच्च आर्द्रता बनाए रखने के लिए पारदर्शी ढक्कन से ढके प्लास्टिक के बॉक्स में पाउच को निलंबित करें। एक जलवायु विकास के कमरे में बॉक्स प्लेस (28 डिग्री सेल्सियस, 16 ज:8 ज दिन:रात).
- बाँझ अल्ट्रा शुद्ध पानी जोड़कर पानी वाष्पीकरण के लिए क्षतिपूर्ति, इस तरह के रूप में है कि कागज की बाती नम रहता है (पाउच के तल पर खड़े पानी से बचें). पहले सप्ताह के बाद, यह आम तौर पर हर 4 दिन 2-3 मिलीलीटर जोड़ने की आवश्यकता है।
- चरण 6.1 पर वर्णित जीवाणु इनोकुलम तैयार करें।
- पाउच में 10-12 दिनों के लिए अंकुर उगाए जाने के बाद, राइजोबियम संस्कृति के 500 डिग्री सेल्सियस के साथ रूट सिस्टम को टीका करें (ओडी600 $ 0.025)।
- समय के माध्यम से नोड्यूल गठन का पालन करें। चार सप्ताह के बाद टीका, nodules गिना जा सकता है और nodulation दक्षता निर्धारित करने के लिए काटा.
9. नोड्यूल साइटोआर्किटेक्चर विश्लेषण
- फिक्सेटिव युक्त 2 एमएल ट्यूब में 10-15 नोड्यूल लीजिए (5% ग्लूटारैल्डिहाइड में 0.1 एम फॉस्फेट बफर, पीएच 7.2)। 1/2-1 एच के लिए वैक्यूम लागू करें और रात में 4 डिग्री सेल्सियस पर इनक्यूबेट करें। इस अवधि के दौरान, नमूने ट्यूब के नीचे करने के लिए सिंक.
नोट:स्थिर समाधान $ 2-4 सप्ताह पूर्व उपयोग के लिए 4 डिग्री सेल्सियस पर संग्रहीत किया जा सकता है। ऊतक fixative के साथ काम करते समय दस्ताने पहनने के लिए सुनिश्चित करें. - 0.1 एम फॉस्फेट बफर, पीएच 7.2 के साथ नोड्यूल 2x को धोएं। प्रत्येक धोने के कदम के बीच 10 मिनट के अंतराल लागू करें।
- बाद में 30%, 50%, 70%, और 100% इथेनॉल में इनक्यूबेट करके नमूनों को निर्जलित करें। यह सुनिश्चित करने के लिए कि सभी पानी नमूनों से हटा दिया जाता है, 100% इथेनॉल चरण 3x दोहराएँ। प्रत्येक निर्जलीकरण चरण के बीच 10 मिनट के अंतराल लागू करें।
- 100 एमएल HEMA (2-hydroxyethyl methacrylate) आधारित राल समाधान के साथ मिश्रित PEG400 के 2.5 एमएल करने के लिए Hardener I के 1 पैक जोड़कर बहुलकीकरण मिश्रण I (PM-I) तैयार करें। $ 15 मिनट के लिए समाधान हिलाओ पूरी तरह से Hardener मैं भंग करने के लिए. इसके बाद, -20 डिग्री सेल्सियस पर पीएम-I की दुकान करें।
- चरण 9.3 से इथेनॉल निकालें. और निम्नलिखित क्रम में नमूनों घुसपैठ: PM-I:100% इथेनॉल (1:3, v/v), PM-I:100% इथेनॉल (1:1, v/v), और PM-I:100% इथेनॉल (3:1, v/v)। 1/2-1 एच के लिए आरटी पर प्रत्येक समाधान में नमूने इनक्यूबेट करें या जब तक नमूने नीचे सिंक नहीं करते हैं।
- 100% PM-I समाधान में 4 डिग्री सेल्सियस पर रात भर के नमूनों को इनक्यूबेट करें।
- 15:1 (v/v) अनुपात में पीएम-I और हार्डनर II को मिलाकर बहुलकीकरण मिश्रण II तैयार करें। बहुलकीकरण समाधान के साथ प्लास्टिक मोल्ड भरें, मोल्ड के तल पर क्षैतिज नमूनों उन्मुख, और लोचदार सील पन्नी के एक टुकड़े के साथ कवर। हवा के बुलबुले के गठन से बचें।
नोट:के रूप में समाधान आरटी के संपर्क में बहुलक के लिए शुरू होता है, प्लास्टिक धारक में जितनी जल्दी हो सके नमूने उन्मुख करने के लिए प्रयास करें। बहुलकीकरण आरटी में रात भर ऊष्मायन के बाद पूरा हो गया है, या 37 डिग्री सेल्सियस पर 1 एच. - चरण 9.7 से लोचदार सील पन्नी कवर निकालें और बहुलक नमूनों के लिए एक धारक रखें। धारक को नमूनों में माउंट करने के लिए, मिथाइल मेथाक्रिलेट-आधारित राल पाउडर के 10 एमएल को मिथाइल मेथाक्रिलेट-आधारित राल समाधान के 5 एमएल में भंग करें। जल्दी से धारक के शीर्ष में छेद करने के लिए समाधान जोड़ें।
नोट:धूआं हुड में बहुलकीकरण कदम प्रदर्शन (आरटी पर 30 मिनट). - माइक्रोटोम खंड 4-5 मीटर की मोटाई के लिए नमूने. एक माइक्रोस्कोप स्लाइड को 58 डिग्री सेल्सियस गर्म प्लेट पर रखें और प्रत्येक स्लाइड में पानी की एक बड़ी बूंद जोड़ें। पानी के शीर्ष पर वर्गों प्लेस. एक बार पानी वाष्पित हो जाने के बाद, अनुभाग स्लाइड का पालन करेंगे।
- दाग स्लाइड 0.05 % (w/v) toluidine नीले में 2 मिनट के लिए डूब द्वारा. बाद में, अल्ट्रा शुद्ध पानी के साथ 3x स्लाइड कुल्ला. स्लाइड एक उज्ज्वल क्षेत्र माइक्रोस्कोप का उपयोग कर मनाया जा सकता है.
10. पी एंडरसनी प्लांटलेट का माइकोरहाइज़न
-
राइजोफैगस अनियमित बीजाणुओं के इनोकुलम को तैयार करें
- निम्नलिखित आकारों के साथ पॉलिएस्टर बुना फिल्टर के एक ढेर तैयार करें (ऊपर से नीचे): 210 डिग्री सेल्सियस, 120 डिग्री मीटर, और 36 डिग्री मी जाल आकार.
- पॉलिएस्टर फिल्टर के ढेर पर एक वाणिज्यिक बीजाणु निलंबन की आवश्यक राशि पिपेट। ऑटोक्लेव्ड विखनिजीकृत पानी के 100 एमएल के साथ फिल्टर 3x कुल्ला। बीजाणु36 डिग्री मीटर फिल्टर की सतह पर रखे जाते हैं।
नोट:संदूषण को रोकने के लिए स्तरीय क्रॉसफ्लो कैबिनेट में बीजाणु निलंबन तैयार करें। - पॉलिएस्टर स्टैक को अलग करें और केवल 36 डिग्री मीटर फ़िल्टर रखें। कम से कम 6x के लिए autoclaved deminralized पानी के साथ धोने के कदम को दोहराएँ.
- एक पेट्री डिश पर फिल्टर प्लेस और autoclaved deminralized पानी में बीजाणुओं को फिर से निलंबित. चरण 10-1.2 में प्रयुक्त बीजाणु निलंबन की मात्रा के बराबर जल की मात्रा का उपयोग करें। बीजाणु निलंबन को पाइपिंग द्वारा एक बाँझ ट्यूब में स्थानांतरित करें।
- एक कांच स्लाइड पर बीजाणु निलंबन के 20 डिग्री सेल्सियस की 5 बूँदें रखें और एक उज्ज्वल क्षेत्र माइक्रोस्कोप का उपयोग कर बीजाणुओं की संख्या की गणना। बीजाणुओं/एमएल के अनुपात में परिवर्तित करें और बीजाणु निलंबन को तब तक पतला करें जब तक कि यह 250 बीजाणुओं/एमएल तक न पहुंच जाए। बीजाणु निलंबन को 4 डिग्री सेल्सियस पर स्टोर करें।
- mycorrhization परख प्रदर्शन. इस अंत में, 800 ग्राम ऑटोक्लेव्ड रेत को 1/2-होगलैंड मध्यम के 70 एमएल के साथ बाँझ पारदर्शी पॉलीप्रोपीलीन बर्तन में जोड़ें (तालिकाओं कोदेखें 5 -6)। तेजी से मिलाते हुए बर्तन में सीधे रेत और मध्यम मिलाएं।
- प्रत्येक बर्तन में एक पी एंडरसनी प्लांटलेट रखें, और बीजाणु निलंबन के पिपेट 1 एमएल सीधे पी एंडरसनी प्लांटलेट की जड़ पर रखें। एक CRISPR नियंत्रण निर्माण के साथ बदल P. andersonii plantlets युक्त कई बर्तन शामिल करने के लिए सुनिश्चित करें (पूरक तालिका 1देखें).
- एक जलवायु विकास के कमरे में इनक्यूबेट बर्तन (28 डिग्री सेल्सियस, 16 h:8 h दिन: रात) 6 सप्ताह के लिए।
- बर्तन से पौधों को बाहर ले लो और संभव के रूप में ज्यादा रेत को दूर करने के लिए चल रहे पानी के साथ जड़ों को धो लें।
- 1 सेमी लंबे टुकड़ों में जड़ों को काटें और जड़ के टुकड़ों को 10% KOH (w/v) में 90 डिग्री सेल्सियस पर 20 मिनट के लिए उबालें। इसके बाद, उबले हुए जड़ों को 100 डिग्री मीटर जाल आकार के साथ एक सेल छलनी पर रखें और 50 एमएल पानी के साथ 3x कुल्ला करें।
- लैक्टोग्लिसरोल में 0.05% (w/v) trypan blue (300 एमएल लैक्टिक एसिड; 300 एमएल ग्लिसरॉल; और 400 एमएल डिमिनरलीकृत पानी) के साथ दाग जड़ें 5 मिनट के लिए 90 डिग्री सेल्सियस पर पानी के स्नान या हीटिंग ब्लॉक में। बाद में, 30% ग्लिसरोल के लिए जड़ों को स्थानांतरित करें। रूट नमूने आरटी में संग्रहीत किया जा सकता है.
- एक माइक्रोस्कोप स्लाइड पर 15-25 जड़ टुकड़े रखें। 30% ग्लिसरोल जोड़ें और एक कवर ग्लास के साथ कवर और प्रेस जब तक रूट टुकड़े फ्लैट हो जाते हैं. एक उज्ज्वल क्षेत्र माइक्रोस्कोप का उपयोग कर जड़ टुकड़े का निरीक्षण करें और mycorrhizal उपनिवेशन स्कोर.
नोट:mycorrhization स्कोर करने के लिए एक विधि Trouvelot एट अल29के अनुसार वर्णित है. इस विधि में कई वर्गों (%F, %M, और %A) का उपयोग किया जाता है, जो प्रत्येक रूट खंड के mycorrhizal उपनिवेशन के स्तर के तेजी से आकलन और arbuscules की बहुतायत की अनुमति देता है.
Representative Results
पी. एंडरसनी टीरीज़ को एक वातानुकूलित ग्रीनहाउस में 28 डिग्री सेल्सियस और $85% सापेक्ष आर्द्रता में उगाया जा सकता है (चित्र 1क)। इन परिस्थितियों में, पेड़ रोपण के बाद 6-9 महीने में फूल शुरू करते हैं। महिला पी andersonii फूल जामुन है कि प्रत्येक एक बीज होता है का उत्पादन. परिपक्वता के दौरान, जामुन रंग बदल; पहले हरे से सफेद और बाद में सफेद से भूरे रंग तक (चित्र 1ख) । पके हुए भूरे जामुन से निकाले गए बीज, 10 दिन के तापमान चक्र के बाद अच्छी तरह से अंकुरित होते हैं और एसएच-0 प्लेटों पर 7 दिन का ऊष्मायन (चित्र 1C)। अंकुरित बीज युवा अंकुरों में विकसित होते हैं जिनका उपयोग 4 सप्ताह के बाद प्रयोग के लिए किया जा सकता है (चित्र 1D) ।
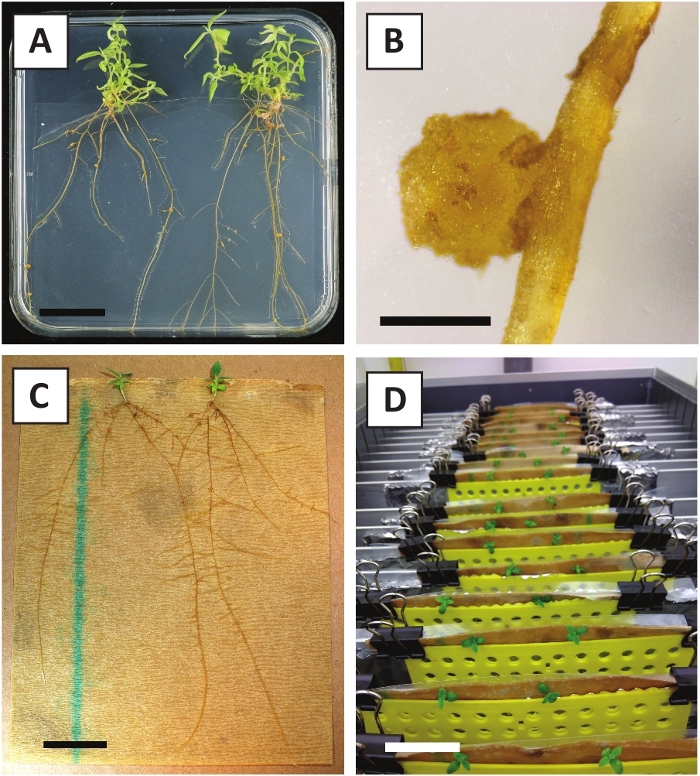
हम पहले से पता चला है कि डंठल और युवा पी andersonii उपजी के क्षेत्रों कुशलता से ए tumefaciens तनाव AGL110का उपयोग कर तब्दील किया जा सकता है. परिवर्तन प्रक्रिया के प्रारंभ में, ऊतक एक्सप्लांट्स को 21 डिग्री सेल्सियस (चित्र2क) में 2 दिनों के लिए ए tumefaciens के साथ सह-कृषि करते हैं। लंबे समय तक सह-कृषि का परिणाम ए tumefaciens द्वारा ऊतक एक्सप्लांट्स के अति उपनिवेशन में होता है और इसलिए इसे रोका जाना चाहिए (चित्र2ख)। सह-कृषि अवधि के बाद, ऊतक एक्सप्लांट्स चयनात्मक मीडिया में स्थानांतरित किए जाते हैं, जो रूपांतरित ऊतक के विकास को बढ़ावा देता है। दो से तीन सप्ताह बाद, छोटे हरे सूक्ष्म कैली आम तौर पर मूल घाव की सतह के साथ मनाया जाता है (चित्र 2C) . इन कैली को परिवर्तन प्रक्रिया शुरू होने के 6-8 सप्ताह बाद 1 या अधिक putatively-transformed shoots विकसित करना जारी रखना चाहिए (चित्र 2D) . इस स्तर पर, परिवर्तन क्षमता आम तौर पर परिपक्व और आंशिक रूप से वुडी शाखाओं से लिया ऊतक explants के साथ शुरू की रूपांतरणों के लिए $ 10-30% से लेकर (तालिका 7) . यदि परिवर्तन ों की शाखाओं है कि अभी तक फूल असर नहीं कर रहे हैं के युवा और तेजी से बढ़ रही सुझावों से लिया explants के साथ शुरू कर रहे हैं, $65-75% की रूपांतरण क्षमता प्राप्त किया जा सकता है (तालिका 7). कभी-कभी, एक एक्सप्लांट के पक्ष में सफेद कैली का गठन किया जाता है जो माध्यम के संपर्क में नहीं है और इसलिए, कनमिसिन चयन का अनुभव नहीं करते हैं। ये कैली प्रायः ट्रांसजेनिक नहीं होते हैं और इन कैली से बनने वाली कोई भी गोली आम तौर पर कानामाइसिन युक्त माध्यम (चित्र2E) के साथ सीधे संपर्क के बाद विरंजन और मर जाएगी। यदि रूपांतरण दर कम है और/या प्रारंभिक सामग्री उपऑप्टिमल थी, तो ऊतक के टुकड़े भूरे रंग के हो सकते हैं (चित्र 2 एफ) और ए tumefaciens द्वारा अति प्रसार से पीड़ित हो सकते हैं (चित्र 2G)। ए tumefaciens फैलने से रोकने के लिए और पास के explants बढ़ रहा है, माध्यम के नियमित रूप से जलपान की आवश्यकता है, और गंभीर रूप से संक्रमित explants को दूर करने की आवश्यकता है. एक बार अलग-अलग ट्रांसजेनिक शूट को प्रचार माध्यम में रखा जाता है, तो ए टयूमफैसिएन्स द्वारा अति प्रसार आमतौर पर अब नहीं होता है (चित्र 2ह) । ट्रांसजेनिक शूट को इन विट्रो प्रोपेगैंडा के माध्यम से गुणा किया जा सकता है, जो एक महीने की अवधि में दसियों शूटिंग को जन्म देगा (चित्र 3ए-बी)। इन शूटों को रूटिंग मीडियम पर रखा जा सकता है, जो र्2 सप्ताह के बाद जड़ निर्माण को प्रेरित करता है (चित्र 3ब्-डी)। जड़ें दार्चित पौधों का प्रयोग करने के लिए बाद में उपयोग किया जा सकता है।
नॉकआउट उत्परिवर्ती रेखाएँ बनाने के लिए, हम CRISPR/Cas9-मध्यस्थ म्यूटजेनेसिस का उपयोग करते हैं. इस उद्देश्य के लिए, हम एक द्विआधारी वेक्टर का उपयोग करते हैं जिसमें कानामीसिन प्रतिरोध जीन NPTIIहोता है, जो CaMV35S प्रमोटर द्वारा संचालित एक Cas9-एन्कोडिंग अनुक्रम और लक्ष्य जीन के अनुसार 2 sgRNAs जो AtU6p छोटे आरएनए प्रमोटर20से व्यक्त किया जाता है। P. Andersonii के CRISPR/Cas9-मध्यस्थ मटजनन के लिए प्रयुक्त निर्माण का आलेखीय निरूपण चित्र 4कमें दिया गया है। इस विधि का उपयोग करते हुए, जीनोम संपादन putatively-transformed shoots10के $40% में मनाया जाता है। उत्परिवर्ती लाइनों की पहचान करने के लिए, putatively बदल गोली लक्षित क्षेत्र फैले प्राइमर का उपयोग कर sgRNA लक्ष्य साइट (ओं) पर उत्परिवर्तन के लिए genotyped हैं. अपेक्षित परिणामों का एक उदाहरण चित्र 4में दिया गया है। के रूप में जेल electrophoresis के बाद लिया तस्वीर से देखा जा सकता है, कई नमूने जंगली प्रकार के लिए इसी तरह के आकार के साथ एक पीसीआर amplicon का उत्पादन (चित्र 4B)। इन पौधों छोटे indels कि agarose जेल electrophoresis द्वारा कल्पना नहीं किया जा सकता है या Cas9 एंजाइम द्वारा अप्रकाशित रह सकते हैं. इसके अतिरिक्त, कई नमूने ऐसे बैंड्स को प्राप्त करते हैं जो जंगली प्रकार से आकार में भिन्न होते हैं (उदा., रेखाएँ 2, 4, 7 और 8 चित्र 4Bमें). इन पंक्तियों में, 1 (रेखा4, 7 और 8) या दोनों (लाइन 2) alleles बड़े indels कि आसानी से कल्पना की जा सकती हो होते हैं. पीसीआर amplicon अनुक्रमण के बाद लक्ष्य स्थल (ओं) पर उत्परिवर्तनों की सटीक प्रकृति का पता चला है। जैसा कि चित्र 4Cसे देखा जा सकता है, 1-4 बीपी के दोनों छोटे indels, साथ ही साथ, बड़े विलोपन CRISPR/Cas9 mutagenesis के बाद प्राप्त किया जा सकता है. चित्र 4Cमें, रेखा 1 का अनुक्रम जंगली प्रकार के समान होता है, जो यह दर्शाता है कि यह पंक्ति संपादन से बच गई और इसलिए उसे छोड़ दिया जाना चाहिए. जिन पंक्तियों में उत्परिवर्तन होते हैं, उनमें विषमयुग्मज, समयुग्मज और द्वि-एलीलिक म्यूटेंट ोंकी पहचान की जा सकती है (चित्र 4ब्) । हालांकि, विषमयुग्मज उत्परिवर्ती आम तौर पर दुर्लभहैं 10. होमोयुग्मज या द्वि-एलीलिक नॉकआउट उत्परिवर्ती को फीनोटाइपिक विश्लेषण के लिए पर्याप्त सामग्री प्राप्त करने के लिए वनस्पति रूप से प्रचारित किया जा सकता है। के रूप में phenotypic विश्लेषण टी0 पीढ़ी में किया जाता है, यह है कि उत्परिवर्ती लाइनों झंकार हो सकता है की जाँच करने के लिए महत्वपूर्ण है. यह अंत करने के लिए, जीनोटाइपिंग प्रत्येक उत्परिवर्ती लाइन से लिया कम से कम 3 अलग अलग नमूनों पर दोहराया जा करने की जरूरत है. यदि जीनोटाइपिंग परिणाम एक-दूसरे के समान हैं और मूल जीनोटाइपिंग नमूना (उदाहरण के लिए, चित्र 4Dमें रेखा 8), रेखा सजातीय रूप से उत्परिवर्तित होती है और आगे के विश्लेषण के लिए उपयोग की जा सकती है। हालांकि, अगर जीनोटाइपिंग परिणाम स्वतंत्र नमूनों के बीच अलग होते हैं (जैसे, चित्र4Dमें रेखा 4), उत्परिवर्ती रेखा झंकार है और इसे खारिज करने की आवश्यकता है।
M. plurifarium BOR2 के साथ पी एंडरसनी का टीका जड़ नोड्यूल के गठन में परिणाम है (चित्र 5) . जैसा कि चित्र 5कमें देखा जा सकता है, ये पिंड मूल प्रणाली के साथ वितरित किए जाते हैं। पी एंडरसनी के पिंड हल्के भूरे रंग के होते हैं लेकिन उनके आकार के आधार पर जड़ ऊतक से आसानी से भेदभाव किया जा सकता है (चित्र 5ख) गमलों में टीका लगाने के प्रयोगों और 4-6 सप्ताह के लिए बाद में वृद्धि आम तौर पर $ 10-30 नोड्यूल के गठन में परिणाम (चित्र 6A) . ईकेएम प्लेट विकसित पी एंडरसनी पौधों को टीका लगाने के 4 सप्ताह बाद टीका लगाने के बाद इसी प्रकार की संख्या में नोड्यूल का गठन किया जाता है (चित्र 6क) । पाउच में, पी एंडरसनी अंकुर आमतौर पर 5 सप्ताह के बाद टीका लगाने पर 5-15 नोड्यूल बनाते हैं (चित्र 5सी-डी, 6ए)। नोड्यूल साइटोआर्किटेक्चर का विश्लेषण करने के लिए, नोड्यूल को उज्ज्वल क्षेत्र माइक्रोस्कोपी का उपयोग करके काट दिया और मनाया जा सकता है। चित्र 6ख एक पी एंडरसनी ग्रंथिका के मध्य के माध्यम से एक अनुदैर्घ्य खंड का एक उदाहरण दिखाता है। यह खंड पी एंडरसनी नोड्यूल के केंद्रीय संवहनी बंडल को दर्शाता है, जो संक्रमित कोशिकाओं से युक्त नोड्यूल लोबों से घिरा हुआ है (चित्र 6ख)।
पी एंडरसनी पौधों को भी mycorrhized किया जा सकता है. आर अनियमितकेसाथ टीका लगाने के 6 सप्ताह बाद , माइकोरिज़ल उपनिवेशन आवृत्ति आमतौर पर 80% तक पहुँचती है (चित्र 6 C) . इस समय में सामान्यतः कोशिकाओं में आर्बुस्कुल्स होते हैं (चित्र 6 ब्)। आर्बसक्लेस युक्त पी एंडरसनी मूल खंड की एक प्रतिनिधि छवि चित्र 6Dमें दिखाई गई है।

चित्र 1: एक के प्रतिनिधि छवियों पी एंडरसनी पेड़, बीज और अंकुर। (क) छह महीने का पी. (ख) प्रतिनिधि छवि परिपक्वता के विभिन्न चरणों में पी एंडरसनी जामुन का चित्रण. युवा पी andersonii जामुन (unripe) हरे रंग से सफेद करने के लिए रंग बदल जाएगा और अंत में पकने पर भूरे रंग (पीप) के लिए. (C) पी एंडरसनी बीज 1 सप्ताह के लिए एसएच -0 माध्यम पर incubated। एक काला वृत्त अंकुरित अंकुर को इंगित करता है। (डी) एसएच-0 माध्यम में उगाए गए चार सप्ताह पुराने पी एंडरसनी पौध। स्केल बार ्स्स 25 से.मी. (ए) और 1 से.मी. कृपया इस चित्र का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
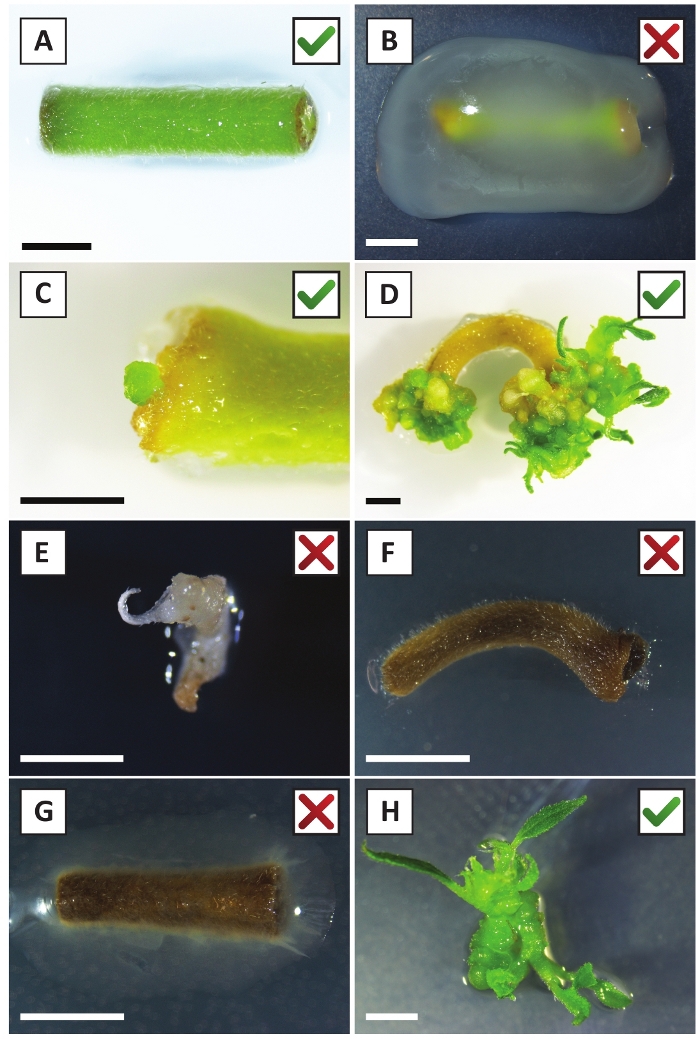
चित्र 2: स्थिर रूपांतरण प्रक्रिया के विभिन्न चरणों में explants के प्रतिनिधि छवियों. (ए) ए tumefaciensके साथ एक्सप्लांट सह -cultivated . (ख ) पहले 2 सप्ताह के बाद परिवर्तन के दौरान ए tumefaciens द्वारा अधिविकसित एक्सप्लांट. (ग) सह-कृषि के बाद 2.5 सप्ताह में एक एक्सप्लांट के घाव स्थल के निकट ट्रांसजेनिक माइक्रो-कॉलस का गठन किया गया। (घ) सह-कृषि के 6 सप्ताह बाद एक एक्सप्लांट की प्रतिनिधि छवि जिसमें (ट्रांसजेनिक) कैली से शूट के उद्भव को दर्शाता है। (ई) एक गोली है कि सफेद हो जाता है और अंत में kanamycin युक्त माध्यम के साथ सीधे संपर्क में मर जाता है की प्रतिनिधि छवि. इस शूटिंग सबसे अधिक संभावना गैर ट्रांसजेनिक है और जब explant से जुड़ी kanamycin चयन से बच. (च) एक असफल रूप से परिवर्तित एक्सप्लांट की प्रतिनिधि छवि। (जी) ए . tumefaciensद्वारा अधिविकसित एक असफल रूप से रूपांतरित एक्सप्लांट की प्रतिनिधि छवि . (एच) ए . tumefaciensके साथ 8 सप्ताह के बाद सह-कृषि पर प्रचार माध्यम पर उगाया गया एकल ट्रांसजेनिक शूट . स्केल बार बराबर 2.5 मिमी बक्से हरे रंग की जांच के निशान या लाल पार युक्त क्रमशः explants के सफल या असफल परिवर्तन का संकेत मिलता है। कृपया इस चित्र का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.

चित्र 3: के प्रतिनिधि चित्र इन विट्रो संचरण. (ए) प्रचार माध्यम पर उगाए जाने वाले शूट। प्लेटें ताज़ा किए जाने के 1 सप्ताह बाद छवि ली गई थी। (ख) प्रचार माध्यम पर उगाए गए शूट। प्लेटों को ताज़ा किए जाने के 4 सप्ताह बाद छवि ली गई थी। (ग) जड़ माध्यम पर रखे गए ताजा कट शूट। (घ) 2 सप्ताह तक रूटिंग माध्यम पर इनक्यूबेट किया गया शूट करता है। जड़ों की उपस्थिति को नोट की। स्केल बार्स 2.5 सेमी के बराबर हैं. कृपया इस आकृति का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
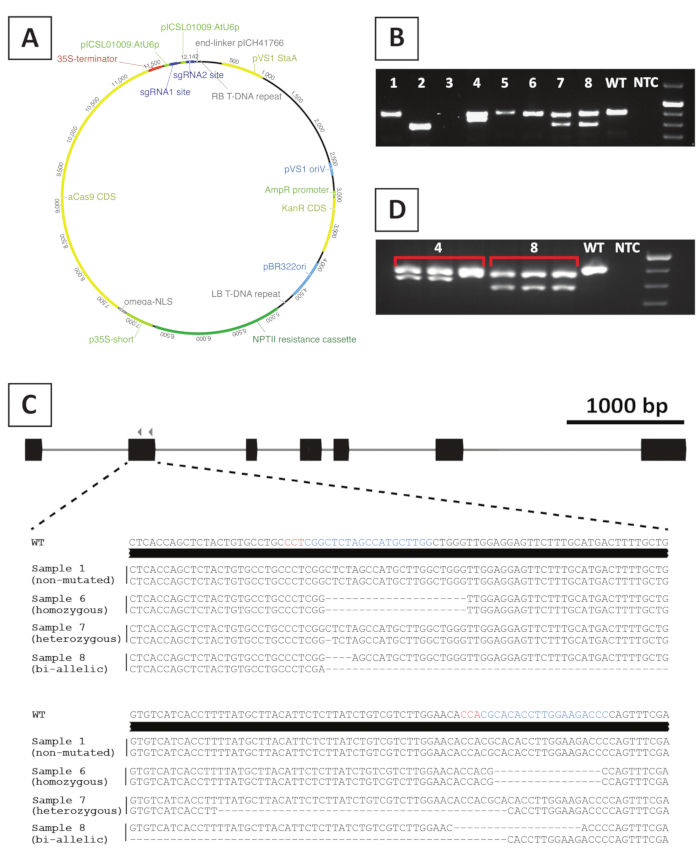
चित्र 4: P. andersonii T0 ट्रांसजेनिक CRISPR/Cas9 उत्परिवर्ती लाइनों के जीनोटाइपिंग के बाद प्रतिनिधि परिणाम। (क) पी एंडरसनीके CRISPR/Cas9-मध्यस्थ मटजनन के लिए प्रयुक्त द्विआधारी वेक्टर का प्रतिनिधि मानचित्र . (बी) पीसीआर के बाद प्रतिनिधि परिणाम संभावित CRISPR/Cas9 उत्परिवर्ती लाइनों के जीनोटाइपिंग sgRNA लक्ष्य साइट (एस) फैले प्राइमर का उपयोग कर। दिखाया amplicons के agarose जेल इलेक्ट्रोफोरोसिस के बाद एक छवि है. अलग-अलग ट्रांसजेनिक लाइनों से लिए गए नमूनों को संख्याओं द्वारा दर्शाया जाता है। वाइल्ड टाइप (डब्ल्यूटी) और कोई टेम्पलेट नियंत्रण (एनटीसी) क्रमशः सकारात्मक और नकारात्मक नियंत्रण वाले लेन को इंगित करते हैं। (ग) CRISPR/Cas9-मध्यस्थ जीन संपादन के बाद प्राप्त उत्परिवर्ती एलेल्स का योजनाबद्ध प्रतिनिधित्व। नीले और लाल रंग में प्रकाश डाला sgRNA लक्ष्य साइटों और पीएएम दृश्यों, क्रमशः कर रहे हैं। (डी) संभावित झंकार उत्परिवर्ती लाइनों के लिए पीसीआर आधारित स्क्रीनिंग के बाद प्रतिनिधि परिणाम। दिखाया उत्परिवर्ती लाइनों से लिया 3 अलग-अलग नमूनों की agarose जेल इलेक्ट्रोफोरोसिस के बाद एक छवि है 4 और 8. ध्यान दें कि ट्रांसजेनिक उत्परिवर्ती लाइन 4 झंकार है. कृपया इस चित्र का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
चित्र 5: प्लेटों और पाउच में नोडुलेशन assays के प्रतिनिधि छवियों. (क) आगर-ठोस ईकेएम माध्यम वाली प्लेटों पर नोडुलेशन और 4 सप्ताह के लिए एम प्लरिफेरियम BOR2 के साथ टीका लगाया गया है। (ख) पी एंडरसनी रूट नोड्यूल की प्रतिनिधि छवि। छवि एम plurifarium BOR2 के साथ 4 सप्ताह के बाद टीका पर लिया गया था. (सी) तरल ईकेएम माध्यम वाले पाउच में नोडुलेशन। बीज 5 सप्ताह के लिए Bradyrhizobium sp. Kelud2A4 के साथ टीका लगाया गया. (डी) पाउच में नोडुलेशन के लिए उपयोग की जाने वाली पूर्ण सेटअप की प्रतिनिधि छवि। स्केल बार्स में 2.5 सेमी (ए , सी), 1 मिमी (बी) और 5 सेमी (डी) के बराबर हैं । कृपया इस चित्र का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
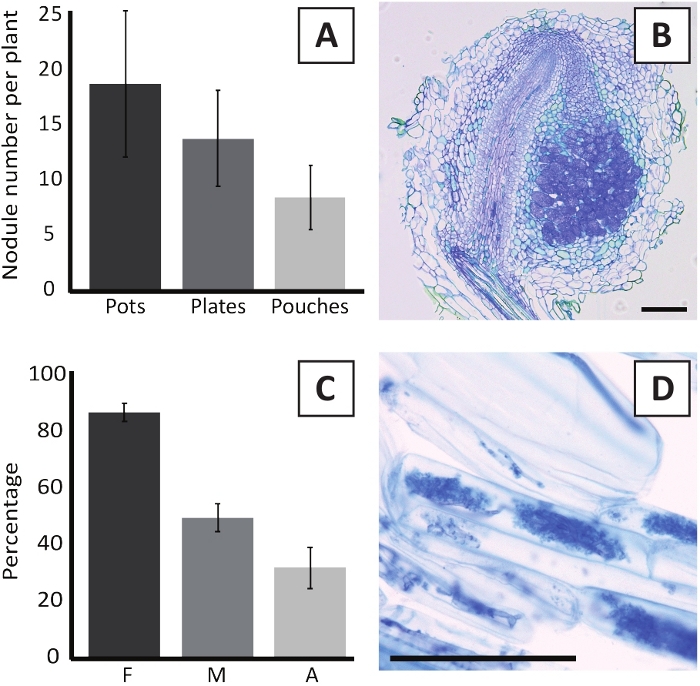
चित्र 6: nodulation और mycorrhization परख के प्रतिनिधि परिणाम. (क) प्रतिनिधि बार ग्राफ जिसमें बर्तन में या प्लेटों में एम प्लूरिफेरियम BOR2 के साथ 4 सप्ताह के बाद टीका लगाए जाने के बाद प्रति पौधे बनाए गए नोड्यूल की संख्या को दर्शाता है और पाउच में ब्रैडीरिजोबियम एसपी केलुड2ए4 के साथ 5 सप्ताह के बाद टीका लगाया गया है। डेटा माध्य का प्रतिनिधित्व करता है - SD (n ] 10). (B) M. plurifarium BOR2 के साथ 4 सप्ताह के बाद टीका के बाद एक नोड्यूल के माध्यम से एक अनुदैर्घ्य अनुभाग के प्रतिनिधि छवि. अनुभाग toluidine नीले रंग के साथ दाग है. (ग) प्रतिनिधि बार ग्राफ जिसमें mycorrhization की मात्रा को दर्शाने के लिए. Trouvelot एट अल29 के अनुसार परिमाणित चर एफ हैं, विश्लेषण रूट टुकड़े कि mycorrhized कर रहे हैं की आवृत्ति; एम, संक्रमण की तीव्रता; एक, कुल जड़ प्रणाली में परिपक्व arbuscules की बहुतायत. Mycorrhization आर अनियमित (तनाव DAOM197198) के साथ 6 सप्ताह के बाद टीका पर परिमाणित किया गया था. डेटा माध्य का प्रतिनिधित्व करता है - SD (n ] 10). (घ) प्रचर्म आर्बसक्यूल्स की प्रतिनिधि छवि जो पी . एंडरसनी रूट कॉर्टिकल कोशिकाओं में 6 सप्ताह के बाद आर अनियमितोंके साथ टीका लगाया जाता है . स्केल बार्स बराबर 75 डिग्री मी. कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
| यौगिक | एसएच-0 | एसएच-10 | प्रचार माध्यम | रूटिंग माध्यम | घुसपैठ माध्यम |
| एसएच-बेसल नमक माध्यम | 3.2 ग्राम | 3.2 ग्राम | 3.2 ग्राम | 3.2 ग्राम | 3.2 ग्राम |
| एसएच विटामिन मिश्रण | 1 ग्राम | 1 ग्राम | 1 ग्राम | 1 ग्राम | 1 ग्राम |
| Sucrose | - | 10 ग्राम | 20 ग्राम | 10 ग्राम | 10 ग्राम |
| बीएपी (1 मिलीग्राम/ | - | - | 1 एमएल (4.44 डिग्री सेल्सियस) | - | - |
| आईबीए (1 मिलीग्राम/ | - | - | 100 $L (0.49 $M) | 1 एमएल (4.92 डिग्री सेल्सियस) | - |
| NAA (1 मिलीग्राम/ | - | - | - | 100 $L (0.54 $M) | - |
| 1 एमईएस पीएच $5.8 | 3 एमएल | 3 एमएल | 3 एमएल | 3 एमएल | 3 एमएल |
| 1 एम कोह | पीएच को 5.8 तक समायोजित करें | पीएच को 5.8 तक समायोजित करें | पीएच को 5.8 तक समायोजित करें | पीएच को 5.8 तक समायोजित करें | पीएच को 5.8 तक समायोजित करें |
| दैशिन ऐगार | 8 ग्राम | - | 8 ग्राम | 8 ग्राम | - |
तालिका 1: Schenk-Hildebrandt आधारित30 मीडिया की संरचना पी andersonii अंकुर, स्थिर परिवर्तन, और इन विट्रो प्रचार के लिए इस्तेमाल किया. तरल स्टॉक जोड़ने से पहले अल्ट्रा-शुद्ध पानी के 750 एमएल में ठोस यौगिकों को भंग करें। बाद में, पूरा माध्यम को 1 एल तैयार करें BAP, आईबीए, एनएए स्टॉक में 0.1 एम KOH और -20 oC पर स्टोर करें।
| ऑटोक्लेविंग से पहले: | ||
| यौगिक | प्रति लीटर राशि | अंतिम एकाग्रता |
| मैनिटॉल | 5 ग्राम | 27.45 एम.एम. |
| ना-ग्लूकोनेट | 5 ग्राम | 22.92 मी. |
| खमीर निकालने | 0.5 ग्राम | - |
| MgSO4$7H2हे | 0.2 ग्राम | 0.81 एम.एम. |
| Nacl | 0.1 ग्राम | 1.71 एम.एम. |
| कश्मीर2एचपीओ4 | 0.5 ग्राम | 2.87 एम.एम. |
| ऑटोक्लेविंग के बाद: | ||
| यौगिक | प्रति लीटर राशि | अंतिम एकाग्रता |
| 1.5 एम CaCl2 | 1 एमएल | 1.5 एम.एम. |
तालिका 2: खमीर-मैनिटॉल (वाईईएम) माध्यम की संरचना जिसका उपयोग राइजोबियम उगाने के लिए किया जाता है। 7.0 करने के लिए पीएच समायोजित करें और 1 एल करने के लिए अल्ट्रा शुद्ध पानी के साथ भरें। आगर-ठोस YEM माध्यम तैयार करने के लिए, autoclaving से पहले 15 ग्राम माइक्रोनगर जोड़ें।
| ऑटोक्लेविंग से पहले: | |||
| यौगिक | स्टॉक एकाग्रता | प्रति लीटर मध्यम राशि | अंतिम एकाग्रता |
| KH2पीओ4 | 0.44 एम | 2 एमएल जोड़ें | 0.88 एम.एम. |
| कश्मीर2एचपीओ4 | 1.03 एम | 2 एमएल जोड़ें | 2.07 एम.एम. |
| 500x सूक्ष्म तत्वों शेयर समाधान | - | 2 एमएल जोड़ें | - |
| एमईएस पीएचजेड 6.6 | 1 एम | 3 एमएल जोड़ें | 3 एम.एम. |
| Hcl | 1 एम | पीएच को 6.6 तक समायोजित करें | - |
| अल्ट्रा शुद्ध पानी | - | 990 एमएल तक भरें | - |
| ऑटोक्लेविंग के बाद: | |||
| यौगिक | स्टॉक एकाग्रता | प्रति लीटर मध्यम राशि | अंतिम एकाग्रता |
| MgSO4$7H2हे | 1.04 एम | 2 एमएल | 2.08 एम.एम. |
| ना2SO4 | 0.35 एम | 2 एमएल | 0.70 एम.एम. |
| एनएच4सं3 | 0.18 एम | 2 एमएल | 0.36 एम.एम. |
| CaCl2$2H2हे | 0.75 एम | 2 एमएल | 1.5 एम.एम. |
| Fe(III)-सिक्रेट | 27 एमएम | 2 एमएल | 54 डिग्री मी. |
तालिका 3: 1 एल संशोधित EKM मध्यम31 की संरचना P. andersonii nodulation परख के लिए इस्तेमाल किया. 500x सूक्ष्म-तत्व स्टॉक विलयन की संरचना सारणी 4 में सूचीबद्ध है। 2% agar-solidified EKM माध्यम तैयार करने के लिए, autoclaving से पहले 20 ग्राम Daishin agar जोड़ें. ऑटोक्लेव MgSO4$7H2O, ना2SO4, CaCl2$2H2O, और Fe(III)-साइटरेट स्टॉक बाँझ करने के लिए। छान NH4NO3 स्टॉक समाधान स्टरलाइज़ करने के लिए।
| यौगिक | प्रति लीटर राशि | स्टॉक एकाग्रता |
| एमएनएसओ4 | 500 मिलीग्राम | 3.31 एम.एम. |
| [nSO4]7H2हे | 125 मिलीग्राम | 0.43 एम.एम. |
| CuSO4$5H2हे | 125 मिलीग्राम | 0.83 एम.एम. |
| एच3बीओ3 | 125 मिलीग्राम | 2.02 मीटर |
| ना2मो4]2H2हे | 50 मिलीग्राम | 0.21 एम.एम. |
तालिका 4: संशोधित EKM माध्यम तैयार करने के लिए इस्तेमाल किया 500x सूक्ष्म तत्वों स्टॉक समाधान की संरचना. सूक्ष्म तत्वों के स्टॉक समाधान को 4 डिग्री सेल्सियस पर संग्रहीत करें।
| यौगिकों | स्टॉक एकाग्रता | प्रति लीटर मध्यम राशि | अंतिम एकाग्रता |
| कश्मीर2एचपीओ4 | 20 एमएम | 1 एमएल | 0.2 एम.एम. |
| एनएच4सं3 | 0.28 एम | 10 एमएल | 2.8 एम.एम. |
| एमजीएसओ4 | 40mm | 10 एमएल | 0.4 एम.एम. |
| कश्मीर2एसओ4 | 40mm | 10 एमएल | 0.4 एम.एम. |
| फे (II)-EDTA | 9 एम.एम. | 10 एमएल | 0.9 एम.एम. |
| CaCl2 | 80 एमएम | 10 एमएल | 0.8 एम.एम. |
| 50x सूक्ष्म तत्वों शेयर समाधान | - | 10 एमएल | - |
तालिका 5: 1/2-Hoagland32 मध्यम की संरचना mycorrhization परख के लिए इस्तेमाल किया. 50x सूक्ष्म-तत्व स्टॉक विलयन की संरचना सारणी 6 में सूचीबद्ध है। Fe(II)-EDTA समाधान FeSO4[7H2O (9 m) और Na2के संयोजन से तैयार करें EDTA (9 mM) 1 स्टॉक समाधान में, और 4 डिग्री सेल्सियस पर दुकान. 1 M KOH का उपयोग करके 6.1 माध्यम के पीएच को समायोजित करें और 1 L तक अल्ट्रा-शुद्ध जल से भरें।
| यौगिकों | प्रति लीटर राशि | स्टॉक एकाग्रता |
| एच3बीओ3 | 71.1 मिलीग्राम | 1.15 एम.एम. |
| MnCl2$4H2हे | 44.5 मिलीग्राम | 0.22 मीटर |
| CuSO4$5H2हे | 3.7 मिलीग्राम | 23.18 डिग्री सेल्सियस |
| [एनसीएल2 | 10.2 मिलीग्राम | 74.84 $M |
| ना2मो4$2H2हे | 1.2 मिलीग्राम | 4.96 $M |
तालिका 6: 50x सूक्ष्म तत्वों स्टॉक समाधान की संरचना 1/2-Hoagland माध्यम तैयार करने के लिए इस्तेमाल किया.
| एक्सप्लांट्स की आयु | परिवर्तन दक्षता |
| युवा | 69.4 - 6.2% (द ] 2) |
| परिपक्व | 18.3 - 10.2% (द ] 15) |
तालिका 7: P. andersoniiकी परिवर्तन दक्षता | यहाँ, रूपांतरण दक्षता explants कि फार्म के प्रतिशत के रूप में परिभाषित किया गया है कम से कम 1 transgenic callus या गोली मार. परिवर्तन दक्षता 6 सप्ताह के बाद परिवर्तन पर रन बनाए गए थे और मतलब के रूप में चित्रित किया गया है - एसडी n परिवर्तन प्रयोगों की संख्या जिसमें से परिवर्तन दक्षता निर्धारित किया गया था इंगित करता है.
पूरक फ़ाइल 1: स्तर 1 और स्तर 2 CONSTRUCT के लिए उपयोग किए जाने वाले Constructs का अवलोकन/ इस फ़ाइल को डाउनलोड करने के लिए कृपया यहाँ क्लिक करें.
Discussion
फलियां और दूर से संबंधित कैनाबसी जीनस पैरास्पोनिया पौधों की प्रजातियों के केवल दो क्लैडों का प्रतिनिधित्व करते हैं जो नाइट्रोजन-फिक्सिंग राइजोबिया के साथ एंडोसिबायोटिक संबंध स्थापित करने और रूट नोड्यूल बनाते हैं। दोनों clades की प्रजातियों के बीच तुलनात्मक अध्ययन अत्यधिक इस सहजीवन की अनुमति कोर आनुवंशिक नेटवर्क में अंतर्दृष्टि प्रदान करने के लिए प्रासंगिक हैं. वर्तमान में, आनुवंशिक अध्ययन मुख्य रूप से फलियां में किया जाता है; विशेष रूप से दो मॉडल प्रजातियों एम truncatula और एल Japonicus. एक अतिरिक्त प्रयोगात्मक मंच प्रदान करने और एक nodulating गैर-लेगम के साथ तुलनात्मक अध्ययन की सुविधा के लिए, हम यहाँ स्थिर परिवर्तन और पी andersoniiमें आनुवंशिक विश्लेषण रिवर्स के लिए एक विस्तृत प्रोटोकॉल का वर्णन. प्रस्तुत प्रोटोकॉल टी0 ट्रांसजेनिक पी एंडरसनी लाइनों के विट्रो प्रचार में उपयोग करता है, phenotypic विश्लेषण ए tumefaciens सह खेती के बाद 4 महीने के भीतर शुरू करने की अनुमति. फलियां33के स्थिर परिवर्तन के लिए स्थापित किए गए वर्तमान प्रोटोकॉलों की तुलना में यह काफी तेज है . यह पी andersonii एक आकर्षक अनुसंधान मॉडल बनाता है.
यहाँ वर्णित प्रोटोकॉल में कई महत्वपूर्ण चरण हैं. जिनमें से पहला बीज अंकुरण से संबंधित है। अंकुरण के लिए पी एंडरसनी बीज तैयार करने के लिए, बीज जामुन से अलग करने की आवश्यकता है। यह ऊतक कागज के एक टुकड़े पर या एक चाय छलनी के अंदर के खिलाफ जामुन रगड़ द्वारा किया जाता है. बीज कोट को नुकसान को रोकने के लिए इस प्रक्रिया को धीरे से किया जाना चाहिए। यदि बीज कोट क्षतिग्रस्त हो जाता है, ब्लीच नसबंदी के दौरान बीज में प्रवेश कर सकता है, जो बीज व्यवहार्यता कम कर देता है. बीज निद्रा को तोड़ने के लिए, बीज एक 10 दिन के तापमान चक्र के अधीन हैं। हालांकि, इस उपचार के बावजूद, अंकुरण पूरी तरह से सिंक्रनाइज़ नहीं है। आम तौर पर, पहले बीज 7 दिनों के बाद मूलांक उद्भव दिखाते हैं, लेकिन दूसरों को अंकुरित होने में कई दिन लग सकते हैं।
परिवर्तन प्रक्रिया में महत्वपूर्ण बिंदु प्रारंभिक सामग्री के चुनाव और सह-कृषि चरण की अवधि से संबंधित हैं। कुशल परिवर्तन तक पहुँचने के लिए, यह सबसे अच्छा है स्वस्थ और युवा उपजी या प्रारंभिक सामग्री के रूप में गैर बाँझ ग्रीनहाउस विकसित पौधों के डंठल का उपयोग करें. युवा शाखाओं के विकास को प्रेरित करने के लिए, हर 2-3 महीने में परपोनिया के पेड़ को ट्रिम करने और साल में एक बार पेड़ों को ताज़ा करने की सलाह दी जाती है। इसके अतिरिक्त, सह-कृषि चरण केवल 2 दिनों के लिए किया जाना चाहिए। लंबे समय तक सह-कृषि ए tumefaciens द्वारा ऊतक explants के अधिक उपनिवेशन को बढ़ावा देता है और आम तौर पर परिवर्तन दक्षता कम कर देता है। ए tumefaciens द्वारा अति उपनिवेशन को रोकने के लिए यह भी नियमित रूप से प्लेटें जिस पर explants खेती कर रहे हैं ताज़ा करने के लिए महत्वपूर्ण है. यदि अधिक उपनिवेशन होता है, तो ऊतक एक्सप्लांट्स को धोया जा सकता है (धारा 3.8 देखें) ए tumefaciens कोशिकाओं को दूर करने के लिए। हम धोने के लिए इस्तेमाल किया एसएच-10 समाधान करने के लिए ब्लीच जोड़ने की सलाह (अंतिम एकाग्रता: $2% hypochlorite). यह ध्यान रखना महत्वपूर्ण है कि यह अतिरिक्त वाशिंग चरण भारी संक्रमित एक्सप्लांट्स पर काम नहीं कर सकता है (चित्र 2ख)। यदि CRISPR/Cas9 के साथ एक रूपांतरण पैदावार केवल putatively रूपांतरित शूटिंग की एक सीमित संख्या में या यदि एक विशेष जीन के mutagenesis पुनर्जनन में समस्याओं के कारण की उम्मीद है, यह एक खाली वेक्टर नियंत्रण निर्माण के रूप में शामिल करने के लिए सलाह दी जाती है सकारात्मक नियंत्रण. अंत में, यह सुनिश्चित करना महत्वपूर्ण है कि चयनित सभी ट्रांसजेनिक लाइनों स्वतंत्र टी-डीएनए एकीकरण घटनाओं से उत्पन्न होते हैं। इसलिए, हम एक एक्सप्लांट के प्रत्येक पक्ष से केवल एक putatively-ट्रांसजेनिक गोली लेने के लिए निर्देश. हालांकि, हम महसूस करते हैं कि यह स्वतंत्र लाइनों की संभावित संख्या कम कर देता है। यदि कई लाइनों की आवश्यकता होती है, तो शोधकर्ताओं ने putatively-transformed calli को मूल एक्सप्लांटसे अलग करने का निर्णय ले सकता है जब ये कैली आकार और संस्कृति में 2 मिमी हैं, तो ये कैली स्वतंत्र रूप से। इस तरह, कई लाइनों प्रत्येक explant, जो संभावित transgenic लाइनों की संख्या को जन्म देती से अलग किया जा सकता है.
वर्तमान प्रोटोकॉल में, पी एंडरसनी की ट्रांसजेनिक लाइनों का इनविट्रो प्रचार के माध्यम से वनस्पति रूप से प्रचारित किया जाता है। इस का लाभ यह है कि कई ट्रांसजेनिक plantlets एक अपेक्षाकृत कम समय अवधि में उत्पन्न किया जा सकता है. हालांकि, इस विधि भी कई सीमाएँ हैं। सबसे पहले, इन विट्रो प्रचार के माध्यम से टी0 ट्रांसजेनिक लाइनों का रखरखाव श्रम गहन है और इसके परिणामस्वरूप अवांछित आनुवंशिक या एपिजेनेटिक परिवर्तन34,35हो सकते हैं. दूसरे, टी0 लाइनों अभी भी टी डीएनए की एक प्रति होते हैं, एंटीबायोटिक प्रतिरोध कैसेट सहित. यह संभव पुन: परिवर्तन की संख्या को सीमित करता है, के रूप में विभिन्न चयन मार्कर प्रत्येक पुन: परिवर्तन के लिए आवश्यक हैं. वर्तमान में, हम केवल kanamycin या hygromycin चयन (डेटा नहीं दिखाया) का उपयोग कर परिवर्तन का परीक्षण किया है. इसके अलावा, T0 ट्रांसजेनिक लाइनों में Cas9-एन्कोडिंग अनुक्रम और sgRNAs की उपस्थिति पूरक अध्ययन जटिल. पूरक परख संभव है, लेकिन sgRNA लक्ष्य साइट (ओं) की आवश्यकता के लिए इस तरह के रूप में उत्परिवर्तित किया जा सकता है कि पूरक निर्माण के जीन संपादन रोका है. तीसरे, टी0 लाइनों के साथ काम करने का एक नुकसान यह है कि CRISPR/Cas9 उत्परिवर्ती झंकार हो सकता है. झंकार उत्परिवर्ती लाइनों के phenotypic विश्लेषण को रोकने के लिए, हम कम से कम 3 अलग अलग शूटिंग पर इन विट्रो प्रचार के बाद जीनोटाइपिंग विश्लेषण दोहराने की सलाह देते हैं। हालांकि, यहाँ वर्णित प्रोटोकॉल का उपयोग कर प्राप्त झंकार उत्परिवर्ती की संख्या सीमित है, वे कभी कभी मनाया जाता है10. टी0 लाइनों के साथ काम करने की सीमाओं को दूर करने के लिए, पी andersonii उत्परिवर्ती लाइनों पैदा करने से प्रचारित किया जा सकता है. पी andersonii पेड़ dioecious और हवा परागण2कर रहे हैं. इसका मतलब यह है कि प्रत्येक ट्रांसजेनिक लाइन के रूप में हेरफेर किया जाना चाहिए कि पुरुष और महिला फूल एक ही व्यक्ति पर उत्पादित कर रहे हैं, और बाद में इस तरह के रूप में हो कि पार परागण नहीं होता है. के रूप में पी andersonii एक तेजी से बढ़ पेड़ यह एक उष्णकटिबंधीय ग्रीनहाउस में अंतरिक्ष की एक पर्याप्त राशि की आवश्यकता है (28 डिग्री सेल्सियस, $ 85% सापेक्ष आर्द्रता). इसलिए, यद्यपि तकनीकी रूप से संभव है, पी एंडरसनी ट्रांसजेनिक लाइनों का उत्पादक प्रचार logically चुनौतीपूर्ण है.
प्रोटोकॉल अनुभाग में, हमने P. andersoniiकी मंजूरी के लिए 3 विधियों का वर्णन किया है। प्लेट और पाउच सिस्टम का लाभ यह है कि जड़ें आसानी से सुलभ हैं, जो बैक्टीरिया के स्पॉट-इनोकेशन की अनुमति दे सकती है और समय के साथ नोड्यूल गठन के बाद हो सकती है। हालांकि, प्लेट प्रणाली काफी श्रम गहन है, जो इसे कम बड़े पैमाने पर नोडुलेशन प्रयोगों के लिए अनुकूल बनाता है। थैली प्रणाली का एक नुकसान यह है कि कवक संदूषण को रोकने के लिए मुश्किल है. पाउच बाँझ नहीं हैं, और इसलिए कवक विकास अक्सर थैली के शीर्ष आधे पर मनाया जाता है। हालांकि, यह पी एंडरसनी विकास को प्रभावित नहीं करता है, और इसलिए मंजूरी परख के साथ हस्तक्षेप नहीं करता है। इसके अतिरिक्त, थैली प्रणाली पौध के लिए ही उपयुक्त है. कई प्रयासों के बावजूद, हम पाउच में इन विट्रो प्रचार के माध्यम से प्राप्त पौधों को विकसित करने में असमर्थ रहे हैं।
यहाँ वर्णित पी एंडरसनी रिवर्स जेनेटिक्स पाइपलाइन मौजूदा ए राइजोजेनेसआधारित रूट ट्रांसफॉर्मेशन विधि11की तुलना में पर्याप्त सुधार प्रदान करता है . वर्णित प्रक्रियाओं का उपयोग करना, स्थिर ट्रांसजेनिक लाइनों कुशलता से उत्पन्न किया जा सकता है और इन विट्रो प्रचार के माध्यम से बनाए रखा जा सकता है. इसके विपरीत, ए राइजोजेन रूपांतरण क्षणिक होता है और केवल ट्रांसजेनिक जड़ों के गठन में परिणाम होता है। क्योंकि प्रत्येक ट्रांसजेनिक रूट एक स्वतंत्र परिवर्तन से परिणाम, ए राइजोजेन रूपांतरण आधारित परख पर्याप्त phenotypic भिन्नता से ग्रस्त हैं. यह भिन्नता स्थिर रेखाओं के मामले में बहुत कम है, यद्यपि इन विट्रो प्रोपेगेशन में भी कुछ भिन्नता का स्तर पैदा होता है। इस कम भिन्नता और तथ्य यह है कि कई plantlets प्रत्येक स्थिर लाइन के लिए phenotyped किया जा सकता है की वजह से, स्थिर लाइनों ए rhizogenes-transformed जड़ों की तुलना में मात्रात्मक परख के लिए अधिक अनुकूल हैं. इसके अतिरिक्त, स्थिर रूपांतरण ए राइजोजेने सितर्णिक पथ (रोल ) की शुरूआत पर निर्भर नहीं करता है जो अंतर्जात हार्मोन संतुलन15को प्रभावित करता है। इसलिए, स्थिर लाइनें ए राइजोजेनेस-रूपांतरण जड़ों की तुलना में हार्मोन होमियोस्टेसिस में शामिल जीनों के रिवर्स आनुवंशिक विश्लेषण के लिए बेहतर अनुकूल हैं। अनुसंधान मॉडल के रूप में पी andersonii का एक अधिक सामान्य लाभ यह है कि यह एक हाल ही में पूरे जीनोम दोहराव (WGD) का अनुभव नहीं किया. फली Papilionoideae उपपरिवार, जो मॉडल फलियां एम truncatula और एल Japonicus,साथ ही साथ Salicaceae (आदेश Malpighiales) कि मॉडल पेड़ Populus trichocarpa अनुभवी WGDs भी शामिल है शामिल हैं दस लाख साल पहले36,37. इन डब्ल्यूजीडी से उत्पन्न होने वाली कई पैरालॉगस जीन प्रतियां एम ट्रंकटुला, एल जैपोनिकस और पी ट्राइकोकार्पा37,38,39के जीनोम में रखी जाती हैं , जो बनाता है अतिरेक जो रिवर्स आनुवंशिक विश्लेषण ों को जटिल बना सकता है। के रूप में पी andersonii हाल ही में WGD का अनुभव नहीं था, पी andersonii पर रिवर्स आनुवंशिक विश्लेषण कम paralogous जीन प्रतियां के अनावश्यक कामकाज से प्रभावित हो सकता है.
एक साथ लिया, हम पी andersoniiमें रिवर्स आनुवंशिक विश्लेषण के लिए एक विस्तृत प्रोटोकॉल प्रदान करते हैं। इस प्रोटोकॉल का उपयोग करना, एकल उत्परिवर्ती लाइनों कुशलतासे 2-3 महीने10की एक समय सीमा में उत्पन्न किया जा सकता है. इस प्रोटोकॉल को विभिन्न जीनों को एक साथ लक्षित करने वाले एसजीआरएन के प्रवर्तकों के माध्यम से उच्च क्रम म्यूटेंट बनाने के लिए बढ़ाया जा सकता है, जैसा कि अन्य पौधों की प्रजातियों40,41,42के लिए दिखाया गया है। इसके अतिरिक्त, यहाँ वर्णित स्थिर रूपांतरण प्रक्रिया CRISPR/Cas9 जीन-लक्ष्यीकरण तक सीमित नहीं है, लेकिन अन्य प्रकार के निर्माणों को लागू करने के लिए भी उपयोग किया जा सकता है (उदाहरण के लिए, प्रमोटर-रिपोर्टर परख के लिए, अस्थानिक अभिव्यक्ति या ट्रांस- पूरक) हमने पी एंड्र्सेनरी को नाइट्रोजन-फिक्सिंग राइजोबिया या एंडोमाइकोरल कवक के साथ पारस्परिक सहजीवन का अध्ययन करने के लिए एक तुलनात्मक अनुसंधान मॉडल के रूप में स्थापित किया। हालांकि, यहाँ वर्णित प्रोटोकॉल भी इस उष्णकटिबंधीय पेड़ के जीव विज्ञान के अन्य पहलुओं का अध्ययन करने के लिए उपकरण प्रदान करते हैं, जैसे लकड़ी के गठन, द्वि-लैंगिक फूलों का विकास या कैनाबी-विशिष्ट माध्यमिक चयापचयों के biosynthesis.
Acknowledgements
लेखकों को स्वीकार करने के लिए मार्क Youles, सोफीन Kamoun और Sylvestre Marillonnet गोल्डन गेट क्लोनिंग भागों Addgene डेटाबेस के माध्यम से उपलब्ध बनाने के लिए पसंद है. इसके अतिरिक्त, हम ई जेम्स, पी Hadobas, और पी एंडरसनी बीज के लिए टी जे Higgens शुक्रिया अदा करना चाहते हैं. इस काम को नीदरलैंड ऑर्गेनाइजेशन फॉर साइंटिफिक रिसर्च (NWO-VICI अनुदान 865.13.001) द्वारा समर्थित किया गया था; NWO-ओपन प्रतियोगिता अनुदान 819.01.007) और इंडोनेशिया गणराज्य के अनुसंधान, प्रौद्योगिकी और उच्च शिक्षा मंत्रालय (RISET-प्रो अनुदान 8245 आईडी).
Materials
| Name | Company | Catalog Number | Comments |
| Sigma-Aldrich | N0640 | NAA | |
| Duchefa Biochemie | M1503.0250 | MES | |
| Sigma-Aldrich | D134406 | Acetosyringone | |
| Duchefa Biochemie | X1402.1000 | X-Gal | |
| Merck | 101236 | For nucleic acid electrophoresis gel | |
| - | - | Pouches box material, hangers | |
| Merck | 101188 | NH4NO3 | |
| Sigma-Aldrich | B3408-1G | BAP | |
| Merck | 100156 | H3BO3 | |
| Thermo-Fisher | ER1011 | Used as restriction enzyme in Golden Gate cloning assembly | |
| Thermo-Fisher | 15561020 | Used in Golden Gate cloning assembly | |
| Merck | 137101 | CaCl2·2H2O | |
| Duchefa Biochemie | C0111.0025 | C16H16N5O7S2Na | |
| Thermo-Fisher | K1231 | Used for cloning the blunt-ended PCR amplicons in genotyping procedure | |
| Agronutrition | AP2011 | Containing Rhizophagus irregularis DAOM 197198 (1,000 spores/mL), used for mychorrization assay | |
| Merck | 102790 | CuSO4·5H2O | |
| Duchefa Biochemie | D1004.1000 | Used for plant tissue culture agar-based medium | |
| Merck | 105101 | K2HPO4 | |
| VWR Chemicals | 20302.293 | Na2·EDTA | |
| Duchefa Biochemie | M0803.1000 | C6H14O6 | |
| Thermo-Fisher | ER0291 | Used as restriction enzyme in Golden Gate cloning assembly | |
| Merck | 100983 | C2H5OH | |
| VWR Chemicals | BDH9232-500G | EDTA | |
| Sigma-Aldrich | Z377600-1PAK | Cellophane membrane | |
| Biomatters, Ltd. | R9 or higher | Bioinformatics software for in silico cloning and designing of sgRNAs | |
| Mega International | - | Technical information at https://mega-international.com/tech-info/ | |
| Sigma-Aldrich | 65882 | Used for fixating nodule tissues | |
| VWR Chemicals | 24385.295 | - | |
| Vink | 219341 | Pouches box material, bottom part | |
| Leica Biosystems | 14702218311 | Used as a template for plastic embedding | |
| Merck | 100317 | HCl | |
| Sigma-Aldrich | I5386-1G | IBA | |
| Merck | 103862 | C6H5FeO7 | |
| Merck | 103965 | FeSO4O·7H2O | |
| Duchefa Biochemie | I1401.0005 | IPTG | |
| Duchefa Biochemie | K0126.0010 | ||
| Sigma-Aldrich | L2000 | ||
| Merck | 105886 | MgSO4O·7H2O | |
| Merck | 105934 | MnCl2·4H2O | |
| Merck | 102786 | MnSO4O | |
| Duchefa Biochemie | M1002.1000 | Used for bacterial culture agar-based medium | |
| Manutan | 92007687 | Pouches material | |
| Paraxisdienst | 130774 | Elastic sealing foil | |
| Pull Rhenen | Agra-Perlite No.3 | Used as growing substrate in pots for nodulation assay | |
| VWR Chemicals | 391-0581 | Used as container for cellophane membranes | |
| Thermo-Fisher | F130WH | For genotyping transgenic lines | |
| Addgene | 50337 | Level 0 terminator, 3'UTR, 35s (Cauliflower Mosaic Virus) | |
| Addgene | 48017 | End-link 2 for assembling 2 level one part into a level 2 acceptor | |
| Addgene | 48018 | End-link 3 for assembling 3 level one part into a level 2 acceptor | |
| Addgene | 48001 | Level 1 acceptor. Position 5. Forward orientation | |
| Addgene | 48007 | Level 1 Acceptor. Position 1. Reverse orientation | |
| Addgene | 50268 | Level 0 promoter (0.4 kb), 35s (Cauliflower Mosaic Virus) + 5'UTR, Ω (Tobacco Mosaic Virus) | |
| Addgene | 46966 | Used for designing CRISPR/Cas9 module | |
| Addgene | 46968 | Used for designing CRISPR/Cas9 module | |
| Addgene | 50334 | Level 0 Kanamycin/Neomycin/Paromomycin resistance cassette | |
| Topzeven | - | Used as filters for washing spore suspension | |
| Sigma-Aldrich | 8.17003 | PEG400 | |
| Duchefa Biochemie | E1674.0001 | Pots to grow Parasponia plantlets/seedlings | |
| Merck | 104871 | KH2PO4 | |
| Merck | 105033 | KOH | |
| Merck | 105153 | K2SO4O | |
| Van Leusden b.v. | - | Used as growing substrate for mychorrhization assay | |
| Duchefa Biochemie | S0225.0050 | SH-basal salt medium | |
| Duchefa Biochemie | S0411.0250 | SH-vitamin mixture | |
| Lehle Seeds | VIS-02 | Used as non-ionic surfactant in the washing step of stable transformation | |
| Merck | 137017 | NaCl | |
| VWR Chemicals | 89230-072 | C6H11NaO7 | |
| Merck | 106521 | Na2MoO4·2H2O | |
| Merck | 106574 | Na2HPO4·7H2O | |
| Merck | 567549 | NaH2PO4·H2O | |
| Sigma-Aldrich | 239313 | Na2SO4O | |
| Duchefa Biochemie | S0809.5000 | C12H22O11 | |
| Thermo-Fisher | B69 | Used in Golden Gate cloning assembly | |
| Thermo-Fisher | EL0013 | Used in Golden Gate cloning assembly | |
| Kulzer-Mitsui Chemicals Group | 64708806 | Methyl methacrylate-based resin powder | |
| Kulzer-Mitsui Chemicals Group | 64709003 | HEMA (2-hydroxyethyl methacrylate)-based resin solution | |
| Kulzer-Mitsui Chemicals Group | 66022678 | Methyl methacrylate-based resin solution | |
| Merck | 1159300025 | ||
| Acros | 189350250 | ||
| VWR Chemicals | 663684B | Polysorbate 20 | |
| Stout Perspex | - | pouches box material, lid | |
| Duchefa Biochemie | Y1333.1000 | ||
| Merck | 108816 | ZnCl2 | |
| Alfa Aesar | 33399 | ZnSO4O·7H2O |
References
- Clason, E. W. THE VEGETATION OF THE UPPER-BADAK REGION OF MOUNT KELUT (EAST JAVA). Bulletin du Jardin Botanique de Buitenzorg. Serie III, 509-518 (1936).
- Soepadmo, E. Ulmaceae. Flora Malesiana-Series 1, Spermatophyta. 8, 31-76 (1974).
- Becking, J. H. The Rhizobium symbiosis of the nonlegume Parasponia. Biological Nitrogen Fixation. , 497-559 (1992).
- Oldroyd, G. E. D. Speak, friend, and enter: signalling systems that promote beneficial symbiotic associations in plants. Nature Reviews Microbiology. 11, 252-263 (2013).
- Gutjahr, C., Parniske, M. Cell and developmental biology of arbuscular mycorrhiza symbiosis. Annual Review of Cell and Developmental Biology. 29, 593-617 (2013).
- van Velzen, R., et al. Comparative genomics of the nonlegume Parasponia reveals insights into evolution of nitrogen-fixing rhizobium symbioses. Proceedings of the National Academy of Sciences of the United States of America. 115, E4700-E4709 (2018).
- van Velzen, R., Doyle, J. J., Geurts, R. A Resurrected Scenario: Single Gain and Massive Loss of Nitrogen-Fixing Nodulation. Trends in Plant Science. 24, 49-57 (2019).
- Griesmann, M., et al. Phylogenomics reveals multiple losses of the nitrogen-fixing root nodule symbiosis. Science. 1743, eaat1743 (2018).
- Becking, J. H. Root-Nodule Symbiosis Between Rhizobium And Parasponia (Ulmaceae). Plant and Soil. 51, 289-296 (1979).
- van Zeijl, A., et al. CRISPR/Cas9-Mediated Mutagenesis of Four Putative Symbiosis Genes of the Tropical Tree Parasponia andersonii Reveals Novel Phenotypes. Frontiers in Plant Science. 9, 284 (2018).
- Cao, Q., et al. Efficiency of Agrobacterium rhizogenes–mediated root transformation of Parasponia and Trema is temperature dependent. Plant Growth Regulation. 68, 459-465 (2012).
- Limpens, E., et al. RNA interference in Agrobacterium rhizogenes-transformed roots of Arabidopsis and Medicago truncatula. Journal of Experimental Botany. 55, 983-992 (2004).
- Boisson-Dernier, A., et al. Agrobacterium rhizogenes-Transformed Roots of Medicago truncatula for the Study of Nitrogen-Fixing and Endomycorrhizal Symbiotic Associations. Molecular Plant-Microbe Interactions. 14, 695-700 (2001).
- Kumagai, H., Kouchi, H. Gene Silencing by Expression of Hairpin RNA in Lotus japonicus Roots and Root Nodules. Molecular Plant-Microbe Interactions. 16, 663-668 (2003).
- Nilsson, O., Olsson, O. Getting to the root: The role of the Agrobacterium rhizogenes rol genes in the formation of hairy roots. Physiologia Plantarum. 100, 463-473 (1997).
- Davey, M. R., et al. Effective Nodulation of Micro-Propagated Shoots of the Non-Legume Parasponia andersonii by Bradyrhizobium. Journal of Experimental Botany. 44, 863-867 (1993).
- Webster, G., Poulton, P. R., Cocking, E. C., Davey, M. R. The nodulation of micro-propagated plants of Parasponia andersonii by tropical legume rhizobia. Journal of Experimental Botany. 46, 1131-1137 (1995).
- Op den Camp, R., et al. LysM-type mycorrhizal receptor recruited for rhizobium symbiosis in nonlegume Parasponia. Science. 331, 909-912 (2011).
- Weber, E., Engler, C., Gruetzner, R., Werner, S., Marillonnet, S. A Modular Cloning System for Standardized Assembly of Multigene Constructs. PLOS ONE. 6, e16765 (2011).
- Nekrasov, V., Staskawicz, B. J., Weigel, D., Jones, J. D. G., Kamoun, S. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease. Nature Biotechnology. 31, 691-693 (2013).
- Bertani, G. Studies On Lysogenesis. I. The Mode Of Phage Liberation By Lysogenic Escherichia Coli. Journal of Bacteriology. 62, 293-300 (1951).
- Engler, C., et al. A Golden Gate modular cloning toolbox for plants. ACS Synthetic Biology. 3, 839-843 (2014).
- Fauser, F., Schiml, S., Puchta, H. Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering in Arabidopsis thaliana. The Plant Journal. 79, 348-359 (2014).
- Lazo, G. R., Stein, P. A., Ludwig, R. A. A DNA Transformation-Competent Arabidopsis Genomic Library in Agrobacterium. Biotechnology. 9, 963-967 (1991).
- Op den Camp, R. H. M., et al. N Nonlegume Parasponia andersonii Deploys A Broad Rhizobium Host Range Strategy Resulting in Largely Variable Symbiotic Effectivenes. Molecular Plant-Microbe Interactions. 25, 954-963 (2012).
- Graham, P. H., Viteri, S. E., Mackie, F., Vargas, A. T., Palacios, A. Variation in acid soil tolerance among strains of Rhizobium phaseoli. Field Crops Research. 5, 121-128 (1982).
- Martinez-Romero, E., et al. Rhizobium tropici, A Novel Species Nodulating Phaseolus vulgaris L. Beans and Leucaena sp. Trees. International Journal of Systematic and Evolutionary Microbiology. 41, 417-421 (1991).
- Felten, J., et al. The Ectomycorrhizal Fungus Laccaria bicolor Stimulates Lateral Root Formation in Poplar and Arabidopsis through Auxin Transport and Signaling. Plant Physiology. 151, 1991-2005 (1991).
- Trouvelot, A., Kough, J. L., Gianinazzi-Pearson, V. Mesure du taux de mycorhization VA d’un systeme radiculaire. Recherche de methods d’estimation ayant une signification fonctionnelle. Aspects Physiologiques et Genetiques des Mycorhizes. , 217-221 (1986).
- Schenk, R. U., Hildebrandt, A. C. Medium and techniques for induction and growth of monocotyledonous and dicotyledonous plant cell cultures. Canadian Journal of Botany. 50, 199-204 (1972).
- Becking, J. H. The Parasponia parviflora - Rhizobium symbiosis. Host specificity, growth and nitrogen fixation under various conditions. Plant and Soil. 75, 309-342 (1983).
- Hoagland, D. R., Arnon Revised, D. I., Arnon, D. I. The Water-Culture Method for Growing Plants without Soil. Circular California Agricultural Experiment Station. 347, 1-32 (1950).
- Wang, K. . Methods in Molecular Biology: Agrobacterium Protocols. 1, (2015).
- Smulders, M. J. M., de Klerk, G. J. Epigenetics in plant tissue culture. Plant Growth Regulation. 63, 137-146 (2011).
- Larkin, P. J., Scowcroft, W. R. Somaclonal variation — a novel source of variability from cell cultures for plant improvement. Theoretical and Applied Genetics. 60, 197-214 (1981).
- Cannon, S. B., et al. Legume genome evolution viewed through the Medicago truncatula and Lotus japonicus genomes. Proceedings of the National Academy of Sciences of the United States of America. 103, 14959-14964 (2006).
- Tuskan, G. A., et al. The Genome of Black Cottonwood, Populus trichocarpa (Torr. & Gray). Science. 313, 1596-1604 (2006).
- Young, N. D., et al. The Medicago genome provides insight into the evolution of rhizobial symbioses. Nature. 480, 520 (2011).
- Sato, S., et al. Genome Structure of the Legume, Lotus japonicus. DNA Research. 15, 227-239 (2008).
- Xing, H. L., et al. A CRISPR/Cas9 toolkit for multiplex genome editing in plants. BMC Plant Biology. 14, 327 (2014).
- Lowder, L. G., et al. A CRISPR/Cas9 Toolbox for Multiplexed Plant Genome Editing and Transcriptional Regulation. Plant Physiology. 169, 971 (2015).
- van Zeijl, A. . Dissecting Hormonal Pathways in Nitrogen-Fixing Rhizobium Symbioses. , (2017).
Explore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved