Transformação, edição do genoma e fenotipagem do nitrogênio-fixação tropical Cannabaceae Tree Parasponia andersonii
Neste Artigo
Resumo
Parasponia andersonii é uma árvore tropical de rápido crescimento que pertence à família cannabis (Cannabaceae) e pode formar nódulos radiculares de fixação de nitrogênio em associação com o Rhizobium. Aqui, descrevemos um protocolo detalhado para análises genéticas reversas em P. andersonii com base na transformação estável mediada por Agrobacterium tumefaciense na edição do GENOMA baseado em crispr/Cas9.
Resumo
Parasponia andersonii é uma árvore tropical de rápido crescimento que pertence à família cannabis (Cannabaceae). Junto com 4 espécies adicionais, forma a única linhagem não legume conhecida capaz de estabelecer uma simbiose de nódulo fixando nitrogênio com rizóbio. Estudos comparativos entre leguminosas e P. andersonii poderiam fornecer insights valiosos sobre as redes genéticas subjacentes à formação de nódulo radicular. Para facilitar estudos comparativos, recentemente sequenciamos o genoma do P. andersonii e estabelecemos a transformação estável mediada por Agrobacterium tumefaciense a edição do GENOMA baseado em crispr/Cas9. Aqui, fornecemos uma descrição detalhada dos procedimentos de transformação e edição de genoma desenvolvidos para P. andersonii. Além disso, descrevemos procedimentos para a germinação de sementes e caracterização de fenótipos simbióticos. Usando este protocolo, as linhas transgénicas estáveis do mutante podem ser geradas em um período de 2-3 meses. A propagação vegetativa in vitro de linhas transgênicas T0 permite que experimentos fenotipados sejam iniciados aos 4 meses após a. tumefaciens co-cultivo. Conseqüentemente, este protocolo toma somente marginalmente mais por muito tempo do que o método transiente do Agrobacterium rhizogenes-baseado da transformação da raiz disponível para o andersonii do P., embora ofereça diversas vantagens desobstruídas. Juntos, os procedimentos descritos aqui permitem que P. andersonii seja utilizado como modelo de pesquisa para estudos que visem a compreensão de associações simbióticas, bem como potencialmente outros aspectos da biologia desta árvore tropical.
Introdução
Parasponia andersonii é uma árvore tropical pertencente à família canábis (Cannabaceae) e é nativa da Papua Nova Guiné e várias ilhas do Pacífico1,2,3. Junto com 4 espécies adicionais de Parasponia , representa a única linhagem não-leguminosa que pode estabelecer uma simbiose de nódulo de fixação de nitrogênio com rizobia. Esta simbiose é bem estudada nas leguminosas (Fabaceae) modelos Medicago Galba e Lotus japonicus, o que resultou na aquisição de conhecimento detalhado da natureza genética molecular da formação de nódulo e funcionamento4. Adicionalmente, demonstrou-se que a simbiose de nódulo radicular em leguminosas é fundada na simbiose micorrízica micorrízicos arbusculares muito mais antiga e disseminada5. As comparações phylogenomic sugerem que os simbioses do nódulo do nitrogênio-fixação das leguminosa, do parasponia, assim como, a espécie as assim chamada da planta que hospedam bactérias diazotróficas de Frankia , têm uma origem evolutiva compartilhada 6,7,8. Para determinar se os genes identificados para serem envolvidos na formação de nódulo de leguminosa são a parte de uma base genética conservada, estudos sobre espécies não-leguminosas são essenciais. Para tanto, propomos o uso de P. andersonii como modelo de pesquisa comparativa, ao lado de leguminosas, para identificar as principais redes genéticas subjacentes à formação e ao funcionamento do nódulo radicular.
P. andersonii é um pioneiro que pode ser encontrado nas encostas das colinas vulcânicas. Pode encontrar velocidades de crescimento de 45 cm por o mês e comprimentos do alcance de até 10 medidores9. As árvores de P. andersonii são vento-polinated, que é facilitada pela formação de flores masculinas e fêmeas separadas3,10. Recentemente, sequenciou e anotou o genoma diploide (2n = 20; 560 MB/1C) de P. andersonii, e montou sequências de genoma de 2 espécies adicionais de parasponia ; P. rigida e p. rugosa6. Isto revelou modelos do gene de ~ 35.000 P. andersonii que podem ser aglomerados em > 20000 orthogroups junto com os genes de M. Galba, de feijão de soja (Glycine max), de Arabidopsis Arabidopsis thaliana, de morango da floresta ( Fragaria vesca), trema orientalis, choupo de algodão preto(Populus neotorularia) eeucalipto (Eucalyptus grandis)6. Adicionalmente, as comparações do transcriptoma entre M. Galba e P. andersonii identificaram um conjunto de 290 ortologues putativos que exibem um padrão de expressão com nódulo aumentado em ambas as espécies6. Isto fornece um recurso excelente para estudos comparativos.
Para estudar a função gênica em raízes e nódulos de P. andersonii , foi estabelecido um protocolo para a transformação da raiz mediada por Rizógenes Agrobacterium11. Usando este protocolo, as plantas compostas que carregam raizes transgênicas podem ser geradas em um frame de tempo relativamente curto. Este método é, também, amplamente aplicado na pesquisa de legume-simbiose12,13,14. No entanto, a desvantagem deste método é que apenas as raízes são transformadas e que cada raiz transgênica representa um evento de transformação independente, resultando em variação substancial. Além disso, a transformação é transitória e as linhas transgênicas não podem ser mantidas. Isso faz com que uma transformação de raiz baseada em rhizogenesseja menos adequada para A edição do genoma mediado por crispr/Cas9. Adicionalmente, a. rhizogenes transfere seus genes de Locus indutores de raiz (rol) para o genoma vegetal, que uma vez expresso interferiu na homeostase hormonal15. Isso torna o estudo do papel dos hormônios vegetais em raízes transformadas em A. rhizogenes. Para superar essas limitações, desenvolvemos recentemente um protocolo para a transformação baseada em Agrobacterium tumefaciense a mutagenese mediada por crispr/Cas9 de P. andersonii10.
Aqui, nós fornecemos uma descrição detalhada do procedimento de transformação a . tumefaciens-based e pipeline de genética reversa desenvolvido para P. andersonii. Adicionalmente, nós fornecemos protocolos para o tratamento a jusante de plântulas transgênicas, incluindo ensaios para estudar interações simbióticas. Usando o protocolo descrito aqui, várias linhas transgênicas podem ser geradas em um período de 2-3 meses. Em combinação com a mutagenese mediada por CRISPR/Cas9, isso permite a geração eficiente de linhas mutantes de nocaute. Estas linhagens mutantes podem ser propagadas vegetativamente in vitro10,16,17, o que permite a geração de material suficiente para iniciar a caracterização fenotípica aos 4 meses após o procedimento de transformação ter foi iniciada10. Juntos, este conjunto de procedimentos deve permitir que qualquer laboratório adote P. andersonii como um modelo de pesquisa para estudos voltados para a compreensão das associações rizóbias e micorrízicas, bem como potencialmente outros aspectos da biologia desta árvore tropical.
Protocolo
1. cresça árvores de P. andersonii na estufa
-
Germinar P. andersonii WU1 sementes18.
- Use bagas de Parasponia frescas ou mergulhe bagas secas em água para 2 h para Rehidratar. Bagas da polpa em uma parte de papel de tecido ou esfregue de encontro ao interior de uma peneira do chá para remover as sementes.
- Desinfecte sementes usando lixívia comercial (~ 4% de hipoclorito) por 15-20 min e depois lave as sementes 6 vezes usando água esterilizada.
- Transfira as sementes para tubos estéreis de 200 μL de PCR. Encha os tubos com água esterilizada, de tal forma que as sementes estejam completamente submersas. Incubar os tubos durante 10 dias em um termocicer executando o seguinte programa: 30 ciclos (7 ° c para 4 h, 28 ° c por 4 h). Não use uma tampa aquecida, pois isso pode matar as sementes.
- Prepare as placas SH-0 (ver tabela 1). Transfira as sementes para as placas SH-0 e incubar a 28 ° c, 16 h: 8 dias de noite. Feche as placas com 2 camadas de folha de vedação elástica para evitar a secagem durante a incubação a 28 ° c.
- Depois que as plântulas desenvolveram seu primeiro jogo das folhas verdadeiras (~ 3-4 semanas após a incubação em 28 ° c), as mudas de transferência aos potenciômetros encheram-se com o solo de potting comercial e cobrem as plântulas com um copo plástico translúcido para impedir o desiccation. Coloc potenciômetros em um quarto do clima de 28 ° c ou em uma estufa, ~ 85% RH, um dia de 16 h: 8 o.
- Após 1 semana, retire o copo de plástico translúcido. Molhe os potenciômetros regularmente e quando as árvores crescerem um suplemento mais grande com fertilizante para sustentar o crescimento.
2. clonagem de construções para Mutagenese mediada por CRISPR/Cas9 de P. andersonii
Nota: Vetores de transformação binária padrão podem ser usados para a transformação estável de P. andersonii. Aqui, por exemplo, é um procedimento para gerar construções para mutagenese mediada por CRISPR/Cas9 usando clonagem modular (por exemplo, Golden Gate)19.
- Identifique as sequências alvo do RNA do guia para o gene (s) do interesse, usando o software da bioinformática que caracteriza uma ferramenta interna do projeto de CRISPR. Escolha as seqüências do RNA do guia situadas no 5 '-fim da seqüência de codificação do gene do alvo para aumentar a possibilidade de obter Knockouts cheios. Certifique-se de verificar se há efeitos fora do alvo pesquisando contra o genoma P. andersonii 6.
Nota: Use 2 sgRNAs por Gene alvo, preferencialmente 200-300 BP separados. Isto pode gerar deleções que podem ser identificadas pelo PCR e subseqüentemente pela electroforese do gel do agarose. - Gere construções de Golden Gate de nível 1 contendo as sequências sgRNA.
- Projete primers para amplificar cada sgRNA individual inserindo a seqüência do guia de 20 BP na posição de n(20) na seguinte seqüência do primer: 5 '-TGTGGTCTCAATTGN(20) gttttagagctagaaatagcaag-3 '.
Nota: se a sequência de guia for igual a GN(19), remova o G na extremidade 5 ' da sequência de guia antes de inserir na sequência de primer. - PCR amplificar sgrnas de pICH86966:: AtU6p:: sgRNA_PDS20 usando os primers para diante projetados na etapa 2.2.1 e o primer reverso universal: 5 '-tgtggtctcaagcgtaatgccaactttgtac-3 '. Use uma polimerase calor-estável de alta fidelidade do ADN e as seguintes condições do PCR: 98 ° c para 30 s; 30 ciclos (98 ° c por 10 s; 53 ° c por 20 s; 72 ° c por 10 s); 72 ° c por 7 min. as reações bem-sucedidas do PCR rendem um amplicon de 165 BP.
- Coluna-purify o amplicon do PCR usando um jogo comercial da purificação do PCR. Subseqüentemente, ajuste acima as reações da porta dourada para clonar sgRNAs atrás do promotor pequeno do RNA de Arabidopsis Arabidopsis thaliana AtU6p: 10 ng do amplicon do PCR de sgRNA, 150 ng de pICSL01009:: AtU6p20, 60 ng do vetor apropriado do aceitador do nível 1, 2 μL de T4 tampão de ligase, 2 μL 0,1% de albumina sérica bovina (BSA), 0,5 μL de BsaI, 0,5 μL de ligase T4, encha a 20 μL com água ultrapura. Assegure-se de que todos os sgRNAs sejam clonados na mesma orientação para evitar a formação de hairpin.
- Incubar reações em um termocicer executando o seguinte programa: 37 ° c por 20 s; 26 ciclos (37 ° c por 3 min; 16 ° c por 4 min); 50 ° c por 5 min; 80 ° c por 5 min. transforme as reações de Golden Gate em Escherichia coli e placa no meio lb21 contendo ampicilina (50 mg/l), X-GAL (200 mg/l) e IPTG (1 mm).
Nota: Prepare soluções de estoque de IPTG e X-GAL em água ultra-pura e dimetilformamida, respectivamente. Filtro esterilizar as soluções de ampicilina e IPTG e armazenar todas as ações em-20 ° c. Use luvas ao manusear dimetilformamida. - Selecione colônias brancas e isole plasmídeo usando um kit de isolamento plasmídico comercial. A seqüência verifica plasmídeos isolados antes de continuar com a montagem dourada do nível 2 da porta.
- Projete primers para amplificar cada sgRNA individual inserindo a seqüência do guia de 20 BP na posição de n(20) na seguinte seqüência do primer: 5 '-TGTGGTCTCAATTGN(20) gttttagagctagaaatagcaag-3 '.
- Monte o nível 2 construções Golden Gate para a transformação estável.
- Realize uma reação Golden Gate usando o nível 1 AtU6p:: sgRNA construções (geradas a seção 2,2), bem como pICH47802::nptII, pICH47742:: 35Spro:: Ωnls-ACas9:: 35Ster, o nível 2 Acceptor pICSL4723 e o apropriado vinculador final (ver Engler et al.22). Realize reações da seguinte forma: use ~ 100 fmol de cada vetor doador e ~ 20 fmol do vetor aceitador e adicione 2 μL de tampão de ligase T4, 2 μL de 0,1% de BSA, 0,5 μL de BpiI, 0,5 μL de ligase T4, encha a 20 μL com água ultrapura.
Nota: o nível 1 plasmídeos pICH47802::nptII, pICH47742:: 35Spro:: ωnls-aCas9:: 35Ster precisa de ser clonado primeiramente (Veja o arquivo suplementar 1), como é descrito para sgrnas a seção 2,220,22 ,23. - Incubar reações como no passo 2.2.4 e transformar em e. coli. Placa no meio da libra que contem o kanamycin. No dia seguinte, selecione colônias brancas e isole plasmímids. Determine o conjunto correto do plasmídeo pela análise da restrição-digestão.
- Realize uma reação Golden Gate usando o nível 1 AtU6p:: sgRNA construções (geradas a seção 2,2), bem como pICH47802::nptII, pICH47742:: 35Spro:: Ωnls-ACas9:: 35Ster, o nível 2 Acceptor pICSL4723 e o apropriado vinculador final (ver Engler et al.22). Realize reações da seguinte forma: use ~ 100 fmol de cada vetor doador e ~ 20 fmol do vetor aceitador e adicione 2 μL de tampão de ligase T4, 2 μL de 0,1% de BSA, 0,5 μL de BpiI, 0,5 μL de ligase T4, encha a 20 μL com água ultrapura.
- Transforme construções de nível 2 em estirpe de Agrobacterium tumefaciens AGL124.
3. transformação estável de P. andersonii
- Inocular 2 placas de LB contendo os antibióticos apropriados com estirpe de a. tumefaciens AGL1 transformadas com o construto de interesse. Incubar as placas a 28 ° c durante 2 dias.
- Colha filiais novas das árvores estufa-crescidas. Use cerca de 5 ramos de 5-8 cm de comprimento para cada transformação. Assegure-se de usar apenas ramificações não infectadas saudáveis. Retire as folhas cortando-as como tal que ~ 1 cm2 de tecido foliar é deixada no final de cada petiolo. Descarte as folhas.
- Desinfecte o tecido por 15 min usando lixívia comercial diluída em 1:1 (~ 2% de hipoclorito após diluição) contendo algumas gotas de polissorbato 20. Em seguida, lave o tecido 6 vezes com água autoclavada.
Nota: esta etapa, assim como, as seguintes etapas precisa de ser conduzida dentro de um armário do para baixo-fluxo laminar para manter o tecido estéril. - Re-suspender as células de a . tumefaciens de 1-2 placas em 25 ml de meio de infiltração (ver tabela 1) contendo acetovone (20 mg/L) e um surfactante não-iônico (0, 1% v/v) para atingir uma densidade óptica (OD600) de ~ 5.
Nota: Prepare a solução de estoque de acetode uma em 70% de etanol e armazene a-20 ° c. O surfactante não-iônico precisa ser esterilizado antes de adicionar ao meio de infiltração. - Corte o tronco e o tecido petiolo em pedaços de ~ 1 cm de comprimento dentro da suspensão a . tumefaciens , criando assim feridas frescas em ambos os lados. Deixe pedaços de tecido na suspensão a . tumefaciens por 10-30 min.
- Prepare o meio de enraizamento (ver tabela 1) e adicione acetosomouma (20 mg/L) após a autoclavagem. Partes do tecido seco em um pedaço estéril de papel de filtro e colocá-lo no meio (~ 10 explants/placa). Incubar as chapas escuras a 21 ° c durante 2 dias.
Nota: Deixe o meio arrefecer até ~ 60 ° c antes de adicionar acetosobiona. - Após 2 dias, inspecione as placas para contaminação bacteriana fúngica ou óbvia (outras bactérias que não A. tumefaciens). As placas contaminadas precisam de ser descartadas.
- Prepare o meio líquido SH-10 (ver tabela 1). Após autoclavagem, adicionar polissorbato 20 (0, 1%, v/v). Transfira peças de tecido para 10 mL de SH-10 contendo polissorbato 20. Durante um período de pelo menos 10 minutos, agitar suavemente a cada 2-3 min para lavar o tecido.
- Lave mais duas vezes com SH-10 fresco contendo polissorbato 20. Estes tempos, um tempo de incubação de 2-3 min por etapa de lavagem é suficiente.
- Prepare o meio de enraizamento (ver tabela 1). Após autoclavagem, adicionar cefotaxima (300 mg/l) e canamicina (50 mg/l) e despeje as placas. Para as transformações secundárias (transformações de linhas transgênicas resistentes à kanamicina), aplique a seleção de higromicina (15 mg/L).
- Partes secas do tecido em partes estéreis de papel de filtro. Depois, transfira peças de tecido para as placas preparadas na etapa 3,9.
- Incubar as placas durante 7 dias a 28 ° c, 16 h: 8 horas de noite. A cada 2 dias, verifique as placas para contaminação fúngica ou bacteriana e crescimento excessivo de A. tumefaciens. Em caso de contaminação, transfira peças não infectadas para uma placa fresca.
- Após 7 dias, transfira peças de tecido para o meio de propagação (ver tabela 1) contendo cefotaxima (300 mg/l) e canamicina (50 mg/l). Incubar as placas a 28 ° c, 16 h: 8 horas de noite. Refresque placas uma vez por semana até que os tiros transgénicos desenvolvam. Assegure para transferir somente partes não-contaminadas do tecido às placas frescas. Descarte as peças que estão sobrecultivadas por a. tumefaciens.
- Uma vez que as brotações putativamente-transgênicas têm ≥ 1 cm de comprimento, os brotos cortados e a cultura independentemente no meio de propagação contendo cefotaxima (300 mg/l) e canamicina (50 mg/l). Para garantir que os rebentos representem transformantes independentes, tome apenas uma única filmagem de cada lado de um explante.
- Propagar vegetativamente brotos putativamente-transgênicos como descrito na etapa 5,2.
4. genotipagem de brotos putativamente-transgênicos
- Iniciadores de Design abrangendo o (s) site (es) de reconhecimento sgRNA. Para permitir o seqüenciamento do amplicon do PCR, escolha primers 150-250 BP longe do (s) site (es) de reconhecimento do sgRNA.
- Corte uma ponta da folha (~ 5 milímetros) de cada tiro transgênicas a ser genotyped. Além disso, colher uma amostra de controle de tipo selvagem.
- Realize 50 μL de reações de PCR usando os primers projetados na etapa 4,1 e um kit comercial para amplificar diretamente o DNA de amostras de plantas. Alternativamente, as reações do PCR podem ser executadas no ADN purified usando um polymerase High-fidelity.
- Separe os amplicões do PCR em um gel do agarose de 1.5-2%.
- Analise os resultados da eletroforese em gel. Verifique se há amostras produzindo várias bandas (mais de 1 alelo) e amplicões de PCR com tamanhos diferentes do tipo selvagem, o que indica a presença de indelos de tamanho médio.
- Amplicões da PCR da seqüência para identificar as mutações exatas. Para amostras que produzem um único amplicon do PCR, os produtos do PCR podem seqüenciado diretamente. As amostras que produzem mais de 1 faixa após a electroforese do gel ou que parecem ser heterozygous depois que arranjar em seqüência direto do amplicon do PCR, precisam de ser clonadas em um vetor do clonagem da Blunt-extremidade primeiramente. Subseqüentemente, sequencia vários clones para cada amostra para identificar todos os alelos possíveis presentes na amostra.
- Alinhe os resultados de sequenciamento ao gene de interesse e inspecione o alinhamento para verificar se há mutações perto do (s) local (es) de destino do sgRNA. Subseqüentemente, verifique se estas mutações criam frameshifts. Descarte linhas com > 2 alelos e linhas contendo mutações no quadro.
- Selecione várias linhas para análise posterior.
- Propagar linhas selecionadas conforme descrito na etapa 5,2.
- Quando as linhas desenvolveram diversos rebentos novos, tome amostras novas de ≥ 3 pontas da folha e repita etapas 4.3-4.7. Determine se as mutações atuais em cada uma das amostras que originam da mesma linha assim como a amostra original do PCR são idênticas. As linhas que produzem as mesmas mutações em todas as amostras são mutado homogênea e podem ser usadas para uma experimentação mais adicional. Descartar linhas que não produzem os mesmos resultados que essas linhas são quiméricos.
5. preparação de plantlets de P. andersonii enraizados para experimentação
-
Iniciar uma nova linha de cultura tecidual de P. andersonii.
- Colha gemas axilares, brotos adventícios jovens ou tecido foliar de árvores saudáveis. Alternativamente, as mudas podem ser usadas como material de partida.
- Desinfecte o tecido usando lixívia comercial diluída em 1:1 (~ 2% de hipoclorito após diluição) contendo algumas gotas de polissorbato 20 por 15 min. depois, enxágüe o tecido 6 vezes usando água autoclavada.
Nota: esta etapa, assim como, as seguintes etapas precisam de ser conduzidas dentro de um Downflow laminar ou de um armário do crossflow laminar para manter o tecido estéril. - Transfira o tecido para o meio de propagação (ver tabela 1). Feche as placas com 2 camadas de folha de selagem elástica e incube placas em 28 ° c, 16 h: dia de 8 h:Night.
- Inspecione as placas a cada poucos dias durante as primeiras 2 semanas para garantir que o tecido esteja livre de contaminação fúngica ou bacteriana.
- Propague o tecido colocando ~ 10 tiros em uma placa fresca do meio da propagação e feche a placa com as 2 camadas de folha de selagem elástica. Incubar as placas a 28 ° c, 16 h: 8 horas de noite. Repita este passo a cada 4 semanas.
- Quando os rebentos tiverem > 1 cm de comprimento, cortar os rebentos na base e colocá-los no meio de enraizamento (ver tabela 1). Cerca de 10 tiros podem ser colocados em uma única placa de enraizamento. A posição dispara na vertical inserindo a ponta basal da parte aérea no meio. As raizes aparecem em 10-14 dias após a incubação das placas em 28 ° c, 16 h 8 h dia: noite.
Nota: não raiz todos os tiros, mas manter parte para a propagação da cultura do tecido (ver passo 5,2).
6. nodulação de P. andersonii plantlets em potes
-
Prepare o inóculo de Rhizobium.
- Inocular 10 mL de meio YEM líquido (ver tabela 2) de uma única colônia de Mesorhizobium plurifarium BOR26 e incubar a 28 ° c por 2 dias.
Nota: M. plurifarium BOR2 é preferível, uma forma eficiente nodulates P. andersonii. No entanto, outras estirpes de rizóbio também podem ser utilizadas para a nodulação de P. andersonii (por exemplo, Bradyrhizobium elkanii WUR325, Rhizobium tropici CIAT89926,27 ou Bradyrhizobium SP. Kelud2A4). - Use a cultura de 10 mL para inocular um volume maior de meio YEM líquido. O volume dessa cultura depende do número de vasos que precisam ser inoculados.
- Prepare o meio líquido EKM (ver tabelas 3, tabela 4). Centrifugue a cultura bacteriana por 10 min a 3.500 x g para colher as células. Subseqüentemente, resuspenda a pelota bacteriana no EKM líquido (use aproximadamente o mesmo volume que a cultura original de YEM) e determine a densidade ótica (OD600).
- Inocular 10 mL de meio YEM líquido (ver tabela 2) de uma única colônia de Mesorhizobium plurifarium BOR26 e incubar a 28 ° c por 2 dias.
- Para o ~ 20 potenciômetros, prepare 3 litros do meio líquido de EKM e inocular com a suspensão rizóbio preparada na etapa 6.1.3. para alcançar OD600 = 0, 25.
- Misture 3 L de EKM contendo rizóbios com 1.250 g de perlita. Subseqüentemente, adicione 210 g desta mistura aos potenciômetros translúcidos estéreis do polypropylene. Alternativamente, em vez de perlita, use areia como substrato para ensaios de nodulação.
- Plantar 1-3 P. andersonii plântulas em cada pote. Além disso, prepare vários potes contendo plântulas de P. andersonii transformados com a construção de controle crispr (ver tabela complementar 1). Pesar vários potes para ser capaz de determinar a perda de água durante o experimento. Cubra a parte inferior de cada pote para proteger as raízes da exposição à luz.
- Incubar vasos em uma sala de crescimento climatizada (28 ° c, 16 h: 8 dias de noite) durante 4-6 semanas. Uma vez por semana, pesar vários potes para determinar a perda de água. Se a perda de água exceder 10 mL, suplemento com água ultrapura para compensar a perda.
- Após 4-6 semanas, limpe as raízes da perlita e determine os números de nódulo usando um binóculo para examinar a eficiência da nodulação.
7. nodulação de P. andersonii plantlets em placas
-
Prepare membranas celofane a 28.
- Corte a membrana do celofane para caber em um prato de Petri quadrado de 12 cm x de 12 cm. Corte as membranas um pouco mais curtas na parte superior para permitir que o espaço para os rebentos cresça.
- Para aumentar a permeabilidade das membranas de celofane, Ferva as membranas em solução de EDTA (1 g/L) por 20 min. depois, enxague pelo menos 6x com água desmineralizada para remover o EDTA.
Nota: como a membrana seca tendem a rugas quando em contato com a água, submergir as membranas secas um por um para a solução. - Organize as membranas horizontalmente em uma fina camada de água em uma placa de vidro redonda. Esterilizar as membranas por autoclavagem duas vezes.
- Coloque 1 membrana de celofane autoclavada em um prato de Petri quadrado de 12 x 12 cm contendo meio de EKM em agar-solidificado (ver tabela 3, tabela 4). Coloque duas plântulas de P. andersonii com 3 semanas de idade (ver secção 5) ou mudas de 4 semanas de idade (ver secção 1,1) no topo da membrana. Certifique-se de escolher apenas plântulas ou mudas com raízes que têm pontas de raiz branca, indicando que essas raízes ainda estão crescendo.
- Cubra delicadamente as raizes com uma segunda membrana do celofane, criando uma camada do sanduíche. Selar a placa com 3 camadas de folha de vedação elástica. Enrole a metade inferior das placas com folha de alumínio, para cobrir as raízes da exposição à luz.
- Incubar as placas em uma sala de crescimento climatizada (28 ° c, 16 h: 8 horas dia, noite) por 3-4 semanas. Marque a posição das dicas de raiz para seguir o crescimento da raiz ao longo do tempo.
- Se as placas EKM começarem a secar devido à incubação prolongada, transfira as plantas para placas EKM frescas alguns dias antes da inoculação bacteriana.
- Prepare o inônio bacteriano como descrito na etapa 6,1.
- Retire a membrana de celofane superior e aplique 1 mL de cultura de rizóbio (OD600 = 0, 25) às raízes. Posteriormente, coloque uma nova membrana de celofane sobre as raízes inoculadas. Envolva a parte externa da placa usando a folha de alumínio para cobrir as raizes da exposição clara.
- Após 4 semanas, examine os números de nódulo usando um binóculo para determinar a eficiência de nodulação.
8. nodulação de mudas de P. andersonii em bolsas
- Germinar sementes de P. andersonii conforme descrito na secção 1,1. Após os cotilédonos terem emergido totalmente (~ 12 dias em placas SH-0 a 28 ° c), transfira as mudas para as bolsas.
- Para preparar os malotes, rasgue a secção dobrada do pavio de papel e adicione 7 mL de meio EKM modificado (ver tabela 3, tabela 4).
- Insira 1 ou 2 mudas colocando as raízes entre ambas as folhas de papel que formam o pavio de papel e a folha plástica dianteira da bolsa.
- Proteja as raízes da exposição à luz, dobrando a folha de alumínio em torno da bolsa. Suspenda os malotes em uma caixa plástica coberta com uma tampa translúcida para manter a umidade elevada. Coloc a caixa em uma sala de crescimento climatizada (28 ° c, 16 h: dia de 8 horas).
- Compensar a evaporação da água, acrescentando estéril água ultra-pura, como tal que o pavio de papel permanece úmido (Evite pé de água na parte inferior da bolsa). Após a primeira semana, isso geralmente requer a adição de 2-3 ml a cada 4 dias.
- Prepare o inônio bacteriano como descrito na etapa 6,1.
- Após o cultivo de mudas por 10-12 dias em bolsas, inocular o sistema radicular com 500 μL de cultura de rizóbio (OD600 = 0, 25).
- Siga a formação do nódulo através do tempo. Quatro semanas após a inoculação, os nódulos podem ser contados e colhidos para determinar a eficiência da nodulação.
9. análise da Citoarquitetura do nódulo
- Colete 10-15 nódulos em um tubo de 2 mL contendo fixador (5% de glutaraldeído em tampão fosfato de 0,1 M, pH 7,2). Aplique vácuo para 1/2-1 h e incubar durante a noite a 4 ° c. Durante este período, as amostras afundam-se na parte inferior do tubo.
Nota: a solução fixador pode ser armazenada em 4 ° c para ~ 2-4 semanas de uso prévio. Certifique-se de usar luvas ao trabalhar com fixador de tecido. - Lave os nódulos 2x com tampão fosfato 0,1 M, pH 7,2. Aplique intervalos de 10 minutos entre cada etapa de lavagem.
- Desidratar as amostras, posteriormente incubando em 30%, 50%, 70%, e 100% etanol. Para garantir que toda a água é removida das amostras, repita a etapa de etanol 100% 3x. Aplique intervalos de 10 minutos entre cada etapa de desidratação.
- Prepare a mistura de polimerização I (PM-I) adicionando 1 pacote de endurecedor I a 2,5 mL de PEG400 misturado com 100 mL de solução de resina com base em HEMA (2-hidroxietil metacrilato). Mexa a solução para ~ 15 min para dissolver completamente o endurecedor I. Posteriormente, armazene PM-I a-20 ° c.
- Retire o etanol da etapa 9,3. e infiltrar as amostras na seguinte ordem: PM-I:100% etanol (1:3, v/v), PM-I:100% etanol (1:1, v/v), e PM-I:100% etanol (3:1, v/v). Incubar as amostras em cada solução em RT para 1/2-1 h ou até que as amostras afundam para o fundo.
- Incubar amostras durante a noite a 4 ° c em 100% PM-I solução.
- Prepare a mistura de polimerização II misturando PM-I e Hardener II em uma proporção de 15:1 (v/v). Encha o molde plástico com a solução da polimerização, Oriente as amostras horizontalmente na parte inferior do molde, e cubra com uma parte de folha de selagem elástica. Evite a formação de bolhas de ar.
Nota: como a solução começa a polimerizar após a exposição a RT, tente orientar as amostras o mais rapidamente possível no suporte de plástico. A polimerização é concluída após a incubação durante a noite em RT, ou 1 h a 37 ° c. - Retire a tampa da folha de vedação elástica da etapa 9,7 e coloque um suporte para as amostras polimerizadas. Para montar o suporte nas amostras, dissolva 10 mL de pó de resina metacrilato de metilo em 5 mL de solução de resina à base de metacrilato de metilo. Adicione rapidamente a solução ao furo na parte superior do suporte.
Nota: Realize a etapa da polimerização na capa das emanações (~ 30 minutos em RT). - Amostras da seção do micrótomo a uma espessura de 4-5 μm. Coloque uma lâmina de microscópio em uma placa quente de 58 ° c e adicione uma grande gota de água a cada slide. Coloque as secções no topo da água. Uma vez que a água evaporou, as seções aderirão ao slide.
- Lâminas de mancha por imersão em 0, 5% (w/v) azul de toluidina por 2 min. Subsequentemente, enxágüe slides 3x com água ultraleve. Slides podem ser observados usando um microscópio de campo brilhante.
10. micorrização de plantlets de P. andersonii
-
Prepare o inóculo de Rhizophagus irregularis spores
- Prepare uma pilha de filtros de tecido de poliéster com os seguintes tamanhos (de cima para baixo): 210 μm, 120 μm e 36 μm de tamanho de malha.
- Pipeta a quantidade exigida de uma suspensão comercial do Spore na pilha de filtros do poliéster. Enxágüe os filtros 3x com 100 mL de água desmineralizada autoclavada. Os esporos são retidos na superfície do filtro de 36 μm.
Nota: Prepare a suspensão do esporo no gabinete de fluxo cruzado laminar para evitar a contaminação. - Desmontar a pilha de poliéster e manter apenas o filtro de 36 μm. Repita a etapa de lavagem com água desmineralizada autoclavada por pelo menos 6x.
- Coloque o filtro em uma placa de Petri e Resuspenda os esporos em água desmineralizada autoclavada. Use um volume de água igual ao volume da suspensão de esporos usado na etapa 10.1.2. Transfira a suspensão de esporos para um tubo estéril com pipetagem.
- Coloque 5 gotas de 20 μL da suspensão de esporos em uma lâmina de vidro e conte o número de esporos usando um microscópio de campo brilhante. Converter contagens de esporos em uma proporção de esporos/mL e diluir a suspensão de esporos até atingir 250 esporos/mL. Conservar a suspensão de esporos a 4 ° c.
- Realizar ensaio de micorrização. Para este fim, adicione 800 g de areia autoclavada suplementada com 70 mL de meio de 1/2-Hoagland a potes de polipropileno translúcidos estéreis (ver tabelas 5-6). Misture a areia e o meio diretamente na panela agitando vigorosamente.
- Coloque um plantlet p. andersonii em cada pote, e Pipetar 1 ml da suspensão de esporos diretamente na raiz do plantlet p. andersonii . Assegure-se de incluir vários potes contendo plântulas de P. andersonii transformados com uma construção de controle crispr (ver tabela complementar 1).
- Incubar vasos em uma sala de crescimento climatizada (28 ° c, 16 h: 8 dias de noite) durante 6 semanas.
- Retire as plantas das panelas e lave as raízes com água corrente para remover o máximo de areia possível.
- Corte as raízes em 1 cm de comprimento e ferva as peças de raiz em 10% KOH (w/v) por 20 min a 90 ° c. Subseqüentemente, coloc as raizes fervidas em um filtro da pilha com um tamanho de engranzamento de 100 μm e enxague 3x com 50 mL da água.
- Raízes de mancha com 0, 5% (w/v) azul de Tripan em lactoglicerol (300 ml de ácido láctico; 300 ml de glicerol; e 400 ml de água desmineralizada) por 5 min a 90 ° c em um banho de água ou bloco de aquecimento. Subsequentemente, transfira raízes para 30% de glicerol. As amostras de raiz podem ser armazenadas em RT.
- Coloque 15-25 fragmentos de raiz em um único slide de microscópio. Adicione 30% de glicerol e cubra com um vidro de cobertura e pressione até que as peças de raiz se tornem planas. Observar os fragmentos de raiz usando um microscópio de campo brilhante e marcar a colonização micorrízica.
Nota: um método para marcar a micorrização é descrito de acordo com Trouvelot et al.29. Este método utiliza várias classes (% F,% M e% A), o que permite estimar rapidamente o nível de colonização micorrízica de cada fragmento radicular e abundância de arbusculos.
Resultados
P. andersonii tRees pode ser cultivado em estufa condicionada a 28 ° c e ~ 85% de umidade relativa (Figura 1a). Nestas condições, as árvores começam a florescer em 6-9 meses após o plantio. As flores fêmeas de P. andersonii produzem as bagas que cada um contem uma única semente. Durante a maturação, as bagas mudam de cor; primeiro de verde para branco e, subsequentemente, de branco para castanho (Figura 1b). Sementes extraídas das bagas marrons amadurecidas, germinam bem após um ciclo de temperatura de 10 dias e uma incubação de 7 dias em placas SH-0 (Figura 1C). As sementes germinadas continuam a se desenvolver em mudas jovens que podem ser utilizadas para experimentação após ~ 4 semanas (Figura 1D).
Nós mostramos previamente que os pecíolos e os segmentos de hastes jovens de P. andersonii podem eficientemente ser transformados usando A tensão de A. tumefaciens AGL110. No início do procedimento de transformação, os explantes teciduais são cocultivados com a. tumefaciens por 2 dias a 21 ° c (Figura 2a). O cocultivo prolongado resulta na sobrecolonização dos explantes teciduais por a. tumefaciens e deve, portanto, ser prevenido (Figura 2b). Após o período de cocultivo, os explantes teciduais são transferidos para meios seletivos, o que promove o crescimento do tecido transformado. Duas a três semanas mais tarde, os micro-calos verdes pequenos são observados geralmente ao longo da superfície original da ferida (Figura 2C). Estes calos devem continuar a crescer e desenvolver 1 ou mais brotos putatively-transformados em 6-8 semanas depois que o procedimento da transformação foi iniciado (Figura 2D). Nesta fase, as eficiências de transformação tipicamente variam de ~ 10-30% para as transformações iniciadas com explantes teciduais retirados de ramos maduros e parcialmente lenhosas (tabela 7). Se as transformações são iniciadas com explantes retirados das pontas jovens e de crescimento rápido de galhos que ainda não carregam flores, eficiências de transformação de ~ 65-75% podem ser alcançadas (tabela 7). Ocasionalmente, calos esbranquiçados são formados no lado de um explante que não está em contato com o meio e, portanto, não experimentam a seleção de kanamicina. Estes calos muitas vezes não são transgênicos e quaisquer brotos formados a partir destes calos geralmente lixívia e morrem após o contato direto com o meio contendo kanamicina (Figura 2e). Caso a taxa de transformação seja baixa e/ou o material inicial tenha sido suboptimal, as peças teciduais podem virar castanhas (Figura 2F) e sofrer de excesso de proliferação por a. tumefaciens (Figura 2G). Para evitar que a . tumefaciens espalhe e sobrecrescendo explantes próximos, o refresco regular do meio é necessário, e explantes severamente infectados precisam ser removidos. Uma vez que os brotos transgênicos individuais são colocados no meio de propagação, a sobreproliferação por a. tumefaciens geralmente não ocorre mais (Figura 2h). Os brotos transgênicos podem ser multiplicados através da propagação in vitro, o que dará origem a dezenas de brotos em um período de um mês (Figura 3a-B). Estes brotos podem ser colocados em meio de enraizamento, o que deve induzir a formação de raízes após ~ 2 semanas (Figura 3C-D). As plântulas enraizadas podem ser usadas subseqüentemente para a experimentação.
Para criar linhas mutantes de nocaute, fazemos uso de mutagenese mediada por CRISPR/Cas9. Para este fim, nós fazemos o uso de um vetor binário que contem o gene nptIIda resistência do canamicina, uma seqüência do Cas9-Encoding conduzida pelo promotor de CaMV35S e por 2 sgrnas por o gene do alvo que são expressos do AtU6p pequeno promotor20do RNA. Uma representação gráfica do construto utilizado para mutagenese mediada por CRISPR/Cas9 de P. andersonii é fornecida na figura 4a. Usando esse método, a edição do genoma é observada em ~ 40% dos brotos putativamente transformados10. Para identificar as linhas mutantes, as brotações putativamente transformadas são genotipadas para mutações no local de destino do sgRNA usando iniciadores que abrangem a região alvo. Um exemplo dos resultados esperados é dado na Figura 4. Como pode ser visto a partir da foto tirada após a eletroforese em gel, várias amostras produzem um amplicon de PCR com tamanho semelhante ao tipo selvagem (Figura 4B). Essas plantas podem conter pequenos indelos que não podem ser visualizados por eletroforese em gel de agarose ou permanecem inéditos pela enzima Cas9. Além disso, várias amostras produzem bandas que são diferentes em tamanho do tipo Wild (por exemplo, linhas 2, 4, 7 e 8 na Figura 4B). Nestas linhas, 1 (linhas 4, 7 e 8) ou ambos (linha 2) alelos contêm os indels maiores que podem facilmente ser visualizados. A natureza exata das mutações no local de alvo (s) é revelada após o seqüenciamento do amplicon do PCR. Como pode ser visto a partir da Figura 4C, ambos os pequenos indels de 1-4 BP, bem como, deleções maiores podem ser obtidos após a mutagenese de crispr/Cas9. Na Figura 4C, a seqüência da linha 1 é idêntica à do tipo Wild, indicando que essa linha escapou da edição e, portanto, deve ser descartada. Entre as linhagens que contêm mutações, podem ser identificados mutantes heterozygous, homozygous e bi-alélicos (Figura 4C). Entretanto, os mutantes heterozygous são geralmente raros10. Os mutantes de nocaute homozygous ou bi-allelic podem ser propagados vegetativamente para obter o suficiente material para a análise fenotípica. Como a análise fenotípica é realizada na geração T0 , é importante verificar se as linhas mutantes podem ser quiméricas. Para este fim, a genotipagem precisa ser repetida em pelo menos 3 amostras diferentes retiradas de cada linha mutante. Se os resultados de genotipagem forem idênticos entre si e a amostra de genotipagem original (por exemplo, linha 8 na Figura 4D), a linha é homogênea mutada e pode ser usada para análise posterior. No entanto, se os resultados de genotipagem diferirem entre amostras independentes (por exemplo, linha 4 na Figura 4D), a linha mutante é quimérica e precisa ser descartada.
A inoculação de P. andersonii com M. plurifarium BOR2 resulta na formação de nódulos radiculares (Figura 5). Como pode ser observado na Figura 5a, esses nódulos são distribuídos ao longo do sistema radicular. Nódulos de P. andersonii são de cor marrom claro, mas podem ser facilmente discriminados a partir do tecido radicular com base em sua forma (Figura 5b). Experimentos de inoculação em vasos e crescimento subseqüente por 4-6 semanas tipicamente resultam na formação de ~ 10-30 nódulos (Figura 6a). Um número semelhante de nódulos é formado após a inoculação de plântulas de P. andersonii cultivadas em placas de EKM às 4 semanas após a inoculação (Figura 6a). Em bolsas, as mudas de P. andersonii formam tipicamente ~ 5-15 nódulos em 5 semanas após a inoculação (Figura 5C-D, 6a). Para analisar a Citoarquitetura do nódulo, os nódulos podem ser seccionados e observados usando microscopia de campo brilhante. A Figura 6B mostra um exemplo de uma seção longitudinal através do meio de um nódulo de P. andersonii . Esta seção mostra o feixe vascular central de um nódulo de P. andersonii , que é ladeado por lóbulos de nódulo contendo células infectadas (Figura 6B).
As plântulas de P. andersonii também podem ser micorrizadas. Após 6 semanas de inoculação com R. irregularis, a frequência de colonização micorrízica atinge tipicamente > 80% (Figura 6C). Neste momento, geralmente ~ 30% das células contêm arbusculos (Figura 6C). Uma imagem representativa de um segmento de raiz de P. andersonii contendo arbuscles é mostrada na Figura 6D.

Figura 1: imagens representativas de um Árvore de P. andersonii , sementes e seedlings. (A) árvore de seis meses de idade P. andersonii cultivada em solo de potting em estufa condicionada a 28 ° c. (B) imagem representativa representando bagas de P. andersonii em vários estágios de maturação. As bagas novas de P. andersonii (unripe) mudarão a cor do verde ao branco e finalmente ao marrom (maduro) em cima do amadurecimento. (C) sementes de P. andersonii incubadas em meio sh-0 por 1 semana. Um círculo preto indica uma plântula germinada. (D) mudas de P. andersonii de quatro semanas de idade cultivadas em meio sh-0. As barras de escala são iguais a 25 cm em (a) e 1 cm em (B-D). Por favor clique aqui para ver uma versão maior desta figura.
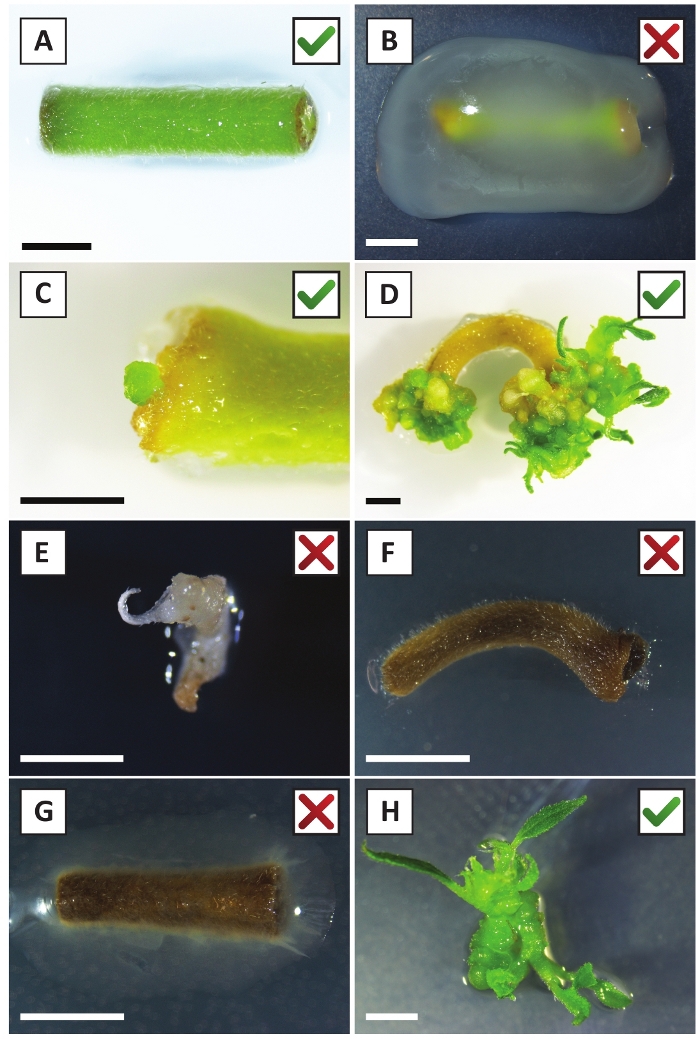
Figura 2: imagens representativas de explantes em diferentes estágios do procedimento de transformação estável. (A) explante co-cultivado com A. tumefaciens. (B) explante overgrown por a. tumefaciens durante as primeiras 2 semanas após a transformação. (C) microcalo transgênico formado perto do local da ferida de um explante em 2,5 semanas após o cultivo. (D) imagem representativa de um explante a 6 semanas após o cultivo, demonstrando o surgimento de brotos de calos (transgênicos). (E) imagem representativa de um tiro que se torna esbranquiçado e eventualmente morre quando em contato direto com o meio contendo canamicina. Este tiro é mais provável não-transgênica e escapou seleção canamicina quando anexado ao explante. (F) imagem representativa de um explante transformado sem sucesso. G) imagem representativa de um explante transformado sem sucesso, sobrecrescido por a. tumefaciens. (H) única parte aérea transgênica cultivada em meio de propagação em 8 semanas pós cocultivo com a. tumefaciens. Barras de escala iguais 2,5 mm. caixas contendo marcas de verificação verdes ou cruzes vermelhas indicam transformação bem-sucedida ou malsucedida de explantes, respectivamente. Por favor clique aqui para ver uma versão maior desta figura.

Figura 3: imagens representativas de in vitro propagação. (A) brotos cultivados em meio de propagação. A imagem foi tomada 1 semana depois que as placas foram refrescadas. (B) brotos cultivados em meio de propagação. A imagem foi tomada 4 semanas depois que as placas foram refrescadas. (C) rebentos recém-cortados colocados no meio de enraizamento. D) brotações incubadas em meio de enraizamento durante 2 semanas. Observe a presença de raízes. As barras de escala são iguais a 2,5 cm. por favor clique aqui para ver uma versão maior desta figura.
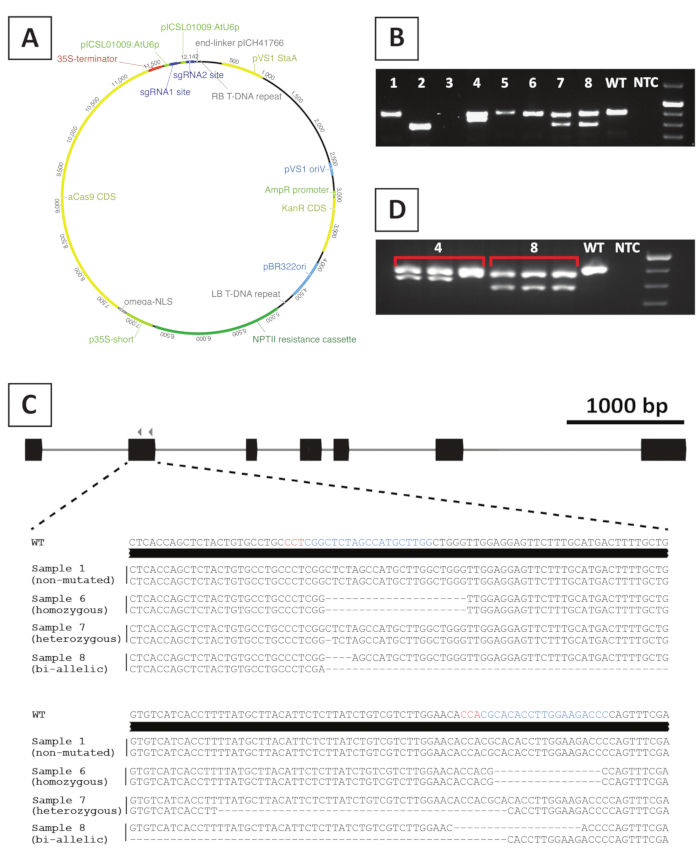
Figura 4: resultados representativos após genotipagem de linhas mutantes transgênicas CRISPR/Cas9 de P. andersonii T0 . (A) mapa representativo de um vetor binário utilizado para mutagenese mediada por crispr/Cas9 de P. andersonii. (B) resultado representativo após a genotipagem baseada em PCR de linhas mutantes potenciais de crispr/Cas9 usando primers que abrangem o (s) local (es) de destino do sgRNA. É mostrada uma imagem após a electroforese do gel do agarose dos amplicons. As amostras retiradas de linhas individuais transgênicas são indicadas por números. O tipo Wild (WT) e nenhum controle de modelo (NTC) indicam faixas contendo controles positivos e negativos, respectivamente. (C) representação esquemática de alelos mutantes obtidos após a edição do gene MEDIADO por crispr/Cas9. Realçado em cores azuis e vermelhas são os locais de destino sgRNA e sequências PAM, respectivamente. (D) resultado representativo após a triagem baseada em PCR para linhas mutantes quiméricas potenciais. É mostrada uma imagem após a electroforese do gel do agarose de 3 amostras individuais retiradas das linhas 4 e 8 do mutante. Note-se que a linha mutante transgênica 4 é quimérico. Por favor clique aqui para ver uma versão maior desta figura.
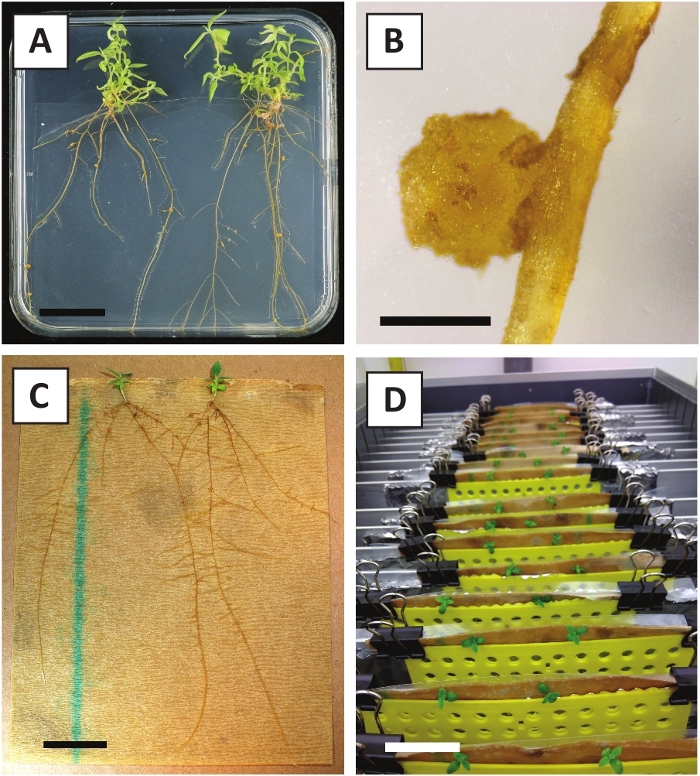
Figura 5: imagens representativas de ensaios de nodulação em placas e bolsas. (A) nodulação em placas contendo meio EKM de agar-solidificado e inoculadas com M. plurifarium BOR2 por 4 semanas. (B) imagem representativa de um nódulo radicular P. andersonii . A imagem foi tomada em 4 semanas após a inoculação com M. plurifarium BOR2. (C) nodulação em bolsas contendo meio EKM líquido. As mudas foram inoculadas com Bradyrhizobium SP. Kelud2A4 por 5 semanas. D) imagem representativa de uma instalação completa utilizada para a nodulação em malotes. As barras de escala são iguais a 2,5 cm em (a, C), 1 mm em (B) e 5 cm em (D). Por favor clique aqui para ver uma versão maior desta figura.
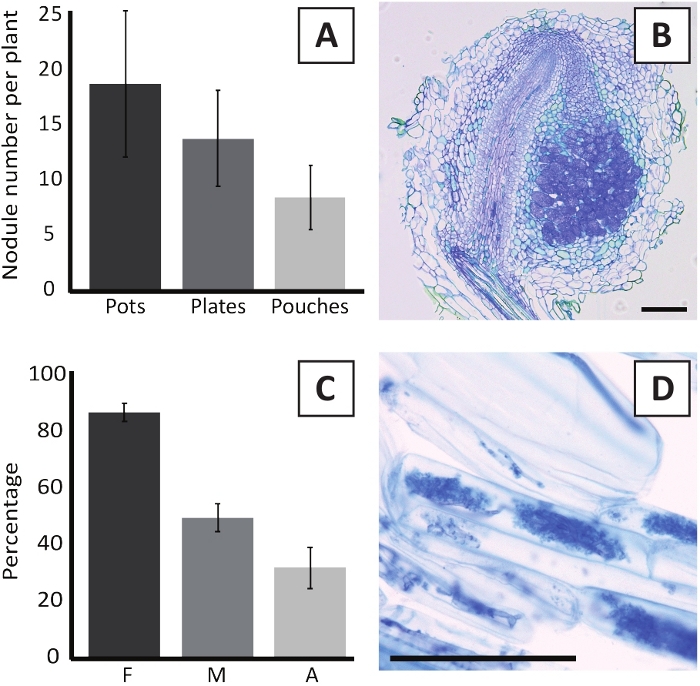
Figura 6: resultados representativos dos ensaios de nodulação e micorrização. (A) gráfico de barras representativo mostrando o número de nódulos formados por planta em 4 semanas após a inoculação com M. plurifarium BOR2 em vasos ou em placas e em 5 semanas após a inoculação com Bradyrhizobium SP. Kelud2A4 em bolsas. Os dados representam média ± DP (n = 10). (B) imagem representativa de uma seção longitudinal através de um nódulo formado em 4 semanas após a inoculação com M. plurifarium BOR2. A seção é manchada com azul do toluidina. (C) gráfico de barras representativo que mostra a quantificação da micorrização. As variáveis quantificadas de acordo com Trouvelot et al.29 são F, a freqüência de fragmentos de raízes analisados que são micorrizados; M, a intensidade da infecção; A, a abundância de arbusculos maduros no sistema radicular total. A micorrização foi quantificada às 6 semanas após a inoculação com R. irregularis (cepa DAOM197198). Os dados representam média ± DP (n = 10). (D) imagem representativa de arbusculos maduros presentes em células corticais da raiz de P. andersonii em 6 semanas após a inoculação com R. irregularis. Barras de escala iguais 75 μm. por favor clique aqui para ver uma versão maior desta figura.
| Composto | SH-0 | SH-10 | Meio de propagação | Meio de enraizamento | Meio de infiltração |
| Meio de sal SH-basal | 3,2 g | 3,2 g | 3,2 g | 3,2 g | 3,2 g |
| Mistura de SH-vitamina | 1 g de | 1 g de | 1 g de | 1 g de | 1 g de |
| Sacarose | - | 10 g de | 20 g de | 10 g de | 10 g de |
| BAP (1 mg/mL) | - | - | 1 mL (4,44 μM) | - | - |
| IBA (1 mg/mL) | - | - | 100 μL (0,49 μM) | 1 mL (4,92 μM) | - |
| NAA (1 mg/mL) | - | - | - | 100 μL (0,54 μM) | - |
| 1 M MES pH = 5,8 | 3 mL de | 3 mL de | 3 mL de | 3 mL de | 3 mL de |
| 1 M DE KOH | Ajuste o pH a 5,8 | Ajuste o pH a 5,8 | Ajuste o pH a 5,8 | Ajuste o pH a 5,8 | Ajuste o pH a 5,8 |
| Daishin Agar | 8 g de | - | 8 g de | 8 g de | - |
Tabela 1: composição dos meios de comunicação baseados em Schenk-Hildebrandt30 utilizados para o cultivo de mudas de P. andersonii , transformação estável e propagação in vitro . Dissolva os compostos sólidos em 750 mL de água ultrapura antes de adicionar estoques líquidos. Em seguida, preencha o meio completo para 1 L. Prepare BAP, IBA, NAA estoques em 0,1 M KOH e armazenar em-20 oC.
| Antes de autoclavagem: | ||
| Composto | Quantidade por litro | Concentração final |
| Manitol | 5 g de | 27,45 milímetros |
| Na-gluconato | 5 g de | 22,92 milímetros |
| Extrato de levedura | 0,5 g | - |
| MgSO4· 7h2O | 0,2 g | 0,81 milímetros |
| Nacl | 0,1 g | 1,71 milímetros |
| K2HPO4 | 0,5 g | 2,87 milímetros |
| Depois de autoclavagem: | ||
| Composto | Quantidade por litro | Concentração final |
| 1,5 M CaCl2 | 1 mL de | 1,5 milímetros |
Tabela 2: composição do meio de levedura-manitol (YEM) utilizado para o cultivo de rizóbio. Ajuste o pH para 7,0 e encha com água ultrapura para 1 L. Para preparar o meio de YEM Agar-solidified, adicione 15 g de microagar antes de autoclavagem.
| Antes de autoclavagem: | |||
| Composto | Concentração de estoque | Quantidade por litro médio | Concentração final |
| KH2po4 | 0,44 M | Adicionar 2 mL | 0,88 milímetros |
| K2HPO4 | 1, 3 M | Adicionar 2 mL | 2, 7 milímetros |
| 500x micro-elementos de solução de ações | - | Adicionar 2 mL | - |
| MES pH = 6,6 | 1 M de | Adicionar 3 mL | de 3 mM |
| Hcl | 1 M de | Ajuste o pH a 6,6 | - |
| Água ultra-pura | - | Preencha até 990 mL | - |
| Depois de autoclavagem: | |||
| Composto | Concentração de estoque | Quantidade por litro médio | Concentração final |
| MgSO4· 7h2O | 1, 4 M | 2 mL de | 2, 8 milímetros |
| Na2so4 | 0,35 M | 2 mL de | 0,70 milímetros |
| NH4no3 | 0,18 M | 2 mL de | 0,36 milímetros |
| CaCl2· 2h2O | 0,75 M | 2 mL de | 1,5 milímetros |
| Fe (III)-citrato | de 27 mM | 2 mL de | 54 μM |
Tabela 3: composição de 1 L modificado EKM médio31 utilizado para o ensaio de nodulação de P. andersonii . A composição da solução de estoque de microelementos 500x está listada na tabela 4. Para preparar 2% de meio de EKM em agar-solidificado, adicione 20 g de agar Daishin antes de autoclavagem. Autoclave o MgSO4· 7h2o, na2so4, CAcl2· 2h2o, e Fe (III)-ações do citrato para esterilizar. Filtro esterilizar NH4no3 solução de estoque para esterilizar.
| Composto | Quantidade por litro | Concentração de estoque |
| MnSO4 | 500 mg | 3,31 milímetros |
| ZnSO4· 7h2O | 125 mg | 0,43 milímetros |
| CuSO4· 5h2O | 125 mg | 0,83 milímetros |
| H3Bo3 | 125 mg | 2, 2 milímetros |
| Na2MoO4· 2h2O | 50 mg | 0,21 milímetros |
Tabela 4: composição da solução de estoque de microelementos 500x usada para preparar o meio EKM modificado. Armazene a solução em estoque de microelementos a 4 ° c.
| Compostos | Concentração de estoque | Quantidade por litro médio | Concentração final |
| K2HPO4 | de 20 mM | 1 mL de | 0,2 milímetros |
| NH4no3 | 0,28 M | 10 mL de | 2,8 milímetros |
| MgSO4 | 40 milímetros | 10 mL de | 0,4 milímetros |
| K2so4 | 40 milímetros | 10 mL de | 0,4 milímetros |
| Fe (II)-EDTA | de 9 mM | 10 mL de | 0,9 milímetros |
| CaCl2 | 80 milímetros | 10 mL de | 0,8 milímetros |
| 50x micro-elementos de solução de ações | - | 10 mL de | - |
Tabela 5: composição do meio 1/2-Hoagland32 utilizado para ensaios de micorrização. A composição da solução de estoque de microelementos 50x está listada na tabela 6. Prepare a solução de Fe (II)-EDTA combinando FeSO4· 7h2O (9 mm) e na2· EDTA (9 mM) em 1 solução de estoque, e armazenar a 4 ° c. Ajuste o pH do meio a 6,1 usando KOH de 1 M e encha com água ultra-pura a 1 L.
| Compostos | Quantidade por litro | Concentração de estoque |
| H3Bo3 | 71,1 mg | 1,15 milímetros |
| MnCl2· 4h2O | 44,5 mg | 0,22 milímetros |
| CuSO4· 5h2O | 3,7 mg | 23,18 μM |
| ZnCl2 | 10,2 mg | 74,84 μM |
| Nd2Moo4· 2h2O | 1,2 mg | 4,96 μM |
Tabela 6: composição da solução de estoque de microelementos 50x utilizada para preparação de meio de Hoagland 1/2.
| Idade dos explantes | Eficiência de transformação |
| Jovem | 69,4 ± 6,2% (n = 2) |
| Maduro | 18,3 ± 10,2% (n = 15) |
Tabela 7: eficiência de transformação de P. andersonii. Aqui, a eficiência de transformação é definida como a porcentagem de explantes que formam pelo menos 1 calo transgênico ou disparar. A eficiência da transformação foi pontuada em 6 semanas após a transformação e é descrita como média ± DP. n indica o número de experimentos de transformação a partir dos quais a eficiência de transformação foi determinada.
Arquivo suplementar 1: visão geral dos constructos de nível 1 e nível 2 usados para mutagenese CRISPR/Cas9. Por favor, clique aqui para baixar este arquivo.
Discussão
As leguminosas e o gênero de Cannabaceae, relacionados à distantly parasponia , representam os únicos dois clados de espécies vegetais capazes de estabelecer uma relação endosimbiótica com rizóbios de fixação de nitrogênio e nódulos radiculares de forma. Estudos comparativos entre espécies de ambos os clados são altamente relevantes para fornecer insights sobre as principais redes genéticas que permitem esta simbiose. Atualmente, os estudos genéticos são feitos principalmente em leguminosas; especialmente as duas espécies-modelo M. Galba e L. japonicus. Para fornecer uma plataforma experimental adicional e facilitar estudos comparativos com um não-legume noduladores, nós descrevemos aqui um protocolo detalhado para a transformação estável e análises genéticas inversas em P. andersonii. O protocolo apresentado utiliza in vitro a propagação de linhas T0 transgênicas de P. andersonii , permitindo que a análise fenotípica seja iniciada dentro de 4 meses após a. tumefaciens co-cultivo. Isto é substancialmente mais rápido do que os protocolos atuais que foram estabelecidos para a transformação estável das leguminosas33. Isso torna o P. andersonii um modelo de pesquisa atraente.
O protocolo descrito aqui contém várias etapas críticas. A primeira delas diz respeito à germinação de sementes. Para preparar sementes de P. andersonii para a germinação, as sementes precisam ser isoladas das bagas. Isto é feito esfregando as bagas em um pedaço de papel de tecido ou contra o interior de uma peneira de chá. Este procedimento precisa de ser executado delicadamente a fim impedir dano ao revestimento de semente. Se o revestimento de semente começ danificado, o alvejante poderia incorporar a semente durante a esterilização, que reduz a viabilidade da semente. Para quebrar a dormência das sementes, as sementes são submetidas a um ciclo de temperatura de 10 dias. No entanto, apesar desse tratamento, a germinação não é totalmente sincronizada. Geralmente, as primeiras sementes mostram a emergência do radícula após 7 dias, mas outro puderam tomar diversos dias mais por muito tempo para germinar.
Os pontos críticos no procedimento de transformação dizem respeito à escolha do material inicial e à duração da etapa de cocultivo. Para alcançar uma transformação eficiente, é melhor usar hastes saudáveis e jovens ou pecíolos de plantas não esterilizadas com efeito de estufa como o material de partida. A fim induzir o crescimento de filiais novas, é aconselhável aparar árvores do Parasponia cada 2-3 meses e refrescar árvores uma vez por ano. Adicionalmente, a etapa do cocultivation precisa de ser executada por 2 dias somente. O cocultivo prolongado promove a sobrecolonização dos explantes teciduais por a. tumefaciens e geralmente reduz a eficiência da transformação. Para evitar a sobrecolonização por a. tumefaciens também é importante atualizar regularmente as placas nas quais os explantes são cultivados. Caso ocorra excesso de colonização, os explantes teciduais podem ser lavados (ver secção 3,8) para remover as células de a. tumefaciens . Aconselhamos a adição de lixívia para a solução SH-10 usada para lavagem (concentração final: ~ 2% de hipoclorito). É importante notar que esta etapa de lavagem adicional pode não funcionar em explantes fortemente infectados (Figura 2b). No caso de uma transformação com uma construção CRISPR/Cas9 produz apenas um número limitado de brotos putativamente transformados ou se a mutagenese de um determinado gene é esperada para causar problemas na regeneração, é aconselhável incluir uma construção de controle de vetor vazio como o controle positivo. Por fim, é importante garantir que todas as linhas transgênicas selecionadas sejam resultantes de eventos de integração de T-DNA independentes. Portanto, instruímos a tomar apenas um único tiro putativamente-transgênico de cada lado de um explante. No entanto, percebemos que isso reduz o número potencial de linhas independentes. Se muitas linhas são exigidas, os investigadores poderiam decidir separar os Calli putatively-transformados dos explantes originais quando estes Calli são ≥ 2 milímetros no tamanho e na cultura estes Calli independentemente. Dessa forma, várias linhas podem ser isoladas de cada explante, o que eleva o número de linhas potenciais transgênicas.
No protocolo atual, linhas transgênicas de P. andersonii são propagadas vegetativamente através da propagação in vitro. A vantagem disso é que muitas plântulas transgênicas podem ser geradas em um período de tempo relativamente curto. No entanto, esse método também tem várias limitações. Em primeiro lugar, a manutenção de linhas transgênicas T0 através da propagação in vitro é trabalhosa e pode resultar em alterações genéticas ou epigenéticas indesejadas34,35. Em segundo lugar, T0 linhas ainda contêm uma cópia do t-DNA, incluindo a gaveta de resistência a antibióticos. Isso limita o número de possíveis re-transformações, como diferentes marcadores de seleção são necessários para cada re-transformação. Atualmente, temos apenas testado transformação usando canamicina ou higromicina seleção (dados não mostrados). Além disso, a presença da seqüência de codificação Cas9 e do sgRNAs nas linhas T0 transgênicas complica os estudos de complementação. Os ensaios de complementação são possíveis, mas exigem que os sites alvo do sgRNA sejam mutados de forma que a edição genética do construto de complementação seja impedida. Em terceiro lugar, uma desvantagem de trabalhar com linhas T0 é que os mutantes Crispr/Cas9 podem ser quiméricos. Para evitar a análise fenotípica de linhagens quiméricas mutantes, recomendamos repetir a análise de genotipagem após a propagação in vitro em pelo menos 3 brotações diferentes. Embora, o número de mutantes quiméricos obtidos usando o protocolo descrito aqui é limitado, são observados ocasionalmente10. Para superar as limitações do trabalho com linhas T0 , as linhas mutantes de P. andersonii poderiam ser propagadas generativamente. As árvores de P. andersonii são dióica e vento-polinated2. Isto significa que cada linha transgénica precisa de ser manipulada como tal que as flores masculinas e fêmeas são produzidas em um único indivíduo, e crescidas subseqüentemente como tais que a polinização transversal não ocorre. Como P. andersonii é uma árvore de crescimento rápido que requer uma quantidade substancial de espaço em uma estufa tropical (28 ° c, ~ 85% umidade relativa). Portanto, embora tecnicamente possível, a propagação generativa de linhas transgênicas de P. andersonii é um desafio logisticamente desafiador.
Na seção protocolo, descrevemos 3 métodos para nodulação de P. andersonii. A vantagem dos sistemas da placa e do malote é que as raizes são facilmente acessíveis, que podem permitir a mancha-inoculação das bactérias e de seguir a formação do nódulo sobre o tempo. No entanto, o sistema de placas é bastante trabalhoso, o que o torna menos adequado para experimentos de nodulação em grande escala. Uma desvantagem do sistema do malote é que é difícil impedir a contaminação fungosa. Os malotes não são estéreis, e conseqüentemente o crescimento fungoso é observado frequentemente na metade superior da bolsa. No entanto, isso não afeta o crescimento de P. andersonii e, portanto, não interfere com os ensaios de nodulação. Adicionalmente, o sistema da bolsa é somente apropriado para seedlings. Apesar de várias tentativas, não conseguimos cultivar plântulas obtidas através da propagação in vitro em bolsas.
O pipeline de genética reversa P. andersonii descrito aqui oferece uma melhoria substancial em comparação com o método de transformação radicular a . rhizogenesexistente11. Usando os procedimentos descritos, as linhas transgénicas estáveis podem ser geradas eficientemente e podem ser mantidas através da propagação in vitro. Em contrapartida, a transformação de A. rhizogenes é transitória e só resulta na formação de raízes transgênicas. Como cada raiz transgênica resulta de uma transformação independente, os ensaios baseados na transformação de A. rhizogenes sofrem de variação fenotípica substancial. Essa variação é muito menor no caso de linhas estáveis, embora a propagação in vitro também crie algum nível de variação. Devido a essa variação reduzida e o fato de que múltiplas plântulas poderiam ser fenotipadas para cada linha estável, linhas estáveis são mais adequadas para ensaios quantitativos em comparação com raízes transformadas de a. rhizogenes. Adicionalmente, a transformação estável não depende da introdução do Locus indutor da raiz de a. rhizogenes (rol) que afeta o equilíbrio hormonal endógeno15. Conseqüentemente, as linhas estáveis são seridas melhor para a análise genética reversa dos genes envolvidos na homeostase da hormona comparada a . rizógenes-raizes transformadas. Uma vantagem mais geral do P. andersonii como modelo de pesquisa é que não teve uma recente duplicação do genoma (wgd). A leguminosa Papilionoideae subfamília, que inclui as leguminosas modelo M. Galba e L. japonicus, bem como o Salicaceae (ordem Malpighiales) que inclui a árvore modelo Populus neotorularia experiente wgds ~ 65 milhões de anos atrás36,37. Muitas cópias genéticas trypsinogen resultantes destes wgds são mantidas nos genomas de M. Galba, L. japonicus e P. neotorularia37,38,39, que cria redundância que possa complicar análises genéticas inversas. Como p. andersonii não experimentaram um wgd recente, as análises genéticas reversas em p. andersonii puderam ser menos afetadas pelo funcionamento redundante de cópias de gene trypsinogen.
Tomados em conjunto, fornecemos um protocolo detalhado para análise genética reversa em P. andersonii. Usando este protocolo, as únicas linhas do mutante podem eficientemente ser geradas em um período de 2-3 meses10. Este protocolo pode ser estendido para criar mutantes de ordem superior através da multiplexação de sgrnas visando diferentes genes simultaneamente, como mostrado para outras espécies de plantas40,41,42. Além disso, o procedimento de transformação estável descrito aqui não se limita à segmentação por genes CRISPR/Cas9, mas também pode ser usado para introduzir outros tipos de construções (por exemplo, para ensaios de promotor-repórter, expressão ectópica ou Trans- complementação). Nós estabelecemos P. andersonii como um modelo de pesquisa comparativa para estudar simbiososes mutualistas com rizobia de fixação de nitrogênio ou fungos endomicorrízicos. No entanto, os protocolos aqui descritos também fornecem ferramentas para estudar outros aspectos da biologia desta árvore tropical, como a formação de madeira, o desenvolvimento de flores bi-sexuais ou a biossíntese de metabólitos secundários específicos de Cannabaceae.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores gostam de reconhecer Mark Youles, Sophien Kamoun e Sylvestre Marillonnet para fazer peças de clonagem Golden Gate disponíveis através do banco de dados Addgene. Além disso, gostaríamos de agradecer a E. James, P. Hadobas, e T. J. Higgens por sementes de p. andersonii . Este trabalho foi apoiado pela organização dos Países Baixos para a investigação científica (NWO-VICI Grant 865.13.001; NWO-concurso aberto 819.01.007) e do Ministério da investigação, tecnologia e ensino superior da República da Indonésia (RISET-PRO Grant 8245-ID).
Materiais
| Name | Company | Catalog Number | Comments |
| Sigma-Aldrich | N0640 | NAA | |
| Duchefa Biochemie | M1503.0250 | MES | |
| Sigma-Aldrich | D134406 | Acetosyringone | |
| Duchefa Biochemie | X1402.1000 | X-Gal | |
| Merck | 101236 | For nucleic acid electrophoresis gel | |
| - | - | Pouches box material, hangers | |
| Merck | 101188 | NH4NO3 | |
| Sigma-Aldrich | B3408-1G | BAP | |
| Merck | 100156 | H3BO3 | |
| Thermo-Fisher | ER1011 | Used as restriction enzyme in Golden Gate cloning assembly | |
| Thermo-Fisher | 15561020 | Used in Golden Gate cloning assembly | |
| Merck | 137101 | CaCl2·2H2O | |
| Duchefa Biochemie | C0111.0025 | C16H16N5O7S2Na | |
| Thermo-Fisher | K1231 | Used for cloning the blunt-ended PCR amplicons in genotyping procedure | |
| Agronutrition | AP2011 | Containing Rhizophagus irregularis DAOM 197198 (1,000 spores/mL), used for mychorrization assay | |
| Merck | 102790 | CuSO4·5H2O | |
| Duchefa Biochemie | D1004.1000 | Used for plant tissue culture agar-based medium | |
| Merck | 105101 | K2HPO4 | |
| VWR Chemicals | 20302.293 | Na2·EDTA | |
| Duchefa Biochemie | M0803.1000 | C6H14O6 | |
| Thermo-Fisher | ER0291 | Used as restriction enzyme in Golden Gate cloning assembly | |
| Merck | 100983 | C2H5OH | |
| VWR Chemicals | BDH9232-500G | EDTA | |
| Sigma-Aldrich | Z377600-1PAK | Cellophane membrane | |
| Biomatters, Ltd. | R9 or higher | Bioinformatics software for in silico cloning and designing of sgRNAs | |
| Mega International | - | Technical information at https://mega-international.com/tech-info/ | |
| Sigma-Aldrich | 65882 | Used for fixating nodule tissues | |
| VWR Chemicals | 24385.295 | - | |
| Vink | 219341 | Pouches box material, bottom part | |
| Leica Biosystems | 14702218311 | Used as a template for plastic embedding | |
| Merck | 100317 | HCl | |
| Sigma-Aldrich | I5386-1G | IBA | |
| Merck | 103862 | C6H5FeO7 | |
| Merck | 103965 | FeSO4O·7H2O | |
| Duchefa Biochemie | I1401.0005 | IPTG | |
| Duchefa Biochemie | K0126.0010 | ||
| Sigma-Aldrich | L2000 | ||
| Merck | 105886 | MgSO4O·7H2O | |
| Merck | 105934 | MnCl2·4H2O | |
| Merck | 102786 | MnSO4O | |
| Duchefa Biochemie | M1002.1000 | Used for bacterial culture agar-based medium | |
| Manutan | 92007687 | Pouches material | |
| Paraxisdienst | 130774 | Elastic sealing foil | |
| Pull Rhenen | Agra-Perlite No.3 | Used as growing substrate in pots for nodulation assay | |
| VWR Chemicals | 391-0581 | Used as container for cellophane membranes | |
| Thermo-Fisher | F130WH | For genotyping transgenic lines | |
| Addgene | 50337 | Level 0 terminator, 3'UTR, 35s (Cauliflower Mosaic Virus) | |
| Addgene | 48017 | End-link 2 for assembling 2 level one part into a level 2 acceptor | |
| Addgene | 48018 | End-link 3 for assembling 3 level one part into a level 2 acceptor | |
| Addgene | 48001 | Level 1 acceptor. Position 5. Forward orientation | |
| Addgene | 48007 | Level 1 Acceptor. Position 1. Reverse orientation | |
| Addgene | 50268 | Level 0 promoter (0.4 kb), 35s (Cauliflower Mosaic Virus) + 5'UTR, Ω (Tobacco Mosaic Virus) | |
| Addgene | 46966 | Used for designing CRISPR/Cas9 module | |
| Addgene | 46968 | Used for designing CRISPR/Cas9 module | |
| Addgene | 50334 | Level 0 Kanamycin/Neomycin/Paromomycin resistance cassette | |
| Topzeven | - | Used as filters for washing spore suspension | |
| Sigma-Aldrich | 8.17003 | PEG400 | |
| Duchefa Biochemie | E1674.0001 | Pots to grow Parasponia plantlets/seedlings | |
| Merck | 104871 | KH2PO4 | |
| Merck | 105033 | KOH | |
| Merck | 105153 | K2SO4O | |
| Van Leusden b.v. | - | Used as growing substrate for mychorrhization assay | |
| Duchefa Biochemie | S0225.0050 | SH-basal salt medium | |
| Duchefa Biochemie | S0411.0250 | SH-vitamin mixture | |
| Lehle Seeds | VIS-02 | Used as non-ionic surfactant in the washing step of stable transformation | |
| Merck | 137017 | NaCl | |
| VWR Chemicals | 89230-072 | C6H11NaO7 | |
| Merck | 106521 | Na2MoO4·2H2O | |
| Merck | 106574 | Na2HPO4·7H2O | |
| Merck | 567549 | NaH2PO4·H2O | |
| Sigma-Aldrich | 239313 | Na2SO4O | |
| Duchefa Biochemie | S0809.5000 | C12H22O11 | |
| Thermo-Fisher | B69 | Used in Golden Gate cloning assembly | |
| Thermo-Fisher | EL0013 | Used in Golden Gate cloning assembly | |
| Kulzer-Mitsui Chemicals Group | 64708806 | Methyl methacrylate-based resin powder | |
| Kulzer-Mitsui Chemicals Group | 64709003 | HEMA (2-hydroxyethyl methacrylate)-based resin solution | |
| Kulzer-Mitsui Chemicals Group | 66022678 | Methyl methacrylate-based resin solution | |
| Merck | 1159300025 | ||
| Acros | 189350250 | ||
| VWR Chemicals | 663684B | Polysorbate 20 | |
| Stout Perspex | - | pouches box material, lid | |
| Duchefa Biochemie | Y1333.1000 | ||
| Merck | 108816 | ZnCl2 | |
| Alfa Aesar | 33399 | ZnSO4O·7H2O |
Referências
- Clason, E. W. THE VEGETATION OF THE UPPER-BADAK REGION OF MOUNT KELUT (EAST JAVA). Bulletin du Jardin Botanique de Buitenzorg. Serie III, 509-518 (1936).
- Soepadmo, E. Ulmaceae. Flora Malesiana-Series 1, Spermatophyta. 8, 31-76 (1974).
- Becking, J. H. The Rhizobium symbiosis of the nonlegume Parasponia. Biological Nitrogen Fixation. , 497-559 (1992).
- Oldroyd, G. E. D. Speak, friend, and enter: signalling systems that promote beneficial symbiotic associations in plants. Nature Reviews Microbiology. 11, 252-263 (2013).
- Gutjahr, C., Parniske, M. Cell and developmental biology of arbuscular mycorrhiza symbiosis. Annual Review of Cell and Developmental Biology. 29, 593-617 (2013).
- van Velzen, R., et al. Comparative genomics of the nonlegume Parasponia reveals insights into evolution of nitrogen-fixing rhizobium symbioses. Proceedings of the National Academy of Sciences of the United States of America. 115, E4700-E4709 (2018).
- van Velzen, R., Doyle, J. J., Geurts, R. A Resurrected Scenario: Single Gain and Massive Loss of Nitrogen-Fixing Nodulation. Trends in Plant Science. 24, 49-57 (2019).
- Griesmann, M., et al. Phylogenomics reveals multiple losses of the nitrogen-fixing root nodule symbiosis. Science. 1743, eaat1743 (2018).
- Becking, J. H. Root-Nodule Symbiosis Between Rhizobium And Parasponia (Ulmaceae). Plant and Soil. 51, 289-296 (1979).
- van Zeijl, A., et al. CRISPR/Cas9-Mediated Mutagenesis of Four Putative Symbiosis Genes of the Tropical Tree Parasponia andersonii Reveals Novel Phenotypes. Frontiers in Plant Science. 9, 284 (2018).
- Cao, Q., et al. Efficiency of Agrobacterium rhizogenes–mediated root transformation of Parasponia and Trema is temperature dependent. Plant Growth Regulation. 68, 459-465 (2012).
- Limpens, E., et al. RNA interference in Agrobacterium rhizogenes-transformed roots of Arabidopsis and Medicago truncatula. Journal of Experimental Botany. 55, 983-992 (2004).
- Boisson-Dernier, A., et al. Agrobacterium rhizogenes-Transformed Roots of Medicago truncatula for the Study of Nitrogen-Fixing and Endomycorrhizal Symbiotic Associations. Molecular Plant-Microbe Interactions. 14, 695-700 (2001).
- Kumagai, H., Kouchi, H. Gene Silencing by Expression of Hairpin RNA in Lotus japonicus Roots and Root Nodules. Molecular Plant-Microbe Interactions. 16, 663-668 (2003).
- Nilsson, O., Olsson, O. Getting to the root: The role of the Agrobacterium rhizogenes rol genes in the formation of hairy roots. Physiologia Plantarum. 100, 463-473 (1997).
- Davey, M. R., et al. Effective Nodulation of Micro-Propagated Shoots of the Non-Legume Parasponia andersonii by Bradyrhizobium. Journal of Experimental Botany. 44, 863-867 (1993).
- Webster, G., Poulton, P. R., Cocking, E. C., Davey, M. R. The nodulation of micro-propagated plants of Parasponia andersonii by tropical legume rhizobia. Journal of Experimental Botany. 46, 1131-1137 (1995).
- Op den Camp, R., et al. LysM-type mycorrhizal receptor recruited for rhizobium symbiosis in nonlegume Parasponia. Science. 331, 909-912 (2011).
- Weber, E., Engler, C., Gruetzner, R., Werner, S., Marillonnet, S. A Modular Cloning System for Standardized Assembly of Multigene Constructs. PLOS ONE. 6, e16765 (2011).
- Nekrasov, V., Staskawicz, B. J., Weigel, D., Jones, J. D. G., Kamoun, S. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease. Nature Biotechnology. 31, 691-693 (2013).
- Bertani, G. Studies On Lysogenesis. I. The Mode Of Phage Liberation By Lysogenic Escherichia Coli. Journal of Bacteriology. 62, 293-300 (1951).
- Engler, C., et al. A Golden Gate modular cloning toolbox for plants. ACS Synthetic Biology. 3, 839-843 (2014).
- Fauser, F., Schiml, S., Puchta, H. Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering in Arabidopsis thaliana. The Plant Journal. 79, 348-359 (2014).
- Lazo, G. R., Stein, P. A., Ludwig, R. A. A DNA Transformation-Competent Arabidopsis Genomic Library in Agrobacterium. Biotechnology. 9, 963-967 (1991).
- Op den Camp, R. H. M., et al. N Nonlegume Parasponia andersonii Deploys A Broad Rhizobium Host Range Strategy Resulting in Largely Variable Symbiotic Effectivenes. Molecular Plant-Microbe Interactions. 25, 954-963 (2012).
- Graham, P. H., Viteri, S. E., Mackie, F., Vargas, A. T., Palacios, A. Variation in acid soil tolerance among strains of Rhizobium phaseoli. Field Crops Research. 5, 121-128 (1982).
- Martinez-Romero, E., et al. Rhizobium tropici, A Novel Species Nodulating Phaseolus vulgaris L. Beans and Leucaena sp. Trees. International Journal of Systematic and Evolutionary Microbiology. 41, 417-421 (1991).
- Felten, J., et al. The Ectomycorrhizal Fungus Laccaria bicolor Stimulates Lateral Root Formation in Poplar and Arabidopsis through Auxin Transport and Signaling. Plant Physiology. 151, 1991-2005 (1991).
- Trouvelot, A., Kough, J. L., Gianinazzi-Pearson, V. Mesure du taux de mycorhization VA d’un systeme radiculaire. Recherche de methods d’estimation ayant une signification fonctionnelle. Aspects Physiologiques et Genetiques des Mycorhizes. , 217-221 (1986).
- Schenk, R. U., Hildebrandt, A. C. Medium and techniques for induction and growth of monocotyledonous and dicotyledonous plant cell cultures. Canadian Journal of Botany. 50, 199-204 (1972).
- Becking, J. H. The Parasponia parviflora - Rhizobium symbiosis. Host specificity, growth and nitrogen fixation under various conditions. Plant and Soil. 75, 309-342 (1983).
- Hoagland, D. R., Arnon Revised, D. I., Arnon, D. I. The Water-Culture Method for Growing Plants without Soil. Circular California Agricultural Experiment Station. 347, 1-32 (1950).
- Wang, K. . Methods in Molecular Biology: Agrobacterium Protocols. 1, (2015).
- Smulders, M. J. M., de Klerk, G. J. Epigenetics in plant tissue culture. Plant Growth Regulation. 63, 137-146 (2011).
- Larkin, P. J., Scowcroft, W. R. Somaclonal variation — a novel source of variability from cell cultures for plant improvement. Theoretical and Applied Genetics. 60, 197-214 (1981).
- Cannon, S. B., et al. Legume genome evolution viewed through the Medicago truncatula and Lotus japonicus genomes. Proceedings of the National Academy of Sciences of the United States of America. 103, 14959-14964 (2006).
- Tuskan, G. A., et al. The Genome of Black Cottonwood, Populus trichocarpa (Torr. & Gray). Science. 313, 1596-1604 (2006).
- Young, N. D., et al. The Medicago genome provides insight into the evolution of rhizobial symbioses. Nature. 480, 520 (2011).
- Sato, S., et al. Genome Structure of the Legume, Lotus japonicus. DNA Research. 15, 227-239 (2008).
- Xing, H. L., et al. A CRISPR/Cas9 toolkit for multiplex genome editing in plants. BMC Plant Biology. 14, 327 (2014).
- Lowder, L. G., et al. A CRISPR/Cas9 Toolbox for Multiplexed Plant Genome Editing and Transcriptional Regulation. Plant Physiology. 169, 971 (2015).
- van Zeijl, A. . Dissecting Hormonal Pathways in Nitrogen-Fixing Rhizobium Symbioses. , (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados