Transformando, Edición del Genoma y Fenotipando el árbol tropical de cannabaceae de fijación de nitrógeno Parasponia andersonii
In This Article
Summary
Parasponia andersonii es un árbol tropical de rápido crecimiento que pertenece a la familia Cannabis (Cannabaceae) y puede formar nódulos radiculares fijados al nitrógeno en asociación con el rizobio. Aquí, describimos un protocolo detallado para análisis genéticos inversos en P. andersonii basado en Agrobacterium tumefaciens-mediada transformación estable y edición del genoma basada en CRISPR/Cas9.
Abstract
Parasponia andersonii es un árbol tropical de rápido crecimiento que pertenece a la familia Cannabis (Cannabaceae). Junto con 4 especies adicionales, forma el único linaje no leguminoso conocido capaz de establecer una simbiosis nódula fijadora de nitrógeno con rizobio. Los estudios comparativos entre las legumbres y P. andersonii podrían proporcionar información valiosa sobre las redes genéticas subyacentes a la formación de nódulos radiculares. Para facilitar los estudios comparativos, recientemente secuenciamos el genoma de P. andersonii y establecimos Agrobacterium tumefaciens-mediada en transformación estable y edición del genoma basada en CRISPR/Cas9. Aquí, proporcionamos una descripción detallada de los procedimientos de transformación y edición del genoma desarrollados para P. andersonii. Además, describimos los procedimientos para la germinación de semillas y la caracterización de fenotipos simbióticos. Usando este protocolo, las líneas mutantes transgénicas estables se pueden generar en un período de 2-3 meses. La propagación vegetativa in vitro de líneas transgénicas T0 permite iniciar experimentos de fenotipado a los 4 meses después del cocultivo de A. tumefaciens. Por lo tanto, este protocolo toma sólo marginalmente más tiempo que el método transitorio de transformación de raíz basado en Agrobacterium rhizogenesdisponible para P. andersonii, aunque ofrece varias ventajas claras. Juntos, los procedimientos descritos aquí permiten que P. andersonii sea utilizado como un modelo de investigación para estudios dirigidos a entender las asociaciones simbióticas, así como potencialmente otros aspectos de la biología de este árbol tropical.
Introduction
Parasponia andersonii es un árbol tropical perteneciente a la familia Cannabis (Cannabaceae) y es nativo de Papúa Nueva Guinea y varias Islas del Pacífico1,2,3. Junto con 4 especies adicionales de Parasponia, representa el único linaje no leguminoso que puede establecer una simbiosis nódula fijante de nitrógeno con rizobia. Esta simbiosis está bien estudiada en los modelos de leguminosas (Fabaceae) Medicago truncatula y Lotus japonicus,lo que ha dado como resultado la adquisición de un conocimiento detallado de la naturaleza genética molecular de la formación de nódulos y el funcionamiento4. Además, se demostró que la simbiosis del nódulo raíz en las legumbres se basa enla simbiosis micorriza arbuscular mucho más antigua y generalizada 5. Las comparaciones filogenómicas sugieren que las simbiosis de nódulos fijadores de nitrógeno de las legumbres, Parasponia,así como las llamadas especies de plantas actinorhizales que albergan bacterias Defrankia diazotroficas, tienen un origen evolutivo compartido 6,7,8. Para determinar si los genes identificados para participar en la formación de nódulos de leguminosas son la parte de una base genética conservada, los estudios sobre especies no leguminosas son esenciales. Con este fin, proponemos utilizar P. andersonii como modelo de investigación comparativa, junto con las legumbres, para identificar las redes genéticas básicas subyacentes a la formación y funcionamiento del nódulo raíz.
P. andersonii es un pionero que se puede encontrar en las laderas de las colinas volcánicas. Puede alcanzar velocidades de crecimiento de 45 cm por mes y alcanzar longitudes de hasta 10 metros9. Los árboles P. andersonii son polinizados por el viento, lo que se ve facilitado por la formación de flores separadas masculinas y femeninas3,10. Recientemente secuenciamos y anotamos el genoma diploide (2n a 20; 560 Mb/1C) de P. andersonii,y ensamblamos secuencias de genoma de borrador de 2 especies adicionales de Parasponia; P. rigida y P. rugosa6. Esto reveló modelos de genes de 35.000 P. andersonii que se pueden agrupar en >20.000 ortogrupos junto con genes de M. truncatula, soja (Glycine max), Arabidopsis thaliana, bosque de fresas ( Fragaria vesca), Trema orientalis, álamo de algodón negro (Populus trichocarpa) y eucalipto (Eucalyptus grandis)6. Además, las comparaciones de transcriptoma entre M. truncatula y P. andersonii identificaron un conjunto de 290 ortologos putativos que muestran un patrón de expresión mejorado con nódulos en ambas especies6. Esto proporciona un excelente recurso para los estudios comparativos.
Para estudiar la función génica en las raíces y nódulos de P. andersonii, se ha establecido un protocolo para la transformación de la raíz mediada por Agrobacterium rhizogenes11. Usando este protocolo, las plantas compuestas que soportan raíces transgénicas se pueden generar en un marco de tiempo relativamente corto. Este método es, también, ampliamente aplicado en la investigación de leguminosis12,13,14. Sin embargo, la desventaja de este método es que sólo las raíces se transforman y que cada raíz transgénica representa un evento de transformación independiente, lo que resulta en una variación sustancial. Además, la transformación es transitoria y las líneas transgénicas no se pueden mantener. Esto hace que la transformación de raíz basada en Rhizogenessea menos adecuada para la edición del genoma mediado por CRISPR/Cas9. Además, A. rhizogenes transfiere sus genes de locus inductor de raíz (rol) al genoma de la planta, que una vez expresado interfieren con la hormona homeostasis15. Esto hace que estudiar el papel de las hormonas vegetales en A. rizogenes-raíces transformadas desafiante. Para superar estas limitaciones, recientemente desarrollamos un protocolo para la transformación basada en Agrobacterium tumefaciensy la mutagénesis mediada por CRISPR/Cas9 de P. andersonii10.
Aquí, proporcionamos una descripción detallada del procedimiento de transformación basado en A. tumefaciensy la canalización de genética inversa desarrollada para P. andersonii. Además, proporcionamos protocolos para el manejo posterior de plantlets transgénicos, incluyendo ensayos para estudiar interacciones simbióticas. Usando el protocolo descrito aquí, se pueden generar múltiples líneas transgénicas en un período de 2-3 meses. En combinación con la mutagénesis mediada por CRISPR/Cas9, esto permite una generación eficiente de líneas mutantes noqueadas. Estas líneas mutantes pueden propagarse vegetativamente in vitro10,16,17, lo que permite generar material suficiente para iniciar la caracterización fenotípica a los 4 meses después de que el procedimiento de transformación iniciado10. Juntos, este conjunto de procedimientos debe permitir que cualquier laboratorio adopte P. andersonii como modelo de investigación para estudios dirigidos a entender las asociaciones rizobialyas y micorrizas, así como potencialmente otros aspectos de la biología de este árbol tropical.
Protocol
1. Cultivar árboles P. andersonii en el invernadero
-
Germinate P. andersonii WU1 semillas18.
- Usa bayas frescas de Parasponia o remoje las bayas secas en agua durante 2 h para rehidratar. Aplasta las bayas en un pedazo de papel tisú o frota contra el interior de un tamiz de té para eliminar las semillas.
- Desinfectar las semillas con lejía comercial (4% hipoclorito) durante 15-20 min y posteriormente lavar las semillas 6 veces usando agua esterilizada.
- Transfiera las semillas a tubos de PCR estériles de 200 l. Llene los tubos con agua esterilizada, de tal forma que las semillas estén completamente sumergidas. Incubar los tubos durante 10 días en un termociclador ejecutando el siguiente programa: 30 ciclos (7 oC durante 4 h, 28 oC durante 4 h). No utilice una tapa calentada, ya que esto podría matar las semillas.
- Prepare las placas SH-0 (véase el Cuadro 1). Transfiera las semillas a placas SH-0 e incubar a 28oC, 16 h:8 h día:noche. Cierre las placas con 2 capas de lámina de sellado elástica para evitar el secado durante la incubación a 28 oC.
- Después de que las plántulas hayan desarrollado su primer conjunto de hojas verdaderas (3-4 semanas después de la incubación a 28 oC), transfiera las plántulas a macetas llenas de tierra de macetas comerciales y cubra las plántulas con una copa de plástico translúcida para evitar la desecación. Colocar macetas en una sala climática o invernadero de 28oC, 85% de humedad relativa, bajo un régimen de 16 h:8 h día:noche.
- Después de 1 semana, retire la taza de plástico translúcido. Agua las ollas regularmente y cuando los árboles crecen suplemento con fertilizante para sostener el crecimiento.
2. Clonación de construcciones para mutagénesis mediada por CRISPR/Cas9 de P. andersonii
NOTA: Los vectores de transformación binaria estándar se pueden utilizar para la transformación estable de P. andersonii. Aquí, por ejemplo, es un procedimiento para generar construcciones para mutagénesis mediada por CRISPR/Cas9 utilizando clonación modular (por ejemplo, Golden Gate)19.
- Identifique las secuencias de destino de ARN guía para los genes de interés, utilizando un software bioinformático con una herramienta de diseño CRISPR integrada. Elija secuencias de ARN guía ubicadas en el extremo 5 de la secuencia de codificación del gen objetivo para aumentar la posibilidad de obtener eliminatorias completas. Asegúrese de comprobar si hay efectos fuera del objetivo mediante la búsqueda contra el genoma de P. andersonii 6.
NOTA: Utilice 2 sgRNA por gen diana, preferiblemente 200-300 bp de separación. Esto puede generar deleciones que pueden ser identificadas por PCR y posteriormente por electroforesis de gel de agarosa. - Genera construcciones Golden Gate de nivel 1 que contienen las secuencias sgRNA.
- Diseñar imprimaciones para amplificar cada sgRNA individual insertando la secuencia guía de 20 bp en la posición de N(20) en la siguiente secuencia de imprimación: 5'-TGTGGTCTCAATTGN(20) GTTTTACTACTAGAAATAGCAAG-3'.
NOTA: Si la secuencia de guía es igual a GN(19),retire la G en el extremo de 5' de la secuencia de guía antes de insertarla en la secuencia de imprimación. - PCR amplifica los sgRNA de pICH86966::AtU6p::sgRNA_PDS20 utilizando las imprimaciones delanteras diseñadas en el paso 2.2.1 y la imprimación inversa universal: 5'-TGTGGTCTCAAGCGTAATGCCAACTTTGTAC-3'. Utilice una polimerasa de ADN de alta fidelidad estable al calor y las siguientes condiciones de PCR: 98 oC durante 30 s; 30 ciclos (98oC para 10 s; 53oC para 20 s; 72oC para 10 s); 72 oC para 7 min. Las reacciones de PCR exitosas producen un amplificador de 165 bp.
- Purificar columnas el amplificador de PCR utilizando un kit de purificación de PCR comercial. Posteriormente, establecer reacciones Golden Gate a los sgRNAs clon detrás del arabidopsis thaliana AtU6p pequeño promotor de ARN: 10 ng del amplicon sgRNA PCR, 150 ng de pICSL01009::AtU6p20, 60 ng del vector de aceptación de nivel 1 apropiado, 2 l de T4 tampón de ligasa, 2 L 0,1% de albúmina sérica bovina (BSA), 0,5 l de BsaI, 0,5 l de ligasa T4, llenar a 20 l con agua ultrapura. Asegúrese de que todos los sgRNA se clonan en la misma orientación para evitar la formación de horquillas.
- Incubar reacciones en un termociclador que ejecuta el siguiente programa: 37 oC para 20 s; 26 ciclos (37oC durante 3 min; 16oC durante 4 min); 50 oC durante 5 min; 80 oC para 5 min. Transformar reacciones Golden Gate a Escherichia coli y placa en lb medio21 que contiene ampicilina (50 mg/L), X-Gal (200 mg/L) e IPTG (1 mM).
NOTA: Prepare soluciones de stock de IPTG y X-Gal en agua ultrapura y dimetilformamida, respectivamente. Filtrar esterilizar las soluciones de stock de ampicilina e IPTG y almacenar todas las existencias a -20 oC. Use guantes cuando manipule dimetilformamida. - Seleccione colonias blancas y aísle los plásmidos utilizando un kit de aislamiento de plásmido comercial. La secuencia verifica los plásmidos aislados antes de continuar con el ensamblaje de nivel 2 de Golden Gate.
- Diseñar imprimaciones para amplificar cada sgRNA individual insertando la secuencia guía de 20 bp en la posición de N(20) en la siguiente secuencia de imprimación: 5'-TGTGGTCTCAATTGN(20) GTTTTACTACTAGAAATAGCAAG-3'.
- Montar construcciones Golden Gate de nivel 2 para la transformación estable.
- Realice una reacción Golden Gate utilizando las construcciones AtU6p::sgRNA de nivel 1 (generadas en la sección 2.2), así como pICH47802::NPTII, pICH47742::35Spro::-NLS-aCas9::35Ster, el propietario de nivel 2 pICSL4723 y el apropiado end-linker (véase Engler et al.22). Realice las reacciones de la siguiente manera: utilice 100 fmol de cada vector de donante y 20 fmol del vector de aceptación y agregue 2 ml de tampón de ligasa T4, 2 l de BSA al 0,1%, 0,5 ml de BpiI, 0,5 l de ligados T4, llene a 20 l con agua ultrapura.
NOTA: Los plásmidos de nivel 1 pICH47802::NPTII, pICH47742::35Spro::-NLS-aCas9::35Ster necesita ser clonado primero (ver Archivo Suplementario 1), como se describe para sgRNAs en la sección 2.220,22 ,23. - Incubar reacciones como en el paso 2.2.4 y transformarse en E. coli. Placa en el medio LB que contiene kanamicina. Al día siguiente, seleccione colonias blancas y aísle los plásmidos. Determine el conjunto de plásmido correcto mediante el análisis de restricción-digestión.
- Realice una reacción Golden Gate utilizando las construcciones AtU6p::sgRNA de nivel 1 (generadas en la sección 2.2), así como pICH47802::NPTII, pICH47742::35Spro::-NLS-aCas9::35Ster, el propietario de nivel 2 pICSL4723 y el apropiado end-linker (véase Engler et al.22). Realice las reacciones de la siguiente manera: utilice 100 fmol de cada vector de donante y 20 fmol del vector de aceptación y agregue 2 ml de tampón de ligasa T4, 2 l de BSA al 0,1%, 0,5 ml de BpiI, 0,5 l de ligados T4, llene a 20 l con agua ultrapura.
- Transform de nivel 2 construye a la cepa Agrobacterium tumefaciens AGL124.
3. Transformación estable de P. andersonii
- Inocular 2 placas LB que contienen los antibióticos apropiados con cepa A. tumefaciens AGL1 transformada con la construcción de interés. Incubar placas a 28oC durante 2 días.
- Cosecha ramas jóvenes de árboles cultivados en invernadero. Utilice aproximadamente 5 ramas de 5-8 cm de longitud para cada transformación. Asegúrese de utilizar únicamente ramas sanas no infectadas. Retire las hojas cortándolas como tal que queden 1 cm2 de tejido de la hoja al final de cada pecíolo. Deseche las hojas.
- Desinfectar el tejido durante 15 minutos utilizando 1:1 lejía comercial diluida (2% hipoclorito después de la dilución) que contiene unas gotas de polisorbato 20. Luego, enjuague el tejido 6 veces con agua autoclave.
NOTA: Este paso, así como, los siguientes pasos deben llevarse a cabo dentro de un gabinete de flujo descendente laminar para mantener el tejido estéril. - Vuelva a suspender las células A. tumefaciens de 1-2 placas en 25 ml de medio de infiltración (ver Tabla 1) que contiene acetosyringona (20 mg/L) y un tensioactivo no iónico (0,001% v/v) para alcanzar una densidad óptica (OD600)de 5 o5.
NOTA: Preparar la solución de stock de acetosyringona en 70% etanol y almacenar a -20 oC. El tensioactivo no iónico necesita ser esterilizado por filtro antes de añadir al medio de infiltración. - Corta tanto el tallo como el tejido pecíolo en trozos de 1 cm de longitud dentro de la suspensión A. tumefaciens, creando así heridas frescas en ambos lados. Deje las piezas de tejido en la suspensión A. tumefaciens durante 10-30 min.
- Preparar el medio de enraizamiento (ver Tabla 1) y añadir acetosyringona (20 mg/L) después del autoclave. Seque las piezas de tejido sobre una pieza estéril de papel de filtro y colóquela en el medio (10 explantas/placa). Incubar placas en la oscuridad a 21oC durante 2 días.
NOTA: Deje que el medio se enfríe a 60 oC antes de añadir acetosíringona. - Después de 2 días, inspeccione las placas en busca de contaminación fúngica o bacteriana obvia (bacterias distintas de A. tumefaciens). Las placas contaminadas deben desecharse.
- Preparar el medio líquido SH-10 (ver Tabla 1). Después del autoclave, añada polisorbato 20 (0,01%, v/v). Transfiera las piezas de tejido a 10 ml de SH-10 que contengan polisorbato 20. Durante un período de al menos 10 minutos, agitar suavemente cada 2-3 minutos para lavar el tejido.
- Lavar dos veces más con SH-10 fresco que contiene polisorbato 20. Estos tiempos, un tiempo de incubación de 2-3 minutos por paso de lavado es suficiente.
- Prepare el medio de enraizamiento (véase el Cuadro 1). Después del autoclave, añadir cefotaxime (300 mg/L) y kanamicina (50 mg/L) y verter las placas. Para las transformaciones secundarias (transformaciones de líneas transgénicas resistentes a la kanamicina), aplicar la selección de higromicina (15 mg/L).
- Piezas de tejido seco sobre trozos estériles de papel de filtro. Después, transfiera las piezas de tejido a las placas preparadas en el paso 3.9.
- Incubar platos durante 7 días a 28oC, 16 h:8 h día:noche. Cada 2 días comprobar las placas para la contaminación fúngica o bacteriana y el crecimiento excesivo de A. tumefaciens. En caso de contaminación, transfiera piezas no infectadas a un plato nuevo.
- Después de 7 días, transfiera las piezas tisulares al medio de propagación (ver Tabla 1) que contenga cefotaxime (300 mg/L) y kanamicina (50 mg/L). Incubar platos a 28oC, 16 h:8 h día:noche. Refresque las placas una vez a la semana hasta que se desarrollen brotes transgénicos. Asegúrese de transferir únicamente piezas de tejido no infectadas a placas nuevas. Deseche las piezas que están cubiertas por A. tumefaciens.
- Una vez que los brotes putativamente transgénicos miden 1 cm de longitud, cortan los brotes y los cultivan de forma independiente en el medio de propagación que contiene cefotaxime (300 mg/L) y kanamicina (50 mg/L). Para asegurarse de que los brotes representan transformadores independientes, tome un solo brote de cada lado de una explanta.
- Propague vegetativamente brotes putativos transgénicos como se describe en el paso 5.2.
4. Genotipado de brotes putativos transgénicos
- Imprimaciones de diseño que abarcan los sitios de reconocimiento de sgRNA. Para permitir la secuenciación del amplicon de PCR, elija las imprimaciones 150-250 bp lejos de los sitios de reconocimiento sgRNA.
- Corte una punta de hoja (5 mm) de cada brote transgénico para ser genotipado. Además, cosecha una muestra de control de tipo silvestre.
- Realice reacciones de PCR de 50 ml utilizando las imprimaciones diseñadas en el paso 4.1 y un kit comercial para amplificar directamente el ADN a partir de muestras de plantas. Alternativamente, las reacciones de PCR se pueden realizar en ADN purificado utilizando una polimerasa de alta fidelidad.
- PcR separado amplifica en un gel de agarosa 1.5-2%.
- Analizar los resultados de la electroforesis de gel. Compruebe si hay muestras que producen múltiples bandas (más de 1 alelo) y amplicons PCR con tamaños diferentes del tipo salvaje, lo que indica la presencia de indels de tamaño medio.
- La SECUENCIA PCR se amplifica para identificar las mutaciones exactas. Para las muestras que producen un solo amplificador de PCR, los productos PCR se pueden secuenciar directamente. Las muestras que producen más de 1 banda después de la electroforesis de gel o que parecen ser heterocigotos después de la secuenciación directa del amplicon DE PCR, deben clonarse primero en un vector de clonación de extremo contundente. Posteriormente, secuencia varios clones para cada muestra para identificar todos los alelos posibles presentes en la muestra.
- Alinee los resultados de la secuenciación con el gen de interés e inspeccione la alineación para comprobar si hay mutaciones cerca del sitio o sitios objetivo del sgRNA. Posteriormente, compruebe si estas mutaciones crean cambios de trama. Deseche las líneas con > 2 alelos y las líneas que contengan mutaciones en el marco.
- Seleccione varias líneas para su análisis posterior.
- Propague las líneas seleccionadas como se describe en el paso 5.2.
- Cuando las líneas hayan desarrollado varios brotes nuevos, tome nuevas muestras de las puntas de las hojas 3 y repita los pasos 4.3-4.7. Determinar si las mutaciones presentes en cada una de las muestras que se originan en la misma línea, así como la muestra de PCR original son idénticas. Las líneas que producen las mismas mutaciones en todas las muestras se mutan homogéneamente y se pueden utilizar para una mayor experimentación. Descartar líneas que no producen los mismos resultados que estas líneas son quiméricas.
5. Preparación de Plantlets P. andersonii arraigados para la experimentación
-
Iniciar una nueva línea de cultivo de tejidos de P. andersonii.
- Cosecha cogollos axilares, brotes jóvenes aventureros o tejido de hojas de árboles sanos. Alternativamente, las plántulas se pueden utilizar como material de partida.
- Desinfectar el tejido con lejía comercial diluida 1:1 (2% hipoclorito después de la dilución) que contenga unas gotas de polisorbato 20 durante 15 minutos después, enjuague el tejido 6 veces con agua autoclavedor.
NOTA: Este paso, así como, los siguientes pasos deben llevarse a cabo dentro de un flujo descendente laminar o gabinete de flujo cruzado laminar para mantener el tejido estéril. - Transfiera el tejido al medio de propagación (ver Tabla 1). Cierre las placas con 2 capas de lámina de sellado elástica e incubar placas a 28oC, 16 h:8 h día:noche.
- Inspeccione las placas cada pocos días durante las primeras 2 semanas para asegurarse de que el tejido esté libre de contaminación por hongos o bacterias.
- Propague el tejido colocando 10 brotes en una placa nueva de medio de propagación y cierre la placa con 2 capas de lámina de sellado elástica. Incubar platos a 28oC, 16 h:8 h día:noche. Repita este paso cada 4 semanas.
- Cuando los brotes estén >1 cm de longitud, corte los brotes en su base y colóquelos en el medio de enraizamiento (ver Tabla 1). Se pueden colocar alrededor de 10 brotes en una sola placa de enraizamiento. La posición se dispara en posición vertical insertando la punta basal del brote en el medio. Las raíces aparecen a los 10-14 días después de la incubación de las placas a 28oC, 16 h:8 h día:noche.
NOTA: No rootee todos los brotes, sino que mantenga parte para la propagación del cultivo de tejidos (ver paso 5.2).
6. Nodulación de P. andersonii Plantlets en potas
-
Preparar el inóculo de rhizobio.
- Inocular 10 ml de medio Líquido YEM (ver Tabla 2) de una sola colonia de Mesorhizobium plurifarium BOR26 e incubar a 28oC durante 2 días.
NOTA: Se prefiere M. plurifarium BOR2 ya que eficientemente dita P. andersonii. Sin embargo, otras cepas de rizobium también se pueden utilizar para la nodulación de P. andersonii (por ejemplo, Bradyrhizobium elkanii WUR325, Rhizobium tropici CIAT89926,27 o Bradyrhizobium kelud2A4). - Utilice el cultivo de 10 ml para inocular un mayor volumen de medio YEM líquido. El volumen de esta cultura depende del número de macetas que deben ser inoculadas.
- Preparar el medio EKM líquido (ver Tablas 3, Tabla 4). Centrifugar el cultivo bacteriano durante 10 min a 3.500 x g para cosechar las células. Posteriormente, vuelva a suspender el pellet bacteriano en EKM líquido (utilice aproximadamente el mismo volumen que el cultivo Original de YEM) y determine la densidad óptica (OD600).
- Inocular 10 ml de medio Líquido YEM (ver Tabla 2) de una sola colonia de Mesorhizobium plurifarium BOR26 e incubar a 28oC durante 2 días.
- Para 20 macetas, prepare 3 L de medio EKM líquido e inocular con la suspensión rizobiana preparada en el paso 6.1.3. para llegar a OD600 a 0,025.
- Mezclar 3 L de EKM que contiene rizobia con 1.250 g de perlita. Posteriormente, añadir 210 g de esta mezcla a las macetas de polipropileno translúcido estériles. Alternativamente, en lugar de perlita, utilice arena como sustrato para ensayos de nodulación.
- Planta 1-3 p. de p. andersonii en cada maceta. Además, prepare varias macetas que contengan plantlets P. andersonii transformados con la construcción de control CRISPR (ver Tabla Suplementaria 1). Pesar varias macetas para poder determinar la pérdida de agua durante el experimento. Cubra la parte inferior de cada maceta para proteger las raíces de la exposición a la luz.
- Incubar macetas en una sala de crecimiento climatizada (28oC, 16 h:8 h día:noche) durante 4-6 semanas. Una vez a la semana, pesar varias macetas para determinar la pérdida de agua. Si la pérdida de agua supera los 10 ml, complemente con agua ultrapura para compensar la pérdida.
- Después de 4-6 semanas, limpie las raíces de la perlita y determine los números de nódulos utilizando un binocular para examinar la eficiencia de la nodulación.
7. Nodulación de P. andersonii Plantlets en Placas
-
Preparar membranas de celofán 28.
- Corte la membrana de celofán para que quepa en un plato Petri cuadrado de 12 cm x 12 cm. Cortar las membranas un poco más corto en la parte superior para permitir el espacio para que los brotes crezcan.
- Para aumentar la permeabilidad de las membranas de celofán, hervir las membranas en la solución EDTA (1 g/L) durante 20 min. Después, enjuagar al menos 6x con agua desmineralizada para eliminar el EDTA.
NOTA: A medida que la membrana seca tiende a arrugarse cuando está en contacto con el agua, sumerja las membranas secas una por una en la solución. - Coloque las membranas horizontalmente en una fina capa de agua en una placa de vidrio redonda. Esterilice las membranas autoclavedos dos veces.
- Coloque 1 membrana de celofán autoclave en una placa Petri cuadrada de 12 x 12 cm que contenga medio EKM solidificado con agar (ver Tabla 3, Tabla 4). Coloque dos plantas P. andersonii de 3 semanas de edad (ver sección 5) o plántulas de 4 semanas (ver sección 1.1) en la parte superior de la membrana. Asegúrese de recoger sólo plantitas o plántulas con raíces que tengan puntas de raíz blanca, lo que indica que estas raíces todavía están creciendo.
- Cubre suavemente las raíces con una segunda membrana de celofán, creando una capa sándwich. Sellar la placa con 3 capas de lámina de sellado elástica. Envuelva la mitad inferior de las placas con papel de aluminio, para cubrir las raíces de la exposición a la luz.
- Incubar las placas en una sala de crecimiento climatizada (28oC, 16 h:8 h día:noche) durante 3-4 semanas. Marque la posición de los consejos de raíz para seguir el crecimiento de la raíz a lo largo del tiempo.
- Si las placas EKM comienzan a secarse debido a la incubación prolongada, transfiera las plantas a placas EKM frescas unos días antes de la inoculación bacteriana.
- Preparar el inóculo bacteriano como se describe en el paso 6.1.
- Retirar la membrana superior del celofán y aplicar 1 ml de cultivo de rizobio (OD600 a 0,025) a las raíces. Posteriormente, coloque una nueva membrana de celofán en las raíces inoculadas. Envuelva el exterior de la placa usando papel de aluminio para cubrir las raíces de la exposición a la luz.
- Después de 4 semanas, examine los números de nódulos utilizando un binocular para determinar la eficiencia de la nodulación.
8. Nodulación de las seedlings de P. andersonii en bolsas
- Germinar las semillas de P. andersonii como se describe en la sección 1.1. Después de que los cotiledons hayan surgido completamente (12 días en placas SH-0 a 28 oC), transfiera las plántulas a las bolsas.
- Para preparar las bolsas, rasgue la sección doblada de la mecha de papel y añada 7 ml del medio EKM modificado (véase el Cuadro 3, Tabla 4).
- Inserte 1 o 2 plántulas colocando las raíces entre ambas hojas de papel que forman la mecha de papel y la hoja de plástico frontal de la bolsa.
- Proteger las raíces de la exposición a la luz, mediante el plegado de papel de aluminio alrededor de la bolsa. Suspenda las bolsas en una caja de plástico cubierta con una tapa translúcida para mantener una alta humedad. Coloque la caja en una sala de crecimiento climatizada (28oC, 16 h:8 h día:noche).
- Compense la evaporación del agua añadiendo agua estéril ultrapura, de tal manera que la mecha de papel permanezca húmeda (evitar el agua estancada en la parte inferior de la bolsa). Después de la primera semana, Esto generalmente requiere añadir 2-3 ml cada 4 días.
- Preparar el inóculo bacteriano como se describe en el paso 6.1.
- Después de que las plántulas hayan sido cultivadas durante 10-12 días en bolsas, inocular el sistema radicular con 500 l de cultivo de rizobio (OD600 a 0,025).
- Siga la formación del nódulo a través del tiempo. Cuatro semanas después de la inoculación, los nódulos se pueden contar y cosechar para determinar la eficiencia de la nodulación.
9. Análisis de citoarquitectura de nódulo
- Recoger 10-15 nódulos en un tubo de 2 ml que contenga fijante (5% glutaraldehído en tampón de fosfato de 0,1 M, pH 7,2). Aplicar al vacío durante 1/2-1 h e incubar durante la noche a 4oC. Durante este período, las muestras se hunden en la parte inferior del tubo.
NOTA: La solución fijativa se puede almacenar a 4 oC durante 2-4 semanas de uso anterior. Asegúrese de usar guantes cuando trabaje con un fijador de tejido. - Lavar los nódulos 2x con tampón de fosfato de 0,1 M, pH 7,2. Aplicar intervalos de 10 minutos entre cada paso de lavado.
- Deshidratar las muestras incubando posteriormente en 30%, 50%, 70% y 100% etanol. Para asegurarse de que se elimina toda el agua de las muestras, repita el paso de etanol 100% 3x. Aplique intervalos de 10 minutos entre cada paso de deshidratación.
- Preparar la mezcla de polimerización I (PM-I) añadiendo 1 envase de Hardener I a 2,5 ml de PEG400 mezclado con 100 ml de hema (2-hidroxietilo metacrilato) solución de resina a base. Revuelva la solución durante 15 minutos para disolver completamente el endurector I. Posteriormente, almacene PM-I a -20 oC.
- Retire el etanol del paso 9.3. e infiltrar las muestras en el siguiente orden: etanol PM-I:100% (1:3, v/v), etanol PM-I:100% (1:1, v/v) y etanol PM-I:100% (3:1, v/v). Incubar las muestras en cada solución en RT durante 1/2-1 h o hasta que las muestras se hundan hasta el fondo.
- Incubar muestras durante la noche a 4oC en solución 100% PM-I.
- Preparar la mezcla de polimerización II mezclando PM-I y Hardener II en una relación de 15:1 (v/v). Llene el molde de plástico con la solución de polimerización, oriente las muestras horizontalmente en la parte inferior del molde y cubra con un pedazo de lámina de sellado elástica. Evitar la formación de burbujas de aire.
NOTA: A medida que la solución comienza a polimerizarse al exponerse a RT, trate de orientar las muestras lo más rápido posible en el soporte de plástico. La polimerización se completa después de la incubación durante la noche a RT, o 1 h a 37oC. - Retire la cubierta de la lámina de sellado elástica del paso 9.7 y coloque un soporte en las muestras polimerizadas. Para montar el soporte en las muestras, disolver 10 ml de polvo de resina a base de metacrilato de metilo en 5 ml de solución de resina a base de metacrilato de metilo. Agregue rápidamente la solución al orificio en la parte superior del soporte.
NOTA: Realice el paso de polimerización en la campana de humos (30 min en RT). - Muestras de sección de microtome a un espesor de 4-5 m. Coloque un portaobjetos de microscopio en una placa caliente de 58 oC y agregue una gran gota de agua a cada diapositiva. Coloque las secciones en la parte superior del agua. Una vez que el agua se ha evaporado, las secciones se adhieren al tobogán.
- Se desliza por la mancha sumergiendo en 0.05 % (w/v) azul toluidina durante 2 min. Posteriormente, enjuague los portaobjetos 3x con agua ultrapura. Las diapositivas se pueden observar con un microscopio de campo brillante.
10. Micorrización de P. andersonii Plantlets
-
Preparar el inóculo de las esporas de Rhizophagus irregularis
- Preparar una pila de filtros tejidos de poliéster con los siguientes tamaños (de arriba a abajo): 210 m, 120 m y 36 m de tamaño de malla.
- Pipetear la cantidad necesaria de una suspensión comercial de esporas en la pila de filtros de poliéster. Enjuagar los filtros 3x con 100 ml de agua autoclaveizada desmineralizada. Las esporas se conservan en la superficie del filtro de 36 m.
NOTA: Prepare la suspensión de esporas en el gabinete de flujo cruzado laminar para evitar la contaminación. - Desmonte la pila de poliéster y mantenga el filtro de 36 m solamente. Repita el paso de lavado con agua autoclaveizada desmineralizada durante al menos 6x.
- Coloque el filtro en una placa Petri y vuelva a suspender las esporas en agua autoclaveizada. Utilice un volumen de agua igual al volumen de la suspensión de esporas utilizada en el paso 10.1.2. Transfiera la suspensión de esporas a un tubo estéril pipeteando.
- Coloque 5 gotas de 20 l de la suspensión de esporas en un portaobjetos de vidrio y cuente el número de esporas utilizando un microscopio de campo brillante. Convierte los recuentos de esporas en una proporción de esporas/ml y diluye la suspensión de esporas hasta que alcance las 250 esporas/ml. Conservar la suspensión de las esporas a 4oC.
- Realizar el ensayo de micorrización. Para ello, añada 800 g de arena autoclave suplementada con 70 ml de ollas de polipropileno translúcido de medio a medio de Hoagland de 1 2 a estéril (ver Tablas 5-6). Mezclar la arena y el medio directamente en la olla agitando vigorosamente.
- Coloque una planta de P. andersonii en cada maceta, y pipeta 1 ml de la suspensión de esporas directamente sobre la raíz de la planta P. andersonii. Asegúrese de incluir varias macetas que contengan plantlets P. andersonii transformados con una construcción de control CRISPR (ver Tabla Suplementaria 1).
- Incubar macetas en una sala de crecimiento climatizada (28oC, 16 h:8 h día:noche) durante 6 semanas.
- Saca las plantas de las macetas y lava las raíces con agua corriente para eliminar la mayor cantidad de arena posible.
- Cortar las raíces en trozos largos de 1 cm y hervir las piezas de raíz en 10% KOH (p/v) durante 20 min a 90 oC. Posteriormente, coloque las raíces hervidas en un colador de células con un tamaño de malla de 100 m y enjuague 3 veces con 50 ml de agua.
- Raíces de mancha con 0,05% (p/v) trippan azul en lactoglicerol (300 ml de ácido láctico; 300 ml de glicerol; y 400 ml de agua desmineralizada) durante 5 min a 90 oC en un baño de agua o bloque de calentamiento. Posteriormente, transferir raíces a 30% glicerol. Las muestras raíz se pueden almacenar en RT.
- Coloque 15-25 fragmentos de raíz en una sola diapositiva de microscopio. Añadir 30% de glicerol y cubrir con un vidrio de cubierta y presionar hasta que las piezas de la raíz se vuelvan planas. Observe los fragmentos de raíz con un microscopio de campo brillante y puntúe la colonización micorriza.
NOTA: Se describe un método para puntuar la micorrización de acuerdo con Trouvelot et al.29. Este método utiliza varias clases (%F, %M y %A), lo que permite una estimación rápida del nivel de colonización micorriza de cada fragmento de raíz y la abundancia de arbuscules.
Representative Results
P. andersonii trees se puede cultivar en un invernadero acondicionado a 28 oC y 85% de humedad relativa (Figura1A). Bajo estas condiciones, los árboles comienzan a florecer a los 6-9 meses después de la siembra. Las flores femeninas de P. andersonii producen bayas que cada una contiene una sola semilla. Durante la maduración, las bayas cambian de color; primero de verde a blanco y posteriormente de blanco a marrón (Figura1B). Las semillas extraídas de las bayas marrones maduras, germinan bien después de un ciclo de temperatura de 10 días y una incubación de 7 días en placas SH-0 (Figura1C). Las semillas germinadas continúan desarrollándose en plántulas jóvenes que se pueden utilizar para la experimentación después de 4 semanas (Figura1D).
Hemos demostrado previamente que los pecíolos y segmentos de tallos jóvenes de P. andersonii se pueden transformar eficientemente utilizando la cepa A. tumefaciens AGL110. Al inicio del procedimiento de transformación, los explantes de tejido se cultivan co-cultivo con A. tumefaciens durante 2 días a 21 oC (Figura2A). El co-cultivo prolongado da lugar a la colonización excesiva de los explantes tisulares por A. tumefaciens y, por lo tanto, debe prevenirse (Figura2B). Después del período de co-cultivo, los explantes de tejido se transfieren a medios selectivos, lo que promueve el crecimiento del tejido transformado. Dos o tres semanas más tarde, se observan generalmente pequeños microcali verdes a lo largo de la superficie original de la herida (Figura2C). Estos calli deben seguir creciendo y desarrollar 1 o más brotes transformados putativamente a las 6-8 semanas después de que se haya iniciado el procedimiento de transformación (Figura2D). En esta etapa, las eficiencias de transformación suelen oscilar entre el 10 y el 30 % para las transformaciones iniciadas con explantes de tejido tomados de ramas maduras y parcialmente leñosas (Tabla7). Si se inician transformaciones con explantes tomados de las puntas jóvenes y de rápido crecimiento de las ramas que aún no llevan flores, se pueden lograr eficiencias de transformación del 65-75% (Tabla7). Ocasionalmente, los calli blanquecinos se forman en el lado de una explanta que no está en contacto con el medio y, por lo tanto, no experimentan la selección de kanamicina. Estos calli a menudo no son transgénicos y cualquier brote formado a partir de estos calli generalmente se blanqueará y morirá después del contacto directo con el medio que contiene kanamicina (Figura2E). En caso de que la tasa de transformación sea baja y/o el material de partida fuera subóptimo, las piezas de tejido podrían volverse marrones (Figura2F)y sufrir de sobreproliferación por Parte De A. tumefaciens (Figura 2G). Para evitar que A. tumefaciens se propaguen y sobrecrezcan los explantes cercanos, se requiere un refrigerio regular del medio, y es necesario eliminar los explantes gravemente infectados. Una vez que los brotes transgénicos individuales se colocan en el medio de propagación, la sobreproliferación por A. tumefaciens generalmente ya no ocurre (Figura2H). Los brotes transgénicos se pueden multiplicar a través de la propagación in vitro, lo que dará lugar a decenas de brotes en un período de un mes (Figura3A-B). Estos brotes se pueden colocar en el medio de enraizamiento, que debe inducir la formación de la raíz después de 2 semanas (Figura3C-D). Las plantitas enraizadas se pueden utilizar posteriormente para la experimentación.
Para crear líneas mutantes noqueadas, hacemos uso de mutagénesis mediada por CRISPR/Cas9. Con este fin, hacemos uso de un vector binario que contiene el gen de resistencia a la kanamicina NPTII, una secuencia de codificación Cas9 impulsada por el promotor CaMV35S y 2 sgRNAs por gen objetivo que se expresan desde el promotor de ARN pequeño AtU6p20. En la Figura 4Ase proporciona una representación gráfica de la construcción utilizada para la mutagénesis mediada por CRISPR/Cas9 de P. andersonii. Usando este método, la edición del genoma se observa en el 40% de los brotes transformados putativamente10. Para identificar las líneas mutantes, los brotes transformados putativamente se genotipan para mutaciones en los sitios objetivo del sgRNA utilizando imprimaciones que abarcan la región objetivo. En la Figura 4se muestra un ejemplo de los resultados esperados. Como se puede ver en la foto tomada después de la electroforesis de gel, varias muestras producen un amplicon PCR con un tamaño similar al tipo salvaje (Figura4B). Estas plantas pueden contener pequeños indels que no pueden ser visualizados por electroforesis de gel de agarosa o permanecer sin editar por la enzima Cas9. Además, varias muestras producen bandas que son diferentes en tamaño del tipo salvaje (por ejemplo, las líneas 2, 4, 7 y 8 en la Figura 4B). En estas líneas, 1 (líneas 4, 7 y 8) o ambos (línea 2) alelos contienen indels más grandes que se pueden visualizar fácilmente. La naturaleza exacta de las mutaciones en el sitio o sitios de destino se revela después de la secuenciación del amplicon de PCR. Como se puede ver en la Figura 4C,tanto los pequeños indels de 1-4 bp, como, eliminaciones más grandes se pueden obtener después de la mutagénesis CRISPR/Cas9. En la Figura 4C, la secuencia de la línea 1 es idéntica a la del tipo comodín, lo que indica que esta línea tiene escape de edición y, por lo tanto, debe descartarse. Entre las líneas que contienen mutaciones, se pueden identificar mutantes heterocigotos, homocigotos y bialélicos (Figura4C). Sin embargo, los mutantes heterocigotos son generalmente raros10. Los mutantes noqueadores homocigotos o bialélicos se pueden propagar vegetativamente para obtener material suficiente para el análisis fenotípico. Como el análisis fenotípico se realiza en la generación T 0, es importante comprobar si las líneas mutantes pueden ser quiméricas. Para ello, el genotipado debe repetirse en al menos 3 muestras diferentes tomadas de cada línea mutante. Si los resultados del genotipado son idénticos entre sí y la muestra de genotipado original (por ejemplo, la línea 8 de la Figura 4D),la línea se muta homogéneamente y se puede utilizar para un análisis posterior. Sin embargo, si los resultados del genotipado difieren entre muestras independientes (por ejemplo, la línea 4 en la Figura 4D),la línea mutante es quimérica y debe descartarse.
La inoculación de P. andersonii con M. plurifarium BOR2 da como resultado la formación de nódulos radiculares (Figura 5). Como se puede ver en la Figura 5A,estos nódulos se distribuyen a lo largo del sistema raíz. Los nódulos de P. andersonii son de color marrón claro, pero pueden ser fácilmente discriminados del tejido radicular basado en su forma (Figura5B). Los experimentos de inoculación en macetas y el crecimiento posterior durante 4-6 semanas suelen dar lugar a la formación de nódulos de 10 a 30 nódulos (Figura6A). Un número similar de nódulos se forma después de la inoculación de las plantas P. andersonii cultivadas en placas EKM a las 4 semanas después de la inoculación (Figura6A). En las bolsas, las plántulas de P. andersonii suelen formar nódulos de 5 a 15 euros a las 5 semanas posteriores a la inoculación (Figura5C-D, 6A). Para analizar la citoarquitectura nódula, los nódulos se pueden seccionar y observar mediante microscopía de campo brillante. La Figura 6B muestra un ejemplo de una sección longitudinal a través de la mitad de un nódulo P. andersonii. Esta sección muestra el haz vascular central de un nódulo P. andersonii, que está flanqueado por lóbulos nódulos que contienen células infectadas (Figura6B).
Las plantitas P. andersonii también se pueden micorrimar. Después de 6 semanas de inoculación con R. irregularis, la frecuencia de colonización micorriza suele alcanzar > 80% (Figura6C). En este momento, generalmente el 30 % de las celdas contienen arbuscules (Figura6C). En la Figura 6Dse muestra una imagen representativa de un segmento de raíz de P. andersonii que contiene arbuscles.

Figura 1: Imágenes representativas de un P. andersonii árbol, semillas y plántulas. (A) Árbol P. andersonii de seis meses de edad cultivado en tierra maceta en un invernadero acondicionado a 28 oC. (B) Imagen representativa que representa las bayas de P. andersonii en varias etapas de maduración. Las bayas jóvenes de P. andersonii (sin madurar) cambiarán de color de verde a blanco y finalmente a marrón (madura) al madurar. (C) Semillas de P. andersonii incubadas en medio SH-0 durante 1 semana. Un círculo negro indica una plántula germinada. (D) Plántulas P. andersonii de cuatro semanas cultivadas en medio SH-0. Las barras de escala son iguales a 25 cm de (A) y 1 cm en (B-D). Haga clic aquí para ver una versión más grande de esta figura.
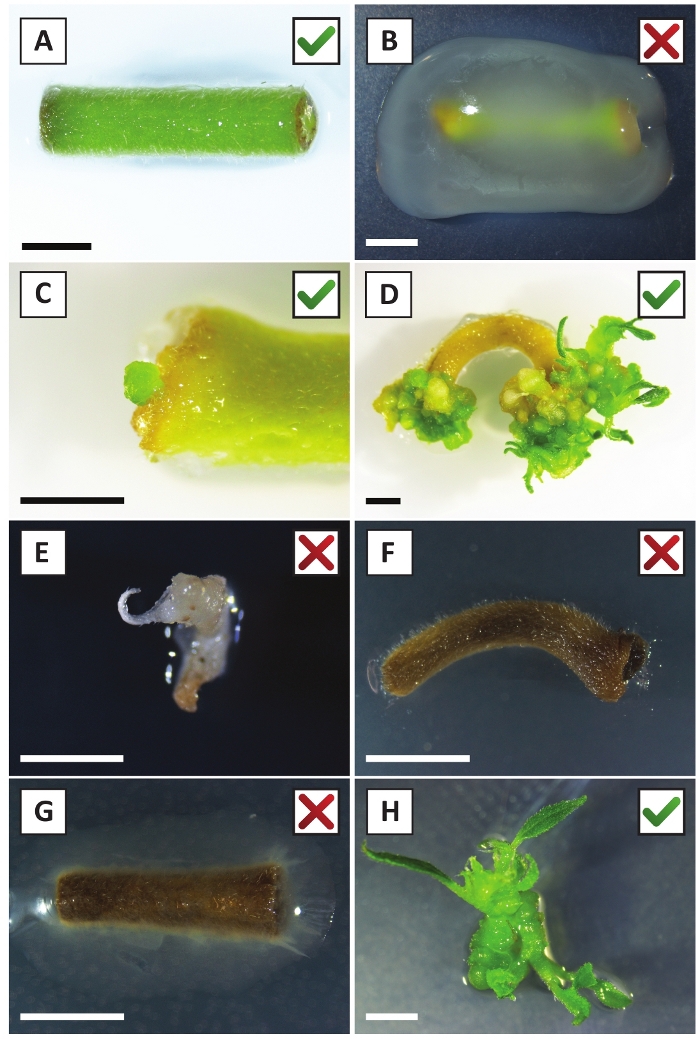
Figura 2: Imágenes representativas de explantes en diferentes etapas del procedimiento de transformación estable. (A) Explantación cocultivada con A. tumefaciens. (B) Explantar sobreplantado por A. tumefaciens durante las primeras 2 semanas después de la transformación. (C) Micro-calilo transgénico formado cerca del lugar de la herida de un explanta a las 2,5 semanas después del co-cultivo. (D) Imagen representativa de un explantar a las 6 semanas posteriores al cocultivo que muestra la aparición de brotes de calli (transgénicos). (E) Imagen representativa de un brote que se vuelve blanquecino y eventualmente muere cuando está en contacto directo con el medio que contiene kanamicina. Este brote es probablemente no transgénico y escapó de la selección de kanamicina cuando se une a la explantación. (F) Imagen representativa de una explanta transformada sin éxito. (G) Imagen representativa de un explant transformado sin éxito cubierto por A. tumefaciens. (H) Brote transgénico único cultivado en medio de propagación a las 8 semanas posteriores al cocultivo con A. tumefaciens. Las barras de escala iguales a 2,5 mm. Las cajas que contienen marcas de verificación verdes o cruces rojas indican una transformación correcta o incorrecta de explantas, respectivamente. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Imágenes representativas de in vitro propagación. (A) Brotes cultivados en medio de propagación. La imagen fue tomada 1 semana después de que las placas se refrescaron. (B) Brotes cultivados en el medio de propagación. La imagen fue tomada 4 semanas después de que las placas se refrescaran. (C) Brotes recién cortados colocados en el medio de enraizamiento. (D) Brotes incubados en el medio de enraizamiento durante 2 semanas. Tenga en cuenta la presencia de raíces. Las barras de escala son iguales a 2,5 cm. Haga clic aquí para ver una versión más grande de esta figura.
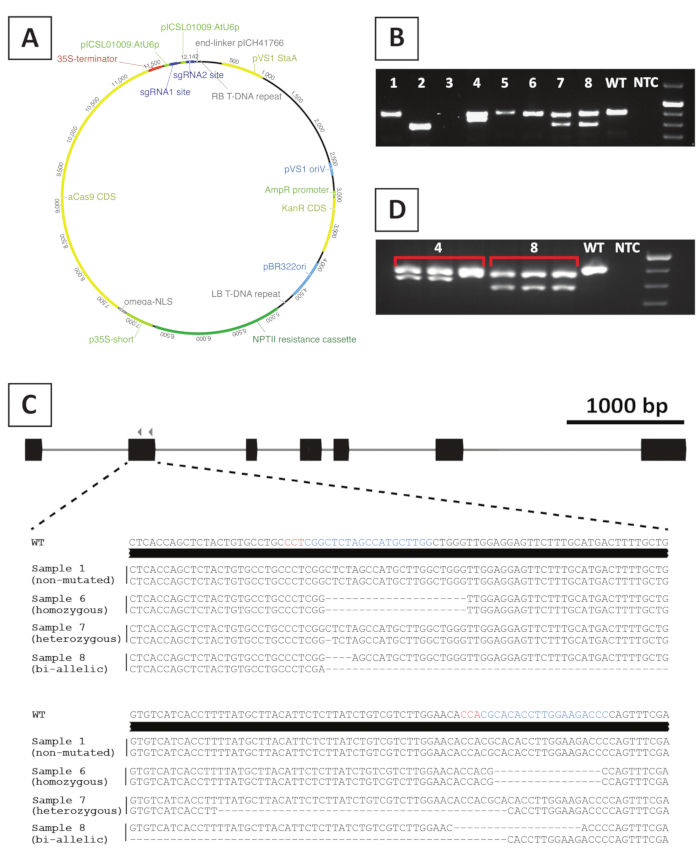
Figura 4: Resultados representativos después del genotipado de las líneas mutantes transgénicas CRISPR/Cas9 transgénicas de P. andersonii T 0. (A) Mapa representativo de un vector binario utilizado para la mutagénesis mediada por CRISPR/Cas9 de P. andersonii. (B) Resultado representativo después del genotipado basado en PCR de posibles líneas mutantes CRISPR/Cas9 utilizando imprimaciones que abarcan los sitios de destino del sgRNA. Se muestra una imagen después de la electroforesis de gel de agarosa de los amplicons. Las muestras tomadas de líneas transgénicas individuales se indican por números. El tipo comodín (WT) y ningún control de plantilla (NTC) indican carriles que contienen controles positivos y negativos, respectivamente. (C) Representación esquemática de alelos mutantes obtenidos después de la edición de genes mediada por CRISPR/Cas9. En colores azul y rojo se destacan los sitios de destino sgRNA y las secuencias de PAM, respectivamente. (D) Resultado representativo después de la detección basada en PCR para posibles líneas mutantes quiméricas. Se muestra una imagen después de la electroforesis de gel de agarosa de 3 muestras individuales tomadas de las líneas mutantes 4 y 8. Tenga en cuenta que la línea mutante transgénica 4 es quimérica. Haga clic aquí para ver una versión más grande de esta figura.
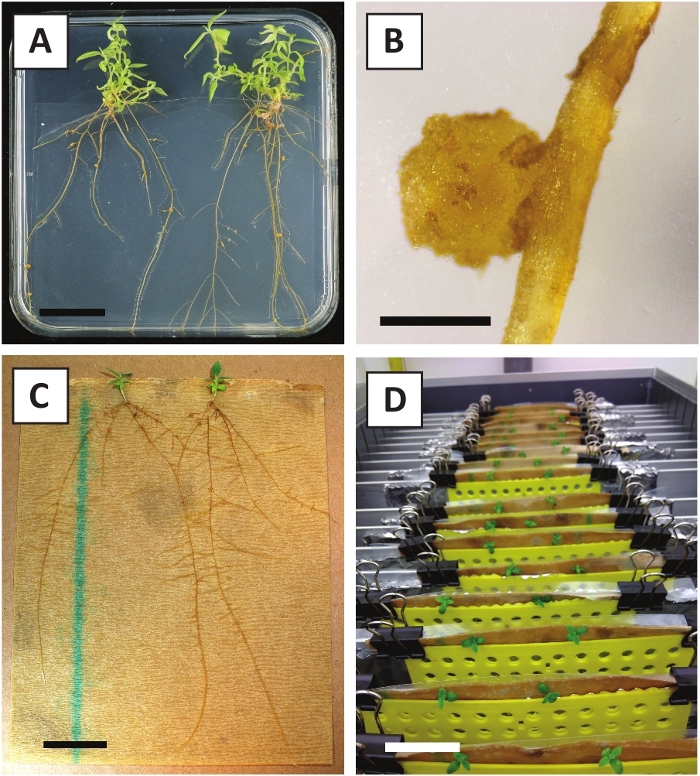
Figura 5: Imágenes representativas de ensayos de nodulación en placas y bolsas. (A) Nodulación en placas que contengan medio EKM solidificado con agar e inoculados con M. plurifarium BOR2 durante 4 semanas. (B) Imagen representativa de un nódulo de raíz de P. andersonii. La imagen fue tomada a las 4 semanas después de la inoculación con M. plurifarium BOR2. (C) Nodulación en bolsas que contengan medio EKM líquido. Las plántulas fueron inoculadas con Bradyrhizobium sp. Kelud2A4 durante 5 semanas. (D) Imagen representativa de una configuración completa utilizada para la nodulación en las bolsas. Las barras de escala son iguales a 2,5 cm de (A,C), 1 mm en (B) y 5 cm en (D). Haga clic aquí para ver una versión más grande de esta figura.
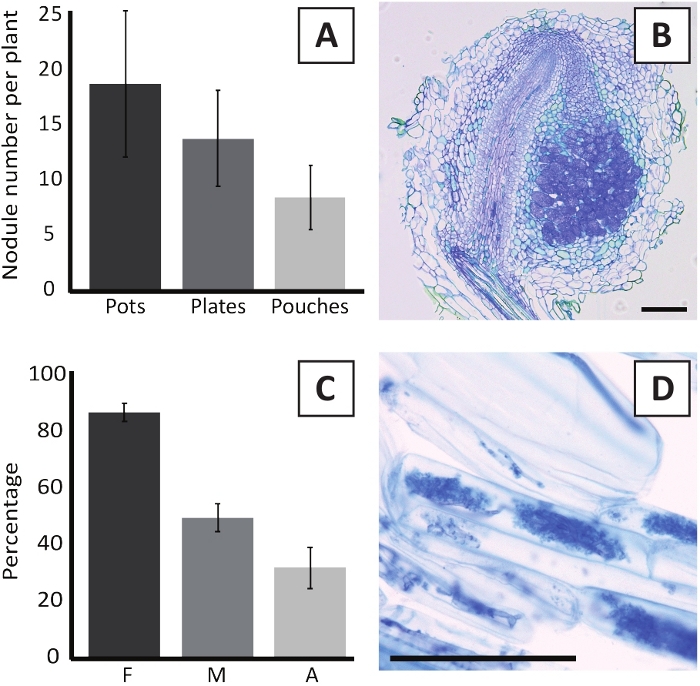
Figura 6: Resultados representativos de los ensayos de nodulación y micorritización. (A) Gráfico de barras representativas que muestra el número de nódulos formados por planta a las 4 semanas posteriores a la inoculación con M. plurifarium BOR2 en macetas o en placas y a las 5 semanas después de la inoculación con Bradyrhizobium sp. Kelud2A4 en bolsas. Los datos representan la media de SD (n a 10). (B) Imagen representativa de una sección longitudinal a través de un nódulo formado a las 4 semanas posteriores a la inoculación con M. plurifarium BOR2. La sección está teñida de azul toluidina. (C) Gráfico de barras representativas que muestra la cuantificación de la micorrización. Las variables cuantificadas según Trouvelot et al.29 son F, la frecuencia de los fragmentos de raíz analizados que son micorrizados; M, la intensidad de la infección; A, la abundancia de arbuscules maduros en el sistema radicular total. La micorrización se cuantificó a las 6 semanas posteriores a la inoculación con R. irregularis (strain DAOM197198). Los datos representan la media de SD (n a 10). (D) Imagen representativa de los arbuscules maduros presentes en las células corticales de raíz de P. andersonii a las 6 semanas posteriores a la inoculación con R. irregularis. Barras de escala iguales a 75 m. Por favor, haga clic aquí para ver una versión más grande de esta figura.
| Compuesto | SH-0 | SH-10 | Medio de propagación | Medio de enraizamiento | Medio de infiltración |
| Medio sal-basal SH | 3,2 g | 3,2 g | 3,2 g | 3,2 g | 3,2 g |
| Mezcla sH-vitamina | 1 g | 1 g | 1 g | 1 g | 1 g |
| Sacarosa | - | 10 g | 20 g | 10 g | 10 g |
| BAP (1 mg/ml) | - | - | 1 mL (4,44 m) | - | - |
| IBA (1 mg/ml) | - | - | 100 l (0,49 m) | 1 mL (4,92 m) | - |
| NAA (1 mg/ml) | - | - | - | 100 l (0,54 m) | - |
| 1 M MES pH 5.8 | 3 ml | 3 ml | 3 ml | 3 ml | 3 ml |
| 1 M KOH | Ajuste el pH a 5,8 | Ajuste el pH a 5,8 | Ajuste el pH a 5,8 | Ajuste el pH a 5,8 | Ajuste el pH a 5,8 |
| Agar Daishin | 8 g | - | 8 g | 8 g | - |
Tabla 1: Composición de30 medios basados en Schenk-Hildebrandt utilizados para el cultivo de plántulas P. andersonii, transformación estable y propagación in vitro. Disuelva los compuestos sólidos en 750 ml de agua ultrapura antes de añadir existencias líquidas. Después, llene el medio completo a 1 L. Preparar las existencias de BAP, IBA, NAA en 0,1 M KOH y almacenar a -20 oC.
| Antes de autoclave: | ||
| Compuesto | Cantidad por litro | Concentración final |
| Manitol | 5 g | 27,45 mM |
| Na-Gluconate | 5 g | 22,92 mM |
| Extracto de levadura | 0,5 g | - |
| MgSO4x 7H2O | 0,2 g | 0,81 mM |
| Nacl | 0,1 g | 1,71 mM |
| K2HPO4 | 0,5 g | 2,87 mM |
| Después del autoclave: | ||
| Compuesto | Cantidad por litro | Concentración final |
| 1.5 M CaCl2 | 1 mL | 1,5 mM |
Tabla 2: Composición del medio de levadura-Mannitol (YEM) utilizado para el cultivo de rizobium. Ajuste el pH a 7.0 y llene con agua ultrapura a 1 L. Para preparar el medio YEM solidificado en agar, añada 15 g de microagar antes de autoclave.
| Antes de autoclave: | |||
| Compuesto | Concentración de stock | Cantidad por litro medio | Concentración final |
| KH2PO4 | 0.44 M | Añadir 2 mL | 0,88 mM |
| K2HPO4 | 1.03 M | Añadir 2 mL | 2,07 mM |
| Solución en stock de microelementos 500x | - | Añadir 2 mL | - |
| ETM pH 6,6 | 1 M | Añadir 3 mL | 3 mM |
| Hcl | 1 M | Ajuste el pH a 6,6 | - |
| Agua ultrapura | - | Llenar hasta 990 mL | - |
| Después del autoclave: | |||
| Compuesto | Concentración de stock | Cantidad por litro medio | Concentración final |
| MgSO4x 7H2O | 1.04 M | 2 ml | 2,08 mM |
| Na2SO4 | 0,35 M | 2 ml | 0,70 mM |
| NH4NO3 | 0,18 M | 2 ml | 0,36 mM |
| CaCl2x 2H2O | 0,75 M | 2 ml | 1,5 mM |
| Fe(III)-citrato | 27 mM | 2 ml | 54 M |
Tabla 3: Composición de 1 L modificado EKM medio31 utilizado para el ensayo de nodulación P. andersonii. La composición de la solución de material de microelementos 500x se enumera en la Tabla 4. Para preparar un medio EKM solidificado al agar al 2%, añade 20 g de agar Daishin antes de autoclave. Autoclave el MgSO47H2O, Na2SO4, CaCl2s 2H2O, y Fe(III)-citrato stocks para esterilizar. Esterilizar el filtro NH4NO3 solución en stock para esterilizar.
| Compuesto | Cantidad por litro | Concentración de stock |
| MnSO4 | 500 mg | 3,31 mM |
| ZnSO4x 7H2O | 125 mg | 0,43 mM |
| CuSO4x 5H2O | 125 mg | 0,83 mM |
| H3BO3 | 125 mg | 2,02 mM |
| Na2MoO4x 2H2O | 50 mg | 0,21 mM |
Tabla 4: Composición de la solución de material de microelementos 500x utilizada para preparar medios EKM modificados. Almacene la solución de material de microelementos a 4 oC.
| Compuestos | Concentración de stock | Cantidad por litro medio | Concentración final |
| K2HPO4 | 20 mM | 1 mL | 0,2 mM |
| NH4NO3 | 0,28 M | 10 mL | 2,8 mM |
| MgSO4 | 40 mM | 10 mL | 0,4 mM |
| K2SO4 | 40 mM | 10 mL | 0,4 mM |
| Fe(II)-EDTA | 9 mM | 10 mL | 0,9 mM |
| CaCl2 | 80 mM | 10 mL | 0,8 mM |
| Solución en stock de 50x microelementos | - | 10 mL | - |
Tabla 5: Composición del medio 1/2-Hoagland32 utilizado para ensayos de micorriza. La composición de la solución de material de microelementos 50x se enumera en la Tabla 6. Preparar la solución Fe(II)-EDTA combinando FeSO4x 7H2O (9 mM) y Na2 EDTA (9 mM) en 1 solución de stock y almacenar a 4 oC. Ajuste el pH del medio a 6.1 usando 1 M KOH y llene con agua ultrapura a 1 L.
| Compuestos | Cantidad por litro | Concentración de stock |
| H3BO3 | 71,1 mg | 1,15 mM |
| MnCl2s4H2O | 44,5 mg | 0,22 mM |
| CuSO4x 5H2O | 3,7 mg | 23,18 m |
| ZnCl2 | 10,2 mg | 74,84 m |
| Na2MoO4x 2H2O | 1,2 mg | 4,96 m |
Tabla 6: Composición de la solución de material de microelementos 50x utilizada para la preparación de medio de 1/2-Hoagland.
| Edad de los explantes | Eficiencia de transformación |
| Joven | 69,4 á 6,2 % (n x 2) |
| Maduro | 18,3 á 10,2% (n a 15) |
Tabla 7: Eficiencia de transformación de P. andersonii. Aquí, la eficiencia de transformación se define como el porcentaje de explantas que forman al menos 1 callos o brote transgénico. La eficiencia de transformación se puntuó a las 6 semanas posteriores a la transformación y se representa como media : SD. n indica el número de experimentos de transformación a partir de los cuales se determinó la eficiencia de transformación.
Archivo suplementario 1: Descripción general de las construcciones de nivel 1 y nivel 2 utilizadas para la mutagénesis CRISPR/Cas9. Haga clic aquí para descargar este archivo.
Discussion
Las legumbres y el género Parasponia de Cannabaceae, distantemente relacionado, representan los dos únicos clados de especies vegetales capaces de establecer una relación endosimbiótica con la rizobia fijadora de nitrógeno y formar nódulos radiculares. Los estudios comparativos entre especies de ambos clados son muy relevantes para proporcionar información sobre las redes genéticas centrales que permiten esta simbiosis. Actualmente, los estudios genéticos se realizan principalmente en legumbres; especialmente las dos especies modelo M. truncatula y L. japonicus. Para proporcionar una plataforma experimental adicional y facilitar los estudios comparativos con un nódulo no leguminoso, describimos aquí un protocolo detallado para la transformación estable y análisis genéticos inversos en P. andersonii. El protocolo presentado utiliza la propagación in vitro de las líneas transgénicas T0 P. andersonii, permitiendo que el análisis fenotípico se inicie dentro de los 4 meses después del co-cultivo de A. tumefaciens. Esto es sustancialmente más rápido que los protocolos actuales que se han establecido para la transformación estable de las legumbres33. Esto hace de P. andersonii un modelo de investigación atractivo.
El protocolo descrito aquí contiene varios pasos críticos. El primero de los cuales se refiere a la germinación de semillas. Para preparar las semillas de P. andersonii para la germinación, las semillas necesitan ser aisladas de las bayas. Esto se hace frotando las bayas en un pedazo de papel tisú o contra el interior de un tamiz de té. Este procedimiento debe realizarse suavemente para evitar daños en la capa de semillas. Si la capa de semilla se daña, la lejía podría entrar en la semilla durante la esterilización, lo que reduce la viabilidad de las semillas. Para romper la latencia de las semillas, las semillas se someten a un ciclo de temperatura de 10 días. Sin embargo, a pesar de este tratamiento, la germinación no está totalmente sincronizada. Generalmente, las primeras semillas muestran la aparición del radio después de 7 días, pero otras pueden tardar varios días más en germinar.
Los puntos críticos del procedimiento de transformación se refieren a la elección del material de partida y a la duración de la etapa de co-cultivo. Para alcanzar una transformación eficiente, lo mejor es utilizar tallos o pecíolos sanos y jóvenes de plantas cultivadas en invernadero no estériles como material de partida. Con el fin de inducir el crecimiento de ramas jóvenes, es aconsejable recortar árboles de Parasponia cada 2-3 meses y refrescar los árboles una vez al año. Además, el paso de co-cultivo debe realizarse solo durante 2 días. El co-cultivo prolongado promueve la colonización excesiva de explantes de tejido por A. tumefaciens y generalmente reduce la eficiencia de transformación. Para evitar la colonización excesiva por A. tumefaciens también es importante refrescar regularmente las placas en las que se cultivan los explantas. En caso de que se produzca una colonización excesiva, los explantes de tejido podrían lavarse (ver Sección 3.8) para eliminar las células A. tumefaciens. Aconsejamos añadir lejía a la solución SH-10 utilizada para el lavado (concentración final: 2% hipoclorito). Es importante tener en cuenta que este paso de lavado adicional podría no funcionar en explantes muy infectados (Figura2B). En caso de que una transformación con una construcción CRISPR/Cas9 produzca sólo un número limitado de brotes transformados putativamente o si se espera que la mutagénesis de un gen en particular cause problemas en la regeneración, es aconsejable incluir una construcción de control vectorial vacía como el control positivo. Por último, es importante asegurarse de que todas las líneas transgénicas seleccionadas sean resultantes de eventos independientes de integración de T-DNA. Por lo tanto, instruimos a tomar sólo un brote putativo-transgénico de cada lado de una explanta. Sin embargo, nos damos cuenta de que esto reduce el número potencial de líneas independientes. Si se requieren muchas líneas, los investigadores podrían decidir separar el calli transformado putativamente de los explantes originales cuando estos calli son de 2 mm de tamaño y cultivo de estos calli de forma independiente. De esta manera, se podrían aislar varias líneas de cada explanta, lo que aumenta el número de líneas transgénicas potenciales.
En el protocolo actual, las líneas transgénicas de P. andersonii se propagan vegetativamente a través de la propagación in vitro. La ventaja de esto es que muchas plantas transgénicas se pueden generar en un período de tiempo relativamente corto. Sin embargo, este método también tiene varias limitaciones. En primer lugar, el mantenimiento de las líneas transgénicas T0 a través de la propagación in vitro requiere mucha mano de obra y podría dar lugar a alteraciones genéticas o epigenéticas no deseadas34,35. En segundo lugar, las líneas T0 todavía contienen una copia del ADN T, incluido el casete de resistencia a antibióticos. Esto limita el número de posibles retransformaciones, ya que se requieren diferentes marcadores de selección para cada retransformación. Actualmente, solo hemos probado la transformación utilizando la selección de kanamicina o higromicina (datos no mostrados). Además, la presencia de la secuencia de codificación Cas9 y los sgRNA en las líneas transgénicas T0 complica los estudios de complementación. Los ensayos de complementación son posibles, pero requieren que los sitios objetivo del sgRNA sean mutados como tales para evitar la edición génica de la construcción de la complementación. En tercer lugar, una desventaja de trabajar con líneas T0 es que los mutantes CRISPR/Cas9 podrían ser quiméricos. Para evitar el análisis fenotípico de las líneas mutantes quiméricas, recomendamos repetir el análisis de genotipado después de la propagación in vitro en al menos 3 brotes diferentes. Aunque, el número de mutantes quiméricos obtenidos usando el protocolo descrito aquí es limitado, ocasionalmente se observan10. Para superar las limitaciones de trabajar con líneas T 0, las líneas mutantes de P. andersonii podrían propagarse generativamente. Los árboles P. andersonii son dioicas y polinizadas por el viento2. Esto significa que cada línea transgénica necesita ser manipulada como tal para que las flores masculinas y femeninas se produzcan en un solo individuo, y posteriormente se cultivan como tal que no se produce la polinización cruzada. Como P. andersonii es un árbol de rápido crecimiento que requiere una cantidad sustancial de espacio en un invernadero tropical (28 oC, 85% de humedad relativa). Por lo tanto, aunque técnicamente posible, la propagación generativa de las líneas transgénicas de P. andersonii es logísticamente desafiante.
En la sección de protocolo, describimos 3 métodos para la nodulación de P. andersonii. La ventaja de los sistemas de placas y bolsas es que las raíces son fácilmente accesibles, lo que puede permitir la inoculación puntual de bacterias y después de la formación de nódulos con el tiempo. Sin embargo, el sistema de placas es bastante laborioso, lo que lo hace menos adecuado para experimentos de nodulación a gran escala. Una desventaja del sistema de la bolsa es que es difícil prevenir la contaminación por hongos. Las bolsas no son estériles, y por lo tanto el crecimiento de hongos se observa a menudo en la mitad superior de la bolsa. Sin embargo, Esto no afecta el crecimiento de P. andersonii, y por lo tanto no interfiere con los ensayos de nodulación. Además, el sistema de bolsa sólo es adecuado para plántulas. A pesar de varios intentos, hemos sido incapaces de cultivar plantitas obtenidas a través de la propagación in vitro en bolsas.
La canalización de genética inversa P. andersonii descrita aquí ofrece una mejora sustancial en comparación con el método de transformación de raíz basado en A. rhizogenesexistente 11. Utilizando los procedimientos descritos, las líneas transgénicas estables se pueden generar eficientemente y se pueden mantener a través de la propagación in vitro. Por el contrario, la transformación de A. rhizogenes es transitoria y sólo resulta en la formación de raíces transgénicas. Debido a que cada raíz transgénica resulta de una transformación independiente, los ensayos basados en la transformación de A. rizogenes sufren de variación fenotípica sustancial. Esta variación es mucho menor en el caso de líneas estables, aunque la propagación in vitro también crea cierto nivel de variación. Debido a esta variación reducida y el hecho de que múltiples plantitas podrían ser fenotipo para cada línea estable, las líneas estables son más adecuadas para ensayos cuantitativos en comparación con A. rhizogenes-raíces transformadas. Además, la transformación estable no depende de la introducción de la raíz A. rizogenes inducir locus (rol) que afecta el equilibrio hormonal endógeno15. Por lo tanto, las líneas estables son más adecuadas para el análisis genético inverso de genes implicados en la homeostasis hormonal en comparación con las raíces transformadas de A. rhizogenes. Una ventaja más general de P. andersonii como modelo de investigación es que no experimentó una duplicación reciente del genoma entero (WGD). La leguminosa Subfamilia Papilionoideae, que incluye el modelo de legumbres M. truncatula y L. japonicus, así como las Salicaceae (orden Malpighiales) que incluye el árbol modelo Populus trichocarpa experimentado WGDs 65 millones de años hace36,37. Muchas copias genéticas parapálogas resultantes de estos GMT se conservan en los genomas de M. truncatula, L. japonicus y P. trichocarpa37,38,39, que crea redundancia que podría complicar los análisis genéticos inversos. Como P. andersonii no experimentó un WGD reciente, los análisis genéticos inversos en P. andersonii podrían verse menos afectados por el funcionamiento redundante de copias genéticas paralocóloas.
En conjunto, proporcionamos un protocolo detallado para el análisis genético inverso en P. andersonii. Usando este protocolo, las líneas mutantes individuales se pueden generar eficientemente en un período de tiempo de 2-3 meses10. Este protocolo se puede ampliar para crear mutantes de orden superior a través de la multiplexación de sgRNAs dirigidos a diferentes genes simultáneamente, como se muestra para otras especies de plantas40,41,42. Además, el procedimiento de transformación estable descrito aquí no se limita a la orientación genética CRISPR/Cas9, sino que también podría utilizarse para introducir otros tipos de construcciones (por ejemplo, para ensayos promotor-reportero, expresión ectópica o trans- complementamiento). Establecimos P. andersonii como un modelo de investigación comparativa para estudiar simbiosis mutuas con rizobia fijador de nitrógeno o hongos endomicorrhizal. Sin embargo, los protocolos descritos aquí también proporcionan herramientas para estudiar otros aspectos de la biología de este árbol tropical, como la formación de madera, el desarrollo de flores bisexuales o la biosíntesis de metabolitos secundarios específicos de Cannabaceae.
Acknowledgements
A los autores les gusta reconocer a Mark Youles, Sophien Kamoun y Sylvestre Marillonnet por hacer que las piezas de clonación de Golden Gate estén disponibles a través de la base de datos Addgene. Además, nos gustaría agradecer a E. James, P. Hadobas y T. J. Higgens por las semillas de P. andersonii. Este trabajo fue apoyado por la Organización Neerlandesa de Investigación Científica (NWO-VICI grant 865.13.001; NWO-Open Competition concede 819.01.007) y el Ministerio de Investigación, Tecnología y Educación Superior de la República de Indonesia (beca RISET-PRO 8245-ID).
Materials
| Name | Company | Catalog Number | Comments |
| Sigma-Aldrich | N0640 | NAA | |
| Duchefa Biochemie | M1503.0250 | MES | |
| Sigma-Aldrich | D134406 | Acetosyringone | |
| Duchefa Biochemie | X1402.1000 | X-Gal | |
| Merck | 101236 | For nucleic acid electrophoresis gel | |
| - | - | Pouches box material, hangers | |
| Merck | 101188 | NH4NO3 | |
| Sigma-Aldrich | B3408-1G | BAP | |
| Merck | 100156 | H3BO3 | |
| Thermo-Fisher | ER1011 | Used as restriction enzyme in Golden Gate cloning assembly | |
| Thermo-Fisher | 15561020 | Used in Golden Gate cloning assembly | |
| Merck | 137101 | CaCl2·2H2O | |
| Duchefa Biochemie | C0111.0025 | C16H16N5O7S2Na | |
| Thermo-Fisher | K1231 | Used for cloning the blunt-ended PCR amplicons in genotyping procedure | |
| Agronutrition | AP2011 | Containing Rhizophagus irregularis DAOM 197198 (1,000 spores/mL), used for mychorrization assay | |
| Merck | 102790 | CuSO4·5H2O | |
| Duchefa Biochemie | D1004.1000 | Used for plant tissue culture agar-based medium | |
| Merck | 105101 | K2HPO4 | |
| VWR Chemicals | 20302.293 | Na2·EDTA | |
| Duchefa Biochemie | M0803.1000 | C6H14O6 | |
| Thermo-Fisher | ER0291 | Used as restriction enzyme in Golden Gate cloning assembly | |
| Merck | 100983 | C2H5OH | |
| VWR Chemicals | BDH9232-500G | EDTA | |
| Sigma-Aldrich | Z377600-1PAK | Cellophane membrane | |
| Biomatters, Ltd. | R9 or higher | Bioinformatics software for in silico cloning and designing of sgRNAs | |
| Mega International | - | Technical information at https://mega-international.com/tech-info/ | |
| Sigma-Aldrich | 65882 | Used for fixating nodule tissues | |
| VWR Chemicals | 24385.295 | - | |
| Vink | 219341 | Pouches box material, bottom part | |
| Leica Biosystems | 14702218311 | Used as a template for plastic embedding | |
| Merck | 100317 | HCl | |
| Sigma-Aldrich | I5386-1G | IBA | |
| Merck | 103862 | C6H5FeO7 | |
| Merck | 103965 | FeSO4O·7H2O | |
| Duchefa Biochemie | I1401.0005 | IPTG | |
| Duchefa Biochemie | K0126.0010 | ||
| Sigma-Aldrich | L2000 | ||
| Merck | 105886 | MgSO4O·7H2O | |
| Merck | 105934 | MnCl2·4H2O | |
| Merck | 102786 | MnSO4O | |
| Duchefa Biochemie | M1002.1000 | Used for bacterial culture agar-based medium | |
| Manutan | 92007687 | Pouches material | |
| Paraxisdienst | 130774 | Elastic sealing foil | |
| Pull Rhenen | Agra-Perlite No.3 | Used as growing substrate in pots for nodulation assay | |
| VWR Chemicals | 391-0581 | Used as container for cellophane membranes | |
| Thermo-Fisher | F130WH | For genotyping transgenic lines | |
| Addgene | 50337 | Level 0 terminator, 3'UTR, 35s (Cauliflower Mosaic Virus) | |
| Addgene | 48017 | End-link 2 for assembling 2 level one part into a level 2 acceptor | |
| Addgene | 48018 | End-link 3 for assembling 3 level one part into a level 2 acceptor | |
| Addgene | 48001 | Level 1 acceptor. Position 5. Forward orientation | |
| Addgene | 48007 | Level 1 Acceptor. Position 1. Reverse orientation | |
| Addgene | 50268 | Level 0 promoter (0.4 kb), 35s (Cauliflower Mosaic Virus) + 5'UTR, Ω (Tobacco Mosaic Virus) | |
| Addgene | 46966 | Used for designing CRISPR/Cas9 module | |
| Addgene | 46968 | Used for designing CRISPR/Cas9 module | |
| Addgene | 50334 | Level 0 Kanamycin/Neomycin/Paromomycin resistance cassette | |
| Topzeven | - | Used as filters for washing spore suspension | |
| Sigma-Aldrich | 8.17003 | PEG400 | |
| Duchefa Biochemie | E1674.0001 | Pots to grow Parasponia plantlets/seedlings | |
| Merck | 104871 | KH2PO4 | |
| Merck | 105033 | KOH | |
| Merck | 105153 | K2SO4O | |
| Van Leusden b.v. | - | Used as growing substrate for mychorrhization assay | |
| Duchefa Biochemie | S0225.0050 | SH-basal salt medium | |
| Duchefa Biochemie | S0411.0250 | SH-vitamin mixture | |
| Lehle Seeds | VIS-02 | Used as non-ionic surfactant in the washing step of stable transformation | |
| Merck | 137017 | NaCl | |
| VWR Chemicals | 89230-072 | C6H11NaO7 | |
| Merck | 106521 | Na2MoO4·2H2O | |
| Merck | 106574 | Na2HPO4·7H2O | |
| Merck | 567549 | NaH2PO4·H2O | |
| Sigma-Aldrich | 239313 | Na2SO4O | |
| Duchefa Biochemie | S0809.5000 | C12H22O11 | |
| Thermo-Fisher | B69 | Used in Golden Gate cloning assembly | |
| Thermo-Fisher | EL0013 | Used in Golden Gate cloning assembly | |
| Kulzer-Mitsui Chemicals Group | 64708806 | Methyl methacrylate-based resin powder | |
| Kulzer-Mitsui Chemicals Group | 64709003 | HEMA (2-hydroxyethyl methacrylate)-based resin solution | |
| Kulzer-Mitsui Chemicals Group | 66022678 | Methyl methacrylate-based resin solution | |
| Merck | 1159300025 | ||
| Acros | 189350250 | ||
| VWR Chemicals | 663684B | Polysorbate 20 | |
| Stout Perspex | - | pouches box material, lid | |
| Duchefa Biochemie | Y1333.1000 | ||
| Merck | 108816 | ZnCl2 | |
| Alfa Aesar | 33399 | ZnSO4O·7H2O |
References
- Clason, E. W. THE VEGETATION OF THE UPPER-BADAK REGION OF MOUNT KELUT (EAST JAVA). Bulletin du Jardin Botanique de Buitenzorg. Serie III, 509-518 (1936).
- Soepadmo, E. Ulmaceae. Flora Malesiana-Series 1, Spermatophyta. 8, 31-76 (1974).
- Becking, J. H. The Rhizobium symbiosis of the nonlegume Parasponia. Biological Nitrogen Fixation. , 497-559 (1992).
- Oldroyd, G. E. D. Speak, friend, and enter: signalling systems that promote beneficial symbiotic associations in plants. Nature Reviews Microbiology. 11, 252-263 (2013).
- Gutjahr, C., Parniske, M. Cell and developmental biology of arbuscular mycorrhiza symbiosis. Annual Review of Cell and Developmental Biology. 29, 593-617 (2013).
- van Velzen, R., et al. Comparative genomics of the nonlegume Parasponia reveals insights into evolution of nitrogen-fixing rhizobium symbioses. Proceedings of the National Academy of Sciences of the United States of America. 115, E4700-E4709 (2018).
- van Velzen, R., Doyle, J. J., Geurts, R. A Resurrected Scenario: Single Gain and Massive Loss of Nitrogen-Fixing Nodulation. Trends in Plant Science. 24, 49-57 (2019).
- Griesmann, M., et al. Phylogenomics reveals multiple losses of the nitrogen-fixing root nodule symbiosis. Science. 1743, eaat1743 (2018).
- Becking, J. H. Root-Nodule Symbiosis Between Rhizobium And Parasponia (Ulmaceae). Plant and Soil. 51, 289-296 (1979).
- van Zeijl, A., et al. CRISPR/Cas9-Mediated Mutagenesis of Four Putative Symbiosis Genes of the Tropical Tree Parasponia andersonii Reveals Novel Phenotypes. Frontiers in Plant Science. 9, 284 (2018).
- Cao, Q., et al. Efficiency of Agrobacterium rhizogenes–mediated root transformation of Parasponia and Trema is temperature dependent. Plant Growth Regulation. 68, 459-465 (2012).
- Limpens, E., et al. RNA interference in Agrobacterium rhizogenes-transformed roots of Arabidopsis and Medicago truncatula. Journal of Experimental Botany. 55, 983-992 (2004).
- Boisson-Dernier, A., et al. Agrobacterium rhizogenes-Transformed Roots of Medicago truncatula for the Study of Nitrogen-Fixing and Endomycorrhizal Symbiotic Associations. Molecular Plant-Microbe Interactions. 14, 695-700 (2001).
- Kumagai, H., Kouchi, H. Gene Silencing by Expression of Hairpin RNA in Lotus japonicus Roots and Root Nodules. Molecular Plant-Microbe Interactions. 16, 663-668 (2003).
- Nilsson, O., Olsson, O. Getting to the root: The role of the Agrobacterium rhizogenes rol genes in the formation of hairy roots. Physiologia Plantarum. 100, 463-473 (1997).
- Davey, M. R., et al. Effective Nodulation of Micro-Propagated Shoots of the Non-Legume Parasponia andersonii by Bradyrhizobium. Journal of Experimental Botany. 44, 863-867 (1993).
- Webster, G., Poulton, P. R., Cocking, E. C., Davey, M. R. The nodulation of micro-propagated plants of Parasponia andersonii by tropical legume rhizobia. Journal of Experimental Botany. 46, 1131-1137 (1995).
- Op den Camp, R., et al. LysM-type mycorrhizal receptor recruited for rhizobium symbiosis in nonlegume Parasponia. Science. 331, 909-912 (2011).
- Weber, E., Engler, C., Gruetzner, R., Werner, S., Marillonnet, S. A Modular Cloning System for Standardized Assembly of Multigene Constructs. PLOS ONE. 6, e16765 (2011).
- Nekrasov, V., Staskawicz, B. J., Weigel, D., Jones, J. D. G., Kamoun, S. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease. Nature Biotechnology. 31, 691-693 (2013).
- Bertani, G. Studies On Lysogenesis. I. The Mode Of Phage Liberation By Lysogenic Escherichia Coli. Journal of Bacteriology. 62, 293-300 (1951).
- Engler, C., et al. A Golden Gate modular cloning toolbox for plants. ACS Synthetic Biology. 3, 839-843 (2014).
- Fauser, F., Schiml, S., Puchta, H. Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering in Arabidopsis thaliana. The Plant Journal. 79, 348-359 (2014).
- Lazo, G. R., Stein, P. A., Ludwig, R. A. A DNA Transformation-Competent Arabidopsis Genomic Library in Agrobacterium. Biotechnology. 9, 963-967 (1991).
- Op den Camp, R. H. M., et al. N Nonlegume Parasponia andersonii Deploys A Broad Rhizobium Host Range Strategy Resulting in Largely Variable Symbiotic Effectivenes. Molecular Plant-Microbe Interactions. 25, 954-963 (2012).
- Graham, P. H., Viteri, S. E., Mackie, F., Vargas, A. T., Palacios, A. Variation in acid soil tolerance among strains of Rhizobium phaseoli. Field Crops Research. 5, 121-128 (1982).
- Martinez-Romero, E., et al. Rhizobium tropici, A Novel Species Nodulating Phaseolus vulgaris L. Beans and Leucaena sp. Trees. International Journal of Systematic and Evolutionary Microbiology. 41, 417-421 (1991).
- Felten, J., et al. The Ectomycorrhizal Fungus Laccaria bicolor Stimulates Lateral Root Formation in Poplar and Arabidopsis through Auxin Transport and Signaling. Plant Physiology. 151, 1991-2005 (1991).
- Trouvelot, A., Kough, J. L., Gianinazzi-Pearson, V. Mesure du taux de mycorhization VA d’un systeme radiculaire. Recherche de methods d’estimation ayant une signification fonctionnelle. Aspects Physiologiques et Genetiques des Mycorhizes. , 217-221 (1986).
- Schenk, R. U., Hildebrandt, A. C. Medium and techniques for induction and growth of monocotyledonous and dicotyledonous plant cell cultures. Canadian Journal of Botany. 50, 199-204 (1972).
- Becking, J. H. The Parasponia parviflora - Rhizobium symbiosis. Host specificity, growth and nitrogen fixation under various conditions. Plant and Soil. 75, 309-342 (1983).
- Hoagland, D. R., Arnon Revised, D. I., Arnon, D. I. The Water-Culture Method for Growing Plants without Soil. Circular California Agricultural Experiment Station. 347, 1-32 (1950).
- Wang, K. . Methods in Molecular Biology: Agrobacterium Protocols. 1, (2015).
- Smulders, M. J. M., de Klerk, G. J. Epigenetics in plant tissue culture. Plant Growth Regulation. 63, 137-146 (2011).
- Larkin, P. J., Scowcroft, W. R. Somaclonal variation — a novel source of variability from cell cultures for plant improvement. Theoretical and Applied Genetics. 60, 197-214 (1981).
- Cannon, S. B., et al. Legume genome evolution viewed through the Medicago truncatula and Lotus japonicus genomes. Proceedings of the National Academy of Sciences of the United States of America. 103, 14959-14964 (2006).
- Tuskan, G. A., et al. The Genome of Black Cottonwood, Populus trichocarpa (Torr. & Gray). Science. 313, 1596-1604 (2006).
- Young, N. D., et al. The Medicago genome provides insight into the evolution of rhizobial symbioses. Nature. 480, 520 (2011).
- Sato, S., et al. Genome Structure of the Legume, Lotus japonicus. DNA Research. 15, 227-239 (2008).
- Xing, H. L., et al. A CRISPR/Cas9 toolkit for multiplex genome editing in plants. BMC Plant Biology. 14, 327 (2014).
- Lowder, L. G., et al. A CRISPR/Cas9 Toolbox for Multiplexed Plant Genome Editing and Transcriptional Regulation. Plant Physiology. 169, 971 (2015).
- van Zeijl, A. . Dissecting Hormonal Pathways in Nitrogen-Fixing Rhizobium Symbioses. , (2017).
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved