使用色马格龙的超分辨率生物成像时代3D色变的高精度校正
摘要
校正三维(3D)多色荧光显微镜图像中的色移对于定量数据分析至关重要。该协议旨在通过获取合适的参考图像和使用开源软件Chromagnon进行处理,测量和校正生物样品的色变。
摘要
定量多色荧光显微镜依赖于不同波长获得的颜色通道的精心空间匹配。由于色差和摄像机的不完全对齐,每个通道中获取的图像可能会移动和放大,并在三个维度中的任何一个中相对旋转。使用经典的校准方法,色变通过附着在盖玻片表面的多色珠子进行测量,并且有许多软件可用于测量此类校准样品的色差。然而,色差随深度变化而变化,随观察条件而变化,由生物样品本身引起,从而阻碍确定感兴趣样品中和体积中染色体变化的真实量。以更高的精度校正色差对于超分辨率显微镜特别相关,其中只有轻微的色移可能会影响定量分析,并改变多色图像的解释。我们开发了一种开源软件Chromagnon和配套方法,用于测量和校正生物样品中的3D色变。在这里,我们提供详细的应用协议,其中包括样品制备、数据采集和软件处理的特殊要求,以测量感兴趣的生物样品的色的变化。
引言
多色成像是生物荧光显微镜的基本方面之一,在不同的分子或结构的空间关系是主要利益的情况下。色差是色散引起的多色光的光学畸变,它改变了感兴趣的彩色物体的表观位置。同样,由于光学元件的差异和通道之间的对齐不完美,配备多台摄像机的显微镜也具有更复杂的色变。因此,除非用户明确更正,否则这种色差可能导致错误的结论。尽管只要显微镜的分辨率受到经典分辨率限制的限制,色差并不是一个主要问题,但最近超分辨率显微镜1的发展促使需要更准确地校正色变。
使用多色珠标片2测量显微镜系统的色移是一种常见做法。基于珠子的校准方法适用于测量从显微镜的整个光学元件到盖玻片2表面的色变。然而,这种方法无法测量感兴趣的生物样品中的色的变化。需要注意的是,许多生物样品是三维(3D),这种样品的色变与盖玻片表面的色变不同。此外,色变随成像条件22、33而变化。我们测量了3D生物样本的色变,发现通过经典的多色珠校准方法3,色移的不确定性通常高达350nm。因此,需要在生物样品中测量色变,在感兴趣的深度和所使用的成像条件下。
在这里,我们描述了测量生物样品中染色体变化的程序,并使用我们的软件Chromagnon3纠正这些变化。为了测量生物样本的色变,我们的方法使用两种数据集,一种是"目标"图像和一个"参考"图像。"目标"图像是感兴趣的多色图像,例如,DNA、核包络和微管的图像。在这样的图像中,通常不可能测量色的变化。因此,我们需要一个专门用于测量样品中色移的"参考"图像。"参考"图像的唯一定义是同一对象的多色图像。从这个意义上说,多色珠图像也是一种参考图像。在这里,我们描述了用于测量生物样本中色变的三种不同类型的参考图像:"串扰参考图像"、"明亮场参考图像"和"生物校准参考图像"。参考图像的类型取决于所使用的显微镜类型或表 1中总结所需的校正精度。
| 串 扰 | 亮场 | 生物校准(在不同的幻灯片上) | 生物校准(在同一张幻灯片上) | |
| 精度a | +++ | + | #b | +++ |
| 简单 | ++ | +++ | ++ | ++ |
| 适用显微镜 | 宽场 | 宽场 | 所有 | 所有 |
| 本地对齐的可用性 | + | + | -c | -c |
|
a: "+"的数量表示评级增加。单加号约为 50 nm,三加在 3D 中约为 15 nm。 b: 精度取决于可变成像条件保持恒定程度。 c:多色珠样品测量的局部校准可以组合在协议第4节中。 | ||||
表 1:选择参考图像类型的参数。
"串扰参考图像"具有最高的校正精度,并且相对简单,只需完成33、4(4表 1)。缺点是由于它们无法测量激励路径的色变,因此在显微镜应用中受到限制。此外,为了获得这样的图像,显微镜应配备多波段二色镜和发射过滤器,这些滤镜独立于激励滤波器或光源控制。合适的显微镜包括传统的广域显微镜、单分子定位显微镜(SMLM),如光活化定位显微镜/随机光学重建显微镜(PALM/STORM)5、6和扩展显微镜7,通过广域显微镜观察。5,6从目标样本本身获取串扰参考图像。它是在所有所需通道中获得的染料的串扰(出血)荧光的图像。荧光发射总是向较长的波长扩展,因此发射波长最短的染料在较长波长的通道中兴奋地获得串扰荧光。例如,当样品被蓝色、绿色和红色染色时,只有蓝色染料是兴奋的,并且发射灯是在蓝色、绿色和红色通道中获得的。在该协议中,用4_6-二酰胺-2-苯胺酚(DAPI)染色的DNA用于获得串扰荧光。
"明亮场参考图像"是"串扰参考图像"的一种更简单、少光毒性的替代品,但最不准确的3(表1)。这些是目标样本的明亮场图像,在目标图像中使用的所有颜色通道中获取。
"生物校准参考图像"的优点适用于任何类型的显微镜,因为他们能够测量激励和发射路径33、88中的色变(表1)。合适的显微镜包括广场显微镜、共聚焦显微镜、光片显微镜、刺激发射耗竭(STED)9、结构照明显微镜(SIM)10、空气扫描/SORA1011、12、SMLM观测与11,12总内部反射荧光(TIRF)模式、奥林巴斯超分辨率(OSR)13等。13生物校准参考图像取自与目标样品类似的校准样品,但具有多种颜色的单一结构染色。校正精度优于大多数超分辨率显微镜的分辨率,制备生物校准样品可能相对简单。另一个优点是可"平均"多个参考映像。因此,即使单个图像包含用于测量色变的不良信息,信息内容可以通过平均多个图像来增加。精度取决于成像条件保持恒定程度。在这方面,当目标和参考样品都在同一张幻灯片上时,使用8口室盖眼镜(表1,最右边)时,可以获得最佳性能。在此协议中,沾有三种颜色的邻苯二甲二甲酮的行为素被用作生物校准。
获得参考图像后,我们的软件Chromagnon测量和校正色变。Chromagnon可以测量和校正色变的通道数、Z 节数和时间帧没有限制。色谱测量两个步骤的色变。第一步获取 X、Y、Z 轴中平移的"全局"或"仿"对齐参数、沿 X、Y、Z 轴的放大倍数以及 Z 轴周围的旋转。全局对齐的计算精度为3D为±16 nm,2D为±8nm。第二步是对投影图像的可选 2D 迭代"局部对齐",以获得更高的精度。在局部对齐过程中,图像被细分为多个区域,并测量这些局部区域的色变。随后,区域进一步划分,并迭代测量子区域中的色移,直到该区域中的像素数达到最小像素数(通常为 60 x 60 像素)。生成的局部对齐贴图与全局对齐参数相结合,并通过弹性变换应用于目标图像。按照此步骤,计算精度在 3D 中提高到 ±14 nm,在 2D 中提高到 ±6 nm。局部对齐不适合生物校准参考图像,因为参考中的生物结构与目标中的结构不同(表1)。因此,只有全局对齐用于生物校准参考图像。
局部色变来自两个来源;显微镜器位局部变形和生物结构不均匀。由于显微镜器器局部失真是恒定的,因此可以从多色珠子参考样品中测量,并作为固定参数进行校正。Chromagnon可以从生物校准中组合显微镜局部失真图和全局对齐参数(表 1)。使用这种方法,生物校准的平均精度有望提高1⁄2nm。
在这里,我们描述了一个协议,用我们的软件Chromagnon来校正3D荧光图像的色差变化,从最简单的低端到最高的精度。我们以HeLa细胞的免疫染色为例,并使用3D宽场显微镜和3D-SIM观察它们。在第一部分中,我们将介绍如何制备目标样品和生物校准样品。协议的这一部分应针对研究的具体目标进行优化。第二部分介绍了显微镜三种参考图像的采集方法。假设是获得蓝色、绿色和红色通道,但通道组成应通过研究的具体目标和显微镜的设置加以修改。显微镜是否配备单个摄像机或多个摄像机并不重要。在第三部分中,我们将介绍如何使用我们的软件使用参考图像测量和校正目标图像的色差。最后,在第四部分,我们描述了一种使用显微镜的仪器局部校准来补充生物校准参考图像的方法。
研究方案
1. 样品制备
- 准备目标样本
- 种子2.5 x 105 HeLa细胞在35毫米玻璃底盘上生长,并在2mL的生长介质中生长(Dulbeco的改良鹰中,含有L-谷氨酰胺和丙酮钠,辅以10%胎儿牛血清[FBS]),在37°C和CO2浓度为5%。或者,在8口腔盖玻片上播种6 x 104 HeLa细胞,并在37°C的0.5mL生长介质中生长,CO2浓度为5%。使用 #1.5 盖玻璃(厚度为 0.170 mm)进行高分辨率显微镜。
注:使用 1/4 卷用于 8 井室盖玻片的进一步步骤,但步骤 1.1.5、1.1.6、1.1.8、1.1.11 和 1.2.5,无论容器类型如何,体积为 100 μl。 - 约24小时后孵育后,在磷酸盐缓冲盐水(PBS)中用2 mL3.7%甲醛代替溶液。轻轻混合后,在室温 (RT) 下继续在旋转平台上固定细胞 15 分钟。
注意:在烟罩中工作。 - 使用 2 mL PBS 清洗电池,在旋转平台上每 3 次 5-10 分钟。
注:遵守当地法规处置甲醛废物。 - 在旋转平台上将 PBS 中的 2 mL 0.1% Triton X-100 的细胞渗透 5 分钟,然后用 2 mL PBS 进行 3 次处理,每个套圈在旋转平台上 5-10 分钟。
- 在PBS中孵育100 μL的1%牛血清白蛋白(BSA),在旋转平台上RT中孵育1小时。
- 在适当的稀释剂(1/500用于抗黄蛋白,1/100用于抗图布林),在PBS中,在1%BSA中,将原抗体混合物(抗埃梅林多克隆抗体14和抗图布林单克隆抗体15)的混合物替换溶液,并在4°C下通宵孵育。
- 在旋转平台上用 2 mL PBS 清洗电池,每个电池 3⁄5x 10 分钟。
- 在PBS中1%BSA中,在1/500稀释时,用100 μL的二次抗体混合物(用Alexa Fluor 488和抗小鼠IgG与Alexa Fluor 555)混合,用100μL的溶液代替溶液,并在RT时孵育3⁄4小时。
- 在旋转平台上用 2 mL PBS 清洗电池,每个电池 3⁄4x 5 分钟。
- 将溶液替换为 PBS 中的 2 mL 0.5 μg/mL DAPI,在旋转平台上在 RT 上染色 DNA 30 分钟。
注:使用同浓度的Hoechst 33342,而不是DAPI。 - 将溶液替换为 ±100 μL 的安装介质(材料表)。
- 种子2.5 x 105 HeLa细胞在35毫米玻璃底盘上生长,并在2mL的生长介质中生长(Dulbeco的改良鹰中,含有L-谷氨酰胺和丙酮钠,辅以10%胎儿牛血清[FBS]),在37°C和CO2浓度为5%。或者,在8口腔盖玻片上播种6 x 104 HeLa细胞,并在37°C的0.5mL生长介质中生长,CO2浓度为5%。使用 #1.5 盖玻璃(厚度为 0.170 mm)进行高分辨率显微镜。
- 生物校准样品的制备
- 按照步骤 1.1.1_1.1.4 中所述的准备固定单元格。
注:最好在室盖玻璃上制备样品,将靶样和参考样品放在同一盖玻片上的单独腔室中(表1,最右边)。当实验要求最高的校正精度时,最好进行这种制备。当样品需要在常规盖玻片上准备时,使用厚度变化率低的精密盖玻片("1.5H号")以确保可重复性。 - 在1.5mL甲醇中制备荧光染料偶联醇素的库存溶液,并储存在-20°C。在PBS混合phalloidin股票溶液在以下稀释:1/100 phalloidin与Alexa Fluor 405,1/1000的phalloidin与Alexa Fluor 488,和1/200的phalloidin与Alexa Fluor 594。
- 将溶液替换为步骤 1.2.2 中准备的 1 mL 的 phalloidin 混合物,并在旋转平台上在 RT 上孵育 30 分钟。
- 在旋转平台上用 2 mL PBS 清洗电池,每个电池 3⁄5x 10 分钟。
- 将溶液更换为 ±100 μL 的安装介质。
注:校准样品可储存在4°C或-20°C下,供重复使用。
- 按照步骤 1.1.1_1.1.4 中所述的准备固定单元格。
2. 获取参考图像
- 串扰参考图像
- 将步骤 1.1 中准备的目标样品放在宽场显微镜上。
- 以蓝色、绿色和红色通道获取目标的荧光图像。
注: 对于 3D 图像,只要图像文件包含步长大小的信息,参考图像中的 Z 步长大小就可以与目标图像中的 Z 步长大小不同。用于参考和目标图像的 Z 步进大小优选小于 Z 轴光学分辨率的一半,该分辨率由衍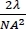 射限制显微镜计算,其中α是纳米波长,NA是目标透镜的数字光圈。例如,如果轴向分辨率为 550 nm,则使用小于 275 nm 的 Z 步长。参考图像不需要时间序列。
射限制显微镜计算,其中α是纳米波长,NA是目标透镜的数字光圈。例如,如果轴向分辨率为 550 nm,则使用小于 275 nm 的 Z 步长。参考图像不需要时间序列。 - 随后,要获取参考的荧光图像,请仅为 DAPI 选择激发光,然后选择以蓝色、绿色和红色通道获取。在 3D 堆栈的情况下,在同一阶段位置和 Z 高度完全获取参考图像。
注:对于较长的发射波长,需要增加照明强度和曝光时间。
- 亮场参考图像
- 将步骤 1.1 中准备的目标样品放在宽场显微镜上。
- 以蓝色、绿色和红色通道获取目标的荧光图像。
注:Z 步长大小最好满足步骤 2.1.2 中所述的奈奎斯特标准。 - 在蓝色、绿色和红色通道中完全在 2.2.2 和 Z 高度相同的阶段位置(在 3D 堆栈中)获取目标的明亮场图像。
- 生物校准参考图像
- 将步骤 1.1 中准备的目标样品放在 3D-SIM 显微镜上。
- 通过 3D-SIM 以蓝色、绿色和红色通道获取目标的荧光图像。重建超解析图像。
注:显微镜的类型可以是任何类型的,如3D-SIM,共聚焦,STED等。Z 步长大小最好满足步骤 2.1.2 中所述的奈奎斯特标准。 - 通过 3D-SIM 在类似于目标图像(步骤 2.3.2)的不同阶段位置获取步骤 1.2 中准备的参考的多个荧光图像。重建超解析的图像。
注: 所有参考图像中的 XY 中的像素总数必须相同。在所有参考图像中,Z 中的步长大小必须相同。Z 节的总数与目标图像中的 Z 节总数相同,但这不是绝对要求。这些参考图像的 XY 位置并不重要,因为位置或盖玻片已与目标样品的位置不同,而且单个盖玻片上的色移差小于 15 nm3。成像条件包括客观透镜、观察温度、浸没油、共聚焦显微镜中的针孔尺寸和高倾斜照明显微镜16中的倾斜角度,应均符合参考,以获得最佳性能。如果显微镜使多个摄像机能够同时获取多个通道,则参考图像应每周一次获得,以校正仪器漂移。
3. 使用色马龙软件校正色移
- 使用 Web 浏览器,转到https://github.com/macronucleus/Chromagnon/releases,并下载Chromagnon的最新二进制版本 。
注意:二进制版本可用于 Windows、Mac 和某些 Linux 版本。 - 提取程序并将可执行文件放在方便的位置。在 Windows 或 Mac 上双击文件以打开该文件,否则从 Linux 系统上的命令行执行二进制文件。
注: 图形用户界面如图1所示将打开。由于安全原因,第一次双击可能会阻止程序打开。在这种情况下,使用与平台相关的方法打开从 Internet 下载的程序。
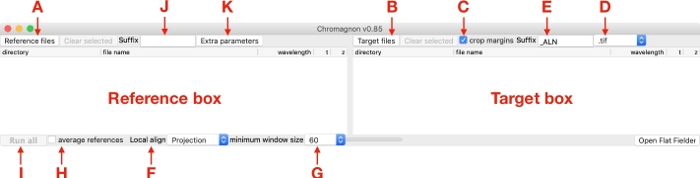
图1:Chromagnon图形用户界面的屏幕截图。请点击此处查看此图形的较大版本。
- 在"参考框"(图 1)中拖放参考文件,或单击参考文件(图 1A)以打开文件选择器对话框。根据文件格式,如果程序要求安装 Java 开发工具包 (JDK),请按"是",用户将导航到下载页面。按照指示下载并安装操作系统的 JDK。重新启动程序后,Chromagnon应该能够读取图像文件格式。
注: Chromagnon可以读取大多数图像文件格式(多页"tif"、"czi"、"nd2"、"oib"、"lif"、"dv"等)。在此步骤中,首选原始显微镜图像格式,因为当图像格式转换为多页 tif 文件时,元数据可能会丢失。如果通道名称显示为 0、1、2 等,而不是波长(如图2A、绿色框),则Chromagnon不知道图像中通道的标识。在这种情况下,请确保引用文件中的通道顺序和像素大小与目标文件中的通道顺序和像素大小匹配。例如,如果参考中的通道顺序为绿色和红色,则目标中的通道顺序也必须为绿色和红色,但不能为红色和绿色。
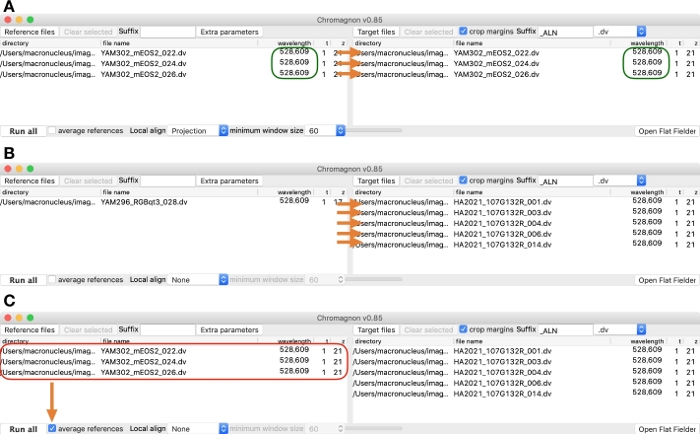
图 2:用于加载多个文件的示例屏幕截图。(A) 所有参考图像都有相应的目标图像的情况。通道名称通过本示例中使用的图像文件中的波长(由绿色框指示)正确标识。(B) 使用单个参考图像校正多个目标图像的情况。(C) 对多个参考图像(由红色框指示)求平均值的情况,以及用于校正多个目标图像后生成的参考图像的情况。请点击此处查看此图形的较大版本。
- 在"目标框"(图 1)中拖放目标文件,或单击"目标文件"(图 1B)以打开文件选择器对话框。
注:如果有多个目标图像,并且每个图像都有相应的参考图像,则相应的参考图像和目标图像必须位于相应参考框和目标框中的同一行上(图 2A)。单个参考图像也可用于对齐多个目标图像(图 2Figure 2B)。 - 如果未选中,请选中裁剪边距复选框(图 1C)。
注: 如果选中此复选框,则裁剪由对齐产生的边距。选中此选项以进行常规使用,但取消选中校准前后图像之间是否需要进行像素级比较。 - 从选择列表中选择输出图像格式(图1D)。
注: 可用的格式是"tIF"(ImageJ17格式)、"dv"和"ome.tif";"ome.tif"格式仅在安装 JDK 后可用。 - 指定输出文件名的后缀(图 1E,默认值为"_ALN"),该默认值将添加到目标文件名中。
- 对于具有高信噪比的串扰参考图像,请使用局部对齐选项列表(图 1F)中的局部对齐,选择"投影"以使用局部对齐和None禁用它。使用最小窗口大小 60 (图 1G)。
注: 使用局部对齐时,所有目标图像都必须具有相应的参考图像(图 2A)。不建议生物校准参考图像进行局部对齐,因为局部色差因样本而异(表 1)。出于同样的原因,单个局部对齐不能应用于许多目标图像(图 2B)。作为例外,当感兴趣的样本仅位于盖玻片表面,并且视野中填充明亮的物体时,可以使用它,就像多色荧光珠样品一样。 - 对于多个生物校准参考图像,请检查平均参考选项 (图 1H),以测量平均图像中的单个对齐参数,然后将单个对齐参数应用于所有目标图像(图 2C)。为局部对齐选择"无"(图 1F)。
- 单击"全部运行" (图 1I) 以开始测量;对齐参数应用于目标图像。
注:在从参考文件中测量色变后,程序制作扩展名"chromagnon.csv"或"chromagnon.tif"的文件。输出文件的类型取决于局部对齐是否有效(图 1F)。如果对齐在没有局部对齐的情况下完成,则输出为"chromagnon.csv",而如果使用局部对齐,则输出为"chromagnon.tif"。文件名与参考文件相同,但指定的后缀(图 1J,默认值没有后缀)和扩展名("chromagnon.csv"或"chromagnon.tif")。在同一文件夹中创建的"Chromagnon.log"文件中可以找到对齐过程的详细说明。 - 等待,直到更正的图像显示在查看器中(图 3)。
注: 用鼠标拖动图像可移动图像,移动鼠标滚轮可更改缩放。移动滑块 (图 3A) 以更改 Z(和/或 T(如果适用)部分以进行显示。如果查看器太慢无法刷新图像,请单击"将整个数据加载到内存"按钮(图3B)以停止访问硬盘上的数据。拖动颜色框的左边缘或右边缘 (图 3C) 可以控制显示的最小值或最大值。单击每个通道的按钮 (图 3D) 在查看器中显示或隐藏所选通道之间切换。右键单击颜色框(图 3D)使用户能够选择显示颜色,或更改颜色栏的显示选项,并且还允许用户通过选择"缩放到 ..."来指定显示缩放的确切最小值和最大值。单击正交视图按钮 (图 3E) 可显示 ZY 和 XZ 视图的图像。移动横线会更改要显示在侧视图中的位置。
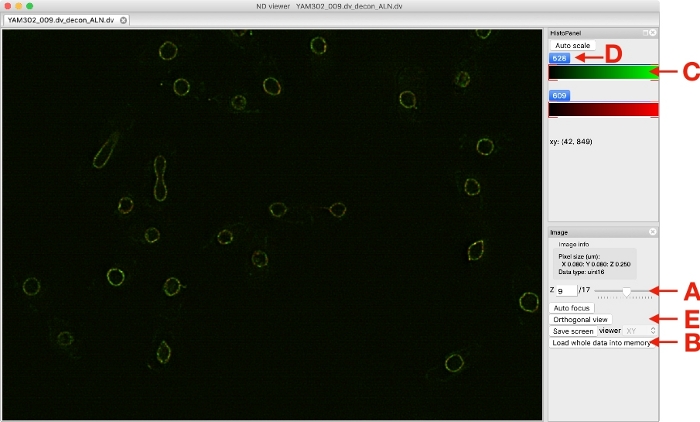
图 3:图像查看器的屏幕截图。请点击此处查看此图形的较大版本。
- 要检查测量是否正确,请将参考图像拖放到"参考框"和"目标框"中。运行程序,并检查图像是否完全重叠。
注:如果可用,"chromagnon.csv"或"chromagnon.tif"文件可用作参考,以便跳过测量。如果图像中的色移仍未校正,请尝试在表 2中汇总的解决方案。 - 要访问示例中的对齐参数,或者当需要手动编辑对齐参数时,使用任何文本编辑器或电子表格软件直接打开"chromagnon.csv"文件。编辑后,将其另存为"csv"。
注:这些值以像素为单位表示"tz"、"ty"、"tx"和"r"的度数,以及"mz"、"my"和"mx"的放大系数。- 或者,将"chromagnon.csv"或"chromagnon.tif"文件加载到Chromagnon的参考或目标框(图1),然后双击打开对齐编辑器(图4)。
- 要查看编辑参数时通道偏移量,请将参考图像文件拖放到底部区域(图 4A)以打开编辑器中的图像。编辑表中的值(图 4Figure 4B)并命中输入/返回键时,观察相应通道的图像在值更改时如何移动。编辑后,单击"保存为..." (图 4C) 以保存更改。
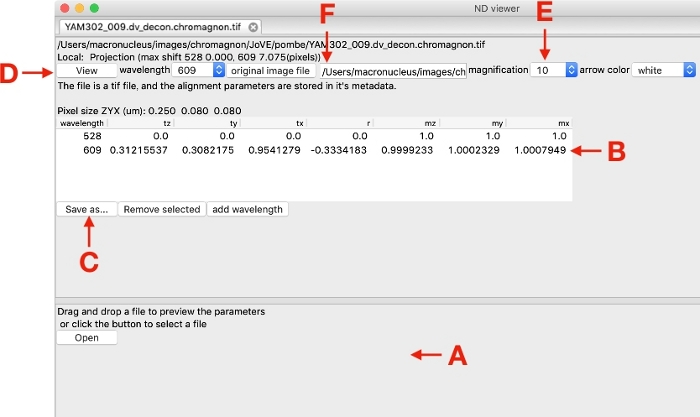
图 4:对齐参数编辑器的屏幕截图。请点击此处查看此图形的较大版本。
- 要检查局部对齐贴图,请通过Chromagnon的参考或目标框加载"chromagnon.tif"(图 1),然后双击以打开对齐编辑器(图 4)。单击视图(图 4D) 以查看由长度被放大的放大因子(图 4E) 的行指示的局部偏移。
注: 通过指定"原始图像文件"(图 4F),这些偏移与原始图像一起映射。
4. 生成显微镜特定的局部对齐图
- 在18μL乙醇中稀释直径为200nm的2μL多色荧光珠2μL。
- 将溶液涡旋,并将 10 μL 的珠溶液放在玻璃底盘的中心。确保使用#1.5 盖玻璃(厚度为 0.170 mm)进行高分辨率显微镜。
- 将菜在 RT 处保持 1 小时,完全干燥。
- 将盘子放在显微镜上进行校准,并聚焦于荧光珠。
- 使用要校准的显微镜获取 2D 或 3D 图像。通过更改舞台位置获取多个图像。
- 打开色图图形用户界面。
- 在"参考框"(图 1)上拖放珠图像文件,或单击参考文件(图 1A)以打开文件选择器对话框。
- 检查平均引用选项(图 1H)。从局部对齐的选择列表中(图1F),选择投影。使用最小窗口大小 60 (图 1G)。单击"全部运行" (图 1I) 以开始测量。
注: 创建一个".chromagnon.tif"文件,其中存储显微镜的局部对齐映射。 - 测量后,单击"额外参数"(图 1K)。从显微镜仪器框的局部失真中,单击选择列表并选择"新建..."以打开对话框。将步骤 4.8 中生成的"chromagnon.tif"文件拖放到文件名框中,或单击"选择文件"按钮以打开文件选择器对话框。输入显微镜的名称,然后单击"确定"。
- 测量来自串扰或生物校准参考图像的色差时,单击"额外参数"(图 1K)。从显微镜仪器箱的局部失真中,选择步骤 4.9 中指定的显微镜名称。单击"确定"。
注:程序关闭后,不会保存"显微镜仪器的局部失真"的选择。 - 在没有局部对齐的情况下继续进行色度校正,如第 3 节所述。
注: 除了显微镜校准外,还可以使用局部校准。在这种情况下,局部校准从显微镜校准开始。
结果
图 5显示了使用串扰参考图像进行色移校正的示例。图像是使用配备单个摄像机的宽场显微镜获得的。DAPI 的荧光发射用作参考(图 5Figure 5A,B),以校正蓝色、绿色和红色通道。图像由 3 个通道组成 60 个 Z 切片,每个通道由 256 x 256 像素组成。在测量色变之前,这些图像被解光化。使用Chromagnon测量本地色变需要使用英特尔酷睿 i7(四核、8 线程、2.7 GHz)、16 GB RAM 和 1 TB 闪存的 Mac 上 51 s。对齐参数应用于目标图像(图 5Figure 5C,D),其体素数与参考图像完全相同。准备对齐的文件需要 3 s。由于在对齐过程中修剪边缘像素(图 1C中的裁剪边距复选框),对齐后体素数量减少(图 5B,51Z 切片,252 x 251 像素)。相位桥中的DNA(由箭头指示)在对齐前在核包络外被错误地看到(图5C,在显示XZ视图的底部面板中显而易见),但对齐后信封内的预期(图5D)。
图6显示了使用生物校准参考图像进行色移校正的示例。这些图像是使用装有三台摄像机的SIM显微镜获得的。平均了三张HeLa细胞的图像,它们与蓝色、绿色和红色染料结合在一起,被染成邻苯二甲a素(图6A)。参考图像包括 3 个通道,包含 76 个 Z 切片,每个通道由 1,024 x 1,024 像素组成。使用Chromagnon测量色变,无需进行局部校准,需要在上述 Mac 系统上测量 194 s。该参数应用于由 3 个通道组成的 73 Z 切片,每个通道由 1,024 x 1,024 像素组成。生成对齐的文件需要 25 s。XZ 视图显示沿 Z 方向的错误通道位置,并稍微沿 X 方向(图 6A,C),但对齐后纠正了此错误注册(图 6B,D)。
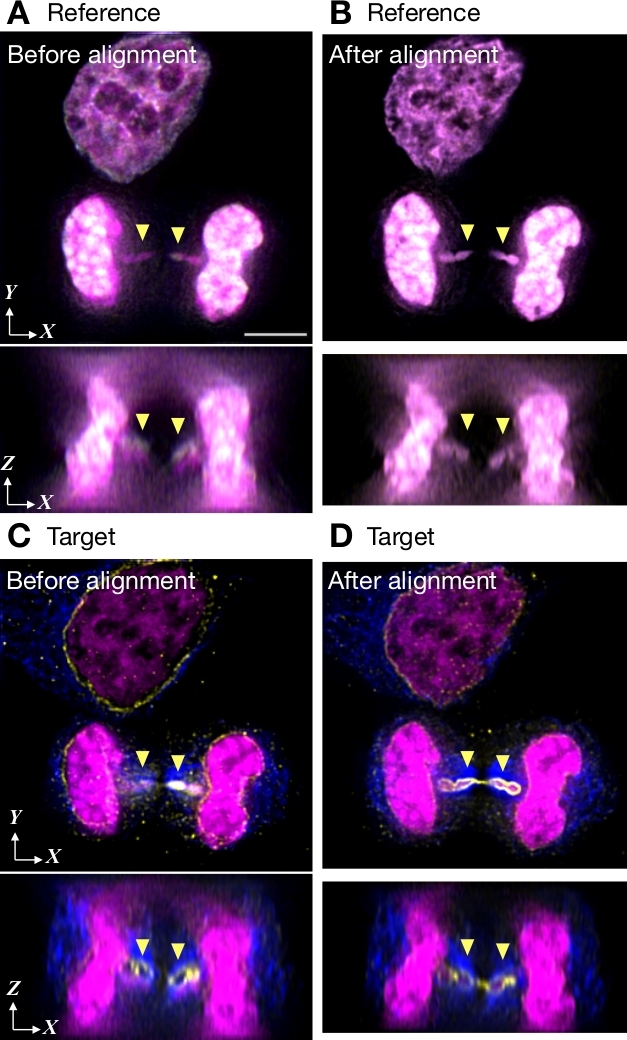
图 5:与串扰参考图像对齐的示例。HeLa细胞被染色DAPI的DNA(在洋红色中显示),Alexa Fluor 488(以黄色显示)用于核包,Alexa Fluor 555(以蓝色显示)用于微管。图像由3D宽场显微镜通过一台相机和装饰获得。(A, B)使用 DAPI 发射(由 Chromagnon进行对齐之前和之后)的代表性串扰参考图像。三个颜色通道显示为叠加。(C, D)对齐前后三个通道中 3D 堆栈的光学部分。轴向色差在箭头显示的相位桥中是显而易见的。面板 A 中的刻度条表示所有面板的 5 μm。请点击此处查看此图形的较大版本。
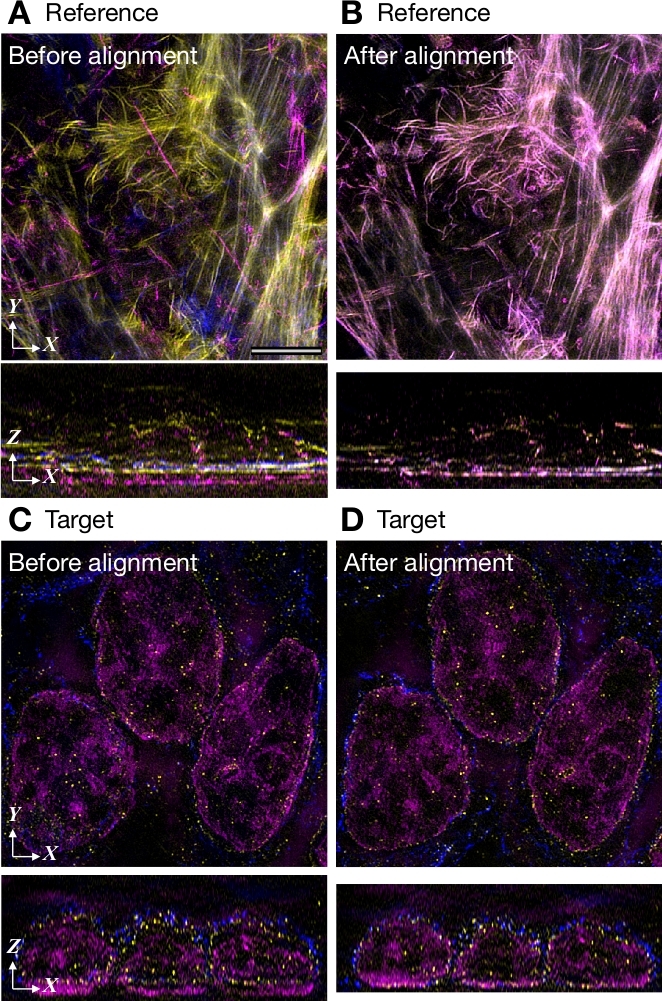
图 6:与生物校准参考图像对齐的示例图像是使用配备三台摄像机的 3D-SIM 获得的。(A, B)参考图像从三个图像的平均值之前 (A) 和之后 (B) 对齐。HeLa细胞与Alexa Fluor 405、488或594结合的邻苯二甲苯素被染色。(C, D)目标图像之前 (C) 和之后 (D) 对齐。HeLa细胞被染色DAPI的DNA(在洋红色中显示),Alexa Fluor 488(以黄色显示)用于核包,Alexa Fluor 594(以蓝色显示)用于微管。面板 A 中的刻度条表示所有面板的 5 μm。请点击此处查看此图形的较大版本。
讨论
色谱校正过程是精度和工作量之间的权衡。为了节省不必要的努力,最好知道学习需要多少准确性。传统的宽场(实时)成像可能不需要最高的精度,因此,明亮的场参考图像通常足以校正色移。同样,当成像条件和环境恒定时,反复使用生物校准将节省时间。另一方面,如果需要高精度的注册,则需要高质量的串扰或生物校准参考图像。为了获得最佳性能,应尽可能使用与目标图像类似的条件和计时获得参考图像。只要参考图像和目标图像都通过同一显微镜获得,较高的空间分辨率就会提高校正精度。如果可同时用于参考图像和目标图像的 deconvol;如果可在校正之前实现此值,从而提高校正精度。此外,为了获得最佳性能,应在参考和目标文件中实现光学 (Z) 轴的采样定理,以便精确进行子像素插值(协议步骤 2.1.3)。
未能纠正色差导致错误的结论。此外,使用错误的校准甚至会使色变恶化,而不是纠正它们,因此需要避免这种情况。我们在表2中总结了失败的可能原因及其共同解决办法。为了检查故障的原因,首先,必须直观地检查参考图像中的色移是否得到精确校正(协议步骤 3.12)。大多数故障是由于参考图像的质量,并且根据表 2中的说明很容易补救。关于参考图像的质量,请务必注意,如果整个视场没有填充样本,则全局对齐的准确性会降低(图7,表2)。与图 7A中所示的好示例相比,图 7B中所示的坏示例在左上角区域中仅包含三个核包络,Chromagnon未能对齐此图像的一部分。这是因为Chromagnon的全局对齐方法将视场分为四个区域(图7C),以便高精度3测量旋转和放大度的差异。如果操作正确,此方法比其他线性方法(如日志极性变换和简单方法3)更准确一个阶。如果四个区域中的任何一个不可用,则Chromagnon将切换到效果较差的线性方法。因此,为了获得最佳性能,图 7B和图 7C中所示的示例是不可取的,并且四个区域应填充对象。用户可以通过查看日志文件("Chromagnon.log";请参阅协议步骤 3.10)来检查视图场的任何二次区域是否不可用进行测量。幸运的是,通过平均多个生物校准图像或使用串扰或明亮场参考图像的局部对齐,这个问题很容易克服(表2)。与无法更正参考图像的情况相反,无法更正目标图像更难识别。由于文件格式、成像条件、成像时间、参考图像和目标图像之间的成像/对齐方法不同(表 2),因此出现此类故障,因此用户在使用从目标图像的不同条件下/计时获得的参考图像时应始终小心谨慎。一些示例图像可用于测试 (https://github.com/macronucleus/Chromagnon), 以获得好和坏示例图像的具体想法.
| 问题 | 原因 | 解决 方案 |
| 无法更正参考图像 | 低对比度 | 如果可能,获取更高的对比度图像。如果使用亮场参考图像,则在水性溶液中重新获取图像以获得更高的细胞对比度。或者,尝试应用计算降噪(例如高斯滤波)。关闭对噪声更敏感的局部对齐。 |
| 不相关图像的污染 | 如果可能,请删除示例中不相关的图像源。对于串扰参考图像,请检查用于目标图像的染料的激发光谱。如果染料在获取相声图像(例如 Alexa Fluor 568 或 594)时兴奋,请考虑其他染料(例如 Alexa Fluor 555)。如果相机芯片上的灰尘产生明显的通道差,请清洁摄像机芯片或使用计算平面法。 | |
| 由宇宙射线构成的极亮点 | 如果可能,再次获取映像。或者,尝试应用计算降噪(例如中位或高斯滤波)。 | |
| 去挥发伪影(轴向和横向边缘的人工信号) | 在去卷后修剪边缘像素或 Z 部分。如果修剪了一侧,则另一侧也应修剪以保持图像中心。 | |
| Z 步长大小太稀疏 | 应获取 Z 堆栈以满足协议 2.1.3 中写入的奈奎斯特标准。 | |
| 光学畸变 | 球形畸变是由用户引起的主要畸变。为样品选择正确的目标镜头,并使用 170 μm 的盖玻片厚度。如果目标透镜配有校正环,请调整它以查找从焦点获得最高荧光计数的位置。在没有校正环的浸油目标的情况下,调整浸入式油的折射率,增加焦点处的荧光计数。 | |
| 未填充视野(图 7) | 在生物校准参考图像的情况下,平均许多图像。对于串扰或亮场参考图像,请使用局部对齐。 | |
| 一个未识别的软件错误 | 通过GitHub(https://github.com/macronucleus/Chromagnon/issues)报告问题 | |
| 无法更正目标图像 | 图像文件的元数据丢失 | 使用包含完整元数据的原始显微镜文件格式,避免在处理之前转换为多页 tff 文件。使用协议 3.3 中写入的通道顺序。 |
| 给定显微镜的对齐方法错误 | 当从生物校准参考图像测量到目标图像时,请勿应用局部对齐方法。请勿使用宽场显微镜以外的串扰参考图像。 | |
| 成像条件的差异 | 保持引用和目标图像之间的成像条件不变,如协议 2.3.3 中写入的。 | |
| 样品差异(包括盖玻片) | 始终使用相同的安装介质、盖玻片(例如 1.5H 号)和类似的对焦深度。 | |
| 自上次校准以来的显微镜漂移 | 每隔两周进行一次校准。保持温度恒定,并使用浮动表以避免显微镜的硬件漂移。 |
表 2:用于色谱校正的故障排除。
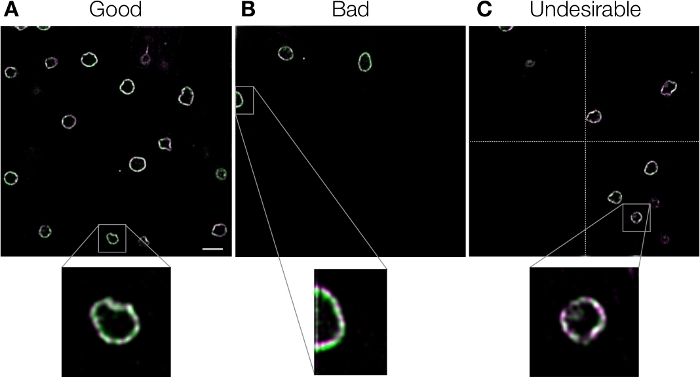
图 7:参考图像的示例。裂变酵母细胞中的核包络,标有GFP和mCherry。图像是用传统的广域显微镜获得的。使用Chromagnon校正色变,无需局部对齐,使用图像本身作为参考图像。然后,图像被解说,以显示细节。(A) 视野中有许多对象的好例子。(B) 一个坏例子,对象只位于左上角。在图像的特定区域,错位是显而易见的。(C) 四节之一(由虚线分隔)为空的不良示例。面板 A 中的刻度条表示整个现场视图为 5 μm,对于放大视图指示 1.25 μm,适用于所有面板。请点击此处查看此图形的较大版本。
在此协议中,我们描述了三种不同的引用类型 (表 1)。其中,相声参考图像和生物校准参考图像需要进一步认真讨论。对于串扰参考图像,可有效使用带有 DAPI 或 Hoechst 33342 的样品,并安装在甘油或商用安装介质中,以对齐蓝色、绿色和红色通道。同样,Alexa Fluor 488 可用于对齐绿色和红色通道。然而,获得相声荧光通常是困难的,因为除了DAPI和Hoechst之外,许多蓝色染料比大多数绿色和红色染料更暗和衰变更快。此外,现代染料的发射光谱较窄,因此采用这种方法对三个以上通道的对齐具有挑战性。还应注意一些常见的红色染料(例如,Alexa Flour 568 和 594,但不是 Alexa Fluor 555),这些染料可以因紫光而兴奋,从而阻止从蓝色染料获得高对比度的串扰图像。另一个缺点是,此方法无法测量多色激发中激励光路径的色差,因为激发只使用单个激励波长(表 1)。由于最先进的显微镜使用改变的照明光学元件,此方法的应用是有限的。尽管如此,其较高的校正精度足以使其在该协议中加以描述。通常,在目标图像后应拍摄串扰图像,以防止漂白或光毒性影响。对于使用宽场模式观察的 SMLM,在获取目标图像之前应获取参考图像,因为在成像时可以漂白荧光染料。
生物校准参考图像允许用户轻松对齐任何所需数量的通道,但代价是额外的样品制备。生物校准参考图像的另一个优点是"平均"多个参考,有助于填充所有视场。如果校准样品是在不同的幻灯片上制备的,则此方法可能会因成像条件不同而受到影响。如果使用商用室盖镜(表 1)在同一张幻灯片上制备目标和参考板,并且其他成像条件保持不变,如协议步骤 2.3.3 中那样,大多数问题都可以解决。在这种情况下,可预期与串扰参考图像类似的校正精度为 3。使用邻苯二甲二甲二甲酮的协议,如此处所示,是用多种颜色染色单个细胞结构的最简单方法之一。准备生物校准样品可能有很多种方案。对于免疫染色,样品可以标记与单个原抗体,然后染色与多种颜色的二次抗体。这样,单个目标结构就可以用多种颜色进行标记。另外,5-ethynyl-2'-脱氧核酸,通过"咔嗒"化学标签检测出,在高密度下以多种颜色新合成的DNA,如前8所述。对于活细胞,制备一种含有两个基因副本的转基因菌株非常有用,该基因与GFP或mCherry融合在一起,用两种颜色标记同一结构。如果基因的拷贝数量与膜蛋白的通常观察量一样至关重要,则基因的单个副本可以串联到GFP和mCherry(图7)。光可转换荧光蛋白,如mEOS218,也可以通过照亮适量的紫光来获得两种蛋白质,无论是否进行光转换。在低氧条件下,GFP也可以用作从绿色到红色19,20,20的光能蛋白。因此,选择正确的校准样品将使实验更加健壮。
披露声明
作者没有什么可透露的。
致谢
这项研究得到了JSPS KAKENHI赠款号码JP19H03202到A.M.的支持, JP18H05528和JP17H03636至T.H.,JP17H01444和JP18H05533对H.Y.L.S.感谢欢迎信托战略奖091911和107457/Z/15/Z在Micron牛津提供高级成像的支持。
材料
| Name | Company | Catalog Number | Comments |
| 16% formaldehyde solution | Polyscience | 18814-10 | |
| 35 mm glass-bottom dish | MatTek | P35G-1.5-10-C | |
| Alexa Fluor 405 phalloidin | Thermo Fisher Scientific | A30104 | |
| Alexa Fluor 488 phalloidin | Thermo Fisher Scientific | A12379 | |
| Alexa Fluor 594 phalloidin | Thermo Fisher Scientific | A12381 | |
| Bovine Serum | Thermo Fisher Scientific | 16170078 | |
| Coverslip | Matsunami | No. 1S HT | |
| DAPI (4',6-Diamidino-2-Phenylindole, Dihydrochloride) | Thermo Fisher Scientific | D1306 | |
| Dulbecco’s Modified Eagle Medium with L-Gln and sodium pyruvate | Nacalai Tesque | 08458-16 | |
| Mounting medium (VECTASHIELD) | Vector Laboratories | H-1000 | |
| Mouse anti-tubulin monoclonal antibody (TAT1) | Described in Ref 15. | ||
| Nunc Lab-Tek II chambered coverglass (8 well) | Thermo Fisher Scientific | 155409 | |
| Rabbit anti-emerin polyclonal antibody (ED1) | A gift from Hiroshi Yorifuji, Gunma University, Gunma, Japan and Kiichi Arahata, National Center of Neurology and Psychiatry, Tokyo, Japan; deceased. | ||
| Secondary antibody with Alexa Fluor 488 | Thermo Fisher Scientific | A-11034 | |
| Secondary antibody with Alexa Fluor 555 | Thermo Fisher Scientific | A-21424 | |
| Secondary antibody with Alexa Fluor 594 | Thermo Fisher Scientific | A-11032 | |
| TetraSpeck Microspheres, 0.2 µm | Thermo Fisher Scientific | T7280 |
参考文献
- Schermelleh, L., et al. Super-resolution microscopy demystified. Nature Cell Biology. 21 (1), 72-84 (2019).
- Manders, E. M. M. Chromatic shift in multicolour confocal microscopy. Journal of Microscopy. 185 (3), 321-328 (1997).
- Matsuda, A., Schermelleh, L., Hirano, Y., Haraguchi, T., Hiraoka, Y. Accurate and fiducial-marker-free correction for three-dimensional chromatic shift in biological fluorescence microscopy. Scientific Reports. 8 (1), 7583 (2018).
- Grünwald, D., Singer, R. H. In vivo imaging of labelled endogenous β-actin mRNA during nucleocytoplasmic transport. Nature. 467 (7315), 604-607 (2010).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Chen, F., Tillberg, P. W., Boyden, E. S. Expansion microscopy. Science. 347 (6221), 543-548 (2015).
- Kraus, F., et al. Quantitative 3D structured illumination microscopy of nuclear structures. Nature Protocols. 2, 1011-1028 (2017).
- Hell, S. W. Far-Field Optical Nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. Journal of Microscopy. 198 (2), 82-87 (2000).
- Schulz, O., et al. Resolution doubling in fluorescence microscopy with confocal spinning-disk image scanning microscopy. Proceedings of the National Academy of Sciences of United States of America. 110 (52), 21000-21005 (2013).
- Müller, C. B., Enderlein, J. Image Scanning Microscopy. Physical Review Letters. 104 (19), 198101 (1981).
- Hayashi, S., Okada, Y. Ultrafast superresolution fluorescence imaging with spinning disk confocal microscope optics. Molecular Biology of the Cell. 26 (9), 1743-1751 (2015).
- Yorifuji, H., et al. Emerin, deficiency of which causes Emery-Dreifuss muscular dystrophy, is localized at the inner nuclear membrane. Neurogenetics. 1 (2), 135-140 (1997).
- Woods, A., Sherwin, T., Sasse, R., MacRae, T. H., Baines, A. J., Gull, K. Definition of individual components within the cytoskeleton of Trypanosoma brucei by a library of monoclonal antibodies. Journal of Cell Science. 93 (3), 491-500 (1989).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells. Nature Methods. 5 (2), 159-161 (2008).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529 (2017).
- McKinney, S. A., Murphy, C. S., Hazelwood, K. L., Davidson, M. W., Looger, L. L. A bright and photostable photoconvertible fluorescent protein for fusion tags. Nature Methods. 6 (2), 131-133 (2009).
- Sawin, K. E., Nurse, P. Photoactivation of green fluorescent protein. Current Biology. 7 (10), 606-607 (1997).
- Elowitz, M. B., Surette, M. G., Wolf, P. E., Stock, J., Leibler, S. Photoactivation turns green fluorescent protein red. Current Biology. 7 (10), 809-812 (1997).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。