Analisi di popolazione e monocellulare della persistenza antibiotica in Escherichia coli
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
La persistenza degli antibiotici descrive la capacità di piccole sottopopolazioni all'interno di una popolazione isogena sensibile di tollerare transitoriamente alte dosi di antibiotici battericidi. Il presente protocollo combina approcci per caratterizzare il fenotipo di persistenza antibiotica a livello molecolare e cellulare dopo aver esposto Escherichia coli a dosi letali di ofloxacina.
Abstract
La persistenza antibiotica si riferisce alla capacità di piccole sottopopolazioni batteriche di tollerare transitoriamente alte dosi di antibiotici battericidi. Dopo il trattamento antibiotico battericida, la maggior parte della popolazione batterica viene rapidamente uccisa. Questa prima fase rapida di uccisione è seguita da una sostanziale diminuzione del tasso di uccisione man mano che le cellule persistenti rimangono vitali. Classicamente, la persistenza è determinata a livello di popolazione da saggi time/kill eseguiti con alte dosi di antibiotici e per tempi di esposizione definiti. Mentre questo metodo fornisce informazioni sul livello delle cellule persistenti e sulla cinetica di uccisione, non riesce a riflettere l'eterogeneità intrinseca cellula-cellula alla base del fenomeno di persistenza. Il protocollo qui descritto combina i classici saggi time/kill con l'analisi a singola cellula utilizzando la microscopia a fluorescenza in tempo reale. Utilizzando appropriati reporter fluorescenti, l'imaging al microscopio delle cellule vive può fornire informazioni sugli effetti dell'antibiotico sui processi cellulari, come la replicazione e la segregazione cromosomica, l'allungamento cellulare e la divisione cellulare. La combinazione dell'analisi della popolazione e di una singola cellula consente la caratterizzazione molecolare e cellulare del fenotipo di persistenza.
Introduzione
Questo protocollo mira ad analizzare il fenotipo di persistenza batterica in risposta a specifici trattamenti antibiotici a livello di singola cellula e di popolazione. La persistenza descrive la capacità di piccole sottopopolazioni all'interno di una popolazione isogena di sopportare alte dosi di antibiotici battericidi (fluorochinoloni, aminoglicosidi, β-lattamici, ecc.), con la concentrazione inibitoria minima (MIC) delle cosiddette cellule persistenti identica a quella della maggior parte della popolazione. Le dinamiche di uccisione bifasica, quando misurano la sopravvivenza batterica nel tempo in presenza di un antibiotico, rivelano la presenza di cellule transitoriamente tolleranti ai farmaci, con una rapida eradicazione iniziale delle cellule non persistenti, seguita da un tasso di uccisione molto più lento delle cellule persistenti. Dopo la rimozione dell'antibiotico, queste cellule danno origine a una popolazione geneticamente identica che mostra dinamiche di uccisione simili quando trattata con lo stesso antibiotico 1,2. In contrasto con la persistenza, la resistenza agli antibiotici è definita a livello di popolazione ed è generalmente una conseguenza di mutazioni de novo o del trasferimento genico orizzontale di un plasmide 3 che conferisceresistenza. Mentre le mutazioni responsabili della resistenza sono per lo più localizzate nel bersaglio del farmaco o nelle regioni promotorie delle pompe di efflusso del farmaco, i geni che alterano la frequenza di persistenza identificati dagli approcci di analisi dei mutanti a livello di genoma e mirati si sono dimostrati numerosi e diversi 2,3,4,5,6,7,8 . Pertanto, è probabile che le cellule batteriche possano entrare nello stato di persistenza attraverso percorsi multipli 9,10,11, e sono necessari approcci per studiare il fenomeno di persistenza a livello di singola cellula per caratterizzare la fisiologia di queste cellule persistenti.
Il recente sviluppo di strumenti microfluidici utilizzati in combinazione con la microscopia a fluorescenza ha aperto la strada alla caratterizzazione del fenotipo di persistenza e ha evidenziato il ruolo dei processi cellulari chiave, come la replicazione cromosomica12, la riparazione del DNA 13 e la divisione cellulare14, nella formazione delle cellule persistenti. In questo articolo, descriviamo un approccio integrato che combina saggi di microbiologia classici con imaging live a singola cellula per caratterizzare le cellule persistenti generate in colture di Escherichia coli in crescita esponenziale trattate con un'alta dose di ofloxacina. Il protocollo qui descritto può essere applicato per studiare il fenomeno della persistenza antibiotica in altre specie batteriche, come il Bacillus subtilis15, o condizioni (ad esempio, persistenza antibiotica dopo trattamento con β-lattamici 16) e può essere facilmente modificato per indagare i numerosi fenomeni che coinvolgono l'eterogeneità fenotipica17,18,19 . Inoltre, la configurazione descritta in questo articolo può essere combinata con altri reporter fluorescenti per studiare parametri cellulari distinti di interesse, come i livelli intracellulari di pH20 o ATP21 a livello di singola cellula, che possono potenzialmente produrre nuove intuizioni sul fenomeno della persistenza antibiotica.
Protocollo
NOTA: Utilizzare vetreria di coltura sterile, punte per pipette e terreno di coltura. Qui, le cellule di E. coli sono state coltivate in un mezzo a bassa autofluorescenza chimicamente definito (vedi Tabella dei materiali). Le inoculazioni sono state eseguite in presenza di un bruciatore Bunsen per ridurre al minimo il rischio di contaminazione.
1. Coltura cellulare e curva di crescita
- Striare il ceppo di interesse da uno stock di glicerolo congelato su una piastra di agar Luria-Bertaini (LB) (integrata con un antibiotico selettivo, se necessario) e incubare a 37 °C durante la notte (tra 15 h e 19 h) per ottenere singole colonie.
NOTA: L'esperimento qui presentato utilizza due ceppi, E. coli K-12 MG1655 corrispondente al ceppo wt e il ceppo isogenico MG1655 hupA-mCherry 22. Quest'ultimo ceppo esprime la α-subunità marcata con fluorescenza della proteina associata al nucleoide HU. Il reporter hupA-mCherry è integrato nel luogo nativo di hupA. HU-mCherry funge da proxy per seguire le dinamiche nucleoidi nelle cellule vive mentre si lega al DNA in modo non specifico. - Inoculare 5 mL di terreno (qui, 3-[N-morfolino] terreno a base di acido propanosolfonico [MOPS]; Tabella 1, Tabella 2 e Tabella 3) integrata con glucosio 0,4% e un antibiotico selettivo (se necessario) con una colonia isolata in un tubo di vetro (≥25 ml) e porre il tubo in un incubatore agitatore impostato a 37 °C e 180 rotazioni al minuto (rpm) durante la notte (tra 15 h e 19 h). In alternativa, è possibile utilizzare tubi di plastica, vetro o fiaschi di plastica (≥ 25 ml) al posto dei tubi di vetro.
NOTA: MOPS mezzo integrato con glicerolo ad una concentrazione finale dello 0,4% è stato utilizzato in tutti gli esperimenti descritti in questo documento, ad eccezione delle colture notturne che sono state eseguite in MOPS glucosio 0,4%. Il tempo di generazione di E. coli in MOPS integrato con glucosio è più breve di quello in MOPS glicerolo. L'utilizzo di glucosio MOPS 0,4% invece di glicerolo MOPS 0,4% in questa fase assicura che le cellule raggiungano la fase stazionaria entro 19 ore. Possono essere utilizzati anche altri substrati di crescita, come M9 o rich defined medium (RDM) integrati con fonti di carbonio distinte. Va tuttavia osservato che il tasso di crescita e la frequenza di persistenza sono diversi a seconda del mezzo e/o del carbonio utilizzato23. - La mattina seguente, centrifugare 1 mL di coltura a 2.300 x g per 3 minuti, scartare il surnatante e risospendere delicatamente il pellet nello stesso volume di soluzione salina tamponata fosfato (PBS). Misurare la densità ottica a 600 nm (OD 600 nm) e calcolare il volume necessario per un OD iniziale di600 nm di 0,01 in un volume finaledi 2 mL.
- Mettere 2 ml di terreno MOPS glicerolo 0,4% in un pozzetto di una piastra trasparente inferiore a 24 pozzetti e inoculare con il volume di coltura notturno calcolato. Posizionare la piastra a 24 pozzetti in un lettore automatico di micropiastre (vedere Tabella dei materiali) per monitorare l'OD600 nm per 24 ore. Impostare il lettore di micropiastre per misurare l'OD600 nm ogni 15 minuti ad una temperatura di 37 °C e con un'elevata rotazione orbitale (140 rpm).
NOTA: Se il ceppo utilizzato nell'esperimento codifica un reporter fluorescente, assicurarsi che il suo tasso di crescita sia paragonabile a quello del wt per evitare qualsiasi artefatto negli esperimenti successivi, poiché il tasso di crescita influisce sulla frequenza di persistenza dell'antibiotico23. A causa delle limitazioni di rivelazione a densità cellulari molto basse e dei potenziali effetti specifici del ceppo sulla relazione OD600 nm/unità formanti colonie (CFU), si raccomanda di valutare la cinetica di crescita monitorando il CFU·mL-1 quando si lavora con ceppi non caratterizzati.
2. Determinazione della concentrazione minima inibitoria degli antibiotici
NOTA: La concentrazione minima inibitoria (MIC) è definita come la dose più bassa di antibiotico alla quale non si osserva crescita batterica. La determinazione della MIC deve essere eseguita per ciascun antibiotico e ceppo. Negli esperimenti qui descritti, è stato utilizzato l'antibiotico fluorochinolone ofloxacina (OFX). La determinazione del MIC consente di confermare che la soluzione antibiotica è stata preparata correttamente, che l'antibiotico è attivo e che i ceppi sono ugualmente sensibili all'antibiotico. Qui, il metodo di diluizione dell'agar pubblicato è stato eseguito per determinare il MIC in OFX dei diversi ceppi utilizzati24. Il MIC di un dato antibiotico per un dato ceppo batterico può anche essere determinato tramite il metodo di diluizione del brodo24.
- Preparazione delle piastre per la determinazione del MIC
- Preparare una soluzione madre per l'antibiotico utilizzato negli esperimenti sciogliendo 5 mg di OFX in 1 ml di acqua ultrapura. Aggiungere 20 μL di HCl al 37% per aumentare la solubilità dell'OFX.
- Fondere 100 mL di agar LB sterile e mantenerlo a 55 °C per evitare la solidificazione. Preparare sei piccoli matraccio di vetro (25 ml) e inpipettare 5 mL di mezzo agar LB liquido in ciascun matraccio utilizzando una pipetta sterile.
- Diluire 10 μL della soluzione madre di 5 mg·mL-1 OFX in 90 μL di acqua ultrapura (diluizione 1:10 del brodo madre). Aggiungere 0 μL, 2 μL, 4 μL, 6 μL, 8 μL o 10 μL della soluzione di 500 μg·mL-1 OFX a ciascuno dei sei matramenti di vetro contenenti 5 mL di terreno di agar LB per generare terreno agar LB con concentrazioni finali di 0 μg·mL−1, 0,02 μg·mL−1, 0,04 μg·mL−1, 0,06 μg·mL−1, 0,08 μg·mL−1 e 0,1 μg·mL-1 OFX, rispettivamente. Mescolare la soluzione ruotando più volte i palloni.
NOTA: Se il MIC dell'antibiotico di interesse è noto, l'intervallo di concentrazioni dovrebbe raggiungere dal basso al di sopra del MIC effettivo. Se la MIC non è nota, si raccomanda un ampio intervallo di concentrazioni con una serie di diluizioni log2 . Assicurarsi che il mezzo agar LB si sia raffreddato prima di aggiungere l'antibiotico, poiché le alte temperature potrebbero inattivarlo. Tuttavia, è importante non lasciare che il terreno di agar LB si solidifichi prima di aggiungere l'antibiotico, poiché ciò potrebbe portare alla distribuzione non omogenea dell'antibiotico nel mezzo di agar LB. - Versare 5 mL di ciascuno dei sei mezzi agar LB preparati al punto 2.1.3 in modo dose-crescente in una piastra di coltura a 6 pozzetti utilizzando una pipetta sterile. Lasciare raffreddare l'agar fino alla solidificazione e asciugare la piastra prima dell'uso.
NOTA: Preparare la soluzione antibiotica e la piastra di coltura il giorno in cui viene eseguito il test.
- Saggio di determinazione MIC
- Inoculare 5 mL di terreno LB con una colonia isolata in un tubo di vetro (≥25 mL) e porre il tubo di vetro in un incubatore agitatore impostato a 37 °C e 180 giri/min durante la notte (tra 15 h e 19 h).
- La mattina seguente, misurare l'OD600 nm e diluire la coltura in una provetta fino ad una densità cellulare finale di 1 x 107 CFU·mL-1 in PBS. Per i ceppi qui testati, 1 x 107 CFU·mL−1 corrisponde a un OD600 nm di 0,0125.
NOTA: Se la correlazione tra CFU·mL-1 e OD 600 nm non è nota, la curva di crescita determinata dalle misurazioni di CFU e OD 600 nm deve essere stabilita per calcolare il fattore di correlazione tra CFU·mL-1 e OD600 nm. - Spot 2 μL della coltura precedentemente diluita su ogni pozzetto della piastra essiccata a 6 pozzetti. Lasciare asciugare le macchie prima di mettere la piastra in un'incubatrice a 37 °C durante la notte (tra 15 h e 19 h).
- Il giorno dopo, conta le colonie formate in ogni pozzo. Il MIC corrisponde al pozzetto con la concentrazione minima di antibiotico dove non viene rilevata alcuna crescita batterica.
3. Saggio a campione
NOTA: Il metodo del saggio spot è un approccio qualitativo che consente la stima del numero di cellule vitali (cellule in grado di generare colonie dopo stress antibiotico). Il test spot viene eseguito prima del test time-kill per fornire informazioni sulla fattibilità del ceppo utilizzato nelle condizioni testate e per informare sulle diluizioni necessarie durante il test time-kill (vedere paragrafo 4).
- Per preparare le piastre di agar LB per i saggi spot, versare 50 mL di agar LB in una capsula di Petri quadrata (144 cm2). Preparare una capsula di Petri quadrata per punto temporale. Lasciare solidificare l'agar LB e asciugare le piastre prima dell'uso.
- Inoculare 5 mL di terreno (glucosio MOPS 0,4%) con una colonia isolata in un tubo di vetro (≥25 mL) e porre il tubo in un incubatore agitatore impostato a 37 °C e 180 rpm durante la notte (tra 15 h e 19 h).
- Il giorno successivo, misurare l'OD 600 nm e diluire la coltura in un mezzo fresco regolato in temperatura (37 °C, MOPS glicerolo 0,4%) in un tubo di vetro (≥25 ml) a un OD finale600 nm di ~0,001 . Lasciare crescere la coltura durante la notte in un'incubatrice a 37 °C a 180 giri/min (tra 15 h e 19 h).
- Il giorno seguente, misurare l'OD 600 nm e incubare la coltura a un OD finale di600 nm di 0,3.
- Durante l'incubazione, preparare piastre a 96 pozzetti ponendo 90 μL di soluzione di 0,01 M MgSO4 in ogni pozzetto tranne i pozzetti della prima fila (fila A). Le piastre a 96 pozzetti saranno utilizzate per la diluizione seriale di 10 volte nella fase 3.7.
NOTA: In questo esperimento, due ceppi (ceppo wt e hupA-mCherry ) sono stati testati in triplice copia in sette diversi punti temporali. Poiché una piastra comprende 12 colonne, una piastra può essere utilizzata per due punti temporali e, quindi, sono state preparate un totale di quattro piastre. - A un OD 600 nm di 0,3, prelevare200 μL di ciascuna coltura batterica. Questi campioni corrispondono al timepoint t0 (non trattato) prima del trattamento antibiotico e consentono la determinazione del CFU·mL-1 prima del trattamento antibiotico.
- Centrifugare i campioni a 2.300 x g per 3 minuti. Durante il tempo di centrifugazione, aggiungere la concentrazione desiderata di OFX alla coltura liquida e continuare a incubare a 37 °C agitando.
NOTA: Qui, OFX è stato utilizzato ad una concentrazione di 5 μg·mL-1 (corrispondente al MIC moltiplicato 83 volte). In uno studio precedente, questa concentrazione è stata utilizzata per caratterizzare il fenomeno di persistenza sotto esposizione OFX25. La concentrazione di antibiotico utilizzata per i test time/kill può variare a seconda dell'antibiotico, del terreno di coltura e dello stato di crescita batterica. - Dopo centrifugazione, risospendere il pellet cellulare in 200 μL di soluzione 0,01 M MgSO4 . Porre 100 μL nel pozzetto vuoto della piastra da 96 pozzetti preparata al punto 3.5. Eseguire una diluizione trasferendo 10 μL del pozzetto nella fila A nel pozzetto della fila B contenente 90 μL di 0,01 M MgSO4. Continuare le diluizioni seriali trasferendo 10 μL del pozzetto nella fila B nel pozzetto nella fila C. Ripetere fino a raggiungere una diluizione di 10−7 (con ogni trasferimento di diluizione di 10 volte).
NOTA: In questo esperimento, sei colture (tre per il ceppo wt e tre per il ceppo hupA-mCherry ) sono state testate per ogni timepoint. Secondo il protocollo, una pipetta multicanale viene utilizzata per eseguire contemporaneamente le diluizioni seriali per i sei ceppi. Per ridurre al minimo l'errore tecnico, le punte delle pipette vengono cambiate tra un trasferimento e l'altro. - Puntare 10 μL di ciascuna diluizione sulle piastre di agar LB preparate al punto 3.1.
NOTA: Una pipetta multicanale viene utilizzata per individuare simultaneamente la stessa diluizione (ad esempio, una diluizione 10−4 ) per le sei colture. Durante lo spotting, il primo stop della pipetta multicanale deve essere utilizzato senza spingere il secondo stop, in quanto ciò può comportare l'erogazione di microgoccioline sulla piastra. - Nei momenti rilevanti dopo l'aggiunta di antibiotico, prelevare 200 μL della coltura ed eseguire diluizioni seriali come descritto al punto 3.8. Spot 10 μL di ciascuna diluizione come descritto al punto 3.9.
NOTA: Per l'esperimento qui descritto, sono stati raccolti sette punti temporali (t0, t1h, t2h, t3h, t4h, t5h, t6h). Per i ceppi che presentano una maggiore suscettibilità agli antibiotici rispetto al wt, possono essere eseguiti lavaggi multipli in MgSO4 0,01 M per rimuovere gli antibiotici residui. - Incubare le piastre a 37 °C durante la notte (tra 15 h e 19 h). Il giorno successivo, conta il numero di colonie alle due diluizioni più alte per le quali è possibile rilevare colonie. Idealmente, le macchie sulle piastre di agar per queste diluizioni contengono da 3 a 30 colonie che consentono la determinazione accurata del CFU·mL-1 per ciascun campione.
- Calcolare il rapporto di sopravvivenza dividendo il CFU·mL-1 calcolato di ciascun timepoint per il CFU·mL-1 della popolazione iniziale a t0.
4. Saggi time-kill
NOTA: Mentre i saggi spot sono un metodo facile da usare per stimare il tasso di sopravvivenza di un dato ceppo per un determinato antibiotico, i saggi time-kill danno un tasso di sopravvivenza ad alta risoluzione e vengono eseguiti per quantificare con precisione la vitalità batterica. Il profilo della curva di uccisione può essere utilizzato per determinare se un determinato ceppo batterico è sensibile, tollerante o resistente all'antibiotico in una determinata condizione. Inoltre, i saggi time-kill consentono di determinare il tempo di esposizione agli antibiotici necessario per rilevare il fenomeno di persistenza (inizio della seconda pendenza della curva di uccisione bifasica) e la frequenza di persistenza.
- Preparare le piastre di agar LB per il test di placcatura time-kill. Versare 25 mL di agar LB in una capsula di Petri (±57 cm2). Preparare almeno due piastre di Petri per timepoint (vengono placcate due diluizioni per timepoint per ceppo).
- Lasciare solidificare l'agar LB e asciugare le piastre prima di aggiungere da cinque a otto perle di vetro sterili a ciascuna piastra. Capovolgere ed etichettare le piastre in base alla deformazione/condizione/timepoint.
NOTA: Le sfere di vetro consentono la diffusione di batteri sulle piastre di agar durante il passaggio 4.7 e il passaggio 4.9. In alternativa, le cellule possono essere disperse sulla piastra di agar usando uno spargitore. - Per ciascun campione, preparare tubi di vetro di diluizione seriale 10 volte contenenti 900 μL di soluzione di 0,01 M MgSO4 . Il numero di tubi di vetro di diluizione per campione che deve essere preparato corrisponde alle diluizioni necessarie per rilevare le colonie nel saggio spot.
- Inoculare 5 mL di terreno (glucosio MOPS 0,4%) con una colonia isolata in un tubo di vetro (≥25 mL) e porre il tubo in un incubatore agitatore impostato a 37 °C e 180 rpm durante la notte (tra 15 h e 19 h).
- Dopo 16 ore di crescita, misurare l'OD 600 nm e diluire la coltura in terreno fresco regolato in temperatura (37 °C, MOPS glicerolo 0,4%) in un tubo di vetro fino a un OD finale600 nm di ~0,001 . Lasciare crescere la coltura durante la notte in un'incubatrice a 37 °C a 180 giri/min (tra 15 h e 19 h).
- Il giorno seguente, misurare l'OD 600 nm e incubare la coltura a un OD finale di600 nm di 0,3.
- A un OD 600 nm di 0,3, prelevare 100 μL della coltura, diluire secondo i dati ottenuti nel saggio spot (a un OD 600 nm di 0,3 e senza antibiotico, una diluizione 10−5 dà solitamente 200-300 colonie) e piastra100 μL sulle piastre di agar LB preparate nei punti 4.1-4.2. Agitare delicatamente le piastre per evitare che le perle entrino in contatto con i bordi del piatto, poiché ciò può portare a una diffusione non omogenea delle cellule batteriche sul mezzo di agar LB. Questo primo campione prima del trattamento antibiotico corrisponde al punto temporale t0 (CFU·mL-1 prima del trattamento antibiotico).
- Aggiungere la concentrazione desiderata di OFX e continuare ad incubare a 37 °C agitando.
NOTA: Qui, OFX è stato utilizzato ad una concentrazione di 5 μg·mL-1 (corrispondente al MIC moltiplicato 83 volte). - Nei momenti rilevanti dopo l'aggiunta di antibiotico, prelevare 100 μL della coltura, diluire in base ai dati ottenuti nel saggio spot e placcare 100 μL sulle piastre di agar LB preparate nei punti 4.1-4.2. Placcare le celle come descritto al punto 4.7.
NOTA: Per l'esperimento qui descritto, sono stati raccolti sette punti temporali (t0, t1h, t2h, t3h, t4h, t5h, t6h). Per i ceppi che presentano una maggiore suscettibilità agli antibiotici rispetto alla wt, possono essere eseguiti lavaggi multipli in MgSO4 0,01 M per rimuovere gli antibiotici residui. - Incubare le piastre a 37 °C durante la notte (tra 15 h e 19 h). Il giorno successivo, conta il numero di colonie alle due diluizioni più alte per le quali è possibile rilevare colonie. Idealmente, le piastre dovrebbero contenere 30-300 colonie per consentire una determinazione accurata del CFU·mL-1 in ciascun campione.
- Calcolare il rapporto di sopravvivenza normalizzando il CFU·mL-1 ad ogni time-point per il CFU·mL-1 a t0. Tracciare il log10 normalizzato CFU·mL-1 in funzione del tempo.
5. Microscopia microfluidica time-lapse
NOTA: La sezione seguente descrive la preparazione della piastra microfluidica e la procedura di acquisizione e analisi delle immagini time-lapse. Lo scopo di questo esperimento è quello di osservare e analizzare il fenotipo di persistenza dopo il trattamento antibiotico a livello di singola cellula. I dati raccolti durante questo esperimento possono essere utilizzati per generare una vasta gamma di risultati a seconda della domanda affrontata e / o dei reporter fluorescenti utilizzati durante l'esperimento. Nell'esperimento qui descritto, è stata effettuata un'analisi quantitativa della lunghezza cellulare e della fluorescenza HU-mCherry22, che riflette l'organizzazione nucleoidale nelle cellule persistenti e non persistenti.
- Coltura cellulare batterica per microscopia time-lapse microfluidica
- Inoculare 5 mL di terreno (MOPS glicerolo 0,4%, integrato con un antibiotico selettivo se necessario) con una colonia isolata in un tubo di vetro (≥25 mL) e porre il tubo in un incubatore agitatore impostato a 37 °C e 180 rpm durante la notte (tra 15 h e 19 h).
- Il giorno successivo, misurare l'OD 600 nm e diluire la coltura in terreno fresco regolato in temperatura (37 °C, glicerolo MOPS 0,4%) in un tubo di vetro fino a un OD finale di600 nm di ~ 0,001. Lasciare crescere la coltura durante la notte (tra 15 h e 19 h) in un incubatore vibrante a 37 °C e 180 rpm per ottenere una coltura in fase esponenziale precoce il giorno successivo.
- Preparazione della piastra microfluidica e microscopia time-lapse
NOTA: Gli esperimenti microfluidici possono essere eseguiti in dispositivi microfluidici disponibili in commercio (come descritto qui) o in sistemi microfluidici prodotti internamente.- Rimuovere la soluzione di conservazione (se presente) da ogni pozzetto della piastra microfluidica e sostituirla con un terreno di coltura fresco.
NOTA: Se la piastra microfluidica contiene un pozzetto di scarico dei rifiuti, la soluzione di conservazione del pozzetto di uscita deve essere rimossa ma non sostituita dal mezzo. - Sigillare la piastra microfluidica con il sistema del collettore facendo clic sul pulsante Seal o tramite il software microfluidico (selezionare prima Tool, quindi Seal Plate).
NOTA: Per sigillare la piastra, è necessario applicare una pressione uniforme alla piastra e al collettore schiacciando manualmente la piastra contro il collettore. Se eseguita correttamente, la nota "sigillato" dovrebbe apparire sull'interfaccia ONIX2. È importante non esercitare alcuna pressione sul vetrino per evitare qualsiasi rischio potenziale di rottura del collettore. - Una volta sigillato, eseguire una prima sequenza di adescamento (fare clic su Run Liquid Priming Sequence sull'interfaccia del software microfluidico).
NOTA: Run Liquid Priming Sequence corrisponde a 5 minuti di perfusione a 6,9 kPa per pozzetti 1-5, seguiti da 5 minuti di perfusione a 6,9 kPa per ben 8 e un ciclo di perfusione finale per ben 6 per 5 minuti a 6,9 kPa. La Run Liquid Priming Sequence permette la rimozione della soluzione conservativa che può essere ancora presente nei canali che collegano i diversi pozzi. - Incubare la piastra in un armadio termostatico del microscopio alla temperatura desiderata (qui, 37 °C) per un minimo di 2 ore prima dell'inizio dell'imaging microscopico.
- Avviare una seconda sequenza di adescamento liquido prima di iniziare l'esperimento.
- Sigillare la piastra microfluidica facendo clic su Sigilla disattivato sull'interfaccia del software microfluidico. Sostituire il terreno nel pozzo 1 e nel pozzetto 2 con 200 μL di terreno fresco, nel pozzo 3 con 200 μL di terreno fresco contenente l'antibiotico (qui, OFX a 5 μg·mL−1), nel pozzo 4 e nel pozzo 5 con 200 μL di terreno fresco, nel pozzo 6 con 200 μL di terreno fresco, e nel pozzo 8 con 200 μL del campione di coltura (dal punto 5.1.2) diluito ad un OD600 nm di 0,01 nel mezzo fresco.
- Sigillare la piastra microfluidica come descritto al punto 5.2.2 e posizionare la piastra sull'obiettivo del microscopio all'interno dell'armadio del microscopio.
NOTA: Assicurarsi di posizionare una goccia di olio ad immersione sull'obiettivo del microscopio prima di posizionare la piastra microfluidica. - Nel software microfluidico, fare clic su Cell Loading (Caricamento cellulare ) per consentire il caricamento della cella nella piastra microfluidica.
NOTA: La fase di carico cellulare comprende 15 s di perfusione a 13,8 kPa per il pozzo 8, seguita da 15 s di perfusione a 27,6 kPa per il pozzo 6 e il pozzo 8, e un ciclo di perfusione finale per il pozzo 6 per 30 s a 6,9 kPa. La densità delle cellule nella piastra microfluidica è fondamentale per l'esperimento. La prima parte di questo protocollo microfluidico consiste nella crescita di batteri per 6 ore in un mezzo fresco prima del trattamento antibiotico. Dopo 6 ore di crescita, la densità cellulare deve essere sufficiente per rilevare le rare cellule persistenti (nelle condizioni utilizzate in questo studio, le cellule persistenti generano ad una frequenza di 10−4). Se la densità cellulare è troppo alta, è difficile distinguere le singole cellule, il che impedisce un'analisi precisa a singola cellula. Poiché il tasso di crescita dipende direttamente dal mezzo, la densità delle cellule nei campi della microscopia dovrebbe essere valutata prima di avviare l'esperimento. - Impostare una messa a fuoco ottimale utilizzando la modalità a luce trasmessa e selezionare diverse regioni di interesse (ROI) in cui viene osservato un numero di celle appropriato (fino a 300 celle per campo).
NOTA: selezionare almeno 40 ROI per assicurarsi che le rare celle persistenti vengano visualizzate nell'immagine. - Sul software microfluidico, fare clic su Crea un protocollo. Programmare l'iniezione di terreno fresco a 6,9 kPa per 6 h (pozzo 1-2), seguita dall'iniezione del terreno contenente l'antibiotico a 6,9 kPa per 6 h (pozzo 3), e, infine, l'iniezione di terreno fresco a 6,9 kPa per 20 h (pozzetti 4-5).
NOTA: Una coltura batterica diluita a un OD600 nm di 0,01 consente ai batteri di crescere nella camera microfluidica per 6 ore, assicurando che le cellule siano nella fase di crescita esponenziale. A seconda del numero di cellule introdotte nel dispositivo microfluidico durante la fase di carico (vedi fase 5.2.8), la durata della fase di crescita può essere adattata per ottenere fino a 300 cellule per ROI. Poiché la persistenza è un fenomeno raro, l'aumento del numero di cellule per ROI migliora la possibilità di osservare le cellule persistenti. Il numero di cellule non dovrebbe, tuttavia, superare le 300 celle per ROI, poiché ciò rende noiosa l'analisi di una singola cellula. - Eseguire l'imaging al microscopio in modalità time-lapse con un fotogramma ogni 15 minuti utilizzando la luce trasmessa e la sorgente luminosa di eccitazione per il reporter fluorescente. Qui è stata utilizzata una sorgente luminosa di eccitazione a 560 nm per il segnale mCherry (LED da 580 nm al 10% di potenza con filtro 00 [530-585 ex, 615LP em, Zeiss] e esposizione a 100 ms per mCherry). Per l'imaging cellulare è stato utilizzato il software Zen3.2 compatibile con Zeiss.
- Rimuovere la soluzione di conservazione (se presente) da ogni pozzetto della piastra microfluidica e sostituirla con un terreno di coltura fresco.
- Analisi delle immagini
NOTA: L'apertura e la visualizzazione delle immagini al microscopio vengono eseguite con il software open source ImageJ/Fiji (https://fiji.sc/)26. L'analisi quantitativa delle immagini viene eseguita utilizzando il software open source ImageJ/Fiji e il plugin gratuito MicrobeJ (https://microbej.com)27. In questo protocollo è stata utilizzata la versione MicrobeJ 5.13I(14).- Aprire il software ImageJ/Fiji sul computer e trascinare le immagini di microscopia time-lapse hyperstack nella barra di caricamento delle Fiji. Utilizzare Immagine > Colore > Crea composito per fondere i diversi canali dell'hyperstack. Se i canali dell'esperimento time-lapse non corrispondono al colore desiderato (ad esempio, il contrasto di fase viene mostrato in rosso anziché in grigio), utilizzare Immagine > Colore > Disponi canali per applicare il colore appropriato ai canali.
- Apri il plug-in MicrobeJ e rileva le cellule batteriche utilizzando l'interfaccia di modifica manuale. Eliminare le celle rilevate automaticamente e delineare manualmente le celle persistenti di interesse fotogramma per fotogramma.
NOTA: è possibile utilizzare impostazioni diverse per rilevare automaticamente le singole celle. Il rilevamento manuale è stato utilizzato qui poiché le cellule persistenti analizzate formano lunghi filamenti, che raramente vengono rilevati correttamente utilizzando il rilevamento automatico. - Dopo il rilevamento, utilizzare l'icona Risultato nell'interfaccia di modifica manuale MicrobeJ per generare una tabella ResultJ. Salvare il file ResultJ e utilizzare la tabella ResultJ per ottenere informazioni dettagliate sui diversi parametri di interesse dell'analisi a cella singola. Nel caso di questo protocollo, sono state esportate la fluorescenza media dell'intensità di HU-mCherry, la lunghezza della cellula e l'area cellulare delle singole cellule.
Risultati
Come descritto sopra, i ceppi utilizzati per l'analisi fenotipica a singola cellula delle cellule persistenti sono stati caratterizzati in MOPS glicerolo 0,4% medio. Il monitoraggio dell'OD600nm nel tempo non ha mostrato alcuna differenza tra i ceppi wt e hupA-mCherry (Figura 1). Ciò indica che l'espressione della proteina di fusione HU-mCherry non ha influito sulla crescita in queste condizioni. Le cellule batteriche di entrambi i ceppi inizialmente inoculati a un OD600 nm di 0,01 hanno raggiunto la fase esponenziale ±8 h dopo l'inoculazione.
Il MIC di OFX è stato determinato con metodi standardizzati (qui, diluizione seriale dell'agar)24. La MIC è definita come la concentrazione minima in cui non viene rilevata alcuna crescita visibile. Il MIC di OFX per entrambi i ceppi è stato determinato essere 0,06 μg·mL−1, indicando che la fusione hupA-mCherry non ha avuto alcun effetto sulla sensibilità all'OFX rispetto al ceppo isogenico wt (Figura 2).
Abbiamo inoltre determinato l'effetto di un trattamento OFX letale (83 volte MIC) sulla vitalità di entrambi i ceppi utilizzati in questo studio. Poiché la conta delle cellule vitali diminuisce nel tempo con l'esposizione a OFX, le diluizioni delle colture batteriche devono essere regolate in modo appropriato per raggiungere da 30 a 300 colonie per piastra. Per determinare le diluizioni appropriate nel tempo, è stato eseguito un test spot, in cui sono stati posizionati 10 μL da 0 a 10−7 diluizioni seriali 10 volte su piastre di Petri quadrate utilizzando una pipetta multicanale. Le diluizioni appropriate erano quelle in cui erano visibili cloni isolati (ad esempio, a t0 = 10−5, t1h = 10−4/10−3, t4h = 10−2/10−1) (Figura 3).
Mentre il test spot è un metodo semplice per ottenere informazioni sulla cinetica dell'uccisione mediata da OFX, non riesce a determinare con precisione le dinamiche di uccisione. Quando la vitalità delle cellule in crescita esponenziale trattate con OFX è stata monitorata dal saggio time-kill, è stata osservata una tipica curva bifasica (Figura 4). La prima pendenza della curva riflette la rapida uccisione della popolazione non persistente (linea tratteggiata rossa). Nelle condizioni qui testate, fino al 99,9% delle cellule non è stato in grado di formare colonie dopo 3 ore in presenza di OFX. Questa prima fase di uccisione è seguita da una seconda fase, che mostra un tasso di uccisione più lento (linea tratteggiata blu), che rivela la presenza di cellule persistenti tolleranti ai farmaci. Nelle condizioni testate, la fase di persistenza è iniziata circa 3 ore dopo l'aggiunta di OFX, evidenziando la necessità di esporre le cellule all'OFX per più di 3 ore per studiare i fenotipi persistenti. È importante sottolineare che la curva time-kill mostra che la proteina di fusione hupA-mCherry non ha avuto alcun effetto sulla cinetica time-kill. Il ceppo che codifica la fusione fluorescente traslazionale può quindi essere utilizzato per monitorare le cellule persistenti utilizzando la microscopia a fluorescenza.
Abbiamo inoltre studiato il fenomeno della persistenza a livello di singola cellula. Per fare ciò, il ceppo hupA-mCherry è stato introdotto in una piastra microfluidica, che ha permesso il cambiamento delle condizioni del mezzo (qui, crescita, trattamento e recupero) durante l'esecuzione della microscopia time-lapse su un dato ROI. Durante la prima fase dell'esperimento microfluidico, le cellule introdotte nel dispositivo microfluidico sono state perfuse con terreno di crescita (MOPS glicerolo 0,4%) e divise con un tempo di generazione di ~ 2 h (Figura 5 e Figura 6). Questa prima fase di crescita indica che le cellule erano vitali e si dividevano attivamente prima del trattamento OFX.
Dopo questa prima fase di crescita, le cellule sono state perfuse con terreno di crescita integrato con 5 μg·mL−1 OFX per 6 ore. Non appena l'antibiotico ha raggiunto le cellule, la divisione cellulare è stata bloccata (Figura 5 e Figura 6). Dopo 6 ore di trattamento OFX, le cellule sono state perfuse con terreno fresco. Mentre la stragrande maggioranza delle cellule non era in grado di riprendere la crescita (Figura 5 e Figura 6), una piccola sottopopolazione di batteri era in grado di allungare e generare cellule filamentose25. Queste cellule, che sono state in grado di dividersi e generare cellule figlie vitali dopo il trattamento OFX, possono essere definite come cellule persistenti.
Poiché questa configurazione consente la visualizzazione delle cellule persistenti prima, durante e dopo il trattamento, non solo fornisce informazioni sul fenotipo persistente durante la fase di recupero, ma anche sullo stato fisiologico delle cellule persistenti prima del trattamento (Figura 6). Nelle condizioni testate, le cellule persistenti si sono divise in modo simile alle cellule non persistenti prima del trattamento OFX, indicando che le cellule persistenti osservate non provenivano da una sottopopolazione dormiente (Figura 6)25.
L'analisi della lunghezza cellulare delle cellule persistenti durante la fase di recupero ha rivelato che ogni filamento aveva una velocità specifica di allungamento. La lunghezza della cella raggiunta da ciascun persistente prima della prima divisione differiva da un persister all'altro. Allo stesso modo, la tempistica dell'evento di prima divisione era altamente eterogenea (Figura 6). Il filamento persistente in divisione ha generato più cellule figlie, che hanno iniziato a crescere e dividersi, per la maggior parte, in modo simile alle cellule non trattate (Figura 7). La successiva divisione del filamento ha poi comportato una progressiva diminuzione della lunghezza della cellula, dando infine origine a cellule figlie con lunghezza cellulare simile a prima del trattamento OFX (Figura 6 e Figura 7B). La stragrande maggioranza delle cellule non è stata in grado di indurre la filamentazione dopo la rimozione di OFX. Questa grande popolazione cellulare corrisponde alle cellule morte (Figura 5 e Figura 6).
La fusione fluorescente della proteina HU associata al nucleoide consente la visualizzazione della dinamica del nucleoide22. L'analisi dell'intensità totale di fluorescenza di HU-mCherry all'interno della cellula può essere utilizzata come proxy per il contenuto di DNA22,25. Durante la fase di crescita (prima del trattamento OFX), l'intensità totale della fluorescenza mCherry variava, riflettendo la dinamica della replicazione cromosomica e della segregazione durante il ciclo cellulare (Figura 8). Dopo l'aggiunta di OFX, la fluorescenza di mCherry è aumentata a metà cellula, indicativa della compattazione nucleoide, che ha dimostrato essere indotta dalla formazione di rotture del DNA a doppio filamento28 (Figura 5). Le rotture del DNA a doppio filamento sono una conseguenza del meccanismo d'azione di OFX, che corrompe le topoisomerasi di tipo II DNA-girasi e la topoisomerasi IV29,30. In E. coli, la DNA-girasi è il bersaglio primario di OFX29,30. Legando il suo bersaglio in una fase critica del meccanismo di passaggio a doppio filamento, OFX inibisce la retrocessione dei filamenti di DNA scissi, portando infine al rilascio di rotture di DNA a doppio filamento30. Come descritto sopra, le cellule persistenti al trattamento OFX hanno iniziato a filarsi durante il recupero25 (Figura 6). L'aumento della lunghezza della cellula è correlato con un aumento dell'intensità totale della fluorescenza mCherry, che riflette il riavvio della replicazione e un aumento dell'abbondanza nucleoidale nel filamento25 (Figura 7a e Figura 8). Per le cellule morte, l'intensità totale della fluorescenza mCherry è rimasta stabile durante il trattamento e durante la fase di recupero, indicando che queste cellule non erano in grado di replicare i loro cromosomi dopo la rimozione di OFX (Figura 8). Viene anche mostrato un video microfluidico (Video 1) delle cellule di E. coli HU-mCherry prima, durante e dopo il trattamento con ofloxacina.
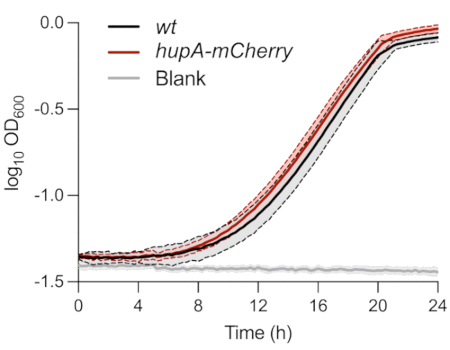
Figura 1: Monitoraggio della crescita dei ceppi di wt e hupA-mCherry E. coli. Monitoraggio della densità ottica (OD600 nm) di wt (nero) e hupA-mCherry (rosso). Le sfumature e le linee tratteggiate indicano le deviazioni standard dei triplicati biologici. Fare clic qui per visualizzare una versione ingrandita di questa figura.
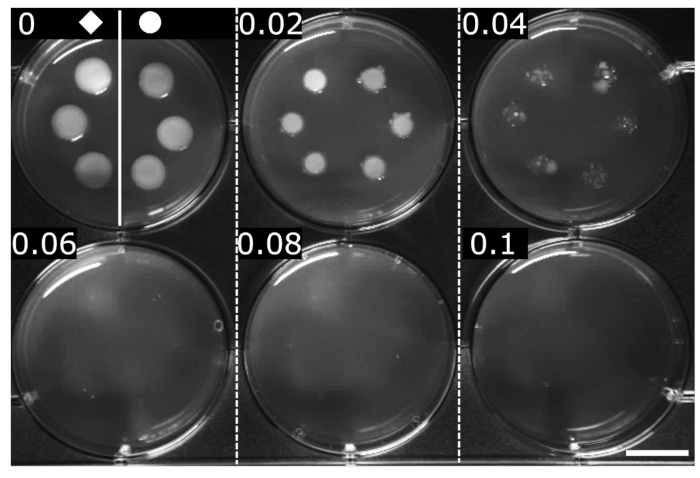
Figura 2: Determinazione della MIC di OFX per i ceppi wt e hupA-mCherry E. coli. La wt () e l'hupA-mCherry (●) sono state coltivate in mezzo LB e 2 μL sono stati individuati su diluizioni seriali di agar LB contenente OFX (♦ concentrazione indicata in ciascun pannello in μg·mL−1). L'inibizione della crescita è visibile ad un minimo di 0,06 μg·mL−1. La figura è un esperimento rappresentativo di triplicati biologici. Barra della scala = 1 cm. Fare clic qui per visualizzare una versione più grande di questa figura.
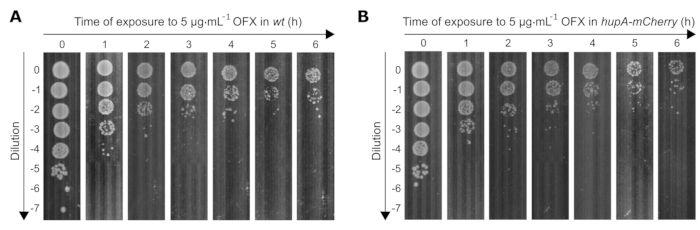
Figura 3: Saggio a campione dei ceppi wt e hupA-mCherry E. coli dopo esposizione a OFX. I ceppi (A) wt e (B) hupA-mCherry sono stati coltivati in glicerolo MOPS 0,4% come descritto nel protocollo (sezione 3) e le cellule a crescita esponenziale (OD600 nm = 0,3) sono state trattate con 5 μg·mL−1 OFX. T0 corrisponde al punto temporale prima dell'aggiunta di OFX. T1, T2, T3, T4, T5 e T6 corrispondono a 1-6 h dopo l'aggiunta di OFX. La figura è un esperimento rappresentativo di triplicati biologici. Fare clic qui per visualizzare una versione ingrandita di questa figura.
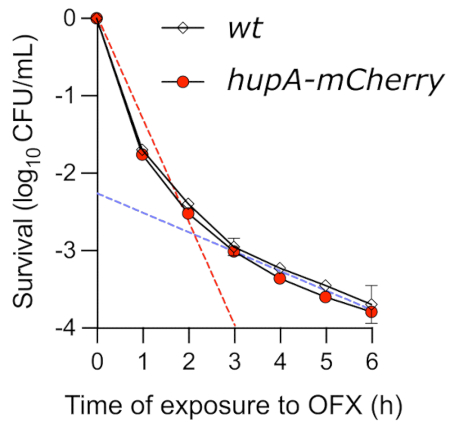
Figura 4: Saggio time-kill dei ceppi wt e hupA-mCherry E. coli dopo esposizione a OFX. I ceppi wt () e hupA-mCherry (●) sono stati coltivati in glicerolo MOPS 0,4% come descritto nel protocollo (sezione 4) e le cellule a crescita esponenziale (♦ OD600 nm = 0,3) sono state trattate con 5 μg·mL−1 OFX. Le linee tratteggiate indicano la prima fase di uccisione "rapida" (rossa) e la seconda fase di uccisione "lenta" (blu), corrispondenti alle sottopopolazioni sensibili e persistenti (ottenute per regressione lineare tra T0 e T2, nonché tra T3 e T6, rispettivamente). Le barre di errore indicano le deviazioni standard dei triplicati biologici. Fare clic qui per visualizzare una versione ingrandita di questa figura.
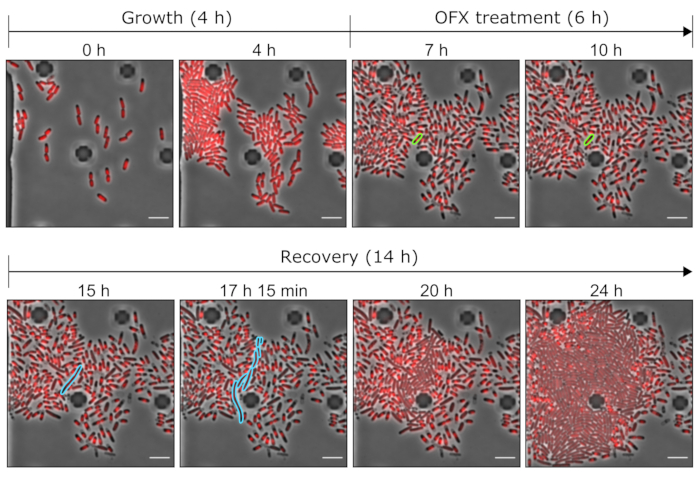
Figura 5: Immagini rappresentative del persistente OFX e delle cellule morte utilizzando strumenti microfluidici. Immagini al microscopio rappresentative che mostrano i punti temporali rilevanti dell'esperimento microfluidico eseguito con il ceppo hupA-mCherry (contrasto di fase in grigio, segnale HU-mCherry in rosso). Le cellule che esprimono l'hupA-mCherry marcato sono state coltivate in una piastra microfluidica (qui, 4 ore), seguita da una sfida OFX (5 μg·mL−1). Dopo 6 ore in presenza di OFX, le cellule sono state perfuse con terreno fresco, consentendo alle cellule persistenti di recuperare. La cellula persistente e le sue cellule progenie durante il trattamento con OFX e dopo la rimozione di OFX sono evidenziate rispettivamente in verde e blu. I punti temporali corrispondenti sono indicati su ciascun pannello. Barra di scala = 5 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
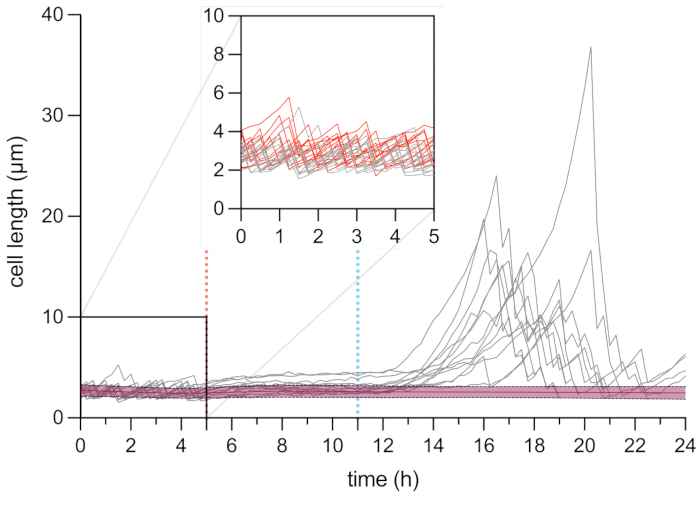
Figura 6: Analisi time-lapse al microscopio della lunghezza delle cellule persistenti e morte. Analisi della lunghezza cellulare delle cellule morte (in rosso, n = 109) e delle cellule persistenti (in grigio, n = 13). L'inizio del trattamento OFX (5 μg·mL−1) è indicato dalla linea tratteggiata rossa (5 ore) e la rimozione di OFX è indicata dalla linea tratteggiata blu. L'inserto corrisponde alla fase di crescita prima dell'aggiunta di OFX. Gli esperimenti sono stati eseguiti in triplice copia. Le sfumature e le linee tratteggiate indicano le deviazioni standard per la popolazione di cellule morte (n = 109). Fare clic qui per visualizzare una versione ingrandita di questa figura.
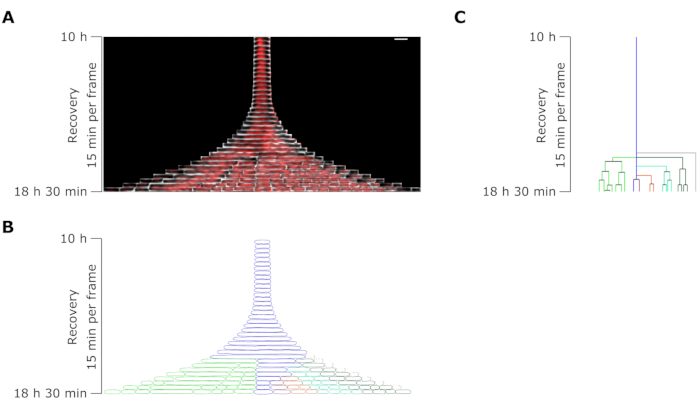
Figura 7: Analisi time-lapse al microscopio di un persistente rappresentativo di OFX. (A) Cimografo di un persistente OFX rappresentativo e delle sue cellule figlie generate da divisioni di filamenti durante 8,5 ore dopo la rimozione di OFX (18,5 ore dopo l'inizio dell'esperimento microfluidico, comprendente 4 ore di crescita, 6 ore di 5 μg·mL−1 di trattamento OFX e 8,5 ore di recupero dopo rimozione di OFX). Un fotogramma corrisponde a 15 min. Barra della scala = 5 μm. (B) Maschera generata dal cimografo persistente in A. La cella persistente monitorata è indicata con un contorno blu e le celle figlie sono evidenziate in colori distinti. (C) Rappresentazione schematica della linea cellulare persistente generata da B. La codifica a colori è identica a B. Fare clic qui per visualizzare una versione ingrandita di questa figura.
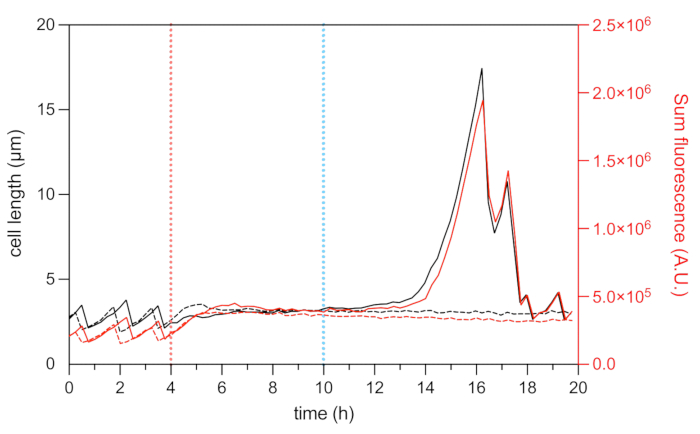
Figura 8: Analisi della lunghezza cellulare e della fluorescenza mCherry di cellule persistenti e morte rappresentative. Analisi della lunghezza della cella (asse sinistro) e dell'intensità totale della fluorescenza HU-mCherry (asse destro, mostrato in unità arbitrarie) di un persistente rappresentativo (linee nere e rosse solide) e di una cellula morta rappresentativa (linea nera e rossa tratteggiata) durante l'esperimento time-lapse microfluidico. L'inizio del trattamento con OFX (5 μg·mL−1) è indicato dalla linea tratteggiata rossa e la rimozione dell'OFX dalla linea tratteggiata blu. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Video 1: Video microfluidico di cellule di E. coli HU-mCherry prima, durante e dopo il trattamento con ofloxacina. Imaging time-lapse microfluidico che mostra cellule HU-mCherry. Le cellule sono state coltivate per 4 ore in MOPS glicerolo 0,4%. Dopo 6 ore di trattamento con OFX (5 μg·mL−1), il mezzo privo di antibiotici è stato perfuso nella piastra microfluidica per consentire alle cellule persistenti di recuperare. Barra della scala = 5 μm. Il tempo (in min) è indicato. Le fasi di crescita e recupero sono indicate da "MOPS-Gly. 0,4%" e il trattamento OFX con "OFX 5 μg/ml". Clicca qui per scaricare questo video.
| MOPS 10x | |||
| Soluzione madre | Volume della soluzione madre per 1 L di 10x MOPS | Concentrazione finale in base 10x MOPS | |
| Acido MOPS | 1 M (regolato a pH 7,4 utilizzando KOH) | 400 ml | 0,4 M |
| Patrizia | 1 M (regolato a pH 7,4 utilizzando KOH) | 40 ml | 0,04 M |
| FeSO4.7H2O | 0,01 M | 10 ml | 0,0001 m |
| NH4Cl | 1,9 M | 50 ml | 0,095 M |
| K2SO4 | 0,276 M | 10 ml | 0,00276 m |
| CaCl2.2H 2O | 0,0005 m | 10 ml | 0,000005 m |
| MgCl 2.6H2O | 0,528 m | 10 ml | 0,00528 M |
| NaCl | aggiungere direttamente 29,2 g | 0,5 M | |
| Acqua distillata | 460 ml | ||
| Micronutrimenti 1000x (vedi Tabella 2) | 10 ml | ||
Tabella 1: Composizione di 10x MOPS.
| Micronutrienti 1000x | ||
| Concentrazione in Micronutrimenti 1000x soluzione madre | Concentrazione finale in base 10x MOPS | |
| (NH4)6Mo7O24.4H2O | 0,000003 m | 0,00000003 m |
| H3 BO3 | 0,0004 m | 0,000004 m |
| CoCl2.6H 2O | 0,00003 m | 0,0000003 m |
| CuSO4.5H 2O | 0,00001 M | 0,0000001 M |
| MnCl2.4H 2O | 0,00008 m | 0,0000008 m |
| ZnSO.7H2O | 0,00001 M | 0,0000001M |
Tabella 2: Composizione di 1.000 micronutrienti.
| MOPS glucosio 0,4% o MOPS glicerolo 0,4% | |||
| Soluzione madre | Volume per 1 L MOPS glucosio 0,4 % o MOPS glicerolo 0,4 % | Concentrazione finale in glucosio MOPS 0,4% o glicerolo MOPS 0,4% | |
| MOPS 10x | vedi tabella 1 | 100 ml | |
| K2HPO4 | 0,132 M | 10 ml | 0,00132 M |
| Glucosio (per MOPS glucosio 0,4%) | 20% (20 g in 100 ml di acqua distillata) | 20 ml | 0.40% |
| Glicerolo (per MOPS glicerolo 0,4%) | ≤99% | 4 ml | 0.40% |
| Acqua distillata | 870 ml per MOPS glucosio 0,4% o 886 ml per MOPS glicerolo 0,4% | ||
Tabella 3: Composizione di glucosio MOPS 0,4% e glicerolo MOPS 0,4%.
Discussione
Il protocollo presentato in questo articolo consente l'analisi del fenotipo di persistenza osservato in risposta al trattamento antibiotico a livello di popolazione e di singola cellula. Gli esperimenti sono stati eseguiti con il ceppo E. coli MG1655, che è stato coltivato in un mezzo chimicamente definito (glicerolo MOPS 0,4%). Sono stati condotti saggi time-kill ed esperimenti di microscopia su colture in fase esponenziale. Abbiamo usato OFX, un fluorochinolone, ad una concentrazione di 5 μg·mL−1 per rivelare le cellule persistenti. Gli approcci qui descritti possono essere applicati ad altri antibiotici battericidi, come β-lattamici, aminoglicosidi o composti antimicrobici31. Di conseguenza, possono essere utilizzati altri ceppi batterici, mezzi o condizioni di crescita. Il monitoraggio di diverse fusioni fluorescenti in una configurazione simile a quella descritta qui può essere utile per seguire processi cellulari come la replicazione del DNA32, la riparazione del DNA25,33 e la divisione cellulare34 prima, durante e dopo il trattamento antibiotico. Allo stesso modo, i reporter fluorescenti possono essere sfruttati per indagare aspetti distinti della fisiologia cellulare, come i livelli intracellulari di pH35, ATP 36 o ROS37. In alternativa alle fusioni fluorescenti, potrebbero essere applicati anche coloranti chimici. Ad esempio, la fusione hupA-mCherry potrebbe essere sostituita da 4',6-diamidino-2-fenilindolo (DAPI), un colorante fluorescente che colora il DNA38. L'esecuzione di microscopia time-lapse accoppiata con tali coloranti fluorescenti dovrebbe, tuttavia, essere evitata, poiché queste tecniche di colorazione possono perturbare la dinamica del ciclo cellulare durante gli esperimenti time-lapse. In alternativa, tali esperimenti possono essere sostituiti da analisi del corso temporale dell'imaging istantaneo in punti temporali rilevanti.
Mentre tali reporter fluorescenti sono utili, la quantità di informazioni che possono essere estratte attraverso l'analisi delle immagini a contrasto di fase non dovrebbe essere trascurata. Qui, abbiamo monitorato l'evoluzione della lunghezza cellulare durante le fasi di crescita, trattamento OFX e recupero. Altri parametri basati su immagini a contrasto di fase, come la larghezza della cella, l'intensità del contrasto di fase e le curvature delle cellule batteriche, possono anche essere estratti con facilità utilizzando software adeguati, come MicrobeJ27.
In sintesi, la procedura qui descritta può essere applicata ad altre condizioni e specie batteriche per monitorare le risposte cellulari a cambiamenti ambientali o fattori di stress18,19. Utilizzando altri reporter fluorescenti (reporter trascrizionali e traslazionali, coloranti chimici) in combinazione con un'analisi di popolazione, come la citometria a flusso / FACS, domande interessanti possono essere affrontate in un quadro multiscala.
Divulgazioni
Gli autori non dichiarano interessi concorrenti.
Riconoscimenti
Il lavoro nel laboratorio Van Melderen è supportato dalle azioni ARC 2018-2023, il Fonds National de la Recherche Scientifique (FNRS CDR J.0182.21F). T.O. è supportato da una borsa di studio ULB. T.S. è supportato da una borsa di studio FRIA (FNRS). J.C. è sostenuto da una borsa di studio post-dottorato "chargé de recherches" (FNRS).
Materiali
| Name | Company | Catalog Number | Comments |
| Axio Observer | Zeiss | Inverted fluorescence microscope | |
| CaCl2.2H2O | Merck | 1.02382.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CellASIC ONIX Microfluidic System | Merck | CAX2-S0000 | Microfluidic system |
| CellASIC ONIX2 FG | Merck | ONIX2 1.0.1 | Microfluidic software |
| CellASIC ONIX2 Manifold Basic | Merck | CAX2-MBC20 | Manifold system |
| CoCl2.6H2O | Merck | 1.02539.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CuSO4.5H2O | Merck | 1.02790.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| D-(+)-glucose | Sigma Aldrich | G7021-1KG | Carbon source for MOPS glucose 0.4% growth medium |
| E. coli K-12 MG1655 CF1648 (fnr+) | BE10 | wt reference strain, lab strain | |
| E. coli K-12 MG1655 CF1648 (fnr+) hupA-mCherry::FRT-kan-FRT | BE16 | HU-mCherry fusion integrated via P1 transduction at the native locus, lab strain | |
| FeSO4.7H2O | VWR Chemicals | 24244.232 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Fiji | ImageJ | https://fiji.sc/ | Image software; Schindelin et al. if used in publication |
| Glycerol | Merck | 56815 | Carbon source for MOPS glycerol 0.4% growth medium |
| Greiner CELLSTAR® multiwell culture plate | Merck | M8812-100EA | 24-well clear flat bottom 2ml volume culture plate for automated plate reader |
| H3BO3 | Sigma-Aldrich | B6768-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Hamamatsu ORCA Flash 4.0 digital camera | Hamamatsu | C13440-20CU | Digital Image Acqusition |
| K2HPO4 | Merck | 1.05099.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| KOH | Merck | 1.05029.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| K2SO4 | Merck | 1.05153.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Luria-Broth agar medium | Invitrogen | 22700041 | Growth medium for plating assay |
| Luria-Broth medium | Invitrogen | 12780029 | Growth medium for MIC determination |
| MgCl2.6H2O | Merck | 1.05832.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MgSO4 | Merck | 7487-88-9 | Dilution solution for bacterial survival assay used at 10mM |
| MicrobeJ | Imagej/Fiji plugin | https://www.microbej.com/ | Microscopy image analysis plugin. Ducret et al.for in publication; Detection settings: For bacteria : Area (μm2): 0,1-max; Length (μm): 0,5-max; Width (μm): 0,6-max; Range (μm): 0,5-max; Angularity (rad): 0-0,3; 0-max for all other parameters. |
| Microfluidic Plates CellASIC ONIX | Merck | B04A-03-5PK | Plate for microfluidic system |
| MnCl2.4H2O | Merck | 1.05927.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MOPS, Free Acid ULTROL® Grade | Merck | 475898-500GM | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NaCl | SIgma-Aldrich | S5886-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| (NH4)6Mo7O24.4H2O | Merck | 1.01180.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NH4Cl | Merck | 1.01145.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Ofloxacin | Merck | 82419-36-1 | Fluoroquinolone antibiotic used to treat the bacterial cells |
| Phosphate Buffered Saline (PBS) pH 7.4, 1x | Merck | P3813 | Dilution buffer |
| GENESYS 10S UV-Visible Spectrophotometer | Thermofisher Scientific | 840-208200 | UV-Visible Spectrophotometer, Single/Six Cell Holder with PrinterMeasurement of optical density OD600nm |
| SoftMax Pro | Molecular Devices | Microplate reader software | |
| SpectraMax i3x | Molecular Devices | Microplate reader | |
| N-[Tris(hydroxyméthyl)-méthyl]-glycine | Merck | 1.08602.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Zeiss® immersion Oil 518F | Zeiss | Immersion oil to increase resolution of microscope | |
| Zen3.2 Pro | Zeiss | Microscopic image acquisition and processing software | |
| ZnSO4.7H2O | Merck | 1.08883.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
Riferimenti
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nature Reviews Microbiology. 17 (7), 441-448 (2019).
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nature Reviews Microbiology. 14 (5), 320-330 (2016).
- Pribis, J. P., Zhai, Y., Hastings, P. J., Rosenberg, S. M. Stress-induced mutagenesis, gambler cells, and stealth targeting antibiotic-induced evolution. mBio. 13 (3), 01074 (2022).
- Molina-Quiroz, R. C., Lazinski, D. W., Camilli, A., Levy, S. B. Transposon-sequencing analysis unveils novel genes involved in the generation of persister cells in uropathogenic Escherichia coli. Antimicrobial Agents and Chemotherapy. 60 (11), 6907-6910 (2016).
- Cameron, D. R., Shan, Y., Zalis, E. A., Isabella, V., Lewis, K. A genetic determinant of persister cell formation in bacterial pathogens. Journal of Bacteriology. 200 (17), 00303-00318 (2018).
- Cui, P., et al. Identification of genes involved in bacteriostatic antibiotic-induced persister formation. Frontiers in Microbiology. 9, 413 (2018).
- Li, T., et al. Identification of novel genes involved in Escherichia coli persistence to tosufloxacin. Frontiers in Cellular and Infection Microbiology. 10, 581986 (2020).
- Verstraeten, N., et al. Obg and membrane depolarization are part of a microbial bet-hedging strategy that leads to antibiotic tolerance. Molecular Cell. 59 (1), 9-21 (2015).
- Hofsteenge, N., van Nimwegen, E., Silander, O. K. Quantitative analysis of persister fractions suggests different mechanisms of formation among environmental isolates of E. coli. BMC Microbiology. 13 (1), 25 (2013).
- Huemer, M., Mairpady Shambat, S., Brugger, S. D., Zinkernagel, A. S. Antibiotic resistance and persistence-Implications for human health and treatment perspectives. EMBO Reports. 21 (12), 51034 (2020).
- Wilmaerts, D., Windels, E. M., Verstraeten, N., Michiels, J. General mechanisms leading to persister formation and awakening. Trends in Genetics. 35 (6), 401-411 (2019).
- Murawski, A. M., Brynildsen, M. P. Ploidy is an important determinant of fluoroquinolone persister survival. Current Biology. 31 (10), 2039-2050 (2021).
- Lemma, A. S., Soto-Echevarria, N., Brynildsen, M. P. Fluoroquinolone persistence in Escherichia coli requires DNA repair despite differing between starving populations. Microorganisms. 10 (2), 286 (2022).
- Balaban, N. Q., Merrin, J., Chait, R., Kowalik, L., Leibler, S. Bacterial persistence as a phenotypic switch. Science. 305 (5690), 1622-1625 (2004).
- Morawska, L. P., Kuipers, O. P. Antibiotic tolerance in environmentally stressed Bacillus subtilis: Physical barriers and induction of a viable but nonculturable state. microLife. 3, (2022).
- Windels, E. M., et al. Enrichment of persisters enabled by a ß-lactam-induced filamentation method reveals their stochastic single-cell awakening. Communications Biology. 2 (1), 426 (2019).
- Leygeber, M., et al. Analyzing microbial population heterogeneity-Expanding the toolbox of microfluidic single-cell cultivations. Journal of Molecular Biology. 431 (23), 4569-4588 (2019).
- Rojas, E., Theriot, J. A., Huang, K. C. Response of Escherichia coli growth rate to osmotic shock. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7807-7812 (2014).
- Shi, H., et al. Starvation induces shrinkage of the bacterial cytoplasm. Proceedings of the National Academy of Sciences of the United States of America. 118 (24), 2104686118 (2021).
- Goode, O., et al. Persister Escherichia coli cells have a lower intracellular pH than susceptible cells but maintain their pH in response to antibiotic treatment. mBio. 12 (4), 00909-00921 (2021).
- Manuse, S., et al. Bacterial persisters are a stochastically formed subpopulation of low-energy cells. PLoS Biology. 19 (4), 3001194 (2021).
- Fisher, J. K., et al. Four-dimensional imaging of E. coli nucleoid organization and dynamics in living cells. Cell. 153 (4), 882-895 (2013).
- Cabral, D., Wurster, J., Belenky, P. Antibiotic persistence as a metabolic adaptation: Stress, metabolism, the host, and new directions. Pharmaceuticals. 11 (1), 14 (2018).
- Wiegand, I., Hilpert, K., Hancock, R. E. W. Agar and broth dilution methods to determine the minimal inhibitory concentration (MIC) of antimicrobial substances. Nature Protocols. 3 (2), 163-175 (2008).
- Goormaghtigh, F., Van Melderen, L. Single-cell imaging and characterization of Escherichia coli persister cells to ofloxacin in exponential cultures. Science Advances. 5 (6), (2019).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Ducret, A., Quardokus, E. M., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nature Microbiology. 1 (7), 16077 (2016).
- Odsbu, I., Skarstad, K. DNA compaction in the early part of the SOS response is dependent on RecN and RecA. Microbiology. 160 (5), 872-882 (2014).
- Pham, T. D. M., Ziora, Z. M., Blaskovich, M. A. T. Quinolone antibiotics. MedChemComm. 10 (10), 1719-1739 (2019).
- Drlica, K., et al. Quinolones: Action and resistance updated. Current Topics in Medicinal Chemistry. 9 (11), 981-998 (2009).
- Kohanski, M. A., Dwyer, D. J., Collins, J. J. How antibiotics kill bacteria: From targets to networks. Nature Reviews Microbiology. 8 (6), 423-435 (2010).
- Reyes-Lamothe, R., Sherratt, D. J., Leake, M. C. Stoichiometry and architecture of active DNA replication machinery in Escherichia coli. Science. 328 (5977), 498-501 (2010).
- Lesterlin, C., Ball, G., Schermelleh, L., Sherratt, D. J. RecA bundles mediate homology pairing between distant sisters during DNA break repair. Nature. 506 (7487), 249-253 (2014).
- Alexeeva, S., Gadella, T. W. J., Verheul, J., Verhoeven, G. S., Den Blaauwen, T. Direct interactions of early and late assembling division proteins in Escherichia coli cells resolved by FRET: Bacterial division studied by spectral FRET. Molecular Microbiology. 77 (2), 384-398 (2010).
- Miesenböck, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Yaginuma, H., et al. Diversity in ATP concentrations in a single bacterial cell population revealed by quantitative single-cell imaging. Scientific Reports. 4, 6522 (2014).
- Ermakova, Y. G., et al. Red fluorescent genetically encoded indicator for intracellular hydrogen peroxide. Nature Communications. 5, 5222 (2014).
- Kapuscinski, J. DAPI: A DNA-specific fluorescent probe. Biotechnic & Histochemistry. 70 (5), 220-233 (1995).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon