Screening ad alta produttività per ottenere colpi cristallini per la cristallografia proteica
In This Article
Summary
Questo protocollo descrive in dettaglio lo screening di cristallizzazione ad alto rendimento, che va dalla preparazione della piastra di microdosaggio di 1.536 alla fine di una finestra temporale sperimentale di 6 settimane. Sono inclusi dettagli sulla configurazione del campione, sull'imaging ottenuto e su come gli utenti possono eseguire analisi utilizzando un'interfaccia utente grafica abilitata all'intelligenza artificiale per identificare in modo rapido ed efficiente le condizioni di cristallizzazione macromolecolare.
Abstract
La cristallografia a raggi X è la tecnica più comunemente impiegata per discernere le strutture macromolecolari, ma la fase cruciale di cristallizzare una proteina in un reticolo ordinato suscettibile di diffrazione rimane impegnativa. La cristallizzazione delle biomolecole è in gran parte definita sperimentalmente e questo processo può essere laborioso e proibitivo per i ricercatori di istituzioni con risorse limitate. Presso il National High-Throughput Crystallization (HTX) Center, sono stati implementati metodi altamente riproducibili per facilitare la crescita dei cristalli, tra cui una configurazione automatizzata di piastre microbatch sotto olio ad alta produttività da 1.536 pozzetti progettata per campionare un'ampia gamma di parametri di cristallizzazione. Le lastre vengono monitorate utilizzando modalità di imaging all'avanguardia nel corso di 6 settimane per fornire informazioni sulla crescita dei cristalli e per distinguere con precisione i preziosi colpi di cristallo. Inoltre, l'implementazione di un algoritmo di punteggio dell'intelligenza artificiale addestrato per identificare i colpi di cristallo, insieme a un'interfaccia open source e user-friendly per la visualizzazione di immagini sperimentali, semplifica il processo di analisi delle immagini di crescita dei cristalli. Qui, vengono descritte le procedure e la strumentazione chiave per la preparazione dei cocktail e delle piastre di cristallizzazione, l'imaging delle piastre e l'identificazione dei colpi in modo da garantire la riproducibilità e aumentare la probabilità di successo della cristallizzazione.
Introduction
Anche in un'epoca di enormi progressi nei metodi di biologia strutturale, la cristallografia a raggi X continua ad essere un metodo affidabile e popolare per generare modelli strutturali di macromolecole di alta qualità. Oltre l'85% di tutti i modelli strutturali tridimensionali depositati nella Protein Data Bank (PDB) provengono da metodi strutturali basati su cristalli (a gennaio 2023). 1 Inoltre, la cristallografia a raggi X rimane indispensabile per risolvere le strutture proteina-ligando, una componente cruciale del processo di scoperta e sviluppo dei farmaci2. Nonostante la cristallizzazione delle proteine sia rimasta la tecnica di biologia strutturale dominante per oltre mezzo secolo, i metodi per prevedere la probabilità di cristallizzazione basati sulle proprietà fisiche3 o sulla sequenza 4,5 sono ancora agli inizi.
La previsione delle condizioni di cristallizzazione è ancora più oscura; Sono stati fatti progressi limitati per prevedere probabili condizioni di cristallizzazione anche per le proteine modello 6,7. Altri studi hanno tentato di identificare le condizioni di cristallizzazione basate sull'omologia proteica e le condizioni estratte dal PDB 8,9,10. Il potere predittivo che si trova nel PDB è limitato, tuttavia, poiché vengono depositate solo le condizioni finali di cristallizzazione di successo, che, per necessità, mancano gli esperimenti di ottimizzazione spesso estesi richiesti per mettere a punto la crescita dei cristalli. Inoltre, molte voci PDB mancano di metadati contenenti questi dettagli, tra cui le formule cocktail, il formato di cristallizzazione, la temperatura e il tempo per cristallizzare11,12. Pertanto, per molte proteine di interesse, il modo più accessibile per determinare le condizioni di cristallizzazione è sperimentalmente, utilizzando il maggior numero possibile di condizioni in una vasta gamma di possibilità chimiche.
Sono stati esplorati diversi approcci per rendere lo screening della cristallizzazione il più fruttuoso e completo possibile con grande effetto, tra cui matrici sparse 13, screening fattoriale incompleto 14, additivi15,16, semina17 e agenti nucleanti 18. Il National HTX Center presso l'Hauptman-Woodward Medical Research Institute (HWI) ha sviluppato una pipeline efficiente per lo screening della cristallizzazione utilizzando l'approccio microbatch-under-oil19, che utilizza modalità automatizzate di gestione dei liquidi e imaging per semplificare l'identificazione delle condizioni iniziali di cristallizzazione utilizzando volumi di campioni e cocktail relativamente minimi (Figura 1). L'insieme di 1.536 cocktail unici si basa su condizioni precedentemente determinate per favorire la crescita dei cristalli proteici e sono progettati per essere chimicamente diversi al fine di campionare una vasta gamma di possibili condizioni di cristallizzazione20,21,22. L'ampio campionamento delle condizioni di cristallizzazione aumenta la probabilità di osservare uno o più elettrocateteri di cristallizzazione.
Poche analisi formali di quante condizioni sono necessarie per lo screening sono apparse in letteratura. Uno studio si è concentrato sul layout di campionamento di diversi schermi e ha scoperto che il campionamento casuale dei componenti (simile a un fattoriale incompleto) rappresentava il metodo di campionamento più completo ed efficiente23. Un altro studio di screening ha rilevato che ci sono stati numerosi casi in cui lo schermo molto completo di 1.536 ha prodotto solo un singolo cristallo colpito24, e uno studio molto recente ha evidenziato che la maggior parte degli schermi commerciali sottocampiona lo spazio di cristallizzazione noto per essere associato allo screening raggiunge25. Non tutti i cavi di cristallizzazione produrranno un cristallo di qualità di diffrazione adatto alla raccolta di dati a causa del disordine intrinseco all'interno del cristallo, limitazioni di diffrazione o difetti cristallini; Pertanto, lanciare una rete più ampia per le condizioni ha l'ulteriore vantaggio di fornire forme cristalline alternative per l'ottimizzazione.
Anche il formato degli esperimenti di cristallizzazione delle proteine ha un impatto sul successo dello schermo. La diffusione del vapore è la configurazione più comunemente utilizzata per le applicazioni di cristallizzazione ad alto rendimento e viene utilizzata nei centri di cristallizzazione all'avanguardia, tra cui i centri di screening ad alta produttività EMBL Hamburg e Institut Pasteur26,27,28. Il Centro HTX utilizza il metodo microbatch-under-oil; Sebbene meno comunemente usato, è un metodo robusto che riduce al minimo il consumo di cocktail di campionamento e cristallizzazione20,21,22. Un vantaggio del metodo microbatch-under-oil, in particolare quando si utilizza un olio di paraffina ad alta viscosità, è che solo una leggera evaporazione si verifica all'interno della goccia durante l'esperimento, il che significa che la concentrazione di equilibrio viene raggiunta durante la miscelazione delle gocce. Se si osservano risultati positivi di cristallizzazione nel metodo microbatch-under-oil, la riproduzione di queste condizioni è tipicamente più semplice rispetto alle configurazioni di diffusione del vapore, in cui la cristallizzazione avviene in un punto indefinito durante l'equilibrio tra la goccia di cristallizzazione e il serbatoio. La riproducibilità dei colpi è auspicabile per approcci di cristallizzazione ad alto rendimento, che producono cristalli proteici proibitivamente minuscoli che in genere devono essere ottimizzati per esperimenti a raggi X a cristallo singolo.
Lo schermo di cristallizzazione ad alta produttività per le proteine solubili è costituito da cocktail preparati internamente, schermi commerciali pronti all'uso e schermi commerciali modificati internamente22. I cocktail sono stati inizialmente sviluppati utilizzando la strategia fattoriale incompleta utilizzando cocktail di cristallizzazione precedentemente di successo20. I reagenti nello schermo disponibili in commercio includono matrici di polimeri, sali di cristallizzazione, PEG e combinazioni ioniche e schermi che utilizzano approcci fattoriali sparsi e incompleti. Ci sono anche reagenti che vengono modificati prima dell'inclusione nello schermo: uno schermo additivo, uno schermo di pH e tampone, uno schermo di additivi liquidi ionici e uno schermo polimerico.
La potenza delle condizioni e delle strategie di cristallizzazione note è stata sfruttata nei 1.536 cocktail di cristallizzazione, insieme ai vantaggi del sistema microbatch-under-oil per generare una pipeline che impiega la gestione automatizzata dei liquidi, l'imaging automatizzato in campo chiaro e l'imaging non lineare del secondo ordine dei cristalli chirali (SONICC). L'automazione della gestione dei liquidi e dell'imaging offre i vantaggi di un minor numero di ore di laboratorio umido e di una maggiore riproducibilità. La natura ad alto rendimento dello screening automatizzato della cristallizzazione richiede l'automazione del processo di monitoraggio della crescita dei cristalli. Questi progressi sono ottenuti con tecnologie di imaging all'avanguardia per aiutare nell'identificazione di colpi di cristallo positivi. Sia l'imaging standard in campo chiaro delle lastre, sia i metodi multi-fotoni per il rilevamento avanzato, vengono utilizzati tramite un sistema di imaging a cristallo con SONICC (Figura 2). SONICC combina la microscopia di seconda generazione armonica (SHG)29 e la microscopia ultravioletta a fluorescenza eccitata a due fotoni (UV-TPEF)30 per rilevare cristalli molto piccoli, nonché quelli oscurati dal precipitato. L'imaging SONICC informa se i pozzetti contengono proteine (tramite UV-TPEF) e cristalli (tramite SHG). Oltre all'identificazione positiva dei cristalli proteici, è possibile ottenere ulteriori informazioni utilizzando metodi di imaging all'avanguardia. L'imaging solo cocktail prima dell'aggiunta del campione funge da controllo negativo; Queste immagini possono identificare l'aspetto del pozzo prima dell'aggiunta del campione, anche in termini di cristalli di sale e detriti. Inoltre, l'imaging SHG e UV-TPEF aiuta a differenziare i cristalli proteici dai cristalli di sale e può essere utilizzato per visualizzare il materiale complessato proteina-acido nucleico31.
Gli esperimenti di cristallizzazione ad alto rendimento sottoposti a monitoraggio ripetuto tramite imaging producono un volume molto elevato di immagini che richiedono un esame. Sono stati sviluppati metodi automatizzati di punteggio dei cristalli per ridurre l'onere per l'utente e aumentare la probabilità di identificare i colpi positivi dei cristalli. L'HTX Center ha partecipato allo sviluppo dell'algoritmo di punteggio MAchine Recognition of Crystallization Outcomes (MARCO), un'architettura di rete neurale convoluzionale profonda addestrata sviluppata da un consorzio di partner accademici, senza scopo di lucro, governativi e industriali per classificare le immagini dei pozzi a campo chiaro32. L'algoritmo è stato addestrato su quasi mezzo milione di immagini in campo chiaro da esperimenti di cristallizzazione di più istituzioni utilizzando diversi metodi di cristallizzazione e diversi imager. L'algoritmo produce un punteggio probabilistico che indica se una data immagine rientra in quattro possibili classi di immagini: "cristallo", "chiaro", "precipitato" e "altro". MARCO ha un'accuratezza di classificazione riportata del 94,5%. Il rilevamento dei cristalli è ulteriormente migliorato con un software che implementa l'algoritmo e fornisce un'interfaccia utente grafica (GUI) per una visualizzazione delle immagini accessibile e semplice, abilitata con le funzionalità di punteggio abilitate all'intelligenza artificiale32,33. La GUI MARCO Polo è progettata per funzionare perfettamente con la configurazione del sistema di imaging e gestione dei dati nel Centro HTX per identificare i riscontri nello schermo a 1.536 pozzetti, con l'impegno umano per esaminare l'output delle liste ordinate. Inoltre, come software open source disponibile su GitHub, la GUI è prontamente disponibile per la modifica per riflettere le esigenze specifiche di altri gruppi di laboratorio.
Qui, viene descritto il processo di creazione di un esperimento microbatch-under-oil ad alta produttività utilizzando la gestione robotica dei liquidi per fornire sia il cocktail che le proteine. Il Centro HTX ha una gamma unica di strumentazione e risorse che non si trovano in altre istituzioni, con l'obiettivo di fornire servizi di screening e risorse educative agli utenti interessati. Dimostrare i metodi e le capacità delle tecniche ad alto rendimento abilitate alla robotica consentirà alla comunità di avere conoscenza delle tecnologie disponibili e prendere decisioni per i propri sforzi di determinazione della struttura.
Protocol
1. Preparazione o acquisto di cocktail per sedici blocchi di pozzi profondi 96 pozzi
- Preparare cocktail chimici generati internamente erogandoli in blocchi di pozzi profondi 96 pozzetti (DW). Utilizzare un manipolatore di liquidi robotizzato per erogare e miscelare soluzioni stock di sali, tamponi, polimeri e acqua.
- Preparare cocktail chimici modificati internamente utilizzando un manipolatore di liquidi robotizzato o una pipetta multicanale per aggiungere componenti aggiuntivi agli schermi a blocchi DW a 96 pozzetti acquistati commercialmente.
- Acquista blocchi DW disponibili in commercio.
- Conservare i blocchi DW a 96 pozzetti etichettati a -20 °C per 12-18 mesi.
NOTA: I cocktail preparati al punto 1.1. e 1.2. riempire 10/16 blocchi DW a 96 pozzetti e 5/16 blocchi DW da 96 pozzetti vengono utilizzati come acquistati. Un blocco DW a 96 pozzetti nello schermo è impostato al momento dell'erogazione della piastra da 1.536 pozzetti per evitare la precipitazione dello schermo additivo (vedere sezione 3).
2. Erogazione dei cocktail in piatti da 384 pozzetti
- Scongelare i blocchi DW a 96 pozzetti a 4 °C durante la notte. Portare a temperatura ambiente (20-23 °C) prima di iniziare la preparazione delle piastre da 384 pozzetti.
NOTA: La temperatura ambiente è adatta per la preparazione dei piatti da cocktail. La preoccupazione principale nella preparazione di queste piastre è quella di evitare precipitati, che possono ostruire i dispositivi di manipolazione dei liquidi e portare a cambiamenti imprevedibili nelle concentrazioni degli ingredienti del cocktail. - Mescolare accuratamente i blocchi per inversione secondo necessità per dissolvere qualsiasi precipitato opaco persistente. Se i pozzetti contengono precipitato, riscaldare i blocchi a 30 °C per dissolverli.
- Erogare 50 μL di soluzione cocktail da quattro blocchi DW da 96 pozzetti a una piastra da 384 pozzetti utilizzando un robot per la gestione dei liquidi dotato di una siringa da 96 o di una testa di pipettatore. I quattro blocchi DW a 96 pozzetti sono stampati nella piastra a 384 pozzetti in modo tale che i quadranti siano riempiti (ad esempio, A1 di 96-DW1 a A1 di 384-plate1, A1 di 96-DW2 a B1 di 384-plate1, ecc.) (Figura 3).
- Consegna di 15 dei 16 blocchi DW da 96 pozzetti a piastre da 384 pozzetti per lo stoccaggio.
- Conservare le piastre da 384 pozzetti a -20 °C per un massimo di 6 mesi per l'uso nella preparazione delle piastre da 1.536 pozzetti.
3. Preparazione delle piastre da 1.536 pozzetti con olio e cocktail di cristallizzazione
- Fornire 5 μL di olio di paraffina a ciascun pozzetto di una piastra da 1.536 pozzetti utilizzando un sistema robotizzato di gestione dei liquidi con la capacità di aspirazione e consegna lente. Conservare le piastre d'olio a 4 °C per un massimo di 6 mesi.
- Scongelare le piastre da 384 pozzetti dalla sezione 2 a 4 °C durante la notte. Capovolgere le piastre per mescolare le soluzioni e sciogliere il precipitato. Incubare le piastre a 30 °C per sciogliere i precipitati persistenti.
- Per preparare i componenti dello schermo additivo, utilizzare il blocco DW finale a 96 pozzetti contenente 0,1 M HEPES pH 6,8, 30% PEG3350 da miscelare con lo schermo additivo commerciale utilizzando un robot di gestione dei liquidi o una pipetta multicanale.
- Preparare le soluzioni di vaglio additivo erogando una miscela 1:1 della soluzione tamponata PEG3350 preparata nella fase 3.3 e del vaglio additivo ad un volume finale di 50 μL nella piastra appropriata da 384 pozzetti.
- Utilizzare un robot per la gestione dei liquidi dotato di una siringa 384 o di una testa per pipettor per erogare 200 nL di soluzione per cocktail in ciascun pozzetto della piastra da 1.536 pozzetti. Eliminare quattro piastre da 384 pozzetti nella piastra da 1.536 pozzetti in modo tale che i quadranti siano riempiti (ad esempio, A1 della piastra da 384 a 1.536 pozzetti, A2 della piastra da 384 pozzetti1 ad A3 della piastra da 1.536 pozzetti, ecc.) (Figura 3).
- Centrifugare le piastre a 150 × g per 5 minuti prima di conservare a 4 °C per un massimo di 4 settimane.
4. Invio del campione
- Per inviare un campione, invia un'e-mail di prenotazione prima della scadenza della prenotazione per la prossima corsa di screening. Includere il numero di esperimenti di screening, il nome, il PI e l'istituzione, nonché eventuali requisiti speciali di gestione per il campione. Le corse di screening vengono condotte circa una volta al mese, risultando in 12 corse all'anno.
- Compila un modulo di invio del campione prima di spedire il campione.
- Per i nuovi utenti, scegliere una password che verrà utilizzata per scaricare le immagini di cristallizzazione nella sezione 7.
- Per gli utenti consolidati, utilizzare una password esistente o modificare la password in questo passaggio.
- Inviare il campione in una provetta da 1,5 ml. Assicurarsi che la macromolecola sia omogenea e adeguatamente concentrata per favorire la cristallizzazione. Utilizzare un test di pre-cristallizzazione, tipicamente composto da solfato di ammonio o PEG 4.000, per indagare la concentrazione appropriata del campione osservando se le concentrazioni del campione testato provocano gocce chiare o precipitano34.
NOTA: I test di qualità appropriati che possono essere eseguiti prima della presentazione del campione per verificare la purezza e l'omogeneità includono SDS-PAGE, filtrazione su gel e diffusione dinamica della luce (DLS), tra gli altri. La cristallizzazione può essere influenzata dalla presenza di impurità anche minori. Attualmente è necessario un volume di campione di 500 μL per installare una piastra da 1.536 pozzetti. Sono in corso test per ridurre il requisito di volume del campione.- Evitare l'uso di concentrazioni tampone superiori a 50 mM, nonché fosfati, che potrebbero cristallizzare all'interno dello schermo.
- Evitare eccessivi agenti solubilizzanti, comprese concentrazioni di glicerolo superiori al 10% p/v.
- Imballare il campione per mantenere in modo sicuro una temperatura appropriata utilizzando ghiaccio secco, ghiaccio umido o pacchetti di raffreddamento in un contenitore sigillato.
- Spedisci la priorità del campione durante la notte dal lunedì al mercoledì durante la corsa.
- Invia via email il numero di tracciamento una volta che il campione è stato spedito.
5. Configurazione del campione nelle piastre da 1.536 pozzetti preparate
- Disimballare i campioni e incubarli immediatamente alla temperatura richiesta dall'utente.
- Una volta scongelato, centrifugare il campione a 10.000 × g per 2 minuti a temperatura ambiente. Osservare visivamente il campione per identificare le precipitazioni, il colore e le condizioni del campione prima della configurazione.
- Riscaldare la piastra da 1.536 pozzetti a 23 °C e centrifugare a 150 × g per 5 min. Immagina il piatto solo cocktail usando l'imaging a campo chiaro come controllo negativo.
NOTA: Tutte le lastre vengono visualizzate con imaging in campo chiaro prima della configurazione del campione, che consente l'identificazione di pozzi che contengono già cristalli o detriti prima dell'aggiunta del campione come controllo negativo. Inoltre, consente l'identificazione di pozzi in cui il cocktail di cristallizzazione non è stato consegnato. Il riscaldamento della piastra a temperatura ambiente elimina la condensa sulla superficie della piastra, portando a immagini nitide. - Erogare 200 nL di campione a ciascun pozzetto nella piastra da 1.536 pozzetti utilizzando un robot per la manipolazione dei liquidi. Centrifugare la piastra a 150 × g e incubare le piastre a 4 °C, 14 °C o 23 °C.
NOTA: Gli esperimenti di microbatch under-oil possono essere impostati a mano erogando la proteina e il cocktail sotto l'olio desiderato. Tuttavia, si raccomanda di utilizzare non meno di 1 μL di ciascuna proteina e cocktail per ottenere risultati riproducibili.
6. Monitorare le piastre da 1.536 pozzetti per la formazione di cristalli
- Dopo che il campione è stato aggiunto alle piastre da 1.536 pozzetti, immagine con imaging in campo chiaro al giorno 1 e alla settimana 1, alla settimana 2, alla settimana 3, alla settimana 4 e alla settimana 6.
- Eseguire l'imaging SONICC con SHG e UV-TPEF al punto temporale di 4 settimane per le piastre incubate a 23 °C e al punto temporale di 6 settimane per le piastre incubate a 14 °C o 4 °C.
NOTA: la tempistica per l'imaging SONICC è programmata ai punti temporali di 4 settimane e 6 settimane per la piastra di microanalisi a 1.536 ad alta produttività perché, in genere, i cristalli appariranno in base a quei punti temporali. Per la modifica di un microbatch a 96 pozzetti sotto esperimenti di diffusione di olio o vapore, è consigliabile eseguire l'imaging SONICC prima nella finestra temporale. - Accedere alle immagini sperimentali che sono state automaticamente trasferite all'account utente utilizzando un sistema LIMS interno. Notifica agli utenti tramite un demone e-mail htslab automatizzato che l'imaging è avvenuto.
7. Analisi delle immagini
- Recuperare le immagini di screening dal sito ftp HWI per ogni file .rar.
NOTA: l'output dell'immagine dallo schermo 1.536 produce una serie di file contenenti immagini in campo chiaro, immagini SHG e immagini UV-TPEF. Ogni modalità di imaging o punto temporale è un file .rar separato. Ogni file .rar, una volta decompresso, contiene un'immagine da ciascun pozzetto della piastra da 1.536 pozzetti in un punto temporale specifico utilizzando una specifica modalità di imaging.- Utilizzare il client FileZilla o altre opzioni per accedere ai dati ftp.
NOTA: FileZilla Client è il modo consigliato per gestire il volume di trasferimento di file di grandi dimensioni per ridurre al minimo gli arresti anomali computazionali.- Se FileZilla Client deve essere installato sul computer dell'utente, scaricare il software FileZilla.
- Se FileZilla Client è già installato o al momento dell'installazione, fare clic sull'icona FileZilla per aprire il software.
- Accedi al server ftp remoto da FileZilla inserendo il sito Web ftp host, il nome utente e la password.
- Scaricare i file .rar nella directory desiderata.
- Utilizzare il client FileZilla o altre opzioni per accedere ai dati ftp.
- Utilizza la GUI open source abilitata per l'intelligenza artificiale per visualizzare, assegnare un punteggio e analizzare le immagini di cristallizzazione.
NOTA: la GUI può essere utilizzata sulla maggior parte dei sistemi operativi Windows, Mac e Linux e le istruzioni specifiche del sistema operativo per il download si trovano sul sito GitHub. MARCO Polo è una GUI open source che incorpora i metadati dello schermo di cristallizzazione 1.536 ad alto throughput implementato presso l'HTX Center. È disponibile per chiunque possa essere scaricato da GitHub per la modifica in modo da riflettere le esigenze specifiche di altri gruppi di laboratorio.- Aprire il file .rar nella GUI dopo che il file è stato scaricato (vedere la figura supplementare S1).
- Fare clic su Importa, selezionare Immagini dal menu a discesa, quindi selezionare Da archivio / directory Rar.
- Fare clic su Cerca cartella nella finestra popup, quindi accedere alla cartella contenente le immagini.
- Selezionare i file desiderati e importarli nella GUI facendo clic su Apri. Attendere che i file vengano visualizzati nella finestra Percorsi selezionati . Selezionare uno o più file da scaricare nella GUI e fare clic su Importa esecuzioni.
- Visualizza l'immagine per il primo pozzetto nella finestra del Visualizzatore presentazioni nella GUI facendo clic sul simbolo > a sinistra del nome del campione e quindi selezionando la lettura appropriata facendo doppio clic su di esso (le letture sono elencate in base alla data e al tipo di campo chiaro dell'immagine, UV-TPEF o SHG).
- Ingrandisci l'immagine ridimensionando l'intera finestra. La casella Dettagli immagine include informazioni sull'immagine , incluse le informazioni sul punteggio (vuoto fino a quando la lettura non è stata valutata). La casella Dettagli cocktail contiene metadati sui componenti del cocktail .
- Passare al riquadro successivo facendo clic sul pulsante Avanti nel pannello di navigazione o premendo il tasto freccia destra sulla tastiera. Passare a un pozzo specifico immettendo il numero del pozzo nella finestra Per numero pozzo .
- Visualizza tutte le letture (di quelle importate nella GUI) selezionando la casella Mostra tutte le date .
- Visualizza tutti gli spettri (di quelli importati nella GUI) selezionando la casella Mostra tutti gli spettri . Fare clic sul pulsante Scambia spettro per visualizzare singolarmente ogni immagine dello spettro.
- Aprire il file .rar nella GUI dopo che il file è stato scaricato (vedere la figura supplementare S1).
- Assegna un punteggio alle immagini di cristallo utilizzando l'algoritmo MARCO evidenziando prima una corsa specifica dall'elenco sul lato sinistro della finestra. Quindi, fai clic sul pulsante Classifica esecuzione selezionata . Visualizza le informazioni sul punteggio MARCO nella finestra Dettagli immagine una volta che è stata valutata una lettura dell'imaging per tutti i 1.536 pozzi.
NOTA: la classificazione richiede in genere da 2 a 5 minuti, a seconda della velocità del computer e della memoria disponibile. L'algoritmo genera punteggi che classificano i contenuti in classi "crystal", "clear", "precipitate" o "other". I valori numerici associati alla classificazione di ciascun pozzo riflettono la probabilità che il pozzo contenga oggetti di tale classe.- Visualizza un sottoinsieme delle immagini con punteggio spuntando le caselle desiderate nel pannello Filtro immagini e facendo clic sul pulsante Invia filtri . Ad esempio, visualizzare solo le immagini classificate da MARCO come cristalli spuntando le caselle Cristalli e MARCO e facendo clic su Invia filtri.
- Segnare manualmente le immagini di cristallo per generare il set "punteggio umano". Assegna un punteggio a un pozzo facendo clic sul pulsante appropriato (i pulsanti "cristallo", "chiaro", "precipita" o "altro" si trovano nel pannello Classificazione nella parte inferiore della finestra). In alternativa, utilizzare il tastierino numerico sulla tastiera per assegnare il punteggio (1 = "cristallo", 2 = "chiaro", 3 = "precipitare", 4 = "altro"). Designare un'immagine con punteggio umano come "preferita" spuntando il preferito? scatola.
NOTA: Visualizza solo le immagini classificate da un essere umano come cristalli spuntando le caselle Cristalli e Umani e facendo clic su Invia filtri. Facendo clic sulla casella Preferiti nel pannello Filtro si restringono ulteriormente le immagini restituite, restituendo solo le immagini di cristallo con punteggio umano che sono anche preferite. - Utilizzare la scheda Visualizzatore piastre per visualizzare più pozzi contemporaneamente. Nella seconda scheda Visualizzatore lastre del pannello Controlli , selezionate 16, 64 o 96 immagini dal menu a discesa nella sezione Immagini per piatto . Utilizzare la scheda Filtro immagini per disattivare le immagini che non sono di interesse. Selezionare la casella Applica filtro per filtrare le immagini.
NOTA: Ad esempio, selezionare le caselle "umano" e "cristallo" e solo i pozzi che sono stati segnati come cristallo da un essere umano saranno facilmente visibili.- Navigare nella scheda Visualizzatore lastre , facendo clic sul pulsante Avanti per visualizzare il prossimo set di immagini 16/64/96. Per impostazione predefinita, le immagini contrassegnate come cristalli sono rosse, quelle contrassegnate come chiare sono blu, quelle segnate come precipitate sono verdi e quelle segnate come altre sono arancioni. Modificare i colori utilizzando i menu a discesa.
- Selezionare le informazioni da visualizzare sui pozzetti spuntando varie caselle nella scheda Etichette .
- Fare clic su Salva vista per salvare un file immagine della vista corrente.
- Fare clic su Scambia spettro per alternare tra immagini a campo chiaro, SHG e UV-TPEF per l'immagine a pozzetti multipli.
- Fare clic su Esporta e selezionare il tipo di file appropriato dal menu a discesa per esportare i file con punteggio per l'utilizzo in altri programmi.
NOTA: i file CSV (valori separati da virgole) sono compatibili con programmi di fogli di calcolo come Microsoft Excel o Fogli Google. I file JSON (JavaScript Object Notation) possono essere aperti con la maggior parte degli editor di testo. PPTX (PowerPoint Presentation) può essere utilizzato per visualizzare immagini da Polo, incluso un confronto di immagini a campo chiaro, UV-TPEF e SHG. I file vengono salvati in formato .xtal per essere riaperti nella GUI di MARCO Polo.- Salvare un file in formato .xtal facendo clic su File nella parte superiore della pagina e selezionando Salva esecuzione o Salva esecuzione con nome. Specificare un nome file e un percorso di directory.
- Apri i file in formato .xtal facendo clic su Importa e selezionando Immagini e quindi Da esecuzione salvata. Cercare il percorso del file appropriato, fare clic sul nome del file, quindi fare clic su Apri.
Representative Results
I risultati dell'esperimento di screening dei cristalli a 1.536 pozzetti consistono in sette set completi di immagini in campo chiaro raccolti al giorno 0 (controllo negativo), giorno 1, settimana 1, settimana 2, settimana 3, settimana 4 e settimana 6 (Figura 4). Le immagini SONICC vengono raccolte al punto temporale di 4 settimane per le piastre incubate a 23 °C e al punto temporale di 6 settimane per le piastre incubate a 4 °C o 14 °C. Complessivamente, una volta che un campione è stato spedito, gli utenti possono anticipare di installare le lastre entro 1 giorno dall'arrivo. Le immagini verranno caricate man mano che vengono raccolte. L'esperimento di screening della cristallizzazione si conclude dopo 6 settimane.
La configurazione della piastra a 1.536 pozzetti consente di condurre tutti gli esperimenti di screening all'interno della stessa piastra, limitando così il consumo del campione e facilitando l'imaging e il confronto diretto tra le modalità di imaging. I risultati rappresentativi per il corso temporale della crescita dei cristalli per una singola condizione di cocktail sono mostrati nella Figura 4. L'imaging automatico delle lastre nel corso dell'esperimento consente l'identificazione di cristalli a crescita rapida e lenta mediante imaging in campo chiaro. L'imaging UV-TPEF e SHG consente la convalida incrociata dei colpi osservati dall'imaging in campo chiaro e indicano che i cristalli osservati sono rispettivamente proteici e cristallini (Figura 5A,B). Inoltre, l'imaging SONICC consente l'identificazione di cristalli che sono visivamente oscurati da precipitati o pellicole (Figura 5C) o microcristalli che potrebbero altrimenti essere scambiati per precipitati (Figura 5D). Per alcuni cristalli, la mancanza di segnale SHG non è squalificante, poiché alcuni gruppi di punti non producono un segnale SHG35,36, come esemplificato dal cristallo tetragonale di taumatina nella Figura 5C. Al contrario, dovrebbe essere anticipata una mancanza di segnale UV-TPEF per le proteine prive di residui di triptofano. L'osservazione dei segnali UV-TPEF e SHG facilita anche l'identificazione di cristalli di sale non proteici, che appariranno in campo luminoso e mostreranno un forte segnale SHG positivo ma mancheranno di un segnale UV-TPEF (Figura 5E).
L'analisi delle immagini per la configurazione della piastra è semplificata con la GUI MARCO Polo, che raggruppa anche il trasferimento dei dati ftp dai server HWI (in alternativa al trasferimento di file con FileZilla). La GUI MARCO Polo consente una visualizzazione facilmente navigabile di lastre e immagini ed esegue il punteggio computazionale delle immagini utilizzando l'algoritmo MARCO in modo che i risultati delle immagini possano essere rapidamente scaricati, visualizzati e analizzati dal Centro HTX. L'algoritmo di punteggio MARCO, implementato nella GUI MARCO Polo, è in grado di segnare immagini dall'intera piastra da 1.536 pozzetti in meno di 5 minuti. Le immagini contrassegnate come cristalline dall'algoritmo MARCO possono essere successivamente ordinate dalla GUI del Polo per la visualizzazione. Poiché l'algoritmo MARCO è stato ottimizzato per l'identificazione dei cristalli e per ridurre al minimo i falsi negativi in modo da non perdere alcun risultato positivo, il punteggio può comportare false flag positive. Tuttavia, la capacità di MARCO di limitare l'insieme di immagini da esaminare focalizzando l'attenzione sui pozzi con un'alta probabilità di contenere cristalli si traduce in una sostanziale riduzione del carico di elaborazione dei dati per gli utenti. La comoda implementazione dell'algoritmo nella piattaforma di visualizzazione MARCO Polo di facile utilizzo, con la sua capacità di ordinare le immagini in base ai punteggi MARCO, migliora notevolmente la capacità dell'utente di analizzare rapidamente il set di dati e di determinare con precisione i colpi di cristallo.
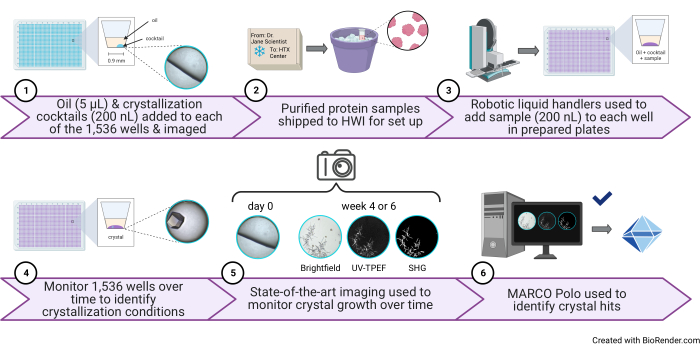
Figura 1: Schema di un esperimento di screening di cristallizzazione ad alta produttività di 1.536 pozzetti eseguito presso il Centro HTX. (1) In questa fase, 5 μL di olio di paraffina e 200 nL di cocktail vengono aggiunti a ciascun pozzetto (fase 3.1 e fase 3.5 del protocollo). Un'illustrazione a fumetti di un pozzo contenente solo olio e cocktail e un'immagine rappresentativa sono mostrati a destra. (2) I campioni arrivano al Centro HTX (fase 5.1 del protocollo). 3) In questa fase, 200 nL di campione vengono aggiunti a ciascun pozzetto (fase 5.4 del protocollo). (4) Tutti i 1.536 pozzi sono monitorati nel tempo utilizzando l'imaging in campo chiaro, 5) così come le modalità UV-TPEF e SHG (fase 6 del protocollo). 6) La GUI open source abilitata all'intelligenza artificiale viene utilizzata per visualizzare, valutare e analizzare le immagini di cristallizzazione (passaggio del protocollo 7). Abbreviazioni: HTX = cristallizzazione ad alta produttività; UV-TPEF = fluorescenza eccitata UV-due-fotoni; SHG = seconda generazione armonica; AI = intelligenza artificiale; GUI = interfaccia utente grafica. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 2: Singole piastre da 1.536 pozzetti contenenti esperimenti di screening, riprese utilizzando l'imaging in campo chiaro, UV-TPEF e SHG. Le piastre da 1.536 pozzetti sono mostrate con un penny americano per scala (in alto). Ogni esperimento di screening viene ripreso una volta prima della configurazione e sei volte dopo l'aggiunta del campione con imaging in campo chiaro (sette set di immagini in campo chiaro totali, a sinistra). Le piastre vengono sottoposte a imaging UV-TPEF (al centro) e SHG (a destra) a 4 settimane o 6 settimane. Abbreviazioni: UV-TPEF = fluorescenza eccitata UV-due-fotoni; SHG = seconda generazione armonica. Fare clic qui per visualizzare una versione ingrandita di questa figura.
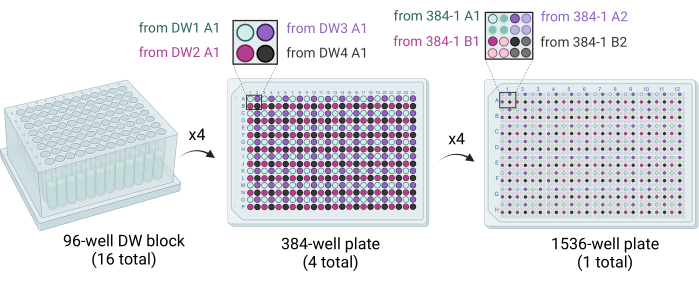
Figura 3: Schema che mostra come vengono generate le piastre da 1.536 pozzetti. Sedici blocchi DW da 96 pozzetti vengono utilizzati per eliminare quattro piastre da 384 pozzetti, con ogni quadrante di ogni piastra da 384 pozzetti riempita erogando cocktail di cristallizzazione. Quattro blocchi DW da 96 pozzetti riempiono una piastra da 384 pozzetti (al centro). Quattro piastre da 384 pozzetti vengono utilizzate per eliminare la singola piastra da 1.536 pozzetti (a destra). Abbreviazione: DW = pozzo profondo. Fare clic qui per visualizzare una versione ingrandita di questa figura.
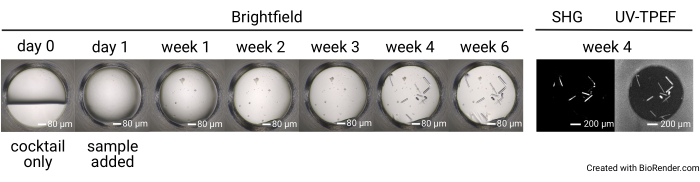
Figura 4: Andamento temporale rappresentativo di un singolo pozzo in un esperimento di screening a 1.536 pozzetti. Le immagini delle lastre vengono visualizzate prima della configurazione del campione (giorno 0), nonché con l'imaging in campo chiaro il giorno 1, la settimana 1, la settimana 2, la settimana 3, la settimana 4 e la settimana 6. Le piastre incubate a 23 °C vengono visualizzate con SONICC alla settimana 4. Barre di scala = 80 μm (campo luminoso), 200 μm (SHG, UV-TPEF). Abbreviazioni: SONICC = imaging non lineare del secondo ordine di cristalli chirali; UV-TPEF = fluorescenza eccitata UV-due-fotoni; SHG = seconda generazione armonica. Fare clic qui per visualizzare una versione ingrandita di questa figura.
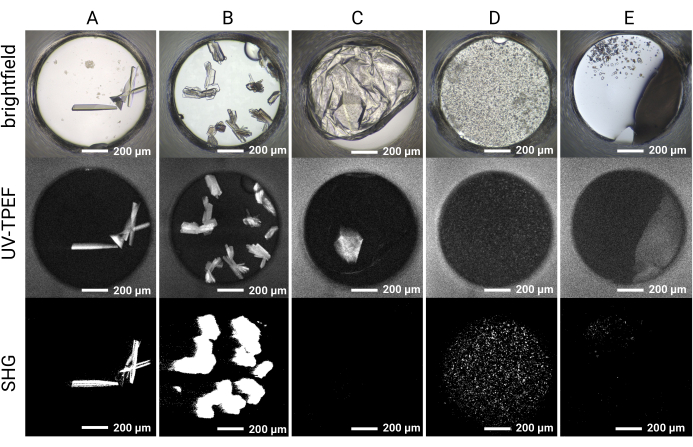
Figura 5: Risultati rappresentativi dell'imaging per gli esperimenti di screening dei cristalli HT 1.536. I risultati delle immagini Brightfield, UV-TPEF e SHG sono mostrati per cinque pozzi di esempio. (A,B) I cristalli proteici osservati dall'imaging in campo chiaro, UV-TPEF e SHG sono chiaramente evidenti in tutte e tre le modalità di imaging. (C) Un cristallo proteico oscurato dalla pellicola nell'imaging in campo chiaro è visibile mediante imaging UV-TPEF; il cristallo non viene osservato dall'imaging SHG a causa dell'incompatibilità del gruppo puntiforme. (D) Esempio di microcristalli verificati mediante imaging UV-TPEF e SHG che potrebbero altrimenti essere considerati precipitati. (E) Esempio di cristalli di sale che appaiono cristallini in campo chiaro e dall'imaging SHG ma non mostrano un segnale UV-TPEF. Barre della scala = 200 μm. Diametro del pozzo = 0,9 mm. Abbreviazioni: UV-TPEF = fluorescenza eccitata UV-due-fotoni; SHG = seconda generazione armonica. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Figura supplementare S1: Apertura di file immagine in MARCO Polo. I file di immagine possono essere aperti all'interno della GUI di MARCO Polo accedendo a Importa | Scheda Immagini in alto (a). Si noti che i file possono anche essere trasferiti tramite lo strumento From FTP direttamente in MARCO Polo (a) o possono essere trasferiti tramite FileZilla come descritto nel passaggio del protocollo 7.2. Per importare file già scaricati, selezionare Immagini | Da Rar Archive/Directory. Nella finestra popup visualizzata, selezionare Cerca cartella (b) e passare alla directory dei file in cui sono salvati i file dell'immagine della lastra. Una volta che i file si trovano nella finestra Percorsi selezionati (c), evidenziare un file e fare clic su Importa esecuzioni (d). La GUI di MARCO Polo identificherà i metadati corretti del Cocktail File da importare con le immagini. Clicca qui per scaricare questo file.
Discussion
Il metodo descrive una pipeline ad alto rendimento per lo screening della cristallizzazione delle proteine che richiede solo 500 μL di campione per 1.536 singoli esperimenti di cristallizzazione nel formato microbatch-under-oil. La pipeline si basa sulla robotica per la gestione dei liquidi per aiutare in modo rapido e riproducibile la configurazione sperimentale, nonché sulla risorsa di analisi computazionale delle immagini MARCO Polo, che è personalizzata per analizzare le immagini di lastre a 1.536 pozzetti utilizzando l'algoritmo MARCO per identificare e isolare i colpi di cristallo.
Il piccolo volume di singole gocce di screening (400 nL totali con un rapporto 1:1 di campione:cocktail) significa che sono necessari volumi di campione estremamente piccoli per identificare le condizioni di cristallizzazione positive. Queste piccole dimensioni delle gocce producono necessariamente piccoli cristalli che non possono essere pescati con il looping tradizionale. Sono stati sviluppati metodi per raccogliere dalle 1.536 piastre37; Inoltre, le piastre con cristalli sono state utilizzate direttamente alle sorgenti di sincrotrone per la raccolta di dati in situ 38. Se venisse sviluppato un metodo robusto per la raccolta di questi cristalli, i progressi nella tecnologia del sincrotrone e nei fasci micro-focalizzati consentirebbero di ottenere ulteriori set di dati utili. Inoltre, i cristalli ottenuti potrebbero potenzialmente essere utilizzati come semi per gli sforzi di ottimizzazione.
L'imaging SONICC è chiaramente vantaggioso nell'identificare sia i piccoli cristalli proteici che i cristalli proteici nascosti sotto il precipitato. Nonostante questi vantaggi, non tutti i tipi di campioni sono suscettibili di imaging SHG e UV-TPEF. Ad esempio, le proteine con pochi o nessun residuo aromatico di triptofano mostreranno un segnale UV-TPEF ambiguo. Inoltre, i cristalli in specifici gruppi spaziali, inclusi i gruppi centrosimmetrici o il gruppo di punti 432, non saranno rilevati dall'imaging SHG. I campioni con fluorofori a volte interferiscono con il segnale SHG, con conseguente cancellazione del segnale o aumento dell'intensità, il che significa che è necessaria un'attenta interpretazione dei segnali SHG per le proteine contenenti metalli e le proteine contenenti porzioni fluorescenti. Tuttavia, in molti casi, è possibile razionalizzare l'assenza di un segnale SHG o UV-TPEF e la mancanza di questi segnali non dovrebbe necessariamente escludere la presenza di un cristallo proteico.
Il formato microbatch-under-oil fornisce un'alternativa al più comune metodo di diffusione del vapore utilizzato per la cristallografia ad alto rendimento. È importante sottolineare che il formato di cristallizzazione influisce sull'identificazione hit39, che fornisce una logica per l'uso di diversi formati di cristallizzazione per gli sforzi di screening ad alto rendimento. L'imaging automatizzato e le modalità abilitate da SONICC aiutano nella rapida identificazione dei cristalli proteici durante il periodo sperimentale di 6 settimane. Infine, la GUI MARCO Polo consente agli utenti di analizzare rapidamente le immagini da 1.536 condizioni per identificare pozzi di successo promettenti per l'ottimizzazione. Le capacità dell'HTX Center, tra cui la configurazione sperimentale ad alto rendimento abilitata alla robotica, insieme agli strumenti di imaging e computazionali all'avanguardia per le analisi, forniscono un importante contributo alla comunità della biologia strutturale consentendo ai ricercatori di affrontare efficacemente un collo di bottiglia primario nel lavoro strutturale basato sui cristalli: trovare condizioni di cristallizzazione.
Disclosures
Gli autori non hanno interessi finanziari concorrenti o altri conflitti di interesse.
Acknowledgements
Vorremmo estendere la nostra gratitudine ai nostri utenti per averci affidato i loro preziosi campioni per lo screening dei cristalli, nonché per aver fornito feedback critici e richieste che ci hanno aiutato a perfezionare e sviluppare le nostre risorse per servire meglio la comunità della biologia strutturale. Vorremmo anche ringraziare Ethan Holleman, Dr. Lisa J Keefe e Dr. Erica Duguid, che hanno guidato lo sviluppo della MARCO Polo GUI. Vorremmo ringraziare i colleghi HWI per il loro supporto e suggerimenti, in particolare la dott.ssa Diana CF Monteiro. Riconosciamo il sostegno finanziario del National Institutes of Health, R24GM141256.
Materials
| Name | Company | Catalog Number | Comments |
| 1536 Well Imp@ct LBR LoBase | Greiner Bio-One | 790 801 | |
| Acetic acid | Hampton Research | HR2-853 | |
| AlumaSeal II Sealing Film | Hampton Research | HR8-069 | |
| Ammonium bromide | Molecular Dimensions | MD2-100-247 | |
| Ammonium chloride | Hampton Research | HR2-691 | |
| Ammonium hydroxide | Hampton Research | HR2-855 | |
| Ammonium nitrate | Hampton Research | HR2-665 | |
| Ammonium phosphate dibasic | Hampton Research | HR2-629 | |
| Ammonium phosphate monobasic | Hampton Research | HR2-555 | |
| Ammonium sulfate | Hampton Research | HR2-541 | |
| Ammonium thiocyanate | Molecular Dimensions | MD2-100-301 | |
| Bicine pH 9.0 | Hampton Research | HR2-723 | |
| Bis-tris propane pH 7.0 | Hampton Research | HR2-993-08 | |
| Calcium acetate | Hampton Research | HR2-567 | |
| Calcium chloride dihydrate | Hampton Research | HR2-557 | |
| CAPS pH 10.0 | Rigaku Reagents | none given | |
| ClearSeal Film | Hampton Research | HR4-521 | |
| Cobalt sulfate heptahydrate | Molecular Dimensions | MD2-100-42 | |
| Crystal Screen HT screen | Hampton Research | HR2-130 | |
| Formulator | Formulatrix | ||
| Glycerol | Hampton Research | HR2-623 | |
| Gryphon liquid handling robot | Art Robbins Instruments | ||
| HEPES pH 7.0 | Hampton Research | HR2-902-03 | |
| HEPES pH 7.5 | Hampton Research | HR2-902-08 | |
| HWI HTX Center sample submission form | https://hwi.buffalo.edu/high-throughput-crystallization-screening-center-sample-submission-form/ | ||
| Hydrochloric acid | Hampton Research | HR2-581 | |
| Index HT screen | Hampton Research | HR2-134 | |
| Ionic Liquid screen | Hampton Research | HR2-214 | |
| Lithium bromide | Molecular Dimensions | MD2-100-312 | |
| Lithium chloride | Hampton Research | HR2-631 | |
| Lithium sulfate monohydrate | Hampton Research | HR2-545 | |
| Magnesium acetate tetrahydrate | Hampton Research | HR2-561 | |
| Magnesium chloride hexahydrate | Hampton Research | HR2-559 | |
| Magnesium nitrate hexahydrate | Hampton Research | HR2-657 | |
| Magnesium sulfate heptahydrate | Hampton Research | HR2-821 | |
| Manganese chloride tetrahydrate | Millipore Sigma | 63535-50G | |
| Manganese sulfate monohydrate | Molecular Dimensions | MD2-100-310 | |
| MARCO Polo GUI download | https://hauptman-woodward.github.io/Marco_Polo/ | ||
| Matrix Platemate 2 x 3 liquid handling robot | Thermo Scientific | ||
| MES pH 6.0 | Hampton Research | HR2-943-09 | |
| Mosquito liquid handling robot | SPTLabtech | ||
| Paraffin Oil/White Mineral Oil Saybolt Viscosity 340-365 at 100 °F | Sigma Aldrich | PX0045-3 | |
| PEG 1000 | Hampton Research | HR2-523 | |
| PEG 2000 | Hampton Research | HR2-592 | |
| PEG 20000 | Hampton Research | HR2-609 | |
| PEG 3350 | Hampton Research | HR2-527 | |
| PEG 400 | Hampton Research | HR2-603 | |
| PEG 4000 | Hampton Research | HR2-529 | |
| PEG 6000 | Hampton Research | HR2-533 | |
| PEG 8000 | Hampton Research | HR2-535 | |
| PEG/Ion HT screen | Hampton Research | HR2-139 | |
| PEGRx HT screen | Hampton Research | HR2-086 | |
| Plate reservations | htslab@hwi.buffalo.edu | ||
| Potassium acetate | Hampton Research | HR2-671 | |
| Potassium bromide | Hampton Research | HR2-779 | |
| Potassium carbonate | Molecular Dimensions | MD2-100-311 | |
| Potassium chloride | Hampton Research | HR2-649 | |
| Potassium nitrate | Hampton Research | HR2-663 | |
| Potassium phosphate dibasic | Hampton Research | HR2-635 | |
| Potassium phosphate-monobasic | Hampton Research | HR2-553 | |
| Potassium phosphate-tribasic | Molecular Dimensions | MD2-100-309 | |
| Potassium thiocyanate | Hampton Research | HR2-695 | |
| Rock Imager 1000 with SONICC | Formulatrix | ||
| Rock Imager 54 | Formulatrix | ||
| Rubidium chloride | Millipore Sigma | R2252-10G | |
| SaltRx HT screen | Hampton Research | HR2-136 | |
| Silver Bullets screen | Hampton Research | HR2-096 | |
| Slice pH screen | Hampton Research | HR2-070 | |
| Sodium acetate pH 5.0 | Hampton Research | HR2-933-15 | |
| Sodium bromide | Hampton Research | HR2-699 | |
| Sodium chloride | Hampton Research | HR2-637 | |
| Sodium citrate pH 4.2 | Hampton Research | HR2-935-01 | |
| Sodium citrate pH 5.6 | Hampton Research | HR2-735 | |
| Sodium hydroxide | Hampton Research | HR2-583 | |
| Sodium molybdate dihydrate | Molecular Dimensions | MD2-100-207 | |
| Sodium nitrate | Hampton Research | HR2-661 | |
| Sodium phosphate monobasic | Hampton Research | HR2-551 | |
| Sodium thiosulfate pentahydrate | Molecular Dimensions | MD-100-307 | |
| StockOptions Polymer screen | Hampton Research | HR2-227 | |
| Tacsimate pH 7 | Hampton Research | HR2-755 | |
| TAPS pH 9.0 | bioWORLD | 40121071 | |
| Tris pH 8 | Hampton Research | HR2-900-11 | |
| Tris pH 8.5 | Hampton Research | HR2-725 | |
| ViaFLO 384 | Integra | ||
| ViaFLO 384 384 channel pipettor head (0.5-12.5µL) | Integra | ||
| ViaFLO 384 96 channel pipettor head (300µL) | Integra | ||
| Zinc acetate dihydrate | Hampton Research | HR2-563 |
References
- PDB data distribution by experimental method and molecular type. RCSB Protein Data Bank Available from: https://www.rcsb.org/stats/summary (2022)
- Maveyraud, L., Mourey, L. Protein X-ray crystallography and drug discovery. Molecules. 25 (5), 1030 (2020).
- Dupeux, F., Röwer, M., Seroul, G., Blot, D., Márquez, J. A. A thermal stability assay can help to estimate the crystallization likelihood of biological samples. Acta Crystallographica Section D: Biological Crystallography. 67 (11), 915-919 (2011).
- Elbasir, A., et al. DeepCrystal: A deep learning framework for sequence-based protein crystallization prediction. Bioinformatics. 35 (13), 2216-2225 (2019).
- Zucker, F. H., et al. Prediction of protein crystallization outcome using a hybrid method. Journal of Structural Biology. 171 (1), 64-73 (2010).
- George, A., Wilson, W. W. Predicting protein crystallization from a dilute solution property. Acta Crystallographica Section D: Biological Crystallography. 50 (4), 361-365 (1994).
- Jia, Y., Liu, X. -. Y. From surface self-assembly to crystallization: prediction of protein crystallization conditions. The Journal of Physical Chemistry B. 110 (13), 6949-6955 (2006).
- Slabinski, L., et al. XtalPred: A web server for prediction of protein crystallizability. Bioinformatics. 23 (24), 3403-3405 (2007).
- Abrahams, G. J., Newman, J. BLASTing away preconceptions in crystallization trials. Acta Crystallographica Section F: Structural Biology Communications. 75 (3), 184-192 (2019).
- Rosa, N., et al. Tools to ease the choice and design of protein crystallisation experiments. Crystals. 10 (2), 95 (2020).
- Newman, J., et al. On the need for an international effort to capture, share and use crystallization screening data. Acta Crystallographica Section F: Structural Biology and Crystallization Communications. 68 (3), 253-258 (2012).
- Lynch, M. L., Dudek, M. F., Bowman, S. E. A searchable database of crystallization cocktails in the PDB: analyzing the chemical condition space. Patterns. 1 (4), 100024 (2020).
- Jancarik, J., Kim, S. -. H. Sparse matrix sampling: a screening method for crystallization of proteins. Journal of Applied Crystallography. 24 (4), 409-411 (1991).
- Carter, C. W. Efficient factorial designs and the analysis of macromolecular crystal growth conditions. Methods. 1 (1), 12-24 (1990).
- McPherson, A., Cudney, B. Searching for silver bullets: An alternative strategy for crystallizing macromolecules. Journal of Structural Biology. 156 (3), 387-406 (2006).
- McPherson, A., Nguyen, C., Cudney, R., Larson, S. The role of small molecule additives and chemical modification in protein crystallization. Crystal Growth & Design. 11 (5), 1469-1474 (2011).
- Luft, J. R., DeTitta, G. T. A method to produce microseed stock for use in the crystallization of biological macromolecules. Acta Crystallographica Section D: Biological Crystallography. 55 (5), 988-993 (1999).
- Thakur, A. S., et al. Improved success of sparse matrix protein crystallization screening with heterogeneous nucleating agents. PLoS One. 2 (10), 1091 (2007).
- Chayen, N. E., Stewart, P. D. S., Blow, D. M. Microbatch crystallization under oil-a new technique allowing many small-volume crystallization trials. Journal of Crystal Growth. 122 (1-4), 176-180 (1992).
- Luft, J. R., et al. A deliberate approach to screening for initial crystallization conditions of biological macromolecules. Journal of Structural Biology. 142 (1), 170-179 (2003).
- Luft, J. R., Snell, E. H., DeTitta, G. T. Lessons from high-throughput protein crystallization screening: 10 years of practical experience. Expert Opinion on Drug Discovery. 6 (5), 465-480 (2011).
- Lynch, M. L., Snell, M. E., Potter, S. A., Snell, E. H., Bowman, S. E. 20 years of crystal hits: Progress and promise in ultrahigh-throughput crystallization screening. Acta Crystallographica Section D: Biological Crystallography. , (2023).
- Segelke, B. W. Efficiency analysis of sampling protocols used in protein crystallization screening. Journal of Crystal Growth. 232 (1-4), 553-562 (2001).
- Luft, J. R., Newman, J., Snell, E. H. Crystallization screening: the influence of history on current practice. Acta Crystallographica Section F. 70 (7), 835-853 (2014).
- Mlynek, G., Kostan, J., Leeb, S., Djinovic-Carugo, K. Tailored suits fit better: Customized protein crystallization screens. Crystal Growth & Design. 20 (2), 984-994 (2019).
- Mueller-Dieckmann, J. The open-access high-throughput crystallization facility at EMBL Hamburg. Acta Crystallographica Section D: Biological Crystallography. 62 (12), 1446-1452 (2006).
- Weber, P., et al. High-throughput crystallization pipeline at the crystallography core facility of the Institut Pasteur. Molecules. 24 (24), 4451 (2019).
- Lin, Y. What's happened over the last five years with high-throughput protein crystallization screening. Expert Opinion on Drug Discovery. 13 (8), 691-695 (2018).
- Haupert, L. M., Simpson, G. J. Screening of protein crystallization trials by second order nonlinear optical imaging of chiral crystals (SONICC). Methods. 55 (4), 379-386 (2011).
- Madden, J. T., DeWalt, E. L., Simpson, G. J. Two-photon excited UV fluorescence for protein crystal detection. Acta Crystallographica Section D: Biological Crystallography. 67 (10), 839-846 (2011).
- Fleming, A. M., et al. Second harmonic generation interrogation of the endonuclease APE1 binding interaction with G-quadruplex DNA. Analytical Chemistry. 94 (43), 15027-15032 (2022).
- Bruno, A. E., et al. Classification of crystallization outcomes using deep convolutional neural networks. PLoS One. 13 (6), 0198883 (2018).
- Holleman, E. T., Duguid, E., Keefe, L. J., Bowman, S. E. Polo: An open-source graphical user interface for crystallization screening. Journal of Applied Crystallography. 54 (2), 673-679 (2021).
- Niesen, F. H., et al. An approach to quality management in structural biology: Biophysical selection of proteins for successful crystallization. Journal of Structural Biology. 162 (3), 451-459 (2008).
- Padayatti, P., Palczewska, G., Sun, W., Palczewski, K., Salom, D. Imaging of protein crystals with two-photon microscopy. Biochemistry. 51 (8), 1625-1637 (2012).
- Haupert, L. M., DeWalt, E. L., Simpson, G. J. Modeling the SHG activities of diverse protein crystals. Acta Crystallographica Section D: Biological Crystallography. 68 (11), 1513-1521 (2012).
- Luft, J. R., Grant, T. D., Wolfley, J. R., Snell, E. H. A new view on crystal harvesting. Journal of Applied Crystallography. 47 (3), 1158-1161 (2014).
- Bruno, A. E., Soares, A. S., Owen, R. L., Snell, E. H. The use of haptic interfaces and web services in crystallography: An application for a 'screen to beam' interface. Journal of Applied Crystallography. 49 (6), 2082-2090 (2016).
- Baldock, P., Mills, V., Stewart, P. S. A comparison of microbatch and vapour diffusion for initial screening of crystallization conditions. Journal of Crystal Growth. 168 (1-4), 170-174 (1996).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved