In vivo Imaging dell'intero cervello delle larve di zebrafish mediante microscopia a fluorescenza tridimensionale
In This Article
Summary
Presentato qui è un protocollo per l'imaging in vivo dell'intero cervello del pesce zebra larvale utilizzando la microscopia a fluorescenza tridimensionale. La procedura sperimentale include la preparazione del campione, l'acquisizione di immagini e la visualizzazione.
Abstract
Come animale modello di vertebrato, i pesci zebra larvali sono ampiamente utilizzati nelle neuroscienze e offrono un'opportunità unica per monitorare l'attività dell'intero cervello alla risoluzione cellulare. Qui, forniamo un protocollo ottimizzato per l'esecuzione dell'imaging dell'intero cervello del pesce zebra larvale utilizzando la microscopia a fluorescenza tridimensionale, compresa la preparazione e l'immobilizzazione del campione, l'incorporamento del campione, l'acquisizione di immagini e la visualizzazione dopo l'imaging. L'attuale protocollo consente l'imaging in vivo della struttura e dell'attività neuronale di un cervello larvale di zebrafish a una risoluzione cellulare per oltre 1 ora utilizzando la microscopia confocale e la microscopia a fluorescenza progettata su misura. Vengono anche discussi i passaggi critici del protocollo, tra cui il montaggio e il posizionamento del campione, la prevenzione della formazione di bolle e polvere nel gel di agarosio e l'evitare il movimento nelle immagini causato dalla solidificazione incompleta del gel di agarosio e dalla paralisi del pesce. Il protocollo è stato convalidato e confermato in più impostazioni. Questo protocollo può essere facilmente adattato per l'imaging di altri organi di un pesce zebra larvale.
Introduction
Il pesce zebra (Danio rerio) è stato ampiamente adottato come animale vertebrato modello nelle neuroscienze, grazie alla sua trasparenza ottica allo stadio larvale, al suo rapido sviluppo, al suo basso costo di manutenzione e alla disponibilità di diversi strumenti genetici 1,2,3,4. In particolare, la trasparenza ottica delle larve combinata con i reporter fluorescenti geneticamente codificati di eventi biologici 5,6,7,8,9 offre un'opportunità unica per visualizzare sia l'attività neuronale che la struttura a livello dell'intero cervello 10,11,12,13,14 . Tuttavia, anche con un microscopio che supporta la risoluzione cellulare, le immagini acquisite non conservano necessariamente le informazioni a livello di singola cellula; La qualità ottica dell'immagine può essere degradata a causa dell'aberrazione introdotta dal gel di agarosio utilizzato per il montaggio del campione, il pesce può essere montato ad angolo, quindi le regioni di interesse non sono completamente contenute all'interno del campo visivo del microscopio e il pesce può muoversi durante la registrazione, causando artefatti di movimento nelle immagini o impedendo un'accurata estrazione del segnale dalle immagini.
Pertanto, è necessario un protocollo efficace e riproducibile per acquisire dati di immagine di alta qualità con rumore e movimento minimi. Sfortunatamente, i protocolli pubblicamente disponibili per l'imaging di un intero cervello di pesce zebra larvale in vivo 15,16,17,18,19 descrivono solo brevemente la procedura, lasciando parti sostanziali dei dettagli, come la solidificazione dell'agarosio, le tecniche di montaggio precise e il posizionamento del campione usando una pinza, a ciascun sperimentatore. Inoltre, le incongruenze nei metodi di concentrazione e immobilizzazione dell'agarosio 10,11,14,15,16,17,18,19 possono portare a sfide derivanti dal movimento dei pesci durante il processo di imaging.
Qui viene fornito un protocollo dettagliato per l'imaging dell'intero cervello del pesce zebra larvale utilizzando la microscopia a fluorescenza tridimensionale. La Figura 1 fornisce una panoramica grafica del protocollo: preparazione e immobilizzazione del campione, incorporamento dei campioni, acquisizione delle immagini e visualizzazione dopo l'imaging. L'imaging strutturale e funzionale del cervello larvale del pesce zebra in vivo è dimostrato utilizzando un microscopio confocale commerciale e un microscopio a fluorescenza progettato su misura. Questo protocollo può essere adattato dai professionisti per l'imaging cerebrale con determinati stimoli sensoriali o contesti comportamentali a seconda delle esigenze sperimentali e del design.
Protocol
Tutti gli esperimenti sul pesce zebra sono stati approvati dall'Institutional Animal Care and Use Committee (IACUC) del KAIST (KA2021-125). Un totale di 12 pesci zebra adulti con espressione pan-neuronale dell'indicatore di calcio GCaMP7a [Tg(huc:GAL4); Tg(UAS:GCaMP7a)] su uno sfondo casper [mitfa(w2/w2);mpv17(a9/a9)] sono stati utilizzati per la riproduzione. Questo gruppo era composto da otto femmine e quattro maschi, con età variabili da 3 a 12 mesi. Gli esperimenti di imaging sono stati eseguiti su pesci zebra larvali a 3-4 giorni dopo la fecondazione (d.p.f.), una fase durante la quale il loro sesso non può essere determinato.
1. Preparazione del campione di zebrafish
- Raccogliere embrioni dopo aver allevato pesci adulti di una linea transgenica desiderata, come Tg (huc: GAL4); Tg(UAS:GCaMP7a)20,21,22, in una capsula di Petri riempita con acqua d'uovo (vedi Tabella dei materiali). Mettere gli embrioni in un'incubatrice a 28 °C e portarli a 3-4 d.p.f. larve23,24,25.
- Se il fondo del pesce zebra non è albino, per inibire una formazione di pigmentazione, trasferire gli embrioni a 24 ore dopo la fecondazione (h.p.f.) nella capsula di Petri riempita con acqua d'uovo contenente 200 μM di 1-fenil 2-tiourea (PTU; vedi tabella dei materiali)25,26. Ogni 24 ore, trasferire il pesce in un nuovo piatto con acqua fresca contenente 200 μM di PTU.
NOTA: I pesci zebra sono mantenuti in condizioni standard a 28 °C e un ciclo luce:buio di 14:10 h. È noto che il trattamento con PTU influenza il comportamento e la funzione tiroidea del pesce zebra larvale27,28. Pertanto, è importante usare la PTU con cautela e controllare attentamente i potenziali fattori confondenti in qualsiasi esperimento.
- Se il fondo del pesce zebra non è albino, per inibire una formazione di pigmentazione, trasferire gli embrioni a 24 ore dopo la fecondazione (h.p.f.) nella capsula di Petri riempita con acqua d'uovo contenente 200 μM di 1-fenil 2-tiourea (PTU; vedi tabella dei materiali)25,26. Ogni 24 ore, trasferire il pesce in un nuovo piatto con acqua fresca contenente 200 μM di PTU.
- Per trovare il campione che esprime proteine fluorescenti di interesse (ad esempio, GCaMP7a pan-neuronale), esaminare il campione al microscopio a epifluorescenza e selezionare un campione con un'espressione luminosa.
- Preparare il campione selezionato di 3-4 d.p.f. zebrafish nella capsula di Petri riempita con acqua d'uovo (Figura 2A).
NOTA: Per registrare l'attività neuronale spontanea, si raccomanda di utilizzare campioni di età compresa tra 80-100 h.p.f., poiché il livello di attività spontanea è basso prima di 80 h.p.f. e lo sviluppo della pigmentazione, anche con uno sfondo più spreco, può degradare la qualità dell'immagine dopo 100 h.p.f. - Preparare una soluzione di bromuro di pancuronio 0,25 mg/ml 14,19,29,30,31 aggiungendo 1 mL di una soluzione madre da 2,5 mg/mL (vedere Tabella dei materiali) a 10 mL di acqua dell'uovo. Aliquotare la soluzione di bromuro di pancuronio in provette da microcentrifuga da 1,5 ml.
- Trasferire il campione pre-vagliato nella capsula di Petri utilizzando una pipetta di trasferimento.
NOTA: Provare a portare con sé un volume minimo di acqua all'uovo. - Trasferire 0,1 mL della soluzione di bromuro di pancuronio nella capsula di Petri per la paralisi.
NOTA: Il bromuro di pancuronio ha un potenziale effetto smorzante sull'attività neurale nel pesce zebra larvale32. È essenziale considerare attentamente la concentrazione e la durata dell'esposizione al bromuro di pancuronio.
2. 2% (wt/vol) preparazione gel di agarosio
- Accendere un blocco termico e impostare la temperatura target a 37 °C. Attendere che l'unità si riscaldi e si bilanci alla temperatura impostata.
- Sciogliere 0,2 g di polvere di agarosio a basso punto di fusione (vedere Tabella dei materiali) in 10 ml di acqua d'uovo.
- Riscaldare la soluzione di agarosio in un microonde e mescolarla agitando e vorticando fino a quando l'agarosio è completamente sciolto.
- Aliquotare il gel di agarosio in provette da microcentrifuga da 1,5 ml e conservare le provette da microcentrifuga sul blocco termico (Figura 2B).
NOTA: Controllare se le bolle nel gel di agarosio sono scomparse.
3. Montaggio e posizionamento del campione
- Controllare il campione sotto uno stereomicroscopio per verificare che il movimento delle larve si sia fermato e per valutare visivamente la salute del campione controllandone il battito cardiaco (Figura 2C). Se il battito cardiaco è troppo lento, eliminare il campione.
NOTA: se il battito cardiaco del pesce ripreso è troppo lento (ad esempio, inferiore a 60 battiti al minuto), potrebbe non essere possibile eseguire immagini a lungo termine. Per valutare il benessere del pesce, la frequenza cardiaca può essere controllata visivamente confrontandola con altri pesci nella stessa capsula di Petri. Ciò contribuirà a garantire che il pesce sottoposto a immagini sia sano e abbastanza stabile per la procedura di imaging. - Utilizzando la pipetta di trasferimento, posizionare un singolo pesce zebra larvale nel gel di agarosio nella provetta da microcentrifuga da 1,5 mL (Figura 2D).
NOTA: Assicurarsi di eliminare la pipetta dopo aver trasferito il campione. - Versare il gel di agarosio nella piastra di Petri per ottenere una mano di 1-2 mm. Trasferire il campione nelle provette della microcentrifuga nella capsula di Petri utilizzando la pipetta di trasferimento, in modo che la larva sia posizionata al centro del piatto.
NOTA: Se ci sono polvere e bolle nel gel di agarosio, rimuoverle usando la pipetta di trasferimento. - Utilizzare una pinza per posizionare il campione nell'orientamento desiderato in modo che la testa e la coda siano piatte (Figura 2E).
- Ruotare il campione usando una pinza in modo che entrambi gli occhi siano in piano (Figura 2F).
NOTA: Le procedure di allineamento (posizione e rotazione) devono essere completate prima che il gel di agarosio inizi a solidificarsi. - Dopo l'allineamento, attendere che il gel di agarosio si sia solidificato (Figura 2G,H).
NOTA: Il tempo di attesa per la solidificazione del gel di agarosio può variare da 5 a 10 minuti, a seconda del volume e delle dimensioni del gel. - Dopo la solidificazione del gel di agarosio, riempire la capsula di Petri con acqua d'uovo e posizionare la capsula di Petri con il campione incorporato sullo stadio del microscopio (Figura 2I).
4. Acquisizione di immagini
- Accendere il sistema di microscopio (ad esempio, laser, controller confocali, microscopio e computer; vedere Tabella dei materiali) e verificare che l'intero sistema funzioni.
- Selezionare un obiettivo a basso ingrandimento e posizionare il campione al centro del campo visivo.
- Selezionare un obiettivo a immersione in acqua o a immersione d'acqua con un ingrandimento adeguato (ad esempio, obiettivo con immersione in acqua 16x 0,8 con apertura numerica (NA); vedere la tabella dei materiali). Apportare regolazioni precise al campo visivo.
- Impostare i parametri di imaging (ad esempio, dimensioni dell'immagine, potenza del laser, tempo di esposizione, numero di fotogrammi) utilizzando il software di acquisizione delle immagini.
NOTA: impostare i parametri di imaging per ottenere i migliori risultati possibili per esigenze specifiche (vedere la configurazione per l'acquisizione delle immagini nella sezione dei risultati rappresentativi). Se l'immagine è satura, ridurre la potenza del laser. - Trova il cervello del campione spostando il palco e determinane lo spessore utilizzando la modalità live view nel software modificando manualmente i piani focali su e giù. Impostare i limiti inferiore e superiore del volume.
NOTA: Assicurarsi che l'intero cervello sia contenuto nel campo visivo lungo entrambe le direzioni laterale e assiale. - Impostare una dimensione z-step considerando la risoluzione assiale del microscopio.
NOTA: La dimensione ottimale dello z-step per l'imaging del cervello del pesce zebra larvale dipende dalla modalità di imaging e dalla risoluzione del microscopio. Ad esempio, è stata utilizzata una dimensione z-step di 5 μm considerando lo spessore del foglio luminoso e il diametro medio dei corpi cellulari10. - Procedere con l'acquisizione dell'immagine per il campo visivo impostato.
- Per l'imaging strutturale volumetrico, acquisire un'immagine 3D (x, y, z) dell'intero cervello cambiando i piani focali e ottenendo immagini 2D di ciascun piano z in sequenza.
- Per l'imaging funzionale di un singolo piano z, acquisire immagini di serie temporali (x, y, t) dell'attività neuronale del cervello a una certa profondità.
- Per l'imaging funzionale volumetrico, acquisire un'immagine 4D (x,y,z,t) dell'attività neuronale nell'intero cervello ottenendo immagini 3D in sequenza.
NOTA: impostare il numero di fotogrammi considerando la dimensione della memoria disponibile del computer. Si raccomanda un tempo di acquisizione inferiore a 1 h a causa della durata dell'effetto del bromuro di pancuronio.
- Dopo aver acquisito le immagini, salvare i risultati e registrare i parametri di imaging (ad esempio, dimensione dei pixel, dimensione z-step, frame rate, potenza del laser) per l'analisi delle immagini.
- Esportare le immagini in un formato appropriato per il rendering dei dati e l'analisi delle immagini.
NOTA: si consiglia di esportare le immagini in formato TIF (Tagged Image File). TIF supporta la compressione delle immagini senza perdita di dati ed è compatibile con la maggior parte dei software di elaborazione delle immagini e dei linguaggi di programmazione. Inoltre, il formato TIF supporta l'inclusione di metadati, come parametri di acquisizione, risoluzione dell'immagine e altre informazioni rilevanti che possono aiutare nell'interpretazione e nella riproducibilità dei dati.
5. Configurazione per le visualizzazioni utilizzando napari
NOTA: napari è un visualizzatore di immagini multidimensionale open source in un ambiente Python con rendering basato su unità di elaborazione grafica (GPU)33. Il plugin napari-animation fornisce una creazione programmatica di filmati. L'utilizzo di Fiji, un programma di elaborazione delle immagini open source, è consigliato per l'elaborazione di immagini generiche, come il filtraggio e la trasformazione geometrica (vedere Tabella dei materiali). Il codice sorgente usato per la visualizzazione tramite napari è disponibile su GitHub (https://github.com/NICALab/Zebrafish-brain-visualization).
- Installa napari e napari-animation usando pip o conda. Dopo l'installazione, creare un nuovo file di notebook jupyter.
Nota : l'esecuzione con jupyter notebook, che è uno strumento interattivo per Python, è consigliato rispetto allo script Python. - Importa napari e napari-animation.
6. Visualizzare le strutture usando napari
- Per visualizzare le immagini e creare filmati del cervello del pesce zebra renderizzato, caricare l'immagine 3D (x, y, z) e aprire la finestra napari. Collegare il plug-in napari-animation (Figura 3A).
- Imposta parametri come la dimensione del voxel, la mappa dei colori e i limiti di contrasto nei controlli dei livelli.
- Regola le impostazioni del visualizzatore (ad esempio, prospettiva, angoli) nell'area di lavoro.
- Per acquisire l'immagine sottoposta a rendering, premere il pulsante di acquisizione nell'animazione guidata.
- Per generare filmati del volume sottoposto a rendering, modificare le impostazioni del visualizzatore e aggiungere fotogrammi chiave (Figura 3B).
- Dopo aver aggiunto i fotogrammi chiave, impostate il numero di fotogrammi (passaggi) tra i fotogrammi chiave nell'Animazione guidata. Salvate l'animazione sottoposta a rendering.
7. Elaborazione delle immagini e visualizzazione dell'attività neuronale utilizzando napari
NOTA: Per visualizzare le immagini della serie temporale dell'attività neuronale come immagini sovrapposte di uno sfondo statico e di un'attività, è necessario applicare un algoritmo di decomposizione alle immagini grezze. Utilizzare un'implementazione MATLAB di un algoritmo di scomposizione chiamato BEAR24. La versione MATLAB di BEAR è disponibile su GitHub (https://github.com/NICALab/BEAR).
- Per scomporre lo sfondo statico e l'attività neuronale, applicare BEAR alle immagini delle serie temporali non elaborate (x,y,t o x,y,z,t). Dopo la decomposizione, salvare le immagini dello sfondo e dell'attività neuronale come file TIF (Figura 3C).
- Carica le immagini di sfondo e di attività neuronale e apri la finestra napari. Collega il plugin napari-animation.
- Utilizzare una mappa a colori grigia per le immagini di sfondo e una mappa a colori calda per le immagini di attività neuronale (Figura 3C).
- Impostate parametri quali i limiti di opacità e contrasto nei controlli dei livelli.
- Per generare filmati di attività neuronale, modificare le impostazioni del visualizzatore e aggiungere fotogrammi chiave per eseguire il rendering dell'animazione.
- Dopo aver aggiunto i fotogrammi chiave, impostate il numero di fotogrammi (passaggi) tra i fotogrammi chiave nell'Animazione guidata. Salvate l'animazione sottoposta a rendering.
Representative Results
La struttura e l'attività neuronale del cervello larvale del pesce zebra che esprime l'indicatore di calcio pan-neuronale GCaMP7a (Tg(huc:GAL4); Tg(UAS:GCaMP7a))20,21,22 con un casper (mitfa(w2/w2);mpv17(a9/a9))34 lo sfondo è stato ripreso a 3-4 d.p.f. seguendo il protocollo descritto.
Per l'imaging strutturale volumetrico, il campione è stato ripreso utilizzando un sistema di microscopia confocale a scansione puntiforme commerciale dotato di un obiettivo a immersione d'acqua 16x 0,8 NA. Un laser di eccitazione a 488 nm è stato utilizzato sia per l'imaging strutturale che funzionale. La frequenza dei fotogrammi, la risoluzione dell'immagine, la dimensione dei pixel e la dimensione del passo assiale erano rispettivamente 0,25 Hz, 2048 x 2048, 0,34 μm e 1,225 μm. L'acquisizione dell'immagine è durata circa 1 ora e 20 minuti. Il campo visivo volumetrico dell'immagine acquisita copriva le regioni cerebrali del proencefalo, del mesencefalo e del retrosencefalo (Figura 4A). I corpi cellulari neuronali del midollo allungato, della piastra cerebellare, del tectum ottico, dell'abenula, e del telencefalo dorsale nell'intero cervello di 4 pesci zebra larvali d.p.f. erano chiaramente visibili nelle immagini di microscopia confocale (Figura 4B). Il rendering 3D delle immagini al microscopio confocale è stato eseguito utilizzando napari27 seguendo il suddetto protocollo (Figura 4C e Video 1).
Per l'imaging funzionale in 2-D, il campione è stato ripreso utilizzando lo stesso sistema di microscopia confocale, dotato di un obiettivo a immersione d'acqua 16x 0,8 NA. La frequenza dei fotogrammi, la risoluzione dell'immagine e la dimensione dei pixel erano rispettivamente di 2 Hz, 512 x 256 e 1,5 μm. I corpi delle cellule neuronali erano chiaramente visibili sia sullo sfondo che nell'attività neuronale sovrapposta (Figura 5A,B).
Per l'imaging funzionale 3D, è stato utilizzato un sistema di microscopia 3D progettato su misura18 , in grado di visualizzare in vivo l'attività neuronale di un intero cervello larvale di zebrafish con un campo visivo di 1.040 μm × 400 μm × 235 μm e risoluzioni laterali e assiali di 1,7 μm e 5,4 μm, rispettivamente. La velocità di imaging era fino a 4,2 volumi al secondo. Analogamente al rendering dei dati di imaging strutturale, napari è stato utilizzato per il rendering 3D dei dati di imaging del calcio dell'intero cervello (Figura 5C e Video 2).
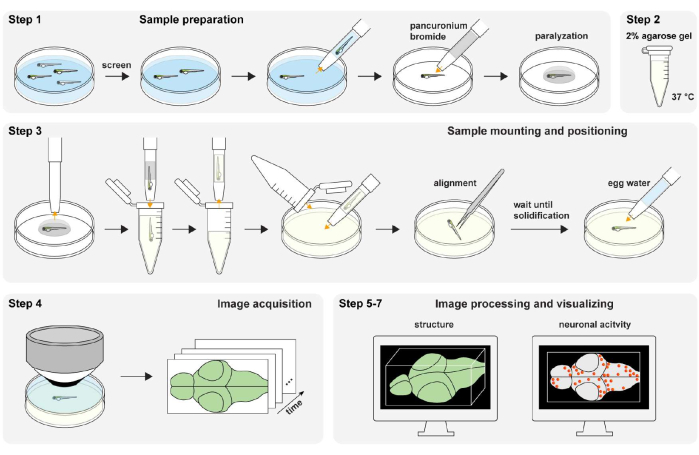
Figura 1: Panoramica della procedura sperimentale. Preparazione e paralisi del campione di zebrafish (fase 1). La preparazione del gel di agarosio al 2% (wt/vol) (fase 2). Montaggio e posizionamento del campione (fase 3). Acquisizione di immagini (fase 4). Elaborazione delle immagini e visualizzazione della struttura e dell'attività neuronale (fase 5-7). Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 2: Procedura sperimentale per la preparazione dell'imaging dell'intero cervello . (A) Campione di zebrafish sottoposto a screening che esprime GCaMP7a pan-neuronale in una capsula di Petri riempita con acqua d'uovo. (B) Attrezzature e materiali necessari per il montaggio e il posizionamento del campione. (1) blocco termico a 37 °C; (2) 2% (wt/vol) gel di agarosio; (3) provetta da microcentrifuga da 1,5 ml; (4) acqua d'uovo; (5) 0,25 mg/mL di soluzione di bromuro di pancuronio; (6) Capsula di Petri; (7) pinze; (8) Pipetta di trasferimento. (C) Immagine stereomicroscopica del campione paralizzato. La freccia nera indica il cuore del campione. D) Il campione nella provetta da microcentrifuga da 1,5 mL viene trasferito mediante una pipetta. L'inserto mostra una vista ingrandita dell'area riquadro. (E) Il campione (freccia nera) viene posto al centro della piastra di Petri usando una pinza. (F) Il campione è allineato con una pinza. (G) Un esempio di campione incorporato disallineato. (H) Un esempio di campione incorporato allineato. (I) Il campione viene posto su un palcoscenico al microscopio sotto la lente dell'obiettivo per l'acquisizione delle immagini. Barra scala: 500 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
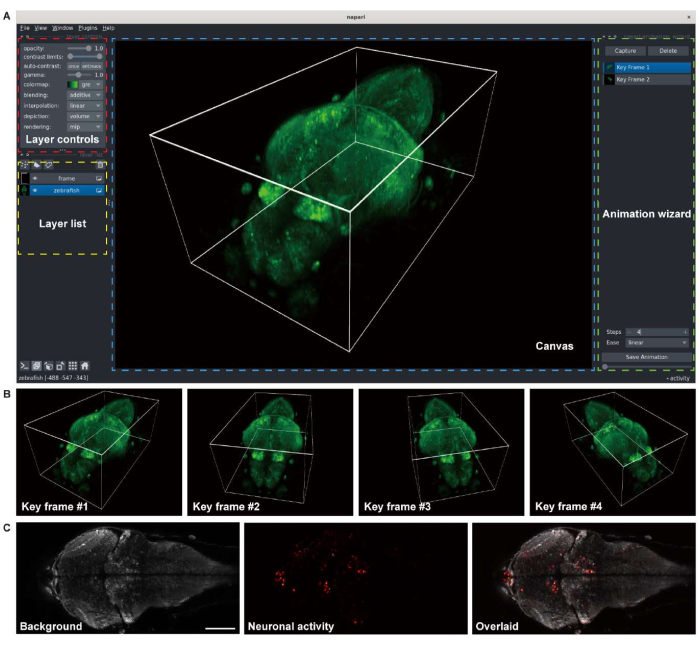
Figura 3: Visualizzazione delle immagini cerebrali del pesce zebra larvale. (A) Il rendering 3D di un'immagine al microscopio confocale di un cervello larvale di zebrafish (4 d.p.f.) che esprime GCaMP7a pan-neuronale. napari, un visualizzatore di immagini multidimensionale open source in un ambiente Python, è stato utilizzato per il rendering. La finestra napari comprende controlli layer (riquadro rosso), un elenco di livelli (riquadro giallo), un canvas (riquadro blu) e una procedura guidata di animazione (riquadro verde). (B) I fotogrammi chiave con più impostazioni del visualizzatore vengono aggiunti per il rendering di un'animazione. (C) Immagine al microscopio confocale dell'imaging time-lapse 2D dell'attività neuronale nel cervello del pesce zebra larvale. L'immagine viene scomposta sullo sfondo (a sinistra) e l'attività neuronale (al centro), quindi sovrapposta (a destra). Barra scala: 100 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
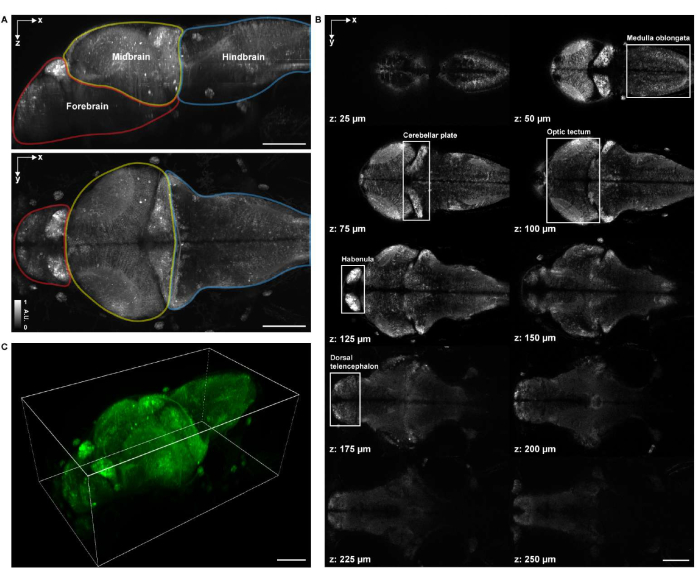
Figura 4: Struttura di imaging del cervello di un pesce zebra larvale. (A) Proiezione della massima intensità (MIP) di un'immagine al microscopio confocale di un cervello larvale di zebrafish (4 d.p.f.) che esprime GCaMP7a pan-neuronale. Parte superiore: MIP laterale. In basso: MIP assiale. Ogni confine contiene il proencefalo (rosso), il mesencefalo (giallo) e il cervello posteriore (blu). (B) Un totale di 10 fette assiali a più profondità da un'immagine volumetrica del cervello (a z = 25 μm, 50 μm, 75 μm, 100 μm, 125 μm, 150 μm, 175 μm, 200 μm, 225 μm, 250 μm, contate verso l'alto dall'alto verso il basso; z = 0 μm indica la superficie superiore del cervello). Ogni scatola bianca rappresenta la regione del cervello (midollo allungato, piastra cerebellare, tectum ottico, abenula, telencefalo dorsale). (C) L'intero cervello reso usando napari. Barra scala: 100 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
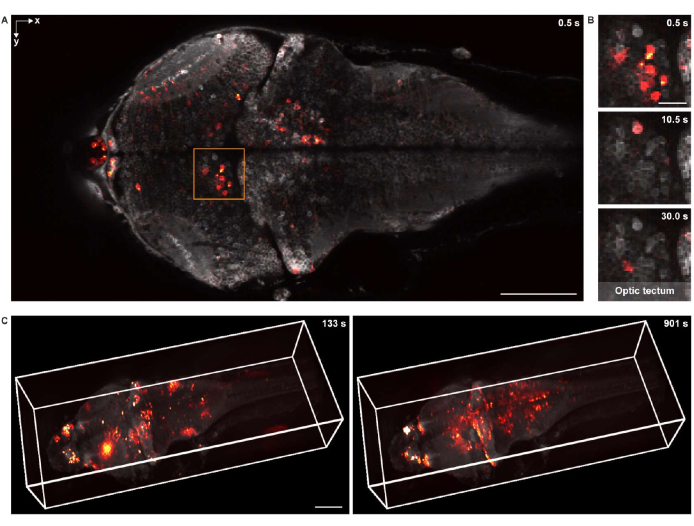
Figura 5: Imaging dell'attività neuronale di un cervello larvale di zebrafish. (A) Immagine al microscopio confocale dell'attività neuronale in un cervello larvale di zebrafish (4 d.p.f.) che esprime GCaMP7a pan-neuronale. Barra della scala: 100 μm. (B) Vista ingrandita dell'area riposta in A che mostra l'attività neuronale nel tectum ottico in più punti temporali. Barra della scala: 20 μm. (C) Il rendering 3D dell'attività neuronale dell'intero cervello nel cervello del pesce zebra larvale acquisito utilizzando un microscopio progettato su misura (sinistra: t = 133 s; destra: t = 901 s). L'attività neuronale è sovrapposta allo sfondo statico. Barra scala: 100 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
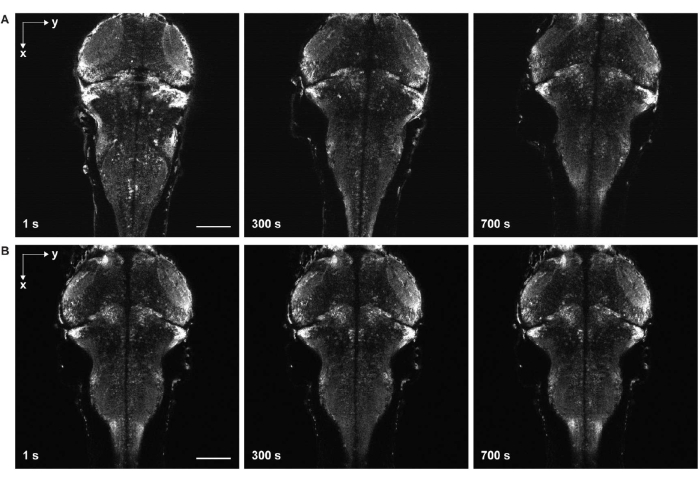
Figura 6: Imaging time-lapse con e senza deriva del campione. (A) Imaging time-lapse del cervello larvale del pesce zebra che esprime GCaMP7a pan-neuronale con deriva del campione. L'acqua dell'uovo è stata aggiunta al campione prima della solidificazione del gel di agarosio (fase 3.6-3.7). Il pesce si muoveva nelle direzioni laterale e assiale. (B) Imaging time-lapse di un cervello larvale di zebrafish senza deriva del campione. L'acqua dell'uovo è stata aggiunta al campione dopo la solidificazione del gel di agarosio (fase 3.6-3.7). Barra scala: 100 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Video 1: Rendering 3D dell'intera struttura cerebrale di un pesce zebra larvale (4 d.p.f.) cerebrale che esprime GCaMP7a pan-neuronale. Clicca qui per scaricare questo video.
Video 2: Rendering 3D dell'attività neuronale dell'intero cervello in un cervello larvale zebrafish (4 d.p.f.) che esprime GCaMP7a pan-neuronale. Clicca qui per scaricare questo video.
Discussion
L'attuale protocollo consente l'imaging in vivo dell'intero cervello del pesce zebra larvale per un periodo prolungato (ad esempio, più di 1 ora) e la visualizzazione dei dati di imaging strutturale e funzionale acquisiti.
I passaggi più critici sono il montaggio e il posizionamento del campione. Durante l'incorporazione del campione, è fondamentale prevenire la formazione di bolle ed evitare la polvere nel gel di agarosio. Se il gel contiene bolle d'aria e polvere, la qualità dell'immagine potrebbe essere gravemente degradata. Durante il posizionamento del campione mediante una pinza, è importante assicurarsi che il campione sia livellato sia orizzontalmente che verticalmente. In caso contrario, le regioni di interesse potrebbero non essere contenute nel campo visivo del microscopio. Inoltre, questo posizionamento dovrebbe essere fatto entro una breve finestra temporale prima della solidificazione del gel di agarosio per evitare di causare danni alla sua superficie, in quanto tali danni compromettono la qualità dell'immagine.
Un'altra grande sfida è evitare il movimento nelle immagini che provengono dalla solidificazione incompleta del gel di agarosio (Figura 6) e dalla paralisi del pesce zebra. Quando l'acqua dell'uovo viene aggiunta al campione prima della completa solidificazione del gel agorse, i pesci si muovono lentamente sia lateralmente che assialmente, manifestandosi come deriva del campione nelle immagini della serie temporale (Figura 6A). Se la dose di paralitici è insufficiente o la durata dell'effetto termina, il campione può tentare di muoversi, il che appare come contrazione rapida nelle immagini time-lapse.
Nonostante l'importanza dei suddetti passaggi per l'acquisizione di immagini di alta qualità senza artefatti di movimento, i protocolli 15,16,17,18,19 disponibili al pubblico forniscono solo una breve panoramica della procedura sperimentale, mancando di questi dettagli. Ad esempio, le immagini acquisite senza protocolli di immobilizzazione11 soffrono di sostanziali artefatti di movimento che rendono difficile l'analisi dell'immagine a valle. Integrando componenti essenziali, come la solidificazione del gel di agarosio e la paralisi del campione, il nostro protocollo migliora significativamente la coerenza nella qualità delle immagini acquisite, riducendo al minimo gli artefatti di movimento.
In sintesi, viene descritta una procedura sperimentale ottimizzata e riproducibile per l'imaging in vivo del cervello del pesce zebra larvale. La validità e la riproducibilità di questo protocollo per l'imaging in vivo dell'attività e della struttura cerebrale sono state confermate in contesti multipli 14,18,29,30,31. Il presente flusso di lavoro si è concentrato sull'imaging dell'intero cervello del pesce zebra larvale, ma può essere facilmente applicato all'imaging di altri organi del pesce zebra larvale35,36.
Disclosures
Gli autori non dichiarano conflitti di interesse.
Acknowledgements
Le linee di zebrafish utilizzate per l'imaging del calcio sono state fornite dal Zebrafish Center for Disease Modeling (ZCDM), Corea. Questa ricerca è stata sostenuta dalla National Research Foundation of Korea (2020R1C1C1009869, NRF2021R1A4A102159411, RS-2023-00209473).
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL microcentrifuge tube | SciLab | SL.Tub3513 | To aliquot agarose gel and pancuronium bromide solution |
| 15 mL Falcon tubes | Falcon | 352096 | To prepare agarose gel and pancuronium bromide solution |
| 16× 0.8NA water dipping objective lens | Nikon | CFI75 LWD 16×W | Objective lens for whole-brain imaging |
| 1-phenyl 2-thiourea (PTU) | Sigma-Aldrich | P7629-10G | 200 μM of 1-phenyl 2-thiourea (PTU) |
| 50 mL Falcon tubes | Falcon | 352070 | To prepare egg water |
| Disposable transfer pipette | SciLab | SL.Pip3032 | To transfer zebrafish larvae |
| Egg water | N/A | N/A | 0.6 g sea salt in 10 L deionized water |
| Forceps | Karl Hammacher GmbH | HSO 010-10 | Forceps used for sample positioning |
| Low melting point agarose | Thermo Scientific | R0801 | 2% (wt/vol) agarose gel |
| Napari | Napari | N/A | To visualize microscopy images in 3-D |
| NIS-Elements C | Nikon | N/A | Imaging software for confocal microscope |
| Pancuronium bromide | Sigma-Aldrich | P1918-10MG | 0.25 mg/mL of pancuronium bromide solution |
| Petri dish, 35 mm | SPL Life Sciences | 11035 | Petri dish used for sample embedding |
| Petri dish, 55 mm | SPL Life Sciences | 11050 | To prepare zebrafish larvae after screening |
| Point-scanning confocal microscopy system (C2 Plus) | Nikon | N/A | Confocal microscope for whole-brain imaging |
| Sea salt | Sigma-Aldrich | S9883-500G | Sea salt used for preparing egg water |
References
- Choi, T. -. Y., Choi, T. -. I., Lee, Y. -. R., Choe, S. -. K., Kim, C. -. H. Zebrafish as an animal model for biomedical research. Experimental & Molecular Medicine. 53 (3), 310-317 (2021).
- Ahrens, M. B., et al. Brain-wide neuronal dynamics during motor adaptation in zebrafish. Nature. 485 (7399), 471-477 (2012).
- Panier, T., et al. Fast functional imaging of multiple brain regions in intact zebrafish larvae using selective plane illumination microscopy. Frontiers in Neural Circuits. 7, 65 (2013).
- Bianco, I. H., Kampff, A. R., Engert, F. Prey capture behavior evoked by simple visual stimuli in larval zebrafish. Frontiers in Systems Neuroscience. 5, 101 (2011).
- Akerboom, J., et al. Optimization of a GCaMP calcium indicator for neural activity imaging. Journal of Neuroscience. 32 (40), 13819-13840 (2012).
- Chen, T. -. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Piatkevich, K. D., et al. A robotic multidimensional directed evolution approach applied to fluorescent voltage reporters. Nature Chemical Biology. 14 (4), 352-360 (2018).
- Tian, L., et al. Imaging neural activity in worms, flies and mice with improved GCaMP calcium indicators. Nature Methods. 6 (12), 875-881 (2009).
- Looger, L., et al. Fast and sensitive GCaMP calcium indicators for imaging neural populations. Biorxiv. , (2021).
- Ahrens, M. B., Orger, M. B., Robson, D. N., Li, J. M., Keller, P. J. Whole-brain functional imaging at cellular resolution using light-sheet microscopy. Nature Methods. 10 (5), 413-420 (2013).
- Prevedel, R., et al. Simultaneous whole-animal 3D imaging of neuronal activity using light-field microscopy. Nature Methods. 11 (7), 727-730 (2014).
- Chhetri, R. K., et al. Whole-animal functional and developmental imaging with isotropic spatial resolution. Nature Methods. 12 (12), 1171-1178 (2015).
- Ahrens, M. B., Engert, F. Large-scale imaging in small brains. Current Opinion in Neurobiology. 32, 78-86 (2015).
- Yoon, Y. -. G., et al. Sparse decomposition light-field microscopy for high speed imaging of neuronal activity. Optica. 7 (10), 1457-1468 (2020).
- Randlett, O., et al. Whole-brain activity mapping onto a zebrafish brain atlas. Nature Methods. 12 (11), 1039-1046 (2015).
- Cong, L., et al. Rapid whole brain imaging of neural activity in freely behaving larval zebrafish (Danio rerio). eLife. 6, e28158 (2017).
- Bruzzone, M., et al. Whole brain functional recordings at cellular resolution in zebrafish larvae with 3D scanning multiphoton microscopy. Scientific Reports. 11 (1), 11048 (2021).
- Cho, E. -. S., Han, S., Lee, K. -. H., Kim, C. -. H., Yoon, Y. -. G. 3DM: deep decomposition and deconvolution microscopy for rapid neural activity imaging. Optics Express. 29 (20), 32700-32711 (2021).
- Zhang, Z., et al. Imaging volumetric dynamics at high speed in mouse and zebrafish brain with confocal light field microscopy. NatureBiotechnology. 39 (1), 74-83 (2021).
- Muto, A., Ohkura, M., Abe, G., Nakai, J., Kawakami, K. Real-time visualization of neuronal activity during perception. Current Biology. 23 (4), 307-311 (2013).
- Köster, R. W., Fraser, S. E. Tracing transgene expression in living zebrafish embryos. Developmental Biology. 233 (2), 329-346 (2001).
- Park, H. C., et al. Analysis of upstream elements in the HuC promoter leads to the establishment of transgenic zebrafish with fluorescent neurons. Developmental Biology. 227 (2), 279-293 (2000).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: an introduction. Journal of Visualized Experiments. (69), e4196 (2012).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). 4th edition. , (2000).
- Morsch, M., et al. Triggering cell stress and death using conventional UV laser confocal microscopy. Journal of Visualized Experiments. (120), e54983 (2017).
- Antinucci, P., Hindges, R. A crystal-clear zebrafish for in vivo imaging. Scientific Reports. 6, 29490 (2016).
- Parker, M. O., Brock, A. J., Millington, M. E., Brennan, C. H. Behavioral phenotyping of casper mutant and 1-pheny-2-thiourea treated adult zebrafish. Zebrafish. 10 (4), 466-471 (2013).
- Elsalini, O. A., Rohr, K. B. Phenylthiourea disrupts thyroid function in developing zebrafish. Development Genes and Evolution. 212 (12), 593-598 (2003).
- Han, S., Cho, E. -. S., Park, I., Shin, K., Yoon, Y. -. G. Efficient neural network approximation of robust PCA for automated analysis of calcium imaging data. Medical Image Computing and Computer Assisted Intervention. , 595-604 (2021).
- Shin, C., et al. Three-dimensional fluorescence microscopy through virtual refocusing using a recursive light propagation network. Medical Image Analysis. 82, 102600 (2022).
- Eom, M., et al. Statistically unbiased prediction enables accurate denoising of voltage imaging data. bioRxiv. , (2022).
- Johnston, L., et al. Electrophysiological recording in the brain of intact adult zebrafish. Journal of Visualized Experiments. (81), e51065 (2013).
- Sofroniew, N., et al. napari: a multi-dimensional image viewer for Python. Zenodo. , 3555620 (2022).
- White, R. M., et al. Transparent adult zebrafish as a tool for in vivo transplantation analysis. Cell Stem Cell. 2 (2), 183-189 (2008).
- Mickoleit, M., et al. High-resolution reconstruction of the beating zebrafish heart. Nature Methods. 11 (9), 919-922 (2014).
- Voleti, V., et al. Real-time volumetric microscopy of in vivo dynamics and large-scale samples with SCAPE 2.0. Nature Methods. 16 (10), 1054-1062 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved