Imunofluorescência Multiplex Combinada com Análise Espacial de Imagens para Avaliação Clínica e Biológica do Microambiente Tumoral
Neste Artigo
Resumo
Neste artigo, um protocolo para amplificação manual do sinal de tiramida (TSA) por imunofluorescência multiplex (mIF) combinado com análise de imagem e análise espacial é descrito. Este protocolo pode ser usado com cortes fixados em formalina e embebidos em parafina (FFPE) para a coloração de dois a seis antígenos por lâmina, dependendo do scanner de lâminas disponível no laboratório.
Resumo
O microambiente tumoral (TME) é composto por uma infinidade de diferentes tipos celulares, como células imunes citotóxicas e células imunomoduladoras. Dependendo de sua composição e das interações entre células cancerosas e células peritumorais, a TME pode afetar a progressão do câncer. A caracterização de tumores e seu microambiente complexo pode melhorar a compreensão das doenças oncológicas e pode ajudar cientistas e clínicos a descobrir novos biomarcadores.
Recentemente, desenvolvemos vários painéis de imunofluorescência multiplex (mIF) baseados em amplificação de sinal de tiramida (TSA) para a caracterização da ETM em câncer colorretal, carcinoma espinocelular de cabeça e pescoço, melanoma e câncer de pulmão. Uma vez concluída a coloração e digitalização dos painéis correspondentes, as amostras são analisadas em um software de análise de imagens. A posição espacial e a coloração de cada célula são então exportadas deste software de quantificação para R. Desenvolvemos scripts R que nos permitem não apenas analisar a densidade de cada tipo celular em vários compartimentos tumorais (por exemplo, o centro do tumor, a margem do tumor e o estroma), mas também realizar análises baseadas na distância entre diferentes tipos celulares.
Esse fluxo de trabalho em particular adiciona uma dimensão espacial à análise clássica de densidade já realizada rotineiramente para vários marcadores. A análise do mIF pode permitir que os cientistas tenham uma melhor compreensão da interação complexa entre as células cancerosas e a TME e descubram novos biomarcadores preditivos de resposta a tratamentos, como inibidores de checkpoints imunológicos e terapias-alvo.
Introdução
Com o desenvolvimento de terapias-alvo e inibidores de checkpoints imunes, tornou-se de extrema importância caracterizar melhor as interações entre células cancerosas e seu microambiente tumoral, e este é atualmente um importante campo de pesquisa translacional. A ETM é composta por uma infinidade de diferentes tipos celulares, com um balanço de células imunocitotóxicas direcionadas às células cancerosas e células imunomoduladoras que poderiam favorecer o crescimento tumoral e a invasividade 1,2,3,4. A caracterização desse ambiente complexo pode melhorar a compreensão das doenças oncológicas e ajudar cientistas e clínicos a descobrir novos biomarcadores preditivos e prognósticos, a fim de melhor selecionar pacientes para tratamentofuturo5,6. Por exemplo, Galon e sua equipe desenvolveram o Immunoscore, que é um método de pontuação reprodutível que pode ser usado como um biomarcador preditivo. O Immunoscore é calculado utilizando-se a densidade de células T CD3+ e CD8+ na margem invasiva e no centro dotumor7,8.
Ao longo das últimas décadas, soluções comerciais para mIF foram desenvolvidas, mas estas são frequentemente caras e projetadas para painéis específicos de antígenos. Para superar a necessidade de painéis específicos de antígenos em pesquisas acadêmicas e translacionais, desenvolvemos um método custo-efetivo para realizar mIF em cortes tumorais FFPE, permitindo a coloração de dois a seis antígenos adicionados à contracoloração de núcleos celulares em amostras humanas e de camundongos.
Uma vez que todos os cortes de tecido são corados e escaneados com um scanner de lâminas de fluorescência, as amostras podem ser analisadas por vários softwares de análise de imagem que suportam grandes conjuntos de dados piramidais. Finalmente, os dados brutos podem ser usados em um ambiente para computação estatística e gráficos como o software R (v.4.0.2), a fim de realizar análises de densidade e baseadas no espaço.
Um protocolo otimizado para coloração de cinco marcadores, bem como truques e dicas para otimizar novos painéis, é apresentado neste manuscrito. Além disso, são explicadas etapas detalhadas da análise das imagens e das funções R utilizadas para a análise estatística e espacial.
Protocolo
Todas as amostras utilizadas no presente protocolo foram provenientes de estudo aprovado pelos comitês de ética locais e autorizado pela autoridade competente. Todos os participantes do estudo assinaram o termo de consentimento livre e esclarecido. O estudo está registrado no ClinicalTrials.gov (NCT03608046).
1. Imunofluorescência multiplex
- Seccionamento FFPE
- Fixar o tecido em paraformaldeído a 4% e embutir o tecido fixado em parafina.
- Corte cortes de 5 μm e coloque-os em lâminas de microscópio adesivo.
- Secar as lâminas durante a noite à temperatura ambiente (TR).
- Desparafinização e inibição de peroxidases endógenas
- Desencefalar os tecidos submergindo as lâminas em tolueno (3x por 5 min cada) e metanol (3x por 5 min cada) sob uma capela de fumaça.
- Inibir as peroxidases endógenas submergindo as lâminas em peróxido de hidrogênio a 3% diluído em metanol por 20 min sob uma capela de fumaça.
- Enxaguar as lâminas em destilado (d) H2O (1x por 3 min).
- Coloração por imunofluorescência multiplex
- Submergir as lâminas em um frasco corante de 300 mL contendo citrato 10 mM (pH 6) ou tampão EDTA (pH 9) complementado com TritonX-100 0,1%.
NOTA: O tampão utilizado (pH 6 ou pH 9) depende do antigénio corado (ver Tabela 1). - Coloque o frasco de coloração com a tampa fechada no micro-ondas por 3-5 minutos na potência máxima (por exemplo, 900 W) até que o tampão comece a ferver.
NOTA: O tempo ideal para ferver depende do micro-ondas e do volume do tampão. Ajustes podem ser necessários para encontrar o momento perfeito. Para alguns antígenos frágeis ou espécimes frágeis e menos aderentes (por exemplo, organoides e esferoides), a ebulição no micro-ondas pode ser muito dura. Neste caso, uma panela de pressão pode ser usada em seu lugar. - Mantenha o tampão em temperatura de quase ebulição, colocando o frasco de coloração fechado no micro-ondas em baixa potência (por exemplo, 90 W) por 15 min.
- Execute a última etapa de aquecimento colocando o micro-ondas na potência máxima por 90 s.
- Retire o frasco do micro-ondas e deixe o tampão esfriar por 15 min no RT.
- Enxaguar as lâminas 3x por 5 min cada em dH2O e 1x por 5 min em solução salina tamponada tris-tamponada contendo Tween 20 0,1% (TBS-T).
- Remova o TBS-T borrando as lâminas em um papel toalha
- Coloque as lâminas (planas) em uma bandeja da câmara de coloração ou caixa de lâmina do microscópio (consulte Tabela de Materiais).
- Circunde o tecido com uma caneta hidrofóbica.
- Bloquear os sítios de ligação inespecíficos cobrindo o tecido com albumina de soro bovino (BSA) a 5% dissolvida em TBS-T por 30 min.
- Remova o buffer de bloqueio borrando as lâminas em um papel toalha.
NOTA: Não enxágue as lâminas após a etapa de bloqueio. - Incubar o tecido durante 60 minutos com o anticorpo primário (ver Tabela 1) diluído em TBS-T a 1% da BSA, cobrindo o tecido com cerca de 300 μL da solução.
- Enxágue as lâminas 3x por 3 min cada com TBS-T.
- Incubar o tecido durante 40 minutos com o anticorpo secundário poli-HRP (ver Tabela 1), cobrindo o tecido com cerca de 300 μL da solução.
- Enxágue as lâminas 3x por 3 min com TBS-T.
- Incubar o tecido durante 10 minutos com o reagente fluorocromo-tiramida (ver Tabela 1) diluído 200 vezes em tampão borato (borato 0,1 M, pH 7,8, NaCl 3 M) suplementado extemporaneamente com 0,003% H 2 O2, cobrindo o tecido com cerca de 300 μL da solução.
- Enxágue as lâminas 3x por 3 min com TBS-T.
- Repita os passos 1.3.1-1.3.16 até que toda a coloração TSA tenha sido realizada.
- Incubar o tecido durante a noite a 4 °C com o último anticorpo primário (ver Tabela 1) diluído em TBS-T a 1% de BSA.
NOTA: Como a incubação é durante a noite, é importante cobrir a bandeja da câmara de coloração ou a caixa de lâmina do microscópio e adicionar dH2O em um papel toalha no fundo da caixa (sob as lâminas) para garantir que os tecidos não sequem durante a incubação. - Enxaguar o tecido 3x por 5 min cada com TBS-T.
- Incubar o tecido por 120 min com o anticorpo secundário (diretamente acoplado ao fluorocromo) diluído 200 vezes em BSA TBS-T a 1%.
- Enxaguar o tecido 3x por 5 min cada com TBS-T.
- Corar os núcleos incubando o tecido por 5 min em bisbenzimida (20 mM) diluída 1.000 vezes em BSA TBS-T a 10%.
NOTA: A bisbenzimida pode ser substituída por DAPI, mas esta última é mais tóxica e tem de ser manuseada cuidadosamente sob um exaustor. - Enxaguar o tecido 3x por 3 min cada em dH2O.
- Monte as lâminas usando um meio de montagem de fluorescência e vidros de cobertura de borossilicato.
- Submergir as lâminas em um frasco corante de 300 mL contendo citrato 10 mM (pH 6) ou tampão EDTA (pH 9) complementado com TritonX-100 0,1%.
2. Digitalização de slides
- Digitalize as lâminas digitalizando-as em um scanner de lâminas de fluorescência com ampliação de 20x (os detalhes do scanner de slides são fornecidos na Tabela de Materiais).
NOTA: Uma varredura representativa de um multiplex ideal é mostrada na Figura 1.
3. Análise das imagens
- Importe as digitalizações para um software de análise de imagem (File > Open Image).
- Vá para a guia Classificadores e selecione o plug-in DenseNet AI V2 .
- Treine o plugin DenseNet AI V2 para reconhecer núcleos cercando cerca de 500 núcleos em uma imagem.
- Treine a IA em várias outras lâminas do mesmo lote e diferentes lotes de coloração mIF, cercando vários núcleos (50) em várias lâminas (cerca de 10).
NOTA: Instruções detalhadas sobre como usar o plugin AI podem ser encontradas no manual do software. O uso de IA para detecção de núcleos é opcional. Outros métodos para detecção de núcleos estão disponíveis dependendo do software de análise de imagem utilizado. - Salve a IA treinada (Ações do Classificador > Salvar).
- Vá para a guia Anotações e crie uma anotação para cada região de interesse (ROI), como o centro do tumor e a margem do tumor, usando a Pen Annotation Tool.
- Se necessário, remova as regiões com dobras e as regiões que aparecem borradas com a Ferramenta de Anotação de Exclusão.
NOTA: A coloração de hematoxilina-eosina de um corte adjacente ao usado para mIF pode ser realizada antes da coloração de mIF para garantir que as células tumorais estejam presentes no espécime e para ajudar os anatomopatologistas a determinar as ROIs. - Vá para a guia Análise e selecione o algoritmo HighPlex FL (Configurações Ações > Carregar > HighPlex FL).
- Selecione a guia Seleção de corante e selecione o corante de interesse.
- Na guia Detecção Nuclear , vá para Tipo de Segmentação Nuclear e selecione AI personalizada.
- Em Classificador de Segmentação Nuclear, selecione a IA salva na etapa 3.5.
- Na aba Detecção de Membrana e Citoplasma, escolheu-se o raio citoplasmático máximo (neste estudo, utilizou-se 1,5) e o número de corantes de membrana.
- Para cada corante, selecione o limiar positivo do núcleo, o limiar positivo do citoplasma e o limiar positivo da membrana.
NOTA: O limiar é diferente para cada coloração e deve ser ajustado para cada lote de lâminas e cada antígeno corado. O uso da ferramenta Configurações de exibição (Exibir > Configurações de exibição) pode ajudar a selecionar um limite adequado usando o valor de intensidade no final do pico de intensidade (à direita). - Para cada corante, selecione os valores de completude do núcleo, membrana e citoplasma.
NOTA: Este parâmetro é importante para evitar a detecção de falso positivo quando duas células com coloração diferente estão próximas uma da outra (Figura 2). - Salve o algoritmo (Ações de configurações > Salvar).
- Analise as ROIs (Analisar > camada de anotação).
- Vá para a guia Resultados e selecione todos os dados em Dados do objeto (ctrl + A).
- Exporte os dados em .csv formato (clique com o botão direito do mouse > Exportar > dados do objeto . CSV).
Observação : esta tabela contém a posição (Xmin, Xmax; Ymin, Ymax) e a positividade de cada marcador de cada célula analisada.
4. Bioinformática usando R
NOTA: Um script R fornecendo mais detalhes sobre as seguintes etapas está disponível no GitHub (benidovskaya/Ring: Pipeline para a análise de colorações de imunofluorescência multiplex. [github.com])
- Usando a tabela exportada, primeiro defina os diferentes tipos de células com base nas colorações de colocalização. Por exemplo, defina células T citotóxicas por células CD3+/CD8+ duplamente positivas.
- Em seguida, reconstrua uma imagem simplificada do slide em um gráfico de pontos usando as coordenadas exportadas do software de análise de imagens e ggplot2 (Figura 3). Usando esses dados, vários tipos de análise podem ser realizados:
- Análise de densidade
NOTA: A análise mais simples é uma análise de densidade.- Realizar uma análise de densidade para todos os tipos celulares usando a lâmina inteira para biópsias ou alguma área específica do tecido. Por exemplo, calcular a densidade de células T CD3+ e CD8+ no centro do tumor e na margem do tumor (Figura 4A-C).
- Para calcular essas densidades, use um software de análise de imagem para produzir um quadro de dados específico por amostra com o fenótipo e as coordenadas de cada célula. Através de uma função de agrupamento (vizinho k-mais próximo) em R, crie um objeto poligonal usando as bordas da biópsia estudada e calcule a densidade dos tipos celulares de interesse dentro dele.
NOTA: Isso permite comparar as densidades de diferentes tipos celulares entre diferentes condições (como diferentes pontos de tempo, tipos de tratamento, tipos de tecido e resposta ao tratamento) e localizações (centro do tumor, margem do tumor, fibrose do estroma e área de necrose), dependendo da hipótese biológica. Devido à alta proximidade entre células cancerosas, células peritumorais e células infiltrantes de tumor, o software de análise de imagem pode detectar células duplamente positivas como células imunes e cancerosas ao mesmo tempo. Nesse caso, é preciso corrigir bioinformaticamente essa questão, mencionando quais são essas células duplamente positivas. Neste caso, as células CD3+CD8+citoqueratinas+ foram marcadas como células citotóxicas porque a positividade da citoqueratina foi devida às células tumorais ao redor dos linfócitos infiltrantes.
- Mapas de calor
- Usando a densidade de cada tipo de célula a partir de diferentes painéis e aplicando uma normalização (por exemplo, centralização de escala), desenhe mapas de calor (Figura 5) representando a abundância celular na população de amostras.
- Usando agrupamento hierárquico não supervisionado com base na densidade celular, agrupar pacientes com composições semelhantes de ETM e correlacionar esses agrupamentos com parâmetros clínicos, como resposta ao tratamento e sobrevida.
NOTA: Heatmaps e clusterização podem ser facilmente executados com o pacote R ComplexHeatmap9.
- Distribuição espacial celular
- Calcular bioinformaticamente as distâncias entre as células (por exemplo, células imunes e tumorais; Figura 6A, B) com base nas coordenadas celulares fornecidas pela análise das imagens. Use as distâncias mediana e média entre os tipos celulares de interesse para comparar a proximidade celular em todas as amostras de uma coorte.
- Funções descritivas espaciais
- Use a função G-cross vizinha mais próxima, disponível através do pacote R spatstat10, para determinar a probabilidade de uma célula de interesse, X (por exemplo, uma célula tumoral), encontrar a célula mais próxima, Y (por exemplo, uma célula T), dentro de um determinado raio em torno da célula X.
- Calcular a área sob a curva empírica para obter um valor numérico que represente a infiltração tumoral de células T CD3+ ao redor das célulastumorais11 (Figura 6C). Use outras funções descritivas espaciais, como a função F ou a função J12.
- Análise do imunoescore
- Calcule o Immunoscore (I), desenvolvido pela equipe de Galon7,8, utilizando a densidade de células T CD3+ e CD8+ no centro do tumor e a margem invasiva do tumor.
OBS: O escore varia de I0 a I4. Uma baixa densidade de células T CD3+ e CD8+ no centro e na margem do tumor está associada a um escore de I0, enquanto uma alta densidade de células T CD3+ e CD8+ em ambas as regiões está associada a um escore de I4. Recentemente, o impacto prognóstico do Immunocore foi validado em um estudo com amostras de 2.681 pacientes com câncer de cólon estádio I-III de 14 centros de 13 países7. No entanto, para ser computado, o Immunoscore requer um espécime ressecado cirurgicamente contendo o centro e a margem do tumor. Para biópsias, que geralmente não têm margem, um Immunoscore adaptado para biópsia foi recentemente desenvolvido13. - Para calcular o Immunoscore adaptado para biópsia, converta o valor da densidade de células T CD3+ e CD8+ em um percentil e, em seguida, use o percentil médio de células T CD3+ e CD8+ para pontuar em uma das três categorias (baixa, intermediária e alta)13.
- Calcule o Immunoscore (I), desenvolvido pela equipe de Galon7,8, utilizando a densidade de células T CD3+ e CD8+ no centro do tumor e a margem invasiva do tumor.
- Análise de hotspots
- Utilizar a análise de hotspots, utilizando a função quadratcount (spatstat)10, para comparar as densidades dos diferentes tipos celulares na área mais infiltrada do tecido. Por exemplo, é possível calcular um escore "Immunoscore-like" usando o valor da densidade de células T CD3 e CD8 dos quadrados mais infiltrados do tecido (Figura 7). Aplicar este método para a análise de qualquer tipo de célula com uma distribuição não homogênea através do tecido.
- Análise de densidade
Resultados
Seguindo esse protocolo, vários parâmetros devem ser investigados para garantir que o tecido seja corado corretamente. Em primeiro lugar, a coloração TSA deve exibir uma boa faixa dinâmica ao usar tempos de exposição baixos (tipicamente 2-100 ms) durante o processo de varredura. Um baixo tempo de exposição implica que a amplificação foi feita corretamente durante a reação com HRP. Para antígenos corados com o anticorpo secundário diretamente acoplado ao fluorocromo, o tempo de exposição poderia ser muito maior, o que poderia levar ao fotoclareamento (diminuição da intensidade do sinal devido a um longo tempo de exposição). Em segundo lugar, é importante verificar se cada coloração apresenta uma SNR alta. Um alto sinal de fundo com baixo sinal antigênico pode ser uma indicação de que o anticorpo primário não é específico o suficiente, que as peroxidases endógenas não foram inativadas corretamente ou que uma etapa do protocolo não foi feita adequadamente. Em terceiro lugar, dependendo do scanner de slides e dos conjuntos de filtros usados para a digitalização, é possível ver sobreposições entre duas cores (por exemplo, AF555, AF594 e AF647). A escolha dos conjuntos de filtros corretos no scanner e a diluição correta de anticorpos primários são cruciais para evitar possíveis detecções cruzadas. O controle de qualidade consiste na detecção de células coradas únicas para cada marcador no arquivo digitalizado. Finalmente, também é importante adicionar um controle positivo e negativo para cada lote de coloração. Para as células imunes, a amígdala é um bom controle positivo. Um resultado representativo da coloração ótima é mostrado na Figura 1.
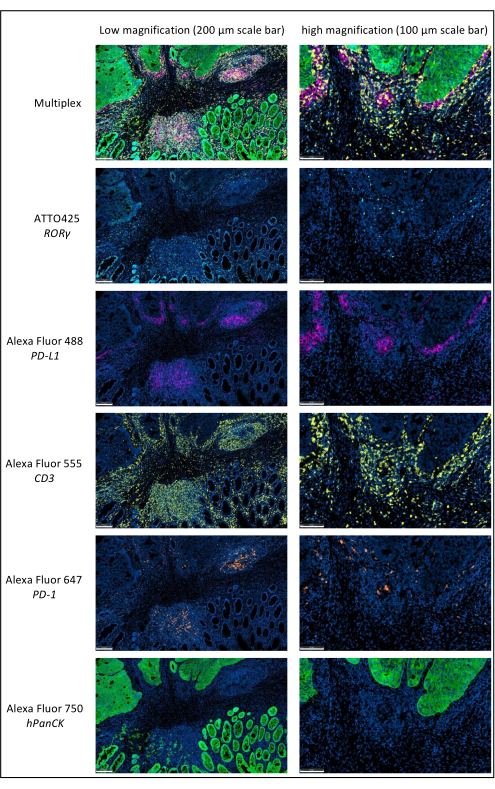
Figura 1: Câncer de reto localmente avançado corado por imunofluorescência multiplex. Abreviações: PD-1 = proteína de morte celular programada 1; PD-L1 = Morte programada-ligante 1; ROR-γ = receptor órfão gama relacionado ao RAR; CD3 = agrupamento de diferenciação 3; hPanCK = pancitoqueratina humana. Cada coloração antigênica é escaneada em escala de cinza, e as cores apresentadas na figura são pseudocores. Ampliação baixa da barra de escala: 200 μm; ampliação da barra de escala: 100 μm. Clique aqui para ver uma versão maior desta figura.
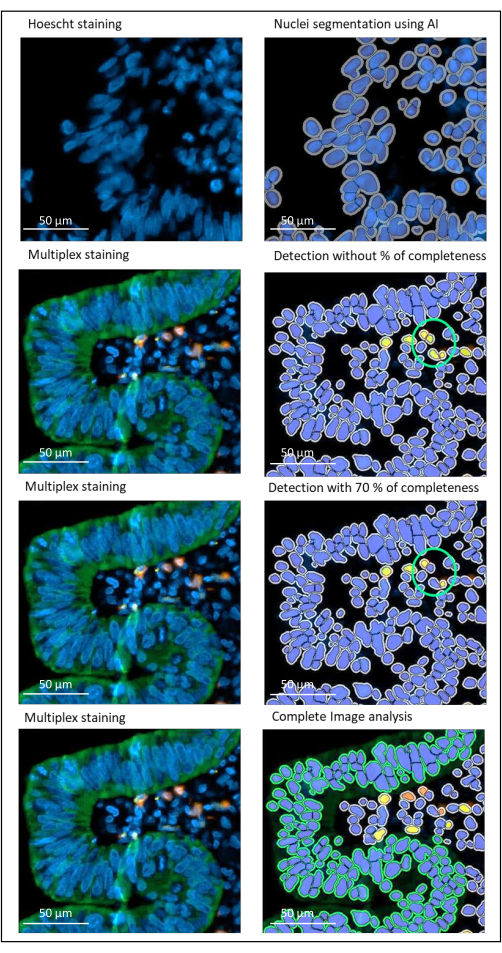
Figura 2: Detecção de núcleos e coloração de câncer retal localmente avançado utilizando um software de análise de imagem. Sem o parâmetro de porcentagem de completude definido corretamente, o software detecta duas células CD8+ (círculo verde) porque elas estão próximas uma da outra, mas apenas uma célula é corada. Usar 70% de completude ajuda a evitar essa detecção de falso positivo. Verde = hPanCK; Amarelo = CD3; Laranja = CD8. Barra de escala: 100 μm Clique aqui para ver uma versão maior desta figura.
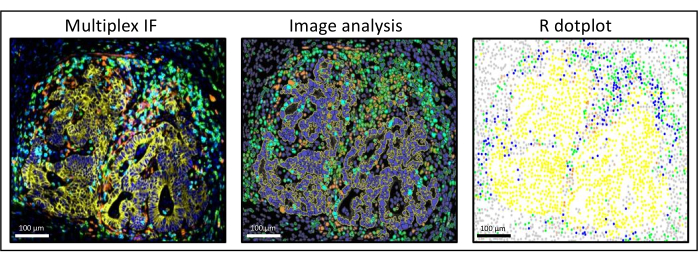
Figura 3: Análise de imagem e reconstituição do dot-plot R de metástase de câncer colorretal hepático. Na coloração multiplex (esquerda), a pancitoqueratina humana está em amarelo, CD3 em verde, CD8 em azul claro e IDO em laranja. No dot-plot (à direita), as células pan-citoqueratinas+ humanas estão em amarelo, as células CD3+CD8− estão em verde, as células CD3+CD8+ estão em azul e as células IDO+ estão em laranja. Clique aqui para ver uma versão maior desta figura.
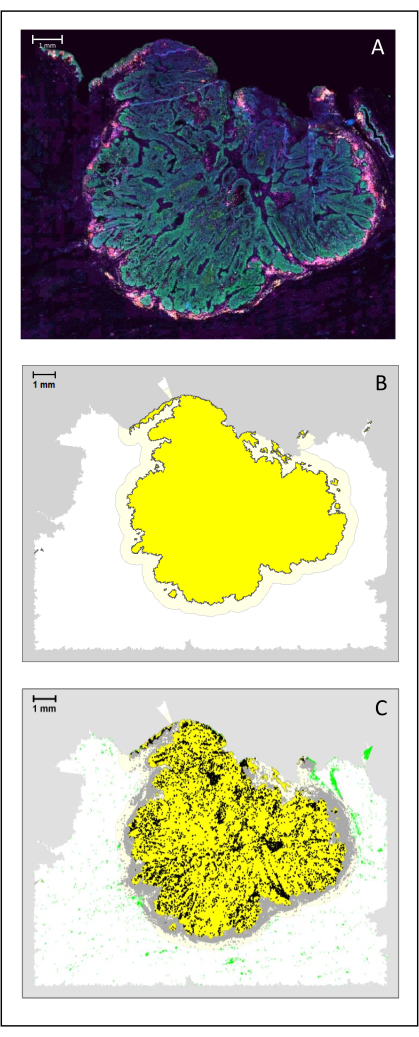
Figura 4: Análise de uma secção cirúrgica de um CECP. (A) Uma secção cirúrgica de um CEC de cabeça e pescoço. As células cancerosas são visíveis em verde. Células peritumorais são visualizadas ao redor das ilhotas tumorais (CD3 em amarelo e CD8 em roxo). (B) O centro do tumor (em amarelo com borda preta) é computado bioinformaticamente pelo algoritmo k-próximo-vizinho baseado na distância entre as ilhas tumorais de uma única área. Ao redor dessa área, uma margem invasora (amarelo claro com uma borda cinza) é calculada em uma base arbitrária de 500 μm. (C) As células T invasivas são destacadas com pontos pretos no centro do tumor e pontos cinzentos na margem invasiva. Outras células T são destacadas em pontos verde-claros. Barra de escala: 1 mm. Clique aqui para ver uma versão maior desta figura.
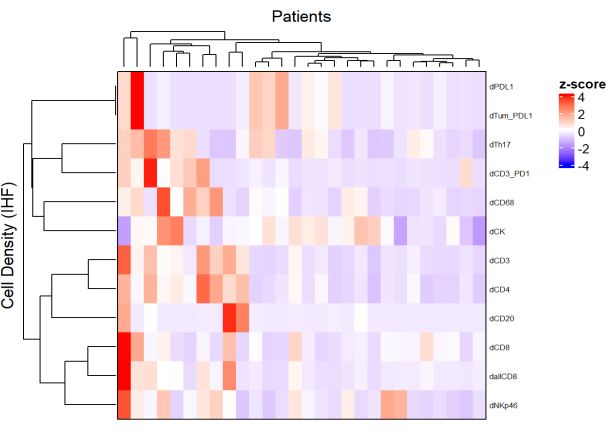
Figura 5: Mapa de calor da densidade de diferentes tipos celulares de biópsias de câncer retal localmente avançado. O mapa de calor foi desenhado usando agrupamento não supervisionado das densidades de diferentes tipos de células de diferentes painéis multiplex com o pacote ComplexHeatmap. Escala e centralização foram usadas para normalização. Clique aqui para ver uma versão maior desta figura.
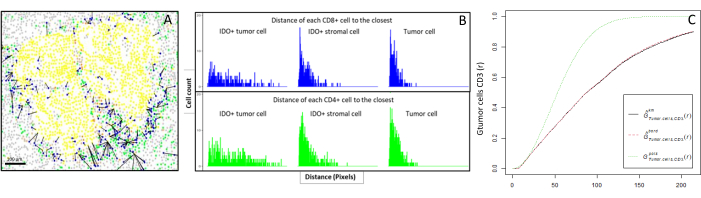
Figura 6: Distância das células CD4+ e CD8+ a cada célula IDO+ ou tumoral. As células humanas pan-citoqueratinas+ estão em amarelo, as células CD3+CD8− estão em verde, as células CD3+CD8+ estão em azul e as células IDO+ estão em laranja. (A) A distância mais próxima entre as células tumorais e cada célula T CD8+. (B) Barplots das distâncias entre as células IDO+ e cada célula T CD8+ (azul) ou célula T CD4+ (verde). (C) Exemplo de uma amostra analisada pela função G-cross. O eixo y mostra a probabilidade de uma célula tumoral encontrar um linfócito CD3+ em um raio que varia de 0-200 μm ao redor da célula tumoral. Três curvas são mostradas; a curva teórica está em verde pontilhado (distribuição de Poisson), a curva empírica corrigida com correção km está em preto e a curva empírica corrigida com correção de borda está em vermelho pontilhado. Clique aqui para ver uma versão maior desta figura.
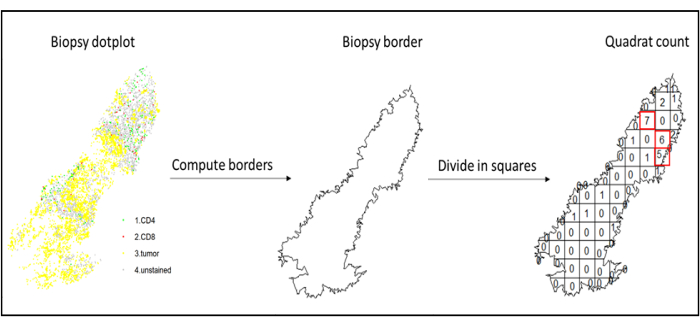
Figura 7: Ilustração de um Quadratcount. O cálculo das bordas e a contagem de quadrados foram realizados usando o pacote spatstats. As praças mais infiltradas (hotspots) podem ser usadas para estatísticas a jusante. CD4 está em verde, CD8 está em vermelho e as células tumorais estão em amarelo. Clique aqui para ver uma versão maior desta figura.
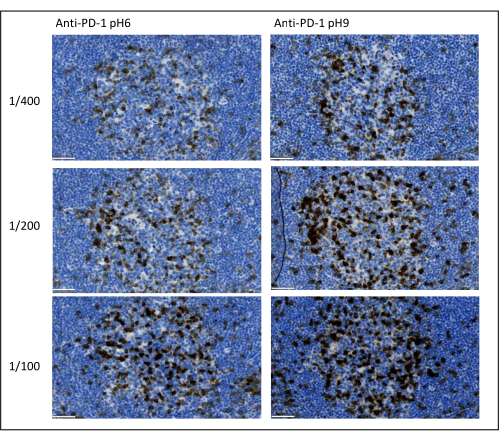
Figura 8: Diluição de anticorpos e otimização da recuperação de antígenos. Detecção cromogênica de PD-1 utilizando três diluições diferentes e duas soluções diferentes de recuperação antigênica do anticorpo primário (Citrato pH 6 e EDTA pH 9). Barra de escala: 50 μm. Clique aqui para ver uma versão maior desta figura.
| Anticorpo primário | Diluição | Recuperação de antígenos | Anticorpo secundário | Fluorochrome | Posição |
| PD-1 | 1/100 | EDTA (pH 9) | Anti-coelho | AF647 | 1 |
| PD-L1 | 1/1000 | EDTA (pH 9) | Anti-coelho | AF488 | 2 |
| ROR-γ | 1/200 | EDTA (pH 9) | Anti-mouse | ATT0-425 | 3 |
| CD3 | 1/100 | Citrato (pH 6) | Anti-coelho | AF555 | 4 |
| hPanCK | 1/50 | Citrato (pH 6) | Anti-mouse acoplado com AF750 | 5 | |
Tabela 1: Exemplo de um painel multiplex otimizado. Abreviações: PD-1 = proteína de morte celular programada 1; PD-L1 = Morte programada-ligante 1; ROR-γ = receptor órfão gama relacionado ao RAR; CD3 = agrupamento de diferenciação 3; hPanCK = pancitoqueratina humana; AF = AlexaFluor; EDTA = ácido etilenodiaminotetracético. CD3 é usado para detectar linfócitos T; PD-1 é usado para detectar linfócitos esgotados; ROR- γ é usado para detectar Th-17; e hPanCK é usado para detectar células tumorais. A coluna de posição indica a ordem em que o multiplex sequencial deve ser executado.
Discussão
Os parâmetros mais importantes a serem levados em consideração para otimizar a coloração multiplex são a diluição, a especificidade e a recuperação antigênica usada para cada anticorpo primário. Antes de iniciar um protocolo multiplex, a diluição ótima de cada anticorpo primário e a recuperação ótima do epítopo (pH 6 ou pH 9) devem ser testadas usando coloração cromogênica (DAB). Aconselhamos testar três diluições para cada tampão de recuperação do antígeno: a diluição que normalmente é especificada pela marca que comercializa o anticorpo, a mesma diluição dividida duas vezes e a mesma diluição multiplicada duas vezes (Figura 8). A escolha da diluição correta é um passo muito importante para verificar a especificidade do anticorpo e otimizar a relação sinal-ruído (SNR) da coloração. Depois de escolher a diluição correta no DAB, a mesma diluição deve ser testada para cada anticorpo primário usando TSA uniplex. Uma vez que o tampão de diluição e recuperação do epítopo são selecionados para cada coloração de antígeno, também é importante configurar corretamente a sequência do multiplex; Especificamente, alguns antígenos são melhor corados na primeira posição e outros na última posição. Aconselhamos testar a marcação multiplex usando todas as permutações de ordem possíveis para escolher qual coloração de antígeno deve vir primeiro, segundo, etc. Este também é um passo muito importante porque alguns antígenos frágeis podem ser degradados após várias rodadas de recuperação de epítopos, e alguns antígenos são melhor corados após várias rodadas de recuperação de epítopos. Por exemplo, a relação S/R é sempre maior na última posição para CD3 e na primeira posição para coloração PD-1. Além disso, a coloração de vários antígenos co-localizados pode ser dificultada por um efeito guarda-chuva (a saturação dos sítios reativos à tiramida). Isso pode ser atenuado pela diminuição da concentração de tiramida. Quando a expressão de um antígeno é condicionada pela expressão de outro (CD8 presente apenas nas células T que expressam CD3), aconselha-se a coloração do antígeno com a expressão mais ampla (CD3 neste caso) após o outro. Finalmente, a escolha do fluorocromo correto para cada coloração antigênica de acordo com as especificidades do scanner também é um passo importante para evitar a detecção cruzada.
As principais vantagens desta técnica são a amplificação e a relação sinal-ruído obtida. No entanto, essa técnica vem com uma limitação, que é que a coloração é sequencial, e os fluorocromos são ligados covalentemente ao tecido. No entanto, após a realização de todas as rodadas de amplificação do sinal da tiramida, também é possível adicionar uma última coloração com um anticorpo secundário diretamente acoplado a um fluorocromo (sem TSA). Em alguns painéis, utilizamos este método para adicionar coloração no canal 750. Isso foi necessário porque nenhuma tiramida-AF750 estava disponível comercialmente naquela época. É importante ressaltar que o tempo de exposição (durante o exame) do antígeno corado com AF750 será muito maior do que para os outros antígenos corados com TSA. Nesse caso, aconselhamos a coloração de uma proteína altamente expressa, como a citoqueratina, ou o aumento da concentração do anticorpo primário. Ao fazer isso, é possível manchar um máximo de cinco a seis antígenos por lâmina em um lote, dependendo do scanner de fluorescência.
Em oposição, várias técnicas comercialmente disponíveis usam coloração seriada com várias rodadas de coloração, varredura e remoção ou fotoclareamento para melhorar o número de antígenos que podem ser corados em um único corte de tecido. No entanto, essas técnicas são muitas vezes demoradas, caras, não têm amplificação de sinal, requerem etapas computacionais avançadas para mesclar corretamente os exames seriados e, em nossa experiência, podem induzir danos teciduais irreversíveis devido às inúmeras etapas do procedimento. No entanto, tem sido relatado que até 30 antígenos podem ser corados em um único tecido usando este método14.
Em conclusão, nosso método é uma técnica de imunohistofluorescência robusta, reprodutível, fácil de usar e custo-efetiva que pode ser usada em qualquer laboratório que possua um scanner de lâminas de fluorescência. Qualquer anticorpo primário comercializado adequado para IHC pode ser usado, e os painéis não são específicos para nenhum kit comercial. A análise de imagens pode ser feita em vários programas diferentes, incluindo programas de código aberto como QuPath e R. No entanto, pensamos que este método poderia até ser melhorado no futuro para grandes painéis de antígenos, permitindo a realização de coloração/varredura seriada da mesma lâmina com diferentes painéis de antígenos e com a vantagem da amplificação do sinal.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Os autores gostariam de agradecer à Dra. Derouane F por sua ajuda e apoio. Nicolas Huyghe é investigador apoiado por uma bolsa do Fundo Nacional de Investigação Científica da Bélgica (Télévie/FNRS 7460918F).
Materiais
| Name | Company | Catalog Number | Comments |
| anti-CD3 primary antibody | Abcam | ab16669 | rabbit monocolonal |
| anti-CD8 primary antibody | DAKO | M710301 | mouse monoclonal |
| anti-hPanCK primary antibody | DAKO | M3515 | mouse monoclonal |
| anti-PD-1 primary antibody | Cell Signalling | D4W2J | rabbit monocolonal |
| anti-PD-L1 primary antibody | Cell Signalling | 13684 | rabbit monocolonal |
| anti-RORC primary antibody | Sigma | MABF81 | mouse monoclonal |
| ATTO-425 | ATTOtec | ||
| Axioscan Z1 | Zeiss | Light source: Colibri 7 (385, 430, 475, 555, 590, 630, 735 nm) Filtersets: Excitation 379/34 – beam splitter 409 – emission 440/40; Excitation 438/24 – beam splitter 458 – emission 483/32; Excitation 490/20 – beam splitter 505 – emission 525/20; Excitation 546/10 – beam splitter 556 – emission 572/23; Excitation 592/21 – beam splitter 610 – emission 630/30; Excitation 635/18 – beam splitter 652 – emission 680/42; Excitation 735/40 – beam splitter QBS 405 + 493 + 611 + 762 - emission QBP 425/30 + 524/51 + 634/38 + 785/38; Objective: Plan-Apochromat 20x/0.8; Camera : Orca Flash 4.0 V3 | |
| Borosilicate Cover Glass | VWR | 631-0146 | |
| Envision+ anti-mouse | DAKO | K4001 | |
| Envision+ anti-rabbit | DAKO | K4003 | |
| Fluorescence mounting medium | DAKO | S3023 | |
| Goat anti-Mouse IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 750 | ThermoFischer | A-21037 | |
| HALO software | Indicalabs | ||
| Hoescht | Sigma | 14533 | |
| Superfrost plus microscope slides | Fisherscientific/Epredia | 10149870 | |
| Tyramide-AF488 | ThermoFischer | B40953 | |
| Tyramide-AF555 | ThermoFischer | B04955 | |
| Tyramide-AF647 | ThermoFischer | B04958 |
Referências
- Ge, P., et al. Profiles of immune cell infiltration and immune-related genes in the tumor microenvironment of colorectal cancer. Biomedicine & Pharmacotherapy. 118, 109228 (2019).
- Fridman, W. H. The immune microenvironment as a guide for cancer therapies. Oncoimmunology. 1 (3), 261-262 (2012).
- Fridman, W. H., Pages, F., Sautes-Fridman, C., Galon, J. The immune contexture in human tumours: Impact on clinical outcome. in Nature Reviews. Cancer. 12 (4), 298-306 (2012).
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: The next generation. Cell. 144 (5), 646-674 (2011).
- Calu, V., et al. Key biomarkers within the colorectal cancer related inflammatory microenvironment. Scientific Reports. 11 (1), 7940 (2021).
- Havel, J. J., Chowell, D., Chan, T. A. The evolving landscape of biomarkers for checkpoint inhibitor immunotherapy. Nature Reviews. Cancer. 19 (3), 133-150 (2019).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Mlecnik, B., et al. Integrative analyses of colorectal cancer show immunoscore is a stronger predictor of patient survival than microsatellite instability. Immunity. 44 (3), 698-711 (2016).
- Gu, Z., Eils, R., Schlesner, M. Complex heatmaps reveal patterns and correlations in multidimensional genomic data. Bioinformatics. 32 (18), 2847-2849 (2016).
- Baddeley, A., Rubak, E., Turner, R. . Spatial Point Patterns: Methodology and Applications with R. , (2022).
- Barua, S., et al. Spatial interaction of tumor cells and regulatory T cells correlates with survival in non-small cell lung cancer. Lung Cancer. 117, 73-79 (2018).
- Parra, E. R. Methods to determine and analyze the cellular spatial distribution extracted from multiplex immunofluorescence data to understand the tumor microenvironment. Frontiers in Molecular Biosciences. 8, 668340 (2021).
- El Sissy, C., et al. A diagnostic biopsy-adapted immunoscore predicts response to neoadjuvant treatment and selects patients with rectal cancer eligible for a watch-and-wait strategy. Clinical Cancer Research. 26 (19), 5198-5207 (2020).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. The Journal of Histochemistry and Cytochemistry. 65 (8), 431-444 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados