Isolamento dei mitocondri senza tag in due fasi per migliorare la scoperta e la quantificazione delle proteine
In This Article
Summary
Presentiamo un protocollo in due fasi per l'isolamento dei mitocondri di alta qualità che è compatibile con la scoperta e la quantificazione delle proteine su scala proteoma. Il nostro protocollo non richiede ingegneria genetica ed è quindi adatto per studiare i mitocondri da qualsiasi cellula e tessuto primario.
Abstract
La maggior parte dei processi fisiologici e patologici, dal metabolismo centrale alla risposta immunitaria alla neurodegenerazione, coinvolgono i mitocondri. Il proteoma mitocondriale è composto da più di 1.000 proteine e l'abbondanza di ciascuna può variare dinamicamente in risposta a stimoli esterni o durante la progressione della malattia. Qui, descriviamo un protocollo per isolare mitocondri di alta qualità da cellule e tessuti primari. La procedura in due fasi comprende (1) omogeneizzazione meccanica e centrifugazione differenziale per isolare i mitocondri grezzi e (2) cattura immunitaria senza tag dei mitocondri per isolare organelli puri ed eliminare i contaminanti. Le proteine mitocondriali di ogni stadio di purificazione vengono analizzate mediante spettrometria di massa quantitativa e vengono calcolati i rendimenti di arricchimento, consentendo la scoperta di nuove proteine mitocondriali mediante proteomica sottrattiva. Il nostro protocollo fornisce un approccio sensibile e completo allo studio del contenuto mitocondriale in linee cellulari, cellule primarie e tessuti.
Introduction
I mitocondri sono organelli complessi e dinamici in grado di percepire e adattarsi alle esigenze metaboliche della cellula. Al centro della complessità del metabolismo cellulare, i mitocondri agiscono come centri metabolici in cui convergono le reazioni di metabolismo di carboidrati, proteine, lipidi, acidi nucleici e cofattori1. Servono anche come organelli di segnalazione per le vie della risposta immunitaria innata e in risposta ai cambiamenti negli ioni e nelle specie reattive dell'ossigeno 2,3. Ad oggi, circa 1.100 proteine sono state mappate sui mitocondri 4,5,6, ma possiamo supporre che molte altre rimangano da scoprire, specialmente quelle espresse solo in determinati tipi di cellule o transitoriamente in specifiche condizioni ambientali. Lo sviluppo di nuovi approcci per quantificare i cambiamenti nella composizione mitocondriale negli stati metabolici di interesse aumenterà la nostra conoscenza di questi organelli ed evidenzierà nuove vie terapeutiche per i disturbi caratterizzati da disfunzione mitocondriale7.
Attualmente sono disponibili diversi protocolli di isolamento dei mitocondri, con rese e livelli di purezza 8diversi. Gli approcci basati sulla centrifugazione sono i più popolari, grazie alla loro semplicità e al basso costo. Sebbene sia adatta alla maggior parte delle applicazioni, la centrifugazione differenziale ha lo svantaggio di ottenere una purezza mitocondriale inferiore e di richiedere grandi quantità di materiale di partenza quando vengono utilizzate applicazioni basate su gradienti di densità più complesse. Negli ultimi anni sono emersi nuovi metodi per l'isolamento dei mitocondri, come la cattura immunitaria basata su tag ("MITO-IP")9 e la selezione di organelli attivati dalla fluorescenza10. Sebbene entrambe le procedure possano generare campioni con elevata purezza, la prima richiede l'ingegneria genetica per marcare i mitocondri per la purificazione di affinità, rendendo i protocolli incompatibili con materiale primario proveniente da organismi non modificati o donatori umani. Nel frattempo, quest'ultimo dipende dall'accesso alla citometria a flusso e agli strumenti di selezione. La combinazione di diversi metodi di isolamento offre la promessa di generare protocolli più robusti e una maggiore purezza.
Qui, presentiamo un nuovo protocollo per l'isolamento dei mitocondri basato sulla combinazione di due metodi esistenti: (1) centrifugazione differenziale per isolare una frazione mitocondriale grezza e (2) cattura immunitaria senza tag dei mitocondri con perline superparamagnetiche legate covalentemente agli anticorpi contro la translocasi della membrana mitocondriale esterna 22 (Tomm22)11, una proteina ubiquitaria della membrana mitocondriale esterna (Figura 1). La procedura che descriviamo è compatibile con la spettrometria di massa quantitativa delle proteine e, poiché è priva di tag e non richiede manipolazione genetica, può essere applicata a una vasta gamma di modelli di ricerca, dalle linee cellulari ai fluidi corporei ai tessuti animali interi. Inoltre, l'uso di due fasi nel protocollo consente l'uso della proteomica sottrattiva 6,12 per la scoperta di nuove proteine mitocondriali e lo studio della loro espressione.
Protocol
I guanti devono essere indossati in ogni momento e le fasi di coltura cellulare eseguite sotto un cappuccio a flusso laminare. Le cellule sono mantenute in un incubatore a 37 °C con il 5% di CO2. La ricerca presentata in questo protocollo è stata approvata ed eseguita in conformità con l'Università di Losanna e le linee guida svizzere per l'uso di animali.
1. Coltura della linea cellulare di macrofagi RAW264.7
- Coltivare le cellule macrofagiche RAW264.7 dei topi nel mezzo di aquila modificato (DMEM) di Dulbecco con alto glucosio e glutammina integrati con siero bovino fetale inattivato dal calore al 5% (HI-FBS) e 100 UI / mL di penicillina e 100 μg / mL di streptomicina (P / S).
NOTA: Per isolare i mitocondri, è sufficiente una singola piastra confluente di 15 cm (circa 70 x 10 7 cellule RAW264.7). - Mantenere le cellule RAW264.7 in piastre di coltura tissutale. Una densità iniziale di semina di 1 x 105 cellule/ml porta ad una piastra confluente in 3 giorni. Utilizzare 25 ml di sospensione cellulare in un mezzo per una piastra di 15 cm. Le cellule RAW264.7 hanno un alto tasso di divisione cellulare e devono essere divise più frequentemente rispetto alla maggior parte delle linee cellulari.
- Scollegamento di celle RAW264.7
- Aspirare il mezzo e lavare le cellule una volta con soluzione salina tamponata fosfato (PBS).
- Per una piastra da 15 cm, aggiungere 8 ml di tampone di dissociazione RAW caldo (270 mM di cloruro di potassio, 30 mM di citrato di sodio diidrato in H2O, filtrato sterile) e incubare le cellule a 37 °C per 5 minuti.
- Aggiungere un volume equivalente di fluido alle piastre (diluizione 1:1 del tampone di dissociazione) e pipettare per staccare e omogeneizzare le celle.
- Trasferire la sospensione cellulare in un tubo conico e centrifugare il tubo a 300 x g per 3 minuti a temperatura ambiente.
- Aspirare il surnatante e risospendere il pellet in un volume appropriato di mezzi (descritto al punto 1.1) per il conteggio delle cellule.
NOTA: è possibile utilizzare altri metodi per staccare le cellule RAW264.7, come la tripsina o un raschietto cellulare. Tuttavia, questi metodi sono più duri sulle cellule e possono portare alla loro polarizzazione in macrofagi simili a M1 nei giorni successivi al distacco.
2. Isolamento e coltura di macrofagi derivati dal midollo osseo (BMDM)
NOTA: il protocollo descritto qui è per un singolo mouse e può essere ridimensionato per più mouse. Protocolli dettagliati per l'isolamento e la coltura BMDM sono stati descritti altrove13,14.
- Sacrificare un topo C57BL/6 di 8-12 settimane con una dose elevata di CO2.
NOTA: È possibile utilizzare topi maschi o femmine. - Spruzzare il mouse con etanolo al 75% per sterilizzarlo.
- Seziona e raccogli i fianchi, i femori e le tibie dal topo15.
- Per raccogliere il midollo osseo dai femori e dalle tibie, rimuovere l'estremità dell'articolazione del ginocchio di entrambe le ossa15. Recuperare il midollo osseo dai fianchi rimuovendo l'acetabolo.
- Trasferire le ossa in un tubo conico da 50 mL con 4 ml di mezzi BMDM mantenuti su ghiaccio (DMEM con alto glucosio e glutammina integrati con 5% HI-FBS, P / S e 10 mM HEPES).
NOTA: È importante mantenere le ossa nei supporti per evitare l'essiccazione del midollo osseo durante la dissezione. - Aggiungere 4 ml di PBS e 4 ml di fluidi BMDM caldi in due diversi pozzetti di una piastra a 6 pozzetti.
- Trasferire le ossa e il supporto dal tubo conico da 50 mL a un pozzetto vuoto della piastra a 6 pozzetti.
- Usando un paio di pinze, trasferire bene le ossa al PBS per lavarle.
- Trasferire le ossa nel pozzetto contenente mezzi BMDM caldi.
- Fare un foro di 1-2 mm di diametro sul fondo di due tubi da 0,5 ml con la pinza e posizionarli in un tubo da 1,5 ml.
NOTA: non è necessario aggiungere supporti al tubo per questo passaggio rapido. - In ogni tubo da 0,5 ml, posizionare un femore, una tibia e un'anca in modo tale che il midollo osseo delle ossa esposte sia rivolto verso il fondo dei tubi.
- Centrifugare i tubi a 13.000 x g per 1 minuto a temperatura ambiente per raccogliere il midollo osseo e il mezzo rimanente attraverso il foro del tubo da 0,5 mL e nel tubo da 1,5 ml. Eliminare i tubi da 0,5 mL con le ossa.
- Risospendere i pellet di midollo osseo in mezzi BMDM e trasferirli in un tubo conico da 15 ml.
- Aggiungere supporti BMDM fino a 10 ml.
- Posizionare un filtro cellulare da 40 μm su un tubo conico da 50 ml e filtrare la sospensione del midollo osseo attraverso di esso.
- Centrifugare la sospensione filtrata a 300 x g per 5 minuti a temperatura ambiente per recuperare le cellule intatte e rimuovere piccoli detriti dalla sospensione cellulare.
- Preparare 70 mL di BMDM media integrati con 50 ng/mL di fattore stimolante le colonie di macrofagi (M-CSF).
- Risospendere il pellet in 10 mL di mezzo BMDM integrato con M-CSF.
- Aggiungere 9 mL di media BMDM integrati con M-CSF a ciascuna delle sette piastre di Petri da 10 cm.
- Piastra 1 mL di cellule (circa 1 x 107 celle) in ciascuna delle sette piastre di Petri da 10 cm.
- Omogeneizzare la sospensione cellulare in ciascuna piastra mediante pipettaggio accurato su e giù sulla piastra e trasferire le piastre nell'incubatore.
- Dopo 3 giorni, aggiungere 5 ml di fluido BMDM caldo integrato con 50 ng/mL M-CSF a ciascuna piastra.
- Dopo 3 giorni (giorno 6, dopo l'isolamento del midollo osseo), verificare l'adesione e la differenziazione del BMDM mediante microscopia.
NOTA: A questo punto, è possibile procedere direttamente all'isolamento dei mitocondri (fase 3). In alternativa, si può riplaccare il BMDM, che consente di trattarli con citochine e piccole molecole. - Scollegamento dei BMDM
- Aspirare il mezzo da ciascuna piastra e aggiungere 7 ml di PBS freddo integrato con 5 mM di acido etilendiamminotetraacetico (EDTA).
- Incubare le cellule a 4 °C per 7-8 min.
- Staccare i BMDM con un pipettaggio accurato su e giù con una pipetta da 10 ml.
- Riunire i BMDM risospesi da tutte e sette le piastre in un unico tubo conico da 50 mL e centrifugare a 300 x g per 3 minuti a temperatura ambiente.
- Aspirare il surnatante e risospendere le cellule in 40 ml di mezzi BMDM caldi per il conteggio delle cellule.
NOTA: si ottengono tra 7-9 x 107 BMDM per mouse. Un minimo di 6 x 107 BMDM per l'isolamento dei mitocondri è raccomandato per la proteomica. - Se lo si desidera, placcare e trattare i BMDM secondo l'obiettivo sperimentale. In caso contrario, procedere direttamente al passaggio 3.3.
3. Preparazione di una frazione mitocondriale grezza mediante centrifugazione differenziale
NOTA: Eseguire tutte le fasi di centrifugazione a 4 °C. Sono necessarie due centrifughe, una con rotore oscillante e adattatori per tubi conici con una forza centrifuga relativa di almeno 300 x g, l'altra con una forza centrifuga relativa di almeno 21.000 x g adatta per tubi da 1,5 ml. Quando si utilizzano celle aderenti, utilizzare un raschietto cellulare.
- Per le cellule aderenti, aspirare il mezzo e aggiungere 10 ml di PBS per piastra da 15 cm.
NOTA: Raschiare le cellule in PBS consente di lavarle contemporaneamente. Se le celle sono già nella sospensione, procedere direttamente al passaggio 3.3. - Staccare le celle usando un raschietto cellulare e raggrupparle in un unico tubo conico da 50 ml. Omogeneizzare la sospensione cellulare mediante pipettaggio su e giù.
NOTA: Le celle possono essere staccate utilizzando un raschietto cellulare, poiché questo è più veloce e verranno lisate poco dopo. - Per ogni condizione sperimentale, trasferire il 5% del volume della sospensione cellulare in un tubo da 1,5 mL e centrifugarlo a 300 x g per 5 minuti a temperatura ambiente.
NOTA: Quando si utilizzano celle di sospensione, assicurarsi di lavare le celle una volta con PBS prima, per rimuovere possibili contaminanti dai supporti come FBS. - Scartare il surnatante e mantenere il pellet sul ghiaccio.
NOTA: Questo rappresenterà la frazione "cellula totale" per la proteomica. - Centrifugare il resto dei campioni dai punti 3.1 o 3.2 a 300 x g per 5 minuti a temperatura ambiente.
- Eseguire tutti i seguenti passaggi sul ghiaccio e utilizzando tamponi ghiacciati.
- Aspirare il surnatante e risospendere il pellet cellulare in 5 mL di tampone mitocondriale ghiacciato (MB) (210 mM mannitolo, 70 mM saccarosio, 10 mM HEPES/NaOH [pH 7,4] e 1 mM EDTA).
- Recuperare le cellule mediante centrifugazione a 300 x g per 5 minuti a 4 °C.
- Risospendere il pellet cellulare in 0,5 ml di MB freddo e trasferirlo in un tubo da 1,5 ml.
NOTA: Questa procedura produce una concentrazione cellulare approssimativa di 1,5 x 10 8 cellule BMDM / ml o 3 x 108 RAW264,7 cellule / ml. - Utilizzando una siringa da 1 mL dotata di un ago da 25 G, omogeneizzare la sospensione cellulare di 30 passaggi attraverso l'ago (Figura 1A).
- Aggiungere 1 mL di MB freddo al tubo e mescolare per inversione. Centrifugare la sospensione cellulare omogeneizzata a 2.000 x g per 5 minuti a 4 °C.
- Trasferire 1 mL di surnatante in un tubo fresco da 1,5 mL su ghiaccio senza disturbare il pellet cellulare. Risospendere il pellet cellulare e omogeneizzarlo di nuovo, come al punto 3.7.
- Raggruppare il pellet cellulare omogeneizzato e il surnatante delle due fasi precedenti in un unico tubo da 1,5 ml e centrifugarlo a 2.000 x g per 5 minuti a 4 °C.
NOTA: A questo punto, il pellet contiene principalmente nuclei e cellule ininterrotte e viene scartato. Il surnatante contiene detriti cellulari, citosol e organelli, compresi i mitocondri (Figura 1B). - Dividere il surnatante tra quattro tubi da 1,5 ml.
NOTA: la divisione del surnatante tra più tubi in questa fase migliora la rimozione dei contaminanti nei passaggi successivi. - Aggiungere MB per creare un volume finale di 1 mL in ciascuna delle quattro provette. Mescolare per inversione e centrifugare le provette a 13.000 x g per 10 minuti a 4 °C.
NOTA: Dopo questo passaggio, è visibile un pellet con due strati. Il pellet inferiore, sodo e marrone, contiene mitocondri e viene conservato per un'ulteriore purificazione (Figura 1C). Il pellet superiore, sciolto, bianco contiene altre strutture cellulari e può essere scartato. - Eseguire questo passaggio con attenzione. Rimuovere il surnatante con la maggior quantità possibile di pellet superiore bianco. Pipettando delicatamente su di esso, è possibile risospendere il pellet bianco e quindi scartarlo, lasciando intatto il pellet mitocondriale marrone.
- Tenere uno dei quattro tubi con il pellet mitocondriale sul ghiaccio. Questo rappresenta la frazione "mitocondri grezzi" per la proteomica.
- Raggruppare gli altri tre pellet in un tubo da 1,5 mL in un volume finale di 1 mL di MB.
4. Purificazione dei mitocondri basata su anticorpi superparamagnetici
NOTA: eseguire tutti i seguenti passaggi in una cella frigorifera a 4 °C.
- Trasferire 1 mL del preparato mitocondriale grezzo dal punto 3.18 a un tubo conico da 15 mL e aggiungere 7 mL di MB integrati con 150 mM NaCl (MB + NaCl).
NOTA: L'aggiunta di NaCl migliora il legame anticorpale e diminuisce il legame non specifico dei contaminanti alle perle e ai mitocondri. - Aggiungere 50 μL di perle Tomm22 alla sospensione di mitocondri grezzi da 8 mL (Figura 1D) e incubare il tubo per 15 minuti a 4 °C su una ruota rotante a bassa velocità.
NOTA: Le perle Tomm22 sono legate covalentemente agli anticorpi monoclonali Tomm22 allevati nel topo, accoppiati a perle superparamagnetiche. - Nel frattempo, posiziona una colonna sul magnete.
- Equilibrare la colonna con 8 mL di MB + NaCl ed eliminare il flowthrough.
- Dopo l'incubazione di 15 minuti a 4 °C del campione con le sfere Tomm22, trasferire il campione nella colonna. Eliminare il flusso continuo.
NOTA: I mitocondri rimarranno attaccati alle sfere magnetiche nella colonna (Figura 1E). - Lavare la colonna tre volte con 8 ml di MB + NaCl.
- Rimuovere la colonna dal magnete e posizionarla in un tubo conico da 15 ml.
- Eluire i mitocondri aggiungendo 1,5 ml di MB + NaCl alla colonna e applicare immediatamente uno stantuffo per eluire i mitocondri purificati nel tubo.
- Trasferire i mitocondri eluiti in una provetta da 1,5 mL e centrifugarli a 21.000 x g per 10 minuti a 4 °C.
NOTA: Si formerà un pellet marrone. Questo contiene i mitocondri isolati e alcune delle perle accoppiate agli anticorpi (Figura 1F). - Rimuovere con cura il surnatante dal pellet. Il pellet rappresenta la frazione "mitocondri puri" per la proteomica. Questo pellet insieme ai pellet delle fasi 3.4 e 3.17 può essere immagazzinato a -20 °C ed è pronto per le applicazioni a valle.
Representative Results
Nel presente protocollo vengono generati tre campioni con gradi crescenti di purezza mitocondriale: cellule totali, mitocondri grezzi ("mito-grezzi") e mitocondri puri ("mito-puri") (Figura 1). Abbiamo convalidato la purificazione dei mitocondri dalla linea cellulare macrofagica RAW264.7 caricando quantità proteiche uguali di ciascuna frazione su un gel e immunoblotting, e abbiamo scoperto che la citrato sintasi mitocondriale (Cs) era arricchita ad ogni fase di purificazione; nel frattempo, le proteine del citosol (GAPDH), della membrana plasmatica (Na/K ATPasi), del nucleo (Lamin B), dei lisosomi (Lamp1) e del reticolo endoplasmatico (ER) (Pdi) sono progressivamente scomparse (Figura 2A). Risultati simili sono stati ottenuti utilizzando BMDM. Per un'ulteriore convalida della purezza e dell'integrità dei mitocondri isolati, è stata eseguita la microscopia elettronica sulla frazione mitocondriale pura. Abbiamo osservato mitocondri con una classica forma ovale e creste intatte circondate da particelle dense di elettroni corrispondenti alle perle rivestite di anticorpi (Figura 2B). Pertanto, si può concludere che il nostro protocollo arricchisce i mitocondri, esaurisce altri componenti cellulari e mantiene l'integrità strutturale mitocondriale.
Successivamente, è stata eseguita un'analisi del proteoma di ciascuna frazione utilizzando la cromatografia liquida accoppiata alla spettrometria di massa (LC / MS). Sono state identificate un totale di 6.248 proteine nell'estratto da cellule totali, 907 delle quali erano state precedentemente annotate come mitocondriali nell'inventario MitoCarta3.05. Dopo aver filtrato le proteine con una soglia di almeno due peptidi unici, abbiamo calcolato un punteggio di arricchimento per ciascuna proteina in ciascun campione in base alla loro intensità rispetto alle cellule totali. Abbiamo quindi assegnato le proteine a sette principali compartimenti subcellulari: mitocondri, ER, lisosomi, apparato di Golgi, citoscheletro, nucleo e citosol, usando Gene Ontology (GO) 16,17 e MitoCarta3.05 come riferimenti. È importante sottolineare che è stato osservato un arricchimento medio per le proteine mitocondriali di oltre 10 volte e più di 20 volte nelle frazioni mitocondriali grezze e pure, rispettivamente, (Figura 2C). Al contrario, i componenti degli altri sei compartimenti cellulari analizzati sono stati esauriti durante la procedura di purificazione. Di particolare rilievo, nella frazione mitocondriale grezza, abbiamo osservato un arricchimento transitorio per ER e proteine lisosomiali, due classi di proteine contaminanti frequentemente presenti seguendo protocolli di centrifugazione differenziale18. Ciò era probabilmente dovuto alle interazioni organello-organello e a coefficienti simili di sedimentazione, specialmente per i lisosomi, che sono molto abbondanti nei macrofagi19. Mentre entrambi erano per lo più esauriti dopo la cattura immunitaria, abbiamo rilevato un piccolo segnale per le proteine dai siti di contatto ER-mitocondri nella frazione mito-pura.
Abbiamo quindi confrontato direttamente l'abbondanza proteica dalle cellule totali e dai campioni mito-puri e abbiamo osservato due popolazioni distinte, corrispondenti a proteine mitocondriali e non mitocondriali (Figura 2D). Mentre la stragrande maggioranza delle proteine MitoCarta si raggruppava insieme, ne abbiamo trovate alcune (<5%) che si raggruppavano con proteine non MitoCarta. Queste proteine possono rappresentare (1) proteine citosoliche che interagiscono con i mitocondri (una nuova categoria annotata nella versione 3.0 di MitoCarta), (2) proteine a doppia localizzazione o (3) proteine erroneamente annotate. Al contrario, abbiamo trovato alcuni casi di proteine non-MitoCarta raggruppate con proteine mitocondriali. Mentre tali proteine possono rappresentare contaminanti della procedura di isolamento, possono anche rappresentare proteine non precedentemente classificate come presenti nei mitocondri.
Per studiare questa nuova classe di potenziali proteine mitocondriali, è stata utilizzata la proteomica sottrattiva, un approccio che si è rivelato utile per la scoperta di proteomi organellari, tra cui i mitocondri 6,12. La proteomica sottrattiva presuppone che i mitocondri dovrebbero arricchirsi durante le fasi di purificazione e che i contaminanti dovrebbero esaurirsi6. Ad esempio, mentre i contaminanti possono accumularsi durante la centrifugazione differenziale (ad esempio, a causa di proprietà di sedimentazione simili) o durante la cattura immunitaria (ad esempio, a causa del legame anticorpale non specifico), solo le proteine mitocondriali in buona fede dovrebbero accumularsi significativamente in entrambi. È quindi possibile filtrare le proteine che sono state trovate nella frazione mitocondriale pura ma hanno mostrato modelli incoerenti di arricchimento. Nel presente esempio con le cellule RAW264.7, impostando una soglia per peptidi unici di ≥1 per i campioni mito-grezzi e mito-puri e utilizzando rigorose soglie di arricchimento, siamo stati in grado di perfezionare l'elenco dei proteomi mitocondriali recuperati da 1.127 proteine inizialmente trovate nella frazione mitocondriale grezza dopo centrifugazione differenziale, fino a 481 proteine dopo il secondo ciclo di purificazione utilizzando l'immunoselezione Tomm22. Il numero ridotto di proteine annotate MitoCarta nella frazione mito-pura riflette l'elevata rigore applicata per la selezione. È interessante notare che 70 delle proteine presenti nella frazione mito-pura non erano presenti nell'inventario MitoCarta3.0 (Figura 3A, B). Queste ultime proteine possono rappresentare potenziali nuove proteine candidate mitocondriali, che possono essere espresse solo nella linea cellulare macrofagica RAW264.7 e nelle cellule correlate, e che meritano ulteriori indagini.
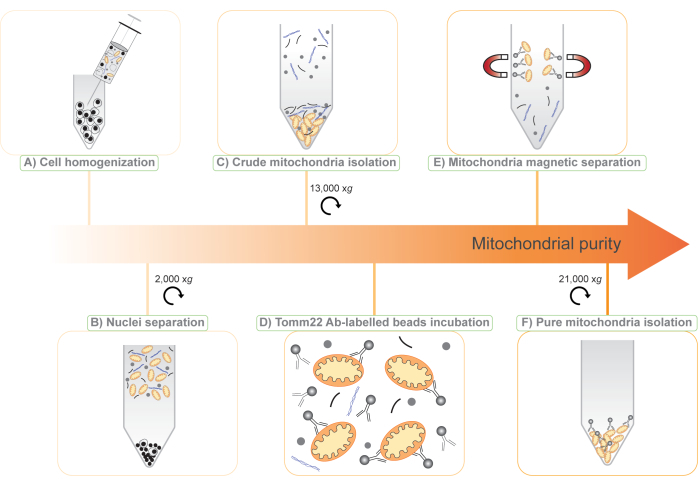
Figura 1: Illustrazione del protocollo di isolamento dei mitocondri in due fasi, senza tag . (A) Una sospensione cellulare viene interrotta attraverso un ago da 25 G. (B) I nuclei e le cellule intere vengono separati mediante centrifugazione a 2.000 x g e il surnatante viene salvato. (C) I mitocondri grezzi sono isolati mediante centrifugazione differenziale del surnatante a 13.000 x g (mito-grezzo). (D) I mitocondri grezzi vengono quindi incubati con anticorpi Tomm22 (Ab) legati covalentemente a perle superparamagnetiche. (E) I complessi mitocondri-Tomm22 anticorpo-perle sono separati dai contaminanti usando colonne magnetiche ed eluiti. (F) I mitocondri puri sono raccolti e concentrati per centrifugazione (mito-pure). Fare clic qui per visualizzare una versione ingrandita di questa figura.
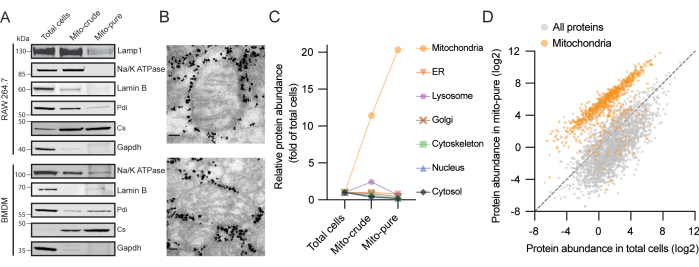
Figura 2. Risultati rappresentativi dell'isolamento dei mitocondri da due fonti di macrofagi. (A) Analisi immunoblot proteica di cellule RAW264.7 (in alto) e BMDM (in basso) utilizzando anticorpi contro la citrato sintasi mitocondriale (Cs - mitocondri), gliceraldeide-3-fosfato deidrogenasi (Gapdh - citosol), pompa sodio-potassio (Na/K ATPasi - membrana plasmatica), Lamin B (Lamin B - nucleo), proteina di membrana lisosomiale associata 1 (Lamp1 - lisosoma) e proteina disolfuro-isomerasi (Pdi - ER). (B) Microscopia elettronica di mitocondri purificati da cellule RAW264.7. Le particelle ad alta densità che circondano i mitocondri corrispondono alle perle Tomm22 che vengono trasportate con campioni mito-puri dopo l'eluizione dalle colonne. Barre scala: 80 nm. (C) Punteggi di arricchimento tra celle totali, mito-grezzo e mito-puro da sette compartimenti cellulari in cellule RAW264.7. MitoCarta3.0 e GO sono stati utilizzati per l'annotazione delle proteine e i punteggi medi sono rappresentati. Abbreviazione: ER = reticolo endoplasmatico. (D) Valori di abbondanza proteica (riBAQ) per proteine in cellule totali e campioni mito-puri da cellule RAW264.7. Le proteine MitoCarta3.0 sono mostrate in arancione. Fare clic qui per visualizzare una versione ingrandita di questa figura.
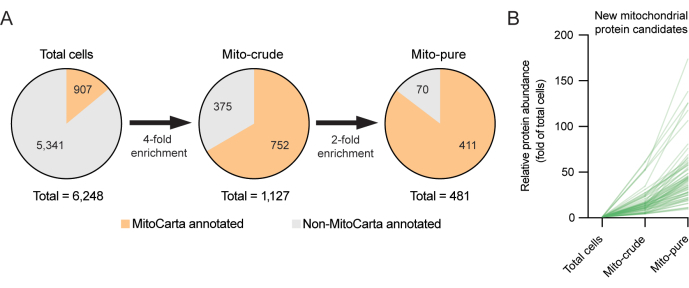
Figura 3. Scoperta di nuove proteine mitocondriali utilizzando la proteomica sottrattiva. (A) Strategia di proteomica sottrattiva per la scoperta di nuove proteine mitocondriali. Vengono applicate soglie di selezione elevate (4x e 2x) per ridurre al minimo la selezione di falsi positivi. (B) Resa di arricchimento (fold of total cells) di nuove proteine candidate mitocondriali non precedentemente annotate nell'inventario MitoCarta3.0. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Discussion
Abbiamo combinato la centrifugazione differenziale e l'immunocattura per ottenere una migliore purezza per l'isolamento dei mitocondri. La nostra procedura consente l'accesso al materiale primario per l'identificazione e la caratterizzazione di nuove proteine mitocondriali. Il protocollo è semplice e robusto e può essere applicato a linee cellulari, cellule primarie e tessuti senza la necessità di modificazioni genetiche. Abbiamo validato il nostro protocollo mediante immunoblotting e analisi proteomiche su campioni prelevati in diverse fasi durante la procedura di purificazione.
Rispetto ai singoli metodi di isolamento, la combinazione di fasi di arricchimento di diversa natura - qui, centrifugazione ed etichettatura immunitaria - genera un protocollo più robusto per isolare i mitocondri. Questo perché, mentre le proteine mitocondriali si arricchiscono in entrambe le purificazioni, è improbabile che anche i contaminanti vengano arricchiti dopo entrambe le fasi di arricchimento. Sebbene un'elevata purezza mitocondriale possa essere raggiunta anche mediante ultracentrifugazione a gradiente di densità, questo approccio richiede una grande quantità di materiale di partenza e l'accesso a un'ultracentrifuga. Infine, a differenza dei recenti metodi basati sull'isolamento mitocondriale basato su tag20, il nostro approccio non richiede la modificazione genetica del campione, rendendolo adatto per materiale primario proveniente da qualsiasi fonte.
Alcune considerazioni tecniche e biologiche devono essere prese in considerazione nel disegno sperimentale quando si applica il nostro protocollo. (1) La quantità di materiale di partenza è fondamentale per ottenere materiale sufficiente. Inevitabilmente, un piccolo numero di mitocondri andrà perso durante l'omogeneizzazione (fase 3.10), poiché non tutte le cellule sono lisate, o durante i lavaggi a tre colonne (fase 4.6). Mentre il nostro protocollo si concentra sulla purezza rispetto alla resa, l'efficienza dell'isolamento dei mitocondri, e quindi la loro resa, non è stata misurata o ottimizzata. L'utilizzo di più perline Tomm22 e più colonne dovrebbe aumentare la resa del recupero dei mitocondri. Allo stesso tempo, un'ottimizzazione completa della fase di omogeneizzazione può anche portare a una migliore resa mitocondriale. Questo protocollo e i numeri di celle iniziali che riportiamo qui per le cellule RAW264.7 e BMDM sono adeguati per la proteomica e possono essere regolati per altre applicazioni. Nel caso dei BMDM primari, abbiamo scoperto che un singolo topo era sufficiente per una replica. Quando necessario, la procedura può essere scalata per isolare i BMDM da più animali, che possono quindi essere raggruppati per ottenere materiale sufficiente. Il numero di cellule può essere ottimizzato in base al tipo di cellula, alle sue dimensioni e al suo contenuto mitocondriale. (2) Tomm22 è espresso sui mitocondri di tutti i tipi di cellule e tessuti21, ma il suo livello di espressione può variare. Pertanto, quando si progetta un esperimento per confrontare condizioni diverse, è importante assicurarsi che i livelli di espressione di Tomm22 siano comparabili. Inoltre, a causa dell'espressione ubiquitaria di Tomm22, non è possibile studiare proteine mitocondriali specifiche del tipo cellulare all'interno di tessuti complessi. (3) Il tempo necessario per generare mitocondri puri (circa 2,5 ore) è incompatibile con uno studio di eventi transitori, come cambiamenti nei profili metabolici. In questo caso, raccomandiamo la cattura immunitaria diretta basata su tag9, che consente anche di studiare i mitocondri specifici del tipo di cellula in vivo20. (4) Sebbene studi su mitocondri isolati ottenuti utilizzando solo perle marcate con anticorpi Tomm22 abbiano mostrato attività nei saggi funzionali11, resta da determinare se i mitocondri generati con il nostro protocollo sono compatibili con i saggi basati sull'attività a valle. MitoTracker o la colorazione con perclorato di estere metile (TMRM) o tetrametilrodamina, o misurazioni respirometriche, sono potenziali approcci per quantificare la funzionalità dei mitocondri isolati22. (5) Dopo aver estratto il campione "mito-puro" dalla colonna, alcune perle di Tomm22 saranno presenti nella frazione mitocondriale pura (Figura 2B). Mentre non abbiamo osservato alcuna interferenza con la digestione della tripsina e la spettrometria di massa proteica, la presenza di queste perle e delle immunoglobuline dovrebbe essere presa in considerazione in altre applicazioni a valle. L'anticorpo Tomm22 è un anticorpo monoclonale prodotto nei topi23, e quindi è importante tenere presente che, quando si utilizzano anticorpi secondari contro i topi in immunoblotting, genererà bande non specifiche alle dimensioni delle catene di immunoglobuline. (6) La completa omogeneizzazione della sospensione cellulare è la chiave per il successo dell'isolamento dei mitocondri. Qui, usiamo una siringa con un ago da 25 G per lisare sia le cellule RAW264.7 che i BMDM. Tuttavia, a seconda del tipo di cellula e delle sue dimensioni, altri metodi di omogeneizzazione meccanica, come l'uso di un omogeneizzatore Dounce, o approcci più controllati come i dispositivi di omogeneizzazione cellulare, possono essere più adatti. Possono essere presi in considerazione anche metodi di omogeneizzazione non meccanici, come la sonicazione delicata. Gli approcci di omogeneizzazione dei tessuti sono ulteriormente discussi in altri studi24,25. (7) Sebbene la convalida mediante immunoblotting sia il metodo più semplice ed economico, i suoi risultati potrebbero non essere sempre correlati con i cambiamenti a livello dell'intero organello. Ecco perché raccomandiamo di utilizzare la proteomica per convalidare pienamente l'arricchimento o l'esaurimento dei mitocondri e di altri organelli, rispettivamente.
Il protocollo di purificazione dei mitocondri in due fasi qui descritto ci ha permesso di generare campioni sequenziali con purezza mitocondriale crescente, e questo ci ha permesso di scoprire nuovi candidati proteici mitocondriali attraverso la proteomica sottrattiva12. Per la nostra analisi, utilizziamo soglie rigorose per selezionare proteine mitocondriali significativamente arricchite e, sebbene ciò possa non riuscire a identificare alcune proteine mitocondriali note (Figura 3A), il tasso di falsi positivi per la scoperta di nuove proteine mitocondriali è diminuito. Tuttavia, è importante sottolineare che qualsiasi proteina candidata rivelata dal nostro protocollo deve essere validata attraverso approcci ortogonali. Si raccomanda la marcatura carbossi-terminale della GFP o l'uso di anticorpi contro la proteina endogena per convalidare l'associazione con i mitocondri microscopicamente o mediante saggi di protezione della proteasi.
L'applicazione diretta del nostro metodo nel caso di cellule e tessuti non modificati offre un potente strumento per studiare come i mitocondri cambiano e si adattano al loro ambiente in condizioni di salute e malattia. L'applicazione del nostro protocollo a linee cellulari, modelli di malattie animali, fluidi umani e persino biopsie chirurgiche può rivelarsi particolarmente utile per migliorare la nostra comprensione dei mitocondri e dei loro disturbi associati.
Disclosures
Gli autori non hanno nulla da rivelare.
Acknowledgements
Ringraziamo Manfredo Quadroni, la Protein Analysis Facility e la Electron Microscopy Facility dell'Università di Losanna per il loro aiuto. Ringraziamo anche H.G. Sprenger, K. Maundrell e i membri del laboratorio Jourdain per i consigli e il feedback sul manoscritto. Questo lavoro è stato sostenuto dalla Fondazione Pierre-Mercier pour la Science e dal Fondo nazionale svizzero per la ricerca scientifica (sovvenzione 310030_200796).
Materials
| Name | Company | Catalog Number | Comments |
| 1 mL syringe | BD Plastipal | 309628 | |
| 25 G Needle | BD Microlance | 300400 | |
| 40 µm cell strainer | Corning | 352340 | |
| Anti-TOM22 Microbeads, mouse | Miltenyi Biotec | 130-127-693 | |
| Cell scraper | FisherScientific | 11577692 | |
| DMEM, high glucose, GlutaMAX | ThermoFisher | 31966 | |
| Ethylenediaminetetraacetic acid | FisherScientific | BP-120-1 | |
| Fetal bovine serum | Gibco | 10270 | |
| HEPES | BioConcept | 5-31F00-H | |
| LS columns and plungers | Miltenyi Biotec | 130-042-401 | |
| Macrophage colony-stimulating factor | Immunotools | 12343115 | |
| Mannitol | Sigma | M4125 | |
| Sodium chloride | Sigma | 71380 | |
| Sucrose | Sigma | S1888 | |
| Penicillin/Streptomycin | BioConcept | 4-01F00-H | |
| Petri dishes | Corning | BH93B-102 | |
| Phosphate-buffered saline 10X | Eurobio Scientific | CS3PBS01-01 | |
| QuadroMACS Separator | Miltenyi Biotec | 130-090-976 | |
| Vi-CELL BLU Cell Viability Analyzer | Beckman Coulter | C19196 |
References
- Spinelli, J. B., Haigis, M. C. The multifaceted contributions of mitochondria to cellular metabolism. Nature Cell Biology. 20 (7), 745-754 (2018).
- West, A. P., Shadel, G. S., Ghosh, S. Mitochondria in innate immune responses. Nature Reviews Immunology. 11 (6), 389-402 (2011).
- Chakrabarty, R. P., Chandel, N. S. Mitochondria as signaling organelles control mammalian stem cell fate. Cell Stem Cell. 28 (3), 394-408 (2021).
- Morgenstern, M., et al. Quantitative high-confidence human mitochondrial proteome and its dynamics in cellular context. Cell Metabolism. 33 (12), 2464-2483 (2021).
- Rath, S., et al. MitoCarta3.0: an updated mitochondrial proteome now with sub-organelle localization and pathway annotations. Nucleic Acids Research. 49, D1541-D1547 (2021).
- Pagliarini, D. J., et al. A mitochondrial protein compendium elucidates complex I disease biology. Cell. 134 (1), 112-123 (2008).
- Diaz-Vegas, A., et al. Is mitochondrial dysfunction a common root of noncommunicable chronic diseases. Endocrine Reviews. 41 (3), 005 (2020).
- Bury, A. G., Vincent, A. E., Turnbull, D. M., Actis, P., Hudson, G. Mitochondrial isolation: when size matters. Wellcome Open Research. 5, 226 (2020).
- Chen, W. W., Freinkman, E., Sabatini, D. M. Rapid immunopurification of mitochondria for metabolite profiling and absolute quantification of matrix metabolites. Nature Protocols. 12 (10), 2215-2231 (2017).
- Daniele, J. R., Heydari, K., Arriaga, E. A., Dillin, A. Identification and characterization of mitochondrial subtypes in Caenorhabditis elegans via analysis of individual mitochondria by flow cytometry. Analytical Chemistry. 88 (12), 6309-6316 (2016).
- Franko, A., et al. Efficient isolation of pure and functional mitochondria from mouse tissues using automated tissue disruption and enrichment with anti-TOM22 magnetic beads. PLoS One. 8 (12), e82392 (2013).
- Yates, J. R., Gilchrist, A., Howell, K. E., Bergeron, J. J. M. Proteomics of organelles and large cellular structures. Nature Reviews Molecular Cell Biology. 6 (9), 702-714 (2005).
- Trouplin, V., et al. marrow-derived macrophage production. Journal of Visualized Experiments. (81), e50966 (2013).
- Ying, W., Cheruku, P. S., Bazer, F. W., Safe, S. H., Zhou, B. Investigation of macrophage polarization using bone marrow derived macrophages. Journal of Visualized Experiments. (76), e50323 (2013).
- Toda, G., Yamauchi, T., Kadowaki, T., Ueki, K. Preparation and culture of bone marrow-derived macrophages from mice for functional analysis. STAR Protocols. 2 (1), 100246 (2020).
- Gene Ontology Consortium. The Gene Ontology resource: enriching a GOld mine. Nucleic Acids Research. 49, D325-D334 (2021).
- Ashburner, M., et al. Gene ontology: tool for the unification of biology. The Gene Ontology Consortium. Nature Genetics. 25 (1), 25-29 (2000).
- Hartwig, S., et al. A critical comparison between two classical and a kit-based method for mitochondria isolation. Proteomics. 9 (11), 3209-3214 (2009).
- Delamarre, L., Pack, M., Chang, H., Mellman, I., Trombetta, E. S. Differential lysosomal proteolysis in antigen-presenting cells determines antigen fate. Science. 307 (5715), 1630-1634 (2005).
- Bayraktar, E. C., et al. MITO-Tag mice enable rapid isolation and multimodal profiling of mitochondria from specific cell types in vivo. Proceedings of the National Academy of Sciences. 116 (1), 303-312 (2019).
- Nusinow, D. P., et al. Quantitative proteomics of the cancer cell line encyclopedia. Cell. 180 (2), 387-402 (2020).
- Iuso, A., Repp, B., Biagosch, C., Terrile, C., Prokisch, H. Assessing mitochondrial bioenergetics in isolated mitochondria from various mouse tissues using seahorse XF96 analyzer. Methods in Molecular Biology. 1567, 217-230 (2017).
- Hornig-Do, H. T., et al. Isolation of functional pure mitochondria by superparamagnetic microbeads. Analytical Biochemistry. 389 (1), 1-5 (2009).
- Liao, P. C., Bergamini, C., Fato, R., Pon, L. A., Pallotti, F. Isolation of mitochondria from cells and tissues. Methods in Cell Biology. 155, 3-31 (2020).
- Lanza, I. R., Nair, K. S. Functional assessment of isolated mitochondria in vitro. Methods in Enzymology. 457, 349-372 (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved