ניתוח תורשה אפיגנטית בין-דורית ב- C. elegans באמצעות כתב פלואורסצנטי ומשקעים חיסוניים של כרומטין (ChIP)
* These authors contributed equally
In This Article
Summary
פרוטוקול זה מתאר התאבכות RNA ובדיקת ChIP כדי לחקור את התורשה האפיגנטית של השתקה הנגרמת על ידי RNAi ושינויים כרומטינים הקשורים ב- C. elegans.
Abstract
תורשה אפיגנטית בין-דורית (TEI) מאפשרת העברת מידע דרך קו הנבט מבלי לשנות את רצף הגנום, באמצעות גורמים כגון RNA לא מקודד ושינויים בכרומטין. תופעת תורשה של הפרעות RNA (RNAi) בנמטודה Caenorhabditis elegans היא מודל יעיל לחקר TEI המנצל את מחזור החיים הקצר של אורגניזם מודל זה, התפשטות עצמית ושקיפות. בתורשה של RNAi, חשיפה של בעלי חיים ל-RNAi מובילה להשתקת גנים ולשינויים בחתימות הכרומטין במיקום המטרה שנמשכים מספר דורות בהיעדר הטריגר הראשוני. פרוטוקול זה מתאר את ניתוח תורשה RNAi ב- C. elegans באמצעות כתב חלבון פלואורסצנטי ירוק גרעיני (GFP) המבוטא על ידי נבט. השתקת הכתבים מתבצעת על ידי האכלת בעלי החיים בחיידקים המבטאים RNA דו-גדילי המכוון ל-GFP. בכל דור עוברים בעלי חיים כדי לשמור על התפתחות מסונכרנת, והשתקת גנים נקבעת במיקרוסקופיה. בדורות נבחרים, אוכלוסיות נאספות ומעובדות עבור תגובת שרשרת פולימראז כמותית של כרומטין (ChIP) (qPCR) כדי למדוד את העשרת שינוי ההיסטון במוקד כתב GFP. פרוטוקול זה לחקר תורשה RNAi ניתן לשינוי בקלות ומשולב עם ניתוחים אחרים כדי להמשיך לחקור גורמי TEI במסלולי RNA וכרומטין קטנים.
Introduction
תורשה אפיגנטית מאפשרת העברת מידע על בקרת גנים בין דורות ולכן יכולה לאפשר לסביבה או לחוויות של ההורים להשפיע על צאצאיהם. ב- C. elegans, השתקת גנים של קו הנבט המופעלת על ידי RNA דו-גדילי אקסוגני (dsRNA) יכולה לעבור בתורשה במשך דורות רבים בצאצאים שלא נחשפו לטריגר המקורי 1,2,3,4. תהליך זה, המכונה RNA interference (RNAi) inheritance, הוא אחת ממספר תופעות השתקה אפיגנטיות קשורות ב- C. elegans, כולל השתקה רב-דורית ביוזמת piRNA 2,5, paramutation/RNAe (השתקה אפיגנטית הנגרמת על ידי RNA) 6,7,8, והשתקה ביוזמת מערך multicopy9, שיש להם דרישות חופפות אך נפרדות למכונות ויסות RNA וכרומטין קטנות . ב-C. elegans exogenous RNAi, dsRNA מעובד ל-RNA מפריע קטן (siRNAs) הפועל בקומפלקס עם חלבוני ארגונאוט ראשוניים כדי לזהות את mRNA המטרה שלהם. מיקוד זה מוביל להגברה של siRNA משני המשויך לארגונאוטים משניים כדי להשתיק mRNA מטרה דרך מסלולי השתקה ציטופלזמיים וגרעיניים כאחד. עבור מטרות RNAi המבוטאות בתאי נבט, המשנה הגרעינית Argonaute HRDE-1 וגורמי RNAi גרעיניים נוספים מכוונים לתעתיקים מתהווים, וכתוצאה מכך דיכוי שעתוק וגיוס של מתילטרנספראזות היסטון להפקדת סימני כרומטין מדכאים, כולל H3K9me310. Histone H3K9me3 מקדם הקמת השתקה תורשתית של טרנסגנים של חלבון פלואורסצנטי ירוק (GFP) המתבטא בנבט על ידי ירושת RNAi11,12.
מטרת פרוטוקול זה היא להשתמש בטרנסגן המבטא חלבון היתוך GFP-היסטון בקו הנבט ככתב לתורשה RNAi ולהעריך שינויים בשינויים בהיסטון במוקד הטרנסגן של הכתב באמצעות משקעים חיסוניים של כרומטין ותגובת שרשרת פולימראז כמותית (ChIP-qPCR). פרוטוקול זה מתאר גישת הזנת RNAi סטנדרטית מבוססת צלחת ליזום השתקת כתבים. הוא גם מספק ציר זמן מפורט להעברת בעלי חיים בין דורות על ידי בידוד עוברי רחם ממבוגרים גרבידיים על ידי טיפול בהיפוכלוריט אלקליין ('הלבנה'). כמו כן מתוארות שיטות ונתונים מייצגים לניטור תדירות השתקת GFP בתת-קבוצה של האוכלוסייה באמצעות מיקרוסקופ פלואורסצנטי ועבור היסטון H3K9me3 ChIP-qPCR. מבחני תורשה מבוססי כתב RNAi מספקים מערכת יעילה ביותר לנתח באופן פונקציונלי את תפקידם של גורמים גנטיים וסביבתיים בוויסות אפיגנטי 13,14, ובדיקות גנטיות באמצעות כתבים כאלה זיהו הן גנים הדרושים עבור 2,3,15 והן גנים המווסתים באופן שלילי 16,17 את משך התורשה האפיגנטית הבין-דורית.
Protocol
הערה: ציר זמן של בדיקה מוצג באיור 1.
1. הכנת לוחות RNAi nematode growth medium (RNAi-NGM)
- הוציאו החוצה את E. coli HT115(DE3) המכיל GFP או וקטורי RNAi בקרה ממלאי גליצרול על לוחות אגר Luria-Bertani (LB) בתוספת 100 מיקרוגרם / מ"ל אמפיצילין. לדגור על החיידקים לילה ב 37 ° C.
- למחרת, הכינו צלחות RNAi-NGM (אגר 1.7% [w/v], 0.3% [w/v] NaCl, 0.25% [w/v] פפטון, 1 mM CaCl2, 5 מיקרוגרם / מ"ל כולסטרול, 25 mM אשלגן פוספט חיץ [pH 6.0], 1 mM MgSO4, 25 מיקרוגרם / מ"ל קרבניצלין, ו 5 mM איזופרופיל β-d-1-thiogalactopyranoside [IPTG]) על פי פרוטוקול סטנדרטי18.
- עבור כל זן בניסוי, הכינו לפחות שש לוחיות RNAi-NGM שנזרעו עם GFP RNAi (שתי לוחיות לכל אחד משלושת העותקים הביולוגיים) ושתי לוחיות RNAi-NGM שנזרעו עם RNAi בקרה (שכפול ביולוגי אחד).
- אוהל רופף את הצלחות עם נייר כסף כדי להגן מפני אור ולהשאיר לילה בטמפרטורת החדר לייבוש.
- באותו יום כמו צלחת RNAi-NGM לשפוך, להכין 4 מ"ל של תרביות נוזליות של חיידקי RNAi.
- בתנאים סטריליים, aliquot 4 מ"ל של מרק LB בתוספת 10 מיקרוגרם / מ"ל טטרציקלין ו 100 מיקרוגרם / מ"ל אמפיצילין לתוך צינורות תרבית. בחרו מושבות בודדות מצלחות האגר LB לכל צינור ודגרו במשך הלילה תוך כדי ניעור במשך כ-16 שעות בטמפרטורה של 37°C.
- זרעו חיידקי RNAi על לוחות RNAi-NGM.
- לאחר גדילת לילה, מדדו את הצפיפות האופטית (OD600) של תרביות באמצעות דילול של 1:5 חיידקים עם ציר LB.
- לדלל את חיידקי ה-RNAi ל-ODיחסית של 600 מתוך 2 באמצעות ציר LB בתוספת טטרציקלין של 10 מיקרוגרם/מ"ל ו-100 מיקרוגרם/מ"ל אמפיצילין.
- בתנאים סטריליים, יש להוסיף 150 μL של חיידקי ה-RNAi של הבקרה או ה-GFP לכל צלחת RNAi-NGM.
- אוהל את הצלחות עם נייר כסף ולאפשר לחיידקים לגדול לפחות 24 שעות בטמפרטורת החדר לפני השימוש.
הערה: ניתן לאחסן לוחות RNAi-NGM שאינם בשימוש הפוכים למשך עד שבוע אחד ב-4°C במיכל אטום בחושך.
2. התחלת בדיקת תורשה RNAi: הלבנה וציפוי עוברים לדור P0
הערה: לפני תחילת בדיקת הירושה RNAi, תולעים המכילות את כתב GFP mjIs134 [mex-5p::gfp-h2b::tbb-2 3'UTR]7 צריכות להישמר ללא רעב ב 21 ° C במשך שני דורות לפחות.
- 4 ימים (96 שעות) לפני תחילת בדיקת התורשה של RNAi (איור 1), בחרו שלושה או ארבעה בוגרים גרבידים על כל צלחת NGM סטנדרטית בקוטר 35 מ"מ עם זרעי חיידקי OP50-1.
הערה: שלוש צלחות NGM לכל זן מספיקות. עבור זנים עם פוריות נפגעת, ייתכן שיהיה צורך ביותר מארבעה בעלי חיים בכל צלחת. - לאחר 4 ימים, ודא כי האוכלוסייה הבוגרת החלה להטיל עוברים על הצלחת. שטפו את התולעים מהלוחות לתוך צינורות של 1.5 מ"ל באמצעות 800 μL של חיץ M9 בתוספת Triton X-100 (TX-100) (22 mM KH 2 PO 4, 42 mM Na2HPO 4, 86 mM NaCl, 1mMMgSO 4, 0.01% (v/v) TX-100). יש לחזור על השטיפה והבריכה לאותו צינור.
- בודדו את העוברים מהאוכלוסייה על ידי הלבנה באופן הבא.
- הכינו תמיסת הלבנה עם אקונומיקה (6% נתרן היפוכלוריט) ו-10 N NaOH ביחס נפח של 1.5:1. הכינו כמות מספקת כך שניתן יהיה להשתמש ב-250 מיקרוליטר לכל דגימה.
- צנטריפוגה את צינורות 1.5 מ"ל עם התולעים ב 1,000 x גרם במשך 1.5-2 דקות.
- שאפו את הסופרנטנט, והשאירו 100 מיקרוליטר מבלי להפריע ל'כדור' התולעת. לחלופין, ניתן להשאיר את הצינורות למשך כ-2 דקות כדי לאפשר לתולעים לשקוע לפני השאיפה.
- הוסף 650 μL של ddH2O לכל צינור כדי להשיג נפח סופי של 750 μL.
הערה: השלבים הבאים הם תלויי זמן. - הוסיפו 250 μL של תמיסת ההלבנה לכל צינור והפעילו טיימר.
- כל 1-2 דקות, מערבלים היטב את הצינורות. לאחר 5 דקות, לבדוק את התולעים מתחת סטריאוסקופ כדי לפקח על השפלה של מבוגרים.
- ממשיכים לערבל עד שהתולעים מומסות לחלוטין ונשארים רק עוברים. צנטריפוגו מיד את הצינורות במהירות של 1,000 x גרם למשך דקה וחצי.
הערה: ההלבנה בדרך כלל מושלמת תוך 6-7 דקות, אך יש לעקוב אחריה תחת סטריאוסקופ בהגדלה מספקת כדי לצפות בעוברים המשוחררים (פי 40). אל תשאירו את התולעים באקונומיקה לתקופה ממושכת, מכיוון שהדבר יפגע בעוברים. - לאחר הצנטריפוגה, שאפו את הסופרנאטנט והשאירו בערך 50-100 μL. הוסיפו 1 מ"ל של חיץ M9 עם TX-100 כדי לשטוף ולערבב על ידי מערבולת. צנטריפוגה שוב ב 1,000 x גרם במשך 1.5-2 דקות ולשטוף לפחות עוד פעמיים.
- שאפו את הסופרנאטנט מהשטיפה הסופית והשאירו כ-100 מיקרוליטר בצינור. מערבבים על ידי מערבולת. פיפטה 2 μL מכל צינור פעמיים על שקופית זכוכית מסומנת. ספרו את העוברים באמצעות מונה ספירה כדי להעריך את ריכוז העוברים למיקרוליטר.
הערה: ניתן להשתמש כאן בשקופיות עם בארות חלביות מרובות כדי לספור העתקים של זנים מרובים. ניתן לשטוף שקופיות ספירה ולעשות בהן שימוש חוזר.
- כדי להתחיל גידול אוכלוסייה מסונכרן למחצה, ערבבו ופיפטה נפח המכיל 250 עוברים על כל צלחת RNAi-NGM. השתמש בשני לוחות עבור כל שכפול. הניחו לנוזל להיספג.
- עבור דור P0 שטופל ב-RNAi, יש לדגור בטמפרטורה של 21°C למשך 4 ימים (96 שעות) (מהעוברים ועד לבגרות). הזמנים עשויים להשתנות בהתאם לגנוטיפ.
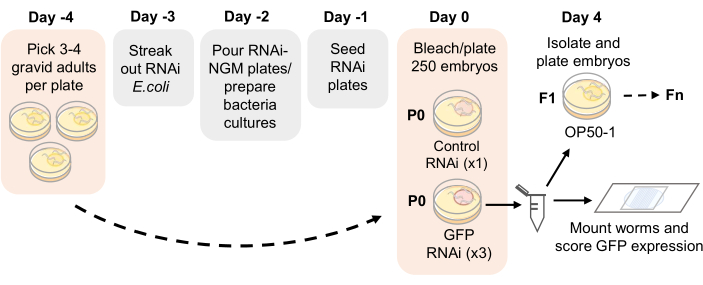
איור 1: סכמת בדיקת תורשה של RNAi. ציר זמן מוצע להכנת לוחות RNAi-NGM והגדרת בדיקת תורשה RNAi. בוגרים Gravid נקטפים ביום -4 על צלחות NGM שנזרעו עם OP50-1. לאחר 4 ימים, צאצאים בוגרים מולבנים והעוברים מצופים על לוחות RNAi-NGM. דור P0 נחשף ל-RNAi במשך 4 ימים בטמפרטורה של 21°C. ברגע שהתולעים מגיעות לבגרות, ההעתקים עוברים על ידי הלבנה ומקבלים ניקוד לביטוי GFP של קו הנבט בכל דור. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
3. מעבר וניקוד בכל דור עבור ביטוי GFP של קו הנבט
הערה: כדי להקל על הניקוד, השתמש בתקליט ויניל כדי ליצור רפידות אגרוז עם רכסים ליניאריים כדי ליישר את התולעים. שיטה זו אומצה על ידי ריברה גומז ושוורצשטיין19.
- מכינים 1% (w/v) אגרוז על ידי המסת אגרוז ב-ddH2O בבקבוק קטן על ידי חימום במיקרוגל. מוסיפים ערבוב ומכסים בנייר כסף. אם נמס מחדש, מחממים את האגרוז על פלטה בחום של 200 מעלות צלזיוס תוך כדי ערבוב. לאחר המסה, להפחית את החום ל 80 ° C.
- שטפו את התולעים מלוחות ה-NGM באמצעות 800 מיקרוליטר של חיץ M9 עם TX-100 לתוך צינור של 1.5 מ"ל. לשטוף את הצלחות פעמיים כדי להסיר את כל המבוגרים gravid. בדוק את הצלחות מתחת לסטריאוסקופ כדי לוודא שבעלי החיים נאספו ביעילות.
- צנטריפוגו את הצינורות במהירות של 1,000 x גרם למשך 1.5-2 דקות או אפשרו לתולעים להסתפק במשך 2 דקות. שאפו את הסופרנאטנט והשאירו 100 מיקרוליטר מבלי להפריע ל'כדור' התולעת.
- הכינו רפידות אגרוז להרכבת התולעים כדלקמן.
- מניחים טיפה של 1% מומס (w/v) agarose על תקליט ויניל (שתואר קודם19) באמצעות פיפטה פסטר זכוכית (עם קצה צר שבור).
- כוון שקופית זכוכית כך שהקווים ברשומה יהיו אופקיים או אנכיים והנח את השקופית במהירות על טיפת אגרוז. המתן כ- 30 שניות לפני הסרת שקופית הזכוכית מהרשומה.
הערה: אם אתה מבקיע גנוטיפים מרובים, ייתכן שיהיה שימושי ליצור כרית אגרוז גדולה יותר בשקופית אחת ולחתוך אותה לשניים באמצעות להב.
- הרכיבו את התולעים על כריות האגרוז.
- תחת הסטריאוסקופ, להוסיף כ 5 μL של 5 mM levamisole כרית agarose. דילול הלבמיסול צריך להיעשות טרי בכל שבוע.
- מעבירים בעדינות את צינורות התולעים כדי לערבב ולהעביר 5-10 מיקרוליטר תולעים על כרית האגרוז. העריכו את מספר התולעים בעין בעת הוספתן, כך שיהיו כ-40 תולעים בכל שכפול.
- סדרו את התולעים בשורות באמצעות פיק ריסים לניקוד קל יותר. הוסיפו כיסוי זכוכית.
הערה: מגלשות עם בעלי חיים המותקנים בדרך זו יכולות להימשך מספר שעות לפני שהן מתייבשות.
- בודדו את העוברים מבעלי החיים הנותרים באמצעות פרוטוקול הלבנה (ראו שלב 2.3) לפני הניקוד של השקפים. צלחת 250 עוברים על לוחות NGM 35 מ"מ שנזרעו עם OP50-120. לאחר שהנוזל נספג, הפכו את הצלחות והחזירו אותן לאינקובטור של 21 מעלות צלזיוס.
- השתמש בסטריאוסקופ תפרחת עם ערכת מסננים GFP. ספור ורשום את מספר התולעים החיוביות של GFP ו- GFP שליליות בשקופיות עבור כל שכפול באמצעות מונה ספירה.
הערה: לתולעים חיוביות של GFP יהיה ביטוי גרעיני של GFP בנבט שלהן ובעוברים ברחם. עם זאת, הנבט של תולעים שליליות של GFP לא יפלוט, עם זאת, כאשר ה-GFP יתחיל לפעול מחדש בדורות מאוחרים יותר, הפלואורסצנטיות עשויה להיות עמומה. זה עשוי להיות מועיל להרכיב זני תולעים נוספים שאינם מהונדסים כדי להסביר כל autofluorescence שנצפה. - מעבר האוכלוסייה על ידי הלבנה, כמתואר לעיל, בערך כל 4 ימים (96 שעות) ב 21 °C (75 °F). לחלופין, ניתן לבצע את המעבר על פי לוח זמנים לסירוגין של 3 ימים / 4 ימים לנוחותכם. צלחת תוספת ~ 50 עוברים עבור הדורות החולפים הקצרים יותר אם לסירוגין, שכן ייתכן שיש יבול נמוך יותר של עוברים לאחר 72 שעות.
הערה: שמור על זמן מעבר עקבי של דור P0 לאחר 4 ימים לאחר ציפוי.
4. אוסף בעלי חיים עבור ChIP
הערה: מספר בעלי החיים והעיתוי תלויים בזן, בשלב ההתפתחותי, באפיטופ ובמספר מטרות המשקעים החיסוניים (IP). בדוגמה שלהלן, אספו בעלי חיים עבור שלושה כתובות IP: H3K9me3, היסטון H3 והדברת IgG. פרוטוקול ChIP הותאם מ-Askjaer et al.21.
- לפני איסוף דגימות ChIP, הרחיבו את הדור הקודם של בדיקת התורשה של RNAi בשלוש לוחיות NGM נוספות (לארבע לוחיות לפחות לכל שכפול). לגדול במשך 4 ימים ב 21 °C (75 °F).
- הלבינו את הבוגרים הגרבידיים כדי לבודד את העוברים (ראו שלב 2.3).
- עבור כל זן, צלחת כ -3,500 עוברים על 14 צלחות (250 לכל צלחת) ולגדול במשך 3 ימים ב 21 ° C לשלב הבוגר הצעיר.
- שטפו את בעלי החיים לתוך צינורות של 1.5 מ"ל עם מלח חוצץ פוספט (PBS) המכיל 0.01% (v/v) TX-100 (PBS/TX). צנטריפוגה ברזולוציה של 1,000 x גרם למשך 2 דקות. יש לשאוף ולאגד את בעלי החיים מזן אחד לתוך צינור אחד ולשטוף שלוש פעמים נוספות עם לפחות 1 מ"ל של PBS/TX.
- לשאוף ל-1,000 μL, להפוך כדי לערבב, ולספור את מספר בעלי החיים ב-3 μL שלוש פעמים (עיין בשלב 2.3.9).
- מחשבים את ריכוז בעלי החיים ומעבירים נפח המתאים ל-3,000 בעלי חיים לצינור חדש להצלבת פורמלדהיד.
- ודא שהנפח הכולל הוא לפחות פי 10 מנפח גלולת התולעת.
5. קרוסלינקינג פורמלדהיד
אזהרה: יש לעבוד עם פורמלדהיד במכסה אדים כדי למנוע חשיפה לאדים.
- מוסיפים פורמלדהיד (ריכוז סופי של 1.8%) לצינור המכיל את התולעים. סובבו בטמפרטורת החדר במשך 6 דקות.
- מיד להקפיא בחנקן נוזלי. ניתן להשהות את הניסוי בשלב זה וניתן לאחסן את הדגימות בטמפרטורה של -80 מעלות צלזיוס.
- הפשירו את הדגימה הצולבת באמבט מים בטמפרטורת החדר למשך 3 דקות וסובבו במשך 16 דקות בטמפרטורת החדר.
- הוסף גליצין 1.25 M לריכוז סופי של 125 mM וסובב במשך 5 דקות.
הערה: עד להדבקת חרוזים מגנטיים, יש לשמור את הדגימות על קרח או בטמפרטורה של 4°C ולהשתמש במאגרים קרים כקרח. - צנטריפוגה את הדגימה ב 1,000 x גרם במשך 3 דקות. יש לשטוף שלוש פעמים עם 1 מ"ל PBS/TX בכל פעם. יש לשטוף פעמיים עם 1 מ"ל של חיץ השעיה (150 mM NaCl, 50 mM HEPES-KOH [pH 7.5], 1 mM ethylenediaminetetraacetic acid [EDTA], 0.01% TX-100, מעכב פרוטאז [טבליה אחת לכל 5 מ"ל]).
- לאחר השטיפה האחרונה, השאירו מספיק חיץ כדי להבטיח שהנפח הכולל יהיה לפחות פי שלושה מנפח הכדוריות ומינימום של ~100 מיקרוליטר.
6. סוניקציה
הערה: פרמטרי הסוניקציה תלויים בסוג ובדגם של הסוניקטור ובמת בעלי החיים. פרמטרים כגון נפח וריכוז הדגימה, מרווחי הפעלה/כיבוי, מספר מחזורים והגדרת צריכת חשמל צריכים להיות ממוטבים אמפירית. לדוגמה, במהלך זמן של סוניקציה, עקוב אחר ליזה תולעת באמצעות בדיקת חלבון וקבע מתי הריכוז מגיע לרמה. בנוסף, יש לעקוב מתי גודל הגזירה הממוצע של הדנ"א הגנומי הוא בערך 200-1,000 bp על ידי אלקטרופורזה של DNA, מטוהר לאחר היפוך crosslinking, על ג'ל agarose/tris-acetate-EDTA (TAE) 1.5%.
- מדוד את נפח הדגימות מהשלב הקודם. מערבבים ומוסיפים 90-120 μL לצינור סוניקציה מפוליסטירן.
- הוסף נפח שווה של חיץ מתלה המכיל 2x דטרגנטים (150 mM NaCl, 50 mM HEPES-KOH [pH 7.5], 1 mM EDTA, 0.2% נתרן deoxycholate, 0.7% sarkosyl).
- סוניק באמבט מים בעוצמה של 50% למשך 7 דקות (20 שניות מופעלות / 40 שניות כבויות) ב- 4 ° C. מערבבים בעדינות על ידי פיפט. חזור על הסוניקציה במשך 7 דקות נוספות.
- מעבירים את הליזט הסוניק לצינור של 1.5 מ"ל. הוסף 0.5 נפחים של חיץ מתלה ללא דטרגנטים (150 mM NaCl, 50 mM HEPES-KOH [pH 7.5], 1 mM EDTA) (למשל, הוסף 100 μL של חיץ ל 200 μL של דגימה).
- צנטריפוגה ב-13,000 x גרם למשך 15 דקות ב-4°C. שומרים את הליזט סופרנאטנט ומעבירים לצינור חדש.
- לחלופין, בצע בדיקת חלבון כדי לקבוע את הריכוזים של כל ליזט. שלב זה עשוי להיות שימושי עבור כמויות מדגם גדולות או השוואות בין מבחנים. עם זאת, סטנדרטיזציה של מספר בעלי החיים כפי שתואר לעיל עובדת היטב עבור כמויות הדגימה המתוארות כאן, שכן יש מתאם טוב יותר עם התשואה של כרומטין/DNA כפי שנקבע על ידי qPCR.
7. משקעים חיסוניים
הערה: שנה את כמות הנוגדנים והחרוזים המגנטיים לנפח ולריכוז של ליזט.
- חלק את סופרנאטנט הליזט לארבעה חלקים: שלושה נפחים שווים לכל IP ו-10% מנפח IP אחד כקלט (למשל, 100 μL של IP #1, 100 μL של IP #2, 100 μL של IP #3, 10 μL של קלט). העבר את ליזה IP לצינור PCR של 200 μL ואחסן את ליזה הקלט ב -20 °C בצינור של 1.5 מ"ל.
- הוסף 0.5 מיקרוגרם של נוגדן נגד H3K9me3, נוגדן H3 נגד היסטון או IgG לדגימת ה- IP המתאימה. יש לדגור ב-4°C (75 °F) למשך הלילה עם סיבוב.
- למחרת, aliquot 9 μL של חלבון G מצופה חרוזים מגנטיים לכל IP לצינור יחיד 1.5 מ"ל. שטפו את החרוזים פעמיים עם 1 מ"ל FA-150 (150 mM NaCl, 50 mM HEPES-KOH [pH 7.5], 1 mM EDTA, 1% TX-100, 0.1% נתרן deoxycholate).
- השהה מחדש את החרוזים המגנטיים במאגר FA-150 לנפח המקורי שנלקח מהמלאי בשלב 7.3 לעיל. הוסף 7.5 μL לכל כתובת IP. יש לדגור ב-4°C למשך שעתיים עם סיבוב.
8. שטיפות ו elution
הערה: כדי להבטיח שהחרוזים המגנטיים לא יתייבשו, הוסיפו במהירות כל חיץ כביסה או חיץ אלוציה לאחר השאיפה של הכביסה הקודמת.
- השתמש 0.2-1 מ"ל של פתרונות החיץ הבאים בכל פעם כדי לשטוף את החרוזים. אספו את החרוזים על מעמד מגנטי כדי לשאוף את השטיפות. יש לדגור על כל כביסה בטמפרטורה של 4°C למשך 5 דקות עם סיבוב. בצע את ההזמנה כמפורט להלן.
- יש לשטוף פעמיים עם FA-150.
- יש לשטוף פעם אחת עם FA-1M (FA-150 עם 1 M NaCl).
- יש לשטוף פעם אחת עם FA-0.5M (FA-150 עם 0.5 M NaCl). העברה לצינור PCR חדש.
- יש לשטוף פעם אחת עם TE-LiCl (250 mM LiCl, 10 mM Tris-Cl [pH 8.0], 1 mM EDTA, 1% IGEPAL CA-630, 1% נתרן deoxycholate).
- שטוף פעמיים עם TE+ (50 mM NaCl, 10 mM Tris-Cl [pH 8.0], 1 mM EDTA, 0.005% IGEPAL CA-630).
הערה: המשך עיבוד הדגימה בטמפרטורת החדר, אלא אם צוין אחרת.
- שאפו את חיץ הכביסה האחרון. השהה מחדש את החרוזים המגנטיים עם 50 μL של חיץ אלוציית ChIP (200 mM NaCl, 10 mM Tris-Cl [pH 8.0], 1 mM EDTA, 1% נתרן דודציל סולפט [SDS]) והעבר לצינור 1.5 מ"ל.
- יש לערבב ב-65°C למשך 15 דקות בתרמומיקסר עם ערבוב ב-1,000 סל"ד למשך 5 שניות בכל דקה.
- אוספים את החרוזים על מעמד מגנטי ומעבירים את הסופרנאטנט לצינור חדש.
- חזור על ה- elution עם עוד 50 μL של מאגר אלוציית ChIP. אגרו את הסופרנאטנטים בסכום כולל של 100 μL. המשיכו לקרוסלינקינג הפוך ופליטת DNA, כמפורט להלן.
9. הצלבה הפוכה ואלוציית DNA
- הפשיר את דגימת הקלט ליזט. למעלה עד 100 μL עם מאגר Elution ChIP.
- הוסף 16.5 מיקרוגרם של RNase A לכל דגימת IP וקלט. לדגור ב 37 ° C במשך 1 שעות.
- מוסיפים 40 מיקרוגרם של פרוטאינאז K ודגורים ב-55°C למשך שעתיים. לאחר מכן, לדגור ב 65 °C (65 °F) במשך הלילה.
- מצננים את הדגימות לטמפרטורת החדר ומטהרים את הדנ"א בעזרת ערכת עמוד ספין.
10. הגדרה והפעלה של תגובת qPCR
הערה: יש לשנות את הפרמטרים פריימר, הגדרת תגובה ותרמוסייקלר כך שיתאימו להמלצות היצרן לתמהיל תגובת qPCR הנמצא בשימוש.
- הכינו ערכות פריימר המכוונות לכתב ה-RNAi של GFP ולאזורי בקרת העשרה חיובית ושלילית של H3K9me3. טמפרטורת ההתכה של הפריימר היא 60 מעלות צלזיוס. ראו טבלת חומרים לרצפי פריימר.
- עבור דנ"א הקלט, בצע ארבעה דילולים סדרתיים פי ארבעה (לדוגמה, 1:5, 1:20, 1:80, 1:320).
הערה: ניתן להשתמש בדנ"א IP ישירות או לדלל אותו (לדוגמה, 1:2 או 1:3) כדי לאפשר תגובות נוספות. התאמת הדילול חייבת להיקבע אמפירית, שכן ביצועי qPCR עשויים להיות מושפעים. - ארגן את כל התגובות המתאימות לערכת פריימר אחת על צלחת PCR אחת. הגדר תגובות טכניות כפולות עבור כל דילול DNA קלט וכל דגימת DNA IP. כל תגובה של 10 μL מכילה 1.5 μL של קלט או IP DNA (או חיץ אלוציה כבקרה), תערובת מאסטר qPCR (ריכוז סופי של 1x), פריימר קדמי ופריימר הפוך (ריכוז סופי של 400 ננומטר עבור כל פריימר).
- על thermocycler בזמן אמת, להפעיל את התוכנית הבאה: דנטורציה ראשונית: 95 °C למשך 4 דקות; הגברה וזיהוי פלואורסצנטי: 40 מחזורים של 95 ° C עבור 10 s ו 60 ° C עבור 30 s עם לוח לקרוא; הארכה סופית: 60°C למשך 5 דקות; עקומת התכה: מ-60°C עד 90°C במרווחים של 0.5°C, 5 שניות לכל צעד.
11. קביעת יעילות הגברה ואימות ספציפיות המוצר
- עבור כל קבוצה של דילולי DNA קלט, התווה את ארבע נקודות הנתונים עם [log10(1/dilution)] על ציר x ו- [Input Cq] על ציר y. לקבוע את השיפוע של הקו של ההתאמה הטובה ביותר.
- חשב את יעילות ההגברה. ערכות פריימר אידיאליות אמורות להציג באופן עקבי יעילות של 95%-100%.
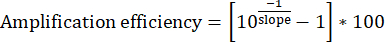
- בדקו שלכל התגובות יש שיא עקומת התכה חד אחד ושלתגובות עם אותה ערכת פריימר יש אותה טמפרטורת התכה. פסגות מרובות או טמפרטורות התכה שונות עשויות להצביע על הגברה לא ספציפית.
- לחלופין, הפעל תגובות על ג'ל סטנדרטי של 2% agarose/TAE בטמפרטורת החדר כדי לוודא את גודל רצועת המוצר.
12. חישוב אחוז הקלט
- חשב את גורם דילול הקלט. מכיוון ש-10% מנפח ה-IP ליזט נשמר כקלט, מקדם הדילול עבור דילול 1:5 של DNA קלט הוא 50.
- קבע את גורם דילול ה- IP. אם ה- DNA IP אינו מדולל לפני qPCR, גורם הדילול הוא 1.
- חשב את הפרש ה- Cq בין ה- IP לקלט המותאם לגורמי דילול.
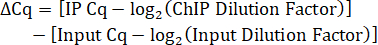
- חשב את אחוז הקלט.
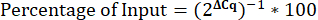
Representative Results
חיות שנשאו את כתב GFP-histone H2B [mex-5p::gfp-h2b::tbb-2 3'UTR]7 (איור 2A) נחשפו ל-GFP RNAi או לרנ"א בקרה באמצעות האכלה, ועברו כמתואר בפרוטוקול ובאיור 1. אות GFP גרעיני בקו הנבט הובחן ידנית באמצעות מיקרוסקופ מנתח פלואורסצנטי עבור דגימה של האוכלוסייה בכל דור. השתקת הטרנסגן חודרת במלואה בחיות P0 ו-F1 שטופלו ב-GFP RNAi (איור 2B). בדור ה-F2, שיעור האוכלוסייה המציגה ירושה של השתקת GFP היה כ-50%. עד דור F5, רוב האוכלוסייה לא הראתה ירושה של השתקה, ודור F10, לא נמצאה ירושה, כפי שכל בעלי החיים ביטאו GFP.
כדי לקבוע את השינוי בהעשרת H3K9me3 של היסטון המתאים להשתקה הנגרמת על ידי RNAi, ChIP-qPCR בוצע על בעלי חיים מדור F1 לאחר טיפול RNAi GFP או RNAi בקרה. כצפוי, האוכלוסייה שטופלה ב-GFP RNAi הציגה רמות גבוהות יותר של היסטון H3K9me3 ביעד ה-GFP ובאזור של 1.3 קילו-בתים במורד הזרם בהשוואה לחיות ביקורת שטופלו ב-RNAi (איור 2C). הספציפיות של ההיסטון H3K9me3 ChIP נתמכת על ידי העשרה במוקד בקרה חיובי (clec-18) הידוע כמועשר בסימן זה, אך לא במוקד בקרה שלילי קרוב (hrp-2). העשרת Histone H3 והעשרת כמעט רקע במשקעים החיסוניים של בקרת IgG זוהו גם הם בכל מוקדי qPCR, כצפוי. כאשר העשרת ChIP אצל המדווח מנורמלת למוקד הבקרה החיובי clec-18 , מוצגת העשרה גבוהה יותר של היסטון H3K9me3 על RNAi GFP, בעוד שהעשרת H3 של היסטון דומה בין הביקורת לבין טיפולי RNAi של GFP (איור 2D). מכיוון ש-GFP RNAi אינו צפוי להשפיע על היסטון H3K9me3 או על תפוסה כוללת של היסטון H3 בלוקוס clec-18 , נורמליזציה זו ממתנת את השונות הטכנית, כגון הבדלים ביעילות ChIP בין RNAi GFP לבין דגימות RNAi בקרה. שינוי הקיפול של רמות ההיסטון H3K9me3 וההיסטון H3 בין טיפולי RNAi מראה העשרה ספציפית של היסטון H3K9me3 לכתב GFP, ללא תלות בתפוסת ההיסטון, בהשתקה הנגרמת על ידי GFP RNAi (איור 2E).
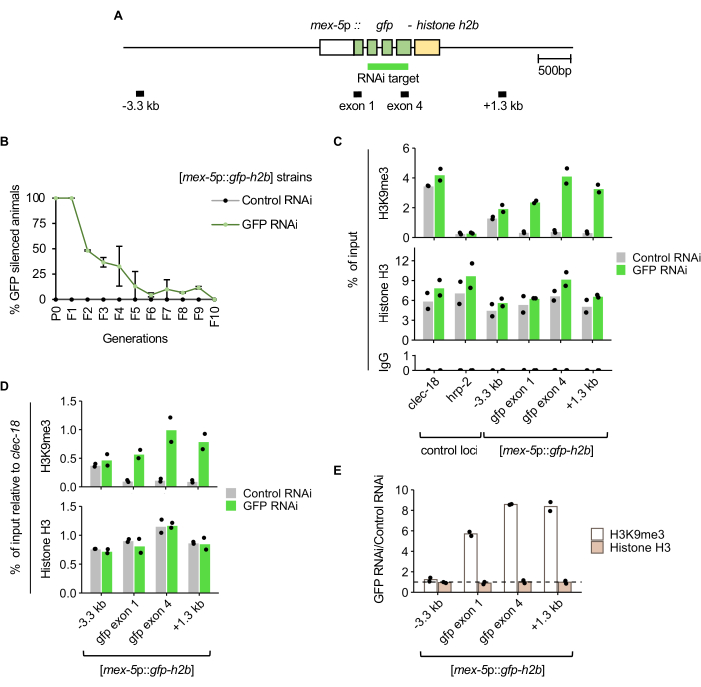
איור 2: השתקה הנגרמת על ידי GFP RNAi מתאימה להעשרה מוגברת של H3K9me3 ביעד ה-RNAi. (A) דיאגרמה של כתב ה-GFP RNAi mjIs134[mex-5p::gfp-h2b::tbb-2 3'UTR] עם אזורי אמפליקון qPCR מסומנים. (B) ביטוי GFP הבקיע לאורך דורות לאחר טיפולי RNAi ב 21 ° C. קווי שגיאה מייצגים את סטיית התקן משני משכפלים ביולוגיים. (C) בקרת ChIP-qPCR של H3K9me3, היסטון H3 ו-IgG בצעירים בפורמולה 1 משני שכפולים ביולוגיים. CLEC-18 ו-HRP-2 הם מוקדי הבקרה החיוביים והשליליים להעשרת H3K9me3, בהתאמה. (D) H3K9me3 והעשרת היסטון H3 בכתב RNAi GFP מנורמל למוקד הבקרה החיובי clec-18. (E) שינוי בהעשרת H3K9me3 וההיסטון H3 בין GFP RNAi ובעלי חיים שטופלו ב-RNAi בקרה, עם נורמליזציה ל-clec-18. קו מקווקו מייצג שינוי קיפול של 1. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Discussion
בפרוטוקול זה, dsRNA מוצג על ידי האכלה, אשר הפך לשיטה סטנדרטית ב- C. elegans18. עבור מבחני תורשה RNAi, גישת ההזנה מספקת שיטה קלה להשגת אוכלוסיית P0 גדולה 2,11,12,22,23,24,25. עם זאת, העיתוי ומשך החשיפה ל-RNAi משפיע על יעילות השתקת הטרנסגנים26, וריכוז חיידקי ה-RNAi משפיע על ההתמדה של השתקת RNAi תורשתית1. לכן, חיידקי RNAi מתוקננים וצמיחת תולעים חשובים להשגת רמה עקבית של השתקת GFP ומשך הירושה. כאן, עוברים מצופים על לוחות RNAi כך שחיות P0 נחשפות לחיידקי RNAi מהבקיעה. גישות חלופיות ציפו חיות שלב מסונכרנות L1 24,27 אוL4 25 על לוחות RNAi-NGM. בנוסף, מכיוון שרעב ולחצים אחרים משפיעים על תחזוקת תורשה RNAi13, יש לפקח על הצלחות כדי למנוע צפיפות ותשישות של אספקת המזון. כחלופה להתחלת RNAi על ידי הזנה, חלק ממחקרי התורשה החלוציים של C. elegans RNAi גרמו ל-RNAi באמצעות הזרקת גונאד, המספקת שליטה גדולה יותר בריכוז dsRNA 1,28.
בפרוטוקול זה, כל דור עובר כאוכלוסייה עם טיפול בהיפוכלוריט אלקליין, כפי שתואר קודם לכן 16,22,23,24. טיפול בהיפוכלוריט מבטיח שדור ה-F1 לא יזוהם בחיידקי RNAi מהסביבה ההורית22 ומונע צוואר בקבוק פוטנציאלי בלתי רצוי באוכלוסייה. עם זאת, מעבר בתפזורת עשוי להיות גם מגבלה, שכן לבעלי חיים בודדים עשויים להיות דפוסי ירושה שונים 1,29. שיטה חלופית להקים כל דור היא לבחור בעלי חיים בודדים 1,2,9,11,25. גישה זו מאפשרת מעקב אחר פנוטיפים בתוך שושלות ושילוב של צלבים גנטיים. ניתוח שושלת עשוי להיות יתרון גם עבור פנוטיפים של חדירה נמוכה12.
ביטוי חלבון פלואורסצנטי הוא קריאה עוצמתית ונוחה של השתקה הנגרמת על ידי RNAi 2,3,4,12,14,15,16,25,30. כמתואר בפרוטוקול זה, ניתן להבקיע ביטוי GFP באופן ידני כ- ON או OFF 11,12,15,16,25. ניתן לשפר עוד יותר את הניקוד הידני על ידי הקצאת רמות עצימות איכותיות 3,4,14. לחלופין, ניקוד עוצמה אוטומטי של תמונות מיקרוסקופיה 11,12,14,25,27 או מדידת פלואורסצנטיות של ציטומטריית זרימה של בעלי חיים2 יכולים לספק קריאה כמותית ובעלת תפוקה גבוהה. עם זאת, מכיוון שהביטוי של הכתב מוגבל לקו הנבט, אזהרה של גישות אוטומטיות היא שהן חייבות גם להבדיל בין בעלי חיים עם ביטוי GFP מושתק לעומת בעלי חיים ללא קו נבט, במיוחד אם המחקר משלב מוטנטים עם התפתחות לא תקינה של קו הנבט. כחלופה לפלואורסנציה של GFP, ניתן להשתמש בשעתוק לאחור (RT)-qPCR כדי לכמת את רמות המטרה של RNAi pre-mRNA ו-mRNA 2,16,23,24. גישה זו מספקת קריאה ישירה יותר של השתקה, המכוונת לרנ"א, והיא שימושית במיוחד עבור מטרות RNAi אחרות שבהן השתקה אינה מייצרת פנוטיפ נראה לעין. מגבלה בשימוש בכתבי GFP מלאכותיים היא שרצפים אקסוגניים ואנדוגניים מווסתים באופן דיפרנציאלי ב-RNAi11 בין-דורי. לפיכך, יש לשקול מחקרים עם מטרות אנדוגניות, כגון האלל הקטלני העוברי הרגיש לטמפרטורה oma-1(zu405)1,11,16,25, כגישה משלימה למדווחי טרנסגנים פלואורסצנטיים.
ניתוח ChIP בהקשר של מבחני תורשה RNAi דורש השוואה בין טיפולים ושכפולים. ראשית, כדי להסביר את ההבדלים בחומר המוצא בין הדגימות, אות ChIP מנורמל לאות קלט באותו מוקד כמו 'אחוז הקלט'. עיבוד של Histone H3 ChIP במקביל יעזור לקבוע אם שינויים כלשהם בשינוי ההיסטון תואמים לשינויים בצפיפות הנוקלאוזומים. בנוסף, מכיוון ש- ChIP הוא תהליך רב-שלבי, היעילות עשויה להשתנות בין דגימות. הבחירה של מוקדי בקרה חיוביים ושליליים מתאימים שימושית כדי להעריך ולהשוות את יחס האות לרעש בין דגימות וניסויים. בנוסף, כדי להקל על השוואות בין דגימות, ערכי מחזור הסף של ChIP DNA qPCR ביעד RNAi מנורמלים לעתים קרובות למוקד בקרה 3,11,15,23,30,31. כדי להעריך את ההשפעות של הטיפול ב-RNAi, משווים גם את היחס בין אות ה-ChIP בטיפול ב-RNAi לעומת תנאי RNAi של בקרה או ללא תנאי RNAi. מגבלה של הגישה הנוכחית היא כי ChIP מבוצע עם בעלי חיים שלמים, בעוד התגובה לטיפול RNAi עשוי להיות ייחודי לקו הנבט. גישה להתגבר על אזהרה זו היא לבצע ChIP באמצעות גרעיני נבט מבודדים. שיקולים טכניים נוספים לאופטימיזציה של ChIP נדונו בהרחבה גם במקומות אחרים32,33.
בסך הכל, פרוטוקול תורשה ו-ChIP זה של RNAi מספק בסיס מפורט וקל להסתגלות שניתן לשלב עם טכניקות אחרות כדי להמשיך לחקור רגולציה אפיגנטית בין-דורית. לדוגמה, ספריות ריצוף בתפוקה גבוהה יכולות להיבנות מהדנ"א ChIP (ChIP-seq) לקבלת תצוגה מפורטת יותר של נוף הכרומטין הן קרוב למטרה RNAi והן בקנה מידה גנומי רחב.
Disclosures
כל המחברים מאשרים כי אין להם התנגשויות לחשוף.
Acknowledgements
ברצוננו להודות למעבדות בקהילת C. elegans שפיתחו ושיתפו את הכלים ושעבודתם מצוטטת בכתב יד זה. חלק מהזנים סופקו על ידי CGC, הממומן על ידי המשרד לתוכניות תשתית מחקר של NIH (P40 OD010440). עבודה זו נתמכה על ידי מענק של פרויקט המכונים הקנדיים לחקר הבריאות (CIHR) ל- A.L.S. (PJT-175245). C.L. נתמכת על ידי מלגת מחקר לתארים מתקדמים של מועצת מדעי הטבע וההנדסה של קנדה (NSERC) (PGS-D).
Materials
| Name | Company | Catalog Number | Comments |
| Agarose | Bioshop | AGA002 | |
| Ampicillin | Bioshop | AMP201 | Make a 100 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Anti-H3K9me3 Rabbit Polyclonal Antibody | Abcam | ab8898 | Concentration is batch-dependent (0.9 - 1 mg/mL). |
| Anti-Histone H3 Rabbit Polyclonal Antibody | Abcam | ab1791 | Concentration is batch-dependent (0.7 - 1 mg/mL). |
| Bleach (6% Sodium hypochlorite) | Lavo | 02358107 | |
| C. elegans strain with GFP RNAi Reporter | NA | SX1263 | Sapetschnig et al. 2015 (ref. 7). A gift from E. Miska lab, University of Cambridge. |
| Carbenicillin | BioShop | CAR544 | Make a 25 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Dynabeads Protein G Magnetic Beads | Invitrogen | 10003D | |
| E. coli strain HT115(DE3) | Caenorhabditis Genetics Center (CGC) | HT115(DE3) | |
| E. coli strain OP50-1 | Caenorhabditis Genetics Center (CGC) | OP50-1 | |
| EDTA (0.5 M, pH 8.0) | Invitrogen | 15575020 | |
| Fluorescence Stereoscope | Zeiss | Axio Zoom.V16 | |
| Formaldehyde (37%) | Sigma | F8775 | |
| Glycine | Sigma | 50046 | Make a 1.25 M solution and store at 4 °C. |
| HEPES-KOH (1 M, pH 7.5) | Teknova | H1035 | |
| Hydrophobic Printed Slides, 10 wells | VWR | 100488-904 | |
| IGEPAL CA-630 (Octylphenol ethoxylate) | BioShop | NON999 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
| IPTG (Isopropyl-β-D-thiogalactoside) | BioShop | IPT001 | Make a 0.2 g/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 1725122 | |
| LB Agar Plates supplemented with 100 µg/mL Ampicillin | NA | NA | Standard lab recipe. |
| Levamisole (Tetramisole hydrochloride) | Sigma | L9756 | Make a 200 mM solution in ultrapure water. Store at -20 °C. |
| LiCl (8 M) | Sigma | L7026 | |
| M9 Buffer | NA | NA | 22 mM KH2PO4, 42 mM Na2HPO4, 86 mM NaCl, 1 mM MgSO4. |
| Magnetic Separator (1.5 mL tubes) | Applied Biosystems | A13346 | |
| Magnetic Separator (0.2 mL tubes) | Permagen | MSR812 | |
| Microscope Cover Glass | Fisher Scientific | 12541B | |
| Microscope Slide | Technologist Choice | LAB-037 | |
| NaCl (5 M) | Promega | V4221 | For ChIP buffers. |
| NaOH | Sigma | S5881 | Make a 10 M solution and store at room temperature. |
| NGM Plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 μg/mL cholesterol, 25 mM Potassium phosphate pH 6.0, 1 mM MgSO4, 50 µg/mL streptomycin. |
| Normal Rabbit IgG (1 mg/mL) | Cell Signaling Technology | 2729 | |
| Petri Dishes (35 mm x 10 mm) | Sarstedt | 82.1135.500 | |
| Phosphate Buffered Saline (10X) | Fisher BioReagents | BP3991 | |
| Plasmid - Control RNAi | Addgene | L4440 (Plasmid #1654) | |
| Plasmid - GFP-targetting RNAi | Addgene | L4417 (Plasmid #1649) | Note, alternative L4440-derived plasmids targeting GFP can be used. |
| Primer pair [-3.3 kb upstream of gfp] | Integrated DNA Technologies | NA | F: AAACCAAAGGACGAGAGATTCA, R: GGCTCGATCAAGTAAAATTTCG |
| Primer pair [+1.3 kb downstream of gfp] | Integrated DNA Technologies | NA | F: TCGACCAGTTCTAAAGTCACCG, R: ACGTGCGGGATCATTTCTTACT |
| Primer pair [clec-18] | Integrated DNA Technologies | NA | F: TGCTCCATGACCTCAACAACA, R: AGTACAGTTCACCGATCCAGA |
| Primer pair [gfp exon 1] | Integrated DNA Technologies | NA | F: CTGGAGTTGTCCCAATTCTTGT, R: GGGTAAGTTTTCCGTATGTTGC |
| Primer pair [gfp exon 4] | Integrated DNA Technologies | NA | F: GATGGCCCTGTCCTTTTACCA, R: ATGCCATGTGTAATCCCAGCA |
| Primer pair [hrp-2] | Integrated DNA Technologies | NA | F: CGTCAACAGGGAGCAGCTG, R: CCTCCGAACTTTCTCTGTCCA |
| Protease Inhibitor Cocktail Tablet | Roche | 11836170001 | |
| Proteinase K | Bioline | BIO-37084 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Real-Time PCR Detection System | Bio-Rad | CFX96 | |
| RNAi-NGM plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 µg/mL cholesterol, 25 mM Potassium phosphate buffer pH 6.0, 1 mM MgSO4, 25 µg/mL carbenicillin and 5 mM IPTG. |
| RNase A | Sigma | R4642 | |
| Sarkosyl (N-Lauroylsarcosine sodium salt) | Sigma | L5777 | Make a 10% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| SDS | Sigma | 74255 | Make a 10% (w/v) solution and store at room temperature. |
| Sodium deoxycholate | Sigma | 30970 | Make a 5% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| Sonication Tube | Evergreen | 214-3721-010 | |
| Sonication Tube Cap | Evergreen | 300-2911-020 | |
| Sonicator | Qsonica | Q800R3-110 | |
| Streptomycin sulfate | Bioshop | STP101 | Make a 50 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| TAE buffer (1X) | NA | NA | 40 mM Tris, 20 mM acetate, 1 mM EDTA |
| Tally counter clicker | Uline | H-7350 | |
| Tetracycline | Bioshop | TET701 | Make a 5 mg/mL solution in ethanol and store at -20 °C. |
| Thermomixer | Eppendorf | 05-400-205 | |
| Tris-HCl (1 M, pH 8.0) | Invitrogen | 15568025 | |
| Triton X-100 | Sigma | T8787 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
References
- Alcazar, R. M., Lin, R., Fire, A. Z. Transmission dynamics of heritable silencing induced by double-stranded RNA in Caenorhabditis elegans. Genetics. 180 (3), 1275-1288 (2008).
- Ashe, A., et al. piRNAs can trigger a multigenerational epigenetic memory in the germline of C. elegans. Cell. 150 (1), 88-99 (2012).
- Buckley, B. A., et al. A nuclear Argonaute promotes multigenerational epigenetic inheritance and germline immortality. Nature. 489 (7416), 447-451 (2012).
- Vastenhouw, N. L., et al. Long-term gene silencing by RNAi. Nature. 442 (7105), 882 (2006).
- Lee, H. -. C., et al. C. elegans piRNAs mediate the genome-wide surveillance of germline transcripts. Cell. 150 (1), 78-87 (2012).
- Luteijn, M. J., et al. Extremely stable Piwi-induced gene silencing in Caenorhabditis elegans. The EMBO Journal. 31 (16), 3422-3430 (2012).
- Sapetschnig, A., Sarkies, P., Lehrbach, N. J., Miska, E. A. Tertiary siRNAs mediate paramutation in C. elegans. PLoS Genetics. 11 (3), e1005078 (2015).
- Shirayama, M., et al. piRNAs initiate an epigenetic memory of nonself RNA in the C. elegans germline. Cell. 150 (1), 65-77 (2012).
- Minkina, O., Hunter, C. P. Stable heritable germline silencing directs somatic silencing at an endogenous locus. Molecular Cell. 65 (4), 659-670 (2017).
- Seroussi, U., et al. Mechanisms of epigenetic regulation by C. elegans nuclear RNA interference pathways. Seminars in Cell & Developmental Biology. 127, 142-154 (2022).
- Lev, I., Gingold, H., Rechavi, O. H3K9me3 is required for inheritance of small RNAs that target a unique subset of newly evolved genes. eLife. 8, e40448 (2019).
- Woodhouse, R. M., et al. Chromatin modifiers SET-25 and SET-32 are required for establishment but not long-term maintenance of transgenerational epigenetic inheritance. Cell Reports. 25 (8), 2259-2272 (2018).
- Houri-Zeevi, L., Teichman, G., Gingold, H., Rechavi, O. Stress resets ancestral heritable small RNA responses. eLife. 10, e65797 (2021).
- Houri-Ze'evi, L., et al. A tunable mechanism determines the duration of the transgenerational small RNA inheritance in C. elegans. Cell. 165 (1), 88-99 (2016).
- Spracklin, G., et al. The RNAi inheritance machinery of Caenorhabditis elegans. Genetics. 206 (3), 1403-1416 (2017).
- Perales, R., et al. Transgenerational epigenetic inheritance is negatively regulated by the HERI-1 chromodomain protein. Genetics. 210 (4), 1287-1299 (2018).
- Shukla, A., Perales, R., Kennedy, S. piRNAs coordinate poly(UG) tailing to prevent aberrant and perpetual gene silencing. Current Biology. 31 (20), 4473-4485 (2021).
- Ahringer, J. Reverse GeneticsWormBook: the Online Review of C. elegans Biology. WormBook. , (2006).
- Rivera Gomez, K., Schvarzstein, M. Immobilization of nematodes for live imaging using an agarose pad produced with a Vinyl Record. microPublication Biology. 2018, (2018).
- Stiernagle, T. Maintenance of C. elegans. WormBook: The Online Review of C. Elegans Biology. , 1-11 (2006).
- Askjaer, P., Ercan, S., Meister, P. Modern techniques for the analysis of chromatin and nuclear organization in C. elegans. WormBook: the Online Review of C. elegans Biology. , 1-35 (2014).
- Gu, S. G., et al. Amplification of siRNA in Caenorhabditis elegans generates a transgenerational sequence-targeted histone H3 lysine 9 methylation footprint. Nature Genetics. 44 (2), 157-164 (2012).
- Kalinava, N., Ni, J. Z., Peterman, K., Chen, E., Gu, S. G. Decoupling the downstream effects of germline nuclear RNAi reveals that H3K9me3 is dispensable for heritable RNAi and the maintenance of endogenous siRNA-mediated transcriptional silencing in Caenorhabditis elegans. Epigenetics & Chromatin. 10, 6 (2017).
- Kalinava, N., et al. elegans heterochromatin factor SET-32 plays an essential role in transgenerational establishment of nuclear RNAi-mediated epigenetic silencing. Cell Reports. 25 (8), 2273-2284 (2018).
- Lev, I., et al. MET-2-dependent H3K9 methylation suppresses transgenerational small RNA inheritance. Current Biology. 27 (8), 1138-1147 (2017).
- Kamath, R. S., Martinez-Campos, M., Zipperlen, P., Fraser, A. G., Ahringer, J. Effectiveness of specific RNA-mediated interference through ingested double-stranded RNA in Caenorhabditis elegans. Genome Biology. 2 (1), (2001).
- Xu, F., et al. A cytoplasmic Argonaute protein promotes the inheritance of RNAi. Cell Reports. 23 (8), 2482-2494 (2018).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287 (5462), 2494-2497 (2000).
- Houri-Zeevi, L., Korem Kohanim, Y., Antonova, O., Rechavi, O. Three rules explain transgenerational small RNA inheritance in C. elegans. Cell. 182 (5), 1186-1197 (2020).
- Burton, N. O., Burkhart, K. B., Kennedy, S. Nuclear RNAi maintains heritable gene silencing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences. 108 (49), 19683-19688 (2011).
- Mao, H., et al. The Nrde pathway mediates small-RNA-directed histone H3 lysine 27 trimethylation in Caenorhabditis elegans. Current Biology. 25 (18), 2398-2403 (2015).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22 (9), 1813-1831 (2012).
- Mukhopadhyay, A., Deplancke, B., Walhout, A. J. M., Tissenbaum, H. A. Chromatin immunoprecipitation (ChIP) coupled to detection by quantitative real-time PCR to study transcription factor binding to DNA in Caenorhabditis elegans. Nature Protocols. 3 (4), 698-709 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved