Análise da Herança Epigenética Transgeracional em C. elegans Usando um Fluorescente Reporter e Imunoprecipitação de Cromatina (ChIP)
* These authors contributed equally
In This Article
Summary
Este protocolo descreve um ensaio de interferência de RNA e ChIP para estudar a herança epigenética do silenciamento induzido por RNAi e modificações associadas da cromatina em C. elegans.
Abstract
A herança epigenética transgeracional (TEI) permite a transmissão de informações através da linha germinativa sem alterar a sequência do genoma, através de fatores como RNAs não codificantes e modificações na cromatina. O fenômeno da herança de RNA de interferência (RNAi) no nematoide Caenorhabditis elegans é um modelo eficaz para investigar TEI que aproveita o curto ciclo de vida, autopropagação e transparência desse organismo modelo. Na herança de RNAi, a exposição dos animais ao RNAi leva ao silenciamento gênico e assinaturas alteradas de cromatina no locus alvo que persistem por várias gerações na ausência do gatilho inicial. Este protocolo descreve a análise da herança de RNAi em C. elegans usando um repórter de proteína fluorescente verde nuclear (GFP) expressa em linhagem germinativa. O silenciamento do repórter é iniciado alimentando os animais com bactérias que expressam RNA de fita dupla visando a GFP. A cada geração, os animais são passados para manter o desenvolvimento sincronizado, e o silenciamento gênico do repórter é determinado por microscopia. Em gerações selecionadas, populações são coletadas e processadas para imunoprecipitação de cromatina (ChIP)-reação em cadeia da polimerase quantitativa (qPCR) para medir o enriquecimento de modificação de histonas no locus repórter da GFP. Este protocolo para estudar a herança de RNAi pode ser facilmente modificado e combinado com outras análises para investigar fatores TEI em pequenas vias de RNA e cromatina.
Introduction
A herança epigenética permite a transmissão de informações reguladoras de genes através de gerações e, portanto, pode permitir que o ambiente ou as experiências dos pais afetem sua progênie. Em C. elegans, o silenciamento gênico germinativo iniciado por RNA exógeno de fita dupla (dsRNA) pode ser herdado por múltiplas gerações em progênies não expostas ao gatilho original 1,2,3,4. Esse processo, denominado herança de interferência de RNA (RNAi), é um dos vários fenômenos de silenciamento epigenético relacionados em C. elegans, incluindo silenciamento multigeracional iniciado por piRNA 2,5, paramutação/RNAe (silenciamento epigenético induzido por RNA)6,7,8 e silenciamento iniciado por array de múltiplas cópias9, que têm requisitos sobrepostos, mas distintos, para pequenas máquinas de regulação de RNA e cromatina . No RNAi exógeno de C. elegans, o dsRNA é processado em pequenos RNAs interferentes (siRNAs) que atuam em um complexo com proteínas primárias de Argonauta para reconhecer seu RNAm alvo. Este alvo leva à amplificação de siRNAs secundários que se associam com Argonautas secundários para silenciar o mRNA alvo através de vias de silenciamento citoplasmático e nuclear. Para alvos de RNAi expressos em linhagem germinativa, o Argonaute HRDE-1 secundário nuclear nuclear e fatores adicionais de RNAi têm como alvo transcritos nascentes, resultando em repressão transcricional e recrutamento de histonas metiltransferases para depositar marcas repressivas de cromatina, incluindo H3K9me310. Histone H3K9me3 promove o estabelecimento do silenciamento hereditário de transgenes de proteína verde fluorescente (GFP) expressa em germinação por herança de RNAi11,12.
O objetivo deste protocolo é usar um transgene expressando uma proteína de fusão GFP-histona na linha germinativa como repórter para herança de RNAi e testar mudanças nas modificações de histonas no locus transgênico repórter usando imunoprecipitação de cromatina e reação em cadeia da polimerase quantitativa (ChIP-qPCR). Este protocolo descreve uma abordagem padronizada de alimentação de RNAi baseada em placas para iniciar o silenciamento do repórter. Também fornece uma linha do tempo detalhada para a passagem de animais entre gerações, isolando embriões in utero de adultos grávidos por tratamento com hipoclorito alcalino ('branqueamento'). Métodos e dados representativos para monitorar a frequência de silenciamento de GFP em um subgrupo da população por microscopia de fluorescência e para a histona H3K9me3 ChIP-qPCR também são descritos. Ensaios de herança de RNAi baseados em repórteres fornecem um sistema altamente tratável para dissecar funcionalmente os papéis de fatores genéticos e ambientais na regulação epigenética 13,14, e rastreios genéticos usando tais repórteres identificaram tanto genes que são necessários para 2,3,15 quanto genes que regulam negativamente16,17 a duração da herança epigenética transgeracional.
Protocol
Observação : uma linha do tempo de ensaio é fornecida na Figura 1.
1. Preparação de placas de meio de crescimento de nematoides RNAi (RNAi-NGM)
- Extrair E. coli HT115(DE3) contendo vetores de GFP ou RNAi de controle de estoques de glicerol em placas de ágar Luria-Bertani (LB) suplementadas com 100 μg/mL de ampicilina. Incubar as bactérias durante a noite a 37 °C.
- No dia seguinte, preparar placas de RNAi-NGM (ágar 1,7% [p/v], NaCl 0,3% [p/v], peptona 0,25% [p/v], CaCl2 mM, 5 μg/mL de colesterol, tampão fosfato de potássio 25 mM [pH 6,0], MgSO4 mM, carbenicilina 25 μg/mL e isopropil 5 mM β-d-1-thiogalactopiranosídeo [IPTG]) de acordo com o protocolo padrão18.
- Para cada cepa do ensaio, preparar um mínimo de seis placas de RNAi-NGM semeadas com RNAi de GFP (duas placas para cada uma das três réplicas biológicas) e duas placas de RNAi-NGM semeadas com RNAi controle (uma réplica biológica).
- Tenda frouxamente os pratos com papel alumínio para proteger da luz e deixe durante a noite em temperatura ambiente para secar.
- No mesmo dia do derramamento da placa de RNAi-NGM, preparar 4 mL de culturas líquidas de bactérias RNAi.
- Em condições estéreis, alíquota 4 mL de caldo LB suplementado com 10 μg/mL de tetraciclina e 100 μg/mL de ampicilina em tubos de cultura. Colher colônias únicas das placas de ágar LB em cada tubo e incubar durante a noite enquanto agita por aproximadamente 16 h a 37 °C.
- Sementes de bactérias RNAi nas placas RNAi-NGM.
- Após o crescimento noturno, medir a densidade óptica (OD600) das culturas usando uma diluição de 1:5 de bactérias com caldo LB.
- Diluir bactérias RNAi para um OD relativo 600 de 2 usando caldo LB suplementado com 10 μg/mL tetraciclina e100 μg/mL ampicilina.
- Sob condições estéreis, adicionar 150 μL da bactéria controle ou GFP RNAi em cada placa RNAi-NGM.
- Tenda as placas com papel alumínio e deixe as bactérias crescerem por pelo menos 24 h à temperatura ambiente antes do uso.
NOTA: As placas RNAi-NGM não utilizadas podem ser armazenadas invertidas por até 1 semana a 4 °C em um recipiente selado no escuro.
2. Início do ensaio de herança de RNAi: branqueamento e plaqueamento de embriões para a geração de P0
NOTA: Antes de iniciar o ensaio de herança RNAi, os worms contendo o repórter GFP mjIs134 [mex-5p::gfp-h2b::tbb-2 3'UTR]7 devem ser mantidos sem fome a 21 °C por pelo menos duas gerações.
- Aos 4 dias (96 h) antes do início do ensaio de herança RNAi (Figura 1), escolher três ou quatro adultos gravídicos em cada placa NGM padrão de 35 mm semeada com bactérias OP50-1.
NOTA: Três placas NGM por cepa são suficientes. Para cepas com fertilidade comprometida, podem ser necessários mais de quatro animais por placa. - Após 4 dias, certifique-se de que a população adulta tenha começado a colocar embriões no prato. Lavar os vermes das placas em tubos de 1,5 mL usando 800 μL de tampão M9 suplementado com Triton X-100 (TX-100) (22 mM KH 2 PO 4, 42 mMNa2HPO 4, 86 mM NaCl, 1mM MgSO 4, 0,01% (v/v) TX-100). Repita a lavagem e coloque na mesma tubulação.
- Isole os embriões da população através do clareamento da seguinte forma.
- Preparar uma solução clareadora com água sanitária (hipoclorito de sódio a 6%) e NaOH 10 N numa relação de volume de 1,5:1. Preparar uma quantidade suficiente para que possam ser utilizados 250 μL para cada amostra.
- Centrifugar os tubos de 1,5 mL com os vermes a 1.000 x g por 1,5-2 min.
- Aspirar o sobrenadante, deixando 100 μL sem perturbar a "pelota" do verme. Alternativamente, os tubos podem ser deixados por aproximadamente 2 minutos para permitir que os vermes se estabeleçam antes de aspirar.
- Adicionar 650 μL de ddH2O a cada tubo para atingir um volume final de 750 μL.
Observação : as etapas a seguir são sensíveis ao tempo. - Adicionar 250 μL da solução clareadora a cada tubo e iniciar um temporizador.
- A cada 1-2 min, vórtice os tubos completamente. Após 5 min, verifique os vermes sob o estereoscópio para monitorar a degradação dos adultos.
- Continue o vórtice até que os vermes estejam completamente dissolvidos e restem apenas embriões. Centrifugar imediatamente os tubos a 1.000 x g por 1,5 min.
NOTA: O clareamento geralmente é completo dentro de 6-7 min, mas deve ser monitorado sob um estereoscópio em uma ampliação suficiente para observar os embriões liberados (40x). Não deixe os vermes em água sanitária por um período prolongado, pois isso comprometerá os embriões. - Após a centrifugação, aspirar o sobrenadante e deixar aproximadamente 50-100 μL. Adicionar 1 mL de tampão M9 com TX-100 para lavar e misturar por vórtice. Centrifugar novamente a 1.000 x g por 1,5-2 min e lavar pelo menos mais duas vezes.
- Aspirar o sobrenadante da lavagem final e deixar aproximadamente 100 μL no tubo. Misture por vórtice. Pipetar 2 μL de cada tubo duas vezes para uma lâmina de vidro marcada. Conte os embriões usando um contador de contagem para estimar a concentração de embriões por microlitro.
NOTA: Lâminas com vários poços gelados podem ser usadas aqui para contar réplicas de várias cepas. As lâminas de contagem podem ser lavadas e reutilizadas.
- Para iniciar um crescimento populacional semi-sincronizado, misturar e pipetar um volume contendo 250 embriões em cada placa RNAi-NGM. Use duas placas para cada réplica. Deixe o líquido absorver.
- Para a geração P0 tratada com RNAi, incubar a 21 °C por 4 dias (96 h) (dos embriões até a idade adulta). Os tempos podem variar de acordo com o genótipo.
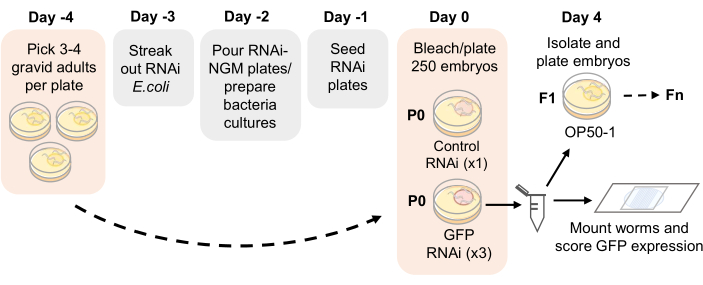
Figura 1: Esquema do ensaio de herança RNAi. Linha do tempo proposta para preparação da placa RNAi-NGM e configuração do ensaio de herança RNAi. Adultos grávidas são colhidos no Dia -4 em placas NGM semeadas com OP50-1. Após 4 dias, a progênie adulta é branqueada e os embriões são plaqueados em placas de RNAi-NGM. A geração P0 é exposta ao RNAi por 4 dias a 21 °C. Uma vez que os vermes atingem a idade adulta, as réplicas são passadas por branqueamento e marcadas para a expressão de GFP germinativa a cada geração. Clique aqui para ver uma versão maior desta figura.
3. Aprovação e pontuação de cada geração para expressão de GFP germinativa
NOTA: Para facilitar a pontuação, use um disco de vinil para criar almofadas de agarose com cristas lineares para alinhar os vermes. Esse método foi adaptado de Rivera Gomez e Schvarzstein19.
- Preparar 1% (p/v) de agarose dissolvendo agarose em ddH2O num balão pequeno por aquecimento no micro-ondas. Adicione uma barra de agitação e cubra com papel alumínio. Se voltar a derreter, aqueça a agarose numa placa de aquecimento a 200 °C enquanto agita. Depois de derretido, reduza o fogo para 80 °C.
- Lave os vermes das placas de NGM usando 800 μL de tampão M9 com TX-100 em um tubo de 1,5 mL. Lave as placas duas vezes para remover todos os adultos gravídicos. Verifique as placas sob o estereoscópio para confirmar se os animais foram coletados de forma eficiente.
- Centrifugar os tubos a 1.000 x g por 1,5-2 min ou deixar os vermes se acomodarem por 2 min. Aspirar o sobrenadante e deixar 100 μL sem perturbar a "pelota" do verme.
- Prepare almofadas de agarose para montar os vermes da seguinte maneira.
- Coloque uma gota de agarose derretida a 1% (p/v) no disco de vinil (descrito anteriormente19) usando uma pipeta de vidro Pasteur (com a extremidade estreita quebrada).
- Oriente uma lâmina de vidro para que as linhas no registro sejam horizontais ou verticais e coloque a lâmina rapidamente sobre a gota de agarose. Aguarde aproximadamente 30 s antes de remover a lâmina de vidro do registro.
NOTA: Se pontuar vários genótipos, pode ser útil fazer uma almofada de agarose maior em uma lâmina e cortá-la ao meio usando uma lâmina.
- Monte os vermes nas almofadas de agarose.
- Sob o estereoscópio, adicione aproximadamente 5 μL de levamisol 5 mM à almofada de agarose. A diluição de levamisol deve ser feita fresca a cada semana.
- Agite suavemente os tubos de vermes para misturar e transferir 5-10 μL de vermes para a almofada de agarose. Estime o número de vermes a olho nu à medida que eles estão sendo adicionados para que haja aproximadamente 40 vermes por replicação.
- Alinhe os vermes em fileiras usando uma picareta de cílios para facilitar a pontuação. Adicione uma tampa de vidro.
NOTA: Lâminas com animais montados desta forma podem durar várias horas antes de secar.
- Isolar os embriões dos demais animais utilizando o protocolo de clareamento (ver etapa 2.3) antes de pontuar as lâminas. Placa 250 embriões em placas NGM 35 mm semeadas com OP50-120. Uma vez absorvido o líquido, inverta as placas e devolva-as à incubadora a 21 °C.
- Use um estereoscópio de florescência com um conjunto de filtros GFP. Conte e registre o número de worms positivos e negativos para GFP nos slides de cada réplica usando um contador de contagem.
NOTA: Vermes positivos para GFP terão expressão nuclear de GFP em sua linha germinativa e embriões no útero. As linhas germinativas de vermes GFP negativos não fluorescem, no entanto, como a GFP começa a voltar a ligar em gerações posteriores, a fluorescência pode ser fraca. Pode ser útil montar cepas adicionais de vermes não transgênicos para explicar qualquer autofluorescência observada. - Passagem da população por branqueamento, como descrito acima, aproximadamente a cada 4 dias (96 h) a 21 °C. Alternativamente, a passagem pode ser feita em um horário alternado de 3 dias / 4 dias para conveniência. Placa de ~50 embriões extras para as gerações de passagem mais curtas se alternando, pois pode haver um menor rendimento de embriões após 72 h.
NOTA: Manter o tempo de passagem da geração P0 consistente em 4 dias pós-plaqueamento dos embriões.
4. Coleta de animais para ChIP
NOTA: O número de animais e o tempo dependem da cepa, estágio de desenvolvimento, epítopo e número de alvos de imunoprecipitação (IP). No exemplo abaixo, colete animais para três IPs: H3K9me3, histona H3 e controle IgG. O protocolo ChIP foi adaptado de Askjaer et al.21.
- Antes da coleta da amostra de ChIP, expanda a geração anterior do ensaio de herança de RNAi por mais três placas de NGM (para pelo menos quatro placas por replicação). Crescer durante 4 dias a 21 °C.
- Branquear os adultos gravídicos para isolar os embriões (ver passo 2.3).
- Para cada cepa, plaquear aproximadamente 3.500 embriões em 14 placas (250 por placa) e crescer por 3 dias a 21 °C até a fase adulta jovem.
- Lavar os animais em tubos de 1,5 mL com solução salina tamponada com fosfato (PBS) contendo TX-100 a 0,01% (v/v). Centrifugar a 1.000 x g por 2 min. Aspirar e agrupar os animais de uma cepa em um tubo e lavar mais três vezes com pelo menos 1 mL de PBS/TX.
- Aspirar até 1.000 μL, inverter para misturar e contar o número de animais em 3 μL três vezes (ver passo 2.3.9).
- Calcular a concentração de animais e transferir um volume correspondente a 3.000 animais para um novo tubo para reticulação de formaldeído.
- Certifique-se de que o volume total seja pelo menos 10 vezes o volume da pastilha de sem-fim.
5. Reticulação de formaldeído
CUIDADO: Trabalhe com formaldeído em um exaustor para evitar a exposição ao vapor.
- Adicionar formaldeído (concentração final de 1,8%) ao tubo contendo as minhocas. Gire à temperatura ambiente durante 6 min.
- Congelar imediatamente em nitrogênio líquido. O experimento pode ser pausado nesta etapa e as amostras podem ser armazenadas a -80 °C.
- Descongelar a amostra reticulada em banho-maria à temperatura ambiente durante 3 minutos e rodar durante 16 minutos à temperatura ambiente.
- Adicionar 1,25 M de glicina a uma concentração final de 125 mM e rodar durante 5 min.
NOTA: Até à eluição do cordão magnético, manter as amostras no gelo ou a 4 °C e utilizar tampões gelados. - Centrifugar a amostra a 1.000 x g por 3 min. Lavar três vezes com 1 mL de PBS/TX de cada vez. Lavar duas vezes com 1 mL de tampão de ressuspensão (NaCl 150 mM, hepes-KOH 50 mM [pH 7,5], ácido etilenodiaminotetracético 1 mM [EDTA], TX-100 a 0,01%, inibidor de protease [um comprimido por 5 mL]).
- Após a última lavagem, deixe tampão suficiente para garantir que o volume total seja pelo menos três vezes o volume do pellet e um mínimo de ~100 μL.
6. Sonicação
NOTA: Os parâmetros de sonicação dependem do tipo e modelo do sonicador e do estágio animal. Parâmetros como volume e concentração da amostra, intervalos liga/desliga, número de ciclos e ajuste de potência precisam ser otimizados empiricamente. Por exemplo, ao longo de um curso de tempo de sonicação, monitore a lise do verme usando um ensaio de proteína e determine quando a concentração atinge um platô. Além disso, monitorar quando o tamanho médio de cisalhamento do DNA genômico é de aproximadamente 200-1.000 pb por eletroforese de DNA, purificado após reversão de ligações cruzadas, em um gel de agarose/tris-acetato-EDTA (TAE) a 1,5%.
- Meça o volume de amostras da etapa anterior. Misture e alíquota 90-120 μL a um tubo de sonicação de poliestireno.
- Adicionar um volume igual de tampão de ressuspensão contendo 2x detergentes (NaCl 150 mM, hepes-koh 50 mM [pH 7,5], EDTA 1 mM, desoxicolato de sódio a 0,2%, sarkosil a 0,7%).
- Sonicate em um sonicador em banho-maria a 50% de potência por 7 min (20 s on/40 s off) a 4 °C. Misture suavemente por pipetagem. Repita a sonicação por mais 7 minutos.
- Transfira o lisado sonicado para um tubo de 1,5 mL. Adicionar 0,5 volumes de tampão de ressuspensão sem detergentes (NaCl 150 mM, hepes-koh 50 mM [pH 7,5], EDTA 1 mM) (por exemplo, adicionar 100 μL de tampão a 200 μL de amostra).
- Centrifugar a 13.000 x g por 15 min a 4 °C. Mantenha o sobrenadante lisado e transfira para um novo tubo.
- Opcionalmente, realizar um ensaio proteico para determinar as concentrações de cada lisado. Esta etapa pode ser útil para grandes quantidades de amostras ou comparações entre ensaios. No entanto, a padronização do número de animais como descrito acima funciona bem para as quantidades de amostra aqui descritas, uma vez que há melhor correlação com o rendimento de cromatina/DNA determinado por qPCR.
7. Imunoprecipitação
NOTA: Dimensione a quantidade de anticorpos e esferas magnéticas para o volume e concentração de lisado.
- Divida o sobrenadante lisado em quatro porções: três volumes iguais para cada IP e 10% do volume de um IP como Entrada (por exemplo, 100 μL de IP #1, 100 μL de IP #2, 100 μL de IP #3, 10 μL de Entrada). Transfira o lisado IP para um tubo de PCR de 200 μL e armazene o lisado de entrada a -20 °C em um tubo de 1,5 mL.
- Adicionar 0,5 μg de anticorpo anti-H3K9me3, anticorpo anti-histona H3 ou IgG à amostra IP apropriada. Incubar a 4 °C durante a noite com rotação.
- No dia seguinte, alíquota de 9 μL de esferas magnéticas revestidas com proteína G por IP para um único tubo de 1,5 mL. Lavar as esferas duas vezes com 1 mL de FA-150 (NaCl 150 mM, Hepes-KOH 50 mM [pH 7,5], EDTA 1 mM, TX-100 1%, desoxicolato de sódio 0,1%).
- Ressuspenda as esferas magnéticas no buffer FA-150 para o volume original retirado do estoque na etapa 7.3 acima. Adicionar 7,5 μL a cada IP. Incubar a 4 °C durante 2 h com rotação.
8. Lavagens e eluição
NOTA: Para garantir que as esferas magnéticas não sequem, adicione cada tampão de lavagem ou eluição rapidamente após aspirar a lavagem anterior.
- Use 0,2-1 mL das seguintes soluções tampão cada vez para lavar as contas. Recolher as contas em um suporte magnético para aspirar as lavagens. Incubar cada lavagem a 4 °C durante 5 minutos com rotação. Siga a ordem abaixo.
- Lave duas vezes com FA-150.
- Lave uma vez com FA-1M (FA-150 com NaCl 1M).
- Lave uma vez com FA-0,5M (FA-150 com NaCl 0,5M). Transferência para um novo tubo de PCR.
- Lavar uma vez com TE-LiCl (250 mM LiCl, 10 mM Tris-Cl [pH 8,0], 1 mM EDTA, 1% IGEPAL CA-630, 1% desoxicolato de sódio).
- Lave duas vezes com TE+ (NaCl 50 mM, Tris-Cl 10 mM [pH 8,0], 1 mM EDTA, 0,005% IGEPAL CA-630).
NOTA: Continuar o processamento da amostra à temperatura ambiente, salvo indicação em contrário.
- Aspirar o último tampão de lavagem. Ressuspender as esferas magnéticas com 50 μL de tampão de eluição ChIP (NaCl 200 mM, Tris-Cl 10 mM [pH 8,0], EDTA 1 mM, dodecil sulfato de sódio a 1% [SDS]) e transferir para um tubo de 1,5 mL.
- Elute a 65 °C por 15 min em um termomisturador com mistura a 1.000 rpm por 5 s a cada minuto.
- Recolha as contas num suporte magnético e transfira o sobrenadante para um novo tubo.
- Repetir a eluição com mais 50 μL de tampão de eluição de ChIP. Junte os sobrenadantes para um total de 100 μL. Proceda à reticulação reversa e à eluição de DNA, conforme detalhado abaixo.
9. Reticulação reversa e eluição de DNA
- Descongelar a amostra de lisado de entrada. Recarregue até 100 μL com buffer de eluição ChIP.
- Adicionar 16,5 μg de RNase A a cada amostra IP e Input. Incubar a 37 °C durante 1 h.
- Adicionar 40 μg de proteinase K e incubar a 55 °C durante 2 h. Em seguida, incubar a 65 °C durante a noite.
- Resfriar as amostras à temperatura ambiente e purificar o DNA com um kit de coluna de spin.
10. Configuração e execução da reação qPCR
NOTA: O primer, a configuração da reação e os parâmetros do termociclador devem ser modificados para corresponder às recomendações do fabricante para a mistura de reação qPCR em uso.
- Preparar conjuntos de primers visando o repórter GFP RNAi e as regiões de controle de enriquecimento positivo e negativo H3K9me3. A temperatura de fusão do primer é de 60 °C. Consulte Tabela de materiais para obter sequências de primers.
- Para o DNA de entrada, faça quatro diluições seriais quatro vezes (por exemplo, 1:5, 1:20, 1:80, 1:320).
NOTA: O DNA IP pode ser usado diretamente ou diluído (por exemplo, 1:2 ou 1:3) para permitir mais reações. A adequação da diluição deve ser determinada empiricamente, uma vez que o desempenho da qPCR pode ser afetado. - Organize todas as reações correspondentes a um primer fixado em uma placa de PCR. Configure reações técnicas duplicadas para cada diluição de DNA de entrada e cada amostra de DNA IP. Cada reação de 10 μL contém 1,5 μL de DNA de entrada ou IP (ou tampão de eluição como controle), mistura mestre de qPCR (concentração final de 1x), primer para frente e primer reverso (concentração final de 400 nM para cada primer).
- Em um termociclador em tempo real, execute o seguinte programa: desnaturação inicial: 95 °C por 4 min; amplificação e detecção de fluorescência: 40 ciclos de 95 °C por 10 s e 60 °C por 30 s com leitura em placa; extensão final: 60 °C por 5 min; curva de fusão: de 60 °C a 90 °C em incrementos de 0,5 °C, 5 s por passo.
11. Determinação da eficiência da amplificação e verificação da especificidade do produto
- Para cada conjunto de diluições de DNA de entrada, plote os quatro pontos de dados com [log10(1/diluição)] no eixo x e [Input Cq] no eixo y. Determine a inclinação da linha de melhor ajuste.
- Calcule a eficiência da amplificação. Os conjuntos de primers ideais devem exibir consistentemente 95%-100% de eficiência.
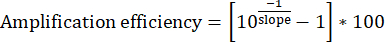
- Verifique se todas as reações têm um pico acentuado de curva de fusão e se as reações com o mesmo conjunto de primers têm a mesma temperatura de fusão. Vários picos ou diferentes temperaturas de fusão podem indicar amplificação não específica.
- Opcionalmente, execute as reações em um gel padrão de agarose/TAE a 2% à temperatura ambiente para verificar o tamanho da banda do produto.
12. Cálculo do percentual de Insumos
- Calcule o fator de diluição de entrada. Como 10% do volume de lisado IP foi salvo como Input, o fator de diluição para a diluição 1:5 do Input DNA é 50.
- Determine o fator de diluição IP. Se o DNA IP não for diluído antes da qPCR, o fator de diluição é 1.
- Calcule a diferença de Cq entre o IP e o Input ajustado para fatores de diluição.
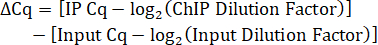
- Calcule a porcentagem de Entrada.
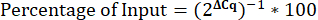
Representative Results
Animais portadores da histona H2B da GFP expressa germinativa [mex-5p::gfp-h2b::tbb-2 3'UTR]7 repórter (Figura 2A) foram expostos ao RNAi da GFP ou RNAi de controle por meio da alimentação e passados conforme descrito no protocolo e na Figura 1. O sinal nuclear de GFP na linha germinativa foi manualmente marcado usando um microscópio dissecante de fluorescência para uma amostra da população em cada geração. O silenciamento do transgene foi totalmente penetrante nos animais pontuados P0 e F1 tratados com GFP RNAi (Figura 2B). Na geração F2, a proporção da população exibindo herança do silenciamento de GFP foi de aproximadamente 50%. Na geração F5, a maioria da população não apresentou herança de silenciamento e, na geração F10, nenhuma herança foi detectada, pois todos os animais expressaram GFP.
Para determinar a mudança no enriquecimento de histona H3K9me3 correspondente ao silenciamento induzido por RNAi, ChIP-qPCR foi realizada em animais da geração F1 após o tratamento com RNAi GFP ou RNAi controle. Como esperado, a população tratada com GFP RNAi exibiu níveis mais altos de histona H3K9me3 no alvo de GFP e na região a jusante de 1,3 kb em comparação com animais tratados com RNAi controle (Figura 2C). A especificidade da histona H3K9me3 ChIP é suportada pelo enriquecimento em um locus de controle positivo (clec-18) conhecido por ser enriquecido nesta marca, mas não em um locus de controle negativo próximo (hrp-2). O enriquecimento com histona H3 e o enriquecimento próximo ao fundo nas imunoprecipitações de controle de IgG também foram detectados em todos os loci de qPCR, como esperado. Quando o enriquecimento de ChIP no repórter é normalizado para o locus de controle positivo clec-18 , maior enriquecimento de histona H3K9me3 sobre o RNAi da GFP é mostrado, enquanto o enriquecimento de histona H3 é semelhante entre os tratamentos controle e RNAi da GFP (Figura 2D). Como não se espera que o RNAi da GFP afete a histona H3K9me3 ou a ocupação total da histona H3 no locus clec-18 , essa normalização atenua a variação técnica, como diferenças na eficiência de ChIP entre as amostras de RNAi da GFP e RNAi de controle. A mudança de dobra dos níveis de histona H3K9me3 e histona H3 entre os tratamentos de RNAi mostra enriquecimento de histona H3K9me3 específico para repórteres de GFP, independente da ocupação de histonas, após silenciamento induzido por RNAi de GFP (Figura 2E).
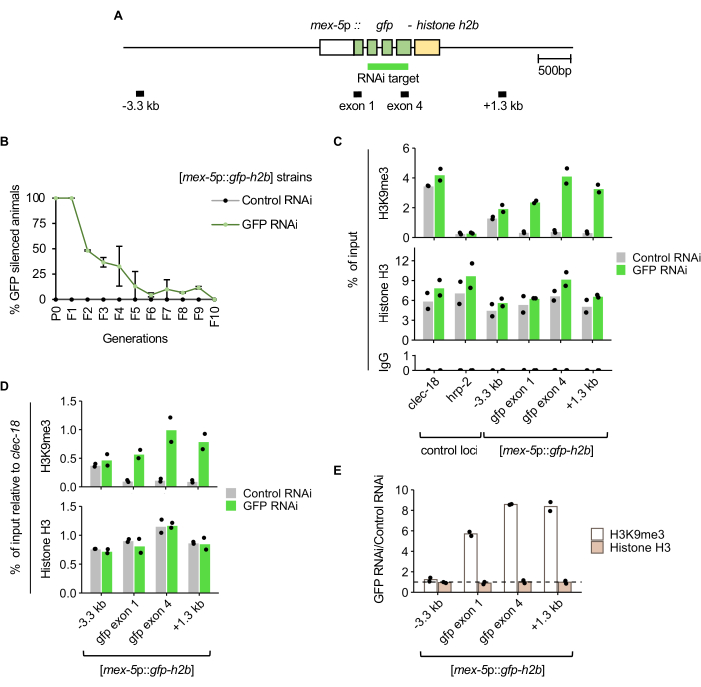
Figura 2: O silenciamento induzido por RNAi da GFP corresponde ao elevado enriquecimento de H3K9me3 no alvo do RNAi. (A) Diagrama do repórter de RNAi de GFP expresso em linhagem germinativa mjIs134[mex-5p::gfp-h2b::tbb-2 3'UTR] com regiões de amplicon de qPCR marcadas. (B) Expressão de GFP pontuada ao longo das gerações após tratamentos com RNAi a 21 °C. As barras de erro representam o desvio padrão de duas réplicas biológicas. (C) ChIP-qPCR do controle de H3K9me3, histona H3 e IgG em adultos jovens F1 de duas réplicas biológicas. clec-18 e hrp-2 são os loci de controle positivo e negativo para o enriquecimento com H3K9me3, respectivamente. (D) O enriquecimento de H3K9me3 e histona H3 no repórter de RNAi da GFP normalizou para o locus de controle positivo clec-18. (E) Alteração no enriquecimento de H3K9me3 e histona H3 entre animais tratados com RNAi de GFP e RNAi controle, com normalização para clec-18. A linha pontilhada representa uma mudança de dobra de 1. Clique aqui para ver uma versão maior desta figura.
Discussion
Nesse protocolo, o dsRNA é introduzido pela alimentação, que se tornou um método padrão em C. elegans18. Para ensaios de herança de RNAi, a abordagem alimentar fornece um método fácil para obter uma grande população de P0 2,11,12,22,23,24,25. No entanto, o tempo e a duração da exposição ao RNAi afetam a eficácia do silenciamento transgênico26, e a concentração de bactérias RNAi afeta a persistência do silenciamento hereditário de RNAi1. Portanto, bactérias RNAi padronizadas e o crescimento de vermes são importantes para obter um nível consistente de silenciamento de GFP e duração da herança. Aqui, os embriões são plaqueados em placas de RNAi para que os animais P0 sejam expostos às bactérias RNAi desde a eclosão. Abordagens alternativas têm plaqueado animais sincronizados nos estágios L1 24,27 ouL4 25 em placas RNAi-NGM. Além disso, como a fome e outros estresses afetam a manutenção da herança do RNAi13, os pratos devem ser monitorados para evitar superlotação e esgotamento da oferta de alimentos. Como alternativa para iniciar RNAi por alimentação, alguns dos estudos pioneiros de herança de RNAi de C. elegans induziram RNAi por meio de injeção de gônadas, o que proporciona maior controle da concentração de dsRNA 1,28.
Nesse protocolo, cada geração é passada como uma população com tratamento com hipoclorito alcalino, conforme descrito anteriormente 16,22,23,24. O tratamento com hipoclorito garante que a geração F1 não será contaminada com bactérias RNAi do ambiente parental22 e evita possíveis gargalos populacionais indesejados. No entanto, a passagem em massa também pode ser uma limitação, uma vez que animais individuais podem ter padrões de herança distintos 1,29. Um método alternativo para estabelecer cada geração é selecionar animais individuais 1,2,9,11,25. Essa abordagem permite o rastreamento de fenótipos dentro de linhagens e a incorporação de cruzamentos genéticos. A análise de linhagens também pode ser vantajosa para fenótipos de baixa penetrância12.
A expressão de proteínas fluorescentes é uma leitura poderosa e conveniente do silenciamento induzido por RNAi 2,3,4,12,14,15,16,25,30. Conforme descrito neste protocolo, a expressão da GFP pode ser pontuada manualmente como ON ou OFF 11,12,15,16,25. A pontuação manual pode ser refinada com a atribuição de níveis qualitativos de intensidade 3,4,14. Alternativamente, a pontuação automatizada da intensidade das imagens de microscopia 11,12,14,25,27 ou a medição da fluorescência da citometria de fluxo de animais vivos2 podem fornecer uma leitura quantitativa e de alto rendimento. No entanto, como a expressão do repórter é restrita à linha germinativa, uma ressalva das abordagens automatizadas é que elas também devem diferenciar entre animais com expressão silenciada de GFP versus animais sem linha germinativa, especialmente se o estudo incorporar mutantes com desenvolvimento germinativo anormal. Como alternativa à fluorescência da GFP, a transcrição reversa (RT)-qPCR pode ser usada para quantificar os níveis de RNAi alvo de pré-mRNA e mRNA 2,16,23,24. Esta abordagem fornece uma leitura mais direta do silenciamento, que tem como alvo o RNA, e é especialmente útil para outros alvos de RNAi onde o silenciamento não produz um fenótipo visível. Uma limitação do uso de repórteres artificiais de GFP é que sequências exógenas e endógenas são reguladas diferencialmente em RNAi transgeracional11. Estudos com alvos endógenos, como o alelo letal embrionário sensível à temperatura oma-1(zu405)1,11,16,25, devem, portanto, ser considerados como uma abordagem complementar aos repórteres de transgenes fluorescentes.
A análise de ChIP no contexto de ensaios de herança de RNAi requer comparação entre tratamentos e réplicas. Primeiro, para levar em conta as diferenças no material de partida entre as amostras, o sinal ChIP é normalizado para o sinal de entrada no mesmo locus que a 'porcentagem de entrada'. O processamento paralelo de uma histona H3 ChIP ajudará a determinar se quaisquer alterações na modificação da histona correspondem a mudanças na densidade nucleossômica. Além disso, como o ChIP é um processo de várias etapas, a eficiência pode variar entre as amostras. A seleção de loci de controle positivo e negativo apropriados é útil para avaliar e comparar a relação sinal-ruído entre amostras e experimentos. Além disso, para facilitar as comparações entre as amostras, os valores do ciclo de limiar de qPCR do DNA ChIP no alvo RNAi são frequentemente normalizados para um locus controle 3,11,15,23,30,31. Para avaliar os efeitos do tratamento com RNAi, a relação do sinal de ChIP em condições de RNAi de tratamento versus controle ou ausência de RNAi também é comparada. Uma limitação da abordagem atual é que o ChIP é realizado com animais inteiros, enquanto a resposta ao tratamento com RNAi pode ser exclusiva da linhagem germinativa. Uma abordagem para superar essa ressalva é realizar ChIP usando núcleos germinativos isolados. Considerações técnicas adicionais para otimizar a ChIP também foram amplamente discutidas em outros estudos32,33.
No geral, esta herança RNAi e protocolo ChIP fornece uma base detalhada e fácil de adaptar que pode ser integrada com outras técnicas para explorar ainda mais a regulação epigenética transgeracional. Por exemplo, bibliotecas de sequenciamento de alto rendimento podem ser construídas a partir do DNA ChIP (ChIP-seq) para uma visão mais detalhada da paisagem da cromatina tanto proximal ao alvo RNAi quanto em escala genômica.
Disclosures
Todos os autores confirmam que não têm conflitos a revelar.
Acknowledgements
Gostaríamos de agradecer aos laboratórios da comunidade de C. elegans que desenvolveram e compartilharam as ferramentas e cujo trabalho é citado neste manuscrito. Algumas cepas foram fornecidas pelo CGC, que é financiado pelo NIH Office of Research Infrastructure Programs (P40 OD010440). Este trabalho foi apoiado por uma bolsa do Projeto Canadian Institutes of Health Research (CIHR) para A.L.S. (PJT-175245). C.L. é apoiado por uma bolsa de pós-graduação do Natural Sciences and Engineering Research Council of Canada (NSERC) (PGS-D).
Materials
| Name | Company | Catalog Number | Comments |
| Agarose | Bioshop | AGA002 | |
| Ampicillin | Bioshop | AMP201 | Make a 100 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Anti-H3K9me3 Rabbit Polyclonal Antibody | Abcam | ab8898 | Concentration is batch-dependent (0.9 - 1 mg/mL). |
| Anti-Histone H3 Rabbit Polyclonal Antibody | Abcam | ab1791 | Concentration is batch-dependent (0.7 - 1 mg/mL). |
| Bleach (6% Sodium hypochlorite) | Lavo | 02358107 | |
| C. elegans strain with GFP RNAi Reporter | NA | SX1263 | Sapetschnig et al. 2015 (ref. 7). A gift from E. Miska lab, University of Cambridge. |
| Carbenicillin | BioShop | CAR544 | Make a 25 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Dynabeads Protein G Magnetic Beads | Invitrogen | 10003D | |
| E. coli strain HT115(DE3) | Caenorhabditis Genetics Center (CGC) | HT115(DE3) | |
| E. coli strain OP50-1 | Caenorhabditis Genetics Center (CGC) | OP50-1 | |
| EDTA (0.5 M, pH 8.0) | Invitrogen | 15575020 | |
| Fluorescence Stereoscope | Zeiss | Axio Zoom.V16 | |
| Formaldehyde (37%) | Sigma | F8775 | |
| Glycine | Sigma | 50046 | Make a 1.25 M solution and store at 4 °C. |
| HEPES-KOH (1 M, pH 7.5) | Teknova | H1035 | |
| Hydrophobic Printed Slides, 10 wells | VWR | 100488-904 | |
| IGEPAL CA-630 (Octylphenol ethoxylate) | BioShop | NON999 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
| IPTG (Isopropyl-β-D-thiogalactoside) | BioShop | IPT001 | Make a 0.2 g/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 1725122 | |
| LB Agar Plates supplemented with 100 µg/mL Ampicillin | NA | NA | Standard lab recipe. |
| Levamisole (Tetramisole hydrochloride) | Sigma | L9756 | Make a 200 mM solution in ultrapure water. Store at -20 °C. |
| LiCl (8 M) | Sigma | L7026 | |
| M9 Buffer | NA | NA | 22 mM KH2PO4, 42 mM Na2HPO4, 86 mM NaCl, 1 mM MgSO4. |
| Magnetic Separator (1.5 mL tubes) | Applied Biosystems | A13346 | |
| Magnetic Separator (0.2 mL tubes) | Permagen | MSR812 | |
| Microscope Cover Glass | Fisher Scientific | 12541B | |
| Microscope Slide | Technologist Choice | LAB-037 | |
| NaCl (5 M) | Promega | V4221 | For ChIP buffers. |
| NaOH | Sigma | S5881 | Make a 10 M solution and store at room temperature. |
| NGM Plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 μg/mL cholesterol, 25 mM Potassium phosphate pH 6.0, 1 mM MgSO4, 50 µg/mL streptomycin. |
| Normal Rabbit IgG (1 mg/mL) | Cell Signaling Technology | 2729 | |
| Petri Dishes (35 mm x 10 mm) | Sarstedt | 82.1135.500 | |
| Phosphate Buffered Saline (10X) | Fisher BioReagents | BP3991 | |
| Plasmid - Control RNAi | Addgene | L4440 (Plasmid #1654) | |
| Plasmid - GFP-targetting RNAi | Addgene | L4417 (Plasmid #1649) | Note, alternative L4440-derived plasmids targeting GFP can be used. |
| Primer pair [-3.3 kb upstream of gfp] | Integrated DNA Technologies | NA | F: AAACCAAAGGACGAGAGATTCA, R: GGCTCGATCAAGTAAAATTTCG |
| Primer pair [+1.3 kb downstream of gfp] | Integrated DNA Technologies | NA | F: TCGACCAGTTCTAAAGTCACCG, R: ACGTGCGGGATCATTTCTTACT |
| Primer pair [clec-18] | Integrated DNA Technologies | NA | F: TGCTCCATGACCTCAACAACA, R: AGTACAGTTCACCGATCCAGA |
| Primer pair [gfp exon 1] | Integrated DNA Technologies | NA | F: CTGGAGTTGTCCCAATTCTTGT, R: GGGTAAGTTTTCCGTATGTTGC |
| Primer pair [gfp exon 4] | Integrated DNA Technologies | NA | F: GATGGCCCTGTCCTTTTACCA, R: ATGCCATGTGTAATCCCAGCA |
| Primer pair [hrp-2] | Integrated DNA Technologies | NA | F: CGTCAACAGGGAGCAGCTG, R: CCTCCGAACTTTCTCTGTCCA |
| Protease Inhibitor Cocktail Tablet | Roche | 11836170001 | |
| Proteinase K | Bioline | BIO-37084 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Real-Time PCR Detection System | Bio-Rad | CFX96 | |
| RNAi-NGM plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 µg/mL cholesterol, 25 mM Potassium phosphate buffer pH 6.0, 1 mM MgSO4, 25 µg/mL carbenicillin and 5 mM IPTG. |
| RNase A | Sigma | R4642 | |
| Sarkosyl (N-Lauroylsarcosine sodium salt) | Sigma | L5777 | Make a 10% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| SDS | Sigma | 74255 | Make a 10% (w/v) solution and store at room temperature. |
| Sodium deoxycholate | Sigma | 30970 | Make a 5% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| Sonication Tube | Evergreen | 214-3721-010 | |
| Sonication Tube Cap | Evergreen | 300-2911-020 | |
| Sonicator | Qsonica | Q800R3-110 | |
| Streptomycin sulfate | Bioshop | STP101 | Make a 50 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| TAE buffer (1X) | NA | NA | 40 mM Tris, 20 mM acetate, 1 mM EDTA |
| Tally counter clicker | Uline | H-7350 | |
| Tetracycline | Bioshop | TET701 | Make a 5 mg/mL solution in ethanol and store at -20 °C. |
| Thermomixer | Eppendorf | 05-400-205 | |
| Tris-HCl (1 M, pH 8.0) | Invitrogen | 15568025 | |
| Triton X-100 | Sigma | T8787 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
References
- Alcazar, R. M., Lin, R., Fire, A. Z. Transmission dynamics of heritable silencing induced by double-stranded RNA in Caenorhabditis elegans. Genetics. 180 (3), 1275-1288 (2008).
- Ashe, A., et al. piRNAs can trigger a multigenerational epigenetic memory in the germline of C. elegans. Cell. 150 (1), 88-99 (2012).
- Buckley, B. A., et al. A nuclear Argonaute promotes multigenerational epigenetic inheritance and germline immortality. Nature. 489 (7416), 447-451 (2012).
- Vastenhouw, N. L., et al. Long-term gene silencing by RNAi. Nature. 442 (7105), 882 (2006).
- Lee, H. -. C., et al. C. elegans piRNAs mediate the genome-wide surveillance of germline transcripts. Cell. 150 (1), 78-87 (2012).
- Luteijn, M. J., et al. Extremely stable Piwi-induced gene silencing in Caenorhabditis elegans. The EMBO Journal. 31 (16), 3422-3430 (2012).
- Sapetschnig, A., Sarkies, P., Lehrbach, N. J., Miska, E. A. Tertiary siRNAs mediate paramutation in C. elegans. PLoS Genetics. 11 (3), e1005078 (2015).
- Shirayama, M., et al. piRNAs initiate an epigenetic memory of nonself RNA in the C. elegans germline. Cell. 150 (1), 65-77 (2012).
- Minkina, O., Hunter, C. P. Stable heritable germline silencing directs somatic silencing at an endogenous locus. Molecular Cell. 65 (4), 659-670 (2017).
- Seroussi, U., et al. Mechanisms of epigenetic regulation by C. elegans nuclear RNA interference pathways. Seminars in Cell & Developmental Biology. 127, 142-154 (2022).
- Lev, I., Gingold, H., Rechavi, O. H3K9me3 is required for inheritance of small RNAs that target a unique subset of newly evolved genes. eLife. 8, e40448 (2019).
- Woodhouse, R. M., et al. Chromatin modifiers SET-25 and SET-32 are required for establishment but not long-term maintenance of transgenerational epigenetic inheritance. Cell Reports. 25 (8), 2259-2272 (2018).
- Houri-Zeevi, L., Teichman, G., Gingold, H., Rechavi, O. Stress resets ancestral heritable small RNA responses. eLife. 10, e65797 (2021).
- Houri-Ze'evi, L., et al. A tunable mechanism determines the duration of the transgenerational small RNA inheritance in C. elegans. Cell. 165 (1), 88-99 (2016).
- Spracklin, G., et al. The RNAi inheritance machinery of Caenorhabditis elegans. Genetics. 206 (3), 1403-1416 (2017).
- Perales, R., et al. Transgenerational epigenetic inheritance is negatively regulated by the HERI-1 chromodomain protein. Genetics. 210 (4), 1287-1299 (2018).
- Shukla, A., Perales, R., Kennedy, S. piRNAs coordinate poly(UG) tailing to prevent aberrant and perpetual gene silencing. Current Biology. 31 (20), 4473-4485 (2021).
- Ahringer, J. Reverse GeneticsWormBook: the Online Review of C. elegans Biology. WormBook. , (2006).
- Rivera Gomez, K., Schvarzstein, M. Immobilization of nematodes for live imaging using an agarose pad produced with a Vinyl Record. microPublication Biology. 2018, (2018).
- Stiernagle, T. Maintenance of C. elegans. WormBook: The Online Review of C. Elegans Biology. , 1-11 (2006).
- Askjaer, P., Ercan, S., Meister, P. Modern techniques for the analysis of chromatin and nuclear organization in C. elegans. WormBook: the Online Review of C. elegans Biology. , 1-35 (2014).
- Gu, S. G., et al. Amplification of siRNA in Caenorhabditis elegans generates a transgenerational sequence-targeted histone H3 lysine 9 methylation footprint. Nature Genetics. 44 (2), 157-164 (2012).
- Kalinava, N., Ni, J. Z., Peterman, K., Chen, E., Gu, S. G. Decoupling the downstream effects of germline nuclear RNAi reveals that H3K9me3 is dispensable for heritable RNAi and the maintenance of endogenous siRNA-mediated transcriptional silencing in Caenorhabditis elegans. Epigenetics & Chromatin. 10, 6 (2017).
- Kalinava, N., et al. elegans heterochromatin factor SET-32 plays an essential role in transgenerational establishment of nuclear RNAi-mediated epigenetic silencing. Cell Reports. 25 (8), 2273-2284 (2018).
- Lev, I., et al. MET-2-dependent H3K9 methylation suppresses transgenerational small RNA inheritance. Current Biology. 27 (8), 1138-1147 (2017).
- Kamath, R. S., Martinez-Campos, M., Zipperlen, P., Fraser, A. G., Ahringer, J. Effectiveness of specific RNA-mediated interference through ingested double-stranded RNA in Caenorhabditis elegans. Genome Biology. 2 (1), (2001).
- Xu, F., et al. A cytoplasmic Argonaute protein promotes the inheritance of RNAi. Cell Reports. 23 (8), 2482-2494 (2018).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287 (5462), 2494-2497 (2000).
- Houri-Zeevi, L., Korem Kohanim, Y., Antonova, O., Rechavi, O. Three rules explain transgenerational small RNA inheritance in C. elegans. Cell. 182 (5), 1186-1197 (2020).
- Burton, N. O., Burkhart, K. B., Kennedy, S. Nuclear RNAi maintains heritable gene silencing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences. 108 (49), 19683-19688 (2011).
- Mao, H., et al. The Nrde pathway mediates small-RNA-directed histone H3 lysine 27 trimethylation in Caenorhabditis elegans. Current Biology. 25 (18), 2398-2403 (2015).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22 (9), 1813-1831 (2012).
- Mukhopadhyay, A., Deplancke, B., Walhout, A. J. M., Tissenbaum, H. A. Chromatin immunoprecipitation (ChIP) coupled to detection by quantitative real-time PCR to study transcription factor binding to DNA in Caenorhabditis elegans. Nature Protocols. 3 (4), 698-709 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved