Полногеномное секвенирование для быстрой характеристики вируса бешенства с использованием технологии нанопор
In This Article
Summary
В этой статье мы представляем быстрый и экономичный рабочий процесс для характеристики геномов вируса бешенства (RABV) с использованием технологии нанопор. Рабочий процесс предназначен для поддержки геномного эпиднадзора на местном уровне, предоставляя информацию о циркулирующих линиях RABV и их размещении в региональных филогенезах для принятия мер по борьбе с бешенством.
Abstract
Геномные данные могут быть использованы для отслеживания передачи и географического распространения инфекционных заболеваний. Тем не менее, возможности секвенирования, необходимые для геномного надзора, остаются ограниченными во многих странах с низким и средним уровнем дохода (СНСД), где бешенство, передаваемое собаками, и/или бешенство, передаваемое дикими животными, такими как летучие мыши-вампиры, представляет собой серьезную проблему для общественного здравоохранения и экономики. Здесь мы представляем быстрый и доступный рабочий процесс от образца до последовательности и интерпретации с использованием технологии нанопор. Кратко описаны протоколы сбора образцов и диагностики бешенства, за которыми следуют подробности оптимизированного рабочего процесса полногеномного секвенирования, включая разработку праймера и оптимизацию для мультиплексной полимеразной цепной реакции (ПЦР), модифицированной, недорогой подготовки библиотеки секвенирования, секвенирование с живым и автономным вызовом оснований, определение генетической линии и филогенетический анализ. Демонстрируется реализация рабочего процесса и выделяются важнейшие шаги для локального развертывания, такие как валидация конвейера, оптимизация праймеров, включение негативных элементов контроля и использование общедоступных данных и геномных инструментов (GLUE, MADDOG) для классификации и размещения в региональных и глобальных филогениях. Время выполнения рабочего процесса составляет 2–3 дня, а стоимость варьируется от 25 долларов США за образец для запуска из 96 образцов до 80 долларов США за образец для запуска из 12 образцов. Мы пришли к выводу, что организация геномного надзора за вирусом бешенства в СНСД осуществима и может способствовать прогрессу в достижении глобальной цели по достижению к 2030 году нулевого уровня смертности людей от бешенства, вызванного собаками, а также усилению мониторинга распространения бешенства в диких животных. Кроме того, платформа может быть адаптирована для других патогенов, что поможет создать универсальный геномный потенциал, способствующий обеспечению готовности к эпидемиям и пандемиям.
Introduction
Вирус бешенства (RABV) является лиссавирусом семейства Rhabdoviridae , вызывающим смертельное неврологическое заболевание у млекопитающих1. Несмотря на то, что бешенство на 100% можно предотвратить с помощью вакцинации, оно остается серьезной проблемой общественного здравоохранения и экономики в эндемичных странах. Из 60 000 случаев смерти людей от бешенства, которые, по оценкам, происходят каждый год, более 95% приходится на Африку и Азию, где собаки являются основным резервуаром бешенства. Напротив, вакцинация собак привела к элиминации бешенства, передаваемого собаками, в Западной Европе, Северной Америке и большей части Латинской Америки. В этих регионах резервуары бешенства в настоящее время ограничены дикими животными, такими как летучие мыши, еноты, скунсы и дикие псовые3. По всей Латинской Америке обыкновенная летучая мышь-вампир является проблематичным источником бешенства из-за регулярной передачи инфекции от летучих мышей как людям, так и домашнему скоту во времяночного кормления кровью. Ежегодный глобальный экономический ущерб от бешенства оценивается в 8,6 миллиарда долларов США, при этом потери скота составляют 6%5.
Данные о последовательностях вирусных патогенов в сочетании с метаданными о времени и источнике инфекции могут обеспечить надежную эпидемиологическую информацию6. Что касается RABV, секвенирование было использовано для расследования происхождения вспышек7,8, выявления ассоциаций хозяина с дикими или домашними собаками 8,9,10,11,12 и отслеживания источников случаев заболевания людей 13,14. Расследования вспышек с использованием филогенетического анализа показали, что бешенство возникло в ранее свободной от бешенства провинции Бали, Индонезия, в результате однократного завоза из близлежащих эндемичных районов Калимантана или Сулавеси15. Между тем, на Филиппинах было доказано, что вспышка на острове Таблас, провинция Ромблон, была завезена с главного острова Лусон16. Вирусные геномные данные также использовались для лучшего понимания динамики передачи патогенов, необходимой для принятия мер по борьбе с вирусом на географическом уровне. Например, геномная характеристика RABV иллюстрирует географическую кластеризацию клад 17,18,19, совместную циркуляцию линий 20,21,22, опосредованное человеком вирусное движение 17,23,24 и динамику метапопуляций 25,26.
Мониторинг заболеваний является одной из важных функций геномного надзора, которая была усилена в связи с глобальным расширением возможностей секвенирования в ответ на пандемию SARS-CoV-2. Геномный надзор способствовал отслеживанию в режиме реального времени вызывающих озабоченность вариантов SARS-COV-227,28 и связанных с ними контрмер6. Достижения в области доступных технологий секвенирования, таких как технология нанопор, привели к созданию улучшенных и более доступных протоколов для быстрого секвенирования патогенов как 29,30,31,32 человека, так и 33,34,35 у животных. Тем не менее, во многих эндемичных по бешенству странах по-прежнему существуют препятствия для внедрения геномного надзора за патогенами, о чем свидетельствуют глобальные различия в возможностях секвенирования SARS-CoV-236. Ограничения в лабораторной инфраструктуре, цепочках поставок и технических знаниях затрудняют создание и рутинизацию геномного надзора. В этой статье мы продемонстрируем, как оптимизированный, быстрый и доступный рабочий процесс полногеномного секвенирования может быть развернут для эпиднадзора за RABV в условиях ограниченных ресурсов.
Protocol
Исследование было одобрено Координационным комитетом медицинских исследований Национального института медицинских исследований (NIMR/HQ/R.8a/vol. IX/2788), Министерство региональной администрации и местного самоуправления (AB.81/288/01) и Институциональный наблюдательный совет Института здравоохранения Ифакары (IHI/IRB/No:22-2014) в Танзании; Институт тропических и инфекционных болезней Найробийского университета (P947/11/2019) и Кенийский медицинский научно-исследовательский институт (KEMRI-SERU; протокол No 3268) в Кении; и Научно-исследовательский институт тропической медицины (RITM), Департамент здравоохранения (2019-023) на Филиппинах. Секвенирование образцов, происходящих из Нигерии, было проведено на основе архивных диагностических материалов, собранных в рамках национального эпиднадзора.
ПРИМЕЧАНИЕ: Разделы 1-4 являются предварительными требованиями. В разделах 5-16 описывается рабочий процесс секвенирования нанопор RABV от образца к последовательности и интерпретации (рис. 1). Для последующих этапов протокола, требующих импульсного центрифугирования, центрифуга при 10-15 000 x g в течение 5-15 с.
1. Настройка вычислительной среды для секвенирования и анализа данных
- Откройте веб-сайт Oxford Nanopore Technology (ONT)37 и создайте учетную запись для доступа к ресурсам, посвященным нанопорам.
- Войдите в систему и установите программное обеспечение для определения последовательности ONT и базового вызова38.
- Откройте GitHub39 и создайте учетную запись.
- Перейдите в репозитории artic-rabv40 и MADDOG41 и следуйте инструкциям по установке.
2. Разработка или обновление мультиплексной схемы праймера
ПРИМЕЧАНИЕ: Существующие схемы RABV доступны в репозитории artic-rabv40. При нацеливании на новую географическую область следует разработать новую схему или изменить существующую, чтобы включить дополнительное разнообразие.
- Выберите референсный набор генома, представляющий разнообразие в исследуемой области; Как правило, это набор общедоступных последовательностей (например, из NCBI GenBank) или предварительные внутренние данные. Выполните шаг 2.1.1, чтобы использовать RABV-GLUE42, ресурс данных последовательности RABV, для фильтрации и загрузки последовательностей NCBI и связанных метаданных.
ПРИМЕЧАНИЕ: Выбирайте референсные последовательности с полными геномами (т.е. без пробелов и маскированных оснований). Рекомендуется выбрать до 10 последовательностей в качестве эталонного набора для проектирования праймера. Если имеющиеся данные о последовательностях являются неполными или нерепрезентативными для исследуемой области, обратитесь к рекомендациям43,44,45 в Дополнительном файле 1.- Перейдите на страницу NCBI RABV Sequences by Clade из раскрывающегося меню Sequence Data в RABV-GLUE. Нажмите на ссылку Вирус бешенства (RABV), чтобы получить доступ ко всем доступным данным или выбрать конкретную интересующую кладу. Используйте параметр фильтра, чтобы добавить фильтры , соответствующие требуемым критериям (например, страна происхождения, длина последовательности). Скачивание последовательностей и метаданных.
- Сгенерируйте схему праймера для мультиплексной полимеразной цепной реакции (ПЦР) в соответствии с инструкциями, приведенными в Primal Scheme46. Для секвенирования образцов низкого качества рекомендуется использовать схему 400.н. с перекрытием 50.н. Загрузите и сохраните все выходные данные (не редактируйте имена файлов или праймеров).
ПРИМЕЧАНИЕ: Схема будет проиндексирована в соответствии с первой последовательностью во входном фасте, которая в дальнейшем будет называться «индексной ссылкой» (рисунок 2). В дополнительном файле 1 приведены параметры оптимизации производительности праймера.
3. Настройка конвейеров биоинформатики RAMPART и ARTIC
- Обратитесь к Дополнительному файлу 2 , чтобы настроить структуру каталогов для управления входными/выходными файлами для RAMPART и конвейера ARTIC bioinformatics.
4. Биобезопасность и лабораторная организация
- Работа с потенциально положительными образцами на бешенство в условиях 2 или 3 уровня биобезопасности (BSL).
- Обеспечить, чтобы сотрудники лабораторий прошли предэкспозиционную вакцинацию против бешенства и мониторинг иммунитета в соответствии с рекомендациями Всемирной организации здравоохранения (ВОЗ)3.
- Обеспечьте наличие в лаборатории специальных стандартных операционных процедур и оценок рисков в соответствии с национальными или международными рекомендациями.
- Необходимое лабораторное оборудование: Сведите к минимуму загрязнение, соблюдая физическое разделение между зонами до и после ПЦР. В лабораториях с ограниченным пространством или в полевых условиях используйте переносные перчаточные ящики или импровизированные лабораторные станции, чтобы свести к минимуму загрязнение.
- В этом протоколе обязательно выделены отдельные области для:
- Извлечение образцов: Установите шкаф/перчаточный ящик BSL2/3 для работы с биологическим материалом и выполнения инактивации и экстракции РНК.
- Область шаблона: Установите шкаф/перчаточный ящик BSL1 для добавления матрицы (РНК/кДНК) в предварительно подготовленную мастер-смесь реакции.
- Зона мастер-микса: Установите специальную чистую зону (шкаф BSL1/перчаточный ящик) для приготовления мастер-смесей реагентов. В этой области не должно быть шаблона.
- Зона после ПЦР: Выделите отдельную зону для работы над ампликонами и секвенированием библиотечной подготовки.
ПРИМЕЧАНИЕ: Все участки должны быть очищены дезинфицирующим средством для поверхностей и стерилизованы ультрафиолетом (УФ) до и после использования.
5. Отбор и диагностика полевых проб
ПРИМЕЧАНИЕ: Отбор проб должен производиться обученным и иммунизированным персоналом, носящим средства индивидуальной защиты и соблюдающим стандартные процедуры47,48,49.
- Собирают пробу через большое затылочное отверстие (т.е. затылочный путь), как подробно описано в Mauti et al.50.
- Диагностировать бешенство в полевых условиях с помощью диагностических экспресс-тестов и подтверждать его в лаборатории с помощью рекомендуемых процедур47, таких как прямой тест на флуоресцентные антитела (DFA), прямой иммуногистохимический тест (DRIT)51,52 или обратная транскрипция в реальном времени (RT)-PCR 53.
- Используйте подтвержденные положительные образцы мозга для экстракции РНК или храните в морозильной камере при температуре -20 °C в течение 2-3 месяцев или -80 °C в течение более длительных периодов времени. Сохраняйте РНК для хранения и транспортировки с помощью подходящей среды стабилизации ДНК/РНК.
6. Пробоподготовка и экстракция РНК (3 ч)
ПРИМЕЧАНИЕ: Используйте набор для экстракции вирусной РНК на основе спиновой колонки, подходящий для типа образца.
- Подготовьте две пробирки с керамическими шариками, заполнив пробирку для ПЦР объемом 2 мл примерно 200 мкл, наполненную керамическими шариками диаметром 1,4 мм, и промаркируйте пробирку.
- Добавьте рекомендуемый объем лизисного буфера, предусмотренный в наборе для экстракции РНК, в маркированную ПЦР-пробирку.
- Возьмите примерно 3-миллиметровый кубик из образца мозга с подтвержденной инфекцией бешенства с помощью деревянного аппликатора и поместите в маркированную пробирку с идентификатором образца и 100 мкл воды, не содержащей нуклеаз, в пробирку с маркировкой «отрицательный контроль».
ПРИМЕЧАНИЕ: Используйте гомогенизацию на основе гранул закрытых пробирок, чтобы ограничить воздействие на образец. Если это невозможно, используйте другие подходящие механические разрушители (например, на основе ротора) или ручной микропестик. Тем не менее, они могут быть менее эффективными, чем удары шариками по твердой поверхности для разрушения тканей (образцы тканей могут затвердевать в определенных средах хранения). - Повредите ткани мозга вручную с помощью деревянного аппликатора, а затем вихрь с максимальной скоростью, пока не будет достигнута полная гомогенизация тканей.
- Центрифугируйте лизат в соответствии с инструкциями производителя и используйте пипетку для переноса надосадочной жидкости в новую пробирку для микроцентрифуги с этикеткой. Используйте эту надосадочную жидкость только на последующих этапах.
- Следуйте инструкциям к спиновой колонке набора для экстракции РНК, чтобы получить очищенную РНК.
- Включите сюда отрицательный контроль экстракции (NEC) и перейдите к этапу секвенирования.
7. Подготовка кДНК (20 мин)
- В зоне мастер-смеси подготовьте мастер-смесь для синтеза кДНК первой цепи в соответствии с количеством образцов и контрольных образцов, подлежащих обработке (с превышением объема на 10% для обеспечения адекватного реагента; Таблица 1). На этом этапе должен быть включен элемент управления без шаблона (NTC).
- Маркируйте пробирки с ПЦР-стрипом объемом 0,2 мл и аликвоту 5 мкл мастер-смеси в пробирки.
- Отнесите подготовленные пробирки в область шаблона. Добавьте 5 мкл РНК в каждую меченую пробирку, включая NEC. Добавьте 5 мкл безнуклеазной воды (NFW) в NTC.
- Инкубируйте в амплификаторе в соответствии с условиями, указанными в таблице 1.
ПРИМЕЧАНИЕ: Дополнительная точка паузы: при необходимости кДНК можно хранить при -20 °C до 1 месяца, но предпочтительнее перейти к ПЦР.
8. Подготовка грунтовочного бассейна (1 ч)
ПРИМЕЧАНИЕ: Этот шаг необходим только при изготовлении новых запасов из отдельных грунтовок, после чего можно использовать предварительно приготовленные исходные растворы.
- Подготовьте грунтовочную ванну объемом 100 мкМ в зоне мастер-микса.
- Ресуспендировать лиофилизированные праймеры в 1x трис-ЭДТА (ТЭ) буфере или NFW в концентрации 100 мкМ каждый. Тщательно взбейте и закрутите вниз.
ПРИМЕЧАНИЕ: На следующих этапах отдельные праймеры разделяются на два пула праймеров - нечетный (называемый пулом А) и четный (называемый пулом Б) - чтобы избежать взаимодействия между праймерами, фланкированными ампликонными перекрытиями. Эти пулы праймеров генерируют перекрывающиеся ампликоны размером 400.о., охватывающие геном-мишень. - Разложите все нечетные капсюли в штативе для тюбиков. Создайте пул праймеров, добавив 5 мкл из каждого праймера в пробирку микроцентрифуги объемом 1,5 мл с маркировкой «Название схемы праймера — пул А (100 мкМ)».
- Повторите процесс для всех праймеров с четными номерами и пометьте их как "Название схемы праймера - Пул B (100 мкМ)".
- Разбавьте каждую пулу праймеров в соотношении 1:10 в молекулярной воде для получения 10 мкМ грунтовки.
ПРИМЕЧАНИЕ: Сделайте несколько аликвот 10 мкМ разбавлений праймера и заморозьте их в случае деградации или загрязнения.
9. Мультиплексная ПЦР (5 ч)
- Подготовьте две мастер-смеси ПЦР, по одной для каждого бассейна праймеров в зоне мастер-смеси.
- Используйте конечную концентрацию 0,015 мкМ на праймер. Рассчитайте необходимый объем пулы праймеров для реакции ПЦР (табл. 2) по следующей формуле:
Объем пулы праймеров = количество праймеров x объем реакции x 0,015/концентрация (мкМ) запаса праймера
- Используйте конечную концентрацию 0,015 мкМ на праймер. Рассчитайте необходимый объем пулы праймеров для реакции ПЦР (табл. 2) по следующей формуле:
- Аликвоту по 10 мкл мастер-смеси пула А и мастер-смеси пула Б в маркированные пробирки для ПЦР-полосок в области шаблона. Для каждого образца добавьте 2,5 мкл кДНК (начиная с шага 3) в каждую из соответствующих меченых реакций пула А и В. Избыток кДНК может храниться при температуре -20 °C.
- Перемешайте, осторожно взмахивая и пульсируя центрифугой.
- Инкубируют образцы в условиях, указанных в таблице 2 , на ПЦР-машине.
ПРИМЕЧАНИЕ: Программа не включает в себя специальную стадию удлинения из-за большого времени отжига 5 мин (требуется из-за большого количества праймеров) и короткой длины ампликонов (400 п.н.), которая достаточна для растяжения.
10. Очистка и количественное определение ПЦР (3,5 ч)
- С этого момента выполняйте все работы в зоне после ПЦР.
- Аликвотные твердофазные обратимые гранулы иммобилизации (SPRI) в микроцентрифужные пробирки из основного флакона. Хранить при температуре 4 °C.
- Нагрейте шарик SPRI аликвотой до комнатной температуры (RT; ~20 °C) и тщательно перемешайте до тех пор, пока шарики не будут полностью ресуспендированы в растворе.
- В пробирках объемом 1,5 мл смешайте праймер Pool A и праймер Pool B PCR для каждого образца. При необходимости долейте воды, чтобы довести объем до 25 мкл.
- Добавьте 25 мкл гранул SPRI в каждый образец (соотношение шарик к образцу 1:1). Перемешайте, пипетируя вверх и вниз или осторожно постукивая по тюбику.
- Инкубируйте при RT в течение 10 минут, время от времени переворачивая или переворачивая пробирки.
- Поместите на магнитную решетку до тех пор, пока шарики и раствор полностью не разделятся. Удалите и выбросьте надосадочную жидкость, стараясь не потревожить гранулу шарика.
- Дважды промыть 80% этиловым спиртом (подогретым до RT).
- Добавьте в гранулу 200 мкл этанола. Подождите 30 с, чтобы убедиться, что бусины вымыты должным образом.
- Осторожно выньте и выбросьте надосадочную жидкость, стараясь не задеть гранулу.
- Повторите шаги 10.8.1-10.8.2, чтобы промыть гранулы во второй раз.
- Удалите все следы этанола с помощью наконечника на 10 мкл. Сушить на воздухе до тех пор, пока следы этанола не испарятся (с мелкими шариками это происходит быстро, ~30 с); Когда это произойдет, гранула должна превратиться из блестящей в матовую. Следите за тем, чтобы не пересушить (если гранула трескается, она слишком сухая), так как это повлияет на восстановление ДНК.
- Повторно суспендируйте шарики в 15 мкл NFW и инкубируйте при RT (вне магнитной стойки) в течение 10 мин.
- Вернитесь на магнитную стойку и перелейте надосадочную жидкость (очищенный продукт) в свежую пробирку объемом 1,5 мл.
- Приготовьте разведение 1:10 каждого образца в отдельной пробирке (2 мкл продукта + 18 мкл NFW).
ПРИМЕЧАНИЕ: Будьте очень осторожны на этом этапе, чтобы избежать перекрестного загрязнения. В каждый момент времени должна быть открыта только одна ампликонная трубка. Сначала в пробирки наливают 18 мкл воды (в чистую зону мастер-смеси). - Измерьте концентрацию ДНК в каждом разбавленном образце с помощью высокочувствительного и специфического флуориметра, как описано в protocols.io54,55.
11. Нормализация (30 мин)
- Используйте шаблон нормализации (Дополнительный файл 3) и концентрацию ДНК (нг/мкл) каждого образца для расчета объема разбавленного (или чистого) образца, необходимого для 200 фмоль каждого образца в общем объеме 5 мкл.
- Пометьте новые пробирки для ПЦР и добавьте рассчитанные объемы NFW и образца для получения нормализованной ДНК.
- Используйте расчетный объем для неразбавленных (чистых) образцов, если для получения 200 фмоль требуется более 5 мкл разбавленного образца.
ПРИМЕЧАНИЕ: Дополнительная точка паузы: На этом этапе очищенный продукт ПЦР можно хранить при 4 °C до 1 недели или поместить при -20 °C для более длительного хранения, если это необходимо.
12. Конечная подготовка и штрихкодирование (1,5 часа)
ПРИМЕЧАНИЕ: Следующие шаги предполагают использование специфических реагентов из наборов для штрих-кодирования и лигирования, специфичных для нанопор (см. Таблицу материалов для получения подробной информации). Протокол может быть перенесен между различными версиями химического состава, но пользователи должны позаботиться об использовании совместимых наборов в соответствии с инструкциями производителя.
- Торцевой ремонт и dA-tail
- Настройте конечную реакцию подготовки для каждого образца, упомянутого в таблице 3. Приготовьте мастер-микс в соответствии с количеством образцов (плюс 10% избытка). Будьте осторожны при пипетировании, так как реагенты вязкие.
- Добавьте 5 мкл мастер-смеси в каждую пробирку нормализованной ДНК (5 мкл). Общий объем реакционной смеси должен составлять 10 мкл. Меняйте наконечники каждый раз и открывайте только одну пробирку за раз.
- Инкубируют в термоциклере в условиях, указанных в таблице 3.
- Штрих-кодирование
- Аликвотируйте штрихкоды из набора штрих-кодирования в пробирки для ПЦР по 1,25 мкл/пробирку и записывайте штрих-код, присвоенный каждому образцу.
- Добавьте 0,75 мкл готового образца к назначенной ему аликвоте штрих-кода.
- Приготовьте мастер-смесь для лигирования в соответствии с количеством образцов (плюс 10% избытка) (табл. 4).
- Добавьте 8 мкл мастер-смеси для лигирования к готовому образцу + штрих-кодам, что даст общую реакцию 10 мкл.
- Инкубируют в амплификаторе в условиях, указанных в таблице 4.
- Очистка шариков SPRI и количественное определение ДНК
- Разморозьте буфер коротких фрагментов (SFB) на RT, перемешайте с помощью вихревой, импульсной центрифуги и поместите на лед.
- Объедините все образцы со штрих-кодом в микроцентрифужную пробирку объемом 1,5 мл. Чтобы не сделать объем очистки слишком большим для использования, объединяйте 12–24 образца (10 мкл/образец), до 48 образцов (5 мкл/образец) или до 96 образцов (2,5 мкл/образец) из каждой нативной реакции штрихкодирования.
- Добавьте 0,4-кратный объем шариков SPRI в пул со штрих-кодом. Осторожно перемешайте (перемешивая или пипетику) и инкубируйте при RT в течение 5 минут.
- Поместите образцы на магнит до тех пор, пока шарики не гранулируются и надосадочная жидкость не станет полностью прозрачной (~2 мин). Выньте и выбросьте надосадочную жидкость. Следите за тем, чтобы не потревожить бусины.
- Дважды промойте 250 мкл SFB.
- Снимите трубку с магнита и полностью ресуспендируйте гранулу в 250 мкл SFB. Инкубируйте в течение 30 с, проведите импульсную центрифугу и верните к магниту.
- Удалите надосадочную жидкость и выбросьте.
- Повторите шаг 12.3.5, чтобы выполнить вторую стирку SFB.
- Импульсная центрифуга и удаление остатков SFB.
- Добавьте 200 мкл 80% (RT) этанола, чтобы промыть гранулу. Удалите и выбросьте этанол, стараясь не повредить гранулу. Сушить на воздухе в течение 30 с или до тех пор, пока гранула не потеряет свой блеск.
- Ресуспендировать в 22 мкл NFW при RT в течение 10 мин.
- Поместите на магнит, оставьте на ~2 минуты, затем осторожно удалите раствор и переложите в чистую пробирку для микроцентрифуг объемом 1,5 мл.
- Используйте 1 мкл для получения концентрации ДНК, как описано ранее (шаг 10.13).
ПРИМЕЧАНИЕ: Дополнительная точка паузы: На этом этапе библиотеку можно хранить при температуре 4 °C до 1 недели или -20 °C для более длительного хранения, но предпочтительнее продолжить лигирование адаптера и секвенирование.
13. Секвенирование (максимум 48 часов)
- Подготовьте компьютер (см. также разделы 1-4 «Предварительные требования»).
- Убедитесь, что достаточно места для хранения новых данных (минимум 150 ГБ), данные из старых запусков резервированы/перемещены на сервер перед удалением, а также установлена последняя версия MinKNOW.
- Извлеките хранящуюся проточную ячейку из холодильника и дайте ей достичь RT.
- Перевязка адаптера (1 ч)
- Импульсной центрифугой адаптер перемешать и лигазировать и поместить на лед.
- Размораживающий элюирующий буфер (EB), SFB и лигирующий буфер на RT. Смешайте с помощью вихревой, импульсной центрифуги и поместите на лед.
- Приготовьте мастер-смесь для перевязки адаптера (табл. 5), комбинируя реагенты в указанном порядке в пробирке с низким связыванием.
ПРИМЕЧАНИЕ: В зависимости от наличия в лаборатории можно использовать альтернативные реагенты для перевязки (Таблица 5). Список альтернатив см. в Дополнительном файле 3 и Таблице материалов . Используйте вычисления на листе Supplementary File 3 , чтобы получить объем библиотеки ДНК, эквивалентный 200 фмоль. Если вычисляется менее 20 мкл, добавьте NFW, чтобы получить до 20 мкл. - Перемешайте легким щелчком и импульсной центрифугой. Инкубируют при RT в течение 20 мин.
ПРИМЕЧАНИЕ: Во время инкубации приступайте к подготовке проточной ячейки (раздел 13.5).
- Очистите с помощью гранул SPRI (не используйте этанол, как в предыдущих чистках).
- Добавьте к образцам 0,4-кратный объем гранул SPRI (RT). Инкубируйте при RT в течение 10 минут, осторожно периодически помешивайте, чтобы облегчить перемешивание.
- Поместите на магнит до тех пор, пока шарики и раствор полностью не разделятся (~5 мин). Удалите и выбросьте надосадочную жидкость; Будьте осторожны, чтобы не потревожить гранулу бисера.
- Дважды промойте 125 мкл SFB.
- Полностью ресуспендируйте гранулу 125 мкл SFB, перемешав с помощью пипетки. Оставить инкубироваться на 30 с.
- Импульсной центрифугой соберите жидкость у основания пробирки и поместите на магнит. Удалите надосадочную жидкость и выбросьте.
- Повторите шаги 13.4.4-13.4.5, чтобы промыть гранулы во второй раз.
- Проведите центрифугу в импульсе и удалите излишки SFB.
- Ресуспендировать в 15 мкл БЭ и инкубировать в течение 10 мин при РТ.
- Вернитесь к магниту на ~2 мин, а затем осторожно перелейте раствор в чистую пробирку для микроцентрифуги объемом 1,5 мл.
- Количественно определите 1 мкл элюированной библиотеки, как описано ранее на шаге 10.13
ПРИМЕЧАНИЕ: Для достижения наилучших результатов переходите непосредственно к секвенированию MinION; однако, при необходимости, окончательная библиотека может храниться в EB при температуре 4 °C до 1 недели.
- Выполните проточную проверку качества.
- Подключите устройство виртуализации к ноутбуку и откройте программное обеспечение для виртуализации.
- Выберите тип проточной ячейки, а затем нажмите кнопку Проверить проточную ячейку и начать тестирование.
- После завершения отобразится общее количество активных (т.е. жизнеспособных) пор. Новая проточная ячейка должна иметь >800 активных пор; Если это не так, обратитесь к производителю за заменой.
- Заливка и загрузка проточной ячейки (20 мин)
- Разморозьте следующие реагенты на RT, а затем поместите буфер секвенирования, промывочный трос, промывочный буфер и загрузочные шарики на лед.
- Встряхните буфер секвенирования и буфер для промывки, импульсную центрифугу и поместите на лед.
- Импульсная центрифуга промывочного троса и смешивание с помощью пипетирования; Затем выложите на лед.
- Приготовьте смесь для заливки проточной ячейки, добавив 30 мкл промывочного троса непосредственно в пробирку промывочного буфера из комплекта для заливки проточной ячейки, и перемешайте пипетированием.
- Смешивайте загрузочные гранулы пипеткой непосредственно перед использованием, так как они быстро оседают.
- В свежей пробирке готовят окончательное библиотечное разведение для секвенирования, как указано в таблице 5.
ПРИМЕЧАНИЕ: Используйте вычисления на листе Supplementary File 3 , чтобы получить объем библиотеки ДНК, эквивалентный 50 фмоль. Если вычисляется менее 12 мкл, добавьте EB, чтобы получить до 12 мкл. - Откиньте крышку устройства последовательности и сдвиньте крышку отверстия для заливки по часовой стрелке так, чтобы отверстие для заливки было видно (Рисунок 3)
- Осторожно удалите пузырьки воздуха, установив пипетку P1000 на 200 мкл, вставьте наконечник в отверстие для заливки и поворачивайте колесико до тех пор, пока не станет виден небольшой объем, входящий в наконечник пипетки (максимальный оборот до 230 мкл).
- Загрузите 800 мкл смеси для заливки в проточную ячейку через отверстие для заливки, стараясь избежать образования пузырьков.
- Оставьте на 5 минут.
- Осторожно поднимите крышку отверстия для образца и загрузите 200 мкл праймерной смеси в проточную ячейку через заливочное отверстие с помощью пипетки P1000.
- Перед загрузкой пипетки перемешайте библиотеку вверх и вниз, убедившись, что загрузочные шарики в основной смеси снова взвешены перед загрузкой.
- Загрузите 75 мкл библиотечной смеси в проточную ячейку через отверстие для образца по каплям. Убедитесь, что каждая капля стекает в порт, прежде чем добавлять следующую.
- Осторожно установите на место крышку отверстия для отбора проб, убедившись, что пробка входит в отверстие для отбора проб.
- Закройте отверстие для заливки и установите на место крышку устройства секвенирования.
- Секвенирование (максимум 48 часов)
- Подключите устройство секвенирования к ноутбуку и откройте программное обеспечение для секвенирования.
- Нажмите кнопку Пуск , а затем нажмите кнопку Начать виртуализацию.
- Нажмите кнопку Создать эксперимент и следуйте рабочему процессу программного обеспечения виртуализации с графическим интерфейсом пользователя (GUI), чтобы настроить параметры выполнения.
- Введите название эксперимента и идентификатор образца (например, rabv_run1) и выберите Тип ячейки потока в раскрывающемся меню.
- Перейдите к выбору набора и выберите соответствующий набор для секвенирования лигирования и используемые наборы для штрихкодирования.
- Перейдите к разделу Параметры выполнения . Сохраняйте значения по умолчанию, если только не требуется, чтобы запуск автоматически останавливался через определенное количество часов (запуски можно остановить вручную в любое время).
- Перейдите к разделу Basecalling. Выберите, включить или выключить Basecalling в зависимости от вычислительных ресурсов (см. Настройка компьютера). Выберите «Параметры редактирования» в разделе «Штрихкодирование» и убедитесь, что параметр «Штрихкод с обоих концов» включен. Сохраните и перейдите к разделу вывода.
- Примите значения по умолчанию и перейдите к окончательной проверке, проверьте настройки и запишите сведения в рабочий лист (дополнительный файл 3). Нажмите кнопку Начать.
ПРИМЕЧАНИЕ: Если проточная ячейка используется повторно, отрегулируйте пусковое напряжение (в расширенном разделе параметров запуска), как показано на схеме в Дополнительном файле 3. - Запишите начальные активные каналы — если он значительно ниже, чем проверка контроля качества (QC), перезапустите программное обеспечение для секвенирования. Если все еще ниже, то перезагрузите компьютер.
- Запишите начальные каналы в виде нити и одной поры, чтобы получить приблизительную занятость пор. Это число будет колебаться, поэтому дайте приблизительную оценку.
- Следите за ходом выполнения.
14. Бейсколлинг в прямом эфире и оффлайн
ПРИМЕЧАНИЕ: В этих инструкциях предполагается, что ранее существовавшая структура каталогов, предоставленная в репозитории artic-rabv, и что были соблюдены разделы 1 и 3 протокола.
- В локальной файловой системе создайте новый каталог с именем analysis, в котором будут храниться все результаты анализа. Для дальнейшей организации: создайте подкаталог с именем вашего проекта, а внутри него новый каталог для запуска, используя идентификатор примера, предоставленный MinKNOW, в качестве run_name. Сделайте это одной командой следующим образом:
mkdir -p
анализ/project_name/run_name
Затем перейдите к его местоположению:
компакт-диск
путь/анализ/project_name/run_name - Бейсколлинг в реальном времени
ПРИМЕЧАНИЕ: Для выполнения вызова нанопор в режиме реального времени ноутбукам требуется графический процессор (GPU), совместимый с NVIDIA CUDA. Убедитесь, что инструкции по настройке базового вызова GPU были выполнены с использованием протокола guppy56.- Во время настройки запуска включите бейсколлинг в реальном времени.
- Используйте RAMPART для мониторинга покрытия последовательности в режиме реального времени, в соответствии с приведенной ниже инструкцией.
- В терминале компьютера активируйте окружение artic-rabv conda:
conda активировать Artic-RABV - Создайте новый каталог для вывода rampart внутри каталога run_name и перейдите в него:
cd /путь/анализ/project_name/run_name
mkdir rampart_output
Компакт-диск rampart_output - Создайте файл штрих-кодов .csv для сопоставления штрихкодов и имен образцов. Он должен иметь одну строку на штрих-код и указывать только те штрих-коды, которые присутствуют в библиотеке, с заголовками «штрих-код» и «образец». Следуйте примеру в каталоге artic-rabv:
анализ/example_project/example_run/rampart_output/штрих-коды.csv - Запустите RAMPART, указав соответствующую папку протокола и путь к папке fastq_pass в выходных данных MinKNOW для выполнения:
rampart --protocol /путь/rampart/scheme_name_V1_protocol - basecalledPath - Откройте окно браузера и перейдите к localhost:3000 в поле URL-адреса. Подождите, пока не будет вызвано достаточное количество данных, прежде чем результаты появятся на экране.
- Автономный вызов базы (выполняется после выполнения)
- Если живой бейсколлинг не был задан, выходными данными MinKNOW будут необработанные сигнальные данные (файлы fast5). Вы не сможете использовать RAMPART во время бега. Преобразуйте файлы fast5 в basecalled data (файлы fastq) после запуска с помощью guppy (см. настройку на шаге 1.1.1.). Запустите RAMPART post-hoc для basecalled data.
- Запустите baspy basecaller:
guppy_basecaller -c dna_r9.4.1_450bps_fast.cfg -i /путь/к/чтению/fast5_* -s /путь/анализ/project_name/run_name -x auto -r
-c - это конфигурационный файл для указания модели basecalling, -i - входной путь, -s - путь сохранения, -x указывает basecalling устройством GPU (исключить при использовании компьютерной версии guppy), а -r указывает на рекурсивный поиск входных файлов.
ПРИМЕЧАНИЕ: Конфигурационный файл (.cfg) можно изменить на высокоточный basecaller, заменив _fast на _hac, хотя это займет значительно больше времени.
15. Промывка проточных ячеек
- Проточные ячейки могут быть промыты и повторно использованы для секвенирования новых библиотек, если поры все еще жизнеспособны. Инструкции по промывке см. в протоколе проточной промывки ячейки ONT57.
16. Анализ и интерпретация
- Генерация согласованной последовательности с помощью конвейера биоинформатики ARTIC
- Следуйте инструкциям, приведенным в репозитории40 GitHub artic-rabv в папке rabv_protocols, чтобы создать последовательности консенсуса из необработанных файлов fast5 или basecalled fastq.
ПРИМЕЧАНИЕ: Для получения дальнейших указаний обратитесь к документу «Арктический трубопровод - основной трубопровод58 ».
- Следуйте инструкциям, приведенным в репозитории40 GitHub artic-rabv в папке rabv_protocols, чтобы создать последовательности консенсуса из необработанных файлов fast5 или basecalled fastq.
- Необязательно: анализ средней глубины считывания на ампликон.
- Адаптируйте скрипты, доступные в репозитории artic-rabv, ссылаясь на Дополнительный файл 1. Вкратце, углубленная статистика генерируется с помощью SAMtools59 и покрытия на нуклеотид на графике R.
- Филогенетический анализ с помощью GLUE
- В RABV_GLUE42 выберите вкладку «Анализ > генотипирование и интерпретация » > «Добавить файлы», выбрав fasta-файл консенсусных последовательностей.
- Нажмите «Отправить» и подождите. После завершения анализа будет доступна кнопка Показать анализ, показывающая присвоение клад и субкладов, покрытие по генам, вариации от референсных последовательностей и ближайшего родственника.
- Соответствующие контекстуальные последовательности также могут быть идентифицированы в разделе Данные последовательностей > NCBI Sequences by Clade .
- Выберите идентифицированную кладу или нажмите Вирус бешенства (RABV), чтобы просмотреть все доступные последовательности.
- Фильтрация по релевантным последовательностям (например, по стране происхождения).
- Загрузите эти последовательности и соответствующие метаданные для анализа и сравнения.
- Присвоение родословной с помощью MADDOG41
- Извлеките репозиторий MADDOG из GitHub, чтобы убедиться, что вы работаете с самой последней версией.
- Создайте папку назначения в локальном репозитории MADDOG (ранее созданном в разделе Предварительные требования) с именем запуска.
- Внутри папки добавьте файл fasta, содержащий последовательности консенсуса.
- Добавьте файл метаданных в папку.
ПРИМЕЧАНИЕ: Этот файл должен быть CSV-файлом с 4 столбцами «ID», «страна», «год» и «назначение», в которых подробно указаны идентификаторы последовательностей, страна выборки и год сбора образцов, а столбец «назначение» должен быть пустым. - В интерфейсе командной строки активируйте окружение conda: conda активирует MADDOG.
- В интерфейсе командной строки перейдите в папку репозитория MADDOG.
- Первоначально выполните присвоение родословной последовательностям, чтобы проверить наличие любых потенциальных аномалий и определить, будет ли целесообразно выполнить более длинный шаг обозначения родословной. Для этого введите в командной строке следующее: sh assignment.sh.
- При появлении запроса введите Y, чтобы указать, что вы извлекли репозиторий и работаете с самой последней версией MADDOG.
- При появлении запроса введите имя папки в папке репозитория MADDOG, содержащей файл fasta.
- Когда присвоение родословной будет завершено, проверьте выходной файл в папке. Если выходные данные соответствуют ожидаемым и одному и тому же происхождению назначено несколько последовательностей, выполните обозначение происхождения.
- Если выполняется обозначение происхождения, удалите только что созданный выходной файл назначения.
- В терминале, внутри папки репозитория MADDOG, выполните команду sh designation.sh.
- При появлении запроса введите Y, чтобы указать, что вы извлекли репозиторий и работаете с самой последней версией MADDOG.
- При появлении запроса введите имя папки в папке репозитория MADDOG, содержащей файл fasta и метаданные. В результате выводится информация о происхождении каждой последовательности, филогения новых и соответствующих предыдущих последовательностей (начиная с 16.3.6), иерархическая информация о линиях, а также сведения о потенциально возникающих линиях и областях недостаточной выборки.
ПРИМЕЧАНИЕ: Полную информацию о протоколе, использовании и выходных данных можно найти в Campbell et al.60. - После завершения первоначального анализа, если вам будет предложено также проверить наличие новых и недостаточных линий, введите Y, если это необходимо. В противном случае введите N.
- Если вам будет предложено подтвердить вновь найденные родословные, введите Y и следуйте инструкциям в результирующем файле NEXT_STEPS.eml. В противном случае введите N.
Representative Results
Описанный в этом протоколе рабочий процесс «образец-последовательность-интерпретация» для RABV успешно используется в различных лабораторных условиях в эндемичных странах, таких как Танзания, Кения, Нигерия и Филиппины (рисунок 4). Протокол был использован на различных типах образцов и условиях (табл. 6): свежая и замороженная ткань мозга, экстракты кДНК и РНК из тканей мозга, транспортируемых в холодовой цепи в течение длительного времени, а также карты FTA с мазками из мозговой ткани.
Вызов базы в реальном времени с использованием RAMPART (рис. 5) показывает генерацию операций чтения практически в реальном времени и процентное покрытие каждой выборки. Это особенно полезно при принятии решения о том, когда следует остановить прогон и сохранить проточную ячейку для повторного использования. Наблюдались различия во времени работы: некоторые из них были завершены за 2 часа, в то время как другие могли занять более 12 часов для достижения достаточной глубины покрытия (x100). Мы также можем просматривать области с плохим усилением; Например, на рисунке 6 показан снимок одного из этапов секвенирования, где профили покрытия показывают некоторые ампликоны с очень низким усилением, что указывает на потенциально проблемные праймеры. Благодаря более тщательному исследованию этих слабо усиливающихся областей мы смогли выявить несоответствия праймеров, что позволит нам перепроектировать и улучшить отдельные праймеры. Некоторые схемы праймеров показали больше несоответствий, чем другие. По сравнению с Филиппинами, в Восточной Африке это проявляется в соответствии с целевым разнообразием, поскольку программа для Восточной Африки направлена на охват гораздо более широкого разнообразия.
Для компиляции и интерпретации результирующих последовательностей RABV использовались RABV-GLUE42, ресурс общего назначения для управления данными генома RABV, и MADDOG60, система классификации и номенклатуры линий. В таблице 7 показаны основные и второстепенные клады, циркулирующие в каждой стране, классифицированные с помощью RABV-GLUE. Также показана классификация локальных линий с более высоким разрешением после присвоения MADDOG.
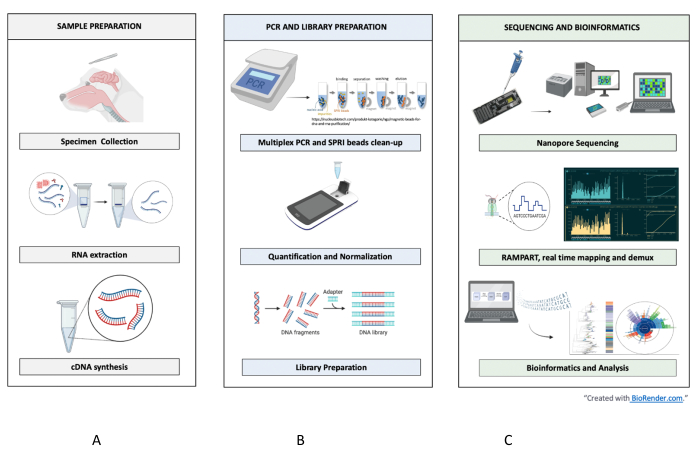
Рисунок 1: Рабочий процесс от образца до последовательности и интерпретации для RABV. Показаны краткие этапы для (А) пробоподготовки, (Б) ПЦР и подготовки библиотеки, и (В) секвенирования и биоинформатики вплоть до анализа и интерпретации. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
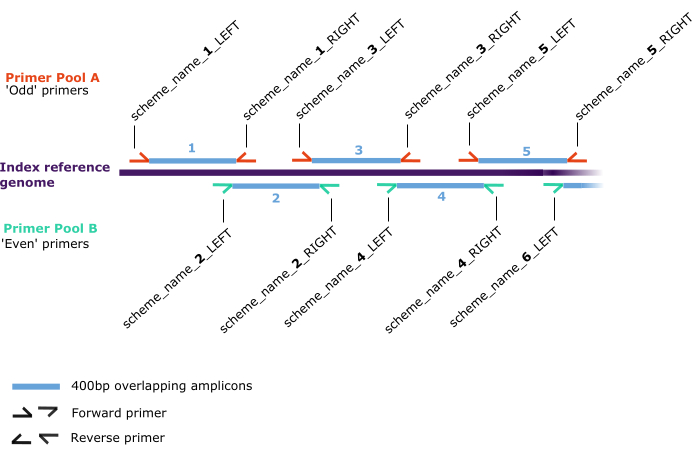
Рисунок 2: Схема праймера. Позиции отжига вдоль «индексного референсного генома» (темно-фиолетовый) для пар прямых и обратных праймеров (полустрелки), которые распределены по двум отдельным пулам: A (красный) и B (зеленый). Пары праймеров генерируют 400 перекрывающихся ампликонов (синий), которые последовательно нумеруются вдоль индексного эталонного генома в формате 'scheme_name_X_DIRECTION', где 'X' - число, относящееся к ампликону, генерируемому праймером, а 'DIRECTION' - это либо 'ВЛЕВО', либо 'ВПРАВО', описывающее прямое или обратное соответственно. Четные или нечетные значения 'X' определяют пул (A или B соответственно). Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
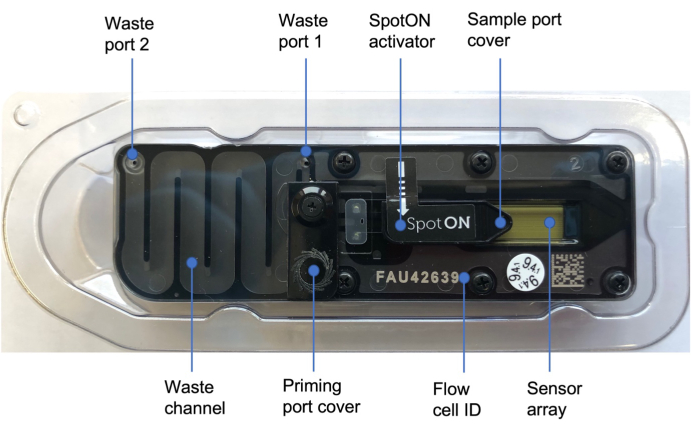
Рисунок 3: Ячейка потока нанопор48. Синие этикетки иллюстрируют различные части проточной ячейки, в том числе крышку заливочного отверстия, которая закрывает заливочное отверстие, куда добавляется заливочный раствор, крышку пробоотборного отверстия SpotON, закрывающую отверстие для отбора проб, куда образец добавляется по каплям, отверстия для отходов 1 и 2, а также идентификатор проточной ячейки. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
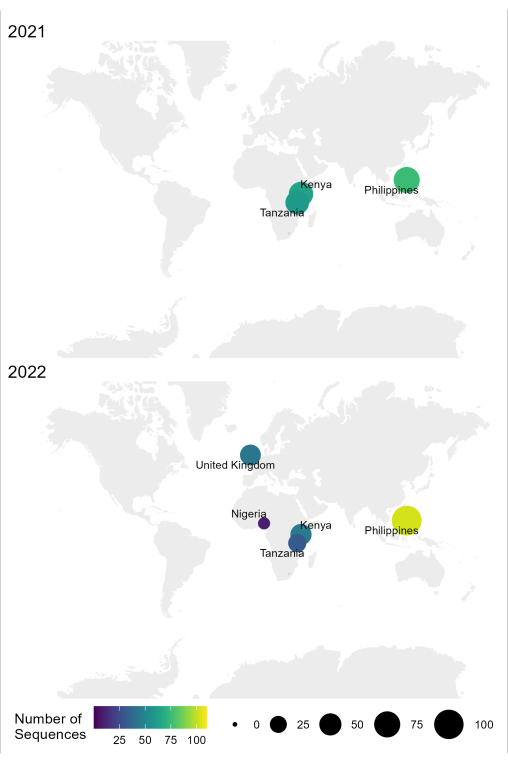
Рисунок 4: Карта, показывающая место, где секвенирование RABV проводилось с использованием оптимизированного рабочего процесса в 2021 и 2022 годах. Размер и цвет пузырьков соответствуют количеству последовательностей в одном местоположении, где чем меньше и темнее, тем меньше, а больше и светлее — больше. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
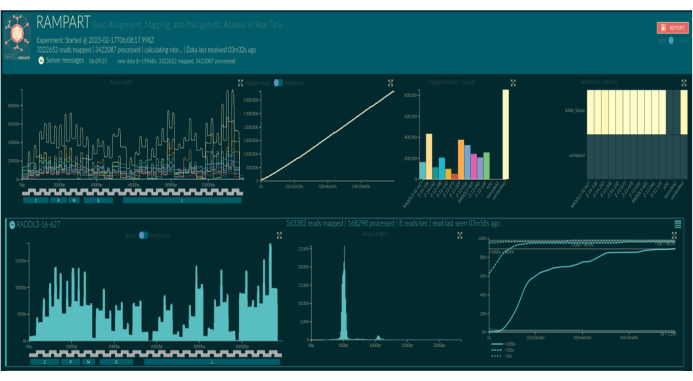
Рисунок 5: Скриншот визуализации RAMPART в веб-браузере. Названия штрих-кодов заменяются названиями образцов в соответствии с биоинформатической установкой. На трех верхних панелях показаны сводные графики за весь прогон: глубина покрытия картированных считываний для каждого штрих-кода на нуклеотидную позицию в индексном эталонном геноме (вверху слева, окрашена штрих-кодом), суммированные сопоставленные считывания всех штрих-кодов с течением времени (вверху посередине) и сопоставленные чтения по штрих-коду (вверху справа, окрашены штрих-кодом). На нижних панелях отображаются ряды графиков для каждого штрих-кода. Слева направо: глубина охвата картированных прочтений на нуклеотидную позицию индексного референсного генома (слева), распределение длины картированных прочтений (посередине) и доля нуклеотидных позиций в индексном референсном геноме, которые получили 10-кратное, 100-кратное и 1000-кратное покрытие картированных прочтений с течением времени (справа). Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
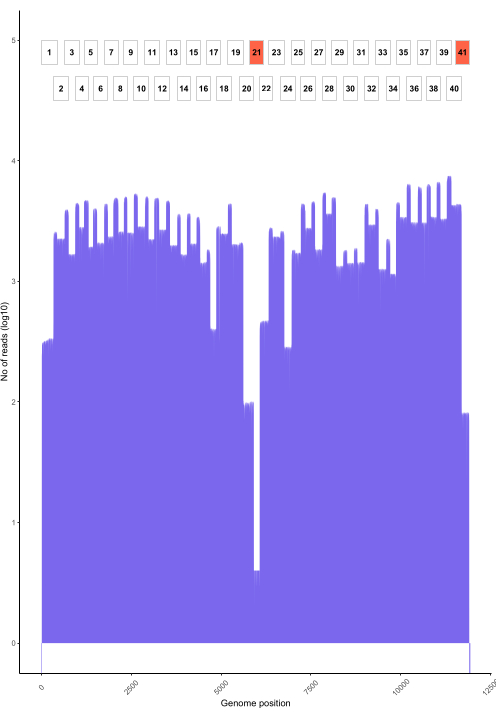
Рисунок 6: Пример считывания по всему геному образца вируса бешенства с Филиппин, секвенированного с использованием протокола. Показано покрытие считывания в каждой нуклеотидной позиции в геноме, а также положение перекрывающихся ампликонов (1-41), используемых для генерации библиотеки. Пики глубины покрытия соответствуют областям перекрытия ампликонов. Ампликоны с малой глубиной покрытия соответствуют областям перекрытия ампликонов. Ампликоны с низкой глубиной покрытия выделены красным цветом с указанием проблемных участков, которые могут потребовать оптимизации. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
Таблица 1: Условия получения кДНК и мастер-смесителя и амплификатора. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Таблица 2: Условия работы мастер-смеси и амплификатора для мультиплексной ПЦР. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Таблица 3: Условия работы мастер-смеси и термоциклера для реакции окончательной подготовки. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Таблица 4: Условия мастер-смеси и термоамплификатора для штрихкодирования. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Таблица 5: Мастер-микс для лигирования адаптера и окончательное библиотечное разведение для секвенирования. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Таблица 6: Количество сгенерированных полногеномных последовательностей вируса бешенства и типы образцов, используемых в разных странах с использованием рабочего процесса «образец-последовательность-интерпретация». Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Таблица 7: Назначения основных и второстепенных клад из RABV-GLUE и присвоения родословных из MADDOG для последовательностей, сгенерированных с помощью рабочего процесса. Пожалуйста, нажмите здесь, чтобы скачать эту таблицу.
Дополнительный файл 1: Разработка и оптимизация схемы праймера, а также анализ глубины считывания ампликонов. Пожалуйста, нажмите здесь, чтобы скачать этот файл.
Дополнительный файл 2: Настройка вычислений Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 3: Рабочий лист протокола RABV WGS Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Discussion
Доступный рабочий процесс полногеномного секвенирования RABV на основе нанопор был разработан Brunker et al.61 с использованием ресурсов сети ARTIC46. Здесь мы представляем обновленный рабочий процесс с полными этапами от образца до последовательности и интерпретации. Рабочий процесс подробно описывает подготовку образцов тканей мозга для полногеномного секвенирования, представляет конвейер биоинформатики для обработки считывания и генерации согласованных последовательностей, а также выделяет два специфичных для бешенства инструмента для автоматизации присвоения родословной и определения филогенетического контекста. Обновленный рабочий процесс также содержит подробные инструкции по настройке соответствующих вычислительных и лабораторных рабочих областей с рекомендациями по реализации в различных контекстах (включая параметры с ограниченными ресурсами). Мы продемонстрировали успешное внедрение рабочего процесса как в академических, так и в научно-исследовательских институтах в четырех эндемичных по СНСД СНСД, не имеющих или ограниченных возможностей геномного надзора. Рабочий процесс доказал свою устойчивость к применению в различных условиях и понятен пользователям с разным опытом.
Этот рабочий процесс для секвенирования RABV является наиболее полным общедоступным протоколом (охватывающим этапы от образца до последовательности и интерпретации) и специально адаптирован для снижения как начальных, так и эксплуатационных расходов. Время и затраты, необходимые для подготовки библиотеки и секвенирования с помощью технологии нанопор, значительно сокращаются по сравнению с другими платформами, такими как Illumina61, а непрерывные технологические разработки улучшают качество и точность последовательностей, чтобы быть сопоставимыми с Illumina62.
Этот протокол разработан для обеспечения устойчивости в различных условиях с ограниченными ресурсами. Обращаясь к рекомендациям по устранению неполадок и изменениям, предоставляемым вместе с основным протоколом, пользователи получают поддержку в адаптации рабочего процесса к своим потребностям. Добавление удобных для пользователя биоинформатических инструментов к рабочему процессу представляет собой серьезное развитие оригинального протокола, обеспечивающее быстрые и стандартизированные методы, которые могут применяться пользователями с минимальным предварительным опытом работы в области биоинформатики для интерпретации данных последовательностей в местных условиях. Возможности для выполнения этой задачи in situ часто ограничены необходимостью обладать специфическими навыками программирования и филогенетики, которые требуют интенсивных и долгосрочных инвестиций в обучение навыкам. Несмотря на то, что этот набор навыков важен для тщательной интерпретации данных последовательностей, базовые и доступные инструменты интерпретации не менее желательны для того, чтобы дать возможность местным «чемпионам по секвенированию», чей основной опыт может быть основан на мокрой лаборатории, что позволяет им интерпретировать свои данные и брать на себя ответственность за них.
Поскольку этот протокол применяется в течение нескольких лет в нескольких странах, теперь мы можем предоставить рекомендации о том, как оптимизировать схемы мультиплексных праймеров для улучшения охвата и борьбы с накопившимся разнообразием. Кроме того, были предприняты усилия для того, чтобы помочь пользователям повысить экономическую эффективность или обеспечить простоту закупок в том или ином регионе, что, как правило, является проблемой для устойчивости молекулярных подходов63. Например, в Африке (Танзания, Кения и Нигерия) мы выбрали мастер-микс тупой/ТА-лигазы на этапе переходного лигирования, который был более доступен у местных поставщиков и был более дешевой альтернативой другим реагентам для лигирования.
Как показывает опыт, существует несколько способов снижения затрат на образец и прогон. Уменьшение количества проб за один проход (например, с 24 до 12 образцов) может продлить срок службы проточных ячеек при нескольких прогонах, в то время как увеличение количества проб за один прогон позволяет максимально увеличить время и количество реагентов. В наших руках мы смогли промыть и повторно использовать проточные ячейки для каждого третьего цикла секвенирования, что позволило секвенировать еще 55 образцов. Промывка проточной ячейки сразу после использования или, если это невозможно, удаление отработанной жидкости из сливного канала после каждого запуска, по-видимому, сохраняло количество пор, доступных для второго запуска. Принимая во внимание начальное количество пор, доступных в проточной ячейке, один прогон также может быть оптимизирован для планирования количества образцов для обработки в конкретной проточной ячейке.
Несмотря на то, что рабочий процесс должен быть как можно более всеобъемлющим, с добавлением подробных инструкций и обозначенных ресурсов, процедура по-прежнему сложна и может быть пугающей для нового пользователя. Пользователю рекомендуется обратиться за личным обучением и поддержкой, в идеале на месте или, в качестве альтернативы, через внешних сотрудников. Например, на Филиппинах в рамках проекта по наращиванию потенциала региональных лабораторий по геномному надзору за SARS-CoV-2 с использованием ONT были разработаны основные компетенции медицинских диагностов, которые могут быть легко перенесены на секвенирование RABV. Важные этапы, такие как очистка шариков SPRI, могут быть трудными для освоения без практического обучения, а неэффективная очистка может повредить проточную ячейку и поставить под угрозу работу. Загрязнение образца всегда является серьезной проблемой при обработке ампликонов в лаборатории, и его может быть трудно устранить. В частности, перекрестное загрязнение между образцами чрезвычайно трудно обнаружить во время биоинформатики после прогона. Надлежащие лабораторные методы и практики, такие как поддержание чистоты рабочих поверхностей, разделение зон до и после ПЦР и включение негативного контроля, являются обязательными для обеспечения контроля качества. Быстрый темп развития секвенирования нанопор является как преимуществом, так и недостатком рутинного геномного надзора за RABV. Постоянное совершенствование точности, доступности и протокольного репертуара нанопор расширяет и улучшает область его применения. Однако те же самые разработки затрудняют поддержание стандартных операционных процедур и биоинформатических конвейеров. В этом протоколе мы предоставляем документ, помогающий перейти от старых к современным наборам для подготовки библиотек нанопор (Таблица материалов).
Распространенным препятствием на пути к секвенированию в СНСД является доступность, включающая не только стоимость, но и возможность своевременной закупки расходных материалов (в частности, реагентов для секвенирования, которые являются относительно новыми для отделов закупок и поставщиков) и вычислительных ресурсов, а также просто наличие доступа к стабильному электроснабжению и Интернету. Использование портативной технологии секвенирования нанопор в качестве основы этого рабочего процесса помогает решить многие из этих проблем доступности, и мы продемонстрировали использование нашего протокола в различных условиях, проведя полный протокол и анализ в стране. По общему признанию, своевременная закупка оборудования и расходных материалов для секвенирования остается проблемой, и во многих случаях мы были вынуждены перевозить или отправлять реагенты из Великобритании. Тем не менее, в некоторых районах мы смогли полностью полагаться на местные маршруты поставок реагентов, благодаря инвестициям в секвенирование SARS-CoV-2 (например, на Филиппинах), которые оптимизировали процессы закупок и начали нормализовать применение геномики патогенов.
Потребность в стабильном интернет-соединении сводится к минимуму за счет одноразовых установок; Например, репозитории GitHub, загрузка программного обеспечения и секвенирование нанопор требуют только доступа к Интернету для запуска запуска (не на всем протяжении) или могут быть выполнены полностью в автономном режиме по согласованию с компанией. При наличии мобильных данных телефон можно использовать в качестве точки доступа к ноутбуку, чтобы начать выполнение последовательности, а затем отключиться на время выполнения. При регулярной обработке образцов требования к хранению данных могут быстро расти, и в идеале данные должны храниться на сервере. В противном случае жесткие диски твердотельных накопителей (SSD) относительно дешевы.
Несмотря на то, что мы признаем, что в странах с низким и средним уровнем дохода по-прежнему существуют препятствия для геномного надзора, увеличение инвестиций в повышение доступности и экспертных знаний в области геномики (например, Африканская инициатива по геномике патогенов [Africa PGI])64 свидетельствует о том, что эта ситуация будет улучшаться. Геномный надзор имеет решающее значение для обеспечения готовности кпандемиям6, и его потенциал может быть создан путем рутинизации геномного надзора за эндемичными патогенами, такими как RABV. Глобальные различия в возможностях секвенирования, выявленные во время пандемии SARS-CoV-2, должны стать движущей силой каталитических изменений для устранения этого структурного неравенства.
Этот рабочий процесс от образца до последовательности и интерпретации для RABV, включая доступные инструменты биоинформатики, может быть использован для принятия мер по борьбе с бешенством, передаваемых собаками, к 2030 году и, в конечном итоге, для элиминации вариантов RABV. В сочетании с соответствующими метаданными геномные данные, полученные на основе этого протокола, способствуют быстрому определению характеристик RABV при расследовании вспышек и выявлении циркулирующих линий в стране или регионе60,61,65. Мы иллюстрируем наш конвейер в основном на примерах бешенства, передаваемого собаками; Тем не менее, рабочий процесс непосредственно применим к бешенству диких животных. Такая трансмиссивность и низкая стоимость сводят к минимуму трудности, связанные с обеспечением доступности рутинного секвенирования не только для бешенства, но и для других патогенов 46,66,67, что позволяет улучшить ведение болезни и контроль над ней.
Disclosures
Авторам нечего раскрывать.
Acknowledgements
Эта работа была поддержана Wellcome [207569/Z/17/Z, 224670/Z/21/Z], финансированием Newton от Совета по медицинским исследованиям [MR/R025649/1] и Министерством науки и технологий Филиппин (DOST), Британским центром исследований и инноваций в области глобальных усилий по борьбе с COVID-19 [MR/V035444/1], Фондом институциональной стратегической поддержки Университета Глазго [204820], Премией Совета по медицинским исследованиям для новых исследователей (KB) [MR/X002047/1], и Фонд развития международного партнерства, стипендия Национального института исследований в области здравоохранения [17/63/82] GemVi (GJ) и стипендии Университета Глазго от MVLS DTP (KC) [125638-06], EPSRC DTP (RD) [EP/T517896/1] и Wellcome IIB DTP (HF) [218518/Z/19/Z]. Мы благодарны коллегам и сотрудникам, которые поддержали эту работу: Даниэлю Стрейкеру, Элис Броос, Элизабет Миранда, DVM, Дарье Манало, DVM, Тумби Мванги, Кеннеди Лушаси, Чарльзу Каюки, Джуду Карло Боливару, Джеромиру Бондоку, Эстевену Бальбину, Роннелю Тонгохану, Агате Уканде, Дэвису Кучаке, Мумбуа Мутунге, Лвитико Сикане и Анне Чуприне.
Materials
| Name | Company | Catalog Number | Comments |
| Brand name | |||
| Software | |||
| Sequencing software (MinKnow) | Oxford Nanopore Technologies | https://community.nanoporetech.com/downloads | |
| Bioinformatics tool kit (Guppy) | Oxford Nanopore Technologies | https://community.nanoporetech.com/docs/prepare/library_prep_protocols/Guppy-protocol/v/gpb_2003_v1_revao_14dec2018 | |
| Equipment | |||
| Thermal cycler (miniPCR™ mini16 thermal cycler) | Cambio | MP-QP-1016-01 | |
| Homogenizer (Precellys Evolution Touch Homogenizer) | Bertin Instruments | EQ02520-300 | |
| Cold Racks (0.2-0.5mL) (PCR Mini-cooler with transparent lid) | BRAND | 781260 | |
| Pipettor | |||
| (Pipetman L Fixed F1000L, 1000 uL) | Gilson | SKU: FA10030 | |
| (Pipetman L Fixed F100L, 100 uL) | Gilson | SKU: FA10024 | |
| (Pipetman L Fixed F10L, 10 uL) | Gilson | SKU: FA10020 | |
| (Pipetman L Fixed F1L, 1 uL) | Gilson | SKU: FA10025 | |
| (Pipetman L Fixed F20L, 20 uL) | Gilson | SKU: FA10021 | |
| (Pipetman L Fixed F250L, 250 uL) | Gilson | SKU: FA10026 | |
| Fluorometer (Qubit 4 Fluorometer) | Thermofisher scientific/Fisher scientific | Q33238 | |
| Laptop (Any brand with ~2 GB of drive space, minimum of 512 GB storage space, msi installer [GPU]) | |||
| Microcentrifuge (Refrigerated centrifuge) | Thermofisher scientific/Fisher scientific | 75004081 | |
| Vortex mixer (Basic vortex mixer) | Thermofisher scientific/Fisher scientific | 88882011 | |
| Magnetic rack (DynaMag -2 Magnet) | Thermofisher scientific/Fisher scientific | 12321D | |
| Sequencing device (MinION) | Oxford Nanopore Technologies | MinION Mk1B | |
| RNA Extraction | |||
| RNA extraction kit (Qiagen RNEasy Mini Kit 250) | Qiagen | 74106 | |
| RNA stabilizing reagents | |||
| (RNA later) | Invitrogen | AM7020 | |
| (DNA/RNA Shield) | Zymo Research | R1100-50 | |
| PCR | |||
| Nuclease-free Water (Nuclease-free Water [not DEPC-treated]) | Thermofisher scientific/Fisher scientific | AM9937 | |
| Master mix for first strand cDNA synthesis (LunaScript RT SuperMix Kit) | New England Biolabs | E3010S | |
| DNA amplification master mix (Q5® Hot Start High-Fidelity 2X Master Mix [NEB]) | New England Biolabs | M0494L | |
| Primer (Scheme) (Custom DNA oligos) | Invitrogen | ||
| SPRI Bead Clean-up | |||
| SPRI beads (Aline Biosciences PCR Clean DX ) | Cambio | AL-AC1003-50 | |
| Ethanol, Pure Absolute, >99.8% (GC) [Riedel-De Haen] | Merck | 818760 | |
| Short Fragment buffer (SFB expansion pack) | Oxford Nanopore Technologies | EXP-SFB001 | |
| DNA Quantification | |||
| DNA quantification kit (Qubit® dsDNA HS Assay Kit) | Thermofisher scientific/Fisher scientific | Q32854 | |
| DNA quantification assay tubes (Qubit™ Assay Tubes) | Thermofisher scientific/Fisher scientific | Q32856 | |
| End Prep and barcoding (Qubit™ Assay Tubes) | |||
| End Prep master mix (NEBNext Ultra End Repair/dA-Tailing Module) | New England Biolabs | E7546L | |
| Barcoding kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 | |
| (Native Barcoding Expansion 1-12) | EXP-NBD104 | ||
| (Native Barcoding Expansion 13-24) | EXP-NBD114 | ||
| (Native Barcoding Expansion 96) | EXP-NBD196 | ||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 | |
| (not compatible) | (not compatible) | ||
| (Native Barcoding Kit 24 V14) | SQK-NBD114.24 | ||
| (Native Barcoding Kit 96 V14) | SQK-NBD114.96 | ||
| Ligation mastermix (Blunt/TA Ligase Master Mix) | New England Biolabs | M0367S | |
| Adapter Ligation | |||
| Adapter ligation master mix | |||
| (NEBNext Quick Ligation Module) | New England Biolabs | E6056S | |
| (NEBNext Ultra II Ligation Module) | New England Biolabs | E7595S | |
| (Blunt/TA Ligase Master Mix) | New England Biolabs | M0367S | |
| Adapter mix | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 EXP-AMII001 | |
| (Adapter Mix II [AMII]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 EXP-NBA114 | |
| (Native adapter [NA]) | |||
| Sequencing | |||
| Flowcell priming kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 EXP-FLP002 | |
| (Flush Buffer [FB]) | |||
| (Flush Tether [FT]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 EXP-FLP004 | |
| (Flow Cell Flush [FCF]) | |||
| (Flow Cell Tether [FCT]) | |||
| Ligation Sequencing Kit | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 SQK-LSK109 | |
| Adapter Mix (Adapter Mix [AMX]) | |||
| Ligation Buffer (Ligation buffer [LNB]) | |||
| Short Fragment Buffer (Short Fragment buffer [SFB]) | |||
| Sequencing Buffer (Sequencing Buffer [SQB]) | |||
| Elution Buffer (Elution buffer [EB]) | |||
| Loading Beads (Loading Beads [LB]) | |||
| Sequencing Tether (Sequencing Tether [SQT]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 SQK-LSK114 | |
| Adapter Mix (Ligation Adapter [LA]) | |||
| Ligation Buffer (Ligation buffer [LNB]) | |||
| Short Fragment Buffer (Short Fragment buffer [SFB]) | |||
| Sequencing Buffer (Sequencing Buffer [SB]) | |||
| Elution Buffer (Elution buffer [EB]) | |||
| Loading Beads (Loading Beads [LIB]) | |||
| Sequencing Tether (Flow Cell Tether [FCT]) | |||
| Library solution (Library solution [LIS]) | |||
| Flush buffer (Flow Cell Flush [FCF]) | |||
| Flow Cell | |||
| *Chemistry 9 | Oxford Nanopore Technologies | *Chemistry 9 FLO-MIN106D | |
| (Flow Cell [R9.4.1]) | |||
| *Chemistry 14 | Oxford Nanopore Technologies | *Chemistry 14 FLO-MIN114 | |
| (Flow Cell [R10.4.1]) | |||
| Flow Cell wash | |||
| Flowcell wash kit (Flow cell wash kit) | Oxford Nanopore Technologies | EXP-WSH004 | |
| Consummables | |||
| Surface decontaminant | |||
| (DNA Away Surface Decontaminant, Squeeze Bottle [Molecular Bio]) | Thermofisher scientific/Fisher scientific | 7010PK | |
| (RNase Away Surface Decontaminant, Bottle [Molecular Bio]) | Thermofisher scientific/Fisher scientific | 7002PK | |
| PCR 8-Tube Strip 0.2ml, individual cap (PCR 8-Tube Strip 0.2ml, with Individual attached Flat Caps, Sterile, DNAse/RNAse, Pyrogen Free,Natural [Greiner]) | Greiner | 608281 | |
| PCR Tube 0.2ml (PCR Tube 0.2ml, Natural [Domed Cap] Bagged in 500s, Non-Sterile [Greiner]) | Greiner | 671201 | |
| 1000µL Filter Tips (500) (Stacked 1000µL Filter Tips [500]) | Thermofisher scientific/Fisher scientific | 11977724 | |
| 100µL Filter Tips (1000) | Thermofisher scientific/Fisher scientific | 11947724 | |
| 10µL Filter Tips (1000) (Stacked 100µL Filter Tips [1000]) | Thermofisher scientific/Fisher scientific | 11907724 | |
| Reinforced tubes tubes (2ml) with screw caps and o-rings (Fisherbrand™ Bulk tubes) | Thermofisher scientific/Fisher scientific | 15545809 | |
| Microcentrifuge tube (1.5ml) (1.5 ml Eppendorf Tubes [500]) | Eppendorf | 1229888 | |
| DNA LoBind Tubes (1.5ml) (DNA LoBind Tubes) | Thermofisher scientific/Fisher scientific | 10051232 | |
| Cryobabies labels | |||
| Gloves (S/M/L) | |||
| Paper towel |
References
- Rupprecht, C. E. Rhabdoviruses: rabies virus. Medical Microbiology. , (1996).
- Rabies. World Health Organization Available from: https://www.who.int/news-room/fact-sheets/detail/rabies (2023)
- WHO Expert Consultation on Rabies: WHO TRS N°1012. World Health Organization Available from: https://www.who.int/publications-detail-redirect/WHO-TRS-1012 (2018)
- Benavides, J. A., et al. Defining new pathways to manage the ongoing emergence of bat rabies in Latin America. Viruses. 12 (9), 1002 (2020).
- Hampson, K. Estimating the global burden of endemic canine rabies. PLoS Neglected Tropical Diseases. 9 (4), e0003709 (2015).
- Global genomic surveillance strategy for pathogens with pandemic and epidemic potential, 2022-2032. World Health Organization Available from: https://www.who.int/publications-detail-redirect/978924004679 (2022)
- Tsai, K. J., et al. Emergence of a sylvatic enzootic formosan ferret badger-associated rabies in Taiwan and the geographical separation of two phylogenetic groups of rabies viruses. Veterinary Microbiology. 182, 28-34 (2016).
- Chiou, H. -. Y., et al. Molecular characterization of cryptically circulating rabies virus from ferret badgers, Taiwan. Emerging Infectious Diseases. 20 (5), 790-798 (2014).
- Sabeta, C. T., Mansfield, K. L., McElhinney, L. M., Fooks, A. R., Nel, L. H. Molecular epidemiology of rabies in bat-eared foxes (Otocyon megalotis) in South Africa. Virus Research. 129 (1), 1-10 (2007).
- Scott, T. P. Complete genome and molecular epidemiological data infer the maintenance of rabies among kudu (Tragelaphus strepsiceros) in Namibia. PLoS One. 8 (3), e58739 (2013).
- Lembo, T., et al. Exploring reservoir dynamics: a case study of rabies in the Serengeti ecosystem. The Journal of Applied Ecology. 45 (4), 1246-1257 (2008).
- Coetzee, P., Nel, L. H. Emerging epidemic dog rabies in coastal South Africa: a molecular epidemiological analysis. Virus Research. 126 (1-2), 186-195 (2007).
- Ou de Munnink, B. B. First molecular analysis of rabies virus in Qatar and clinical cases imported into Qatar, a case report. International Journal of Infectious Diseases. 96, 323-326 (2020).
- Smith, J., et al. Case report: Rapid ante-mortem diagnosis of a human case of rabies imported into the UK from the Philippines. Journal of Medical Virology. 69, 150-155 (2003).
- Mahardika, G. N. K., et al. Phylogenetic analysis and victim contact tracing of rabies virus from humans and dogs in Bali, Indonesia. Epidemiology and Infection. 142 (6), 1146-1154 (2014).
- Tohma, K., et al. Molecular and mathematical modeling analyses of inter-island transmission of rabies into a previously rabies-free island in the Philippines. Infection, Genetics and Evolution. 38, 22-28 (2016).
- Tohma, K., et al. Phylogeographic analysis of rabies viruses in the Philippines. Infection, Genetics and Evolution. 23, 86-94 (2014).
- Saito, M., et al. Genetic diversity and geographic distribution of genetically distinct rabies viruses in the Philippines. PLoS Neglected Tropical Diseases. 7 (4), e2144 (2013).
- Biek, R., Henderson, J. C., Waller, L. A., Rupprecht, C. E., Real, L. A. A high-resolution genetic signature of demographic and spatial expansion in epizootic rabies virus. Proceedings of the National Academy of Sciences. 104 (19), 7993-7998 (2007).
- Reddy, G. B., et al. Molecular characterization of Indian rabies virus isolates by partial sequencing of nucleoprotein (N) and phosphoprotein (P) genes. Virus Genes. 43, 13-17 (2011).
- David, D., Dveres, N., Yakobson, B. A., Davidson, I. Emergence of dog rabies in the northern region of Israel. Epidemiology and Infection. 137 (4), 544-548 (2009).
- Benjathummarak, S. Molecular genetic characterization of rabies virus glycoprotein gene sequences from rabid dogs in Bangkok and neighboring provinces in Thailand, 2013-2014. Archives of Virology. 161 (5), 1261-1271 (2016).
- Denduangboripant, J., et al. Transmission dynamics of rabies virus in Thailand: implications for disease control. BMC Infectious Diseases. 5, 52 (2005).
- Talbi, C., et al. Phylodynamics and human-mediated dispersal of a zoonotic virus. PLoS Pathogens. 6 (10), e1001166 (2010).
- Bourhy, H., et al. Revealing the micro-scale signature of endemic zoonotic disease transmission in an African urban setting. PLoS Pathogens. 12 (4), e1005525 (2016).
- Zinsstag, J., et al. Vaccination of dogs in an African city interrupts rabies transmission and reduces human exposure. Science Translational Medicine. 9 (421), (2017).
- Yakovleva, A., et al. Tracking SARS-COV-2 variants using Nanopore sequencing in Ukraine in 2021. Scientific Reports. 12, 15749 (2022).
- Mannsverk, S., et al. SARS-CoV-2 variants of concern and spike protein mutational dynamics in a Swedish cohort during 2021, studied by Nanopore sequencing. Virology Journal. 19, 164 (2022).
- Soufi, M., et al. Fast and easy nanopore sequencing workflow for rapid genetic testing of familial Hypercholesterolemia. Frontiers in Genetics. 13, 836231 (2022).
- Cabibbe, A. M. Application of targeted next-generation sequencing assay on a portable sequencing platform for culture-free detection of drug-resistant tuberculosis from clinical samples. Journal of Clinical Microbiology. 58 (10), 00632 (2020).
- Xu, Y., et al. Nanopore metagenomic sequencing of influenza virus directly from respiratory samples: diagnosis, drug resistance and nosocomial transmission, United Kingdom, 2018/19 influenza season. Euro Surveillance. 26 (27), 2000004 (2021).
- Stubbs, S. C. B., et al. Assessment of a multiplex PCR and Nanopore-based method for dengue virus sequencing in Indonesia. Virology Journal. 17, 24 (2020).
- Croville, G., et al. Rapid whole-genome based typing and surveillance of avipoxviruses using nanopore sequencing. Journal of Virological Methods. 261, 34-39 (2018).
- Theuns, S., et al. Nanopore sequencing as a revolutionary diagnostic tool for porcine viral enteric disease complexes identifies porcine kobuvirus as an important enteric virus. Scientific Reports. 8, 9830 (2018).
- O'Donnell, V. K., et al. Rapid sequence-based characterization of African swine fever virus by use of the Oxford Nanopore MinION sequence sensing device and a companion analysis software tool. Journal of Clinical Microbiology. 58, 01104-01119 (2019).
- Brito, A. F. Global disparities in SARS-CoV-2 genomic surveillance. Nature Communications. 13, 7003 (2022).
- ONT login/register. Oxford Nanopore Technology Available from: https://nanoporetech.com/login-register (2023)
- Software Downloads. Oxford Nanopore Technology Available from: https://community.nanoporetech.com/downloads (2023)
- . GitHub Available from: https://github.com/ (2023)
- . Artic-rabv Available from: https://github.com/kirstyn/artic-rabv (2022)
- . MADDOG: Method for Assignment, Definition and Designation of Global Lineages Available from: https://github.com/KathrynCampbell/MADDOG (2022)
- RABV-GLUE. Centre for Virus Research Available from: https://github.com/KathrynCampbell/MADDOG (2022)
- Itokawa, K., Sekizuka, T., Hashino, M., Tanaka, R., Kuroda, M. Disentangling primer interactions improves SARS-CoV-2 genome sequencing by multiplex tiling PCR. PLoS ONE. 15 (9), e0239403 (2020).
- Davis, M. W., Jorgensen, E. M. ApE, A plasmid editor: A freely available DNA manipulation and visualization program. Frontiers in Bioinformatics. 2, 818619 (2022).
- Döring, M., Pfeifer, N. . openPrimeR: Multiplex PCR primer design and analysis. , (2023).
- Quick, J. Multiplex PCR method for MiniON and Illumina sequencing of Zika and other virus genomes directly from clinical samples. Nature Protocols. 12 (6), 1261-1276 (2017).
- Laboratory Techniques in Rabies. World Health Organization Available from: https://apps.who.int/iris/bitstream/handle/10665/310836/9789241515153-eng.pdf (2018)
- Lembo, T. Partners for Rabies Prevention. The blueprint for rabies prevention and control: a novel operational toolkit for rabies elimination. PLoS Neglected Tropical Diseases. 6 (2), e1388 (2012).
- Terrestrial Manual Online Access. World Organization for Animal Health Available from: https://www.woah.org/en/what-we-do/standards/codes-and-manuals/terrestrial-manual-online-access/ (2023)
- Mauti, S. Field postmortem rabies rapid immunochromatographic diagnostic test for resource-limited settings with further molecular applications. Journal of Visualized Experiments. (160), e60008 (2020).
- Patrick, E., et al. Enhanced rabies surveillance using a direct rapid immunohistochemical test. Journal of Visualized Experiments. (146), e59416 (2019).
- Lembo, T., et al. Evaluation of a direct, rapid immunohistochemical test for rabies diagnosis. Emerging Infectious Diseases. 12 (2), 310-313 (2006).
- Marston, D. A., et al. Pan-lyssavirus real time RT-PCR for rabies diagnosis. Journal of Visualized Experiments. (149), e59709 (2019).
- . DNA quantification using the Qubit fluorometer Available from: https://www.protocols.io/view/dna-quantification-using-the-qubit-fluorometer-bc6vize6 (2020)
- . DNA quantification using the Quantus fluorometer Available from: https://www.protocols.io/view/dna-quantification-using-the-quantus-fluorometer-7pzhmp6 (2020)
- Guppy protocol. Nanopore Community Available from: https://community.nanoporetech.com/protocols/Guppy-protocol/v/gpb_2003_v1_revaq_14dec2018 (2023)
- Flow Cell Wash Kit (EXP-WSH004). Nanopore Community Available from: https://community.nanoporetech.com/protocols/flow-cell-wash-kit-exp-wsh004/v/wfc_9120_v1_revh_08dec2020 (2023)
- . Core Pipeline - arctic pipeline Available from: https://artic.readthedocs.io/en/latest/minion/ (2023)
- . Samtools Available from: https://www.htslib.org (2023)
- Campbell, K., et al. Making genomic surveillance deliver: A lineage classification and nomenclature system to inform rabies elimination. PLoS Pathogens. 18 (5), e1010023 (2022).
- Brunker, K., et al. Rapid in-country sequencing of whole virus genomes to inform rabies elimination programmes. Wellcome Open Research. 5, 3 (2020).
- Bull, R. A., et al. Analytical validity of nanopore sequencing for rapid SARS-CoV-2 genome analysis. Nature Communications. 11, 6272 (2020).
- Okeke, I. N., Ihekweazu, C. The importance of molecular diagnostics for infectious diseases in low-resource settings. Nature Reviews. Microbiology. 19 (9), 547-548 (2021).
- Inzaule, S. C., Tessema, S. K., Kebede, Y., Ouma, A. E. O., Nkengasong, J. N. Genomic-informed pathogen surveillance in Africa: opportunities and challenges. The Lancet Infectious Diseases. 21 (9), 281-289 (2021).
- Kennedy, L. Integrating contact tracing and whole-genome sequencing to track the elimination of dog-mediated rabies: an observational and genomic study. eLife. , (2023).
- Pallerla, S. R. Diagnosis of pathogens causing bacterial meningitis using Nanopore sequencing in a resource-limited setting. Annals of Clinical Microbiology and Antimicrobials. 21, 39 (2022).
- Quick, J. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228-232 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved