Método LipidUNet-Machine Learning de Caracterização e Quantificação de Depósitos Lipídicos Usando Epitélio Pigmentado Retiniano Derivado de iPSC
In This Article
Summary
As doenças oculares degenerativas que afetam a camada do epitélio pigmentado da retina do olho têm origem monogênica e poligênica. Vários modelos de doença e um software, LipidUNet, foram desenvolvidos para estudar mecanismos de doença, bem como potenciais intervenções terapêuticas.
Abstract
O epitélio pigmentado da retina (EPR) é uma monocamada de células hexagonais localizada na parte posterior do olho. Fornece nutrição e suporte a fotorreceptores e capilares coroidais, realiza fagocitose de segmentos externos (POS) fotorreceptores e secreta citocinas de forma polarizada para manter a homeostase da retina externa. A EPR disfuncional, causada por mutações, envelhecimento e fatores ambientais, resulta na degeneração de outras camadas da retina e causa perda da visão. Uma característica fenotípica marcante da degeneração do EPR são os depósitos ricos em lipídios intra e subcelulares. Esses depósitos são um fenótipo comum em diferentes doenças degenerativas da retina. Para reproduzir in vitro o fenótipo de depósito lipídico das degenerações retinianas monogênicas, o EPR derivado de células-tronco pluripotentes induzido (iRPE) foi gerado a partir de fibroblastos dos pacientes. Linhagens celulares geradas a partir de pacientes com doença de Stargardt e degeneração retiniana de início tardio (L-ORD) foram alimentadas com POS por 7 dias para replicar a função fisiológica do EPR, que causou patologia induzida por fagocitose POS nessas doenças. Para gerar um modelo para a degeneração macular relacionada à idade (DMRI), uma doença poligênica associada à ativação alternativa do complemento, a iRPE foi desafiada com anafilatoxinas alternativas do complemento. Os depósitos lipídicos intra e subcelulares foram caracterizados utilizando-se Vermelho do Nilo, boro-dipirrometeno (BODIPY) e apolipoproteína E (APOE). Para quantificar a densidade dos depósitos lipídicos, foi desenvolvido um software baseado em aprendizado de máquina, o LipidUNet. O software foi treinado em imagens de projeção de máxima intensidade de iRPE em superfícies de cultura. No futuro, será treinado para analisar imagens tridimensionais (3D) e quantificar o volume de gotículas lipídicas. O software LipidUNet será um recurso valioso para a descoberta de fármacos que diminuam o acúmulo lipídico em modelos de doenças.
Introduction
O epitélio pigmentado da retina (EPR) é uma monocamada de células localizada na parte posterior do olho adjacente aos fotorreceptores da retina. O EPR desempenha um papel vital na manutenção da visão adequada, fornecendo suporte metabólico e estrutural aos fotorreceptores. As células saudáveis do EPR são caracterizadas por uma morfologia hexagonal distinta. Eles são conectados por tight junctions, que permitem que o PSE atue como uma barreira entre os coriocapilares localizados em sua face basal e os fotorreceptores localizados apicamente. Para manter o ecossistema da retina, o EPR transporta metabólitos-chave, por exemplo, glicose, para fotorreceptores de forma a minimizar o consumo de glicose no EPR1. Devido a essa limitação, o EPR depende de outros metabólitos para manter suas necessidades metabólicas, incluindo os ácidos graxos, que o EPR converte em cetonas por meio da β-oxidação2. Dada a propensão do EPR a utilizar ácidos graxos, que provavelmente são reciclados da digestão do segmento externo (SOP) dos fotorreceptores, como fonte de energia, alterações prejudiciais nas vias de processamento lipídico no EPR frequentemente levam ou estão implicadas em doenças degenerativas retinianas monogênicas e poligênicas3.
A degeneração macular relacionada à idade (DMRI), uma doença ocular degenerativa poligênica que causa degeneração do EPR, também tem sido associada à autofagia aberrante e ao metabolismo lipídico na monocamada do EPR. A falha de uma monocamada disfuncional de EPR em processar a SOP e executar outras funções críticas leva a depósitos extracelulares (sub-PSE) chamados depósitos lineares basais (BLinD) localizados entre o EPR e a membrana de Bruch - uma característica das patologias da DMRI. Os principais componentes da LBinD incluem as lipoproteínas, sendo a apolipoproteína E (APOE) a mais abundante4. O acúmulo de finas camadas de LBinD pode levar a drusas moles, o que é reconhecido como sintoma clínico da DMRI 5,6.
Vários grupos têm demonstrado que modelos de doenças in vitro derivadas de células-tronco que causam disfunção do EPR apresentam acúmulo lipídico sub-EPR 7,8,9. (2017) geraram EPR pluripotente induzido derivado de células-tronco (iRPE) de pacientes com alto risco de DMRI devido a um polimorfismo do gene CFH. A iPSE apresentou acúmulo de drusa, como marcado pela APOE, e a PSE de alto risco acumulou depósitos maiores do que a iRPE gerada em pacientes de baixo risco10.
Para criar um modelo in vitro que recapitule as características celulares da DMRI, como gotículas lipídicas e deposição de drusas, linhagens de iRPE geradas a partir de amostras de sangue de pacientes foram estabelecidas usando um protocolo guiado pelo desenvolvimento previamente publicado11. Os iRPE foram submetidos ao soro humano competente para o complemento (CC-HS), uma solução contendo anafilatoxinas que mimetizam uma possível causa de DMRI: aumento da sinalização alternativa do complemento8. A deposição celular e subcelular resultante dos depósitos lipídicos foi medida usando marcadores lipídicos e lipoprotéicos comumente usados, APOE, Nile Red e BODIPY. Por meio desses marcadores, demonstrou-se que a sinalização ativada do complemento via CC-HS exacerbou o acúmulo lipídico nas células iRPE8.
Para desenvolver um modelo de doença para uma doença degenerativa retiniana monogênica, linhagens de iRPE foram desenvolvidas a partir de pacientes com doença de Stargardt, uma doença causada por mutações no gene ABCA4 em EPR. Foi demonstrado anteriormente que, quando o ABCA4 é nocauteado, a lipofuscina A2E, um depósito intracelular conhecido por conter altos níveis de fosfolipídios e produtos de peroxidação lipídica dependentes de luz, se acumula dentro do EPR12. Linhagens knockout ABCA4 foram desenvolvidas ao lado das linhagens dos pacientes, e ambas foram submetidas à alimentação por PDV. O Stargardt iRPE demonstrou patologia induzida por fagocitose POS, exibindo aumento do acúmulo lipídico quantificado pela coloração BODIPY. PSE derivados de iPSCs ABCA4 KO foram submetidos ao tratamento CC-HS; a quantificação do sinal BODIPY também mostrou defeito no manuseio lipídico no modelo da doença de Stargardt9.
Dada a prevalência dessas doenças e a necessidade de terapêuticas eficazes, juntamente com os modelos de doenças relevantes descritos acima, há necessidade de estabelecer métodos robustos para quantificar a eficácia de potenciais tratamentos. Para quantificar os depósitos lipídicos de forma objetiva, automatizada e padronizada, um software baseado em aprendizado de máquina, LipidUNet, foi criado para que, quando emparelhado com ferramentas de análise de máscaras, a deposição lipídica possa ser rápida e efetivamente identificada usando os marcadores comuns Nile Red, BODIPY e APOE. As estatísticas resumidas obtidas usando este pipeline de análise podem então ser analisadas e exibidas graficamente, permitindo uma fácil comparação das condições de tratamento. O esquema do protocolo é mostrado na Figura 1.
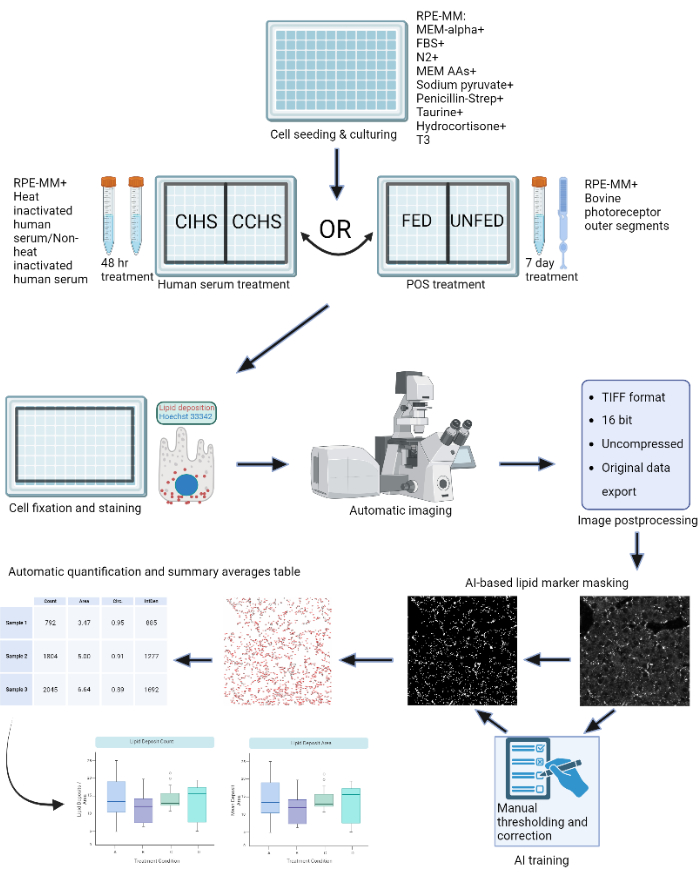
Figura 1: Esquema do protocolo: As células do EPR são cultivadas em uma placa de 96 poços e desafiadas com soro humano ativo ou segmentos externos bovinos purificados para modelar diferentes tipos de degenerações retinianas in vitro. As células do EPR são fixadas e coradas para depósitos de lipoproteínas com Vermelho do Nilo, BODIPY e APOE. Um microscópio confocal é usado para adquirir pilhas Z de partículas lipídicas fluorescentemente marcadas, que são subsequentemente processadas em projeções de intensidade máxima 2D. Um algoritmo de aprendizado de máquina foi treinado para reconhecer e segmentar corretamente as partículas de lipoproteínas. Tabelas de resumo contendo contagem de partículas e várias métricas de forma são geradas e podem ser usadas para plotagem subsequente e análise estatística. Clique aqui para ver uma versão maior desta figura.
Protocol
Todas as etapas do protocolo seguem as diretrizes estabelecidas pelo comitê de ética em pesquisa humana do NIH. O trabalho com células-tronco e a coleta de amostras de pacientes foram aprovados pelo Combined NeuroScience Institutional Review Board (CNS IRB) sob o Office of Human Research Protection (OHRP), NIH, de acordo com as diretrizes 45 CFR 46 do governo dos EUA. As amostras dos pacientes foram coletadas por meio do termo de consentimento livre e esclarecido aprovado pelo CNS de acordo com os critérios estabelecidos pela Declaração de Helsinque sob o número de protocolo NCT01432847 (https://clinicaltrials.gov/ct2/show/NCT01432847?cond=NCT01432847&draw=2&rank=1).
1. Geração de iRPE
- Gerar iRPE a partir de iPSC derivada do sangue do paciente seguindo o protocolo publicado por Sharma et al., 202211 (Figura 2 e Figura 3).
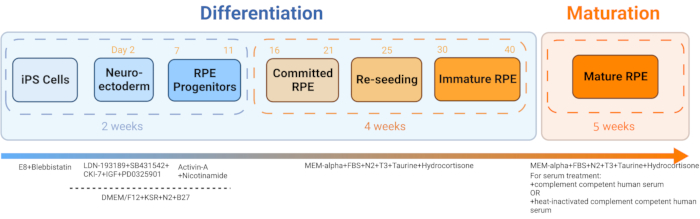
Figura 2: Esquema de diferenciação e maturação do iRPE. Para gerar a iRPE, um protocolo de diferenciação estabelecido foi seguido, e as células foram deixadas amadurecer por 5 semanas. A cultura celular resultante atua como um modelo in vitro que pode ser manipulado com vários tratamentos para mimetizar a disfunção do EPR em doenças como DMRI e doença de Stargardt. Clique aqui para ver uma versão maior desta figura.
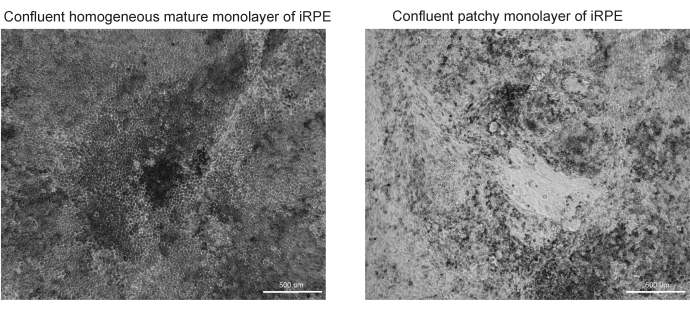
Figura 3: Imagens representativas da diferenciação e maturação bem-sucedidas e malsucedidas da PSE. Duas imagens de campo brilhante com aumento de 10x da PSE TJP1 são mostradas no Dia 42 do protocolo iRPE. (A) A diferenciação e maturação bem-sucedidas mostrarão PSE confluente com pigmentação e morfologia poligonal. (B) A diferenciação e a maturação malsucedidas mostrarão aglomerados de células moribundas, como mostrado aqui. Clique aqui para ver uma versão maior desta figura.
2. Preparação do meio de manutenção RPE (RPE-MM)
- Descongele o suplemento de N2 a 4 °C durante a noite. Descongelar todos os outros reagentes à temperatura ambiente (RT).
- Em condições estéreis, adicionar os reagentes listados na Tabela 1 nos fatores de diluição listados, conforme protocolo estabelecido por Sharma et al., 202211.
- Misture bem o meio e filtre-o usando uma unidade de filtragem de 0,22 μm.
NOTA: Os suportes são adequados para utilização no prazo de 2 semanas se armazenados a 4 °C.
3. Semeadura de placa de 96 poços
- Descongele uma alíquota de vitronectina em RT por 3-5 min ou até que o gelo esteja completamente derretido.
- Diluir a vitronectina com 1x solução salina tamponada com fosfato (DPBS) de Dulbecco para obter a solução de trabalho desejada usando uma diluição de 1:200 (vitronectina: DPBS). Para uma placa de 96 poços, cubra cada poço com 200 μL da solução de trabalho.
- Combinar o inibidor de ROCK descongelado (dicloridrato de Y-27632) com o RPE-MM numa diluição de 1:1000 para atingir uma concentração final de 10 μM. Este é o meio de revestimento para células de EPR.
- Descongelar o frasco para injetáveis de iRPE utilizando um sistema automatizado de descongelamento celular e transferir a suspensão de células de iRPE para um tubo de 50 ml.
- Diluir a suspensão celular com o meio de chapeamento numa diluição de 1:10. Centrifugar o tubo a 400 x g por 5 min.
- Aspirar cuidadosamente o sobrenadante e ressuspender as células em 10 mL do meio de plaqueamento.
- Misturar 400 μL do meio de plaqueamento com 100 μL da solução celular ressuspensa para contagem celular. Use esta alíquota para determinar a concentração de células viáveis da suspensão celular usando um contador de viabilidade celular.
- Diluir a suspensão celular com o meio de plaqueamento até uma concentração final de 60.000 células/mL.
- Aspirar completamente a solução de revestimento de vitronectina da placa de 96 poços e distribuir 200 μL da suspensão celular em cada poço. Haverá aproximadamente 12.000 células/poço ou ~200 células/mm2.
- Incubar as placas celulares semeadas durante 48 h a 37 °C e 5% de CO2. Após 48 h, trocar o meio para PSE-MM sem suplemento inibidor de ROCK. Troque a mídia a cada 2-3 dias durante o período de maturação de 5 semanas.
4. Modelos de doenças in vitro
- Tratamento com soro humano competente do complemento (CC-HS)
- Descongelar o soro competente do complemento humano a 4°C durante a noite.
- Preparar CC-HS e complementar meios de soro humano incompetente (CI-HS).
- Para preparar o meio CC-HS a 5%, misturar o soro humano competente do complemento descongelado com o PSE-MM na diluição de 1:20. Filtrar a solução através de um filtro de meio de 0,22 μm antes de usar.
- Para preparar o meio de soro humano incompetente (CI-HS) de complemento a 5%, primeiro inative o CC-HS em banho-maria a 57 °C por 30 min e, em seguida, misture com o meio de cultura na diluição de 1:20. Filtrar a solução através de um filtro de meio de 0,22 μm antes de usar.
- O soro trata as células com 200 μL de CC-HS a 5% ou CI-HS a 5% por um tempo total de incubação de 48 h, refrescando o meio após 24 h.
- Lave as células com 1x DPBS e fixe-as com paraformaldeído a 4% por 20 min em RT. Lave novamente com 1x DPBS e armazene as amostras a 4 °C, imersas em 200 μL de DPBS.
- OPCIONAL: Se desejar, lisar as células da placa para mostrar apenas a deposição lipídica sub-RPE.
- Para lisar as células e deixar apenas depósitos lipídicos, remova o meio e adicione 200 μL de água deionizada a cada poço.
- Incubar por 10-15 min, pipetar para cima e para baixo até que as células sejam removidas. Lavar novamente com 200 μL de água deionizada e fixar imediatamente as células com paraformaldeído a 4%.
- Confirmar a eficácia da remoção celular com coloração nuclear usando Hoechst. Adicionar Hoechst na diluição de 1:2000 a uma solução de 1x DPBS contendo 1% de albumina de soro bovino (BSA), 0,5% de Tween 20 e 0,5% de Triton-X 100. Incubar em TR por 1 h no escuro. Posteriormente, lave com 1x DPBS.
- Tratamento do segmento externo (POS) fotorreceptor em iRPE
- Preparação do PDV
- Remova o tubo de pellet POS do armazenamento de -80 °C e descongele durante a noite a 4 °C em um balde de gelo coberto.
- Preparar o tampão de lavagem do PDV misturando 10 g de sacarose em 40 mL de H 2 O (ddH2O) duplamente deionizado.
- Aqueça a mistura a 40-50 °C enquanto mexe suavemente durante 15 minutos. Adicione 840 mg de bicarbonato de sódio à mistura e mexa enquanto aquece durante 10 minutos.
- Ajustar o volume total do tampão de lavagem do PDV para 100 mL com ddH2O e ajustar o pH da solução para 8,3 com 1 N HCl ou 1 N NaOH, conforme necessário. Filtrar a solução de lavagem utilizando um filtro de 0,22 μm.
NOTA: O protocolo pode ser pausado aqui; o tampão de lavagem do PDV pode ser armazenado a 4 °C durante a noite. - Uma vez descongelado, suspender o pellet em 15 mL de tampão de lavagem POS. Seja gentil durante as suspensões de pellets para garantir a integridade do PDV. Centrifugar a suspensão de PDV a 600 x g a 4 °C durante 20 min e, em seguida, aspirar o sobrenadante.
- Ressuspender o pellet de PDV em 10 mL do tampão de lavagem do PDV.
- Remova uma alíquota de 100 μL do tampão de lavagem POS + POS (solução POS) e dilua em 400 μL de 1x DPBS. Espalhe 50 μL da solução diluída de POS numa placa de ágar sangue e numa placa de agarose para verificar a existência de contaminantes bacterianos e fúngicos. Preparar controlos positivos para cada um e incubar todas as placas durante 48 h a 37 °C.
- Realizar um ensaio de qPCR adicionando 1 μL de solução de POS ao poço de detecção para testar a presença de micoplasma. Para amplificar fragmentos de DNA, realizar 40 ciclos de desnaturação (95°C, 15 s), recozimento e alongamento (60°C, 1 min). Os primers forward e reverse para detecção de micoplasma na amostra POS são os seguintes:
Cartilha para a frente: GGA TTA GAT ACC CTG GTA GTC CAC G
Primer reverso: CGT CAA TTC CTT TAA GTT TCA CTC TTG GC - Meça a concentração de PDV usando um analisador de células e alíquota de acordo com a necessidade. Para um poço de placas de 96 poços com células de EPR, 3 x 106 POS é suficiente. A proporção desejada é de 10 células POS/RPE. Conservar as alíquotas a 80 °C para utilização futura.
- Adição de PDV às células
- Descongele frascos de PDV em banho de gelo.
- Misturar a quantidade calculada de POS preparado com PSE-MM e tratar as células com POS uma vez ao dia por 7 dias.
NOTA: Prepare a solução de PDV fresca diariamente. - Lave as células com 1x DPBS e depois fixe-as com paraformaldeído a 4% por 20 min em RT. Lave com DPBS mais uma vez e armazene as amostras a 4 °C, imersas em 200 μL de DPBS.
- Preparação do PDV
5. Coloração para depósitos sub-RPE
- Protocolo de coloração vermelho do Nilo
- Após a fixação com PFA, lavar as amostras 3 vezes com 1x DPBS.
NOTA: Se não for usado imediatamente, o protocolo pode ser pausado aqui, mas as amostras devem ser armazenadas em uma solução de 1x DPBS + azida de sódio a 0,02% a 4 °C. - Para preparar a solução-mãe de Nilo Vermelho, dissolver o pó de Vermelho do Nilo em acetona na concentração de 3 mg/mL. Incubar por 15 min em TR com mistura periódica. Filtrar as soluções com um filtro de 0,22 μm uma ou duas vezes, dependendo do nível de precipitado restante na solução.
NOTA: Proteja a solução-mãe da luz. - Para preparar a solução de trabalho, dilua a solução-estoque na proporção de 1:500 em 1x DPBS. Adicionar 200 μL da solução de trabalho à amostra durante 30 minutos em RT num agitador e protegê-la da luz.
- Lavar 3 vezes com 1x PBS e armazenar as amostras a 4 °C, imersas em 200 μL de DPBS.
NOTA: Se estiver realizando um experimento em transwells em vez de uma placa de 96 poços, as amostras podem ser montadas em uma lâmina com mídia de montagem, cobertas com uma lamínula de vidro e seladas com esmalte transparente. Deve-se tomar cuidado para montar a amostra com as células voltadas para cima.
- Após a fixação com PFA, lavar as amostras 3 vezes com 1x DPBS.
- Protocolo de coloração BODIPY
- Para a solução-mãe, dissolver BODIPY em dimetilsulfóxido anidro (DMSO) para atingir uma concentração de estoque de 3,8 mM.
- Para amostras fixas de PFA, diluir o estoque de BODIPY a 1:300 em 1x DPBS. Adicione 200 μL às células e incube durante a noite em um balancim em RT.
- Lavar 3 vezes com 1x DPBS e armazenar as amostras a 4 °C, imersas em 200 μL de DPBS.
NOTA: Se estiver realizando um experimento em transwells em vez de uma placa de 96 poços, as amostras podem ser montadas em uma lâmina com mídia de montagem, cobertas com uma lamínula de vidro e seladas com esmalte transparente. Deve-se tomar cuidado para montar a amostra com as células voltadas para cima.
- Protocolo de imunomarcação da APOE
- Combine 1x DPBS com 1% de albumina de soro bovino (BSA), 0,5% Tween 20 e 0,5% Triton-X 100 para criar uma solução tampão.
- Para amostras fixas em PFA, bloquear e permeabilizar a amostra em 200 μL da solução tampão por 1 h no TR.
- Adicionar o anticorpo primário APOE diluído a 1:100 na solução tampão e incubar durante a noite na RT.
- No dia seguinte, lave as amostras 3 vezes com 1x DPBS.
- Adicionar um anticorpo secundário na diluição de 1:1000 na solução tampão e adicionar 200 μL da solução às células durante 1 h em RT.
- Lavar 3 vezes com 1x DPBS e armazenar amostras a 4 °C, imersas em 200 μL de DPBS.
NOTA: Se estiver realizando um experimento em transwells em vez de uma placa de 96 poços, as amostras podem ser montadas em uma lâmina com mídia de montagem (Fluoromount), cobertas com uma tampa de vidro e seladas com esmalte transparente. Deve-se tomar cuidado para montar a amostra com as células voltadas para cima.
6. Automação e processamento de imagens
- Varredura automatizada de imagens
OBS: Microscópio de varredura confocal invertido Zeiss LSM 800 e software ZEN 3.2 (edição azul) foram utilizados neste estudo. Certifique-se de que a placa de 96 poços seja aquecida para RT por um mínimo de 60 minutos antes da aquisição de imagens para evitar desvio do plano focal durante a varredura devido a uma mudança no índice de refração do meio com a mudança de temperatura.- Usando um microscópio confocal e uma objetiva de 40x, crie um perfil de varredura com os canais fluorescentes apropriados para o marcador lipídico usado e quaisquer anticorpos adicionais.
- Use a caixa de seleção Blocos para configurar a automação de imagem. Para calibrar a placa de 96 poços, certifique-se de que as medições corretas do portador da amostra sejam inseridas e selecionadas. Em seguida, clique no botão Calibrar para calibrar a placa de acordo com as instruções, o que requer o uso da objetiva de 10x.
- Escolha a visualização Configuração Avançada para selecionar poços apropriados e adicionar 3 pontos de imagem diferentes perto do centro do poço usando a função Posições . Isso pode ser feito manualmente na subguia Posição ou aleatoriamente usando a guia Configuração de posição e selecionando Configuração por operadora. Repita para todos os poços da mesma mancha.
- Para obter o foco ideal e o posicionamento da pilha Z durante a automação, vá para a guia Estratégia de foco para selecionar Usar valores de superfície de foco/Z definidos pela configuração de blocos. Métodos alternativos podem usar outras estratégias de foco, mas recomenda-se usar essa configuração para obter os resultados mais consistentes.
- Na guia Blocos , clique em Verificar Posições e defina manualmente o plano Z central para cada posição. As configurações na subguia Opções ordenarão a aquisição de imagens, portanto, verifique isso antes de iniciar a imagem. Para adquirir imagens na ordem em que as posições foram selecionadas, desmarque as caixas de seleção Regiões de bloco/posições e Poços transportadores/Contêiner . Selecione Dividir cenas em arquivos separados para facilitar o processamento de imagens.
- Verifique se a guia Z-Stack está definida como Central, um intervalo é inserido para as preferências do usuário e o botão Optimal está selecionado para definir o intervalo de fatia.
- Depois de otimizar as guias Modo de aquisição, Canais, Estratégia de foco, Z-Stack e Blocos , inicie o experimento.
- Processamento de imagens
- Usando um método de processamento de imagem em lote, crie projeções máximas de cada pilha Z com o método Profundidade de Foco Estendida .
- Usando um método de processamento de imagem em lote, exporte o máximo de arquivos de projeção como imagens TIFF de 16 bits. Defina a compactação como Nenhum e verifique se Dados Originais estão marcados. A imagem resultante deve ser um TIFF máximo de escala de cinza de projeção apenas do canal de fluorescência no qual o marcador lipídico é expresso.
7. Segmentação e quantificação
NOTA: O programa LipidUNet foi treinado em imagens de 40x de uma placa de 96 poços. É altamente recomendável o uso de imagens obtidas com objetiva de 40x.
- Instale o software LipidUNet. LipidUNet pode ser baixado do seguinte repositório GitHub: https://github.com/RPEGoogleMap/LipidUNet
- Identifique as imagens TIFF que representam Nile Red, Bodipy ou APOE e mova-as para uma pasta chamada imgs dentro de um diretório chamado Nile_Red, Bodipy ou APOE, dependendo do método que está sendo usado.
Observação : convenções de nomenclatura exatas devem ser usadas para o programa LipidUNet para reconhecer os diretórios. - Abra o software LipidUNet (Figura 4).
- Na guia Prever do software, selecione o diretório relevante (Nile_Red, Bodipy ou APOE) clicando nas reticências e navegando até o diretório nomeado. Confirme se o programa LipidUNet identificou corretamente as imagens verificando a entrada de classe.
- Selecione um limite de probabilidade para o algoritmo entre 0,01 e 0,99. Um valor mais alto eliminará mais falsos positivos, mas pode causar mais falsos negativos, e valores mais baixos podem introduzir mais falsos positivos, eliminando mais falsos negativos. Um valor de 0,65 é o padrão e é recomendado.
- Clique em Prever.
NOTA: O software irá iterar através de todas as imagens automaticamente e criar uma nova pasta chamada predicted_masks no diretório selecionado. - Use uma ferramenta de Análise de Máscara para iterar pelas máscaras geradas e fornecer uma contagem quantitativa dos depósitos lipídicos limítrofes das imagens da máscara.
- Analise os dados de contagens gerados para comparar as condições de tratamento.
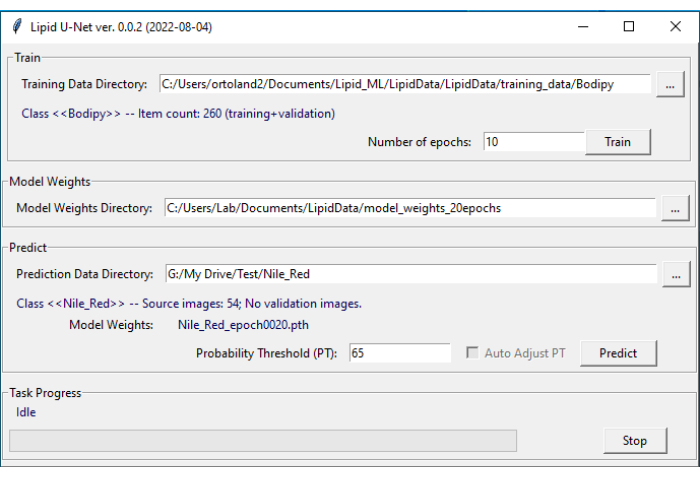
Figura 4: Interface do usuário do LipidUNet. O software LipidUNet possui diferentes seções para selecionar para o diretório de dados de treinamento, onde as imagens dos depósitos lipídicos foram identificadas corretamente; o diretório de pesos do modelo, que é produzido a partir dos dados de treinamento; e o diretório de dados de previsão no qual o usuário inserirá suas imagens para segmentação. Clique aqui para ver uma versão maior desta figura.
Representative Results
Este protocolo fornece um fluxo de trabalho para identificar depósitos lipídicos corados por Nile Red, BODIPY e APOE. O software desenvolvido pode identificar e quantificar automaticamente os depósitos lipídicos e tem melhor desempenho quando o protocolo delineado é otimizado. Incluem-se exemplos de PSE diferenciado com sucesso (Figura 3A) e EPR pouco diferenciado (Figura 3B), pois a qualidade do modelo celular impacta sobremaneira a qualidade da segmentação adequada da imagem.
Dois dos três marcadores descritos no protocolo, Nile Red e BODIPY, são identificados como pequenos pontos circulares que são nitidamente brilhantes nas imagens fluorescentes (Figura 5 e Figura 6). Uma imagem "positiva" do protocolo seria uma identificação apropriada desses depósitos distintos (Figura 5A-D e Figura 5E-H). Um resultado "negativo" mostraria segmentação incorreta da imagem ao confundir fluorescência de fundo com depósito, seja pela fraca coloração (Figura 6A-C e Figura 6D-F) ou pela alta intensidade de fundo (Figura 6G-I).
Os depósitos de APOE têm uma variedade de tamanhos e formas, parecendo mais ovais ou irregulares do que os depósitos circulares de Nile Red e BODIPY. Esses depósitos também são menos pontuais, e a intensidade de sinal pode diferir entre os depósitos devido a variações na permeabilização da amostra. A identificação correta identificará cada depósito, incluindo aqueles que estão menos saturados (Figura 5I-L), enquanto a segmentação incorreta não captará esses depósitos (Figura 6J-L). Portanto, é importante otimizar os métodos de coloração e imagem para evitar variações drásticas. Uma maneira de fazer isso é prestando muita atenção às etapas de permeabilização da amostra durante a imunomarcação. Para otimizar o sinal fluorescente, as células podem ser lisadas antes da fixação e imunomarcação para APOE, o que resulta em saturação uniforme e melhor segmentação dos depósitos de APOE.
Também são fornecidas imagens segmentadas de células amadurecidas em uma plataforma de cultura diferente de uma placa de 96 poços. O software LipidUNet foi executado em imagens de células cultivadas em um transwell, e enquanto os depósitos lipídicos são limiares, os poros na membrana do transwell também o são (Figura 6M-O). Devido à semelhança na forma e tamanho, o software LipidUNet em sua forma atual irá mascarar os depósitos lipídicos e transwell poros indiscriminadamente.
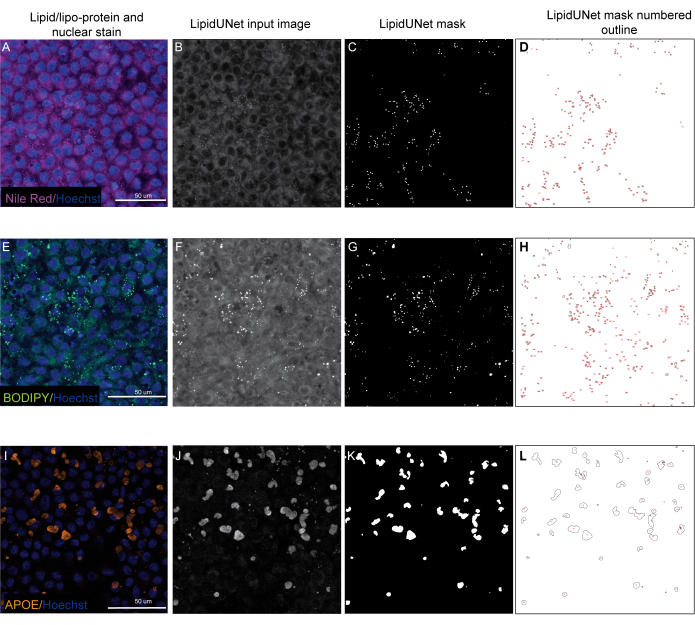
Figura 5: Resultados representativos. (A,E,I) 96 poços de RPE são corados com coloração nuclear Hoechst (azul) e vermelho Nilo (magenta), BODIPY (verde) ou APOE (laranja) e são as projeções de intensidade máxima de uma pilha Z. (B,F,J) As imagens de entrada em escala de cinza para o software LipidUNet após o processamento da imagem. (C,G,K) Máscaras geradas pelo LipidUNet, onde todos os depósitos são identificados corretamente. (D,H,L) Os contornos de cada partícula mascarada são numerados. Esses rótulos permitem conectar cada partícula na imagem a uma entrada no spreadheet com os dados brutos. (A-D) mostra a coloração de vermelho do Nilo, e o software é capaz de reconhecer os depósitos contra o fundo com precisão, apesar de um sinal mais fraco. (E-H) mostra um forte contraste entre o sinal BODIPY e o fundo, o que é ideal. LipidUNet identifica corretamente cada depósito na imagem. (I-L) mostra um forte sinal APOE e representa a variabilidade da saturação do sinal que é frequentemente observada com esta coloração. No entanto, a segmentação das imagens é capaz de identificar as bordas de cada depósito de APOE. Clique aqui para ver uma versão maior desta figura.
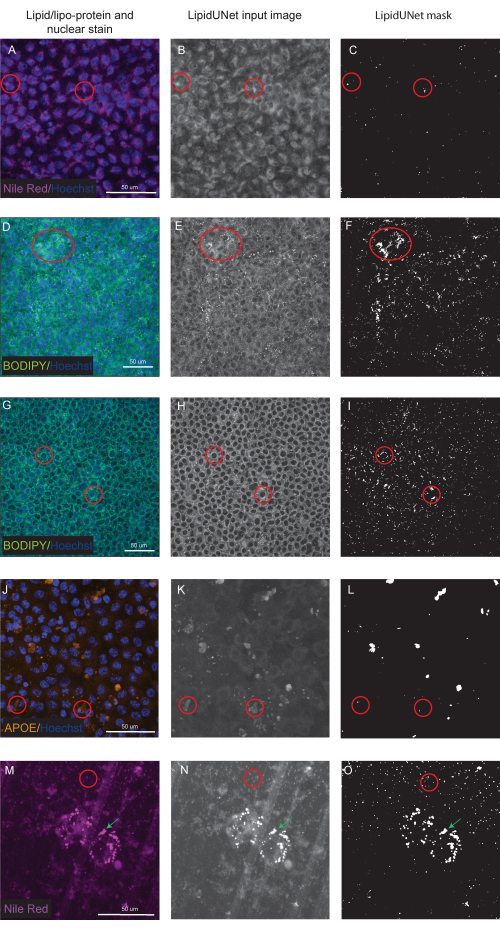
Figura 6: Resultados subótimos. (A,D,G,J,M) 96-bem chapeado RPE são corados com coloração nuclear Hoechst (azul) e vermelho Nilo (magenta), BODIPY (verde) ou APOE (laranja) e são as projeções de intensidade máxima de uma pilha Z. (B,E,H,K,N) As imagens de entrada em escala de cinza para o software LipidUNet após o processamento da imagem. (C, F, I, L, O) As máscaras incorretas geradas pelo LipidUNet. Círculos vermelhos indicam onde o software identificou incorretamente um depósito lipídico. (A-C) O processamento do Nile Red está incorreto porque o software identificou a coloração de fundo como um depósito. Isso pode acontecer com mais frequência quando há fundo alto, mas poucos depósitos lipídicos na imagem. Dois exemplos de coloração BODIPY são mostrados: uma imagem de baixa qualidade devido à coloração BODIPY fraca (D-F) e (G - I) um sinal BODIPY forte com alto fundo. Em ambos os casos, o software é incapaz de distinguir pequenos depósitos lipídicos circulares do anel circular de fundo que envolve o núcleo. Embora a coloração e a imagem devam ser otimizadas para evitar esses erros, a versão mais recente do LipidUNet foi amplamente melhorada para essas imagens. (J-L) Segmentação APOE incorreta. Como os depósitos são mais variáveis em tamanho e saturação de sinal, o software tem dificuldade em reconhecer alguns depósitos. (M-O) RPE semeado em um transwell e corado com Nile Red. Uma fatia da pilha Z é mostrada aqui com depósitos lipídicos vermelho-do-nilo e poros transwell. O software é incapaz de distinguir entre os dois, como mostrado pelo círculo vermelho contendo poros transwell e a seta verde apontando para depósitos de Nile Red. Clique aqui para ver uma versão maior desta figura.

Figura 7: Comparação da ferramenta de máscara. (A,B,C) PSE de 96 poços com quantidades variáveis de deposição lipídica são identificados com vermelho do Nilo (vermelho). As imagens são mascaradas usando três diferentes métodos de mascaramento comuns, Find Maxima, Max Entropy e Renyi Entropy, e comparadas com a máscara gerada pelo LipidUNet. A imagem original é acompanhada por uma contagem manual dos depósitos lipídicos, enquanto as máscaras exibem as contagens previstas por cada método de segmentação. A taxa de erro média foi calculada para cada método de segmentação usando a seguinte fórmula: média[(|Contagem Prevista - Contagem Manual|/Contagem Manual) x 100]. A máscara gerada pelo LipidUNet identifica com mais precisão os depósitos lipídicos nas imagens com deposição variável quando comparada a outros métodos de mascaramento (Taxas de erro médias: 23% LipidUnet, 1164% Find Maxima, 851% Max Entropy, 203% Renyi Entropy). Clique aqui para ver uma versão maior desta figura.
| Componente | Número do gato | Estoque Conc. | Final Conc. | Ml |
| MEM alfa | 12571-063 | NA | 500 | |
| Suplemento N2 | 17502-048 | NA | 1% | 5 |
| FBS inativado por calor | SH30071.03 | NA | 5% | 25 |
| NMEM NEAA | 11140-050 | 10mM | 0.01mM | 5 |
| Piruvato de Sódio | 11360-070 | 100mM | 1mM | 5 |
| Penicilina-estreptomicina | 15140-122 | 10000u/mL | 100U/mL | 5 |
| Taurina | T4571 | 50mg/mL | 250ug/mL | 2.5 |
| Hidrocortisona | H6909 | 18,1mg/L | 20ug/l | 0.553 |
| T3 | T5516 | 20ug/l | 0.013ug/l | 0.33 |
| Volume total, mL | 548.383 | |||
Tabela 1: Composição do reagente PSE-MM. Uma lista de reagentes e concentrações ótimas para PSE-MM.
Discussion
Este protocolo fornece um método para marcar, imagear e quantificar eficientemente depósitos lipídicos em modelos de doenças in vitro monogênicas e poligênicas para doenças oculares degenerativas. O software baseado em IA, LipidUNet, pode ser aplicado a três marcadores lipídicos comuns, APOE, Nile Red e BODIPY, e fornece um método rápido e automático de análise que permite que a quantificação seja padrão e imparcial.
A principal limitação do LipidUNet é o fato de que o conjunto de dados de treinamento para a IA foi limitado a imagens de aumento de 40x de células cultivadas em uma placa de 96 poços. Como resultado do conjunto de imagens de treinamento, o LipidUNet, em sua forma atual, limita-se a analisar imagens de aumento de 40x. O software pode ser usado para analisar imagens de 40x de células cultivadas em outras superfícies de cultura além de uma placa de 96 poços, mas deve-se ter cuidado ao examinar as máscaras de saída geradas para verificar o limiar preciso pelo software. Mais conjuntos de imagens (em diferentes ampliações) serão necessários para expandir o escopo de quais amostras/imagens ele pode analisar.
O protocolo tem várias etapas críticas. Na etapa do marcador lipídico, o usuário deve confirmar que seu composto de rotulagem escolhido (BODIPY, APOE, Nile Red) rotulou sua amostra de forma eficaz. As células maduras do EPR são frequentemente fortemente pigmentadas, o que pode prejudicar o sinal fluorescente da imunomarcação de anticorpos. Quando o sinal de fluorescência é fraco ou quando há muita coloração de fundo, LipidUNet não pode discernir gotículas lipídicas com precisão. Por uma razão semelhante, configurações de aquisição adequadamente selecionadas para a etapa de aquisição automática do protocolo devem ser usadas. Se as imagens adquiridas forem de baixa qualidade, o LipidUNet terá dificuldades para mascarar adequadamente as imagens e, portanto, a quantificação será imprecisa (Figura 6A-L). Finalmente, o pós-processamento das imagens é um passo importante, pois o LipidUNet possui requisitos específicos para que o software funcione.
Quando comparado a fluxos de trabalho para análise lipídica que usam limiares manuais ou técnicas que envolvem limiares automáticos em softwares como Fiji, o LipidUNet oferece uma segmentação não tendenciosa e confiável em imagens com deposição lipídica variável, refletida por uma pequena taxa de erro na identificação de partículas lipídicas (Figura 7). O software permite a entrada pelo usuário de imagens adicionais de treinamento, permitindo a análise de conjuntos de imagens além daquelas que utilizam uma objetiva de aumento de 40x ou mesmo aquelas que utilizam um marcador lipídico diferente, conforme descrito no protocolo. No futuro, o software será treinado para analisar imagens 3D para que a quantificação do volume de depósito lipídico seja possível. Doenças oculares degenerativas que implicam a deposição lipídica como um dos principais contribuintes para a patologia são prevalentes, e prevê-se que os casos aumentem à medida que a população idosa está se expandindo13. Modelos de doença precisos e ferramentas de análise eficientes, como delineamos neste protocolo, permitirão o desenvolvimento de novas intervenções terapêuticas.
Disclosures
Sem divulgações.
Acknowledgements
Agradecemos ao núcleo de histologia do National Eye Institute (NEI) pelo uso do sistema confocal Zeiss. Este trabalho foi apoiado por fundos do NEI IRP (número de processo ZIA EY000533-04).
Materials
| Name | Company | Catalog Number | Comments |
| 0.22 µm Steriflip filter system | EMD Millipore | SCGP00525 | |
| 1x Dulbecco's Phosphate Buffered Saline | Gibco | 14190-144 | |
| 3,3',5-Triiodo-L-thyronine | Sigma | T5516 | |
| Albumin Bovine, Fraction V | MP Biomedical | 160069 | |
| Alexa Fluor 555 rabbit anti-goat IgG (H+L) | Invitrogen | A21431 | APOE secondary antibody |
| APOE primary antibody | Millipore Sigma | AB947 | |
| BODIPY 493/503 | Invitrogen | D3922 | Protect from light |
| Complement competent human serum | Millipore Sigma | S1-LITER | |
| CTS N2 Supplement | Life Technologies | A13707-01 | |
| Fetal Bovine Serum | Hyclone | SH30071.03 | |
| Fluoromount-G | SouthernBiotech | 0100-01 | Slide mounting media |
| Glass Cover Slips #1 1/2 22 mm x 22 mm | Electron Microscopy Sciences | 72204-01 | |
| Glass Microscope Slide 25 mm x 75 mm- 1.2 mm Thick | Electron Microscopy Sciences | 71870-01 | |
| Hydrocortisone | Sigma | H0396 | |
| MEM Alpha | Life Technologies | 12571-063 | |
| MEM non-essential Amino Acids | Life Technologies | 11140 | |
| Nile Red | Sigma | 72485-100MG | Protect from light |
| Paraformaldehyde 16% Solution, EM Grade | Electron Microscopy Sciences | 15710 | |
| Penicillin-Strep | Life Technologies | 15140-148 | |
| Phosphate Buffered Saline 10x | Gibco | 70011-044 | |
| Rod Outer Segments (OS) | InVision Bioresources | 98740 | |
| Sodium bicarbonate | Sigma Aldrich | S5761 | |
| Sodium Pyruvate | Life Technologies | 11360-070 | |
| Sucrose | Sigma Aldrich | S1888 | |
| SYBR Green Master Mix | Bio-Rad | 1725274 | |
| Taurine | Sigma | T0625 | |
| Triton X-100 | Sigma | 9002-93-1 | |
| Tween 20 Ultrapure | Affymetrix | 9005-64-5 | |
| Vitronectin | Life Technologies | A14701SA | |
| Y-27632 dihydrochloride | R&D Systems | 1254 |
References
- Kanow, M. A., et al. Biochemical adaptations of the retina and retinal pigment epithelium support a metabolic ecosystem in the vertebrate eye. eLife. 6, e28899 (2017).
- Adijanto, J., et al. The retinal pigment epithelium utilizes fatty acids for ketogenesis. The Journal of Biological Chemistry. 289 (30), 20570-20582 (2014).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Curcio, C. A. Soft drusen in age-related macular degeneration: Biology and targeting via the oil spill strategies. Investigative Ophthalmology & Visual Science. 59 (4), AMD160-AMD181 (2018).
- Cankova, Z., Huang, J. D., Kruth, H. S., Johnson, M. Passage of low-density lipoproteins through Bruch's membrane and choroid. Experimental Eye Research. 93 (6), 947-955 (2011).
- Curcio, C. A., et al. Esterified and unesterified cholesterol in drusen and basal deposits of eyes with age-related maculopathy. Experimental Eye Research. 81 (6), 731-741 (2005).
- Miyagishima, K. J., et al. AMPK modulation ameliorates dominant disease phenotypes of CTRP5 variant in retinal degeneration. Communications Biology. 4 (1), 1360 (2021).
- Sharma, R., et al. Epithelial phenotype restoring drugs suppress macular degeneration phenotypes in an iPSC model. Nature Communications. 12 (1), 7293 (2021).
- Farnoodian, M., et al. Cell-autonomous lipid-handling defects in Stargardt iPSC-derived retinal pigment epithelium cells. Stem Cell Reports. 17 (11), 2438-2450 (2022).
- Hallam, D., et al. An induced pluripotent stem cell patient specific model of complement factor H (Y402H) polymorphism displays characteristic features of age-related macular degeneration and indicates a beneficial role for UV light exposure. Stem Cells (Dayton, Ohio). 35 (11), 2305-2320 (2017).
- Sharma, R., Bose, D., Montford, J., Ortolan, D., Bharti, K. Triphasic developmentally guided protocol to generate retinal pigment epithelium from induced pluripotent stem cells. STAR Protocols. 3 (3), 101582 (2022).
- Issa, P. C., Barnard, A. R., Herrmann, P., Washington, I., MacLaren, R. E. Rescue of the Stargardt phenotype in Abca4 knockout mice through inhibition of vitamin A dimerization. Proceedings of the National Academy of Sciences of the United States of America. 112 (27), 8415-8420 (2015).
- GBD 2019 Blindness and Vision Impairment Collaborators. Blindness and Vision Impairment Collaborators. Causes of blindness and vision impairment in 2020 and trends over 30 years, and prevalence of avoidable blindness in relation to VISION 2020: the Right to Sight: an analysis for the Global Burden of Disease Study. Lancet Global Health. 9 (2), e144-e160 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved