Usando imagens STED de células vivas para visualizar a ultraestrutura da membrana interna mitocondrial em modelos de células neuronais
In This Article
Summary
Este protocolo apresenta um fluxo de trabalho para a propagação, diferenciação e coloração de células SH-SY5Y cultivadas e neurônios primários do hipocampo de ratos para visualização e análise da ultraestrutura mitocondrial usando microscopia de depleção de emissão estimulada (STED).
Abstract
As mitocôndrias desempenham muitos papéis essenciais na célula, incluindo produção de energia, regulação da homeostase de Ca2+ , biossíntese lipídica e produção de espécies reativas de oxigênio (EROs). Esses processos mediados por mitocôndrias assumem papéis especializados nos neurônios, coordenando o metabolismo aeróbico para atender às altas demandas energéticas dessas células, modulando a sinalização de Ca2+ , fornecendo lipídios para o crescimento e regeneração do axônio e ajustando a produção de ROS para o desenvolvimento e a função neuronal. A disfunção mitocondrial é, portanto, um fator central nas doenças neurodegenerativas. A estrutura e a função mitocondrial estão inextricavelmente ligadas. A membrana interna morfologicamente complexa com dobras estruturais chamadas cristas abriga muitos sistemas moleculares que realizam os processos de assinatura da mitocôndria. As características arquitetônicas da membrana interna são ultraestruturais e, portanto, muito pequenas para serem visualizadas pela microscopia resolvida tradicional limitada por difração. Assim, a maioria dos conhecimentos sobre a ultraestrutura mitocondrial provém da microscopia eletrônica em amostras fixas. No entanto, tecnologias emergentes em microscopia de fluorescência de super-resolução agora fornecem resolução de até dezenas de nanômetros, permitindo a visualização de características ultraestruturais em células vivas. A imagem de super-resolução, portanto, oferece uma capacidade sem precedentes de obter imagens diretas de detalhes finos da estrutura mitocondrial, distribuições de proteínas em nanoescala e dinâmica de cristas, fornecendo novos insights fundamentais que ligam as mitocôndrias à saúde e à doença humanas. Este protocolo apresenta o uso de microscopia de super-resolução de depleção de emissão estimulada (STED) para visualizar a ultraestrutura mitocondrial de células vivas de neuroblastoma humano e neurônios primários de ratos. Este procedimento está organizado em cinco seções: (1) crescimento e diferenciação da linhagem celular SH-SY5Y, (2) isolamento, plaqueamento e crescimento de neurônios primários do hipocampo de ratos, (3) procedimentos para coloração de células para imagens STED vivas, (4) procedimentos para experimentos STED de células vivas usando um microscópio STED para referência, e (5) orientação para segmentação e processamento de imagens usando exemplos para medir e quantificar características morfológicas da membrana interna.
Introduction
As mitocôndrias são organelas eucarióticas de origem endossimbiótica responsáveis pela regulação de vários processos celulares chave, incluindo metabolismo intermediário e produção de ATP, homeostase iônica, biossíntese lipídica e morte celular programada (apoptose). Essas organelas são topologicamente complexas, contendo um sistema de membrana dupla que estabelece múltiplos subcompartimentos1 (Figura 1A). A membrana mitocondrial externa (MOO) faz interface com o citosol e estabelece contatos diretos interorganelas 2,3. A membrana mitocondrial interna (MIM) é uma membrana conservadora de energia que mantém gradientes iônicos armazenados principalmente como um potencial de membrana elétrica (ΔΨm) para conduzir a síntese de ATP e outros processos que requerem energia 4,5. O IMM é subdividido em membrana de contorno interno (IBM), que é intimamente aprimida ao OMM, e estruturas salientes chamadas cristas que são ligadas pela membrana de cristas (CM). Esta membrana delineia o compartimento mais interno da matriz a partir do espaço intracristal (ICS) e do espaço intermembrana (IMS).
As mitocôndrias possuem uma morfologia dinâmica baseada em processos contínuos e equilibrados de fissão e fusão que são governados por mecanoenzimas da superfamília dasdinaminas6. A fusão permite maior conectividade e formação de redes reticulares, enquanto a fissão leva à fragmentação mitocondrial e possibilita a remoção de mitocôndrias danificadas pela mitofagia7. A morfologia mitocondrial varia de acordo com o tipode tecido 8 e o estágio de desenvolvimento9 e é regulada para permitir que as células se adaptem a fatores como necessidades energéticas 10,11 e estressores 12. Características morfométricas padrão das mitocôndrias, como a extensão da formação da rede (interconectada vs. fragmentada), perímetro, área, volume, comprimento (relação de aspecto), arredondamento e grau de ramificação, podem ser medidas e quantificadas por microscopia óptica padrão, pois os tamanhos dessas características são maiores que o limite de difração da luz (~200 nm)13.
A arquitetura das cristas define a estrutura interna das mitocôndrias (Figura 1B). A diversidade de morfologias das cristas pode ser amplamente categorizada em plana (lamelar ou discoidal) ou tubular-vesicular14. Todas as cristas se fixam na IBM através de estruturas tubulares ou semelhantes a slots, denominadas cristae junctions (CJs) que podem servir para compartimentar o IMS do ICS e o IBM do CM15. A morfologia das cristas é regulada por complexos proteicos chave do IMM, incluindo (1) o sítio de contato mitocondrial e o sistema organizador de cristas (MICOS) que reside nas CJs e estabiliza os contatos IMM-OMM 16, (2) a GTPase da atrofia óptica 1 (OPA1) que regula o remodelamento das cristas17,18,19 e (3) a F1FO ATP sintase que forma conjuntos oligoméricos estabilizadores nas pontas das cristas (CTs)20, 21º. Além disso, o IMM é enriquecido em fosfolipídios não bicamadas, fosfatidiletanolamina e cardiolipina, que estabilizam o IMM altamente curvo22. As cristas também são dinâmicas, demonstrando alterações morfológicas sob diversas condições, como diferentes estados metabólicos 23,24, com diferentes substratos respiratórios 25, sob inanição e estresse oxidativo 26,27, com apoptose 28,29 e com o envelhecimento 30. Recentemente, foi demonstrado que as cristas poderiam sofrer grandes eventos de remodelação em uma escala de tempo de segundos, ressaltando sua natureza dinâmica31. Várias características das cristas podem ser quantificadas, incluindo dimensões das estruturas dentro das cristas individuais (por exemplo, largura da CJ, comprimento e largura da crista) e parâmetros que relacionam cristas individuais com outras estruturas (por exemplo, espaçamento intra-cristas e ângulo incidente das cristas em relação à OMM)32. Estes parâmetros quantificáveis das cristas mostram uma correlação direta com a função. Por exemplo, a extensão da produção de ATP mitocondrial está positivamente relacionada com a abundância de cristas, quantificada como densidade de cristas ou número de cristas normalizado para outra característica (por exemplo, cristas por área de OMM)33,34,35. Como a morfologia do IMM é definida por características nanométricas, compreende a ultraestrutura mitocondrial, que requer técnicas de imagem que forneçam resolução maior que o limite de difração da luz. Como descrito a seguir, tais técnicas incluem microscopia eletrônica e microscopia de super-resolução (nanoscopia).
As células neurais e gliais do sistema nervoso central (SNC) são particularmente dependentes da função mitocondrial. Em média, o cérebro constitui apenas 2% do peso corporal total, mas utiliza 25% da glicose corporal total e é responsável por 20% do consumo de oxigênio corporal, tornando-o vulnerável a prejuízos no metabolismo energético36. As doenças neurodegenerativas progressivas (DNs), incluindo a doença de Alzheimer (DA), a esclerose lateral amiotrófica (ELA), a doença de Huntington (DH), a esclerose múltipla (EM) e a doença de Parkinson (DP), são algumas das patologias mais extensamente estudadas até o momento, com esforços de pesquisa que vão desde a compreensão dos fundamentos moleculares dessas doenças até a busca de potenciais intervenções e prevenção terapêutica. Os DEs estão associados ao aumento do estresse oxidativo originado, em parte, por espécies reativas de oxigênio (EROs) geradas pela cadeia de transporte de elétrons mitocondrial (ETC)37, bem como a alterações no manuseio do cálcio mitocondrial 38 e no metabolismo lipídico mitocondrial39. Essas alterações fisiológicas são acompanhadas por defeitos observados na morfologia mitocondrial que estão associados com DA 40,41,42,43,44, ELA 45,46, HD47,48,49, EM 50 e DP 51,52,53. Esses defeitos estruturais e funcionais podem ser acoplados por complexas relações de causa e efeito. Por exemplo, dado que a morfologia das cristas estabiliza as enzimas OXPHOS54, as ROS mitocondriais não são geradas apenas pela ETC, mas também atuam para danificar a infraestrutura na qual a ETC reside, promovendo um ciclo de ROS feed-forward que aumenta a suscetibilidade a danos oxidativos. Além disso, foi demonstrado que a desorganização das cristas desencadeia processos como a liberação de DNA mitocondrial (mtDNA) e vias inflamatórias ligadas a distúrbios autoimunes, metabólicos e relacionados à idade55. Portanto, a análise da estrutura mitocondrial é a chave para uma compreensão completa dos DEs e seus fundamentos moleculares.
Métodos populares de visualização de cristas, incluindo microscopia eletrônica de transmissão, tomografia eletrônica e tomografia crioeletrônica (crio-TE), e tomografia de raios-X, em particular a tomografia de raios X criomole, têm revelado achados importantes e trabalhos com uma variedade de tipos de amostras56,57,58,59,60. Apesar dos recentes avanços em direção à melhor observação da ultraestrutura organelar, esses métodos ainda vêm com a ressalva de exigir a fixação da amostra e, portanto, não podem capturar diretamente a dinâmica em tempo real das cristas. A microscopia de fluorescência de super-resolução, particularmente nas formas de microscopia de iluminação estruturada (SIM), microscopia de reconstrução óptica estocástica (STORM), microscopia de localização fotoativada (PALM), microscopia de expansão (ExM) e microscopia de depleção de emissão estimulada (STED), tornaram-se formas populares de visualização de estruturas que requerem resolução abaixo do limite de difração que restringe os métodos clássicos de microscopia óptica. Quando a ExM é usada em conjunto com outra técnica de super-resolução, os resultados são impressionantes, mas a amostra deve ser fixada e corada em gel61. Em comparação, SIM, PALM/STORM e STED foram usados com sucesso com amostras vivas, e novos e promissores corantes que geralmente coram o IMM fornecem uma abordagem nova e fácil para imagens ao vivo da dinâmica das mitocôndrias cristas 62,63,64,65,66. Avanços recentes em corantes vivos para imagens STED melhoraram o brilho e a fotoestabilidade do corante, e esses corantes têm como alvo o IMM em um grau mais alto de especificidade do que seus antecessores. Esses desenvolvimentos permitem a coleta de experimentos de longo prazo em timelapse e z-stack com imagens de super-resolução, abrindo as portas para uma melhor análise de células vivas da ultraestrutura e dinâmica mitocondrial.
Neste estudo, são apresentados protocolos para obtenção de imagens de células SH-SY5Y indiferenciadas e diferenciadas coradas com o corante PKmito Orange (PKMO) usando STED63. A linhagem celular SH-SY5Y é um derivado subclonado três vezes da linhagem celular parental, SK-N-SH, gerada a partir de biópsia de medula óssea de neuroblastoma metastático67,68,69,70. Essa linhagem celular é um modelo in vitro comumente utilizado na pesquisa de DN, particularmente em doenças como DA, DH e DP, nas quais a disfunção mitocondrial está fortemente implicada10,43,71,72,73. A capacidade de diferenciar células SH-SY5Y em células com fenótipo semelhante a um neurônio por meio da manipulação de meios de cultura tem se mostrado um modelo adequado para pesquisas em neurociência sem depender de células neuronais primárias10,74. Nesse protocolo, ácido retinóico (AR) foi adicionado ao meio de cultura celular para induzir a diferenciação de células SH-SY5Y. A AR é um derivado da vitamina A e demonstrou regular o ciclo celular e promover a expressão de fatores de transcrição que regulam a diferenciação neuronal75. Um protocolo para cultivo e imagem de células vivas de neurônios isolados do hipocampo de ratos também é fornecido. Demonstrou-se que o hipocampo é afetado pela degeneração mitocondrial e, juntamente com o córtex, desempenha um papel importante no envelhecimento e na DN76,77,78,79,80.
Protocol
1. Propagação e diferenciação de células SH-SY5Y
- Preparação de meios para crescimento e manutenção celular
- Preparar meio de águia modificado de Dulbecco completo e com alto teor de glicose (DMEM, 4,5 g/L de D-glicose, 4 mM de L-glutamina, 110 mg/L de piruvato de sódio) suplementado com um antibiótico-antimicótico final de 1% (v/v) (10.000 unidades/mL de penicilina, 10.000 μg/mL de estreptomicina e 25 μg/mL de anfotericina B) e quantidades variáveis de soro fetal bovino (FBS) (ver Tabela de Materiais). As quantidades de FBS nos meios de diferenciação variam entre 10%, 5% ou 2% (v/v) de SFB final.
- Manutenção celular
- Manter as células em DMEM suplementado com FBS a 10% (v/v) e colocá-las a 37 °C e 5% CO2, em seguida, semear em DMEM contendo 5% (v/v) de SFB para diferenciação. Os estoques de células congeladas foram armazenados em FBS com 10% (v/v) de dimetilsulfóxido (DMSO) a 1-2 x 107 células/mL.
- Preparação de ácido retinóico (AR)
- Dissolver 7,51 mg de all-trans-RA (ver Tabela de Materiais) em 5 mL de etanol 95% recém-preparado para obter 5 mM de solução-mãe. Verificar a concentração com absorbância a 350 nm (ɛ = 44.300 M-1 cm-1), obtida da ficha de informações do produto do protocolo81 do fabricante, utilizando uma diluição da solução-mãe a 5 μM em etanol. Conservar 5 mM de existências protegidas da luz a 4 °C durante um período máximo de 6 semanas.
- Preparação de poli-D-lisina para revestimento de lamínulas
NOTA: O protocolo de produto poli-D-lisina (PDL), encontrado na seção Documentos e downloads do site do fornecedor82, fornece informações para revestir uma variedade de vasos de cultura.- Este protocolo inclui volumes baseados em um recipiente com câmara de 2 poços com uma área de 4 cm2 por poço com fundos de vidro de borossilicato estéril #1,5 (ver Tabela de Materiais). Diluir a solução-mãe de PDL duas vezes para 50 μg/mL com PBS de Dulbecco (DPBS; sem cálcio, sem magnésio).
NOTA: #1.5 ou #1.5H coverglass são ambas espessuras aceitáveis, que são essenciais para a qualidade da imagem. Outras espessuras induzirão aberração esférica e devem ser evitadas.
- Este protocolo inclui volumes baseados em um recipiente com câmara de 2 poços com uma área de 4 cm2 por poço com fundos de vidro de borossilicato estéril #1,5 (ver Tabela de Materiais). Diluir a solução-mãe de PDL duas vezes para 50 μg/mL com PBS de Dulbecco (DPBS; sem cálcio, sem magnésio).
- Revestimento de deslizamento com PDL
NOTA: As lamínulas podem ser expostas à luz ultravioleta (UV) por 10-15 minutos em um armário de biossegurança para esterilização adicional.- Aplicar 1,2 mL de solução de PDL 50 μg/mL em cada poço de lamínulas estéreis em um gabinete de cultura celular e incubar à temperatura ambiente por 1 h. Retire a solução de PDL e lave três vezes com 3,6 mL de água destilada. Depois de completar a lavagem final, deixe a câmara revestida secar por 2 h exposta ao ar antes de enxaguar e usar imediatamente ou armazenar com um recipiente hermético a 4 °C por até 2 semanas.
NOTA: Enxágue bem as tampas, pois o excesso de PDL pode ser tóxico para as células.
- Aplicar 1,2 mL de solução de PDL 50 μg/mL em cada poço de lamínulas estéreis em um gabinete de cultura celular e incubar à temperatura ambiente por 1 h. Retire a solução de PDL e lave três vezes com 3,6 mL de água destilada. Depois de completar a lavagem final, deixe a câmara revestida secar por 2 h exposta ao ar antes de enxaguar e usar imediatamente ou armazenar com um recipiente hermético a 4 °C por até 2 semanas.
- Diferenciação de células SH-SY5Y com AR
NOTA: Não utilize células acima da passagem 15. As células são passadas em 80%-90% de confluência. Os procedimentos de diferenciação diferem, mas seguem etapas semelhantes. Diferenciação adicional de neuroblastomas para neurônios maduros é obtida com tratamento adicional com fator neurotrófico derivado do cérebro (BDNF)68,83,84,85, mas não foi realizada neste protocolo.
OPCIONAL: Estabelecer células por pelo menos 24 h antes da semeadura em vidro de cobertura. Para preparar as células a partir de estoques congelados, descongele rapidamente 1 mL de frasco congelado de células e adicione a 9 mL de meio pré-aquecido suplementado com FBS a 10%, depois gire para baixo a 350 x g por 10 min (à temperatura ambiente) e descarte o sobrenadante para remover o DMSO. Ressuspender o pellet de células em 5 mL de meio pré-aquecido e células de sementes em frasco T-25. Quando as células atingirem 80%-90% de confluência, passe as células por contagem e semeadura para diferenciação, quando aplicável.- Dia 0: Células da semente.
- Semeando as células numa tampa de câmara a partir de existências congeladas ou de um balão de trabalho. Use uma densidade de semeadura de 1,5 x 104 células/cm2.
NOTA: Um único poço em um vidro de cobertura padrão de 2 poços com 4 cm2 de área de cultura exigirá 6,0 x 104 células. As células que permanecerão indiferenciadas devem ser semeadas com DMEM suplementado com 10% (v/v) de SFB, e as células que serão diferenciadas devem ser semeadas com DMEM suplementado com 5% (v/v) de SFB.
- Semeando as células numa tampa de câmara a partir de existências congeladas ou de um balão de trabalho. Use uma densidade de semeadura de 1,5 x 104 células/cm2.
- Dia 1: Iniciar o tratamento de diferenciação da AR.
- Preparar DMEM suplementado com 5% (v/v) de SFB, 1% (v/v) antibiótico-antimicótico e uma concentração final de 10 μM de RA ou etanol do mesmo volume de aditivo para servir como controle do veículo para este procedimento de diferenciação. Remova o meio da tampa de câmara usada para semeadura, enxágue com 1x PBS e adicione o novo DMEM aos poços.
- Dia 3: Substitua o meio por meios frescos contendo AR ou etanol.
- Remover o meio do Dia 1 e substituir por meio fresco suplementado com 2% (v/v) de SFB, 1% (v/v) antibiótico-antimicótico e 10 μM de AR ou 95% de etanol do mesmo volume de aditivo para servir como controle do veículo para este procedimento de diferenciação. Remova o meio da tampa de câmara usada para semeadura, enxágue com 1x PBS e adicione novos meios aos poços.
- Dia 6: Realizar a imagem das células.
NOTA: Os tempos de diferenciação celular variam de acordo com o protocolo, mas seis dias de exposição à AR são suficientes para induzir um fenótipo semelhante a um neurônio em células SH-SY5Y86.- Realizar imagens ao vivo, com detalhes nas seções 3 e 4 (Figura 2).
- Dia 0: Células da semente.
2. Cultura primária de neurônios hipocampais de ratos
- Preparação de materiais para isolamento de neurônios hipocampais de ratos.
- Preparar DMEM fresco suplementado.
- Filtrar-esterilizar uma mistura de DMEM (glicose alta, sem piruvato de sódio) suplementada com FBS inativado pelo calor a 10% (v/v), solução de piruvato de sódio a 1% (v/v) e penicilina-estreptomicina a 1% (10.000 U/mL). Conservar até 2 semanas a 4 °C.
- Preparar meios de crescimento de neurônio frescos suplementados.
- Filtrar-esterilizar uma mistura de meios de crescimento de neurônios suplementados com 2% (v/v) suplemento B27, 0,25% (v/v) suplemento de glutamina e 1% (v/v) penicilina-estreptomicina (ver Tabela de Materiais). Conservar até 2 semanas a 4 °C.
- Preparar DMEM fresco suplementado.
- Preparar a cultura primária do neurônio hipocampal.
- Preparar a cultura primária do neurônio hipocampal seguindo o trabalho publicado anteriormente87 e a partir do protocolo do produto no local do fabricante a partir do qual o hipocampo dissecado do rato E18 é obtido88 (ver Tabela de Materiais). Esse protocolo resulta em uma população majoritariamente neuronal com <2% de astrócitos.
NOTA: O meio de hibernação com o qual este tecido é enviado será usado para etapas futuras neste protocolo. Não descarte ele. - Preparar materiais e meios para dissociação tecidual.
- Chame uma pipeta Pasteur para diminuir o diâmetro da abertura e guarde-a em papel alumínio para evitar contaminação até o uso. Pré-aqueça o DMEM preparado, a solução tamponada de sal de 1X Hank (HBSS) e o meio de crescimento de neurônios a 37 °C. Adicionar 2 flocos de DNAase com pinça estéril a um tubo cônico de 15 mL.
- Realizar dissociação tecidual.
- Remover o máximo possível do meio de hibernação no hipocampo dissecado de ratos E18 antes de colocar o tecido no tubo cônico de 15 mL contendo DNAase e incubar brevemente a 37 °C. Adicionar 900 μL de 1X HBSS seguido de 100 μL de tripsina a 0,5%. Incubar o tecido a 37 °C durante 15 minutos.
NOTA: As placas revestidas com PDL podem ser retiradas do armazenamento e colocadas em uma incubadora até o uso durante este tempo de incubação.
- Remover o máximo possível do meio de hibernação no hipocampo dissecado de ratos E18 antes de colocar o tecido no tubo cônico de 15 mL contendo DNAase e incubar brevemente a 37 °C. Adicionar 900 μL de 1X HBSS seguido de 100 μL de tripsina a 0,5%. Incubar o tecido a 37 °C durante 15 minutos.
- Realizar homogeneização tecidual e contagem celular.
- Após incubação com tripsina, retirar o meio e adicionar 1 mL de meio de hibernação-DNAase pré-aquecido da etapa anterior ao tecido e homogeneizar com a pipeta de Pasteur. A mídia parecerá opaca e, em seguida, gradualmente clara à medida que a homogeneização continua.
- Adicione neurônios dissociados a um novo tubo com 4 mL de DMEM pré-aquecido e, em seguida, conte as células usando um contador de células.
- Realizar plaqueamento celular e crescimento de células primárias.
- Células de placa a uma densidade de aproximadamente 1,65 x 104 células/cm2 em DMEM. Para uma cobertura de vidro com câmara de 2 poços (4 cm 2), semeie 65.000 – 70.000 células por poço com2 mL de DMEM. Incubar as células a 37 °C e 5% CO 2 durante2 h antes de verificar a aderência. Quando as células começarem a aderir, remova 1 mL de meio e substitua-o pelo mesmo volume de meio de hibernação, agite suavemente para misturar. Uma vez que o meio é misturado, repita este processo e remova metade do meio presente e substitua pelo mesmo volume de meio de crescimento de neurônio, em seguida, misture suavemente.
NOTA: O dia do plaqueamento é considerado dia in vitro (DIV) 0, e as células estão prontas para obter imagens na DIV 7 (Figura 2).
- Células de placa a uma densidade de aproximadamente 1,65 x 104 células/cm2 em DMEM. Para uma cobertura de vidro com câmara de 2 poços (4 cm 2), semeie 65.000 – 70.000 células por poço com2 mL de DMEM. Incubar as células a 37 °C e 5% CO 2 durante2 h antes de verificar a aderência. Quando as células começarem a aderir, remova 1 mL de meio e substitua-o pelo mesmo volume de meio de hibernação, agite suavemente para misturar. Uma vez que o meio é misturado, repita este processo e remova metade do meio presente e substitua pelo mesmo volume de meio de crescimento de neurônio, em seguida, misture suavemente.
- Preparar a cultura primária do neurônio hipocampal seguindo o trabalho publicado anteriormente87 e a partir do protocolo do produto no local do fabricante a partir do qual o hipocampo dissecado do rato E18 é obtido88 (ver Tabela de Materiais). Esse protocolo resulta em uma população majoritariamente neuronal com <2% de astrócitos.
3. Preparação de células para imagens de células vivas
NOTA: Os tipos celulares e a origem (ou seja, células cultivadas e primárias) podem diferir nos requisitos de coloração; ver relatórios publicados para mais detalhes62,63.
- Preparo de PKmito Orange
NOTA: Outros corantes que geralmente coram o IMM foram relatados64,65,66 e estão disponíveis comercialmente. A PKMO é a única utilizada neste protocolo.- Ressuspender PKMO em pó (ver Tabela de Materiais) em DMSO de acordo com as instruções do fabricante89. Aspirar o meio das células e lavar em meio pré-aquecido sem vermelho de fenol. Preparar um estoque de PKMO em DMEM pré-aquecido, livre de fenol e sem vermelho suplementado com 2% (v/v) de FBS ou 10% (v/v), dependendo do estado de diferenciação, HEPES para uma concentração final de 20 mM, e 1% (v/v) antibiótico-antimicótico antes de colorir as células seguindo as instruções do fabricante. Esta formulação, sem PKMO, é o meio de imagem de células vivas.
- Coloração celular com PKMO
- Incubar as células com o corante a 37 °C e 5% CO2, durante 30 min. Lavar as células três vezes com meios de imagem de células vivas e, para a lavagem final, incubar durante 30 minutos a 37 °C, 5% de CO2.
- Adicione mídia de imagem de célula viva fresca e pré-aquecida. As células agora estão prontas para exames de imagem.
NOTA: Tratamentos agudos (por exemplo, medicamentos e/ou estressores), quando usados, são adicionados antes de imagens ao vivo; veja a seção Discussão e a Figura Suplementar 1.
4. Imagem de células vivas por microscopia STED
NOTA: Este protocolo utiliza um sistema STED construído em torno de um microscópio invertido, com o sistema especificado na Tabela de Materiais. Este sistema é equipado com lasers de excitação pulsada (laser de 561 nm com potência nominal ~300 μW) e um laser de depleção STED pulsado de 775 nm (potência nominal de 1,2 W), um scanner de galvano continuamente ajustável e um detector de fotodiodo de avalanche (APD) baseado em filtro de 615/20 nm. Uma lente de imersão em óleo 100x/1.40 para STED é usada aqui. O software Lightbox é utilizado para aquisição de imagens. Todos os detalhes fornecidos estão relacionados diretamente a este software e configuração do sistema.
- Diretrizes gerais e etapas para exames de imagem
- Use uma incubadora de estágio ou câmara ambiental para manter a viabilidade celular, mas experimentos de curto prazo à temperatura ambiente também são aceitáveis. Essas etapas são específicas para a configuração do STED descrita acima.
- Selecione os conjuntos de laser e filtro para geração de imagens.
- Use parâmetros para um corante laranja selecionando o(s) corante(s) usado(s) na coloração da Lista de Corantes, ou aquele com propriedades espectrais mais próximas do(s) corante(s) usado(s). Torne-os ativos clicando duas vezes ou arrastando-os para a lista de exemplo, onde diz "Arraste o(s) corante(s) aqui".
- Selecione uma região para a imagem.
- Em uma visão geral, crie uma região de interesse (ROI) ao redor de uma mitocôndria de interesse selecionando o botão ROI retangular e clicando e arrastando para moldar a região. O ROI pode ser redimensionado e girado usando os cantos da ROI ou bordas curvas que aparecem enquanto passa o mouse sobre um canto.
NOTA: Um resumo dos parâmetros de imagem sugeridos pode ser encontrado na Tabela 1. Esses parâmetros foram ajustados empiricamente usando aqueles previamente relatados para essa configuração STED e combinação de corantes63.
- Em uma visão geral, crie uma região de interesse (ROI) ao redor de uma mitocôndria de interesse selecionando o botão ROI retangular e clicando e arrastando para moldar a região. O ROI pode ser redimensionado e girado usando os cantos da ROI ou bordas curvas que aparecem enquanto passa o mouse sobre um canto.
- Configurar o gating.
- Ao lado do menu Geral , selecione o menu Fechar ou clique e segure para adicionar o menu à exibição. Recomenda-se que o acoplamento do detector STED seja ajustado para 1-1,05 a 7,8-7,85 ns, como apresentado aqui. Os tempos de fechamento podem variar e ser tão curtos quanto 1-1,05 a 7-7,05 ns.
- Ajustar a intensidade adequadamente de acordo com a amostra.
NOTA: Geralmente, o poder de excitação usado para STED é 2-3 vezes a potência usada para confocal, de modo que uma amostra que requer 5% de potência de laser de excitação para confocal pode usar 10%-15% de potência de laser de excitação durante a aquisição de STED.- Ajuste o laser de excitação para STED para 15%-20% e o laser de depleção STED para 20%-25% com acúmulos de 10 linhas. Use um tempo de permanência de pixel de 4 μs com um tamanho de pixel de 20-25 nm. O orifício foi mantido a 1,0 UA para células cultivadas e 0,7 UA para células primárias do hipocampo de ratos, a fim de melhorar a secção óptica nas mitocôndrias mais densamente embaladas.
NOTA: Imagens confocais e STED podem ser adquiridas para comparação lado a lado (Figura 3A, B, Figura 4A) ou somente STED podem ser adquiridas.
- Ajuste o laser de excitação para STED para 15%-20% e o laser de depleção STED para 20%-25% com acúmulos de 10 linhas. Use um tempo de permanência de pixel de 4 μs com um tamanho de pixel de 20-25 nm. O orifício foi mantido a 1,0 UA para células cultivadas e 0,7 UA para células primárias do hipocampo de ratos, a fim de melhorar a secção óptica nas mitocôndrias mais densamente embaladas.
- Informações adicionais para séries temporais/séries z
- Selecione a série temporal.
- Selecione o menu suspenso Hora. Defina o número de iterações (5 usadas aqui) e o intervalo de tempo (25 ou 30 s usados aqui) desejados para um timelapse (Figura 3C, D, Figura 4B).
Observação : se o intervalo for menor do que o tempo de aquisição, as iterações continuarão sem qualquer atraso. Ao executar um timelapse, o acionamento de uma unidade de foco perfeita ou rastreamento de foco semelhante é altamente recomendado para evitar possíveis desvios.
- Selecione o menu suspenso Hora. Defina o número de iterações (5 usadas aqui) e o intervalo de tempo (25 ou 30 s usados aqui) desejados para um timelapse (Figura 3C, D, Figura 4B).
- Defina o intervalo de volume.
- Defina o intervalo de volume z conforme desejado, ativando a opção Volume e ajustando as duas extremidades do intervalo. Os tamanhos de passo utilizados neste protocolo para aquisição de imagens 2D foram de 150-200 nm. O tamanho do passo recomendado em relação à amostragem de Nyquist necessária para a deconvolução de STED bruto pode ser calculado com ferramentas on-line90.
NOTA: A potência do laser de depleção STED e o número de planos fotografados podem aumentar o fotobranqueamento do corante e a exposição à luz da célula a níveis prejudiciais. Verifique se há sinais de fototoxicidade e fotodano após a aquisição.
- Defina o intervalo de volume z conforme desejado, ativando a opção Volume e ajustando as duas extremidades do intervalo. Os tamanhos de passo utilizados neste protocolo para aquisição de imagens 2D foram de 150-200 nm. O tamanho do passo recomendado em relação à amostragem de Nyquist necessária para a deconvolução de STED bruto pode ser calculado com ferramentas on-line90.
- Selecione a série temporal.
5. Ferramentas analíticas e de processamento para ultraestrutura mitocondrial
NOTA: O processamento de imagens (ou seja, deconvolução) é opcional, mas normalmente usado ao criar e analisar imagens STED para publicação. A deconvolução para melhorar o contraste e reduzir o ruído é altamente sugerida para a segmentação ideal das cristas individuais, como descrito a seguir (Figura 2).
- Deconvolução da imagem STED
NOTA: O software utilizado para a deconvolução neste protocolo é fornecido na Tabela de Materiais.- Defina os parâmetros microscópicos da imagem.
NOTA: Certifique-se de que os metadados do microscópio são inseridos corretamente nos parâmetros microscópicos da imagem. Isso inclui a montagem do índice de refração médio; meio de imersão; tamanho do pixel; comprimentos de onda de excitação, emissão e depleção; e qualquer outra informação relevante. Modelos com esses parâmetros podem ser salvos para reutilização. - Desconvolute imagens STED brutas com o algoritmo do software.
- O acesso a algoritmos de deconvolução automatizados em software de deconvolução permite o processamento de imagens de deconvolução hands-off. Selecione o botão Expresso e defina o tipo de deconvolução como Rápido, Padrão, Agressivo ou Conservador para diferentes graus de poder de deconvolução. Imagens representativas usando configurações Express Deconvolution with Aggressive são mostradas (Figura 3, Figura 4 e Figura 5).
- Salve as imagens do software de deconvolução no formato ICS2.
- Para deconvolução manual, execute as seguintes etapas.
- Resumidamente, ao executar a deconvolução manual, salve os modelos do assistente de deconvolução para consistência e tenha a opção de carregar um modelo ao iniciar o assistente. Use informações de PSF (função de propagação de ponto medido) se geradas com a configuração e os parâmetros de aquisição.
- Recorte a imagem STED bruta, se necessário, antes de fazer com que o software de deconvolução estabilize automaticamente a imagem. Complementos aos pacotes de software de deconvolução permitem a compensação específica de possíveis artefatos de imagem, como deriva térmica e aberrações cromáticas.
- Em seguida, gere um histograma logarítmico para executar manualmente ou automaticamente a subtração em segundo plano. Selecione o algoritmo clássico de deconvolução de estimativa de máxima verossimilhança (CMLE).
Observação : para deconvolução, os valores chave a serem ajustados são o limite de relação sinal-ruído, o número de iterações e o limite de qualidade. Esses valores podem ser ajustados e a deconvolução pode ser visualizada para determinar as configurações ideais.
- Defina os parâmetros microscópicos da imagem.
- Segmentação e análise de partículas
NOTA: Este protocolo utiliza FIJI (Is Just ImageJ), um software de código aberto (consulte Tabela de Materiais), para segmentação e análise. Outros softwares comparáveis, incluindo CellProfiler, Icy, Ilastik e QuPath, estão disponíveis para esses fins.- Preparar imagens para segmentação.
- Abra as imagens STED raw .obf ou arquivos .ics2 do software de deconvolução indo para Arquivo → Abrir ou clicando e arrastando os arquivos para a barra de ferramentas do ImageJ. A partir daqui, qualquer processamento que facilite a segmentação das imagens pode ser realizado antes da segmentação.
- Para manter as alterações consistentes, registre funções usando Plug-ins → Macros → Grave e copie e cole comandos de tecla em uma nova macro, de Plug-ins → Nova Macro →. Certifique-se de selecionar a imagem a ser processada antes de executar a macro.
NOTA: As alterações comumente aceitáveis para quantificação de tamanho e forma incluem corte, projeção de uma pilha z e subtração em plano de fundo com suavização desabilitada. As alterações devem ser realizadas de forma consistente entre as imagens dentro de um conjunto de dados e relatadas.
- Ajustar configurações de segmentação Weka treinável
NOTA: Detalhes adicionais com instruções passo a passo para o uso da ferramenta de segmentação semi-automatizada e análises a jusante para mitocôndrias foram publicados91.- Abra as imagens STED desconvolvidas no plugin Trainable Weka Segmentation (TWS)92 , localizado em Plugins → Segmentation → Trainable Weka Segmentation. Nas Configurações de segmentação, selecione os recursos de desfoque gaussiano, projeções de membrana e filtro Sobel . O padrão de espessura de membrana é 1 e o padrão de tamanho de patch de membrana é 19.
- Rotule a classe 1 ou 2 como "Cristae" e a outra como "Background" (Figura 2). Os modelos também podem ser salvos com o botão Salvar classificador . Selecione o classificador Load para reutilizar essas configurações para outras imagens.
- Execute rastreamentos de classe TWS.
- Use a ferramenta de linha ou outras formas para realçar algumas das cristas ou plano de fundo. Pelo menos algumas seleções de fundo devem incluir espaços entre as cristas. Desenhe uma linha sobre a estrutura para atribuir a qualquer classe e, em seguida, selecione o botão Adicionar a no lado direito para cristas ou fundo. Clique duas vezes em um rastreamento para remover a estrutura desse rótulo.
- Realizar treinamento de classificação TWS.
- Selecione o botão Classificar trem no lado esquerdo para gerar um mapa com base nas informações fornecidas ao plugin. A sobreposição de classes segmentadas pode ser ativada e desativada com o botão Alternar sobreposição, e a opacidade da sobreposição pode ser ajustada em Configurações. O classificador pode ser retreinado com etiquetas adicionais. Quando estiver satisfeito, selecione o botão Obter Probabilidade.
- Meça as partículas.
- Usando o mapa de probabilidade cristae, limite a imagem para gerar uma máscara e, em seguida, vá para Analisar → Analisar partículas. Geralmente, o tipo de limite padrão pode ser usado e o intervalo ajustado para garantir que todas as cristas sejam contabilizadas. As medidas fornecidas pela análise de partículas podem ser ajustadas por Analisar → Definir Medidas.
NOTA: Exemplos de parâmetros de tamanho e forma, como área, perímetro, circularidade e proporção das cristas, são medidos e exibidos com base nas medidas selecionadas (Figura 2, Figura 5A, Tabela Suplementar 1). - OPCIONAL: Selecione a imagem STED bruta com as mesmas dimensões da imagem desconvolvida e aplique as ROIs do gerente e, em seguida, selecione Medir no gerenciador de ROI para obter valores de intensidade.
- Usando o mapa de probabilidade cristae, limite a imagem para gerar uma máscara e, em seguida, vá para Analisar → Analisar partículas. Geralmente, o tipo de limite padrão pode ser usado e o intervalo ajustado para garantir que todas as cristas sejam contabilizadas. As medidas fornecidas pela análise de partículas podem ser ajustadas por Analisar → Definir Medidas.
- Obter gráficos de linha.
- Gráficos de linhas foram gerados a partir das imagens STED desconvolvidas. Desenhe uma linha multiponto, ajuste a espessura da linha para vários pixels de largura para calcular a média do ruído e gire a linha para se ajustar às mitocôndrias (Figura 2, Figura 5B). O gráfico de linhas resultante gerado é usado para medir distâncias pico-a-pico para relatar a periodicidade e distribuição das cristas em um determinado intervalo.
NOTA: Relacionadamente, a densidade de cristas pode ser relatada como a contagem de cristas independentes dentro de uma determinada área, conforme determinado pela medição da parte externa da mitocôndria. A área mitocondrial pode ser determinada aplicando-se um filtro à imagem STED desconvolvida ou crua para gerar uma máscara. Certifique-se de que a máscara se ajusta com precisão ao contorno das mitocôndrias antes de medir a área.
- Gráficos de linhas foram gerados a partir das imagens STED desconvolvidas. Desenhe uma linha multiponto, ajuste a espessura da linha para vários pixels de largura para calcular a média do ruído e gire a linha para se ajustar às mitocôndrias (Figura 2, Figura 5B). O gráfico de linhas resultante gerado é usado para medir distâncias pico-a-pico para relatar a periodicidade e distribuição das cristas em um determinado intervalo.
- Preparar imagens para segmentação.
Representative Results
Este protocolo descreve as condições de crescimento celular para células cultivadas e primárias com foco em imagens STED de células vivas e subsequentes análises de cristas mitocondriais. Projeções feitas com ImageJ de mitocôndrias de células SH-SY5Y indiferenciadas (Figura 3A) e SH-SY5Y diferenciadas por AR (Figura 3B) podem ser coletadas como pilhas z com confocal e STED tradicionais, e as imagens STED brutas podem então ser desconenvolvidas. Imagens com timelapse também podem ser realizadas e posteriormente desconvolvidas (Figura 3C,D). Usando parâmetros de imagem ligeiramente diferentes para neurônios primários do hipocampo de ratos (Tabela 1), imagens STED confocais e brutas podem ser adquiridas como pilhas z, e as imagens STED brutas podem ser desconvolvidas (Figura 4A). Imagens em timelapse de mitocôndrias de neurônios primários também são possíveis (Figura 4B). Em geral, as imagens de lapso de tempo devem ser capazes de mostrar eventos dinâmicos mitocondriais.
Quando as projeções STED brutas e STED z-stack desconvolvidas das amostras usadas para segmentação parecem consistentes, medições quantitativas são realizadas. O plugin TWS usa a imagem STED desconvolvida para segmentar para fazer uma máscara de probabilidade, que é então usada para criar uma máscara binária das cristas para obter parâmetros de tamanho e forma (Figura 5A). As regiões dessa máscara são salvas no gerenciador de ROI e podem ser aplicadas à imagem STED bruta se desejado para medir diferenças na intensidade relativa. As projeções STED deconvolved também podem ser usadas para determinar a periodicidade e densidade das cristas em uma determinada área (Figura 5B).
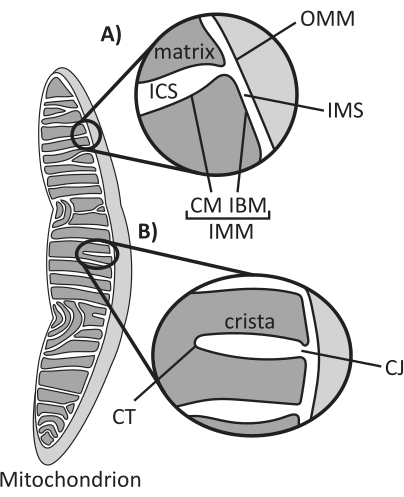
Figura 1: Morfologia mitocondrial. As mitocôndrias possuem um sistema de duas membranas que define diferentes subcompartimentos (A). Cristae são dobras da membrana interna com características definidas (B). Abreviações: OMM, membrana mitocondrial externa; ICS, espaço intracristal; IMS, espaço intermembrana; MC, membrana das cristas; IBM, membrana de contorno interno; MIM, membrana mitocondrial interna; TC, ponta das cristas; CJ, junção de cristae. Clique aqui para ver uma versão maior desta figura.
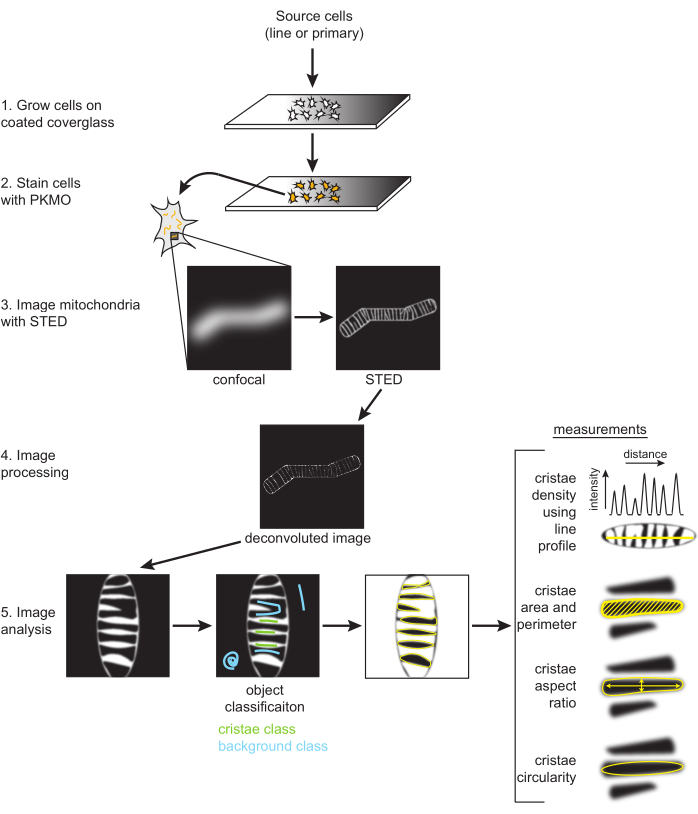
Figura 2: Esquema do fluxo de trabalho. As células SH-SY5Y ou neurônios primários do hipocampo de rato são cultivados em uma cobertura revestida de PDL. As células SH-SY5Y são cultivadas em paralelo para permanecerem indiferenciadas ou submetidas à diferenciação da AR ao longo de seis dias. Neurônios primários do hipocampo de ratos foram cultivados em uma cobertura revestida com PDL após serem isolados de seções hipocampais por sete dias. Uma vez prontas para serem imageadas, as células foram coradas com PKMO e imageadas com STED. As imagens STED brutas são então desconvolvidas e as imagens desconvolvidas são processadas em FIJI para obter medidas de tamanho e forma, como densidade de cristas, área, perímetro, circularidade e proporção de cristas. Clique aqui para ver uma versão maior desta figura.
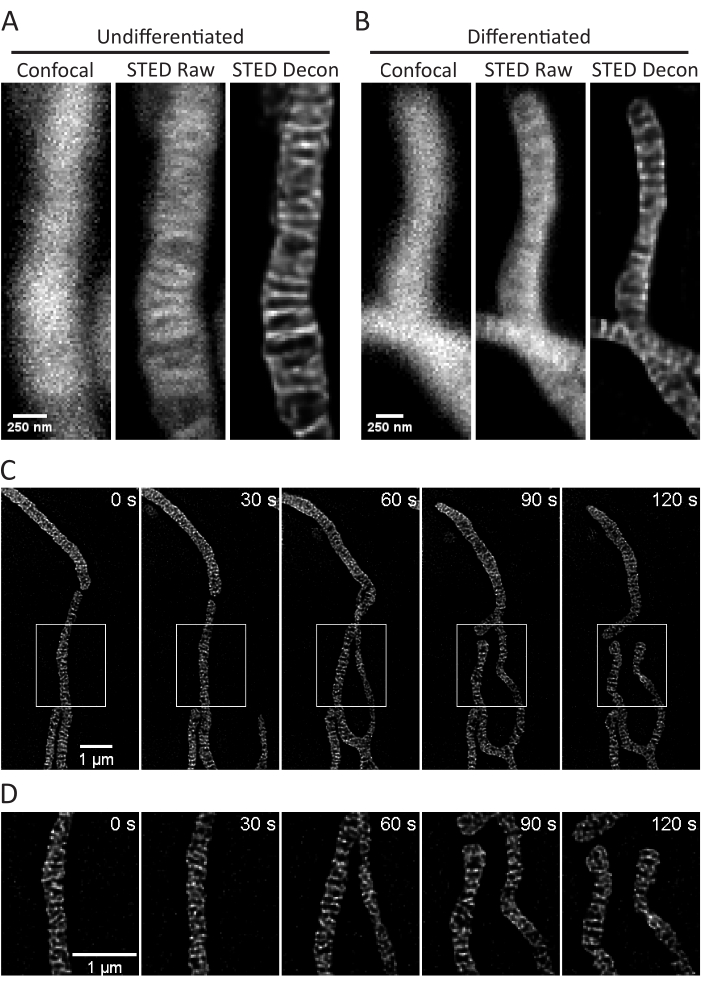
Figura 3: Imagem das mitocôndrias em células SH-SY5Y. Projeções representativas de STED confocal (esquerda), STED bruta (centro) e desconvolvida de Huygens (direita) de mitocôndrias de células SH-SY5Y não diferenciadas (A) e diferenciadas por AR (B) com coloração PKMO são mostradas. Um timelapse com intervalos de 30 s e 5 iterações de células SH-SY5Y diferenciadas por AR é mostrado (C) com regiões selecionadas (caixas brancas) expandidas sobre (D) usando imagens dimensionadas dessas regiões sem interpolação. Barras de escala: A,B, 250 nm; C,D, 1 μm. Clique aqui para ver uma versão maior desta figura.
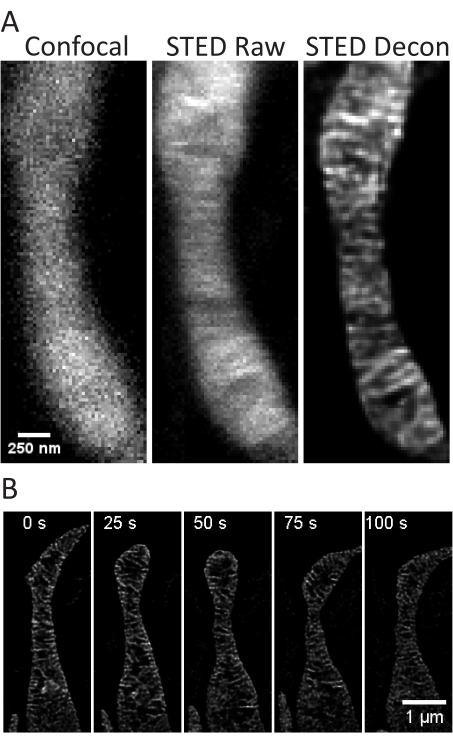
Figura 4: Imagem das mitocôndrias em neurônios primários do hipocampo de ratos. Projeções representativas de STED (esquerda), STED bruto (centro) e STED desconvolvido de Huygens (direita) de mitocôndrias de neurônios primários do hipocampo de ratos são mostradas (A). Um timelapse com intervalos de 25 s e 5 iterações de mitocôndrias nesses neurônios é mostrado (B). Barras de escala: A, 250 nm; B, 1 μm. Clique aqui para ver uma versão maior desta figura.
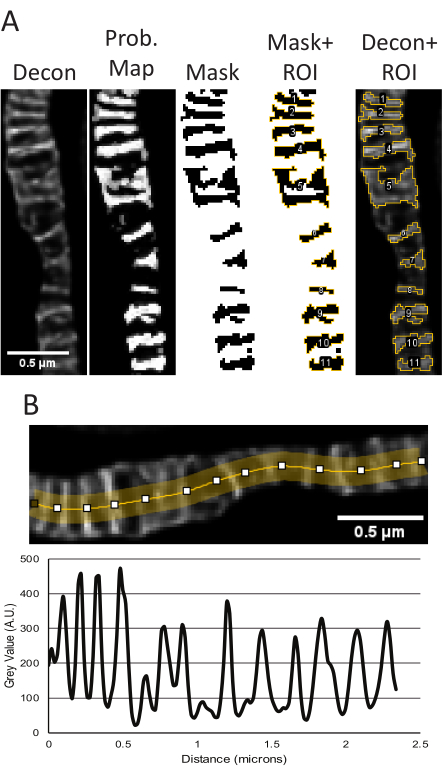
Figura 5: Processamento de imagens STED desconvolvidas no ImageJ. O uso representativo do plugin Trainable Weka Segmentation para medir o tamanho e a forma das cristas é mostrado (A). Da esquerda para a direita, as seguintes imagens são mostradas: a imagem STED desconvolvida, o mapa de probabilidade baseado na segmentação do plugin TWS, a máscara de limiar em FIJI usando o mapa de probabilidade como entrada, a máscara com as ROIs delineadas e as ROIs sobrepostas à imagem STED desconvolvida original. As medidas resultantes de área, perímetro, circularidade e proporção correspondentes a esses objetos podem ser encontradas na Tabela Suplementar 1. Um gráfico de linhas usando a imagem STED desconvolvida para medir distâncias pico-a-pico como uma leitura para a densidade de cristas é mostrado (B). Barras de escala: 0,5 μm. Clique aqui para ver uma versão maior desta figura.
| Tamanho do pixel (nm) | Tempo de permanência (μs) | Linha acc. | Excitação de 561 nm durante a aquisição de STED (%) | Potência de depleção STED de 775 nm (%) | Tamanho do passo (nm) | Pinhole (AU) | Intervalo(s) de timelapse | Iterações de timelapse | |
| SH-SY5Y indiferenciado | 20 | 4 | 10 | 15 | 20 | 200 | 1.0 | 30 | 5 |
| RA-Differe-ntiated SH-SY5Y | 20-25 | 4 | 10-12 | 15 | 20-22 | 150-200 | 1.0 | 30 | 5 |
| Neurônios primários | 20-25 | 4 | 10 | 10 | 25 | 200 | 0.7 | 30 | 5 |
| NOTA: O tamanho do pixel pode variar com base nos requisitos de imagem e na intenção de desconvolver imagens. A amostragem adequada é necessária para a deconvolução. Os tamanhos de pixel para imagens STED brutas sem deconvolução podem ir até 30 nm. | |||||||||
Tabela 1: Resumo dos parâmetros de aquisição de STED. As configurações usadas para imagens STED 2D para cada tipo de célula, SH-SY5Y indiferenciado, SH-SY5Y diferenciado por AR e neurônios primários do hipocampo de ratos são exibidos. Para todos os lapsos de tempo, 5 iterações foram tomadas com intervalos variáveis com base no tamanho do ROI.
Figura suplementar 1: Imagem de células SH-SY5Y com adição de β amiloide (Aβ). Imagens representativas confocais (esquerda), STED brutas (centro) e STED desconvolvidas (direita) de células SH-SY5Y diferenciadas por AR com coloração PKMO (superior) e Aβ-HiLyte647 (inferior) são mostradas (A). Projeções z-stack mescladas de PKMO STED bruto (verde) com STED Aβ bruto (magenta) (B) ou PKMO STED desconvolvido (verde) com STED Aβ desconvolvido (magenta) (C) são mostradas. Barras de escala: 0,5 μm. Clique aqui para baixar este arquivo.
Tabela Suplementar 1: Medidas de tamanho e forma das cristas segmentadas. As medidas de tamanho e forma de área (μm2), perímetro (μm), circularidade e proporção, correspondentes aos objetos delineados na Figura 5A a partir de mitocôndrias segmentadas, são mostradas. Clique aqui para baixar este arquivo.
Tabela Suplementar 2: Resumo dos parâmetros de aquisição com amostras de β amiloide. As configurações usadas para imagens STED 2D de PKMO e Aβ-HiLyte647 em células SH-SY5Y indiferenciadas e diferenciadas por AR são exibidas. Confocal de Aβ-HiLyte647 pode ser usado sozinho, pois não há estrutura específica para resolver; Imagens STED de Aβ-HiLyte647 são mostradas aqui para tamanhos de partículas menores. Clique aqui para baixar este arquivo.
Arquivo Suplementar 1: Protocolo de tratamento com β amiloide. Clique aqui para baixar este arquivo.
Discussion
Este protocolo apresenta o uso da linhagem celular SH-SY5Y do neuroblastoma humano e neurônios primários do hipocampo de ratos com o novo corante PKMO direcionado para IMM para imagens STED de células vivas. Devido à novidade da PKMO, atualmente há pouca publicação usando este corante para imagens STED ao vivo. O uso desses tipos celulares para imagens STED apresenta desafios, especificamente porque as células neuronais têm mitocôndrias mais estreitas. Uma limitação desse protocolo é o corante PKMO utilizado, pois pode ser tóxico para as células. Diferentes células e linhagens celulares respondem diferentemente ao corante, assim, ajustes na concentração do corante e no tempo de incubação para otimizar os resultados para um sinal forte sem prejudicar as células podem ser necessários. Uma solução sugerida é diminuir a concentração e aumentar o tempo de coloração63; no entanto, isso pode resultar em coloração mais pobre sem aumentar a viabilidade celular.
Da mesma forma que o PKMO, o corante comercial Live Orange mito (Tabela de Materiais) também exibe alguma toxicidade celular. Este corante foi usado para uma variedade de células cultivadas, mas foi incapaz de exibir coloração comparável em células SH-SY5Y diferenciadas de AR com sucesso com os mesmos parâmetros que suas contrapartes indiferenciadas (nossas observações não publicadas). No entanto, protocolos de coloração amable podem ser otimizados para esta sonda e tipos celulares escolhidos. Com esse corante, foram utilizados tempos de fechamento do detector de 1-1,05 a 7-7,05 ns, permanecendo os mesmos todos os outros parâmetros da Tabela 1 . Geralmente, a coloração de células com 200-250 nM de Mito de Laranja Viva por 45 min produziu resultados comparáveis como mostraram os resultados de PKMO. Coloração de maior concentração por menos tempo ou coloração de menor concentração pelo mesmo período de tempo ou um pouco mais pode produzir resultados diferentes e pode ser favorável a outros tipos celulares ou condições de crescimento.
A imagem de neurônios primários do hipocampo de ratos difere das células imortalizadas devido à natureza das projeções do axônio e dendrito, bem como à distribuição mitocondrial no momento da imagem. Uma dificuldade nesta parte do protocolo é que a densidade de semeadura determina se as culturas primárias serão capazes de aderir e crescer de forma saudável, e em densidades mais altas, as projeções tendem a crescer demais por DIV 10. Portanto, as mitocôndrias imageadas desses neurônios primários provavelmente virão do corpo celular e não das projeções; no entanto, o crescimento bem-sucedido a partir de uma densidade celular inicial mais baixa produz melhores resultados de imagem em tempos de crescimento posteriores. A chave é garantir fundo baixo e luz fora de foco para ter o melhor contraste para STED. Para abordar as preocupações em relação à população celular, a cultura de células hipocampais primárias em meios de crescimento de neurônios suplementados com B27 impede o crescimento de células gliais, e a fonte relata que <5% das células são astrócitos e a ausência de suplemento de NbActiv1 nos meios de crescimento reduz o número de astrócitos em culturas para <2%87. Tanto para células SH-SY5Y cultivadas quanto para neurônios primários do hipocampo de ratos, o revestimento PDL usado para o crescimento contribui para a névoa de fundo nas imagens. Sinal-ruído suficiente é realizado com as configurações relatadas na (Tabela 1) e a deconvolução remove a maior parte do fundo observado.
Além das imagens abordadas aqui, também é possível adicionar tratamentos ou estresse às células antes ou durante a imagem. Por exemplo, a adição de peróxido de hidrogênio terc-butila (tBHP) induz estresse oxidativo, e é possível monitorar mudanças nas mitocôndrias ao longo do tempo após a adição. A adição de β amiloide (Aβ) com um tag fluorescente permite monitorar a distribuição deste peptídeo em relação às mitocôndrias, bem como a estrutura mitocondrial ao longo do tempo. A saúde mitocondrial tem sido fortemente implicada na DA e é amplamente apoiada por desempenhar um papel na toxicidade da Aβ 43,71,72. Notavelmente, o status de diferenciação das células SH-SY5Y afeta a localização do precursor da proteína Aβ (AβPP)85, e experimentos usando AβPP devem ser cuidadosamente construídos.
Como exemplo de como este protocolo pode ser adaptado, mostra-se que a variante fluorescente Aβ(1-42)-HiLyte 647 pode ser adicionada às células coradas com PKMO 15 minutos antes da aquisição de imagens (Figura 1 Suplementar). Os parâmetros de imagem são semelhantes (Tabela Suplementar 2), com a principal diferença sendo que um orifício menor é necessário na obtenção de imagens de mitocôndrias mais estreitas. A aquisição de imagens Aβ-HiLyte647 com STED requer menos excitação geral (6%-8%) e depleção de STED (10%-12%), potência do laser e menos acúmulos (seis). O fechamento do detector também é estendido de 0,1 para 10 ns. Embora a resolução STED de Aβ não seja necessária, a relação sinal-ruído global e o tamanho das partículas Aβ de STED bruto foram melhores do que os das imagens confocais, e a deconvolução subsequente também pode ser realizada. A coleta de imagens STED e a desconvolvência de projeções brutas STED z-stack de Aβ parecem particularmente úteis quando se fundem com imagens STED brutas ou STED desconvolvidas da coloração PKMO (Figura suplementar 1B,C). Ambos os canais foram coletados em uma única etapa de quadro. Medidas de localização dependente do tempo, semelhantes às listadas na Figura 2 e mostradas na Figura 5, quando aplicável, e diferenças na arquitetura das cristas podem ser obtidas após o tratamento de estresse ou outras adições.
Outros métodos possíveis para marcação dupla em STED de células vivas de mitocôndrias não relatados aqui, mas foram relatados por outros incluem o uso de proteínas marcadas com SNAP93, proteínas marcadas com Halo e o uso de outros corantes permeáveis a células com alvos genéricos, como o mtDNA63. Notavelmente, a estratégia de marcação de SNAP e Halo influencia a intensidade e longevidade do sinal de fluorescência resultante na obtenção de imagens94. Além disso, embora este protocolo apresente vários exemplos de análises que podem ser aplicadas a mitocôndrias segmentadas, existem muitas outras análises que pacotes de software podem realizar nessas imagens.
Disclosures
Os autores não têm nada a revelar.
Acknowledgements
Os neurônios primários do hipocampo de ratos foram fornecidos pelo Dr. George Lykotrafitis e Shiju Gu do Departamento de Engenharia Biomédica da Universidade de Connecticut (Storrs, CT, EUA). O instrumento Abberior STED alojado no Advanced Light Microscopy Facility no Center for Open Research Resources and Equipment foi adquirido com S10OD023618 de concessão do NIH concedida a Christopher O'Connell. Esta pesquisa foi financiada pelo NIH grant R01AG065879 concedido a Nathan N. Alder.
Materials
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | Gibco | 25300054 | |
| 0.4% Trypan blue | Invitrogen | T10282 | |
| 0.5% Trypsin-EDTA, no phenol red | Gibco | 15400054 | |
| 100 X antibiotic-antimycotic | Gibco | 15240062 | |
| 100 X/1.40 UPlanSApo oil immersion lens | Olympus | Equipped in Olympus IX83 microscope for STED setup described in Section 4 | |
| All-trans-retinoic acid | Sigma | R2625 | |
| Amyloid-β (1-42, HiLyte Fluor647, 0.1 mg) | AnaSpec | AS-64161 | Other fluorescent conjugates available |
| B27 supplement (50 X), serum free | Gibco | 17504044 | |
| Cell Counter (Countess II FL) | Life Technologies | AMQAF1000 | |
| Centrifuge | Eppendorf | 5804-R | |
| Counter slides | Invitrogen | C10283 | |
| Conical tubes (15 mL) | Thermo Fisher Scientific | 339650 | |
| Cuvettes (Quartz Cells) | Starna Cells, Inc. | 9-Q-10 | Used with Spectrometer as described in Section 1.3 |
| DMEM (high glucose with sodium pyruvate) | Gibco | 11995073 | Used for SH-SY5Y cell materials as described in Section 1 |
| DMEM (high glucose no sodium pyruvate) | Gibco | 11965092 | Used for primary cell materials as described in Section 2 |
| DMEM (phenol red-free) | Gibco | 31053028 | Used for imaging as described in Section 3 |
| DMSO | Sigma | D8418 | |
| DNAase I from bovine pancreas | Sigma | DN25 | Used for primary cell materials as described in Section 2.2.1 and 2.2.2 |
| DPBS (no calcium, no magnesium) | Gibco | 14190144 | |
| E18 Rat Hippocampus | Transnetyx Tissue | SDEHP | |
| Ethanol (200 proof) | Fisher Bioreagents | BP28184 | |
| Fetal bovine serum (FBS), not heat-inactivated | Gibco | 26140079 | For cultured cells, in Section 1 |
| Fetal bovine serum (heat inactivated) | Gibco | 10082147 | For primary cell culture, Section 2 |
| Filter sterilization unit (0.1 µm, 500 mL) | Thermo Fisher Scientific | 5660010 | |
| FIJI (Is Just ImageJ) and Trainable Weka Segmentation (TWS) plug-in | -- | -- | Free, open-source image analysis software that includes plug-ins including Trainable Weka Segmentation described in Section 5; TWS plug-in from ref. 90 of the main text |
| GlutaMAX supplement (100 X) | Gibco | 35050061 | Glutamine supplement used for primary cell materials described in Section 2.1.2 |
| Hausser Scientific bright-Line and Hy-Lite Counting Chambers | Hausser Scientific | 267110 | |
| HBSS (no calcium, no magnesium) | Gibco | 14170120 | Used for primary cell materials described in Section 2.2.1 and 2.2.2 |
| HEPES | Gibco | 15630080 | |
| Huygens Professional deconvolution software (V. 20.10) | Scientific Volume Imaging (SVI) | -- | The deconvolution software used in this protocol and described in Section 5 |
| IX83 inverted microscope with Continuous Autofocus | Olympus | -- | This paper uses a STED Infinity Line system built around an Olympus IX83 inverted microscope, described in Section 4 |
| Lightbox software (V. 16.3.16118) | Abberior | -- | Vendor software used for STED image acquisition, described in Section 4 |
| Live Orange Mito dye | Abberior | LVORANGE-0146-30NMOL | Live cell imaging IMM-targeting dye described in Discussion |
| Neurobasal media | Gibco | 21103049 | Used for primary cell materials referred to in Section 2.1.2 |
| Nunc Lab-Tek II 2-well chambered coverglass | Nunc | 155379 | Can purchase a variety of chambers but make sure the coverglass is #1.5 |
| Pasteur Pipets (Fisherbrand) | Thermo Fisher Scientific | 22183632 | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140122 | |
| PKmito Orange dye | Spirochrome | SC053 | |
| Poly-D-lysine | Gibco | A3890401 | |
| SH-SY5Y Cell line | ATCC | CRL2266 | |
| Sodium pyruvate (100 mM) | Gibco | 11360070 | Used for primary cell materials described in Section 2 |
| Spectrometer (GENESYS 180 UV-Vis) | Thermo Fisher Scientific | 840309000 | |
| STED Expert Line microscope | Abberior | -- | STED setup can be customized, but at time of purchase instrument was considered Abberior’s Expert Line; brief description of setup is available in Section 4 of protocol |
| T25 flask (TC-treated, filter cap) | Thermo Fisher Scientific | 156367 | Other culture vessels and sizes available |
References
- Iovine, J. C., Claypool, S. M., Alder, N. N. Mitochondrial compartmentalization: emerging themes in structure and function. Trends in Biochemical Sciences. 46 (11), 902-917 (2021).
- Gupta, A., Becker, T. Mechanisms and pathways of mitochondrial outer membrane protein biogenesis. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1862 (1), 148323 (2021).
- Gordaliza-Alaguero, I., Cantó, C., Zorzano, A. Metabolic implications of organelle-mitochondria communication. EMBO Reports. 20 (9), e47928 (2019).
- Klecker, T., Westermann, B. Pathways shaping the mitochondrial inner membrane. Open Biology. 11 (12), 210238 (2021).
- Navarro, A., Boveris, A. The mitochondrial energy transduction system and the aging process. American Journal of Physiology-Cell Physiology. 292 (2), C670-C686 (2007).
- Yu, R., Lendahl, U., Nistér, M., Zhao, J. Regulation of mammalian mitochondrial dynamics: opportunities and challenges. Frontiers in Endocrinology. 11, 374 (2020).
- Horn, A., Raavicharla, S., Shah, S., Cox, D., Jaiswal, J. K. Mitochondrial fragmentation enables localized signaling required for cell repair. The Journal of Cell Biology. 219 (5), e201909154 (2020).
- Glancy, B., Kim, Y., Katti, P., Willingham, T. B. The functional impact of mitochondrial structure across subcellular scales. Frontiers in Physiology. 11, 541040 (2020).
- Bahat, A., et al. MTCH2-mediated mitochondrial fusion drives exit from naïve pluripotency in embryonic stem cells. Nature Communications. 9 (1), 5132 (2018).
- Detmer, S. A., Chan, D. C. Functions and dysfunctions of mitochondrial dynamics. Nature Reviews Molecular Cell Biology. 8, 870-879 (2007).
- Bertholet, A. M., et al. Mitochondrial fusion/fission dynamics in neurodegeneration and neuronal plasticity. Neurobiology of Disease. 90, 3-19 (2016).
- Zemirli, N., Morel, E., Molino, D. Mitochondrial dynamics in basal and stressful conditions. International Journal of Molecular Sciences. 19 (2), 564 (2018).
- Harwig, M. C., et al. Methods for imaging mammalian mitochondrial morphology: a prospective on mitograph. Analytical Biochemistry. 552, 81-99 (2018).
- Pánek, T., Eliáš, M., Vancová, M., Lukeš, J., Hashimi, H. Returning to the fold for lessons in mitochondrial crista diversity and evolution. Current Biology. 30 (10), R575-R588 (2020).
- Gottschalk, B., Madreiter-Sokolowski, C. T., Graier, W. F. Cristae junction as a fundamental switchboard for mitochondrial ion signaling and bioenergetics. Cell Calcium. 101, 102517 (2022).
- Khosravi, S., Harner, M. E. The MICOS complex, a structural element of mitochondria with versatile functions. Biological Chemistry. 401 (6-7), 765-778 (2020).
- Frezza, C., et al. OPA1 controls apoptotic cristae remodeling independently from mitochondrial fusion. Cell. 126 (1), 177-189 (2006).
- Meeusen, S., et al. Mitochondrial inner-membrane fusion and crista maintenance requires the dynamin-related GTPase Mgm1. Cell. 127 (2), 383-395 (2006).
- Patten, D. A., et al. OPA1-dependent cristae modulation is essential for cellular adaptation to metabolic demand. The EMBO Journal. 33 (22), 2676-2691 (2014).
- Paumard, P., et al. The ATP synthase is involved in generating mitochondrial cristae morphology. The EMBO Journal. 21 (3), 221-230 (2002).
- Strauss, M., Hofhaus, G., Schröder, R. R., Kühlbrandt, W. Dimer ribbons of ATP synthase shape the inner mitochondrial membrane. The EMBO Journal. 27 (7), 1154-1160 (2008).
- Basu Ball, W., Neff, J. K., Gohil, V. M. The role of nonbilayer phospholipids in mitochondrial structure and function. FEBS Letters. 592 (8), 1273-1290 (2018).
- Hackenbrock, C. R. Ultrastructural bases for metabolically linked mechanical activity in mitochondria. I. Reversible ultrastructural changes with change in metabolic steady state in isolated liver mitochondria. The Journal of Cell Biology. 30 (2), 269-297 (1966).
- Dlasková, A., et al. Mitochondrial cristae narrowing upon higher 2-oxoglutarate load. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1860 (8), 659-678 (2019).
- Pérez-Hernández, C. A., et al. Mitochondrial ultrastructure and activity are differentially regulated by glycolysis-, krebs cycle-, and microbiota-derived metabolites in monocytes. Biology. 11 (8), 1132 (2022).
- Mannella, C. A. Structural diversity of mitochondria: functional implications. Annals of the New York Academy of Sciences. 1147, 171-179 (2008).
- Plecitá-Hlavatá, L., Ježek, P. Integration of superoxide formation and cristae morphology for mitochondrial redox signaling. The International Journal of Biochemistry & Cell Biology. 80, 31-50 (2016).
- Scorrano, L., et al. A distinct pathway remodels mitochondrial cristae and mobilizes cytochrome c during apoptosis. Developmental Cell. 2 (1), 55-67 (2002).
- Heath-Engel, H. M., Shore, G. C. Mitochondrial membrane dynamics, cristae remodelling and apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1763 (5-6), 549-560 (2006).
- Brandt, T., et al. Changes of mitochondrial ultrastructure and function during ageing in mice and Drosophilia. eLife. 6, e24662 (2017).
- Kondadi, A. K., et al. Cristae undergo continuous cycles of membrane remodelling in a MICOS-dependent manner. EMBO Reports. 21, e49776 (2020).
- Quintana-Cabrera, R., Mehrotra, A., Rigoni, G., Soriano, M. E. Who and how in the regulation of mitochondrial cristae shape and function. Biochemical and Biophysical Research Communications. 500 (1), 94-101 (2018).
- Nielsen, J., et al. Plasticity in mitochondrial cristae density allows metabolic capacity modulation in human skeletal muscle: Enlarged mitochondrial cristae density in athletes. The Journal of Physiology. 595 (9), 2839-2847 (2017).
- Afzal, N., Lederer, W. J., Jafri, M. S., Mannella, C. A. Effect of crista morphology on mitochondrial ATP output: A computational study. Current Research in Physiology. 4, 163-176 (2021).
- Heine, K. B., Parry, H. A., Hood, W. R. How does density of the inner mitochondrial membrane influence mitochondrial performance. American Journal of Physiology-Regulatory, Integrative and Comparative Physiology. 324 (2), R242-R248 (2023).
- Wang, W., Zhao, F., Ma, X., Perry, G., Zhu, X. Mitochondria dysfunction in the pathogenesis of Alzheimer's disease: recent advances. Molecular Neurodegeneration. 15, 30 (2020).
- Singh, A., Kukreti, R., Saso, L., Kukreti, S. Oxidative stress: a key modulator in neurodegenerative diseases. Molecules. 24, 1583 (2019).
- Pchitskaya, E., Popugaeva, E., Bezprozvanny, I. Calcium signaling and molecular mechanisms underlying neurodegenerative diseases. Cell Calcium. 70, 87-94 (2018).
- Estes, R. E., Lin, B., Khera, A., Davis, M. Y. Lipid metabolism influence on neurodegenerative disease progression: is the vehicle as important as the cargo. Frontiers in Molecular Neuroscience. 14, 788695 (2021).
- Calkins, M. J., Manczak, M., Mao, P., Shirendeb, U., Reddy, P. H. Impaired mitochondrial biogenesis, defective axonal transport of mitochondria, abnormal mitochondrial dynamics and synaptic degeneration in a mouse model of Alzheimer's disease. Human Molecular Genetics. 20 (23), 4515-4529 (2011).
- Petersen, C. A. H., et al. The amyloid beta-peptide is imported into mitochondria via the TOM import machinery and localized to mitochondrial cristae. Proceedings of the National Academy of Sciences of the United States of America. 105 (35), 13145-13150 (2008).
- Gan, X., et al. Inhibition of ERK-DLP1 signaling and mitochondrial division alleviates mitochondrial dysfunction in Alzheimer's disease cybrid cell. Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease. 1842 (2), 220-231 (2014).
- Baloyannis, S. J., Costa, V., Michmizos, D. Mitochondrial alterations Alzheimer's disease. American Journal of Alzheimer's Disease & Other Dementias. 19 (2), 89-93 (2004).
- Tillement, L., Lecanu, L., Papadopoulos, V. Alzheimer's disease: Effects of β-amyloid on mitochondria. Mitochondrion. 11 (1), 13-21 (2011).
- Choi, S. Y., et al. C9ORF72-ALS/FTD-associated poly(GR) binds Atp5a1 and compromises mitochondrial function in vivo. Nature Neuroscience. 22 (6), 851-862 (2019).
- Smith, E. F., Shaw, P. J., De Vos, K. J. The role of mitochondria in amyotrophic lateral sclerosis. Neuroscience Letters. 710, 132933 (2019).
- Costa, V., et al. Mitochondrial fission and cristae disruption increase the response of cell models of Huntington's disease to apoptotic stimuli. EMBO Molecular Medicine. 2 (12), 490-503 (2010).
- Costa, V., Scorrano, L. Shaping the role of mitochondria in the pathogenesis of Huntington's disease: Mitochondrial and Huntington's disease. The EMBO Journal. 31 (8), 1853-1864 (2012).
- Vanisova, M., et al. Mitochondrial organization and structure are compromised in fibroblasts from patients with Huntington's disease. Ultrastructural Pathology. 46 (5), 462-475 (2022).
- de Barcelos, I. P., Troxell, R. M., Graves, J. S. Mitochondrial dysfunction and multiple sclerosis. Biology. 8 (2), 37 (2019).
- Park, J., et al. Mitochondrial dysfunction in Drosophila PINK1 mutants is complemented by parkin. Nature. 441, 1157-1161 (2006).
- Meng, H., et al. Loss of Parkinson's disease-associated protein CHCHD2 affects mitochondrial crista structure and destabilizes cytochrome c. Nature Communications. 8, 15500 (2017).
- Lu, L., et al. CHCHD2 maintains mitochondrial contact site and cristae organizing system stability and protects against mitochondrial dysfunction in an experimental model of Parkinson's disease. Chinese Medical Journal. 135 (13), 1588-1596 (2022).
- Cogliati, S., et al. Mitochondrial Cristae shape determines respiratory chain supercomplexes assembly and respiratory efficiency. Cell. 155 (1), 160-171 (2013).
- He, B., et al. Mitochondrial cristae architecture protects against mtDNA release and inflammation. Cell Reports. 41 (10), 111774 (2022).
- Polo, C. C., et al. Three-dimensional imaging of mitochondrial cristae complexity using cryo-soft X-ray tomography. Scientific Reports. 10, 21045 (2020).
- Rybka, V., et al. Transmission electron microscopy study of mitochondria in aging brain synapses. Antioxidants. 8 (6), 171 (2019).
- Mannella, C. A. Structure and dynamics of the mitochondrial inner membrane cristae. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1763 (5-6), 542-548 (2006).
- Fry, M. Y., et al. In situ architecture of Opa1-dependent mitochondrial cristae remodeling. biorxiv. , (2023).
- Barad, B. A., Medina, M., Fuentes, D., Wiseman, R. L., Grotjahn, D. A. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. The Journal of Cell Biology. 222 (4), 202204093 (2023).
- Kunz, T. C., Götz, R., Gao, S., Sauer, M., Kozjak-Pavlovic, V. Using expansion microscopy to visualize and characterize the morphology of mitochondrial cristae. Frontiers in Cell and Developmental Biology. 8, 617 (2020).
- Yang, Z., et al. Cyclooctatetraene-conjugated cyanine mitochondrial probes minimize phototoxicity in fluorescence and nanoscopic imaging. Chemical Science. 11 (32), 8506-8516 (2020).
- Liu, T., et al. Multi-color live-cell STED nanoscopy of mitochondria with a gentle inner membrane stain. Proceedings of the National Academy of Sciences. 119 (52), e2215799119 (2022).
- Yang, X., et al. Mitochondrial dynamics quantitatively revealed by STED nanoscopy with an enhanced squaraine variant probe. Nature Communications. 11, 3699 (2020).
- Zheng, S., et al. Long-term, super-resolution HIDE imaging of the inner mitochondrial membrane in live cells with a cell-permeant lipid probe. biorxiv. , (2022).
- Wang, C., et al. A photostable fluorescent marker for the superresolution live imaging of the dynamic structure of the mitochondrial cristae. Proceedings of the National Academy of Sciences of the United States of America. 116 (32), 15817-15822 (2019).
- Feles, S., et al. Streamlining culture conditions for the neuroblastoma cell line sh-sy5y: a prerequisite for functional studies. Methods and Protocols. 5 (4), 58 (2022).
- Shipley, M. M., Mangold, C. A., Szpara, M. L. Differentiation of the SH-SY5Y human neuroblastoma cell line. Journal of Visualized Experiments. (108), e53193 (2016).
- Kovalevich, J., Langford, D., Amini, S., White, M. K. Considerations for the use of SH-SY5Y neuroblastoma cells in neurobiology. Neuronal Cell Culture. 1078, 9-21 (2013).
- Biedler, J. L., Helson, L., Spengler, B. A. Morphology and growth, tumorigenicity, and cytogenetics of human neuroblastoma cells in continuous culture. Cancer Research. 33 (11), 2643-2652 (1973).
- Swerdlow, R. H. Mitochondria and mitochondrial cascades in alzheimer's disease. Journal of Alzheimer's Disease. 62 (3), 1403-1416 (2018).
- Wang, W., Zhao, F., Ma, X., Perry, G., Zhu, X. Mitochondria dysfunction in the pathogenesis of Alzheimer's disease: recent advances. Molecular Neurodegeneration. 15, 30 (2020).
- Reddy, P. H. Mitochondrial dysfunction in aging and alzheimer's disease: strategies to protect neurons. Antioxidants & Redox Signaling. 9 (10), 1647-1658 (2007).
- Horn, A., Raavicharla, S., Shah, S., Cox, D., Jaiswal, J. K. Mitochondrial fragmentation enables localized signaling required for cell repair. Journal of Cell Biology. 219 (5), e201909154 (2020).
- Korecka, J. A., et al. Phenotypic characterization of retinoic acid differentiated SH-SY5Y cells by transcriptional profiling. PLoS ONE. 8 (5), e63862 (2013).
- Baghel, M. S., Thakur, M. K. Vdac1 downregulation causes mitochondrial disintegration leading to hippocampal neurodegeneration in scopolamine-induced amnesic mice. Molecular Neurobiology. 56 (3), 1707-1718 (2019).
- Jiang, S., et al. Mfn2 ablation causes an oxidative stress response and eventual neuronal death in the hippocampus and cortex. Molecular Neurodegeneration. 13 (1), 5 (2018).
- Mu, Y., Gage, F. H. Adult hippocampal neurogenesis and its role in Alzheimer's disease. Molecular Neurodegeneration. 6, 85 (2011).
- Rao, Y. L., et al. Hippocampus and its involvement in Alzheimer's disease: a review. 3 Biotech. 12 (2), 55 (2022).
- Weerasinghe-Mudiyanselage, P. D. E., Ang, M. J., Kang, S., Kim, J. -. S., Moon, C. Structural Plasticity of the hippocampus in neurodegenerative diseases. International Journal of Molecular Sciences. 23 (6), 3349 (2022).
- . Poly-D-Lysine Available from: https://www.thermofisher.com/order/catalog/product/A3890401 (2023)
- Dravid, A., Raos, B., Svirskis, D., O'Carroll, S. J. Optimised techniques for high-throughput screening of differentiated SH-SY5Y cells and application for neurite outgrowth assays. Scientific Reports. 11, 23935 (2021).
- Hromadkova, L., et al. Brain-derived neurotrophic factor (BDNF) promotes molecular polarization and differentiation of immature neuroblastoma cells into definitive neurons. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1867 (9), 118737 (2020).
- Riegerová, P., et al. Expression and Localization of AβPP in SH-SY5Y cells depends on differentiation state. Journal of Alzheimer's Disease. 82 (2), 485-491 (2021).
- Hoffmann, L. F., et al. Neural regeneration research model to be explored: SH-SY5Y human neuroblastoma cells. Neural Regeneration Research. 18 (6), 1265-1266 (2022).
- Abiraman, K., Tzingounis, A. V., Lykotrafitis, G. K. Ca 2 channel localization and regulation in the axon initial segment. The FASEB Journal. 32 (4), 1794-1805 (2018).
- E18 Rat Hippocampus. Transnetyx Tissue Available from: https://tissue.transnetyx.com/E18-Rat-Hippocampus_4 (2023)
- Kmito ORANGE - Probe for live cell imaging of mitochondria. Spirochrome Available from: https://spirochrome.com/product/pkmito_orange/ (2023)
- Nyquist Calculator. Scientific Volume Imaging Available from: https://svi.nl/Nyquist-Calculator (2023)
- Segawa, M., et al. Quantification of cristae architecture reveals time-dependent characteristics of individual mitochondria. Life Science Alliance. 3 (7), e2019000620 (2020).
- Arganda-Carreras, I., et al. Trainable Weka Segmentation: a machine learning tool for microscopy pixel classification. Bioinformatics. 33 (15), 2424-2426 (2017).
- Stephan, T., Roesch, A., Riedel, D., Jakobs, S. Live-cell STED nanoscopy of mitochondrial cristae. Scientific Reports. 9, 12419 (2019).
- Erdmann, R. S., et al. Labeling strategies matter for super-resolution microscopy: A comparison between HaloTags and SNAP-tags. Cell Chemical Biology. 26 (4), 584-592 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved