Un protocollo semplice, rapido e parzialmente automatizzato per l'isolamento di singoli nuclei da tessuti di mammiferi congelati per il sequenziamento di singoli nuclei
In This Article
Summary
Lo studio descrive un protocollo semplice, rapido e parzialmente automatizzato per isolare nuclei di alta qualità da tessuti di mammiferi congelati per il sequenziamento dell'RNA di singoli nuclei a valle.
Abstract
Il sequenziamento dell'RNA a singola cellula e a singolo nucleo è diventato un'applicazione comune in laboratorio grazie alla ricchezza di informazioni trascrittomiche che fornisce. Il sequenziamento dell'RNA a nucleo singolo, in particolare, è utile per studiare l'espressione genica in tessuti difficili da dissociare. Inoltre, questo approccio è compatibile anche con il materiale congelato (d'archivio). In questo articolo, descriviamo un protocollo per isolare singoli nuclei di alta qualità da tessuti di mammiferi congelati per il sequenziamento a valle dell'RNA di un singolo nucleo in modo parzialmente automatizzato utilizzando strumenti e reagenti disponibili in commercio. In particolare, viene utilizzato un dissociatore robotico per automatizzare e standardizzare l'omogeneizzazione dei tessuti, seguito da un gradiente chimico ottimizzato per filtrare i nuclei. Infine, contiamo in modo accurato e automatico i nuclei utilizzando un contatore di cellule fluorescente automatizzato. Le prestazioni di questo protocollo sono dimostrate sul cervello di topo, sul rene di ratto e sul tessuto epatico e milza di cynomolgus. Questo protocollo è semplice, rapido e facilmente adattabile a vari tessuti di mammiferi senza richiedere un'ampia ottimizzazione e fornisce nuclei di buona qualità per il sequenziamento dell'RNA di singoli nuclei a valle.
Introduction
Il sequenziamento dell'RNA a singola cellula (sc) e a singolo nucleo (sn) è diventato un protocollo comunemente usato in biologia molecolare e cellulare a causa della maggiore risoluzione dell'espressione genica rispetto al sequenziamento di massa dell'RNA. Tuttavia, l'isolamento di preparazioni di buona qualità a singola cellula e a singolo nucleo da tessuti solidi rimane una sfida ed è spesso la fase limitante negli esperimenti sc/sn-RNAseq. Infatti, è stata sviluppata una pletora di protocolli che utilizzano varie procedure chimiche e meccaniche per ottenere sospensioni cellulari/nucleiche 1,2,3,4,5,6,7,8,9,10,11,12,13,14,15 . Inoltre, le strategie per ripulire tali preparati da detriti/grumi, ecc., vanno dalla cernita del flusso alla filtrazione al lavaggio. Tali protocolli sono spesso manuali (con conseguente variabilità correlata all'utente), possono richiedere molto tempo (con conseguente riduzione della vitalità cellulare/nucleo) e/o possono richiedere l'accesso a un citometro a flusso per l'ordinamento di cellule/nuclei. Questo studio si è concentrato sullo sviluppo di un protocollo di isolamento di singoli nuclei semplice, rapido e parzialmente automatizzato da tessuti di mammiferi congelati per applicazioni di sequenziamento dell'RNA a valle. Ci siamo concentrati in particolare sull'isolamento dei nuclei rispetto all'isolamento cellulare in quanto è compatibile con l'uso di tessuti congelati, rendendo più pratica la raccolta/elaborazione dei campioni e consentendo un dosaggio imparziale dei campioni, specialmente negli esperimenti a tempo determinato. Inoltre, sebbene il trascrittoma nucleare non rifletta completamente il trascrittoma cellulare, diversi studi hanno ora dimostrato che i dati di sequenziamento dell'RNA a singolo nucleo sono paragonabili ai dati di sequenziamento dell'RNA a singola cellula per l'identificazione del tipo di cellula, anche se le proporzioni dei tipi di cellule possono variare 6,16,17,18,19.
L'isolamento dei nuclei consiste in diverse fasi: 1) disgregazione meccanica o chimica del tessuto per rilasciare i nuclei, 2) pulizia di detriti e grumi e 3) conteggio accurato dei nuclei per la preparazione per le applicazioni a valle. In un certo numero di protocolli, la fase 1 prevede spesso l'uso di un omogeneizzatore Dounce per distruggere il tessuto 3,20. In alternativa, possono essere utilizzati metodi chimici, anche se spesso devono essere ottimizzati per tessuti diversi 2,5,6. Abbiamo sperimentato che una procedura manuale di disgregazione tissutale è soggetta a variabilità associata all'operatore, portando a una qualità e a una resa dei nuclei variabili. Al fine di ridurre al minimo la variabilità tecnica e di avere un protocollo più coerente e riproducibile che funzioni su tutti i tessuti, è stato sviluppato un protocollo che utilizza un dissociatore tissutale robotico disponibile in commercio21. Per la fase 2, sebbene lo scambio di tamponi sia di solito il mezzo più semplice per lavare i nuclei, abbiamo adottato l'uso di una fase di centrifugazione a gradiente di saccarosio relativamente breve per avere una rimozione più completa dei detriti. Per il tessuto cerebrale in particolare, utilizziamo un gradiente colloidale di silice invece di un gradiente di saccarosio per una rimozione più efficace della mielina. Infine, per il conteggio, l'uso di un emocitometro è il gold standard per il conteggio e l'ispezione visiva dei nuclei. Nel nostro protocollo, questa fase può essere automatizzata in modo affidabile utilizzando un contatore di cellule fluorescenti automatizzato disponibile in commercio22. Questo protocollo è stato testato ed è compatibile con diversi tessuti congelati di mammiferi, tra cui cervello, rene, milza e fegato, di diverse specie di mammiferi (ratto, topo e primate non umano) e fornisce nuclei di buona qualità per il sequenziamento dell'RNA di singoli nuclei a valle con una piattaforma commerciale basata su goccioline. Il protocollo impiega circa 75 minuti dalla preparazione del tessuto all'inizio del flusso di lavoro di sequenziamento dell'RNA a singolo nucleo.
Protocol
Tutti gli studi sugli animali sono stati condotti con l'approvazione dell'autorità veterinaria cantonale di Basilea Città nel rigoroso rispetto delle disposizioni federali svizzere sulla protezione degli animali o con l'approvazione del Comitato istituzionale per la cura e l'uso degli animali in conformità con la legge tedesca sul benessere degli animali.
1. Preparazione di tessuti e reagenti/strumenti
- Pulizia e preparazione degli strumenti
- Pulire i piani di lavoro e le pinzette con etanolo al 70% e soluzione di decontaminazione RNasi. Preraffreddare le centrifughe a 4 °C.
- Preraffreddare le cartucce di isolamento dei nuclei in frigorifero a 4 °C per almeno 30 minuti.
- Avvia il dissociatore robotico e accendi il raffreddamento impostando il cursore in alto a destra dello schermo su raffreddamento e facendo clic su di esso per avviare il raffreddamento in modo che il cursore appaia arancione. Verificare che il flacone del reagente per la conservazione dei nuclei (NSR) collegato e il flacone del tampone di isolamento dei nuclei (NIB) abbiano abbastanza liquido e siano adeguatamente raffreddati.
- Preparare una scatola di polistirolo espanso riempita di ghiaccio secco e preraffreddare le piastre di Petri e le lame del bisturi su ghiaccio secco.
- Preparazione del tampone
- Preparare la soluzione a cuscino di saccarosio 1,5 M (SCS) come mostrato nella Tabella 1. Distribuire l'SCS in aliquote da 500 μL in provette DNasi/RNasi da 2 mL per ottenere quattro aliquote SCS da 500 μL per campione. Conservare le aliquote sul ghiaccio fino al nuovo utilizzo.
- In caso di trattamento del tessuto cerebrale, preparare invece una soluzione colloidale di silice al 18% come descritto nella Tabella 2, diluendo la soluzione madre di colloidi di silice in NSR e aggiungendo un inibitore della RNasi. Preparare 3 ml di soluzione colloidale di silice al 18% per campione e mantenerla in ghiaccio.
2. Omogeneizzazione tissutale e isolamento dei nuclei
- Rimuovere il campione dal congelatore a -80 °C e metterlo immediatamente su ghiaccio secco.
- Tagliare il campione su una piastra di Petri preraffreddata o su una piastra metallica su ghiaccio secco con un bisturi preraffreddato in un pezzo da 15-50 mg (se non è già della dimensione giusta). Assicurarsi di tagliare il campione nella direzione corretta in modo che sia ancora rappresentativo delle strutture organiche di interesse.
NOTA: Con questo protocollo, 15-50 mg è la dimensione ottimale del campione per l'estrazione nucleare. Per ottenere una buona resa dopo la pulizia, si raccomanda una dimensione del campione di almeno 25 mg. Per campioni più piccoli, sono disponibili cartucce speciali ottimizzate per l'elaborazione di piccoli ingressi con il dissociatore robotico. Sono state impiegate piccole cartucce di isolamento dei nuclei in ingresso per dissociare campioni di tessuto di peso minimo di 4 mg con una resa sufficiente per il sequenziamento dell'RNA a singolo nucleo a valle. Un esempio di resa dei nuclei utilizzando la cartuccia a basso input dal tessuto epatico di ratto è mostrato nella Tabella 3. - Rimuovere la cartuccia di isolamento dei nuclei dal frigorifero, disimballarla, rimuovere il macinacaffè e pipettare 15 μL di inibitore della RNasi (40 U/μL) sul fondo della cartuccia.
NOTA: Durante l'estrazione dei nuclei, il dissociatore robotico aggiungerà NIB e NSR alla cartuccia per un volume totale di 3 mL (con il protocollo di estrazione dei nuclei a basso volume). Aggiungendo 15 μL di inibitore della RNasi (40 U/μL) alla cartuccia prima dell'estrazione, la sospensione avrà la concentrazione desiderata di inibitore della RNAsi di 0,2 U/μL. - Posizionare il campione di tessuto sul fondo della cartuccia utilizzando una pinzetta. Non posizionare il campione esattamente al centro della cartuccia per avere un'efficienza di interruzione ottimale.
- Selezionare Esegui un protocollo sullo strumento e fare clic sull'opzione Nuclei nell'angolo in alto a sinistra.
- Dal menu, selezionare il protocollo Low Volume Nuclei Isolation e verificare che la velocità di interruzione sia impostata su veloce facendo clic su Modifica. Caricare la cartuccia sullo strumento aprendo lo sportello e facendo scorrere fuori il tavolino sollevando la manopola rossa.
- Inserire la cartuccia nella posizione designata, ruotare il blocco della cartuccia e far scorrere il stage finché la manopola rossa non scatta in posizione. Chiudere lo sportello e avviare l'estrazione dei nuclei sullo strumento facendo clic su Avanti. La corsa dura circa 7 minuti.
- Una volta terminata la corsa, rimuovere la cartuccia dallo strumento sollevando la manopola rossa ed estraendo il tavolino. Posizionare immediatamente la cartuccia sul ghiaccio.
- Per tutti i tessuti tranne il cervello, procedere al passaggio 3.1. Per i campioni cerebrali, procedere direttamente al passaggio 3.2.
3. Pulizia dei nuclei
- Pulizia del gradiente di saccarosio
NOTA: Per il tessuto cerebrale, saltare questo passaggio e andare direttamente al passaggio 3.2. Tutte le fasi di pulizia vengono eseguite su ghiaccio al fine di ridurre al minimo la degradazione dell'RNA. I tamponi e le provette, così come le centrifughe, devono essere preraffreddati. Tutte le fasi di risospensione e miscelazione vengono eseguite solo mediante un attento pipettaggio, in quanto il vortex potrebbe compromettere la qualità e l'integrità dei nuclei.- Forare con cautela la lamina rotonda sulla cartuccia del dissociatore con un puntale per pipetta.
NOTA: Dopo la dissociazione, la sospensione di nuclei risultante ha un volume approssimativo di 2 mL. Per facilitare la pulizia del gradiente di saccarosio, dividere la sospensione dei nuclei in due aliquote da 900 μL, ottenendo un volume totale di 1,8 mL di sospensione dei nuclei utilizzato durante la purificazione. - Rimuovere la prima aliquota di 900 μL della sospensione di nuclei dalla cartuccia e aggiungerla a un'aliquota SCS da 500 μL precedentemente preparata in una provetta da 2 mL. Mescolare bene pipettando fino ad ottenere un composto omogeneo.
- Rimuovere i 1400 μL di sospensione di nuclei - miscela SCS e sovrapporla con cura su una nuova aliquota SCS da 500 μL tenendo la provetta inclinata e aggiungendo la miscela goccia a goccia, creando una separazione di fase chiaramente visibile (vedere la Figura 1A).
- Chiudere con cautela il tubo e riposizionarlo sul ghiaccio senza disturbare la separazione di fase.
- Ripetere i passaggi da 3.1.2 a 3.1.4 con la seconda aliquota di sospensione da 900 μL e una nuova aliquota SCS per avere due provette da 2 mL per campione con una separazione di fase chiaramente visibile per la centrifugazione in gradiente.
- Aggiungere con cautela le provette a una centrifuga preraffreddata e centrifugare a 13.000 x g per 15 minuti a 4 °C.
- Nel frattempo, preparare l'NSR descritto nella Tabella 4, aggiungendo l'inibitore della RNasi ad un'aliquota di NSR. Preparare 1 mL di NSR per campione.
NOTA: A questo punto, le perle di gel del reagente per l'espressione genica a singola cellula possono essere rimosse dal congelatore a -80 °C, consentendo loro di equilibrarsi a temperatura ambiente (RT) e l'oligo dell'interruttore del modello può essere risospeso in un tampone a basso contenuto di TE. - Dopo la centrifugazione, rimuovere il surnatante da entrambe le provette senza disturbare il pellet e risospendere con cautela il pellet in 50 μL di NSR ghiacciato secondo le raccomandazioni del produttore. Raggruppare i due pellet dello stesso campione in una nuova provetta da 1,5 mL e aggiungere 900 μL di NSR ghiacciato a un volume totale di 1 mL. Mescolare bene pipettando.
- Centrifugare il campione a 500 x g per 5 minuti a 4 °C con una centrifuga a rotore a secchiello oscillante.
NOTA: Si consiglia vivamente di utilizzare un rotore a benna oscillante per ridurre al minimo la perdita di nuclei, soprattutto quando si prevede che la resa dei nuclei sia bassa o quando si inizia con piccole quantità di tessuto. - Nel frattempo, preparare 500 μL per campione di 1x PBS (senza Ca2+ e Mg2+) con albumina sierica bovina (BSA) allo 0,04% e inibitore della RNasi 0,2 U/μL come descritto nella Tabella 5.
- Rimuovere con cautela il surnatante senza scartare il pellet e risospendere il pellet in 100 μL di soluzione PBS come preparato sopra (1x PBS + 0,04% BSA + inibitore della RNasi a 0,2 U/μL).
NOTA: Per piccoli campioni di tessuto, la concentrazione di nuclei può essere bassa e, pertanto, si consiglia di risospendere il pellet in soli 50 μL di soluzione PBS per garantire concentrazioni sufficientemente elevate per il sequenziamento dell'RNA a nucleo singolo. - Procedere direttamente al passaggio 4.
- Forare con cautela la lamina rotonda sulla cartuccia del dissociatore con un puntale per pipetta.
- Pulizia del gradiente colloidale di silice
NOTA: Per il tessuto cerebrale, un gradiente colloidale di silice è più adatto di un gradiente di saccarosio per rimuovere la mielina e i detriti dalla sospensione dei nuclei. Tutte le fasi di pulizia vengono eseguite su ghiaccio per ridurre al minimo la degradazione dell'RNA. I tamponi e le provette, così come le centrifughe, devono essere preraffreddati. Tutte le fasi di risospensione e miscelazione vengono eseguite solo mediante un attento pipettaggio, in quanto il vortex potrebbe compromettere la qualità e l'integrità dei nuclei.- Forare con cautela la lamina rotonda sulla cartuccia del dissociatore con un puntale per pipetta.
- Rimuovere la sospensione di nuclei dalla cartuccia e aggiungerla a una provetta da 5 ml.
- Centrifugare a 500 x g per 5 minuti a 4 °C in una centrifuga preraffreddata.
- Rimuovere con cautela il surnatante senza disturbare il pellet e risospendere il pellet in 1 mL di soluzione colloidale ghiacciata al 18% di silice.
- Aggiungere altri 2 mL di soluzione colloidale di silice al 18% per ottenere un volume totale di 3 mL e mescolare bene pipettando.
- Centrifugare il campione a 700 x g per 5 minuti a 4 °C in un rotore a benna oscillante con il freno disinserito.
- Nel frattempo, preparare l'NSR descritto nella Tabella 4 aggiungendo l'inibitore della RNasi ad un'aliquota di NSR. Preparare 1 mL di NSR per campione.
NOTA: A questo punto, le perle di gel del reagente per l'espressione genica a singola cellula possono essere rimosse dal congelatore a -80 °C, consentendogli di equilibrarsi a RT e l'oligo switch del modello può essere risospeso in tampone a basso contenuto di TE. - Rimuovere con cautela il campione dalla centrifuga senza disturbare lo strato di mielina che galleggia sopra.
- Per prima cosa, rimuovere lo strato di mielina dalla parte superiore e scartare; Quindi, rimuovere con cautela l'intero surnatante senza disturbare il pellet.
NOTA: Lo strato di mielina può essere facilmente rimosso avvolgendo una salvietta sterile priva di lanugine attorno a un puntale di pipetta da 1 mL per aspirare lo strato di mielina insieme a 1-2 mL di surnatante. - Risospendere il pellet in 1 mL di NSR ghiacciato secondo le raccomandazioni del produttore.
- Centrifugare il campione a 500 x g per 5 minuti a 4 °C in una centrifuga con rotore a secchiello oscillante.
NOTA: Si consiglia vivamente di utilizzare un rotore a benna oscillante per ridurre al minimo la perdita di nuclei, soprattutto quando si prevede che la resa dei nuclei sia bassa o quando si inizia con piccole quantità di tessuto. - Nel frattempo, preparare 500 μL per campione di 1x PBS (senza Ca2+ e Mg2+) con albumina sierica bovina (BSA) allo 0,04% e inibitore della RNasi 0,2 U/μL come descritto nella Tabella 5.
- Rimuovere con cautela il surnatante senza disturbare il pellet e risospendere il campione in 100 μL di soluzione di PBS come preparato sopra (1x PBS + 0,04% BSA + inibitore della RNasi a 0,2 U/μL).
NOTA: Per piccoli campioni di tessuto, la concentrazione di nuclei può essere bassa e, pertanto, si consiglia di risospendere il pellet in soli 50 μL di soluzione PBS per garantire concentrazioni sufficientemente elevate per il sequenziamento dell'RNA a nucleo singolo.
4. Conteggio
- Per ogni campione da contare, diluire 10 μL di sospensione di nuclei in 20 μL di soluzione PBS per ottenere una diluizione 1:3.
- Per il conteggio, aggiungere 25 μL di soluzione colorante allo ioduro di propidio (PI) in un pozzetto di miscelazione della piastra contapezzi fluorescente. Aggiungere 25 μL della sospensione di nuclei diluiti e miscelare bene mediante pipettaggio. Trasferire il campione colorato da 50 μL dal pozzetto di miscelazione al pozzetto di carico.
- Caricare la piastra di conteggio sul contatore di celle e avviare il conteggio.
NOTA: La conta dei nuclei viene rilevata dal canale fluorescente rosso con un tempo di esposizione di 700 ms. Questo canale è stato ottimizzato per ottenere un conteggio accurato dei nuclei mediante un confronto incrociato con il conteggio manuale con una camera di Neubauer e la colorazione Trypan Blue al microscopio. Ad alte concentrazioni, i nuclei sono molto vicini tra loro, rendendo difficile per il software separarli. In questo caso, si consiglia di ricontare il campione in una diluizione adeguata. L'integrità dei nuclei e la pulizia possono essere valutate dall'immagine in campo chiaro o al microscopio. - Diluire i campioni con PBS (1x PBS + 0,04% BSA + inibitore della RNasi a 0,2 U/μL) alla concentrazione desiderata per il sequenziamento dell'RNA a nucleo singolo.
NOTA: Concentrazioni comprese tra 700 e 1.200 nuclei/μL sono considerate ottimali per il sequenziamento dell'RNA a singolo nucleo. Concentrazioni cellulari più basse, come 700 nuclei/μL, possono comportare una riduzione della contaminazione di fondo da RNA ambientale.
5. Preparazione della biblioteca
- Eseguire il sequenziamento dell'RNA di singoli nuclei con i reagenti di espressione genica a singola cellula utilizzando il protocollo del produttore che mira a un recupero di nuclei di 8000-10.000 nuclei per campione.
6. Sequenziamento
- Sequenziare le librerie con la profondità di sequenziamento desiderata con l'indicizzazione doppia a due estremità e le seguenti letture di sequenziamento: Lettura 1: 28 cicli, Indice i7: 10 cicli, Indice i5: 10 cicli e Lettura 2: 90 cicli.
Representative Results
Le prestazioni e la versatilità di questo protocollo sono dimostrate dall'esecuzione del sequenziamento dell'RNA a singolo nucleo su tessuto della corteccia occipitale cerebrale congelato fresco di tre topi B6, tessuto renale fresco congelato tagliato trasversalmente da tre ratti Wistar, fegato archiviato (11 anni) e tessuto della milza di tre macachi Cynomolgus delle Mauritius. Tutti gli animali non erano perfusi.
Come mostrato nelle Figure 1B,C, sono stati ottenuti nuclei di buona qualità privi di segni di blebbing, detriti e aggregazioni. La filtrazione basata sul gradiente di saccarosio è stata ottimizzata per rimuovere la maggior parte dei detriti testando diverse densità, velocità e tempi di rotazione e valutando la purezza/integrità nucleare al microscopio, nonché valutando la distribuzione delle dimensioni e la resa dei nuclei (Figura 1D). Questo ci ha permesso di scegliere una densità di gradiente di saccarosio di 1,5 M e di utilizzare un breve tempo di centrifuga di 15 minuti. Successivamente, per valutare ulteriormente la qualità dei nuclei, i dati sono stati pre-elaborati utilizzando 10X Cell Ranger e un'ulteriore analisi dei dati a valle è stata eseguita utilizzando Besca23. I nuclei con il >5% di contenuto mitocondriale (poiché questi tendono ad essere nuclei danneggiati/stressati) sono stati filtrati e i nuclei con 500-7.000 geni (per ridurre al minimo le goccioline vuote e i multipletti) sono stati mantenuti. Abbiamo incluso solo i geni che erano presenti in almeno 30 nuclei. Abbiamo preso di mira 8.000 nuclei per campione di corteccia cerebrale e 10.000 nuclei per campione di rene, fegato e milza. Dopo la filtrazione, sono stati ottenuti 10.644 nuclei di alta qualità dai tre campioni di cervello, 14.960 nuclei di alta qualità dai tre campioni di rene, 18.795 nuclei di alta qualità dai tre campioni di fegato e 13.882 nuclei di alta qualità dai tre campioni di milza. Le Figure 2A,D,G,J mostrano i grafici a violino che rappresentano la distribuzione della conta UMI, della conta genica e del contenuto mitocondriale in ciascun campione. Il numero mediano di conteggi in tutti i campioni cerebrali è stato di 7.563 UMI/nucleo e 3.208 geni/nucleo. Il numero mediano di conteggi in tutti i campioni renali è stato di 3.841 UMI/nucleo e 1.915 geni/nucleo. Il numero mediano di conteggi in tutti i campioni di fegato è stato di 2.649 UMI/nuclei e 1.676 geni/nuclei. Il numero mediano di conteggi in tutti i campioni di milza è stato di 1.609 UMI/nuclei e 1.138 geni/nuclei. Abbiamo quindi generato cluster utilizzando geni altamente variabili e li abbiamo annotati utilizzando i geni marcatori noti 17,24,25,26. Come si vede nella Figura 2B,E,H,K, siamo stati in grado di identificare i tipi di cellule attesi da ciascun tessuto. Inoltre, come si vede nella Figura 2B,E,H,K, tutti gli animali hanno contribuito a tutti i cluster, indicando una bassa variabilità tecnica complessiva introdotta dal protocollo. Inoltre, le proporzioni cellulari erano comparabili in tutti e tre i campioni per tipo di tessuto, così come l'UMI e la conta genica (Figura 2A, C, D, F, G, I, J, L). Un'eccezione degna di nota è il fegato, dove le popolazioni di epatociti tra i tre campioni di fegato erano diverse nelle proporzioni e nel profilo. Ciò è probabilmente dovuto alle differenze biologiche tra gli animali (sesso, età, stato metabolico).
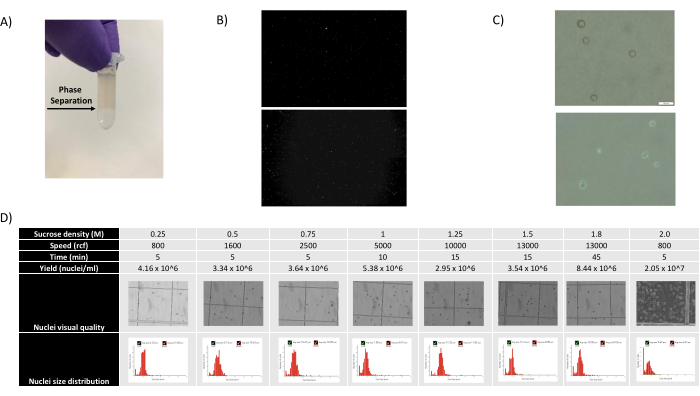
Figura 1: Valutazione della qualità dei nuclei e ottimizzazione del gradiente di saccarosio. (A) La separazione di fase prevista durante la centrifugazione in gradiente di saccarosio è indicata con una freccia. (B) Immagini fluorescenti rappresentative dei nuclei di rene di ratto (in alto) e milza di cynomolgus (in basso) colorati con ioduro di propidio ottenuti con il protocollo. (C) Immagini rappresentative al microscopio in campo chiaro di nuclei isolati dal fegato di topo (in alto) e dal cervello di topo (in basso), barra graduata 500 μm. Si noti la superficie regolare e liscia dei nuclei che indica una buona qualità nucleare. (D) Ottimizzazione del gradiente di saccarosio. Sono state testate diverse densità, velocità di centrifuga e tempi di centrifuga. Per ciascuna condizione vengono mostrate immagini al microscopio in campo chiaro dei nuclei, della distribuzione dimensionale dei nuclei e della resa dei nuclei. Fare clic qui per visualizzare una versione più grande di questa figura.
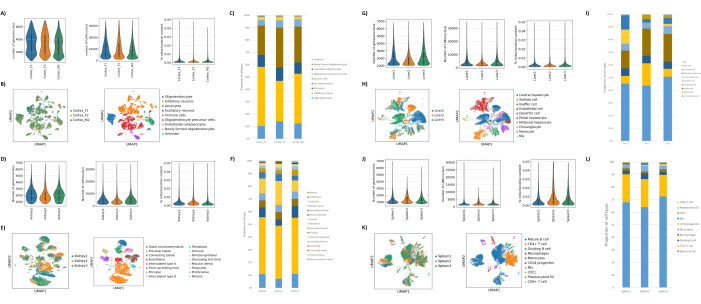
Figura 2: Dati rappresentativi di snRNAseq sulla corteccia occipitale cerebrale di topo, sul rene di ratto (corteccia e midollo) e sul fegato e sulla milza del macaco cynomolgus. (A) Grafici a violino che mostrano la distribuzione di geni/nucleo, UMI/nucleo e percentuale di contenuto mitocondriale per campione di cervello. (B) Pannello di sinistra: grafico UMAP che mostra il contributo di ciascun campione ai cluster identificati nel cervello. Pannello di destra: UMAP che mostra le identità dei cluster annotati in base ai geni marcatori nel tessuto cerebrale. (C) Proporzioni cellulari osservate nei 3 campioni cerebrali. (D) Grafici a violino che mostrano la distribuzione di geni/nucleo, UMI/nucleo e percentuale di contenuto mitocondriale per campione di rene. (E) Pannello di sinistra: grafico UMAP che mostra il contributo di ciascun campione ai cluster identificati nel rene. Pannello di destra: UMAP che mostra le identità dei cluster annotati in base ai geni marcatori nel tessuto renale. (F) Proporzioni cellulari osservate nei 3 campioni di rene. (G) Grafici a violino che mostrano la distribuzione di geni/nucleo, UMI/nucleo e percentuale di contenuto mitocondriale per campione di fegato. (H) Pannello di sinistra: grafico UMAP che mostra il contributo di ciascun campione ai cluster identificati nel fegato. Pannello di destra: UMAP che mostra le identità dei cluster annotati in base ai geni marcatori nel tessuto epatico. (I) Proporzioni cellulari osservate nei 3 campioni di fegato. (J) Grafici a violino che mostrano la distribuzione di geni/nucleo, UMI/nucleo e percentuale di contenuto mitocondriale per campione di milza. (K) Pannello di sinistra: grafico UMAP che mostra il contributo di ciascun campione ai cluster identificati nella milza. Pannello di destra: UMAP che mostra l'identità dei cluster annotati in base ai geni marcatori nel tessuto della milza. (L) Proporzioni cellulari osservate nei 3 campioni di milza. Fare clic qui per visualizzare una versione più grande di questa figura.
| Componenti | Concentrazione delle scorte | Volume per campione | Concentrazione finale |
| Soluzione di cuscino di saccarosio | 2 Mio: | 1500 μL | 1,5 Mio. |
| Tampone cuscino di saccarosio | - | 500 μL | - |
| Ditiotreitolo (DTT) | 1 M | 2 μL | 1 millimetro |
| Inibitore dell'RNAsi | 40 U/μL | 10 μL | 0,2 U/μL |
Tabella 1: Preparazione della soluzione a cuscino di saccarosio 1,5 M (SCS). Questa soluzione viene utilizzata per la centrifugazione a gradiente di saccarosio durante la purificazione nella fase 3.1 e deve essere preparata ogni volta prima di iniziare il protocollo. Tenere sempre l'SCS sul ghiaccio durante il protocollo. Le soluzioni menzionate in questa tabella sono indicate nella Tabella dei materiali.
| Componenti | Concentrazione delle scorte | Volume per campione | Concentrazione finale |
| Soluzione madre colloidale di silice | 90% | 600 μL | 18% |
| Reagente per la conservazione dei nuclei (genomica S2) | - | 2400 μL | - |
| Inibitore dell'RNAsi | 40 U/μL | 15 μL | 0,2 U/μL |
Tabella 2: Preparazione della soluzione colloidale di silice al 18%. Questa soluzione viene utilizzata per la centrifugazione a gradiente colloidale di silice durante la pulizia nella fase 3.2 e deve essere preparata ogni volta prima di iniziare il protocollo. Tenere sempre la soluzione colloidale di silice al 18% sul ghiaccio durante il protocollo.
| Fazzoletto | Peso del campione | Cartuccia | Cedere |
| Fegato di ratto | magnesio 25 | Cartuccia di isolamento dei nuclei | 65.000 nuclei per mg di tessuto |
| Fegato di ratto | 4 magnesio | Cartuccia di isolamento dei nuclei di ingresso di piccole dimensioni | 32.000 nuclei per mg di tessuto |
Tabella 3: Resa dei nuclei dalla cartuccia di isolamento dei nuclei a basso ingresso rispetto alla cartuccia di isolamento dei nuclei dopo la pulizia del gradiente di saccarosio.
| Componenti | Concentrazione delle scorte | Volume per campione | Concentrazione finale |
| Reagente per la conservazione dei nuclei | - | 1000 μL | - |
| Inibitore dell'RNAsi | 40 U/μL | 5 μL | 0,2 U/μL |
Tabella 4: Preparazione del reagente per la conservazione dei nuclei (NSR). Questa soluzione viene utilizzata durante l'isolamento dei nuclei nei passaggi 3-5 e durante la pulizia nel passaggio 3.1.8. Può essere conservato a 4 °C per un massimo di 4 mesi. Preparare un'aliquota fresca con l'inibitore della RNasi durante la fase di centrifugazione nella fase di purificazione 6. Le soluzioni menzionate in questa tabella sono indicate nella Tabella dei materiali.
| 1x PBS + soluzione madre BSA allo 0,04% | |||
| Componenti | Concentrazione delle scorte | Volume per magazzino | Concentrazione finale |
| PBS (senza Ca 2+, senza Mg2+) | 1x | 30 ml | - |
| Albumina sierica bovina (BSA) | 30% | 40 μL | 0.04% |
| 1x PBS + 0,04% BSA + 0,2 U/μL RNAsi inibitore | |||
| Componenti | Concentrazione delle scorte | Volume per campione | Concentrazione finale |
| 1x PBS + 0,04% soluzione madre BSA | - | 500 μL | - |
| Inibitore dell'RNAsi | 40 U/μL | 2,5 μL | 0,2 U/μL |
Tabella 5: Preparazione di PBS + 0,04% BSA. Questa soluzione viene utilizzata alla fine della purificazione nella fase 3.1.10 e dopo il conteggio per diluire la sospensione di nuclei alla concentrazione desiderata per il sequenziamento di RNA a 10 volte a nucleo singolo (fase di conteggio 4.4). La soluzione madre può essere conservata a 4 °C per un massimo di 1 mese. Preparare un'aliquota fresca con l'inibitore della RNasi durante la fase di centrifugazione nella fase di purificazione 6.
Discussion
Abbiamo sviluppato un protocollo versatile e parzialmente automatizzato per ottenere singoli nuclei di alta qualità da tessuti congelati di mammifero e abbiamo dimostrato il protocollo su cervello di topo, rene di ratto e tessuto epatico e milza di cynomolgus.
Confrontando le prestazioni di questo protocollo con quelle di altri protocolli pubblicati per il sequenziamento dell'RNA a nucleo singolo nel cervello, nei reni, nella milza e nel tessuto epatico 6,7,20,24,25,26, osserviamo che siamo in grado di rilevare un numero simile di geni e conteggi UMI per nucleo e siamo in grado di recuperare i tipi di cellule attesi. Rispetto ai metodi esistenti, questo protocollo presenta diversi vantaggi. In primo luogo, il protocollo in questo studio automatizza l'omogeneizzazione dei tessuti e l'isolamento dei singoli nuclei. Ciò si ottiene con l'uso di un disgregatore tissutale robotico21. Nella maggior parte dei protocolli, il tessuto viene omogeneizzato con un omogeneizzatore Dounce per liberare i singoli nuclei 3,20. Tuttavia, abbiamo notato che questo passaggio manuale può portare a variabilità sperimentale nella resa e nell'integrità dei nuclei a seconda della quantità di forza esercitata durante l'omogeneizzazione, compromettendo la riproducibilità degli esperimenti. In questo caso, utilizzando una smerigliatrice automatica per tessuti con impostazioni fisse, sono stati ottenuti una buona qualità dei nuclei e una resa con maggiore coerenza in tutti gli esperimenti. Inoltre, l'automazione di questa fase riduce anche il tempo di esecuzione del protocollo (la fase di rottura del tessuto richiede circa 7 minuti), consentendo all'utente di prepararsi per le fasi successive. In secondo luogo, il protocollo descritto in questo studio è versatile, cioè è compatibile con diversi tessuti di specie diverse. Questo ci permette di evitare lunghe ottimizzazioni del protocollo, ad esempio per identificare tamponi/detergenti di omogeneizzazione per diversi tessuti 2,5,6. In terzo luogo, questo protocollo non dipende dall'accesso a un selezionatore a flusso per ottenere nuclei puliti, rendendolo così più accessibile per i laboratori che non dispongono delle attrezzature/competenze necessarie per lo smistamento a flusso. Invece, abbiamo ottimizzato la filtrazione basata sul gradiente di saccarosio per rimuovere la maggior parte dei detriti. Tuttavia, per il tessuto cerebrale in particolare, si raccomanda l'uso di un gradiente colloidale di silice invece di un gradiente di saccarosio per una rimozione più efficiente della mielina. Abbiamo anche scoperto che l'uso di un rotore a secchiello oscillante alla fine della fase di centrifugazione a gradiente colloidale saccarosio/silice riduce al minimo la perdita di nuclei. Pertanto, l'uso di un tale rotore è altamente raccomandato. In quarto luogo, dopo aver testato più metodi per contare i nuclei (conteggio manuale al microscopio, uso di diversi contatori automatici), si raccomanda l'uso di un contatore automatico di cellule fluorescenti22. L'uso di un colorante intercalante del DNA, come lo ioduro di propidio, aumenta l'accuratezza del conteggio dei nuclei. In quinto luogo, questo protocollo richiede circa 75 minuti dall'inizio al caricamento del chip microfluidico. Ciò contribuisce a garantire che l'integrità dei nuclei rimanga elevata durante l'elaborazione di più campioni. Infine, abbiamo riscontrato che il protocollo è compatibile anche con il tessuto incorporato nel composto a temperatura di taglio ottimale (OCT). Se si utilizza tale materiale, il tessuto può essere rimosso dal blocco OCT utilizzando un bisturi prima dell'omogeneizzazione.
Una sfida frequente nei set di dati di sequenziamento dell'RNA a singolo nucleo è la presenza di RNA ambientale, che può essere non nucleare (ad esempio, mitocondriale) e di derivazione nucleare27,28. Nel nostro protocollo, l'RNA mitocondriale (un proxy per l'RNA ambientale non nucleare) è basso anche prima del filtraggio (0,1-1,6% per i tessuti mostrati). Tuttavia, analogamente ad altri protocolli e set di dati, la contaminazione dell'RNA ambientale da geni altamente espressi nei nuclei di abbondanti tipi di cellule (come gli epatociti nel fegato, i neuroni nel cervello, ecc.) è ancora presente27. Esistono diversi strumenti bioinformatici, come CellBender, SoupX, ecc., in grado di rimuovere tale contaminazione dell'RNA ambientale prima dell'annotazione dei nuclei 29,30,31. Un'altra limitazione di questo protocollo è che, sebbene le fasi di disgregazione dei tessuti e di isolamento dei nuclei siano automatizzate, la produttività di questa fase è ancora limitante in quanto è possibile processare un solo campione alla volta. Tuttavia, poiché questa fase richiede solo circa 7 minuti per pezzo di tessuto, è comunque possibile elaborare più campioni in un lotto. In genere elaboriamo quattro campioni per lotto, ma abbiamo eseguito fino a sei campioni per lotto con buoni risultati. I recenti miglioramenti nel dissociatore robotico per consentire l'elaborazione parallela di due campioni contemporaneamente consentiranno l'elaborazione di 8-12 campioni per lotto, il che è compatibile con la produttività del chip microfluidico utilizzato per l'incapsulamento di singoli nuclei.
Sebbene non abbiamo utilizzato i nuclei isolati da questo protocollo per altre applicazioni a valle come ATAC-seq o snRNAseq utilizzando altre piattaforme, sulla base della qualità dei dati ottenuti con i reagenti di espressione genica qui utilizzati, riteniamo che il nostro protocollo dovrebbe essere compatibile con ulteriori applicazioni a valle. Tuttavia, il lavoro futuro comporterà il test di questo protocollo con altre applicazioni a valle, come ATAC-seq.
In conclusione, abbiamo sviluppato un protocollo di isolamento dei nuclei rapido, semplice e parzialmente automatizzato per il sequenziamento a valle dell'RNA di un singolo nucleo che si è dimostrato compatibile con diversi tipi di tessuti congelati di mammifero.
Disclosures
Tutti gli autori sono/erano dipendenti di F. Hoffmann-La Roche durante la conduzione dello studio.
Acknowledgements
Gli autori desiderano ringraziare Filip Bochner, Marion Richardson, Petra Staeuble e Matthias Selhausen per aver fornito i tessuti animali che sono stati analizzati in questo manoscritto. Vorremmo anche ringraziare Petra Schwalie, Klas Hatje, Roland Schmucki e Martin Ebeling per il loro supporto bioinformatico.
Materials
| Name | Company | Catalog Number | Comments |
| 1 M DTT | Thermo Fisher Scientific | P2325 | |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 10x Magnetic Separator | 10x genomics | PN-120250 | |
| 10x Vortex Adapter | 10x genomics | PN-120251 | |
| 1x DPBS (10x), no calcium, no magnesium | Thermo Fisher Scientific | 14190144 | stored at 4°C |
| 30% Bovine Serum Albumin | Sigma-Aldrich | A9576_50ML | |
| 400 mM Tris-HCl, pH 8.0 | Thermo Fisher Scientific | 15568025 | |
| 40U/μl RNaseOUT Recombinant Ribonuclease Inhibitor | Thermo Fisher Scientific | 10777019 | Stored at -20 °C |
| Agilent High Sensitivity DNA Kit | Agilent | 5067-4626 | |
| Cellaca MX High-throughput Automated Cell Counter | Nexcelom Bioscience | CELMXSYSF2 | Automated fluorescent cell counter |
| Chromium Next GEM Chip G Single Cell Kit, 16 rxns | 10x genomics | PN-1000127 | Single cell gene expression reagent, stored at room temperature |
| Chromium Next GEM Secondary Holder | 10x genomics | PN-1000195 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1, 4 rxns | 10x genomics | PN-1000129 | Single cell gene expression reagent, stored at -80 °C |
| Chromium Next GEM Single Cell 3' GEM, Library & Gel Bead Kit v3.1, 4 rxns | 10x genomics | PN-1000128 | Single cell gene expression reagent |
| Chromium Next GEM Single Cell 3' Library Kit v3.1 4 rxns | 10x genomics | PN-1000158 | Single cell gene expression reagent, stored at -20 °C |
| Chromium Next GEM Single Cell 3'GEM Kit v3.1 4 rxns | 10x genomics | PN-1000130 | Single cell gene expression reagent, stored at -20 °C |
| Divided Polystyrene Reservoirs | VWR | 41428-958 | |
| DNA LoBind Tubes 1.5ml Eppendorf | Sigma-Aldrich | EP0030108051 | |
| DNA LoBind Tubes 2ml Eppendorf | Sigma-Aldrich | EP0030108078 | |
| Dry ice | - | - | |
| Dynabeads MyOne SILANE | 10x genomics | PN-2000048 | Single cell gene expression reagent, stored at 4 °C |
| Ethanol Pure | Sigma-Aldrich | E7023 | |
| Glycerin (Glycerol), 50% (v/v) | Ricca Chemical Company | 3290-16 | |
| Heatblock | |||
| High-Throughput Nexcelom Counting Plates | Nexcelom Bioscience | CHM24-A100-001 | Cell counter counting plate |
| Low TE Buffer (10 mM Tris-HCl pH 8.0, 0.1 mM EDTA) | Thermo Fisher Scientific | 12090015 | |
| Mini Centrifuge | - | - | |
| NovaSeq 6000 SP Reagent Kit v1.5 (100 cycles) | Illumina | 2002840 | |
| Nuclei Isolation Buffer | S2 Genomics | 100-063-396 | Stored at 4 °C |
| Nuclei Isolation Cartridge | S2 Genomics | 100-063-287 | Precooled at 4 °C before use |
| Nuclei PURE 2 M Sucrose Cushion Solution | Sigma-Aldrich | NUC201-1KT | Sucrose cushion solution |
| Nuclei PURE Sucrose Cushion Buffer | Sigma-Aldrich | NUC201-1KT | |
| Nuclei Storage Reagent | S2 Genomics | 100-063-405 | Stored at 4 °C |
| PCR Tubes 0.2 ml 8-tube strips | Eppendorf | 30124359 | |
| Percoll | GE Healthcare | 17-0891-02 | Silica colloid solution |
| PhiX Control v3 | Illumina | FC-110-3001 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific | Q32854 | |
| Refrigerated Centrifuge (Eppendorf 5804R) | Eppendorf | 5805000010 | |
| Refrigerated Centrifuge with Swinging-Bucket Rotor (Eppendorf 5810R) | Eppendorf | 5811000015 | |
| RNAseZap | Ambion | AM9780 | RNAse decontamination solution |
| Round cell culture petri dish | SPL | 330005 | |
| Scalpel disposable | Aesculap AG | BA210 | pre-cooled on dry ice before use |
| Single Index Kit T Set A, 96 rxns | 10x genomics | PN-1000213 | Single cell gene expression reagent, stored at -20 °C |
| Singulator 100 System | S2 Genomics | - | Commercially available robotic tissue dissociator |
| Sodium Hydroxide 1M | Sigma-Aldrich | 72068 | |
| SPRIselect Reagent Kit | Beckman Coulter | b23318 | |
| Sterile tweezers | - | - | |
| UltraPure DNase/RNase-Free Distilled Water | Thermo Fisher Scientific | 10977049 | |
| ViaStain PI Staining Solution | Nexcelom Bioscience | CS1-0109-5mL | Propidium iodide staining solution |
| Vortex Mixer+A2:D44 | VWR | - |
References
- Burja, B., et al. An Optimized Tissue Dissociation Protocol for Single-Cell RNA Sequencing Analysis of Fresh and Cultured Human Skin Biopsies. Front Cell Dev Biol. 10, 872688 (2022).
- Kimbley, L. M., et al. Comparison of optimized methodologies for isolating nuclei from esophageal tissue. Biotechniques. 72 (3), 104-109 (2022).
- Maitra, M., et al. Extraction of nuclei from archived postmortem tissues for single-nucleus sequencing applications. Nature Protocols. 16 (6), 2788-2801 (2021).
- Nadelmann, E. R., et al. Isolation of nuclei from mammalian cells and tissues for single-nucleus molecular profiling. Current Protocols. 1 (5), e132 (2021).
- Rousselle, T. V., et al. An optimized protocol for single nuclei isolation from clinical biopsies for RNA-seq. Scientific Reports. 12, 9851 (2022).
- Slyper, M., et al. A single-cell and single-nucleus RNA-Seq toolbox for fresh and frozen human tumors. Nature Medicine. 26 (5), 792-802 (2020).
- Leiz, J., et al. Nuclei isolation from adult mouse kidney for single-nucleus RNA-sequencing. Journal of Visualized Experiments: JoVE. (175), 62901 (2021).
- Alvarez, M., et al. Isolation of nuclei from human snap-frozen liver tissue for single-nucleus RNA sequencing. Bio-Protocol. 13 (3), e4601 (2023).
- Ayhan, F., Douglas, C., Lega, B. C., Konopka, G. Nuclei isolation from surgically resected human hippocampus. STAR Protocols. 2 (4), 100844 (2021).
- Joshi, N., Misharin, A. Single-nucleus isolation from frozen human lung tissue for single-nucleus RNA-seq. Protocols.io. , (2019).
- Martelotto, L. G., Luciano Martelotto, L. 'Frankenstein' protocol for nuclei isolation from fresh and frozen tissue for snRNAseq. Protocols.io. , (2020).
- Masilionis, I., Chaudhary, O., Chaligne, R., Mazutis, L. Nuclei extraction for single-cell RNAseq from frozen tissue using Singulator™ 100. Protocols.io. , (2022).
- Matson, K. J. E., et al. Isolation of adult spinal cord nuclei for massively parallel single-nucleus RNA sequencing. Journal of Visualized Experiments: JoVE. (140), 58413 (2018).
- Mendelev, N., et al. Multi-omics profiling of single nuclei from frozen archived postmortem human pituitary tissue. STAR Protocols. 3 (2), 101446 (2022).
- Soule, T. G., et al. A protocol for single nucleus RNA-seq from frozen skeletal muscle. Life Science Alliance. 6 (5), e202201806 (2023).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648 (2018).
- Ding, J., et al. Systematic comparison of single-cell and single-nucleus RNA-sequencing methods. Nature Biotechnology. 38, 737-746 (2020).
- Hu, P., et al. Single-nucleus transcriptomic survey of cell diversity and functional maturation in postnatal mammalian hearts. Genes & Development. 32 (19-20), 1344-1357 (2018).
- Lake, B. B., et al. A comparative strategy for single-nucleus and single-cell transcriptomes confirms accuracy in predicted cell-type expression from nuclear RNA. Scientific Reports. 7, 6031 (2017).
- Narayanan, A., et al. Nuclei Isolation from Fresh Frozen Brain Tumors for Single-Nucleus RNA-seq and ATAC-seq. Journal of Visualized Experiments: JoVE. (162), 61542 (2020).
- Jovanovich, S., et al. . Automated processing of solid tissues into single cells or nuclei for genomics and cell biology applications with the Singulator™ 100 and 200 systems. , (2022).
- Bell, J., et al. Characterization of a novel high-throughput, high-speed and high-precision plate-based image cytometric cell counting method. Cell & Gene Therapy Insights. 7 (4), 427-447 (2021).
- Madler, S. C., et al. Besca, a single-cell transcriptomics analysis toolkit to accelerate translational research. NAR Genomics and Bioinformatics. 3 (4), lqab102 (2021).
- Wu, H., et al. Mapping the single-cell transcriptomic response of murine diabetic kidney disease to therapies. Cell Metabolism. 34 (7), 1064-1078 (2022).
- Han, L., et al. Cell transcriptomic atlas of the non-human primate Macaca fascicularis. Nature. 604 (7907), 723-731 (2022).
- Madissoon, E., et al. scRNA-seq assessment of the human lung, spleen, and esophagus tissue stability after cold preservation. Genome Biology. 21 (1), 1 (2019).
- Caglayan, E., Liu, Y., Konopka, G. Neuronal ambient RNA contamination causes misinterpreted and masked cell types in brain single-nuclei datasets. Neuron. 110 (24), 4043-4056 (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Molecular Systems Biology. 15 (6), e8746 (2019).
- Fleming, S. J., et al. Unsupervised removal of systematic background noise from droplet-based single-cell experiments using CellBender. bioRxiv. , (2022).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biology. 21 (1), 57 (2020).
- Young, M. D., Behjati, S. SoupX removes ambient RNA contamination from droplet-based single-cell RNA sequencing data. Gigascience. 9 (12), giaa151 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved