Простой, быстрый и частично автоматизированный протокол выделения одиночных ядер из замороженных тканей млекопитающих для секвенирования одного ядра
In This Article
Summary
В исследовании описывается простой, быстрый и частично автоматизированный протокол выделения высококачественных ядер из замороженных тканей млекопитающих для последующего секвенирования одноядерной РНК.
Abstract
Секвенирование одноклеточной и одноядерной РНК стало распространенным лабораторным применением из-за большого количества транскриптомного информации, которую они предоставляют. Секвенирование одноядерной РНК, в частности, полезно для изучения экспрессии генов в трудно диссоциированных тканях. Кроме того, этот подход совместим и с замороженными (архивными) материалами. В данной статье мы опишем протокол выделения высококачественных одиночных ядер из замороженных тканей млекопитающих для последующего секвенирования одноядерной РНК частично автоматизированным способом с использованием коммерчески доступных инструментов и реагентов. В частности, роботизированный диссоциатор используется для автоматизации и стандартизации гомогенизации тканей с последующим оптимизированным химическим градиентом для фильтрации ядер. Наконец, мы точно и автоматически подсчитываем ядра с помощью автоматического счетчика флуоресцентных клеток. Эффективность этого протокола демонстрируется на мозге мыши, почках крыс, а также на ткани печени и селезенки циномолгуса. Этот протокол прост, быстр и легко адаптируется к различным тканям млекопитающих, не требуя обширной оптимизации, и обеспечивает хорошее качество ядер для последующего секвенирования одноядерной РНК.
Introduction
Секвенирование одноклеточной (sc) и одноядерной (sn) РНК стало широко используемым протоколом в молекулярной и клеточной биологии из-за более высокого разрешения экспрессии генов по сравнению с секвенированием объемной РНК. Тем не менее, выделение высококачественных одноклеточных и одноядерных препаратов из твердых тканей остается сложной задачей и часто является этапом, ограничивающим скорость в экспериментах sc/sn-RNAseq. Действительно, было разработано множество протоколов, в которых используются различные химические и механические процедуры для получения клеточных/ядерных суспензий 1,2,3,4,5,6,7,8,9,10,11,12,13,14,15 . Кроме того, стратегии очистки таких препаратов от мусора/комков и т. д. варьируются от сортировки по потоку до фильтрации и промывки. Такие протоколы часто являются ручными (что приводит к изменчивости, связанной с пользователем), могут занимать много времени (приводя к снижению жизнеспособности клетки/ядра) и/или могут требовать доступа к проточному цитометру для сортировки клеток/ядер. Это исследование было сосредоточено на разработке простого, быстрого и частично автоматизированного протокола выделения отдельных ядер из замороженных тканей млекопитающих для последующего секвенирования РНК. Мы сосредоточились именно на выделении ядер, а не на выделении клеток, поскольку оно совместимо с использованием замороженных тканей, что делает сбор/обработку образцов более практичным и позволяет беспристрастно группировать образцы, особенно в экспериментах с течением времени. Кроме того, несмотря на то, что ядерный транскриптом не полностью отражает клеточный транскриптом, несколько исследований показали, что данные секвенирования РНК одного ядра сопоставимы с данными секвенирования РНК одной клетки для идентификации типа клеток, хотя пропорции типов клеток могут варьироваться 6,16,17,18,19.
Выделение ядер состоит из нескольких этапов: 1) механическое или химическое разрушение ткани для высвобождения ядер, 2) очистка от мусора и комков и 3) точный подсчет ядер для подготовки к последующему применению. В ряде протоколов этап 1 часто включает в себя использование гомогенизатора Dounce для того, чтобы разрушить ткань 3,20. В качестве альтернативы можно использовать химические методы, хотя они часто должны быть оптимизированы для различных тканей 2,5,6. Мы убедились в том, что мануальная процедура разрушения тканей подвержена изменчивости, связанной с оператором, что приводит к изменению качества и выхода ядер. Для того, чтобы свести к минимуму техническую вариабельность и иметь более последовательный и воспроизводимый протокол, работающий в разных тканях, был разработан протокол, в котором используется коммерчески доступный роботизированный диссоциатор тканей21. На этапе 2, несмотря на то, что буферный обмен обычно является простейшим способом промывки ядер, мы использовали относительно короткую стадию центрифугирования градиента сахарозы для более тщательного удаления мусора. В частности, для мозговой ткани мы используем коллоидный градиент кремнезема вместо градиента сахарозы для более эффективного удаления миелина. Наконец, для подсчета использование гемоцитометра является золотым стандартом подсчета и визуального осмотра ядер. В нашем протоколе этот шаг может быть надежно автоматизирован с помощью коммерчески доступного автоматизированного счетчика флуоресцентных клеток22. Этот протокол был протестирован и совместим с несколькими замороженными тканями млекопитающих, включая мозг, почки, селезенку и печень, от различных видов млекопитающих (крыс, мышей и приматов) и обеспечивает ядра хорошего качества для последующего секвенирования одноядерной РНК с помощью коммерческой платформы на основе капель. Протокол занимает около 75 минут от подготовки ткани до начала процесса секвенирования одноядерной РНК.
Protocol
Все исследования на животных проводились с одобрения кантонального ветеринарного управления земли Базель-Штадт в строгом соответствии с федеральными правилами Швейцарии по защите животных или с одобрения Комитета по уходу за животными и их использованию в соответствии с Законом о благополучии животных.
1. Подготовка тканей и реагентов/инструментов
- Очистка и подготовка инструмента
- Очистите столешницы и пинцет 70% раствором для обеззараживания этанолом и РНКазы. Предварительно охладите центрифуги до 4 °C.
- Предварительно охладите картриджи с выделением ядер в холодильнике при температуре 4 °C в течение не менее 30 минут.
- Запустите роботизированный диссоциатор и включите охлаждение, установив ползунок в правом верхнем углу экрана на охлаждение и нажав на него, чтобы начать охлаждение, чтобы ползунок стал оранжевым. Убедитесь, что в прикрепленном флаконе с реагентом для хранения ядер (NSR) и бутылке с буфером для изоляции ядер (NIB) осталось достаточно жидкости и они должным образом охлаждены.
- Подготовьте пенополистирольную коробку, наполненную сухим льдом, и предварительно охладите чашки Петри и лезвия скальпеля на сухом льду.
- Подготовка буфера
- Приготовьте 1,5 М раствор сахарозы (SCS), как показано в таблице 1. Распределите ГКС по 500 мкл аликвот в пробирках ДНКаза/РНКазы по 2 мл для получения четырех аликвот ГКС по 500 мкл на образец. Держите аликвоты на льду до дальнейшего использования.
- В случае обработки мозговой ткани вместо него готовят 18%-ный коллоидный раствор кремнезема, как описано в таблице 2, разбавляя раствор коллоида кремнезема в NSR и добавляя ингибитор РНКазы. Приготовьте 3 мл 18% коллоидного раствора кремнезема на образец и храните его на льду.
2. Гомогенизация тканей и выделение ядер
- Извлеките образец из морозильной камеры при температуре -80 °C и сразу же поместите его на сухой лед.
- Нарежьте образец на предварительно охлажденной чашке Петри или металлической пластине на сухом льду предварительно охлажденным скальпелем на кусочек по 15-50 мг (если он еще не подходящего размера). Обязательно разрежьте образец в правильном направлении, чтобы образец по-прежнему был репрезентативным для интересующих вас структур органов.
ПРИМЕЧАНИЕ: При использовании этого протокола оптимальным размером образца для ядерной экстракции является 15-50 мг. Для достижения хорошего урожая после очистки рекомендуется размер образца не менее 25 мг. Для небольших образцов доступны специальные картриджи, оптимизированные для обработки небольших входных данных с помощью робота-диссоциатора. Для диссоциации образцов тканей массой всего 4 мг с достаточным выходом для последующего секвенирования РНК одного ядра использовались картриджи для выделения небольших входных ядер. Пример выхода ядер с использованием картриджа с низким входом из ткани печени крысы приведен в таблице 3. - Достаньте картридж с выделением ядер из холодильника, распакуйте его, извлеките измельчитель и запишите пипеткой 15 мкл ингибитора РНКазы (40 Ед/мкл) на дно картриджа.
ПРИМЕЧАНИЕ: Во время экстракции ядер роботизированный диссоциатор добавляет NIB и NSR в картридж до общего объема 3 мл (с протоколом экстракции ядра малого объема). Добавив 15 мкл ингибитора РНКазы (40 Ед/мкл) в картридж перед экстракцией, суспензия будет иметь желаемую концентрацию ингибитора РНКазы 0,2 Ед/мкл. - Поместите образец ткани на дно картриджа с помощью пинцета. Не размещайте образец точно в центре картриджа, чтобы обеспечить оптимальную эффективность разрушения.
- Выберите «Запустить протокол» на приборе и нажмите на опцию «Ядра» в левом верхнем углу.
- В меню выберите протокол Low Volume Nuclei Isolation (Изоляция ядра малого объема ) и убедитесь, что скорость прерывания установлена на низкую, нажав кнопку Изменить. Зарядите картридж на инструмент, открыв дверцу и выдвинув сцену, подняв красную ручку.
- Вставьте картридж в указанное место, поверните фиксатор картриджа и сдвиньте его в предметный столик до щелчка. Закройте дверцу и запустите процесс извлечения ядер на приборе, нажав кнопку Далее. Продолжительность пробежки составляет около 7 минут.
- После завершения прогона извлеките картридж из прибора, подняв красную ручку и вытащив столик. Сразу же поместите картридж на лед.
- Для всех тканей, кроме головного мозга, переходите к шагу 3.1. Для образцов мозга перейдите непосредственно к шагу 3.2.
3. Очистка ядер
- Очистка градиента сахарозы
ПРИМЕЧАНИЕ: Для мозговой ткани пропустите этот шаг и перейдите сразу к шагу 3.2. Все этапы очистки выполняются на льду, чтобы свести к минимуму деградацию РНК. Буферы и пробирки, а также центрифуги необходимо предварительно охладить. Все этапы ресуспендирования и смешивания выполняются только путем осторожного пипетирования, так как вихрь может нарушить качество и целостность ядер.- Осторожно проткните наконечником пипетки круглую фольгу на картридже диссоциатора.
ПРИМЕЧАНИЕ: После диссоциации полученная суспензия ядер имеет приблизительный объем 2 мл. Чтобы облегчить очистку градиента сахарозы, разделите суспензию ядер на две аликвоты по 900 мкл, в результате чего во время очистки используется общий объем суспензии ядер 1,8 мл. - Первую 900 мкл аликвоты суспензии ядер извлеките из картриджа и добавьте ее к 500 мкл аликвоты ГКС, предварительно приготовленной в пробирке объемом 2 мл. Хорошо перемешайте пипеткой, пока смесь не станет однородной.
- Извлеките 1400 мкл суспензии ядер - смеси ГКС и осторожно наложите ее на новую аликвоту ГКС объемом 500 мкл, держа трубку под углом и добавляя смесь по каплям, создавая хорошо видимое разделение фаз (см. Рисунок 1А).
- Осторожно закройте пробирку и поместите ее обратно на лед, не нарушая разделения фаз.
- Повторите шаги 3.1.2-3.1.4 со второй аликвотой суспензии 900 мкл и новой аликвотой SCS, чтобы получить две пробирки по 2 мл на образец с четко видимым разделением фаз для градиентного центрифугирования.
- Осторожно добавьте пробирки в предварительно охлажденную центрифугу и отжим при 13 000 x g в течение 15 минут при 4 °C.
- Тем временем приготовьте СМП, описанный в таблице 4, добавив ингибитор РНКазы к аликвоте СМР. Приготовьте 1 мл NSR на образец.
ПРИМЕЧАНИЕ: На этом этапе гранулы геля реагента экспрессии одноклеточных генов могут быть извлечены из морозильной камеры -80 °C, что позволяет им уравновеситься до комнатной температуры (RT), а шаблонный переключатель олиго может быть ресуспендирован в буфере с низким TE. - После центрифугирования удалите надосадочную жидкость из обеих пробирок, не повредив гранулу, и осторожно ресуспендируйте гранулу в 50 мкл ледяного NSR в соответствии с рекомендациями производителя. Объедините две гранулы одного образца в новую пробирку объемом 1,5 мл и добавьте 900 мкл ледяного NSR до общего объема 1 мл. Хорошо перемешайте пипеткой.
- Центрифужируют образец при 500 x g в течение 5 мин при 4 °C с помощью роторной центрифуги с поворотным ковшом.
ПРИМЕЧАНИЕ: Настоятельно рекомендуется использовать ротор с качающимся ковшом, чтобы свести к минимуму потерю ядер, особенно когда ожидается низкий выход ядер или при запуске с небольшим количеством ткани. - Тем временем приготовьте 500 мкл на образец 1x PBS (без Ca2+ и Mg2+) с 0,04% бычьим сывороточным альбумином (BSA) и ингибитором РНКазы 0,2 U/мкл, как описано в таблице 5.
- Осторожно удалите надосадочную жидкость, не выбрасывая гранулу, и повторно суспендируйте гранулу в 100 мкл раствора PBS, как было приготовлено выше (1x PBS + 0,04% BSA + ингибитор РНКазы в дозе 0,2 U/мкл).
ПРИМЕЧАНИЕ: Для небольших образцов тканей концентрация ядер может быть низкой, поэтому рекомендуется повторно суспендировать гранулу только в 50 мкл раствора PBS, чтобы обеспечить достаточно высокие концентрации для секвенирования одноядерной РНК. - Переходите непосредственно к шагу 4.
- Осторожно проткните наконечником пипетки круглую фольгу на картридже диссоциатора.
- Очистка коллоидного градиента диоксида кремния
ПРИМЕЧАНИЕ: Для мозговой ткани коллоидный градиент кремнезема больше подходит, чем градиент сахарозы, для удаления миелина и мусора из суспензии ядер. Все этапы очистки выполняются на льду, чтобы свести к минимуму деградацию РНК. Буферы и пробирки, а также центрифуги необходимо предварительно охладить. Все этапы ресуспендирования и смешивания выполняются только путем осторожного пипетирования, так как вихрь может нарушить качество и целостность ядер.- Осторожно проткните наконечником пипетки круглую фольгу на картридже диссоциатора.
- Извлеките суспензию ядер из картриджа и добавьте ее в пробирку объемом 5 мл.
- Центрифуга при 500 x g в течение 5 мин при 4 °C в предварительно охлажденной центрифуге.
- Осторожно удалите надосадочную жидкость, не повредив гранулу, и повторно суспендируйте гранулу в 1 мл ледяного 18% раствора коллоида диоксида кремния.
- Добавьте дополнительно 2 мл 18% раствора коллоида кремнезема, чтобы получить общий объем 3 мл, и хорошо перемешайте пипетированием.
- Центрифугируют образец при 700 x g в течение 5 мин при 4 °C в роторе с качающимся ковшом с выключенным тормозом.
- Тем временем приготовьте NSR, описанный в таблице 4 , добавив ингибитор РНКазы к аликвоте NSR. Приготовьте 1 мл NSR на образец.
ПРИМЕЧАНИЕ: На этом этапе гелевые шарики реагента экспрессии генов одиночных клеток могут быть извлечены из морозильной камеры при температуре -80 °C, что позволяет им уравновеситься до RT, а олиго переключателя шаблона может быть ресуспендировано в буфере с низким TE. - Осторожно извлеките образец из центрифуги, не нарушая плавающий сверху слой миелина.
- Во-первых, снимите миелиновый слой сверху и выбросьте; Затем осторожно удалите всю надосадочную жидкость, не потревожив гранулу.
ПРИМЕЧАНИЕ: Миелиновый слой можно легко удалить, обернув стерильную безворсовую салфетку вокруг наконечника пипетки объемом 1 мл, чтобы аспирировать миелиновый слой вместе с 1-2 мл надосадочной жидкости. - Ресуспендируйте гранулу в 1 мл ледяного NSR в соответствии с рекомендациями производителя.
- Центрифугу образца при 500 x g в течение 5 мин при 4 °C в центрифуге с ротором с качающимся ковшом.
ПРИМЕЧАНИЕ: Настоятельно рекомендуется использовать ротор с качающимся ковшом, чтобы свести к минимуму потерю ядер, особенно когда ожидается низкий выход ядер или при запуске с небольшим количеством ткани. - Тем временем приготовьте 500 мкл на образец 1x PBS (без Ca2+ и Mg2+) с 0,04% бычьим сывороточным альбумином (BSA) и ингибитором РНКазы 0,2 U/мкл, как описано в таблице 5.
- Осторожно удалите надосадочную жидкость, не повредив гранулу, и повторно суспендируйте образец в 100 мкл раствора PBS, как было приготовлено выше (1x PBS + 0,04% BSA + ингибитор РНКазы в концентрации 0,2 Ед/мкл).
ПРИМЕЧАНИЕ: Для небольших образцов тканей концентрация ядер может быть низкой, поэтому рекомендуется повторно суспендировать гранулу только в 50 мкл раствора PBS, чтобы обеспечить достаточно высокие концентрации для секвенирования одноядерной РНК.
4. Подсчет
- Для каждого образца, подлежащего подсчету, разводят 10 мкл суспензии ядер в 20 мкл раствора PBS до получения разведения 1:3.
- Для подсчета добавьте 25 мкл раствора окрашивания йодида пропидия (PI) в смесительную лунку флуоресцентного счетчика. Добавьте 25 мкл разбавленной суспензии ядер и хорошо перемешайте пипетированием. Переместите окрашенный образец объемом 50 мкл из смесительного колодца в загрузочный колодец.
- Загрузите счетную пластину на счетчик ячеек и начните подсчет.
ПРИМЕЧАНИЕ: Количество ядер берется из красного флуоресцентного канала с временем экспозиции 700 мс. Этот канал был оптимизирован для получения точного подсчета ядер путем перекрестного сравнения с ручным подсчетом с помощью камеры Нойбауэра и окрашивания трипановым синим под микроскопом. При высоких концентрациях ядра расположены очень близко друг к другу, что затрудняет их разделение программным обеспечением. В этом случае рекомендуется пересчитать пробу в подходящем разведении. Целостность ядер, как и чистоту, можно оценить по яркому полевому изображению или под микроскопом. - Разбавляют образцы PBS (1x PBS + 0,04% BSA + ингибитор РНКазы в концентрации 0,2 U/мкл) до желаемой концентрации для секвенирования одноядерной РНК.
ПРИМЕЧАНИЕ: Концентрации от 700 до 1200 ядер/мкл считаются оптимальными для секвенирования одноядерной РНК. Более низкие концентрации в клетках, такие как 700 ядер/мкл, могут привести к уменьшению фонового загрязнения окружающей РНК.
5. Подготовка библиотеки
- Выполняйте секвенирование одноядерной РНК с помощью реагентов экспрессии генов одной клетки, используя протокол производителя, нацеленный на восстановление ядер 8000-10 000 ядер на образец.
6. Секвенирование
- Упорядочивайте библиотеки с требуемой глубиной секвенирования с помощью парного двойного индексирования и следующего чтения последовательности: чтение 1: 28 циклов, индекс i7: 10 циклов, индекс i5: 10 циклов и чтение 2: 90 циклов.
Representative Results
Эффективность и универсальность этого протокола продемонстрированы при выполнении секвенирования одноядерных РНК на свежезамороженной ткани затылочной коры головного мозга от трех мышей B6, свежезамороженной поперечно разрезанной ткани почек от трех крыс линии Wistar, архивной (11-летней) ткани печени и селезенки от трех маврикийских маврикийских макак Cynomolgus. Все животные не подвергались перфузии.
Как показано на рисунках 1В, В, были получены ядра хорошего качества, которые не имели признаков блеббинга, обломков и слипания. Фильтрация на основе градиента сахарозы была оптимизирована для удаления большей части мусора путем тестирования различных плотностей, скоростей и времени вращения, а также оценки чистоты/целостности ядра под микроскопом, а также оценки распределения ядер по размерам и выхода (рис. 1D). Это позволило нам выбрать плотность градиента сахарозы 1,5 М и использовать короткое время отжима 15 минут. Затем, для дальнейшей оценки качества ядер, данные были предварительно обработаны с помощью 10X Cell Ranger, а дальнейший анализ данных был выполнен с помощью Besca23. Ядра с содержанием митохондрий >5% (так как они, как правило, являются поврежденными/стрессовыми ядрами) отфильтровывались, а ядра с 500-7000 генами (чтобы свести к минимуму пустые капли и мультиплеты) сохранялись. Мы включили только те гены, которые присутствовали, по крайней мере, в 30 ядрах. Мы нацелились на 8 000 ядер на образец коры головного мозга и 10 000 ядер на образец почки, печени и селезенки. После фильтрации было получено 10 644 высококачественных ядра из трех образцов мозга, 14 960 высококачественных ядер из трех образцов почек, 18 795 высококачественных ядер из трех образцов печени и 13 882 высококачественных ядра из трех образцов селезенки. На рисунках 2A, D, G, J показаны графики скрипки, представляющие распределение количества UMI, количества генов и содержания митохондрий в каждом образце. Среднее количество подсчетов во всех образцах мозга составило 7 563 UMI на ядро и 3 208 генов на ядро. Среднее количество подсчетов во всех образцах почек составило 3841 UMI/ядро и 1915 генов/ядро. Среднее число подсчетов во всех образцах печени составило 2649 UMI/ядер и 1676 генов/ядер. Медиана количества подсчетов во всех образцах селезенки составила 1609 UMI/ядер и 1138 генов/ядер. Затем мы сгенерировали кластеры с использованием высоковариабельных генов и аннотировали их, используя известные гены-маркеры 17,24,25,26. Как видно на рисунке 2B, E, H, K, мы смогли определить ожидаемые типы клеток из каждой ткани. Кроме того, как видно на рисунке 2B, E, H, K, все животные внесли свой вклад во все кластеры, что указывает на общую низкую техническую вариативность, введенную протоколом. Кроме того, клеточные пропорции были сопоставимы во всех трех образцах для каждого типа ткани, как и количество UMI и генов (рис. 2A, C, D, F, G, I, J, L). Заметным исключением является печень, где популяции гепатоцитов среди трех образцов печени различались по пропорциям и профилю. Скорее всего, это связано с биологическими различиями между животными (пол, возраст, метаболический статус).
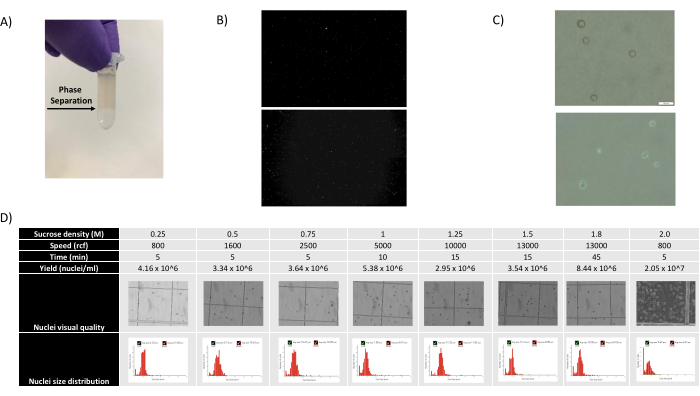
Рисунок 1: Оценка качества ядер и оптимизация градиента сахарозы . (A) Ожидаемое разделение фаз при градиентном центрифугировании сахарозы показано стрелкой. (B) Репрезентативные флуоресцентные изображения ядер почек крысы, окрашенных йодидом пропидия (вверху) и циномолгусной селезенки (внизу), полученные с помощью протокола. (C) Репрезентативные изображения ядра ядра, выделенного из печени мыши (вверху) и мозга мыши (внизу), масштабная линейка 500 мкм. Обратите внимание на правильную гладкую поверхность ядер, указывающую на хорошее качество ядра. (D) Оптимизация градиента сахарозы. Были протестированы несколько плотностей сахарозы, скорости вращения и времени отжима. Для каждого условия показаны изображения ядра с помощью светлопольной микроскопии, распределения ядер по размерам и выхода ядер. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
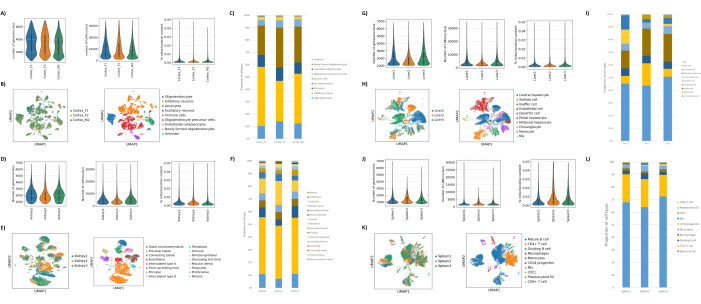
Рисунок 2: Репрезентативные данные snRNAseq по затылочной коре головного мозга мышей, почкам крыс (кора и мозговое вещество), а также печени и селезенке циномолгусных макак. (A) Графики Скрипки, показывающие распределение генов/ядер, UMI/ядра и процентное содержание митохондрий в образце мозга. (B) Левая панель: график UMAP, показывающий вклад каждого образца в кластеры, идентифицированные в мозге. Правая панель: UMAP, показывающий идентичность кластеров, аннотированных на основе маркерных генов в тканях мозга. (C) Клеточные пропорции, наблюдаемые в 3 образцах мозга. (D) Графики Скрипки, показывающие распределение генов/ядер, UMI/ядра и процентное содержание митохондрий в образце почки. (E) Левая панель: график UMAP, показывающий вклад каждого образца в кластеры, идентифицированные в почке. Правая панель: UMAP, показывающий идентификаторы кластеров, аннотированные на основе маркерных генов в почечной ткани. (F) Клеточные пропорции, наблюдаемые в 3 образцах почек. (G) Графики Скрипки, показывающие распределение генов/ядра, UMI/ядра и процентное содержание митохондрий в образце печени. (H) Левая панель: график UMAP, показывающий вклад каждого образца в кластеры, идентифицированные в печени. Правая панель: UMAP, показывающий идентификаторы кластеров, аннотированные на основе маркерных генов в ткани печени. (I) Клеточные пропорции, наблюдаемые в 3 образцах печени. (J) Графики Скрипки, показывающие распределение генов/ядра, UMI/ядра и процентное содержание митохондрий в образце селезенки. (K) Левая панель: график UMAP, показывающий вклад каждого образца в кластеры, идентифицированные в селезенке. Правая панель: UMAP, показывающий идентичность кластеров, аннотированных на основе маркерных генов в ткани селезенки. (L) Клеточные пропорции, наблюдаемые в 3 образцах селезенки. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
| Компоненты | Концентрация запасов | Объем на образец | Конечная концентрация |
| Раствор подушки сахарозы | 2 М | 1500 мкл | 1,5 м |
| Буфер подушки сахарозы | - | 500 мкл | - |
| Дитиотрейтол (DTT) | 1 М | 2 мкл | 1 мМ |
| Ингибитор РНКазы | 40 Ед/мкл | 10 мкл | 0,2 Ед/мкл |
Таблица 1: Приготовление 1,5 М раствора сахарозной подушки (СКС). Этот раствор используется для градиентного центрифугирования сахарозы во время очистки на этапе 3.1 и должен быть приготовлен заново каждый раз перед началом протокола. Всегда держите СКС на льду во время протокола. Решения, упомянутые в этой таблице, приведены в таблице материалов.
| Компоненты | Концентрация запасов | Объем на образец | Конечная концентрация |
| Кремнеземный коллоидный исходный раствор | 90% | 600 мкл | 18% |
| Реагент для хранения ядер (S2 Genomics) | - | 2400 мкл | - |
| Ингибитор РНКазы | 40 Ед/мкл | 15 мкл | 0,2 Ед/мкл |
Таблица 2: Приготовление 18% раствора кремнезема коллоидного. Этот раствор используется для коллоидного градиентного центрифугирования кремнезема во время очистки на этапе 3.2 и должен быть приготовлен заново каждый раз перед началом протокола. Всегда держите 18% коллоидный раствор кремнезема на льду во время протокола.
| Ткань | Масса образца | Патрон | Урожай |
| Печень крысы | 25 мг | Картридж для выделения ядер | 65 000 ядер на мг ткани |
| Печень крысы | 4 мг | Малый входной картридж для изоляции ядер | 32 000 ядер на мг ткани |
Таблица 3: Выход ядра из картриджа для выделения ядер с низким входом по сравнению с картриджем с выделением ядер после очистки градиента сахарозы.
| Компоненты | Концентрация запасов | Объем на образец | Конечная концентрация |
| Реагент для хранения ядер | - | 1000 мкл | - |
| Ингибитор РНКазы | 40 Ед/мкл | 5 мкл | 0,2 Ед/мкл |
Таблица 4: Подготовка реагента для хранения ядер (СМП). Этот раствор используется при выделении ядер на шагах 3-5, а также при очистке на шагах 3.1.8. Его можно хранить при температуре 4 °C до 4 месяцев. Приготовьте свежую аликвоту с ингибитором РНКазы на этапе центрифугирования на этапе очистки 6. Решения, упомянутые в этой таблице, приведены в таблице материалов.
| 1x PBS + 0,04% исходный раствор BSA | |||
| Компоненты | Концентрация запасов | Объем на складе | Конечная концентрация |
| PBS (без Ca 2+, без Mg2+) | В 1 раз | 30 мл | - |
| Бычий сывороточный альбумин (BSA) | 30% | 40 мкл | 0.04% |
| 1x PBS + 0,04% BSA + 0,2 U/мкл ингибитора РНКазы | |||
| Компоненты | Концентрация запасов | Объем на образец | Конечная концентрация |
| 1x PBS + 0,04% BSA Stock Solution | - | 500 мкл | - |
| Ингибитор РНКазы | 40 Ед/мкл | 2,5 мкл | 0,2 Ед/мкл |
Таблица 5: Приготовление ПБС + 0,04% БСА. Этот раствор используют в конце очистки на стадии 3.1.10 и после подсчета разбавляют суспензию ядер до требуемой концентрации для 10-кратного секвенирования РНК одиночных ядер (шаг подсчета 4.4). Исходный раствор можно хранить при температуре 4 °C до 1 месяца. Приготовьте свежую аликвоту с ингибитором РНКазы на этапе центрифугирования на этапе очистки 6.
Discussion
Мы разработали универсальный и частично автоматизированный протокол получения высококачественных одиночных ядер из замороженных тканей млекопитающих и продемонстрировали протокол на мозге мыши, почках крыс, циномольгусной ткани печени и селезенки.
Сравнивая эффективность этого протокола с другими опубликованными протоколами секвенирования одноядерной РНК в тканях головного мозга, почек, селезенки и печени 6,7,20,24,25,26, мы видим, что мы можем обнаружить аналогичное количество генов и количество UMI в ядре и можем восстановить ожидаемые типы клеток. По сравнению с существующими методами, у этого протокола есть несколько преимуществ. Во-первых, протокол в этом исследовании автоматизирует гомогенизацию тканей и выделение отдельных ядер. Это достигается с помощью роботизированного разрушителя тканей21. В большинстве протоколов ткань гомогенизируется с помощью гомогенизатора Dounce с целью высвобождения одиночных ядер 3,20. Однако мы заметили, что этот ручной шаг может привести к экспериментальной изменчивости выхода ядер и целостности в зависимости от величины силы, приложенной во время гомогенизации, что ставит под угрозу воспроизводимость экспериментов. Здесь, с помощью автоматизированного измельчителя тканей с фиксированными настройками, было получено хорошее качество ядер и выход с большей стабильностью в ходе экспериментов. Кроме того, автоматизация этого этапа также сокращает время, затрачиваемое на выполнение протокола (этап разрушения тканей занимает около 7 минут), что позволяет пользователю подготовиться к последующим шагам. Во-вторых, протокол, описанный в этом исследовании, универсален, т.е. совместим с разными тканями разных видов. Это позволяет избежать длительной оптимизации протокола, например, для определения гомогенизационных буферов/детергентов для различных тканей 2,5,6. В-третьих, этот протокол не зависит от доступа к поточному сортировщику для получения чистых ядер, что делает его более доступным для лабораторий, которые не имеют необходимого оборудования/опыта для поточной сортировки. Вместо этого мы оптимизировали фильтрацию на основе градиента сахарозы, чтобы удалить большую часть мусора. Однако, в частности, для мозговой ткани рекомендуется использовать коллоидный градиент кремния вместо градиента сахарозы для более эффективного удаления миелина. Мы также обнаружили, что использование ротора с качающимся ковшом в конце стадии коллоидного градиентного центрифугирования сахарозы/кремнезема сводит к минимуму потерю ядер. Следовательно, использование такого ротора настоятельно рекомендуется. В-четвертых, после тестирования нескольких методов подсчета ядер (ручной подсчет под микроскопом, использование нескольких автоматических счетчиков) рекомендуется использовать автоматизированный флуоресцентный счетчик клеток22. Использование интеркалирующего красителя ДНК, такого как йодид пропидия, повышает точность подсчета ядер. В-пятых, этот протокол занимает около 75 минут от запуска до загрузки микрофлюидного чипа. Это помогает гарантировать, что целостность ядер остается высокой при обработке нескольких образцов. Наконец, мы обнаружили, что протокол также совместим с тканями, внедренными в компаунд с оптимальной температурой резания (OCT). При использовании такого материала ткань может быть удалена из ОКТ-блока с помощью скальпеля перед гомогенизацией.
Одной из частых проблем в наборах данных секвенирования одноядерных РНК является наличие эмбиентной РНК, которая может быть как неядерной (например, митохондриальной), так и ядерной. В нашем протоколе уровень митохондриальной РНК (аналог неядерной окружающей РНК) низок даже до фильтрации (0,1-1,6% для показанных тканей). Однако, как и в случае с другими протоколами и наборами данных, загрязнение окружающей среды РНК высокоэкспрессируемыми генами в ядрах распространенных типов клеток (таких как гепатоциты в печени, нейроны в мозге и т. д.) всееще присутствует. Существует несколько биоинформатических инструментов, таких как CellBender, SoupX и т.д., которые могут удалить такое загрязнение окружающей РНК до аннотации ядер 29,30,31. Еще одно ограничение этого протокола заключается в том, что, несмотря на то, что этапы разрушения тканей и выделения ядер автоматизированы, пропускная способность этого этапа по-прежнему ограничена, поскольку одновременно может быть обработан только один образец. Однако, поскольку этот этап занимает всего около 7 минут на каждый кусочек ткани, несколько образцов все еще могут быть обработаны в партии. Обычно мы обрабатываем четыре образца за партию, но с хорошими результатами мы получаем до шести образцов за партию. Недавние усовершенствования в роботизированном диссоциаторе, позволяющие параллельно обрабатывать два образца одновременно, позволят обрабатывать 8-12 образцов за партию, что совместимо с пропускной способностью микрофлюидного чипа, используемого для инкапсуляции отдельных ядер.
Несмотря на то, что мы не использовали ядра, выделенные по этому протоколу, для других нисходящих приложений, таких как ATAC-seq или snRNAseq с использованием других платформ, основываясь на качестве данных, полученных с помощью используемых здесь реагентов экспрессии генов, мы считаем, что наш протокол должен быть совместим с другими нисходящими приложениями. Однако будущая работа будет включать в себя тестирование этого протокола с другими последующими приложениями, такими как ATAC-seq.
В заключение, мы разработали быстрый, простой и частично автоматизированный протокол выделения ядер для последующего секвенирования одноядерной РНК, который, как было продемонстрировано, совместим с различными типами замороженных тканей млекопитающих.
Disclosures
Все авторы являются/были сотрудниками F. Hoffmann-La Roche во время проведения исследования.
Acknowledgements
Авторы хотели бы поблагодарить Филипа Бохнера, Марион Ричардсон, Петру Штойбле и Маттиаса Зельхаузена за предоставленные ткани животных, которые были проанализированы в этой рукописи. Мы также хотели бы поблагодарить Петру Швали, Класа Хатье, Роланда Шмуки и Мартина Эбелинга за их поддержку в области биоинформатики.
Materials
| Name | Company | Catalog Number | Comments |
| 1 M DTT | Thermo Fisher Scientific | P2325 | |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 10x Magnetic Separator | 10x genomics | PN-120250 | |
| 10x Vortex Adapter | 10x genomics | PN-120251 | |
| 1x DPBS (10x), no calcium, no magnesium | Thermo Fisher Scientific | 14190144 | stored at 4°C |
| 30% Bovine Serum Albumin | Sigma-Aldrich | A9576_50ML | |
| 400 mM Tris-HCl, pH 8.0 | Thermo Fisher Scientific | 15568025 | |
| 40U/μl RNaseOUT Recombinant Ribonuclease Inhibitor | Thermo Fisher Scientific | 10777019 | Stored at -20 °C |
| Agilent High Sensitivity DNA Kit | Agilent | 5067-4626 | |
| Cellaca MX High-throughput Automated Cell Counter | Nexcelom Bioscience | CELMXSYSF2 | Automated fluorescent cell counter |
| Chromium Next GEM Chip G Single Cell Kit, 16 rxns | 10x genomics | PN-1000127 | Single cell gene expression reagent, stored at room temperature |
| Chromium Next GEM Secondary Holder | 10x genomics | PN-1000195 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1, 4 rxns | 10x genomics | PN-1000129 | Single cell gene expression reagent, stored at -80 °C |
| Chromium Next GEM Single Cell 3' GEM, Library & Gel Bead Kit v3.1, 4 rxns | 10x genomics | PN-1000128 | Single cell gene expression reagent |
| Chromium Next GEM Single Cell 3' Library Kit v3.1 4 rxns | 10x genomics | PN-1000158 | Single cell gene expression reagent, stored at -20 °C |
| Chromium Next GEM Single Cell 3'GEM Kit v3.1 4 rxns | 10x genomics | PN-1000130 | Single cell gene expression reagent, stored at -20 °C |
| Divided Polystyrene Reservoirs | VWR | 41428-958 | |
| DNA LoBind Tubes 1.5ml Eppendorf | Sigma-Aldrich | EP0030108051 | |
| DNA LoBind Tubes 2ml Eppendorf | Sigma-Aldrich | EP0030108078 | |
| Dry ice | - | - | |
| Dynabeads MyOne SILANE | 10x genomics | PN-2000048 | Single cell gene expression reagent, stored at 4 °C |
| Ethanol Pure | Sigma-Aldrich | E7023 | |
| Glycerin (Glycerol), 50% (v/v) | Ricca Chemical Company | 3290-16 | |
| Heatblock | |||
| High-Throughput Nexcelom Counting Plates | Nexcelom Bioscience | CHM24-A100-001 | Cell counter counting plate |
| Low TE Buffer (10 mM Tris-HCl pH 8.0, 0.1 mM EDTA) | Thermo Fisher Scientific | 12090015 | |
| Mini Centrifuge | - | - | |
| NovaSeq 6000 SP Reagent Kit v1.5 (100 cycles) | Illumina | 2002840 | |
| Nuclei Isolation Buffer | S2 Genomics | 100-063-396 | Stored at 4 °C |
| Nuclei Isolation Cartridge | S2 Genomics | 100-063-287 | Precooled at 4 °C before use |
| Nuclei PURE 2 M Sucrose Cushion Solution | Sigma-Aldrich | NUC201-1KT | Sucrose cushion solution |
| Nuclei PURE Sucrose Cushion Buffer | Sigma-Aldrich | NUC201-1KT | |
| Nuclei Storage Reagent | S2 Genomics | 100-063-405 | Stored at 4 °C |
| PCR Tubes 0.2 ml 8-tube strips | Eppendorf | 30124359 | |
| Percoll | GE Healthcare | 17-0891-02 | Silica colloid solution |
| PhiX Control v3 | Illumina | FC-110-3001 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific | Q32854 | |
| Refrigerated Centrifuge (Eppendorf 5804R) | Eppendorf | 5805000010 | |
| Refrigerated Centrifuge with Swinging-Bucket Rotor (Eppendorf 5810R) | Eppendorf | 5811000015 | |
| RNAseZap | Ambion | AM9780 | RNAse decontamination solution |
| Round cell culture petri dish | SPL | 330005 | |
| Scalpel disposable | Aesculap AG | BA210 | pre-cooled on dry ice before use |
| Single Index Kit T Set A, 96 rxns | 10x genomics | PN-1000213 | Single cell gene expression reagent, stored at -20 °C |
| Singulator 100 System | S2 Genomics | - | Commercially available robotic tissue dissociator |
| Sodium Hydroxide 1M | Sigma-Aldrich | 72068 | |
| SPRIselect Reagent Kit | Beckman Coulter | b23318 | |
| Sterile tweezers | - | - | |
| UltraPure DNase/RNase-Free Distilled Water | Thermo Fisher Scientific | 10977049 | |
| ViaStain PI Staining Solution | Nexcelom Bioscience | CS1-0109-5mL | Propidium iodide staining solution |
| Vortex Mixer+A2:D44 | VWR | - |
References
- Burja, B., et al. An Optimized Tissue Dissociation Protocol for Single-Cell RNA Sequencing Analysis of Fresh and Cultured Human Skin Biopsies. Front Cell Dev Biol. 10, 872688 (2022).
- Kimbley, L. M., et al. Comparison of optimized methodologies for isolating nuclei from esophageal tissue. Biotechniques. 72 (3), 104-109 (2022).
- Maitra, M., et al. Extraction of nuclei from archived postmortem tissues for single-nucleus sequencing applications. Nature Protocols. 16 (6), 2788-2801 (2021).
- Nadelmann, E. R., et al. Isolation of nuclei from mammalian cells and tissues for single-nucleus molecular profiling. Current Protocols. 1 (5), e132 (2021).
- Rousselle, T. V., et al. An optimized protocol for single nuclei isolation from clinical biopsies for RNA-seq. Scientific Reports. 12, 9851 (2022).
- Slyper, M., et al. A single-cell and single-nucleus RNA-Seq toolbox for fresh and frozen human tumors. Nature Medicine. 26 (5), 792-802 (2020).
- Leiz, J., et al. Nuclei isolation from adult mouse kidney for single-nucleus RNA-sequencing. Journal of Visualized Experiments: JoVE. (175), 62901 (2021).
- Alvarez, M., et al. Isolation of nuclei from human snap-frozen liver tissue for single-nucleus RNA sequencing. Bio-Protocol. 13 (3), e4601 (2023).
- Ayhan, F., Douglas, C., Lega, B. C., Konopka, G. Nuclei isolation from surgically resected human hippocampus. STAR Protocols. 2 (4), 100844 (2021).
- Joshi, N., Misharin, A. Single-nucleus isolation from frozen human lung tissue for single-nucleus RNA-seq. Protocols.io. , (2019).
- Martelotto, L. G., Luciano Martelotto, L. 'Frankenstein' protocol for nuclei isolation from fresh and frozen tissue for snRNAseq. Protocols.io. , (2020).
- Masilionis, I., Chaudhary, O., Chaligne, R., Mazutis, L. Nuclei extraction for single-cell RNAseq from frozen tissue using Singulator™ 100. Protocols.io. , (2022).
- Matson, K. J. E., et al. Isolation of adult spinal cord nuclei for massively parallel single-nucleus RNA sequencing. Journal of Visualized Experiments: JoVE. (140), 58413 (2018).
- Mendelev, N., et al. Multi-omics profiling of single nuclei from frozen archived postmortem human pituitary tissue. STAR Protocols. 3 (2), 101446 (2022).
- Soule, T. G., et al. A protocol for single nucleus RNA-seq from frozen skeletal muscle. Life Science Alliance. 6 (5), e202201806 (2023).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648 (2018).
- Ding, J., et al. Systematic comparison of single-cell and single-nucleus RNA-sequencing methods. Nature Biotechnology. 38, 737-746 (2020).
- Hu, P., et al. Single-nucleus transcriptomic survey of cell diversity and functional maturation in postnatal mammalian hearts. Genes & Development. 32 (19-20), 1344-1357 (2018).
- Lake, B. B., et al. A comparative strategy for single-nucleus and single-cell transcriptomes confirms accuracy in predicted cell-type expression from nuclear RNA. Scientific Reports. 7, 6031 (2017).
- Narayanan, A., et al. Nuclei Isolation from Fresh Frozen Brain Tumors for Single-Nucleus RNA-seq and ATAC-seq. Journal of Visualized Experiments: JoVE. (162), 61542 (2020).
- Jovanovich, S., et al. . Automated processing of solid tissues into single cells or nuclei for genomics and cell biology applications with the Singulator™ 100 and 200 systems. , (2022).
- Bell, J., et al. Characterization of a novel high-throughput, high-speed and high-precision plate-based image cytometric cell counting method. Cell & Gene Therapy Insights. 7 (4), 427-447 (2021).
- Madler, S. C., et al. Besca, a single-cell transcriptomics analysis toolkit to accelerate translational research. NAR Genomics and Bioinformatics. 3 (4), lqab102 (2021).
- Wu, H., et al. Mapping the single-cell transcriptomic response of murine diabetic kidney disease to therapies. Cell Metabolism. 34 (7), 1064-1078 (2022).
- Han, L., et al. Cell transcriptomic atlas of the non-human primate Macaca fascicularis. Nature. 604 (7907), 723-731 (2022).
- Madissoon, E., et al. scRNA-seq assessment of the human lung, spleen, and esophagus tissue stability after cold preservation. Genome Biology. 21 (1), 1 (2019).
- Caglayan, E., Liu, Y., Konopka, G. Neuronal ambient RNA contamination causes misinterpreted and masked cell types in brain single-nuclei datasets. Neuron. 110 (24), 4043-4056 (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Molecular Systems Biology. 15 (6), e8746 (2019).
- Fleming, S. J., et al. Unsupervised removal of systematic background noise from droplet-based single-cell experiments using CellBender. bioRxiv. , (2022).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biology. 21 (1), 57 (2020).
- Young, M. D., Behjati, S. SoupX removes ambient RNA contamination from droplet-based single-cell RNA sequencing data. Gigascience. 9 (12), giaa151 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved