Изучение динамики мышечной транскрипции в одномолекулярных масштабах у дрозофилы
In This Article
Summary
Дрозофила является хорошо зарекомендовавшей себя моделью для изучения ключевых молекул, регулирующих миогенез. Однако существующих методов недостаточно для определения транскрипционной динамики мРНК и пространственного распределения в синцитии. Чтобы устранить это ограничение, мы оптимизировали метод флуоресценции РНК in situ , позволяющий обнаруживать и количественно определять мРНК в масштабе одной молекулы.
Abstract
Скелетные мышцы представляют собой большие синцитии, состоящие из множества пучков миоволокон, которые вырабатывают силы и обеспечивают движение тела. Дрозофила является классической моделью для изучения биологии мышц. Сочетание генетики дрозофилы и передовых омиксных подходов привело к идентификации ключевых консервативных молекул, которые регулируют морфогенез и регенерацию мышц. Однако транскрипционная динамика этих молекул и пространственное распределение их матричной РНК в синцитии не могут быть оценены обычными методами. Здесь мы оптимизировали существующий метод флуоресценции одномолекулярной РНК in situ (smFISH), чтобы обеспечить обнаружение и количественное определение отдельных молекул мРНК в летных мышцах взрослых особей и их мышечных стволовых клетках. В качестве доказательства концепции мы проанализировали экспрессию мРНК и распределение двух эволюционно консервативных транскрипционных факторов, Mef2 и Zfh1/Zeb. Мы показываем, что этот метод может эффективно обнаруживать и количественно определять одиночные молекулы мРНК как для транскриптов в мышечных клетках-предшественниках, так и во взрослых мышцах и мышечных стволовых клетках.
Introduction
Скелетные мышцы взрослого человека состоят из дифференцированных многоядерных миоволокон, сократительные свойства которых порождают движения. Мышечный рост, поддержание и регенерация зависят от мышечных предшественников и мышечных стволовых клеток (MuSC), которые определяются во время эмбрионального развития1. Миогенная программа тонко контролируется набором основных миогенных транскрипционных факторов (ТФ) (например, Pax3/7, MYOD, Mef2 и ZEB)2,3,4. Расшифровка молекулярных механизмов, регулирующих мышечную биологию, важна для выяснения фундаментальных вопросов, связанных с миологией, и для возможного терапевтического использования в лечении мышечно-дегенеративных заболеваний.
Дрозофила имеет долгую историю в качестве генетической модели для изучения миогенеза5. Недавно он появился в качестве новой модели для исследования регенерации мышц 6,7,8. Мышечная структура и основные миогенные программы у мух и млекопитающих очень консервативны. Например, ТФ Mef2 и Zfh1/ZEB выполняют консервативную функцию в регуляции развития и регенерации мышц 3,9,10,11. Мышцы взрослой дрозофилы, такие как мышцы непрямого полета (IFM), формируются из специфической популяции MuSC, известных как взрослые мышечные предшественники (AMP)12. Эти АМПБ определяются во время эмбриогенеза и связываются с эпителиальными структурами, такими как диски крыльев и ног. На протяжении эмбриональной и личиночной стадий АМП остаются недифференцированными до метаморфоза, когда они вступают в дифференцировку и слияние с образованием IFM13,14. TF Spalt major (spalt, salm) экспрессируется во время развития летных мышц и необходим для определения их структурной идентичности15. Mef2 является еще одним важным миогенным ТФ, который необходим для формирования мышц у взрослых 16,17. Он экспрессируется в AMP и поддерживается во взрослых IFM10,18. В то время как большинство АМП дифференцируются в функциональные мышцы, часть из них избегает дифференцировки и образует взрослые MuSC11. Как и у позвоночных, TF Zfh1/ZEB необходим для предотвращения преждевременной дифференцировки взрослых MuSC и поддержания их стволовости 9,10.
Доказано, что динамика экспрессии генов регулирует различные мышечные биологические процессы19,20. Появление высокопроизводительных методов секвенирования одноклеточных и одноядерных РНК позволило всесторонне исследовать эту транскрипционную динамику21,22. Одним из заметных ограничений этих подходов является их неспособность обеспечить пространственное распределение молекул мРНК в многоядерных мышечных волокнах. Эти особенности могут быть исследованы с помощью одномолекулярной флуоресцентной гибридизации in situ (smFISH), которая выявляет два типа мРНК-тел: 1. отдельные молекулы мРНК, распространяющиеся в цитоплазме и представляющие зрелую РНК и 2. Зарождающемуся транскрипту соответствуют максимум два ярких ядерных очага, в которых обнаруживаются транскрипционно активные аллели23. Таким образом, smFISH является методом выбора для количественной оценки отдельных молекул мРНК, исследования их пространственного распределения и получения моментальных снимков транскрипционной динамики генов.
Метод smFISH основан на наборе коротких флуоресцентных олигонуклеотидных зондов, специально разработанных для того, чтобы быть комплементарными к целевому транскрипту. При отжиге генерируются точечные источники высокой интенсивности, позволяющие обнаруживать мРНК на уровне одной молекулы с помощью конфокальной микроскопии24. Этот метод все чаще применяется для широкого спектра типов клеток, включая мышечные ткани млекопитающих19,20. Тем не менее, у дрозофилы, как и у других животных моделей, большая часть знаний об экспрессии генов взрослых мышц получена из объемных молекулярных анализов, в которых отсутствует количественная информация о пространственном расположении молекул мРНК. В данной работе мы оптимизировали метод выполнения smFISH на мышечных клетках-предшественниках и мышцах взрослых дрозофил 23,25. Этот протокол включает в себя полностью автоматизированный конвейер анализа для сегментации ядер, подсчета и локализации мРНК.
Protocol
1. Рассечение и препарирование межпозвоночных дисков крыла и мышц взрослого человека
ПРИМЕЧАНИЕ: Очистите диссекционный стенд и все инструменты для диссекции раствором ингибитора РНКазы (Таблица материалов). Лабораторные перчатки следует носить в течение всего эксперимента.
- Диск крыла
- Поместите личинок L3 в холодный 1x фосфатно-солевой буфер (PBS) (Рисунок 1A).
- Промойте личинки 3 раза 1x PBS и подержите их в этом растворе в течение 5 минут.
- С помощью двух пар острых щипцов зажмите личинку за переднюю часть, потяните и выбросьте остальные ткани тела (примерно на 2/3).
- Удерживайте первую 1/3 личинки одной парой щипцов и ротовые крючки другой парой. Втягивайте ротовые крючки внутрь личинки до тех пор, пока она полностью не перевернется.
ПРИМЕЧАНИЕ: Препарируйте примерно 30 личинок за эксперимент, чтобы обеспечить достаточный размер выборки для анализа. - Обратите внимание, что диски крыльев тесно связаны с двумя первичными ветвями трахеи, которые проходят по обе стороны от личинки.
- Удалите все остальные компоненты личинки (диски ног и мозг), чтобы получить чистую перевернутую тушу личинки с парой крыльевых дисков, прикрепленных к трахее (рис. 1B).
- Поместите образцы в центрифужную пробирку объемом 1 мл и зафиксируйте их в 4% параформальдегиде (PFA), разбавленном в 1x PBS, в течение 45 мин при комнатной температуре при перемешивании.
ПРИМЕЧАНИЕ: Перемешивание образца осуществляется с помощью осциллирующей мешалки (Таблица материалов). - Промывают образцы 3 х 5 мин 70% этанолом (EtOH, разбавленным в воде без РНКазы) и хранят их в том же моющем растворе при 4 °C не менее 2 дней и до 1 недели.
- Мышцы непрямого полета
- Обезболивайте взрослых мух на подушке для мух сCO2 и удаляйте головы, брюшко, ноги и крылья.
- Поместите препарированные грудные клетки в холодный 1x PBS и продолжайте делать это до тех пор, пока все образцы не будут препарированы (рис. 1D).
- Префикс грудной клетки 4% PFA, (1% Triton) в течение 20 мин при комнатной температуре при перемешивании.
- Промывайте образцы 3 x 5 мин 1x моющим раствором PBT (1% Triton в 1x PBS).
- Расположите грудные клетки на двустороннем скотче (Таблица материалов) на предметном стекле и разделите их пополам острым лезвием микротома, чтобы получить два полутораса (Рисунок 1E, F).
- Закрепляют образцы в 4% PFA (1% Triton) в течение 45 мин при комнатной температуре при перемешивании.
- Промывайте 2 x 20 мин 1x моющим раствором PBT (1% Triton, 1x PBS) при комнатной температуре при перемешивании.
- Поместите полуторас в холодный 1x PBS и с помощью щипцов аккуратно изолируйте IFM от полутораса (рис. 1G).
ПРИМЕЧАНИЕ: Для этого важно удалить кутикулу как можно тщательнее. - Для пермеабилизации образцов выделенные IFM хранят в 70%-ном растворе EtOH не менее 2 дней и до 1 недели при 4 °C.
ПРИМЕЧАНИЕ: Изолируйте IFM примерно из 10 грудных клеток за эксперимент, чтобы обеспечить достаточный размер выборки для анализа.
2. Гибридизация, иммуноокрашивание и монтаж
ПРИМЕЧАНИЕ: Аналогичная процедура гибридизации применяется как для дисков крыла, так и для образцов IFM. Очень важно изолировать диски крыльев от тушек личинок и поместить их в 200 мкл буфера А до начала гибридизации.
- Ресуспендируйте зонды в 400 мкл TE-буфера (10 мМ Tris-HCl, 1 мМ ЭДТА, pH 8,0) для получения конечного исходного раствора 12,5 мкМ.
- Переложите образцы в центрифужную пробирку объемом 0,5 мл, содержащую буфер А (таблица материалов).
ПРИМЕЧАНИЕ: Уменьшая объем контейнера, становится легче визуализировать образцы и избежать их потери в процессе мойки. - Промывают образцы 2 x 20 мин буфером А.
- Отсасывайте буфер А, заменяйте его 400 мкл буфера для гибридизации (таблица материалов) и предварительно гибридизируйте образцы в течение 30 мин при 37 °C в термосмесителе (таблица материалов).
- Разбавьте зонды до конечной концентрации 125 нМ для дисков крыльев и 200 нМ для взрослых мышц в буфере гибридизации и добавьте первичные антитела.
ПРИМЕЧАНИЕ: Разведение антител варьируется в зависимости от конкретного используемого антитела (Таблица материалов). Добавление антител не является обязательным. Пользователи могут отказаться от белкового окрашивания и вместо этого использовать аналогичный протокол smFISH, который не включает добавление антител. - Аспирируйте гибридизационный буфер и замените его 100 мкл гибридизационного буфера, содержащего зонд и антитело.
- Инкубируют образцы в темноте при температуре 37 °C не менее 16 ч при перемешивании со скоростью вращения 300 об/мин в термомиксере.
- Нагрейте аликвоту буфера А при 37 °C.
- Промывают образцы в течение 3 x 10 мин в термомиксере при 37 °C при перемешивании 300 об/мин.
- Во время последней промывки разбавляют вторичные антитела (1/200) и DAPI (1/10 000) в буфере А и инкубируют образцы в этом растворе при 37 °C не менее 1 ч.
- Промывайте образцы в течение 3 x 20 минут буфером B (таблица материалов).
- Осторожно перенесите образцы на предметное стекло микроскопа, сотрите остаточный буфер B, добавьте 30 мкл монтажной среды и накройте защитным стеклом размером 18 x 18 мм. Используйте лак для ногтей, чтобы закрепить подготовку.
- Храните образцы до 1 недели при температуре 4 °C.
ПРИМЕЧАНИЕ: Тем не менее, рекомендуется выполнить визуализацию как можно скорее, чтобы избежать ухудшения сигнала датчика.
3. Визуализация
- Получение изображений в режиме съемки xyz с помощью конфокального микроскопа, оснащенного 40-кратными и 63-кратными иммерсионными объективами (таблица материалов). Сигнал smFISH регистрируется детектором HyD в режиме подсчета фотонов.
- Найдите образцы с помощью сигнала DAPI и УФ-лампы.
- Для получения изображения мышечных предшественников и IFM, ассоциированных с дисками крыла (рис. 2 и рис. 3), примените следующую настройку для лазерных линий и эмиссионных фильтров: выберите DAPI (возбуждение (например, 405 нм), излучение (эм.) 450 нм), Alexa Fluor 488 (ex. 496 нм, эм. 519 нм) и Quasar 670 для зондов smFISH (например, 647 нм, em. 670 нм).
- Чтобы получить изображение взрослых MuSC (рис. 2D), примените следующую настройку для лазерных линий и эмиссионных фильтров: выберите DAPI (например, 405 нм, em. 450 нм), Alexa Fluor 488 (ex. 496 нм, эм. 519 нм), Alexa Fluor 555 (ex. 555 нм, эм. 565 нм) и Quasar 670 для зондов smFISH (например, 647 нм, em. 670 нм).
ПРИМЕЧАНИЕ: Для визуализации длины волн могут быть адаптированы в соответствии с красителями, связанными со вторичными антителами и зондами. - Сохраните конфокальные изображения как . Файлы TIFF.
4. Анализ после визуализации
- Для начала запустите ImageJ и перейдите в меню Plugins . Затем выберите Макросы | Изменить , чтобы открыть исходный код макросов.
ПРИМЕЧАНИЕ: Это позволит настроить параметры по мере необходимости и, при необходимости, изменить каталог папок для каждого канала в исходном коде макроса. - Чтобы следовать этому протоколу и количественно оценить пятна Mef2 smFISH в личинках, используйте следующие настройки: Log_radius = 3,0 и Log_quality = 20,0. Сегемент Mef2-положительных ядер с размытием = 2; nucleus_scale_parameter = 30; nucleus_threshold = -8,0; nucleus_size = 300.
- В мышечных волокнах используют следующие параметры: Log_radius = 2,5; Log_quality = 60,0; размытие = 2; nucleus_scale_parameter = 100; nucleus_threshold = 0,0; nucleus_size = 300.
- Чтобы инициировать макрос, просто выполните команду run и подождите, пока макрос автоматически загрузит все изображения из указанной папки и подсчитает их последовательно.
ПРИМЕЧАНИЕ: Обратите внимание, что продолжительность этого процесса может варьироваться от нескольких секунд до нескольких часов, в зависимости от объема анализируемых данных. - Найдите результаты, отображаемые в двух новых файлах .csv: file_FISH_results и file_nuclei_results. Первый показывает количество транскрипционных пятен и их среднюю и максимальную интенсивность. Второй дает как общее количество ядер, так и количество пятен внутри каждого ядра.
Representative Results
В этом протоколе мы использовали коммерчески произведенные зонды, нацеленные на мРНК Mef2 и zfh1. Мы разработали датчики с помощью конструктора FISH-зондов производителя (таблица материалов). Набор Mef2 нацелен на экзоны, общие для всех изоформ РНК Mef2 (от экзона 3 до экзона 10). Набор zfh1 нацелен на третий экзон, общий для изоформ 10 zfh1-RB и zfh1-RE. Оба зонда сопряжены с флуоресцентным красителем Quasar 670.
Мы создали собственный макрос, совместимый с программным обеспечением ImageJ для автоматического анализа необработанных .tif данных. Макрос позволяет проводить 3D-сегментацию ядер с помощью Cellpose26 с помощью плагина BIOP для Фиджи и количественную оценку транскрипционного пятна с помощью лапласиана Гаусса, как это реализовано в плагине Trackmate27. Плагин MorpholibJ28 используется для постобработки (Дополнительный файл 1).
В качестве первого шага мы провели smFISH на имагинальных дисках крыла, где, как известно, Mef2 и zfh1 экспрессируются в AMP. Эти данные показывают, что оба транскрипта одинаково обнаруживаются в популяции АМП, окрашенных совместно с антителами Mef2 или Zfh1 (рис. 2A, B). Активные сайты транскрипции (ТС) могут быть выявлены и отличимы от зрелых мРНК с помощью smFISH, поскольку они, как правило, крупнее и имеют более интенсивные сигналы, чем отдельные цитоплазматические транскрипты или зрелые ядерные транскрипты. Последовательно, при более высоких увеличениях АМП различают фокусы TS и зрелые мРНК, рассеянные в цитоплазме (рис. 2A',B'), что подтверждает чувствительность этого протокола smFISH. Следует, однако, отметить, что мы наблюдали по одному активному TS на ядро как для zfh1 , так и для Mef2 (рис. 2A',B'). Эти данные еще раз подтверждают точность конструкции зонда, поскольку транскрипционные паттерны Mef2 и zfh1 в АМП локализуются совместно с обнаружением соответствующих белков (рис. 2).
На втором этапе мы изучили сайт транскрипции и распределение мРНК Mef2 и zfh1 в дифференцированных взрослых IFM и ассоциированных стволовых клетках (рис. 2C, D). Эти данные наглядно иллюстрируют наличие Mef2 TS в ядрах синцитиальных мышц и распределение мРНК Mef2 по всей цитоплазме (рис. 2C,C'). Zfh1 конкретно помечает популяцию MuSCs10,11. Используя этот протокол smFISH, мы успешно обнаружили транскрипцию zfh1 в MuSC, помеченных экспрессией Zfh1-Gal4 > GFP. Большее увеличение иллюстрирует обнаружение как zfh1 TS, так и цитоплазматических одиночных мРНК (рис. 2D').
Наконец, мы использовали макрос ImageJ собственной разработки для количественного анализа транскрипционной динамики и пространственного распределения Mef2. Вычислительный конвейер позволяет эффективно обнаруживать и сегментировать пятна Mef2 и мышечные ядра (рис. 3A-C). Однако из-за проблем с соотношением сигнал/шум некоторые пятна могут быть не обнаружены в некоторых случаях (рис. 3A',B'). Мы использовали этот автоматизированный метод для изучения того, варьируется ли содержание мРНК Mef2 между мышечными клетками-предшественниками (АМП) и дифференцированными взрослыми IFM. Проведенный нами анализ показывает, что количество мРНК Mef2 на ядро у взрослых IFM и АМП существенно не отличается (рис. 3D).
Чтобы получить представление о пространственном распределении транскриптов Mef2 в мышцах IFM у взрослых, мы количественно оценили долю цитоплазматических и ядерных пятен мРНК Mef2 (рис. 3E, F). С помощью нашей программы мы посчитали как общее количество пятен мРНК Mef2 , так и количество пятен мРНК Mef2 , перекрывающихся окрашиванием ядер (Mef2). Вычитание ядра-ассоциированной мРНК из общего числа мРНК дает количество цитоплазматических пятен мРНК. Наши результаты показывают, что примерно 92% мРНК Mef2 присутствует в цитоплазме, в то время как остальные 8% связаны с мышечными ядрами, как показано на рисунке 3E, F.
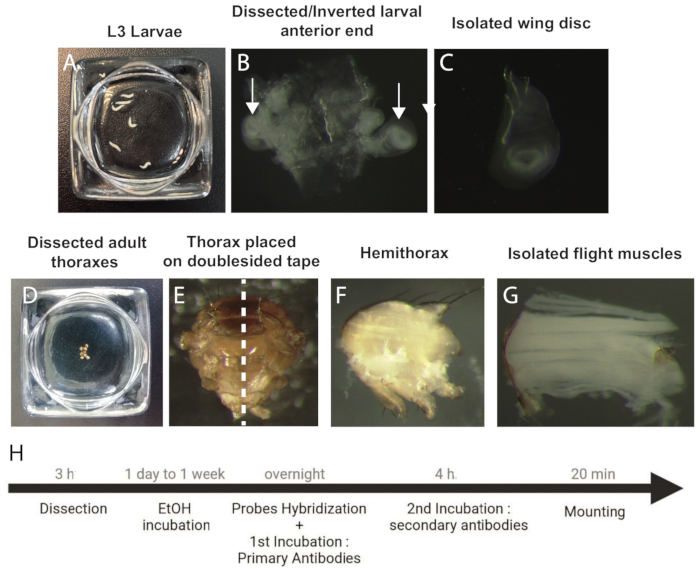
Рисунок 1: Процедура препарирования и подготовки образцов крыльевых дисков и IFM для smFISH. (A) Личинки L3. (Б) Рассеченный и перевернутый передний конец личинки. Стрелками обозначены диски крыла. (C) Изолированный диск крыла. (D) Рассеченная грудная клетка взрослой особи. (E) Грудная клетка взрослого человека, ориентированная на двустороннюю ленту перед рассечением (пунктирная линия). (F) Взрослый гемиторакс. (G) Изолированные IFM. (H) Рабочий процесс протокола smFISH. Сокращения: IFMs = мышцы непрямого полета; smFISH = флуоресцентная одномолекулярная РНК-гибридизация in situ ; EtOH = этанол. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
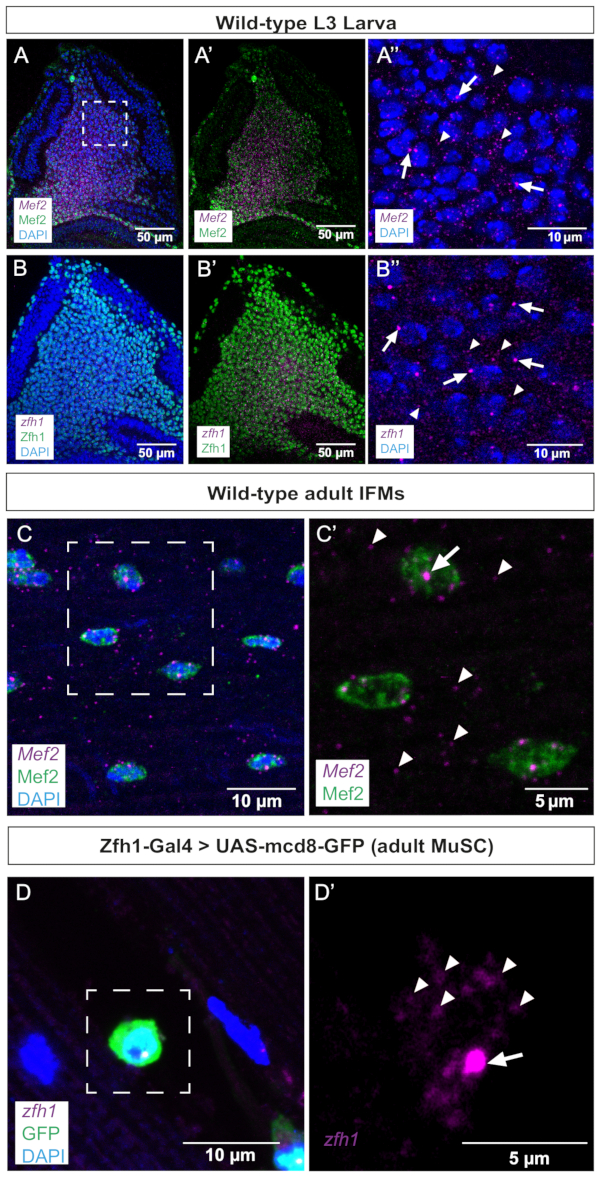
Рисунок 2: Репрезентативные изображения Mef2 и zfh1 smFISH в дисках крыльев и IFM взрослых особей. мРНК (A,B) Mef2 и zfh1 (фиолетовый) обнаруживаются равномерно в AMP и локализуются совместно с белками (A,A') Mef2 и (B,B') Zfh1 (зеленый) соответственно. (А'',Б'') Более высокое увеличение AMP. (C) МРНК Mef2 и белок Mef2 обнаруживаются у взрослых IFM. (с') Более высокое увеличение области в коробке в C. (D) Экспрессия Zfh1-Gal4 >UAS-mCD8GFP (зеленая) помечает взрослые MuSC. ЗФХ1 РНК обнаруживается во взрослых MuSC. (Д') Более высокое увеличение области в коробке в D. Сайты начала транскрипции Zfh1 и одиночные мРНК обозначены стрелками и наконечниками стрелок соответственно. Окрашивание DAPI (синий). Масштабные линейки = 50 мкм (A,B,A',B'), 10 мкм (A",B",C,D), 5 мкм (C',D'). Сокращения: IFMs = мышцы непрямого полета; smFISH = флуоресцентная одномолекулярная РНК-гибридизация in situ ; AMPs = взрослые мышечные предшественники; MuSCs = мышечные стволовые клетки; DAPI = 4',6-диамидино-2-фенилиндол; GFP = зеленый флуоресцентный белок. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
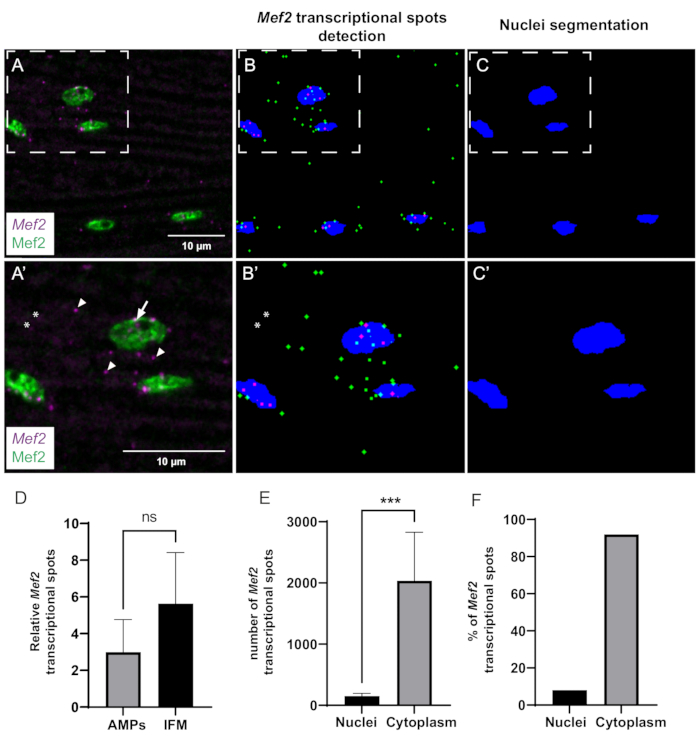
Рисунок 3: Количественная оценка транскрипции Mef2 и цитоплазматического накопления мРНК с помощью smFISH. (A) Распределение транскриптов Mef2 (фиолетовый) и белка Mef2 (зеленый) в взрослых IFM дикого типа. (А') Более высокое увеличение области в коробке в А. Сайты начала транскрипции и одиночные мРНК обозначены стрелками и наконечниками стрелок соответственно. (В,В) Автоматический поиск Mef2-пятен и ядерная сегментация. Цитоплазматическая и ядерная мРНК Mef2 обозначены зеленым и пурпурным цветом соответственно. (B',C') Более высокие увеличения областей в коробках в B и C. Звездочками обозначены места, которые не учитываются макросом. (D) Пример количественного определения пятен Mef2 в АМП и взрослых IFM по отношению к общему числу ядер. (p = 0,0785, t-критерий Стьюдента, диски крыла (n = 4), IFM (n = 11)). (E) Пример количественной оценки распределения пятен Mef2 в IFM у взрослых. (*** p< 0,0007, t-критерий Стьюдента, n = 5). (F) Процент пятен мРНК Mef2, обнаруженных внутри и снаружи ядер (n = 5). Масштабные линейки = 10 мкм (A,A'). Сокращения: IFMs = мышцы непрямого полета; smFISH = флуоресцентная одномолекулярная РНК-гибридизация in situ; АМП = взрослые мышечные предшественники. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
Дополнительный файл 1: Макрос, используемый для постобработки. Пожалуйста, нажмите здесь, чтобы скачать этот файл.
Discussion
Применение метода smFISH приобрело популярность в последнее время, его использование распространяется на различные типы клеток и модельные организмы. Однако в случае с дрозофилой большая часть знаний об экспрессии генов взрослых мышц получена из объемных молекулярных анализов, которые не дают количественной информации о точном пространственном расположении молекул мРНК. Чтобы восполнить этот пробел, мы оптимизировали метод выполнения smFISH на мышечных клетках-предшественниках и мышцах взрослых дрозофил. Этот подход был адаптирован из ранее опубликованного протокола23 и оптимизирован для мышечных тканей дрозофилы .
Основным препятствием для получения высококачественных изображений smFISH является толщина мышц взрослого человека, которая препятствует оптимальному проникновению зондов. Поэтому крайне важно отделить мышцы взрослой особи от других тканей животного и провести процесс гибридизации с легким перемешиванием. Именно этот этап обеспечивает эффективную пермеабилизацию тканей.
Еще один важный аспект заключается в том, что отношение сигнал/шум может привести к сбою вычислительного конвейера в обнаружении определенных пятен мРНК. Мы обнаружили, что увеличение отношения сигнал/шум может быть достигнуто путем нахождения оптимальной концентрации зондов. Оптимальное разведение может отличаться в зависимости от состава каждого набора зондов, включая конъюгированный краситель и олигонуклеотидный состав. Мы рекомендуем попробовать разные разведения; Окончательное разбавление 200 нМ дало наилучшее соотношение сигнал/шум для взрослых тканей в этих экспериментах.
С помощью метода smFISH можно количественно оценить количество вновь синтезированных РНК в очагах ТС. Когда ген активно транскрибируется, в ТС одновременно продуцируются несколько зарождающихся РНК. В результате интенсивность ТС превзойдет интенсивность зрелой цитоплазматической РНК, и эта особенность в сочетании с ее ядерной локализацией может дифференцировать ТС от отдельных цитоплазматических РНК. В контексте биологии мышц обнаружение и количественная оценка TS особенно важны для определения синхронизации транскрипции специфического гена между мышечными ядрами в пределах одного синцития. Однако этот вычислительный конвейер не предназначен для дифференциации цитоплазматических мРНК и TS-сигналов. В качестве альтернативы мы предлагаем комбинировать этот метод smFISH с другими хорошо зарекомендовавшими себя инструментами анализа smFISH, такими как BayFISH или FISH-quant29,30. Было доказано, что эти инструменты облегчают автоматизированную сегментацию и расчет интенсивности флуоресценции агрегатов РНК с замечательной точностью.
Наконец, несмотря на то, что smFISH обнаруживает молекулы мРНК с высоким пространственным разрешением, он ограничен одновременным анализом небольшого количества мРНК. Многомасштабные методы, такие как merFISH (мультиплексная флуоресцентная гибридизация in situ ), позволяют проводить одновременный анализ большого количества различных мРНК31. Комбинируя олигонуклеотидные зонды и коды, исправляющие ошибки, этот метод облегчает обнаружение сотен видов РНК в отдельных клетках.
Disclosures
У авторов нет конфликта интересов, который можно было бы раскрыть.
Acknowledgements
Мы благодарим Алена Винсента за критическое прочтение рукописи. Мы благодарим Рут Леманн (Ruth Lehmann) за предоставление антител к Zfh1. Мы выражаем признательность Стефани Дутертре (Stephanie Dutertre) и Ксавье Пинсону (Xavier Pinson) за помощь в проведении конфокальной микроскопии на платформе МРической визуализации. EL поддерживается докторской стипендией от ministère de l'enseignement supérieur et de la recherche scientifique. АН проводится при поддержке AFM-Telethon Ph.D. fellowship 23846. TP финансируется за счет гранта DAF Chan Zuckerberg Initiative для T.P. (2019-198009). HB поддерживается CNRS, программой Atip Avenir и грантом AFM-Telethon на батуте 23108.
Materials
| Name | Company | Catalog Number | Comments |
| Confocal microscope SP8 | Leica | SP8 DMI 6000 | |
| DAPI | ThermoFisher | 62248 | |
| Double-sided tape | Tesa | 57910-00000 | |
| Dumont #55 Forceps | Fine Science Tools | 11255-20 | |
| Formaldehyde 16% | Euromedex | EM-15710 | |
| Mef2 antibody | DHSB | NA | |
| PBS 10x | Euromedex | ET330 | |
| RNaseZAP | Sigma-Aldrich | R2020 | |
| Stellaris probes | LGC | NA | |
| Stellaris RNA FISH Hybridization Buffer | LGC | SMF-HB1-10 | |
| Stellaris RNA FISH Wash Buffer A | LGC | SMF-WA1-60 | |
| Stellaris RNA FISH Wash Buffer B | LGC | SMF-WB1-20 | |
| Swann-Morton Blades | Fisher scientific | 0210 | |
| ThermoMixer | Eppendorf | 5382000015 | |
| Triton X-100 | Euromedex | 2000 | |
| UAS-mCD8::GFP line | Bloomington | RRID:BDSC_5137 | |
| Ultrapure water | Sigma-Aldrich | 95284 | |
| Watch Glass, Square, 1 5/8 in | Carolina | 742300 | |
| Zfh1 antibody | Gifted by Ruth Leahman | ||
| Zfh1-Gal4 | Bloomington | BDSC: 49924, FLYB: FBtp0059625 |
References
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nature Communications. 12 (1), 692 (2021).
- Chal, J., Pourquie, O. Making muscle: skeletal myogenesis in vivo and in vitro. Development. 144 (12), 2104-2122 (2017).
- Taylor, M. V., Hughes, S. M. Mef2 and the skeletal muscle differentiation program. Seminars in Cell and Developmental Biology. 72, 33-44 (2017).
- Siles, L., et al. ZEB1 imposes a temporary stage-dependent inhibition of muscle gene expression and differentiation via CtBP-mediated transcriptional repression. Molecular and Cellular Biology. 33 (7), 1368-1382 (2013).
- Weitkunat, M., Schnorrer, F. A guide to study Drosophila muscle biology. Methods. 68 (1), 2-14 (2014).
- Boukhatmi, H. Drosophila, an integrative model to study the features of musclestem cells in development and regeneration. Cells. 10 (8), 2112 (2021).
- Bothe, I., Baylies, M. K. Drosophila myogenesis. Current Biology. 26 (17), R786-R791 (2016).
- Catalani, E., et al. RACK1 is evolutionary conserved in satellite stem cell activation and adult skeletal muscle regeneration. Cell Death Discovery. 8 (1), 459 (2022).
- Siles, L., Ninfali, C., Cortes, M., Darling, D. S., Postigo, A. ZEB1 protects skeletal muscle from damage and is required for its regeneration. Nature Communications. 10 (1), 1364 (2019).
- Boukhatmi, H., Bray, S. A population of adult satellite-like cells in Drosophila is maintained through a switch in RNA-isoforms. Elife. 7, e35954 (2018).
- Chaturvedi, D., Reichert, H., Gunage, R. D., VijayRaghavan, K. Identification and functional characterization of muscle satellite cells in Drosophila. Elife. 6, e30107 (2017).
- Gunage, R. D., Reichert, H., VijayRaghavan, K. Identification of a new stem cell population that generates Drosophila flight muscles. Elife. 3, e03126 (2014).
- Figeac, N., Daczewska, M., Marcelle, C., Jagla, K. Muscle stem cells and model systems for their investigation. Developmental Dynamics. 236 (12), 3332-3342 (2007).
- Aradhya, R., Zmojdzian, M., Da Ponte, J. P., Jagla, K. Muscle niche-driven insulin-Notch-Myc cascade reactivates dormant adult muscle precursors in Drosophila. Elife. 4, e08497 (2015).
- Schonbauer, C., et al. Spalt mediates an evolutionarily conserved switch to fibrillar muscle fate in insects. Nature. 479 (7373), 406-409 (2011).
- Bryantsev, A. L., Baker, P. W., Lovato, T. L., Jaramillo, M. S., Cripps, R. M. Differential requirements for Myocyte Enhancer Factor-2 during adult myogenesis in Drosophila. Developmental Biology. 361 (2), 191-207 (2012).
- Soler, C., Han, J., Taylor, M. V. The conserved transcription factor Mef2 has multiple roles in adult Drosophila musculature formation. Development. 139 (7), 1270-1275 (2012).
- Soler, C., Taylor, M. V. The Him gene inhibits the development of Drosophila flight muscles during metamorphosis. Mechanisms of Development. 126 (7), 595-603 (2009).
- Pinheiro, H., et al. mRNA distribution in skeletal muscle is associated with mRNA size. Journal of Cell Science. 134 (14), jcs256388 (2021).
- Denes, L. T., Kelley, C. P., Wang, E. T. Microtubule-based transport is essential to distribute RNA and nascent protein in skeletal muscle. Nature Communications. 12 (1), 6079 (2021).
- Li, H., et al. Fly Cell Atlas: A single-nucleus transcriptomic atlas of the adult fruit fly. Science. 375 (6584), eabk2432 (2022).
- Santos, M. D., et al. Extraction and sequencing of single nuclei from murine skeletal muscles. STAR Protocols. 2 (3), 100694 (2021).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877-879 (2008).
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of single RNA transcripts in situ. Science. 280 (5363), 585-590 (1998).
- Raj, A., Tyagi, S. Detection of individual endogenous RNA transcripts in situ using multiple singly labeled probes. Methods in Enzymology. 472, 365-386 (2010).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: a generalist algorithm for cellular segmentation. Nature Methods. 18 (1), 100-106 (2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Legland, D., Arganda-Carreras, I., Andrey, P. MorphoLibJ: integrated library and plugins for mathematical morphology with ImageJ. Bioinformatics. 32 (22), 3532-3534 (2016).
- Mueller, F., et al. FISH-quant: automatic counting of transcripts in 3D FISH images. Nature Methods. 10 (4), 277-278 (2013).
- Gomez-Schiavon, M., Chen, L. F., West, A. E., Buchler, N. E. BayFish: Bayesian inference of transcription dynamics from population snapshots of single-molecule RNA FISH in single cells. Genome Biology. 18 (1), 164 (2017).
- Chen, K. H., Boettiger, A. N., Moffitt, J. R., Wang, S., Zhuang, X. RNA imaging. Spatially resolved, highly multiplexed RNA profiling in single cells. Science. 348 (6233), aaa6090 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved