Preparação de Núcleos Cerebrais e de Medula Óssea de Alta Qualidade para Ensaios Multioma de Núcleos Únicos
In This Article
Summary
O sucesso da transcriptômica de célula única/núcleo único e multi-ômica depende em grande parte da qualidade das células/núcleos. Portanto, o isolamento de células/núcleos do tecido e sua purificação devem ser altamente padronizados. Este protocolo descreve a preparação de núcleos do cérebro e da medula óssea para o ensaio de multioma de núcleo único a jusante.
Abstract
A análise de célula única tornou-se a abordagem de escolha para desvendar a complexidade dos processos biológicos que requerem a avaliação da variabilidade das respostas celulares individuais ao tratamento ou infecção com resolução de célula única.
Muitas técnicas para perfil molecular de célula única foram desenvolvidas nos últimos 10 anos, e várias tecnologias dedicadas foram comercializadas. O perfil de célula única baseado em gotículas da 10X Genomics é uma tecnologia amplamente difundida que oferece reagentes prontos para uso para perfis de célula única transcriptômicos e multi-ômicos. A tecnologia inclui fluxos de trabalho para sequenciamento de RNA de célula única e núcleo único (scRNA-Seq e snRNA-Seq, respectivamente), scATAC-Seq, perfil imunológico de célula única (sequenciamento BCR/TCR) e multioma. Este último combina informações transcricionais (scRNA-Seq) e epigenéticas (scATAC-Seq) provenientes da mesma célula.
A qualidade (viabilidade, integridade, pureza) de suspensões unicelulares ou de núcleo único isoladas de tecidos e analisadas por qualquer uma dessas abordagens é crítica para a geração de dados de alta qualidade. Portanto, os protocolos de preparo das amostras devem ser adaptados às particularidades de cada tecido biológico e garantir a geração de suspensões celulares e nucleares de alta qualidade.
Este artigo descreve dois protocolos para preparar amostras de cérebro e medula óssea para o pipeline 10X Genomics do multioma a jusante. Os protocolos são realizados passo a passo e abrangem dissociação tecidual, triagem celular, isolamento de núcleos e controle de qualidade da suspensão de núcleos preparada que é usada como material de partida para particionamento celular e código de barras, preparação de biblioteca e sequenciamento. Esses protocolos padronizados produzem bibliotecas de núcleos de alta qualidade e dados robustos e confiáveis.
Introduction
Por muitos anos, as técnicas unicelulares têm sido o padrão-ouro para a análise de processos biológicos. Inicialmente, restringiram-se à fenotipagem unicelular por meio de microscopia, citometria de fluxo e ensaios similares. Um avanço na análise de célula única veio com o desenvolvimento de abordagens para perfil molecular de célula única, em particular o sequenciamento de RNA de célula única (scRNA-Seq) que permitiu caracterizar todo o transcriptoma de células individuais. Altamente poderoso, scRNA-Seq gera informações sobre o status transcricional de uma célula em uma condição e ponto de tempo específicos. No entanto, não fornece visibilidade sobre a regulação gênica que impulsiona a transcrição, ou sobre as modificações moleculares que ocorrem ao longo do tempo. Para superar essa limitação, muitos esforços têm sido investidos no desenvolvimento de ensaios multiômicos unicelulares que possibilitem a análise de múltiplos fatores e processos de uma mesma célula1,2,3,4. A primeira medida bem-sucedida de duas modalidades dentro de células individuais veio através da ligação de padrões de expressão de proteínas de superfície multiplex com o transcriptoma completo de células individuais na abordagem CITE-Seq5. Evoluções mais recentes combinam a expressão gênica com a acessibilidade da cromatina (Assay for Transposase-Accessible Chromatin using sequencing, ATAC-Seq), capturando simultaneamente modalidades transcriptômicas e epigenômicas nas mesmas células (por exemplo, sci-CAR)6. As primeiras soluções comerciais que permitiram associar a transcriptômica ao fenótipo celular ou a alterações epigenéticas de uma mesma célula vieram da 10X Genomics.
Os experimentos para perfil molecular de célula única contêm as seguintes etapas: (1) dissociação tecidual ou preparação de suspensões de célula única; (2) purificação e/ou isolamento de núcleos celulares; (3) particionamento e código de barras; (4) construção de bibliotecas e controle de qualidade; (5) sequenciamento de próxima geração; (6) análise dos dados. Enquanto os passos (3)-(6) podem variar significativamente dependendo da tecnologia empregada, os passos iniciais são geralmente comuns a todos eles. A qualidade da suspensão de células/núcleos preparada determinará o resultado global do experimento. Dependendo do tipo de tecido, a obtenção de suspensões unicelulares/núcleos de alta qualidade pode ser um desafio. As particularidades de alguns tecidos, como coração, músculo, cérebro, pulmão, intestino, entre outros, requerem métodos de ruptura tecidual e isolamento de núcleos adaptados a cada tipo de amostra, a fim de garantir a produção de núcleos de alta qualidade para análise molecular7,8,9,10. Os métodos de ruptura tecidual e os protocolos de dissociação podem ser mecânicos, enzimáticos (por exemplo, uma mistura de colagenases e DNase) ou uma combinação dos dois, e podem ser realizados manualmente ou por instrumentos (por exemplo, Qiagen DSC-400, gentleMACS).
As técnicas unicelulares tornaram-se uma ferramenta de escolha para a pesquisa biomédica. Em neurobiologia, a diversidade celular no cérebro e a complexidade de suas funções requerem análises de alta resolução e alto rendimento para visualização de populações celulares raras e para avaliação de sua heterogeneidade 11,12,13,14. Ligar a identidade celular e os mecanismos de regulação gênica de células individuais fornece insights sobre o desenvolvimento e a fisiologia do cérebro. Outro exemplo são os estudos de resposta imune no contexto de doenças infecciosas, autoimunes ou oncológicas, que dependem fortemente de análises unicelulares. A heterogeneidade dos subgrupos de células imunes e a complexidade de sua atividade e interações com outros tipos celulares requerem resolução de célula única para decifrar os mecanismos subjacentes à resposta imune. As células imunes se originam da medula óssea, onde os progenitores hematopoiéticos são compostos por células que gradualmente se diferenciam e adquirem e perdem marcadores de superfície celular ao longo de um processo gradual antes de sair da medula óssea para a periferia. A análise unicelular permite a caracterização minuciosa dos estágios de desenvolvimento celular. Pode ser realizada através da fenotipagem unicelular, convencionalmente realizada por citometria de fluxo multiparâmetro. No entanto, foi demonstrado que assinaturas transcriptômicas de célula única revelam uma identificação mais precisa dos subtipos de células progenitoras, uma vez que essas células estão distribuídas em agrupamentos que caem umas nas outras e, portanto, podem ser erroneamente identificadas quando se usa uma abordagem de marcador de superfície celular grossa15. Um número crescente de estudos revela as modificações epigenéticas que as células-tronco hematopoéticas e as células progenitoras (HSPCs) podem adquirir a partir da exposição a vários agentes, levando a um impacto significativo na responsividade do sistema imune em longo prazo 16,17,18,19. As novas tecnologias multi-ômicas permitem estudar esses processos com resolução de célula única.
Vários protocolos de isolamento celular e nuclear têm sido descritos para amostras de cérebro11,20,21,22 e medulaóssea23,24. Para minimizar o viés devido à variabilidade experimental, é necessário validar protocolos otimizados de preparação de núcleos únicos para sequenciamento epigenômico e transcriptômico de célula única, garantindo assim a reprodutibilidade de ensaios multiômicos de célula única.
Aqui, dois protocolos robustos para a preparação de núcleos a partir de (1) tecido cerebral fresco congelado e (2) HSPCs de medula óssea fresca para análise de multioma de célula única a jusante são descritos (Figura 1).
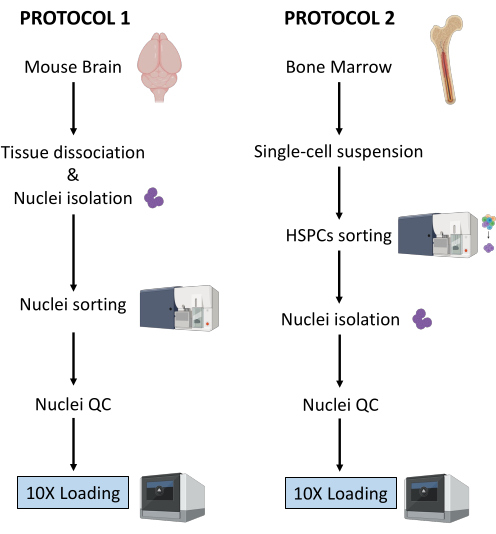
Figura 1: Representação esquemática dos protocolos de isolamento de núcleos de tecidos cerebrais e medulares frescos congelados. Clique aqui para ver uma versão maior desta figura.
Protocol
Os procedimentos experimentais foram conduzidos sob a estrita conformidade regulatória de protocolos aprovados pelo Comitê de Ética em Experimentação Animal (CETEA). Para o isolamento dos núcleos cerebrais, foram utilizados camundongos C57BL/6 com 3 meses de idade. Para o isolamento da medula óssea, foram utilizados camundongos fêmeas C57BL/6J, com 8 semanas de idade, pesando 18 g.
1. Purificação de núcleos do cérebro de camundongos
OBS: Usar luvas de látex ou nitrílica em todos os momentos durante o procedimento. É altamente recomendável ter duas pessoas realizando o experimento, para que as etapas 1 a 3 (ou seja, preparação da suspensão de núcleo único) sejam realizadas por uma pessoa, e a etapa 4 (ou seja, preparação do classificador) seja executada em paralelo por outra pessoa. Como o protocolo é altamente sensível ao tempo, é fundamental minimizar o tempo de processamento da amostra tendo o classificador pronto assim que a suspensão de núcleo único é preparada.
- Preparação de reagentes e materiais
- Esterilizar cuidadosamente as ferramentas de dissecção em autoclave (a 121 °C por 20 min) e lavá-las com etanol 70% imediatamente antes do uso. Preparar uma placa de Petri por amostra, preenchida com 2-3 mL de solução salina tamponada com fosfato (DPBS) gelada 1x Dulbecco.
- Resfriar a microcentrífuga a 4 °C, encher um balde com gelo e colocar o homogeneizador de vidro no gelo.
- Preparar tampão de lise nuclear adicionando digitonina para uma concentração final de 0,01%, 10 mL por amostra.
- Preparar o tampão de coloração adicionando inibidores de RNase ao tampão de coloração celular para uma concentração final de 0,2 U/μL, 20 mL por amostra.
- Preparar DPBS 0,04% BSA adicionando inibidores de RNase para uma concentração final de 0,2 U/μL, 2 mL por amostra.
- Preparar 1 mL de tampão de núcleo diluído de acordo com o protocolo Multiome25.
- Mantenha todos os reagentes e amostras no gelo.
- Dissecção tecidual
- Sacrificar camundongos utilizando protocolos aprovados pela instituição. Neste protocolo, os camundongos foram decapitados após uma overdose de cetamina/xilazina.
- Cortar a cabeça do camundongo com tesoura e remover o cérebro do crânio como descrito em Meyerhoff et al.26. Transfira imediatamente o cérebro para uma placa de Petri preparada com o 1x DPBS gelado sob um microscópio estereomicroscópio iluminado por diodo emissor de luz (LED).
- Corte o tecido cerebral com um bisturi para separar as áreas cerebrais de interesse (por exemplo, córtex entorrinal, hipocampo, córtex pré-frontal) e transfira cada região para uma placa de Petri separada contendo 1x DPBS gelados. Continue no gelo.
- Com um bisturi, pique o tecido em pedaços de <0,5 cm para facilitar a homogeneização na etapa seguinte.
- Com uma micropipeta P1000, transfira o tecido picado e o 1x DPBS da placa de Petri para um tubo de 1,5 mL. Certifique-se de usar tubos feitos de plástico de baixa ligação de proteínas. Permita que os pedaços de tecido se separem por gravidade. Remova cuidadosamente o excesso de 1x DPBS usando uma micropipeta P1000.
NOTA: Após esta etapa, é possível congelar rapidamente o tecido picado, transferindo os tubos de baixa ligação de proteínas para o gelo seco e, em seguida, armazenar a -80 °C até prosseguir com o isolamento dos núcleos.
- Isolamento de núcleos
- Encher a dounce de vidro com 2 mL de tampão de lise de núcleos gelados com digionina a 0,01%. Adicione os pedaços de tecido na dounce.
NOTA: se estiver trabalhando com tecido fresco congelado, adicione o tecido congelado picado diretamente ao tampão de lise nuclear 0,01% digitonina; Não deixe o tecido descongelar antes. - Homogeneizar com homogeneizador de tecido de dounce de vidro 25 vezes com pilão A e depois 25 vezes com pilão B. Transfira o homogeneizado para um tubo de 15 mL.
- Adicionar mais 2 mL de tampão de lise de núcleos gelados com digionina a 0,01% e incubar no gelo por 5 min. Centrífuga de núcleos a 500 x g durante 5 min a 4 °C.
- Remover o sobrenadante com uma micropipeta e adicionar 4 mL de tampão de lise de núcleos gelados com digionina a 0,01%. Incubar no gelo durante 5 min e filtrar através de um filtro celular de 40 μm.
- Centrifugar os núcleos a 500 x g durante 5 min a 4 °C e remover o sobrenadante com uma micropipeta.
- Adicionar 4 ml de tampão de coloração para lavar os núcleos e centrifugar a 500 x g durante 5 minutos a 4 °C. Retire o sobrenadante com uma micropipeta e ressuspenda o pellet em 4 mL de tampão de coloração.
- Filtrar através de um filtro celular de 40 μm e centrifugar a 500 x g durante 5 min a 4 °C. Ressuspender em 1 mL de PBS com BSA 0,04%.
- Conte núcleos para garantir a consistência da preparação de tecidos/núcleos em diferentes amostras. Espera-se obter contagens de núcleos semelhantes das mesmas regiões cerebrais:
- Adicionar 10 μL de azul de tripano a 0,4% a um tubo vazio de 0,5 ml. Adicionar 10 μL dos núcleos e misturar 5x por pipetagem.
- Conte núcleos usando um contador de células automatizado seguindo as recomendações do fornecedor. Mantenha os núcleos no gelo.
- Preparar núcleos para triagem.
NOTA: Os núcleos extraídos incorporam 7-AAD, e esta coloração é usada para sua purificação por classificador de células ativado por fluorescência (FACS).- Transferir 100 μL de núcleos para um tubo FACS para o controle sem coragem. Adicionar 10 μL de 7-AAD aos núcleos restantes e manter 5 min a 4 °C.
- Classifique um mínimo de 0,5 x 106 núcleos por FACS para eliminar duplos e detritos.
- Encher a dounce de vidro com 2 mL de tampão de lise de núcleos gelados com digionina a 0,01%. Adicione os pedaços de tecido na dounce.
- Classificação de núcleos usando um FACS
NOTA: Embora a classificação de núcleos possa ser realizada em uma ampla variedade de classificadores de células, o procedimento para usar os instrumentos BD FACSAria Fusion ou BD FACSAria III é descrito aqui. É altamente recomendável que a calibração e a configuração do classificador de células sejam realizadas sob a supervisão, ou por um usuário experiente do instrumento. Para reduzir o tempo de processamento da amostra, é fundamental ter o classificador pronto assim que a suspensão de núcleo único for preparada.- Calibração do instrumento FACS
- Ligue o classificador de células e o computador. Uma vez que o software está conectado ao instrumento, inicie o procedimento de inicialização fluídica. Selecione Citômetro > inicialização fluídica no menu principal e siga as quatro etapas. Clique em Concluído após concluir cada uma delas.
- Insira o bocal de 70 μm, ligue o fluxo e deixe o fluxo estabilizar por 15 min. Ajuste a amplitude para obter a formação de gotas e clique em Sweet Spot.
- Coloque o filtro de densidade neutra (N.D) 1.0 e abra a interface de configuração e rastreamento do citômetro (CST).
- Controle de qualidade diário: Diluir contas CST em meio FACS (consulte as recomendações do fornecedor) e realizar o controle CST. Depois de concluído, substitua o N.D 1.0 pelo N.D 2.0.
- Dilua Accudrops em meio FACS (consulte as recomendações do fornecedor) e execute o atraso de queda conforme descrito nas etapas 6 a 10.
- No modelo de experimento, selecione o experimento Accudrop Drop Delay e abra o Layout de Classificação do tubo.
- Dentro da janela inferior da câmera, clique em Tensão e, em seguida, em Filtro Óptico para habilitar a aplicação de carga nas placas de deflexão e usar um filtro óptico específico na frente da câmera. Certifique-se de que o quadrante do lado direito indique 100. Se necessário, ajuste o parafuso laser vermelho para otimizar o impacto do laser.
- Ajuste a taxa de fluxo para atingir a velocidade de 1.000 a 3.000 eventos por segundo.
- Clique em Classificar e Cancelar. Certifique-se de que o quadrante esquerdo é igual a 100 e o quadrante direito é 0. Se o quadrante esquerdo estiver abaixo de 95, execute o Atraso Automático.
- Clique em Tensão e, em seguida, em Testar Classificação. Controlar a qualidade dos fluxos laterais depositados nos tubos de coleta. Se necessário, ajuste a posição dos fluxos laterais movendo os controles deslizantes.
- Configuração do instrumento FACS para triagem de núcleos.
- Iniciar a aquisição dos núcleos não manchados. Estes são usados para definir os espalhamentos para frente e lateral, e a tensão do detector para o parâmetro 7-ADD. Defina os parâmetros para que o sinal 7-AAD da amostra não corada caia dentro da primeira década da escala logarítmica no gráfico de pontos.
- Comece a adquirir o tubo de núcleos corados com 7-AAD e defina as populações de núcleos usando uma estratégia de agrupamento baseada em (1) FSC-A/SCC-A e, em seguida, FSC-H/SSC-H para tamanho e granularidade, (2) FSC-H/FSC-A para discriminação de duplos e (3) SSC-A/7-AAD para núcleos positivos para 7-AAD (ver Figura 2A).
- Certifique-se de que o fluxo e a deflexão estejam estáveis.
- Na câmera de fluxo lateral, ligue a classificação de teste, tensão ON e confirme a classificação precisa de gotas em um tubo de 1,5 mL instalado no lado esquerdo.
- Na janela Layout de Classificação , selecione a população de interesse, conforme definido na etapa 2 (acima). Em Eventos de Destino, selecione o limite em Contínuo para obter um mínimo de 0,5 x 106 núcleos por amostra. Em Precisão, selecione Pureza de 4 vias.
- Depois de pronto, clique em Classificar e OK para começar com a classificação de núcleos.
- Calibração do instrumento FACS
- Controle de qualidade e contagem de núcleos purificados
NOTA: Esta etapa deve ser feita apenas durante o experimento piloto para otimização das etapas de preparação da amostra, com o objetivo de testar a pureza dos núcleos classificados que serão carregados no chip de cromo 10X. Uma vez que o protocolo esteja totalmente otimizado, não é aconselhável realizar esta etapa de controle de qualidade nos experimentos de acompanhamento para evitar desperdício desnecessário de núcleos coletados que podem estar disponíveis em baixo número.- Controle de pureza por citometria de fluxo
- Transferir 10 μL dos núcleos triados para um novo tubo FACS contendo 90 μL de DPBS com 2% de soro fetal bovino inativado pelo calor (HI-FBS).
- Adquira e registre dados pós-classificação para verificar a pureza e viabilidade da classificação. Garantir que pelo menos 98% dos núcleos apareçam na porta de interesse, conforme definido no item 4.2 (ver Figura 2B).
- Contagem dos núcleos purificados
- Centrifugar os núcleos separados por 5 min a 500 x g e a 4 °C e remover cuidadosamente completamente o sobrenadante usando uma micropipeta. Ressuspender em 100 μL de tampão de núcleo diluído.
- Adicionar 10 μL de azul de tripano a 0,4% a um tubo vazio de 0,5 ml. Adicionar 10 μL dos núcleos triados e misturar 5x por pipetagem.
- Conte núcleos usando um contador de células automatizado seguindo as recomendações do fornecedor. Ajustar a concentração de núcleos para 3,5 x 106/mL, ou seja, 16.000 núcleos por 5 μL.
- Controle de qualidade de núcleos purificados por microscopia
NOTA: Esta etapa deve ser feita apenas durante o experimento piloto para otimização das etapas de preparação da amostra para testar a qualidade dos núcleos que serão carregados no chip de cromo 10X. Uma vez que o protocolo esteja totalmente otimizado, não é aconselhável realizar esta etapa de controle de qualidade nos experimentos de acompanhamento para evitar desperdício desnecessário de núcleos coletados que podem estar disponíveis em baixo número.- Certifique-se de que as lâminas e as tampas do microscópio estejam limpas e livres de poeira. Se necessário, lave e enxágue as lamínulas com etanol absoluto e seque-as com lenços sem fiapos.
- Distribuir 25 μL de poli-l-lisina nos poços deslizantes que serão utilizados e incubar por 10 min à temperatura ambiente (TR), protegidos da poeira.
- Retire o excesso de poli-L-lisina e adicione 10 μL da suspensão de núcleos purificados. Incubar por 5 min em TR, protegido da poeira.
- Adicione uma gota de meio de montagem a cada poço, evitando bolhas.
- Coloque uma tampa em cima dos poços semeados. Cubra com lenços de papel e pressione a tampa firmemente para remover o excesso de suporte. Tenha cuidado para não mover a tampa e não limpe o meio de montagem em excesso.
- Tire várias imagens com um microscópio invertido com luz de campo brilhante e uma ampliação mínima de 40x.
- Controle de pureza por citometria de fluxo
- Realizar ensaio multiome.
- Prossiga imediatamente para o Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide (CG000338 - Rev F)25.
2. Purificação de núcleos de células-tronco hematopoéticas e progenitoras de medula óssea de camundongos (HSPCs)
NOTA Este protocolo descreve a purificação de núcleos de três subgrupos de HSPCs da medula óssea: linhagem-c-Kit+Sca-1+ células-tronco hematopoéticas (HSC), linhagem-c-Kit+Sca-1-CD34+FcγR- progenitores mieloides comuns (CMP) e linhagem- c-Kit+Sca-1-CD34+FcγR+ progenitores de granulócitos e monócitos (GMP). Use luvas de látex ou nitrilo em todos os momentos durante o procedimento. Este protocolo é uma adaptação do 10X Genomics Demonstrated Protocol - Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 - Rev C)27. Modificações foram introduzidas no protocolo original para maximizar a recuperação dos núcleos. É altamente recomendável ter duas pessoas realizando o experimento, para ter os passos 1. até 3. (i.e., preparação da solução unicelular) realizada por uma pessoa, e a etapa 4 (i.e., preparação do classificador) realizada em paralelo por outra pessoa. Como o protocolo é altamente sensível ao tempo, é fundamental minimizar o tempo de processamento da amostra tendo o classificador pronto assim que a suspensão de célula única é preparada.
- Preparação de reagentes e materiais
- Encha um balde com gelo.
- Preparar o tampão FACS: DPBS com solução HI-FBS a 2% (aproximadamente 500 mL para 6 amostras) e filtrar através de um filtro de 0,2 μm.
- Preparar o meio de coleta: DPBS com solução de HI-FBS a 10% (500 μL por amostra) e filtrar através de um filtro de 0,2 μm.
- Isolamento de células da medula óssea
- Sacrificar camundongos utilizando protocolos aprovados pela instituição. Neste experimento, os camundongos foram sacrificados por deslocamento cervical após uma overdose de cetamina/xilazina.
- Borrife o abdômen e as patas traseiras dos camundongos com etanol 70%.
- Use pinça e tesoura estéreis para fazer uma pequena incisão no meio do abdome inferior e abrir o peritônio da base das patas traseiras até o diafragma (Figura 1 suplementar).
- Faça um corte adicional para cada perna traseira perpendicular ao peritônio aberto, em seguida, pegue cada lado de um desses cortes adicionais e separe-o para descascar a pele de ambas as patas traseiras além da articulação do tornozelo para expor os músculos de ambas as patas traseiras (Figura 1A Suplementar).
- Forre a tesoura ao longo da coluna vertebral na articulação do quadril de uma perna traseira para cortar a perna sem cortar o fêmur (Figura suplementar 1B, C). Repita o mesmo para a outra perna.
- Para isolar o fêmur, corte a maior parte do tecido muscular para fora e, em seguida, segure o fêmur e a tíbia em cada mão com as pontas dos dedos na articulação (Figura 1D Suplementar, E). Dobre suavemente a perna contra a curvatura natural para deslocar a tíbia do fêmur (Figura 1E Suplementar) e, em seguida, corte cuidadosamente o tecido conjuntivo com tesoura para separar o fêmur e a tíbia.
- Use a tesoura com movimentos leves de torção para deslocar o pedaço de espinha dorsal da extremidade superior do fêmur (Figura 1E suplementar).
- Limpe o fêmur isolado com papel higiênico para remover o restante do músculo e tecido conjuntivo.
- Manter gelado no gelo em uma placa de 12 poços bem preenchida com 2 mL de DMEM (1x) + GlutaMAX-I.
- Uma vez que todos os fêmures são coletados, certifique-se de que os tecidos musculares e fibrosos são completamente removidos do osso. Não corte o osso aberto para (a) manter a medula dentro estéril e (b) evitar a perda de células no poço. Use os seguintes passos para liberar as células de dois fêmures de um camundongo, adaptado de Haag e Murthy28.
- Prepare um tubo de 1,5 mL e outro de 0,5 mL. Adicione 150 μL do tampão FACS ao tubo de 1,5 mL, faça um orifício no fundo do tubo de 0,5 mL usando uma agulha de 18 G e encaixe o tubo de 0,5 mL no tubo de 1,5 mL.
- Abra a parte distal de cada fêmur com tesoura cirúrgica de camundongo (Figura 1F Suplementar): Trave a epífise distal entre as lâminas e aplique uma pressão suave enquanto vira a tesoura para desprender suavemente a epífise distal sem abrir o osso bruscamente. Se bem sucedida, 4 saliências devem ser visíveis na extremidade da fise agora exposta (Figura 1G suplementar).
- Encaixar os dois fêmures com a extremidade aberta voltada para baixo no tubo preparado de 0,5 mL colocado dentro de um tubo de 1,5 mL contendo tampão FACS (Figura suplementar 1H).
- Colocar um filtro celular de 70 μm em um tubo de 50 mL e pré-molhar o filtro com 2 mL do tampão FACS.
- Para lavar a medula óssea, centrifugar os tubos (tampas abertas) a 12.000 x g até que a centrífuga atinja o valor de 12.000 x g e, em seguida, pare imediatamente a centrífuga.
- Verifique se as células da medula óssea estão peletizadas no tubo de 1,5 mL e se os fêmures estão brancos (antes da lavagem celular, estão vermelhos) (Figura 1I suplementar). Descarte os tubos de 0,5 mL com os 2 fêmures.
- Descarte o sobrenadante de 150 μL usando uma pipeta.
- Ressuspender o pellet com uma micropipeta em 1 mL de tampão de lise Amônio-Cloreto de Potássio (ACK) por 1-2 minutos em RT para lisar as hemácias. Evite tempos de incubação mais longos, pois eles podem resultar em menor viabilidade das células nucleadas.
- Transferir para o tubo de 50 mL através do filtro de células pré-molhado de 70 μm.
- Adicionar 10 ml de tampão FACS para diluir o tampão de lise ACK e, por conseguinte, parar a lise.
- Centrifugar a 400 x g durante 5 min a 4 °C. Ressuspender em tampão FACS de 10 mL, primeiro ressuspendendo em 1 mL e, em seguida, completando com 9 mL.
- Preparar as células para contagem conforme descrito no ponto 1.3.8.
- Conte as células usando um contador de células automatizado seguindo as recomendações do fornecedor. Espera-se coletar aproximadamente 40 milhões de células de 2 fêmures.
- Coloração da HSPC da medula óssea
- Centrifugar as células a 400 x g durante 5 min a 4 °C e ressuspender o pellet com uma micropipeta em tampão FACS até uma concentração final de 1 x 107 células/ml.
- Com uma micropipeta P1000, transfira a suspensão para um tubo FACS, filtrando através de uma tampa de filtro de células de 35 μm.
- Preparar amostras de tubo de ensaio de coloração única para cada anticorpo listado na Tabela 1 para estabelecer compensações de fluorocromos no classificador celular:
- Preparar um tubo FACS por anticorpo e encher os tubos com 200 μL de PBS.
- Adicionar 15 μL de esferas de compensação de fluorocromo em cada tubo FACS de anticorpo conjugado com fluorocromo. Nos tubos FACS para células não coradas e para células vivas/mortas de coloração única, adicione 500.000 células em vez de contas.
- Adicionar 1 μL de cada anticorpo conjugado ao fluorocromo (ver Tabela 1) no tubo FACS correspondente. Adicione 0,5 μL de mancha Viva/Morto no tubo FACS de mancha única Viva/Morta.
- Manter no gelo por 15 minutos protegido da luz.
- Preparar as misturas 1 e 2 conforme indicado na Tabela 2.
NOTA: Os volumes de anticorpos indicados na Tabela 2 são válidos para os anticorpos referenciados na Tabela de Materiais. Eles precisam ser otimizados para qualquer nova referência de anticorpo ou um lote diferente da mesma referência de anticorpo. - Adicionar 300 μL de Mix 1 no tubo de amostra, ressuspender e manter por 15 min no gelo protegido da luz.
- Adicionar 300 μL de Mix 2 no tubo de amostra, ressuspender e manter durante 20 min no gelo protegido da luz.
- Adicionar 3 ml do tampão FACS aos tubos corados com um único e aos tubos de amostra corados com mistura. Gire para baixo a 400 x g por 5 min a 4°C.
- Descarte cuidadosamente o sobrenadante usando uma micropipeta e ressuspenda o pellet em 500 μL do tampão FACS.
- Preparar um tubo de 1,5 ml pré-preenchido com 500 μL de meio de recolha.
NOTA: A mistura 1 é preparada em DPBS, uma vez que contém a mancha Viva/Morto significativamente afetada pelo HI-FBS. Uma vez que as células são coradas por Live/Dead, é adicionado o Mix 2, que contém os anticorpos conjugados ao fluorocromo ressuspensos em tampão FACS contendo HI-FBS. A única exceção é o anticorpo anti-CD16/32 que está incluído no Antibody Mix 1 para servir como bloqueador do receptor Fc que impede a ligação inespecífica dos outros anticorpos adicionados na etapa seguinte.
- Classificação de células usando um FACS
NOTA: Embora a classificação de células possa ser realizada em uma ampla variedade de classificadores de células, aqui, o procedimento para usar os instrumentos BD FACSAria Fusion ou BD FACSAria III é descrito. É altamente recomendável que a calibração e a configuração do classificador de células sejam realizadas sob supervisão ou por um usuário experiente do instrumento.- Calibração do instrumento FACS: Consulte o protocolo 1 passo 4.1.
- Configuração do instrumento FACS para classificação de células:
- Iniciar a aquisição das células não coradas. Estes são usados para definir os espalhamentos para frente e para os lados e a tensão do detector para cada fluoróforo. Defina os parâmetros para que o sinal fluorescente de cada fluoróforo caia dentro da primeira década da escala logarítmica no gráfico de pontos.
- Adquira controles de cor única para configurar compensações manualmente (a mediana das populações positivas e negativas deve ser alinhada) ou use o software de cálculo automático (medições de inclinação). Certifique-se de que os controles de compensação correspondam às configurações experimentais de fluorocromos e detectores. Registre 10.000 eventos para células e 5.000 eventos para contas.
- Use o tubo de amostra (ou seja, células multicoloridas) para definir populações celulares de interesse usando a estratégia de gating mostrada na Figura 3A. Siga os passos 4 - 6 (abaixo).
- Para identificar os três HSPCs de medula óssea de interesse (HSC, CMP e GMP), inicie o gating usando o tamanho (FSC-A) e granularidade (SSC-A) para gate em leucócitos e, em seguida, FSC-H/FSC-A para discriminar duplos.
- Baseado no SSC-A/marcador de células mortas, gate células vivas. Use Lineage/c-Kit para selecionar células que são negativas de linhagem e expressam níveis intermediários a altos de c-Kit. Através do c-Kit/Sca-1, porta sobre as HSCs da linhagem-c-Kit+ Sca-1+ (LKS+), uma das três populações de interesse.
- Dentre os progenitores mieloides (linhagem-c-Kit+Sca-1-), utilizar FcγR/CD34 para excluir megacariócitos CD34-FcγR- e progenitores eritroides (MEP), enquanto inclui CD34+ FcγR- CMP, bem como GMP CD34+FcγR+ nas populações celulares a serem classificadas.
- Certifique-se de que o fluxo e a deflexão estejam estáveis.
- Na câmera de fluxo lateral, ligue a classificação de teste, a tensão ON e confirme a classificação precisa de gotas em um tubo de 1,5 mL instalado no lado esquerdo.
- Na janela Layout de classificação , selecione a(s) população(s) de interesse (ou seja, "LKS+" e "progenitores mieloides CD34+ " mostrados neste exemplo). Em Dispositivo, selecione 2 Tubo. Em Precisão, selecione Pureza. Em Eventos de destino, selecione Contínuo para classificar entre 160.000 e 200.000 progenitores mieloides LKS+ e CD34+ .
- Adicionar 500 μL de tampão FACS à suspensão celular e transferir o 1 mL da amostra filtrando para um novo tubo FACS com tampa de filtro de 35 μm de células para garantir que todas as células estejam em uma única suspensão imediatamente antes da aquisição. Isso elimina aglomerados celulares que poderiam entupir o instrumento.
- Depois de pronto, clique em Classificar e OK para iniciar a classificação. Ajuste a Taxa de Fluxo para manter a velocidade abaixo de 10.000 eventos por segundo.
NOTA: A proporção esperada de progenitores mieloides LKS+ para CD34+ é de 1:3 para uma ratinha adulta (8-12 semanas de idade) C57BL/6J fêmea em estado estacionário. Os números de células classificados direcionados geralmente são alcançados dentro de 30 minutos após a classificação.
- Controle de qualidade e contagem de células classificadas
NOTA: Esta etapa deve ser feita apenas durante o experimento piloto para otimização das etapas de preparação da amostra, com o objetivo de testar a pureza das células classificadas que serão usadas para o isolamento de núcleos. Uma vez que o protocolo esteja totalmente otimizado, não é aconselhável realizar esta etapa de controle de qualidade nos experimentos de acompanhamento para evitar desperdício desnecessário de material de partida que pode estar disponível em baixo número para isolamento de núcleos.- Controle de pureza por citometria de fluxo
- Transferir 10 μL das células selecionadas para um novo tubo FACS contendo 90 μL de tampão FACS.
- Adquira e registre dados pós-classificação para verificar a pureza e viabilidade da classificação. Certifique-se de que pelo menos 95% das células apareçam no portão de interesse, conforme definido em 3 - 6 e ilustrado na Figura 3B.
- Controle de pureza por citometria de fluxo
- Isolamento de núcleos de HSPCs de medula óssea triados
- Utilizar o protocolo "Low Cell Input Nuclei Isolation" do Apêndice do 10X Genomics Demonstrated Protocol - Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 - Rev C)27, com as seguintes modificações realizadas para otimizar a recuperação de núcleos:
- Tempo de lise: Executar um experimento piloto para este protocolo para identificar o melhor tempo de lise para isolamento de núcleos. Garantir uma lise celular completa, mantendo os núcleos intactos.
NOTA: O passo f do protocolo 10X Genomics27 acima mencionado instrui a "incubar [em Lysis Buffer] por 3-5 min no gelo". Durante o experimento piloto, teste pelo menos por 3 min, 4 min e 5 min e avalie a quantidade de núcleos recuperados por contagem e qualidade por citometria de fluxo e microscopia para escolher a duração ideal da lise (veja a descrição dessas verificações de controle de qualidade abaixo). Para poupar reagentes, substitua o tampão de núcleo diluído por PBS 0,04% BSA no experimento piloto. Para as HSPCs de medula óssea, 3 min foi identificado como a duração ideal da lise. - Centrifugações celulares: Para todas as centrifugações em suspensão celular, centrifugar a 300 x g durante 7 min (em vez dos 5 min em CG000365 - Rev C)27 a 4 °C.
- Centrifugações de núcleos: Realizar centrifugações de suspensão de todos os núcleos a 500 x g por 5 min conforme CG000365 - Rev C27.
- Coleta de núcleos: Na etapa b, após ressuspender em 50 μL de PBS 0,04% BSA e transferir para um tubo de 0,2 mL, adicionar 50 μL de PBS 0,04% BSA ao tubo original e pipeta-mistura para coletar as células remanescentes. Transfira para o tubo de 0,2 mL para atingir um volume total de 100 μL.
- Doravante, o volume total será de 100 μL em vez dos 50 μL do protocolo. Ajuste as etapas a jusante de acordo (por exemplo, para a etapa d, remova 90 μL em vez de 45 μL; para a etapa e, adicione 90 μL de tampão de lise em vez de 45 μL).
- Para a etapa m, ressuspenda a pelota de núcleos em 12 μL de tampão de núcleo diluído em vez de 7 μL.
- Conte os núcleos isolados. Em um tubo vazio de 0,5 mL, adicionar 10 μL de azul de tripano a 0,4% e 8 μL de PBS BSA 0,04%.
- Adicionar 2 μL de núcleos ao tubo e contar os núcleos conforme descrito no ponto 1.3.8. Use um contador de células automatizado seguindo as recomendações do fornecedor.
- Tempo de lise: Executar um experimento piloto para este protocolo para identificar o melhor tempo de lise para isolamento de núcleos. Garantir uma lise celular completa, mantendo os núcleos intactos.
- Utilizar o protocolo "Low Cell Input Nuclei Isolation" do Apêndice do 10X Genomics Demonstrated Protocol - Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 - Rev C)27, com as seguintes modificações realizadas para otimizar a recuperação de núcleos:
- Controle de pureza por citometria de fluxo
NOTA: Esta etapa deve ser feita apenas durante o experimento piloto para otimização das etapas de preparação da amostra para testar a pureza dos núcleos que serão carregados no chip Chromium 10X. Uma vez que o protocolo esteja totalmente otimizado, não é aconselhável realizar esta etapa de controle de qualidade nos experimentos de acompanhamento para evitar desperdício desnecessário de núcleos coletados que podem estar disponíveis em baixo número.- Após completar o isolamento dos núcleos, transfira 6 μL de ressuspensão dos núcleos em um novo tubo FACS pré-preenchido com 150 μL de tampão FACS. Adicionar 3 μL de 7-AAD e incubar por 5 min no gelo.
- Adquira e registre dados pós-classificação para verificar a pureza e viabilidade da classificação. Certifique-se de que pelo menos 95% dos núcleos apareçam no portão de interesse, conforme definido na etapa 4.2 do Protocolo 1 (ver Figura 4).
- Controle de qualidade de núcleos purificados por microscopia:
NOTA: Esta etapa deve ser feita apenas durante o experimento piloto para otimização das etapas de preparação da amostra para testar a qualidade dos núcleos que serão carregados no chip Chromium 10X. Uma vez que o protocolo esteja totalmente otimizado, não é aconselhável realizar esta etapa de controle de qualidade nos experimentos de acompanhamento para evitar desperdício desnecessário de núcleos coletados que podem estar disponíveis em baixo número.- Proceda como descrito na etapa 1.5.3.
- Realizar ensaio multioma
- Prossiga imediatamente para o Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide (CG000338 - Rev F)25.
Representative Results
Os dois protocolos descritos acima detalham o isolamento de núcleos a partir de dois tipos diferentes de tecido. As diferenças e semelhanças entre os dois protocolos estão esquematicamente representadas na Figura 1.
Purificação de núcleos de cérebro de camundongos
No protocolo aqui descrito, propomos um método suave para a preparação de núcleos a partir de amostras cerebrais. Inicia-se com uma dissociação mecânica do tecido cerebral em um tampão de lise, seguida por etapas de lavagem e filtragem do filtro que removem o tecido restante da suspensão. A remoção subsequente de detritos, células não lisadas e pequenas partículas é conseguida pelo FACS para garantir que apenas núcleos purificados sejam carregados para o protocolo Multiome a jusante. A Figura 2 mostra o perfil dos núcleos antes e após a triagem. Após a filtragem e antes da triagem dos núcleos, a amostra contém uma alta quantidade de debris, com mais de 99% dos "singletes" positivos para coloração nuclear (7-AAD), indicando lise celular ótima (Figura 2A). Os núcleos são classificados com base no portão positivo de 7-AAD. Uma fração de material triado é adquirida para verificar a pureza dos núcleos preparados. A Figura 2B mostra o perfil dos núcleos cerebrais após a triagem. A triagem dos núcleos permitiu um aumento na pureza dos núcleos dos 36% iniciais (Figura 2A) para quase 100% (Figura 2B).
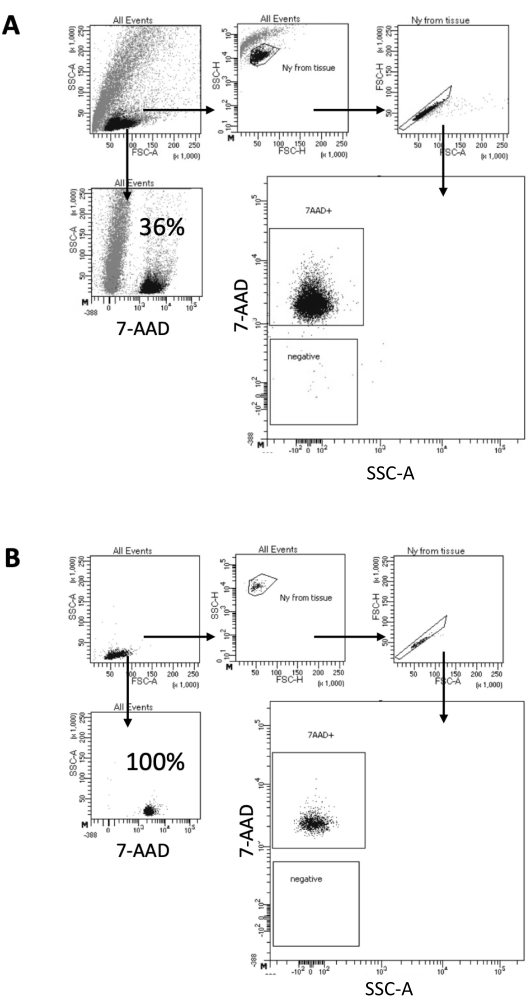
Figura 2: Estratégia de agrupamento para classificação de núcleos e teste de pureza após a triagem. Os núcleos foram corados com 7-AAD e adquiridos pelo classificador celular. (A) Os núcleos são primeiramente fechados com base em seu tamanho e granularidade (FSC-A e SSC-A, respectivamente). Partículas individuais são então selecionadas com base em suas propriedades FSC-A/FSC-H e coloração 7-AAD. (B) Após a triagem celular, uma fração dos núcleos do tubo de coleta é testada quanto à pureza usando a mesma estratégia de fechamento que em A. Clique aqui para ver uma versão maior desta figura.
Purificação de progenitores de células-tronco hematopoéticas (HSPCs) de medula óssea de camundongos
Após o isolamento da medula óssea, até 2 x 105 HSPCs são classificados por FACS, de acordo com a estratégia de gating mostrada na Figura 3A. A eficácia da triagem e a pureza da amostra são avaliadas (Figura 3B).
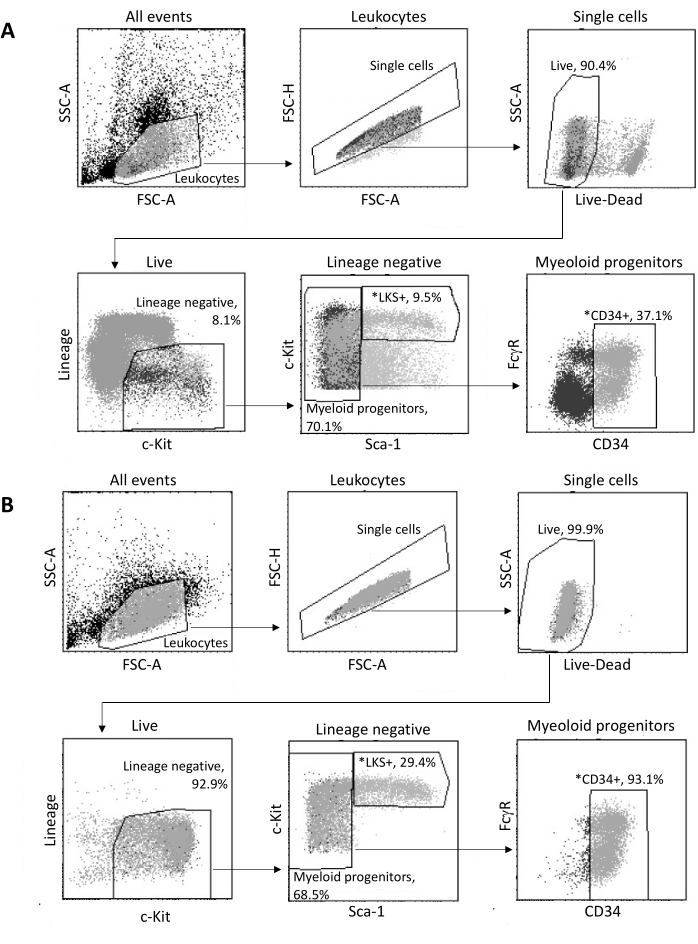
Figura 3: Estratégia de gating para triagem de HSPCs de medula óssea. (A) Uma estratégia representativa de agrupamento FACS para a triagem de células-tronco hematopoéticas LKS+ viáveis e progenitores mieloides CD34+ para isolamento de núcleos. (B) Parcelas FACS representativas utilizadas para a verificação da pureza da população celular classificada. São mostradas as proporções de diferentes subconjuntos celulares em relação à população parental. *As duas populações classificadas. Clique aqui para ver uma versão maior desta figura.
O protocolo "Low Cell Input Nuclei Isolation" permite o isolamento de núcleos de amostras com um máximo de 10 a5 células. Inclui um baixo número de etapas de centrifugação, minimizando assim a perda de células/núcleos. Ajustamos o volume de tampões de lise e lavagem proporcionalmente à entrada celular e aumentamos o tempo de centrifugação para máxima recuperação dos núcleos. Realizamos um experimento piloto para avaliar a quantidade de núcleos recuperados por contagem e sua qualidade por citometria de fluxo e microscopia. A Figura 4 mostra a amostra de HSPCs após lise celular. Esse protocolo gerou núcleos de alta qualidade, como observado na Figura 5A, sem detritos que pudessem impactar o protocolo de multioma a jusante.
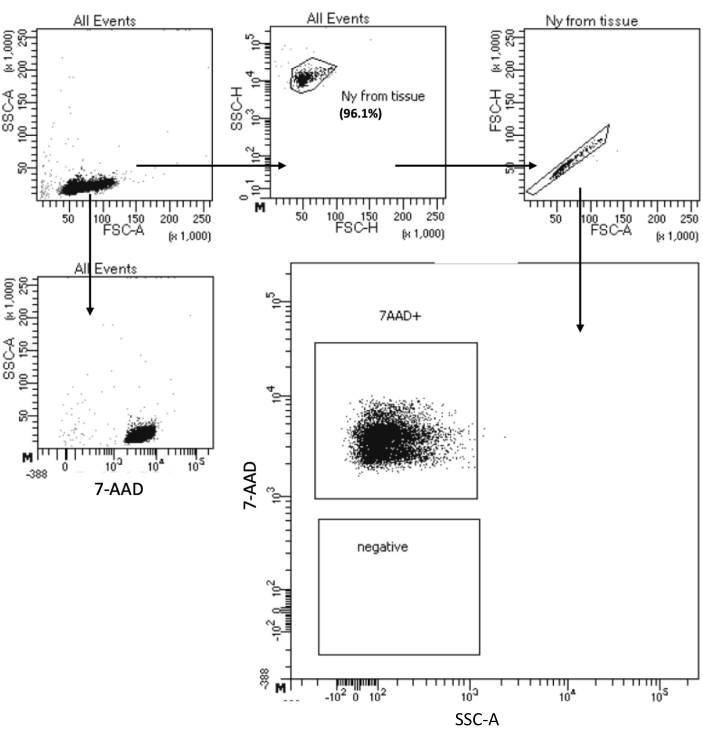
Figura 4: Classificação do teste de pureza dos núcleos isolados de HSPC da medula óssea. Os núcleos foram corados com 7-AAD e adquiridos pelo classificador celular. Os núcleos foram primeiramente fechados com base em seu tamanho e granularidade (FSC-A e SSC-A, respectivamente) para avaliar a pureza da amostra. A proporção de núcleos é indicada em relação à população parental. Clique aqui para ver uma versão maior desta figura.
| Número do tubo | Nome do tubo | Entidade manchada | Quantidade de entidade manchada | Anticorpo/corante (μL) | Buffer de coleta (μL) |
| 1 | Sem manchas | Células | 5,00,000 | N/A | 200 |
| 2 | LIVE/DEAD Mancha Aqua Dead Cell | Células | 5,00,000 | 0.5 | |
| 3 | APC/Cyanine 7 anti-camundongo CD16/32 (FcγR) | OneComp eBeads | 15 μL | 1 | |
| 4 | Coquetel de linhagem anti-rato azul do Pacífico | OneComp eBeads | 15 μL | 1 | |
| 5 | PE anti-mouse Ly-6A/E (Sca-1) | OneComp eBeads | 15 μL | 1 | |
| 6 | APC anti-mouse CD117 (c-Kit) | OneComp eBeads | 15 μL | 1 | |
| 7 | FITC anti-mouse CD34 | OneComp eBeads | 15 μL | 1 |
Tabela 1: Controles de coloração única para ajustes de compensação no citômetro de fluxo. São indicados os controles de coloração única necessários, o número de células ou contas a serem coradas e as quantidades de anticorpos.
| Master Mix | Reagente | Diluição final | Anticorpo/corante (μL) | Tipo de buffer | Tampão (μL) |
| Mistura 1 | APC/Cyanine 7 anti-camundongo CD16/32 (FcγR) | 1/500 | 1.2 | DPBS | 300 |
| LIVE/DEAD Mancha Aqua Dead Cell | 1/250 | 2.4 | |||
| Mistura 2 | Coquetel de linhagem anti-rato azul do Pacífico | 1/20 | 30 | Buffer FACS | 300 |
| PE anti-mouse Ly-6A/E (Sca-1) | 1/200 | 3 | |||
| APC anti-mouse CD117 (c-Kit) | 1/200 | 3 | |||
| FITC anti-mouse CD34 | 1/50 | 12 | |||
| Volume total de coloração | 600 |
Tabela 2: Composição da mistura de coloração para HSPCs de medula óssea. São mostrados os volumes de reagentes necessários para a coloração de uma amostra contendo 40 milhões de células. Para colorir um número maior de amostras, multiplique o volume indicado pelo número necessário de amostras e adicione metade de um volume de amostra extra para garantir um volume suficiente da mistura principal.
Figura suplementar 1: Protocolo de isolamento de células da medula óssea. (A) Abra o peritônio. Linhas pontilhadas brancas indicam a linha a ser cortada. (B) Depois de descascar a pele da pata traseira, forre a tesoura ao longo da coluna vertebral na articulação do quadril para cortar a perna sem cortar o fêmur. (C) A aparência da perna separada do corpo antes da remoção do músculo. (D) O aparecimento da perna após a remoção do músculo. (E) O procedimento para separar o fêmur na articulação do joelho, depois na articulação do quadril, tomando cuidado para não abrir o fêmur. As setas curvas brancas mostram o movimento necessário. (F) Procedimento para abrir a parte distal do fêmur (ou seja, a parte previamente presa à tíbia na articulação do joelho) segurando firmemente a cartilagem e a epífise distal com tesoura e virando-a para trás para expor a medula óssea. (G) Quatro saliências, indicadas por setas pretas, devem ser visíveis na extremidade exposta da fise. (H) A aparência de um fêmur com a extremidade aberta voltada para baixo no tubo preparado de 0,5 mL colocado dentro de um tubo de 1,5 mL contendo 150 μL de tampão FACS. (I) O aparecimento das células da medula óssea peletizadas e do fêmur agora branco após rápida centrifugação a 12.000 x g. Clique aqui para baixar este arquivo.
Discussion
A preparação de suspensão de células ou núcleos de alta qualidade é de importância crucial para o sucesso de análises multiômicas de RNA-Seq de célula única ou de núcleo único 29,30,31. Aqui, descrevemos protocolos de preparo de amostras e isolamento de núcleos para ensaios de multioma de dois tipos de tecido: cérebro e medula óssea.
O protocolo cerebral descrito neste trabalho permite a recuperação de núcleos de alta qualidade a partir de tecido cerebral fresco-congelado. Inclui as seguintes etapas: ruptura do tecido congelado, isolamento dos núcleos, purificação dos núcleos e controle de qualidade do material preparado. O tecido cerebral é composto por diversos tipos celulares, e o procedimento de dissociação tecidual e isolamento de núcleos deve preservar as proporções das populações celulares presentes no tecido inicial. Aqui, a composição do tampão de lise e o tempo de incubação foram otimizados para permitir a lise completa e suave de todas as populações celulares que compõem o tecido.
O protocolo de HSPCs de medula óssea é um pouco diferente, pois requer uma etapa adicional no início do experimento para isolar a população celular de interesse de uma suspensão celular heterogênea. Após a coleta do tecido fresco, as hemácias são lisadas e a amostra é enriquecida para o subconjunto celular de interesse. As células-alvo são lisadas, os núcleos são isolados e a qualidade do material preparado é controlada.
A 10X Genomics fornece vários protocolos validados para isolamento de núcleos em diversos tecidos 32,33. A empresa também comercializa um kit de isolamento de núcleos com uma tubulação simples para isolar núcleos de tecidos validados34. No entanto, esses protocolos necessitam de otimização adicional para adequar as particularidades de determinadas amostras. Um exemplo são as amostras que exigem trabalhar com baixa entrada de células. Para essas amostras, as etapas mais desafiadoras são as centrifugações que precisam ser suficientemente rigorosas para limpar a amostra e suaves o suficiente para evitar a perda de células/núcleos. Com o protocolo aqui descrito, adaptamos o 10X Genomics Demonstrated Protocol - Nuclei Isolation for Single Cell Multiome ATAC + GEX Sequencing (CG000365 - Rev C)27 para encontrar um equilíbrio fino entre esses dois requisitos. Como demonstrado no exemplo da preparação de núcleos a partir de HSPCs triados, melhoramos a recuperação de núcleos sem impacto na qualidade da amostra.
Um desafio adicional é a etapa de lise das células purificadas para isolamento dos núcleos. Condições de lise mais severas e tempos de incubação mais longos podem danificar os núcleos e, assim, afetar a qualidade dos dados de sequenciamento. A Figura 5 mostra imagens representativas de núcleos de amostras de medula óssea em diferentes tempos de incubação com tampão de lise e ilustra como o estado dos núcleos pode ser diferente dependendo da lise celular. No exemplo dos HSPCs, identificamos a lise de 3 min como a condição que resulta na maior proporção de núcleos intactos com aparência saudável e a menor proporção de núcleos danificados. Os tempos de incubação da lise devem ser otimizados para cada novo tipo de amostra.
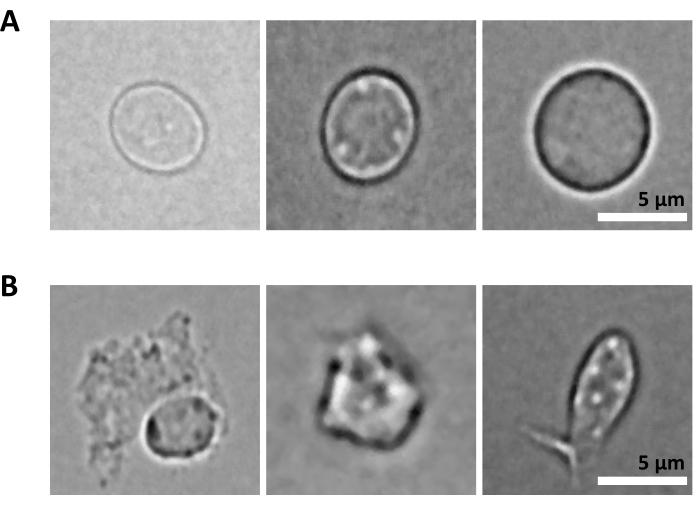
Figura 5: Controle de qualidade dos núcleos por microscopia. São mostradas imagens representativas de campo brilhante de núcleos isolados da medula óssea de camundongos com (A) núcleos intactos e (B) danificados. Barra de escala 5 μm. As imagens foram obtidas com microscópio invertido, utilizando-se objetiva ELWD NA, 0,60 e zoom digital de 1,5x. Clique aqui para ver uma versão maior desta figura.
Ambos os protocolos detalhados neste trabalho baseiam-se na purificação de células-alvo ou núcleos por instrumentos FACS de alto rendimento. Esta etapa é de importância crucial para protocolos de preparação de células únicas/núcleos onde subconjuntos raros de células devem ser isolados de suspensões heterogêneas. Nestes, como no exemplo mostrado aqui para a classificação de HSPCs, um painel de citometria de fluxo de alta dimensão pode ser necessário para permitir o "gating" na população celular de interesse. A classificação é extremamente rápida e precisa, levando a mais de 95% de pureza dos subconjuntos de células classificadas. Essa abordagem expõe a suspensão celular a uma pressão de até 70 psi e, portanto, pode ser limitante para a triagem de células frágeis (por exemplo, células dendríticas, neutrófilos), uma vez que pode causar a ruptura de sua membrana celular. Nesses casos, soluções alternativas devem ser selecionadas para purificação celular, incluindo classificação magnética, aplicação de instrumentos de nova geração (por exemplo, CellenOne, Cellenion; MACSQuant Tyto, Miltenyi)35,36, ou sistemas baseados em gotículas (por exemplo, ODIN, Sensific)37. No entanto, a lenta velocidade de classificação dessas tecnologias, com a triagem celular que dura horas em vez de minutos, é um forte fator limitante para a aplicação dessas abordagens na preparação de células viáveis para o Multiome e outras aplicações unicelulares baseadas na análise de grandes números de células.
Para a purificação de núcleos isolados do tecido, o FACS é o método de escolha devido ao seu rendimento e à pureza do material isolado. Os núcleos não são sensíveis à pressão, e os isolados de tecido filtrado podem ser facilmente purificados através do classificador celular. Se o laboratório não estiver equipado com um instrumento FACS, existem outras alternativas, um pouco menos eficientes, mas suficientemente boas. Exemplos incluem ultracentrifugação ou o uso de pequenos equipamentos como o MARS (Applied Cell) que separa partículas com base em sua diferença de tamanho, usando ondas acústicas; Lavadora laminar CURIOX que utiliza propriedades hidrofóbicas de suspensões de células/núcleos; ou LEVITAS bio que se baseia em propriedades físicas das células (levitação) para separá-las dos detritos.
Aqui, descrevemos protocolos para obtenção de um alto número de núcleos e a melhor pureza para o protocolo Multiome a jusante. A classificação FACS e as etapas repetidas de centrifugação resultam em uma perda substancial do material inicial. Por esta razão, no protocolo de preparação de núcleos do cérebro que descrevemos aqui requer material de partida suficientemente abundante para resultar na coleta de pelo menos 500.000 núcleos após a triagem FACS. Protocolos alternativos devem ser aplicados se esse critério não puder ser correspondido. Ao trabalhar com populações celulares raras ou pequenos cortes de tecido, a quantidade disponível de material inicial pode ser um fator limitante. Para resolver essa questão, é possível melhorar a recuperação de núcleos (a) reduzindo o volume de lise, (b) reduzindo o volume de lavagem, (c) usando uma única lavagem com tempo de centrifugação prolongado para tentar melhorar a recuperação, conforme indicado nos protocolos de Genômica 10X para isolamento de núcleos de baixa entrada celular. Para análise multiômica de material de baixo conteúdo, vale a pena considerar aplicações baseadas em placas, como scNMT, SNARE-seq e Paired-seq38 , que exigem muito menos amostras de entrada.
Em resumo, descrevemos dois protocolos robustos para a preparação de núcleos do cérebro e da medula óssea HSPCs para análise do Multiome a jusante. Esses protocolos são aplicáveis em qualquer projeto científico que exija suspensões de núcleo único de alta qualidade desses dois tipos de tecido, independentemente da questão científica colocada. Nosso grupo vem aplicando o protocolo de isolamento de núcleos cerebrais em estudos do desenvolvimento cerebral após a inativação de vários genes-alvo e em estudos de resposta imune no contexto de doenças neurológicas. Estamos utilizando o protocolo de isolamento de núcleos da medula óssea para decifrar a participação de várias subpopulações hematopoéticas no estabelecimento do sistema imune.
Disclosures
Os autores não têm nada a revelar.
Acknowledgements
Ana Jeemin Choi foi apoiada por uma bolsa do Programa de Doutorado Internacional Pasteur - Universidade de Paris (PPU).
Materials
| Name | Company | Catalog Number | Comments |
| 18 G x 1 ½ (1.2 mm x 38 mm) Agani needles | Terumo | AN*1838S1 | |
| 15 mL tubes | Falcon | 352097 | |
| 5 mL round bottom FACS tube with cell strainer cap 35 µm | falcon | 352235 | |
| 50 mL tubes | Falcon | 352070 | |
| 7-AAD | BD pharmagen | 559925 | |
| ACK Lysing Buffer | Gibco | A10492-01 | |
| APC anti-mouse CD117 (c-Kit) | BioLegend | 105812 | Clone: 2B8 |
| APC/Cyanine 7 anti-mouse CD16/32 (FcγR) | BioLegend | 101328 | Clone: 93 |
| BD FACSAria III | BD Biosciences | non-applicable | |
| BD FACSDiva Software v8.0.1 | BD Biosciences | non-applicable | |
| Bovine Serum Albumin stock solution 10% | Miltenyi Biotec | 130-091-376 | |
| Cell staining buffer | Biolegend | 420201 | |
| CFI Suprplan Fluor ELWD 40XC ON 0.6 | Nikon | non-applicable | |
| CMOS camera Prime 95B 25 mm | Photometrix | non-applicable | |
| Countess II FL Automated Cell Counter | Invitrogen | AMQAF1000 | |
| Countess cell counting chamber slide | Invitrogen | C10283 | |
| Coverglass 24 mm x 24 mm 0.13-0.17 mm | Brand | BR470819 | |
| Digitonine 5% | Invitrogen | BN2006 | |
| Disposable Scalpels | Swann-Morton | 0508 | |
| DMEM (1x) + GlutaMAX-I | Gibco | 31966-021 | |
| DPBS (10x) | Gibco | 14200-067 | |
| DTT | Sigma aldrich | 646563 | |
| Epifluorescence inverted microscope Nikon Ti2 -E | Nikon | non-applicable | |
| Eppendorf Safe-Lock Tubes 0.5 mL | Eppendorf | 30123603 | |
| Ethanol 70% | VWR | 83801.290 | |
| FITC anti-mouse CD34 | Invitrogen | 11-0341-85 | Clone: RAM34 |
| Forceps for dissection | FST | 11152-10 | |
| Heat-inactivated Fetal Bovine Serum (FBS) | Gibco | 11533387 | |
| Dounce Homogeniser 2 mL | Bellco glass | 1984-10002 | Pestle “A” Large Clearance: .0030-.0050″ and Pestle “B” Small Clearance: .0005-.0025″ |
| LIVE/DEAD fixable aqua dead cell stain kit | Invitrogen | L34957 | |
| Magnesium chloride solution 1 M | Sigma aldrich | M1028 | |
| Microcentrifuge | Eppendorf | 5424R | |
| Mounting medium Fluoromount-G | invitrogen | 00-4958-02 | |
| Nonidet P40 substitute | Sigma aldrich | 74385 | |
| Nuclease free water | ThermoFischer | AM9932 | |
| Nuclei buffer 20x | 10X Genomics | 2000153/2000207 | |
| Nuclei isolation kit EZ prep | Sigma Aldrich | NUC-101 | |
| OneComp eBeads compensation beads | Invitrogen | 01-1111-41 | |
| Pacific Blue anti-mouse lineage cocktail (including anti-mouse CD3, Ly-6G/Ly-6C, CD11b, CD45R/B220, TER-119) | BioLegend | 133310 | Clones (in the same order as the antibodies listed): 17A2, RB6-8C5, M1/70, RA3-6B2, Ter-119 |
| PCR Tube Strips 0.2 mL | Eppendorf | 951010022 | |
| PE anti-mouse Ly-6A/E (Sca-1) | BioLegend | 122507 | Clone: E13-161.7 |
| Petri dish 100 mm x 20 mm OPTILUX | Falcon | 353003 | |
| Ply-L-lysine 0.01% sterile-filtered suitable for cell culture | Sigma | P4707 | |
| Printed microscope slides 8 well 6 mm numbered | Epredia | ER-301B-CE24 | |
| Protein LoBind Tubes 1.5 mL | Eppendorf | 30108116 | |
| Recombinant Rnase inhibitor 5000 U | Takara | 2313A | |
| Scissors for dissection | FST | 14090-09 | |
| Sodium chloride solution 5 M | Sigma aldrich | 59222C | |
| Syringe filters, PES, 0.2 µm | Fisher Scientific | 15206869 | |
| Transparent nail polish | any | non-applicable | |
| Trizma Hydrochloride solution pH 7.4 | Sigma aldrich | T2194 | |
| Trypan Blue 0.4% | gibco | 15250061 | |
| Tween 20 | Biorad | 1662404 | |
| UltraPure Distilated Water Dnase/Rnase Free | Invitrogen | 10977-035 |
References
- Clark, S. J., et al. scNMT-seq enables joint profiling of chromatin accessibility DNA methylation and transcription in single cells. Nature Communications. 9 (1), 781 (2018).
- Lee, J., Hyeon, D. Y., Hwang, D. Single-cell multiomics: technologies and data analysis methods. Experimental & Molecular Medicine. 52 (9), 1428-1442 (2020).
- Cerrizuela, S., et al. High-throughput scNMT protocol for multiomics profiling of single cells from mouse brain and pancreatic organoids. STAR Protocols. 3 (3), 101555 (2022).
- Dimitriu, M. A., Lazar-Contes, I., Roszkowski, M., Mansuy, I. M. Single-cell multiomics techniques: From conception to applications. Frontiers in Cell and Developmental Biology. 10, 854317 (2022).
- Stoeckius, M., et al. Simultaneous epitope and transcriptome measurement in single cells. Nature Methods. 14 (9), 865-868 (2017).
- Cao, J., et al. . Joint profiling of chromatin accessibility and gene expression in thousands of single cells. 361 (6409), 1380-1385 (2018).
- Narayanan, A., et al. Nuclei Isolation from Fresh Frozen Brain Tumors for Single-Nucleus RNA-seq and ATAC-seq.Journal of Visualized Experiments. JoVE. 162, 61542 (2020).
- Kim, M., et al. Single-nucleus transcriptomics reveals functional compartmentalization in syncytial skeletal muscle cells. Nature Communications. 11 (1), 6375 (2020).
- Santos, M. D., et al. Extraction and sequencing of single nuclei from murine skeletal muscles. STAR Protocols. 2 (3), 100694 (2021).
- Safabakhsh, S., et al. Isolating nuclei from frozen human heart tissue for single-nucleus RNA sequencing. Current Protocols. 2 (7), (2022).
- Lau, S. -. F., Cao, H., Fu, A. K. Y., Ip, N. Y. Single-nucleus transcriptome analysis reveals dysregulation of angiogenic endothelial cells and neuroprotective glia in Alzheimer's disease. Proceedings of the National Academy of Sciences of the United States of America. 117 (41), 25800-25809 (2020).
- Armand, E. J., Li, J., Xie, F., Luo, C., Mukamel, E. A. Single-cell sequencing of brain cell transcriptomes and epigenomes. Neuron. 109 (1), 11-26 (2021).
- Morabito, S., et al. Single-nucleus chromatin accessibility and transcriptomic characterization of Alzheimer's disease. Nature Genetics. 53 (8), 1143-1155 (2021).
- Chen, S., et al. Spatially resolved transcriptomics reveals genes associated with the vulnerability of middle temporal gyrus in Alzheimer's disease. Acta Neuropathologica Communications. 10 (1), (2022).
- Paul, F., et al. Transcriptional heterogeneity and lineage commitment in myeloid progenitors. Cell. 163 (7), 1663-1677 (2015).
- Kaufmann, E., et al. BCG educates hematopoietic stem cells to generate protective innate immunity against tuberculosis. Cell. 172 (1-2), 176-190 (2018).
- Christ, A., et al. diet triggers NLRP3-dependent innate immune reprogramming. Cell. 172 (1-2), 162-175 (2018).
- Moorlag, S. J. C. F. M., et al. β-Glucan Induces protective trained immunity against mycobacterium tuberculosis infection: A key role for IL-1. Cell Reports. 31 (7), 107634 (2020).
- de Laval, B., et al. C/EBPβ-dependent epigenetic memory induces trained immunity in hematopoietic stem cells. Cell Stem Cell. 26 (5), 657-674 (2020).
- Renthal, W., et al. Characterization of human mosaic Rett syndrome brain tissue by single-nucleus RNA sequencing. Nature Neuroscience. 21 (12), 1670-1679 (2018).
- Yang, A. C., et al. A human brain vascular atlas reveals diverse mediators of Alzheimer's risk. Nature. 603 (7903), 885-892 (2022).
- Lee, D. R., Zhang, Y., Rhodes, C. T., Petros, T. J. Generation of single-cell and single-nuclei suspensions from embryonic and adult mouse brains. STAR Protocols. 4 (1), 101944 (2022).
- Corces, M. R., et al. Lineage-specific and single-cell chromatin accessibility charts human hematopoiesis and leukemia evolution. Nature Genetics. 48 (10), 1193-1203 (2016).
- Ranzoni, A. M., et al. Integrative single-cell RNA-seq and ATAC-seq analysis of human developmental hematopoiesis. Cell Stem Cell. 28 (3), 472-487 (2021).
- . 10X Genomics Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide. Document Number CG000338 Rev F. At. , (2022).
- J, , et al. Microdissection of mouse brain into functionally and anatomically different regions. Journal of Visualized Experiments: JoVE. 168, 61941 (2021).
- . . 10X Genomics 10X Genomics Demonstrated Protocol - Nuclei Isolation for Single Cell Multiome ATAC + GEX sequencing (CG000365 - Rev C). At. , (2022).
- Haag, S., Murthy, A. Murine monocyte and macrophage culture. Bio-Protocol. 11 (6), (2021).
- Haque, A., Engel, J., Teichmann, S. A., Lönnberg, T. A practical guide to single-cell RNA-sequencing for biomedical research and clinical applications. Genome Medicine. 9 (1), (2017).
- Jiang, P. Quality control of single-cell RNA-seq. Methods in Molecular Biology. , 1-9 (1935).
- Regan, C., Preall, J. Practical considerations for single-cell genomics. Current Protocols. 2 (8), (2022).
- . . 10X Genomics 10X Genomics Demonstrated Protocol - Nuclei Isolation for Single Cell ATAC Sequencing (CG000169 - Rev E). At. , (2022).
- . 10X Genomics 10X Genomics Demonstrated Protocol - Nuclei Isolation from Complex Tissues for Single Cell Multiome. ATAC + Gene Expression Sequencing. (CG000375 - Rev C). At. , (2022).
- . . 10X Genomics 10X Genomics - Chromium Nuclei Isolation Kit (CG000505 - Rev A). AT. , (2022).
- Shomroni, O., et al. A novel single-cell RNA-sequencing approach and its applicability connecting genotype to phenotype in ageing disease. Scientific Reports. 12 (1), 4091 (2022).
- Ocañas, S. R., Pham, K. D., Blankenship, H. E., Machalinski, A. H., Chucair-Elliott, A. J., Freeman, W. M. Minimizing the ex vivo confounds of cell-isolation techniques on transcriptomic and translatomic profiles of purified microglia. 9 (2), 0348-0321 (2022).
- Gérard, A., et al. High-throughput single-cell activity-based screening and sequencing of antibodies using droplet microfluidics. Nature Biotechnology. 38 (6), 715-721 (2020).
- Vandereyken, K., Sifrim, A., Thienpont, B., Voet, T. Methods and applications for single-cell and spatial multi-omics. Nature Reviews. Genetics. 24, 494-515 (2023).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved